文章信息
- 迟艳红, 王玮, 王文章, 黄有松. 2018.
- CHI Yan-hong, WANG Wei, WANG Wen-zhang, HUANG You-song. 2018.
- 实验室海月水母大规模解体的初步研究
- Preliminary studies on large-scale moon jellyfish Aurelia aurita decomposition under laboratory conditions
- 海洋科学, 42(11): 43-50
- Marine Sciences, 42(11): 43-50.
- http://dx.doi.org/10.11759/hykx20180606002
-
文章历史
- 收稿日期:2018-06-06
- 修回日期:2018-10-28
2. 青岛农业大学, 山东 青岛 266109
2. Qingdao Agricultural University, Qingdao 266109, China
海月水母(Aurelia aurita Linnaeus, 1758)隶属于刺胞动物门(Cnidaria)钵水母纲(Scyphozoa)旗口水母目(Semaeostomeae)洋须水母科(Uimaridae)海月水母属(Aurelia), 广泛分布于70°N, 40°S海区水域中[1]。在我国青岛、烟台、威海、大连等沿海均有分布, 是我国海域大型水母优势种之一[2]。近几十年来, 水母暴发事件在世界海域频繁发生[3-5], 这严重威胁了海洋生态系统, 并对海洋渔业、滨海旅游业、沿海工业等造成了严重危害, 带来了巨大的经济损失。海月水母作为大型水母暴发的主要种类之一, 广泛分布于世界各地, 并自20世纪中末期开始在全球诸多地区频繁暴发, 包括日本、印度、希腊、英国、澳大利亚等各国海域[4, 6-7]。近年来, 我国烟台、威海、青岛、秦皇岛等沿海海域也多次出现海月水母异常暴发现象[8-10], 引起了人们的关注。
虽然海月水母暴发带来了巨大的海洋生态危机, 但与此同时, 海月水母作为一种常见的水母种类, 泳姿优雅飘逸, 具有较高的观赏价值。并且随着水母饲养繁殖技术的不断成熟, 海月水母逐渐实现大规模的饲养繁殖, 成为各大水族馆展示以及水族爱好者的宠儿。
在海月水母人工养殖过程中, 作者发现由细菌引起的水母大规模发病解体是制约海月水母饲养繁殖顺利进行的重要因素。目前, 国内外关于海月水母病原菌的相关研究鲜见报道。本实验利用分子生物学技术对从发病海月水母中分离出的病原菌进行16S rRNA基因序列克隆和DNA测序, 并对所获得的序列进行相关生物信息学分析, 进而完成海月水母病原菌的分类鉴定。同时, 实验测定分析了海月水母病原菌的蛋白酶活性, 为进一步探讨海月水母细菌致病机理以及自然条件下水母暴发后水母消亡机制奠定了基础。
1 材料与方法 1.1 材料实验所用材料来自青岛海产博物馆实验室内繁殖饲养的海月水母。
1.2 实验方法 1.2.1 发病海月水母病灶处的菌株培养青岛海产博物馆实验室分别于2015年2月15日与2017年4月24日突发两次饲养海月水母大规模伞体烂洞现象。从实验缸中随机取3只烂洞海月水母, 从烂洞伞体处用无菌手术刀取下病灶组织, 利用灭菌后的玻璃匀浆器在冰上进行研磨匀浆, 将所获得的组织匀浆液均匀涂布在2216e培养基平板上, 放置25℃培养箱培养过夜, 至生长出密度大小适中的菌落。同时以未烂洞的正常海月水母、源水样品、发病水母缸中海水样品进行相同条件下细菌培养实验作为对照。
1.2.2 PCR扩增菌落16S rRNA基因序列观察平板上菌落生长情况, 挑取不同表型的优势菌落进行菌落PCR从而获得目的基因。使用细菌16S rRNA基因序列通用引物, 其正向引物为16S rDNA-F (5'-AGAGTTTGATC CTGGCTCAG-3')[11], 反向引物为16S rDNA-R (5'-GGTTACCTTGTTACG ACTT-3') [12], 并由生工生物工程(上海)股份有限公司合成。通过PCR扩增获得16S rRNA基因序列目的片段, PCR反应体系为25 μL︰l μL菌液模板, 1×reaction缓冲液, 1.5 mmol/L MgCl2, 0.2 mmol/L dNTP, 2 mmol/L正向引物, 2 mmol/L反向引物以及1.25 U Taq DNA聚合酶(TaKaRa), ddH2O补足到25 μL。PCR扩增条件: 94℃预变性4 min, 1个循环; 94℃变性50 s, 55℃退火50 s, 72℃延伸60 s, 35个循环; 72℃延伸10 min, 1个循环。
1.2.3 PCR产物的回收纯化, 目的基因的获得及生物信息学分析PCR产物在1%的琼脂糖凝胶中进行电泳, 使用凝胶成像系统观察照相后, 切下特异性目的条带, 并利用DNA胶回收试剂盒(上海生工公司)对目的片段进行回收, 然后克隆到pMD19-T载体(大连宝生物公司)。重组质粒转化至大肠杆菌TOP10菌株, 产生的阳性克隆经PCR验证后进行测序。所获得的目的DNA序列利用NCBI BLAST程序(http://www.ncbi. nlm.nih.gov)进行序列同源性比对以及相似性分析, 利用MEGA 4.1软件进行系统发生和进化分析, 系统树采用Neighbor-joining方法构建。
1.2.4 优势菌的发酵及蛋白酶活性测定将细菌接种到100 mL的2216e液体培养基中进行扩大培养, 于25℃摇床中200 r/min恒温振荡培养过夜。
利用福林-酚试剂法[3]进行蛋白酶活性的测定。以1 mL样品为反应测定溶液, 向试管中加入0.5%的酪素溶液2 mL, 37℃水浴准确反应15 min, 立即加入10%的三氯乙酸3 mL终止反应, 取出反应液常温离心(6000 r/min, 2 min), 去除沉淀后吸取上清液1 mL, 向其中加入0.55 mol/L Na2CO3溶液5 mL, 再加入福林-酚试剂1 mL, 于37℃恒温水浴中显色15 min, 然后在680 nm波长下比色测定, 以蒸馏水作为对照, 读取反应液OD值。
使用蛋白定量测试盒-考马斯亮蓝(南京建成)进行样品蛋白质浓度的测定。
细菌中蛋白酶活力的定义:每mg细菌蛋白每分钟分解酪蛋白生成1 µg酪氨酸相当于1个酶活力单位(U/mg)。
2 结果与分析 2.1 发病海月水母的观察及菌株培养结果分别于2015年2月和2017年4月两次出现的养殖海月水母发病状况具有相同的外部表征:多个饲养缸中的海月水母同时大规模发病, 各缸发病率为95%~100%。染病的海月水母伞体出现烂洞(图 1), 随着时间的推移, 烂洞面积越来越大, 在不到15 h的时间内, 海月水母伞体大面积消亡解体。
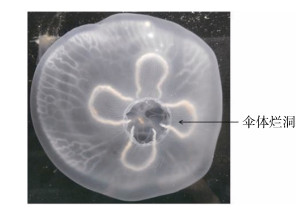 |
| 图 1 发病海月水母伞体烂洞症状 Fig. 1 Symptom of pathogenetic moon jellyfish Aurelia aurita |
分别对两次发病的海月水母烂洞处组织进行细菌培养, 结果显示: 2015年发病海月水母经过细菌培养后, 在2216e固体培养基平板上生长出大量的乳白色菌落(编号为Paa1)和淡黄色菌落(编号为Paa2)(图 2a~b)。2017年发病海月水母烂洞组织匀浆液培养后长有大量透明菌落(编号为Vaa1)和白色细菌菌落(编号为Vaa2)(图 2d~e), 同时, 其饲养水体中也存在大量透明和白色菌落(图 2f), 而未烂洞海月水母伞体组织无菌落出现, 另外, 作为对照组的海水源水样品的培养基平板中无菌落(图 2c)。
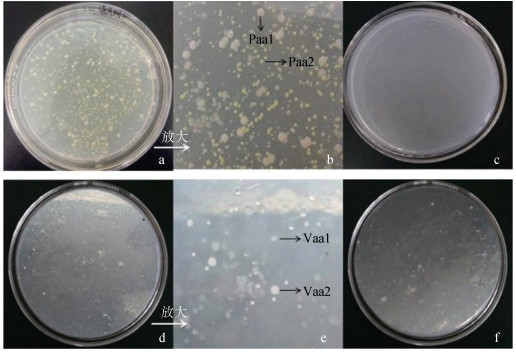 |
| 图 2 海月水母组织及水体细菌培养结果 Fig. 2 Bacterial culture of ambient water and lesions of moon jellyfish Aurelia aurita 注: a~b: 2015年海月水母发病烂洞组织细菌培养结果; c:实验室海水源水水样细菌培养结果; d~e: 2017年海月水母发病烂洞组织细菌培养结果; f: 2017年发病海月水母饲养水体水样细菌培养结果 |
分别以通过细菌培养获得的四种不同表型的优势菌株为DNA模板, 以16S rDNA-F/R引物进行目的基因PCR扩增。扩增产物经过1%的琼脂糖凝胶电泳检测, 结果显示:经过PCR扩增, 均获得了长1500 bp左右的目的产物。测序结果显示:以2015年发病海月水母中分离的2种细菌Paa1和Paa2为模板进行的PCR扩增所获得的16S rRNA基因序列目片段分别为1 498 bp和1 500 bp; 以2017年发病海月水母中分离的2种细菌Vaa1和Vaa2为模板进行的PCR扩增所获得的目的基因长度分别为1 514 bp和1 516 bp。
2.3 病原菌16S rRNA基因同源序列比对及系统发育分析将从2015年发病海月水母中分离得到的Paa1、Paa2两种细菌16S rRNA基因序列在NCBI数据库中进行Blast比对, 分析结果显示, Paa1、Paa2两种细菌均与嗜冷杆菌属中多个种的16S rRNA基因序列具有较高相似度, 且与一种嗜冷杆菌(Psychrobacter sp. DQ064630.1)的16S rRNA基因序列相似度最高, 分别高达99.86%和99.93%。基于16S rRNA基因序列, 利用MEGA4.1软件构建了嗜冷杆菌16S rRNA基因的系统发育树(图 3), 结果显示, 由于嗜冷杆菌属16S rRNA基因序列的种间差异不显著, 导致嗜冷杆菌不具有唯一的聚类特征。菌株Paa1和Paa2与多种嗜冷杆菌Psychrobacter frigidicola、Psychrobacter glacincola、Psychrobacter alimentarius、Psychrobacter adeliensis等均聚为一支, 从而无法确定到种, 因此经过本次实验仅能确定分离到的Paa1菌株和Paa2菌株均属于嗜冷杆菌属(Psychrobacter)。
 |
| 图 3 以16S rRNA基因构建的嗜冷杆菌系统发育树 Fig. 3 Phylogenetic tree of Psychrobacter based on 16S rRNA gene |
同时, 将从2017年发病海月水母中分离得到的Vaa1、Vaa2两种细菌16S rRNA基因序列进行同源性比对, 分析结果显示, Vaa1、Vaa2两种细菌均与弧菌属细菌的16S rRNA基因序列相似度较高, 其中Vaa1菌株的16S rRNA基因序列与海神弧菌(Vibrio neptunius NR_025476.1)相似度最高, 高达99.9%, Vaa2菌株的16S rRNA基因序列与灿烂弧菌(Vibrio splendidus EU091325.1)相似度最高, 高达99.7%。通过构建的弧菌16S rRNA基因系统发育树(图 4)结果显示, 菌株Vaa1与Vaa2分别类聚为两个不同的分支, 其中菌株Vaa1与海神弧菌(Vibrio neptunius)聚为一支, 菌株Vaa2与灿烂弧菌(Vibrio splendidus)聚为一支, 综合16S rRNA基因序列同源性及系统发育学分析结果, Vaa1可鉴定为海神弧菌Vibrio neptunius, Vaa2可鉴定为灿烂弧菌Vibrio splendidus。
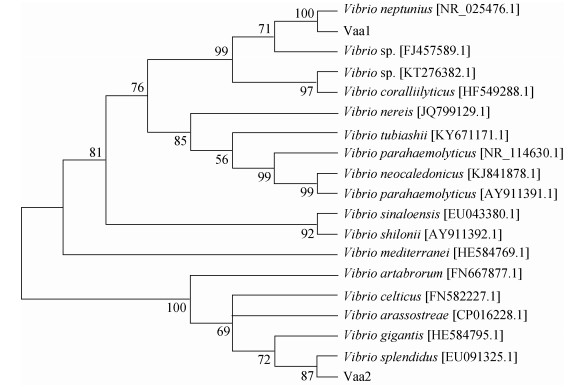 |
| 图 4 以16S rRNA基因构建的弧菌系统发育树 Fig. 4 Phylogenetic tree of Vibrio based on 16S rRNA gene |
将Paa1、Paa2、Vaa1、Vaa2四种细菌菌株接种于100 mL 2216e液体培养基中培养过夜后, 利用福林-酚试剂法分别测定各细菌培养液的蛋白酶活性, 结果显示:四种细菌培养液均具有蛋白酶活性, 其中Paa1与Paa2细菌培养液蛋白酶活性分别为20.57 U/mg、40.50 U/mg, Vaa1和Vaa2的细菌培养液蛋白酶活性分别为509.7 U/mg、321.3 U/mg。通过实验结果显示, 从2015年发病海月水母中分离的菌株Paa1与Paa2培养液的蛋白酶活性要小于从2017年发病海月水母中分离得到的Vaa1与Vaa2菌株培养液的蛋白酶活性。经过分析认为, 有可能是由于菌株Paa1与Paa2在此实验条件下产蛋白酶能力较弱, 或者是由于错过了最佳产蛋白酶的菌株培养时间, 从而导致所测产蛋白酶活性偏低。
3 讨论 3.1 实验室养殖条件下细菌对海月水母大规模发病解体的作用分析目前, 国内外对海月水母的相关研究主要集中于其生态学影响、生活史、生活习性、水母体成分组成以及饲养繁殖条件等方面。对于人工养殖条件下, 由细菌导致的海月水母大规模发病的相关研究鲜见报道。本实验对人工饲养条件下分别于2015年与2017年突然出现的两次大规模发病烂洞的海月水母各自进行细菌检测分析, 以企图寻找致病原因。经过细菌培养, 2015年与2017年烂洞海月水母样品分别在2216e固体培养基中各获得了2种不同种类的优势菌株(Paa1 & Paa2、Vaa1 & Vaa2)。经分子生物学鉴定确定2015年烂洞海月水母分离到的优势菌株为2种嗜冷杆菌属细菌(Psychrobacter sp.), 而从2017年烂洞海月水母中分离到的2种优势菌株隶属弧菌属细菌, 分别为海神弧菌(Vibrio neptunius)和灿烂弧菌(Vibrio splendidus)。
海洋细菌在海洋中分布广泛、种类和数量繁多, 其中弧菌和嗜冷杆菌是海洋细菌的重要组成部分。目前关于嗜冷杆菌作为病原菌致病的案例较少, 曾有研究报道嗜冷杆菌能够引起条斑紫菜褐斑病[13], 另有研究显示嗜冷杆菌感染可导致人类脑膜炎[14]。研究显示, 嗜冷杆菌广泛存在于海水中[15], 多种嗜冷杆菌属细菌具有产脂肪酶、蛋白酶、丙酮酸脱氢酶等各种酶类的性能[16-18]。本实验通过利用福林-酚试剂法检测到从2015年烂洞海月水母样品中分离到的2种嗜冷杆菌Paa1和Paa2均具有蛋白酶活性, 这与此前关于嗜冷杆菌具有蛋白酶活性的报道相一致[17]。弧菌作为海洋微生物的优势细菌类群之一, 也是海洋生物致病的最常见病原菌, 国内外学者对其研究颇多。其中海神弧菌和灿烂弧菌可引发鱼类、贝类等水产养殖动物患多种疾病[19-21]。本实验经过检测显示从2017年烂洞海月水母样品中分离到的2种优势菌株海神弧菌Vaa1和灿烂弧菌Vaa2均具有较强的蛋白酶活性。海月水母通体透明, 体内含97%~98%的水分[22], 此外还有少量的蛋白质和盐分, 其中蛋白质是海月水母细胞结构的重要组成成分。根据实验结果认为, 导致2015年和2017年养殖海月水母大规模伞体烂洞解体的原因, 可能是由于外来饵料携带大量细菌, 并且养殖海水(温度为25℃)达到了适宜病原菌(嗜冷杆菌或弧菌)快速繁殖的条件, 从而导致病原菌大量存在, 产蛋白酶的病原菌使海月水母伞体蛋白质降解, 破坏水母细胞的蛋白结构进而引起水母解体消亡。这与刘全永等[23]关于渤海水母体细菌的相关研究以及水母采集后快速解体的猜测相吻合。在此后的实验室养殖过程中, 当有海月水母伞体出现轻微烂洞情况时, 尝试在养殖缸的水循环装置中添加紫外灯, 发现伞体烂洞逐渐缩小直至自愈。因此, 我们在所有养殖缸中都添加了紫外灯进行灭菌, 至此再未发生大规模海月水母烂洞事件。
3.2 关于水母暴发后水母快速消亡的原因推测目前, 水母暴发机制及其生态学影响成为国内外科研工作者的研究热点, 在大规模暴发后水母的消亡也越来越引起学者的关注。研究表明, 水母消亡会对海洋生物群落、溶解氧、酸碱度及生源要素等产生重要影响[24]。但是对水母消亡的原因尚未有确切定论。有专家学者认为, 水母暴发后由于食物缺乏和低养分环境等原因导致水母老化进而消亡[3], 另有学者认为是由于长期的负面因素如寄生虫、细菌感染、饥饿等的累积效应而导致水母消亡[25], 还有研究认为低温会导致水母活力下降从而大规模下沉(Jelly-fall)后消亡[26]。本实验通过人工养殖环境下偶然出现的海月水母大规模烂洞消亡事件认为, 在自然海域中发生的水母暴发后的快速消亡与致病菌大量存在有密切关系。
海洋中存在种类丰富、数量庞大的微生物, 约占海洋生物的90%[27], 且其中有很多致病性的细菌、病毒和真菌等。水母作为较低等的无脊椎动物, 体腔开放, 结构简单, 体表和机体组织长期接触各种病原微生物, 因其缺乏后天免疫体系, 仅能依靠在漫长的自然进化过程中逐渐形成的强大的先天免疫系统来抵抗病原体的入侵。研究显示, 水母体内含有丰富的胶原蛋白, 具有较好的免疫增强作用[28]。另外, 水母体内还含有较多的蛋白酶抑制剂、抗菌肽、凝集素类等[29-31], 这对于生存在复杂海水表层环境中的水母抵御外界病原体入侵具有举足轻重的作用。而在水母大量暴发后, 由于水母老化、食物匮乏、低氧环境等因素导致水母的先天性免疫系统弱化, 抵抗力降低[3, 25]。另外, 随着水母的暴发, 会引起海洋细菌丰度和细菌生产力的增加[32]。研究表明, 水母代谢产生的溶解性有机物(DOM)为细菌提供更多的物质来源, 能够快速促进细菌的生长[33-34]。Hansson曾报道海月水母释放的溶解性有机物能够显著促进异养细菌的生长[35]。与此同时, 由于水母对主要优势微型生物类群纤毛虫的摄取, 导致纤毛虫对细菌的摄食作用降低[36]。另有研究表明, 主要由于温度的影响, 一般规律下夏季海域中浮游细菌的丰度和生物量较其他季节较高[37-38], 而夏季也是水母暴发后快速消亡的高峰期。综合以上因素推测, 自然海域中, 水母暴发后的食物缺乏、低氧等压力环境导致水母机体退化、抵抗力降低, 同时水生环境中浮游细菌的大量存在且快速生长, 最终致使水母没有足够能力抵抗大量外来细菌的侵蚀, 从而导致水母被细菌解体, 随着机体消解的进行, 水母体释放的有机物又为细菌的繁殖生长提供更多的物质来源, 进一步加速了水母暴发后的消亡。
| [1] |
Dawson M N, Sen G A, England M H. Coupled biophysical global ocean model and molecular genetic analyses identify multiple introductions of cryptogenic specie[J]. PNAS, 2005, 102(34): 11968-11973. DOI:10.1073/pnas.0503811102 |
| [2] |
周永东, 刘子藩, 薄治礼, 等. 东、黄海大型水母及其调查监测[J]. 水产科技情报, 2004, 31(5): 224-227. Zhou Yongdong, Liu Zifan, Bo Zhili, et al. Large jellyfish in the East China Sea and Yellow Sea and its investigation and monitor[J]. Fisheries Science and Technology, 2004, 31(5): 224-227. DOI:10.3969/j.issn.1001-1994.2004.05.018 |
| [3] |
Purcell J E, Arai M N. Interactions of pelagic cnidarians and ctenophores with fish:a review[J]. Hydrobiologia, 2001, 451(1-3): 27-44. |
| [4] |
Mills C E. Jellyfish blooms:are populations increasing globally in response to changing ocean conditions[J]. Hydrobiologia, 2001, 451(1-3): 55-68. |
| [5] |
Link J S, Ford M D. Widespread and persistent increase of Ctenophora in the northeast US shelf ecosystem off NE USA[J]. Marine Ecology Progress, 2006, 320(11): 153-159. |
| [6] |
Matsueda N. Presentation of Aurelia aurita at thermal power station[J]. Bulletin of the Marine Biology Station, Asamushi, 1969, 13: 187-191. |
| [7] |
Papathanassiou E, Panayotidis P, Anagnostaki K. Notes on the biology and ecology of the jellyfish Aurelia aurita Lam. in Elefsis Bay(Saronikos Gulf, Greece)[J]. Marine Biology, 2010, 8(1): 49-58. |
| [8] |
郑凤英, 陈四清, 倪佳. 海月水母的生物学特征及其爆发[J]. 海洋科学进展, 2010, 28(1): 126-132. Zheng Fengying, Chen Shiqing, Ni Jia. Biological characteristics of moon jellyfish (Aurelia aurita Linnaeus, 1758) and its bloom[J]. Advances in Marine Science, 2010, 28(1): 126-132. DOI:10.3969/j.issn.1671-6647.2010.01.017 |
| [9] |
付志璐, 董婧, 孙明, 等. 温度、盐度对黄海北部海月水母碟状幼体生长的影响[J]. 水产科学, 2011, 30(4): 221-224. Fu Zhilu, Dong Jing, Sun Ming, et al. Effects of water temperature and salinity on growth of ephyrae in moon jellyfish Aurelia sp. in North Yellow Sea in China[J]. Fisheries Science, 2011, 30(4): 221-224. DOI:10.3969/j.issn.1003-1111.2011.04.008 |
| [10] |
郑向荣, 李燕, 饶庆贺, 等. 秦皇岛近海大型水母暴发性增长原因探析[J]. 河北渔业, 2014, 2: 16-20. Zheng Xiangrong, Li Yan, Rao Qinghe, et al. The cause analysis of giant jellyfish bloom in inshore of Qinhuangdao[J]. Hebei Fisheries, 2014, 2: 16-20. |
| [11] |
Hogg J C, Lehane M J. Identification of bspecies associated with the sheep scab mite (Psoroptes ovis) by using amplified genes coding for 16S rRNA[J]. Applied and Environment Microbiology, 1999, 65(9): 4227-4229. |
| [12] |
Persing D H, Tenover F C, Tang Y W. Molecular microbiology:Dignostic Principles and Practice[M]. Washington DC: American Society for Microbiology, 2003: 454-483.
|
| [13] |
陈丽, 白雪松, 张晓君, 等. 条斑紫菜褐斑病病原嗜冷杆菌的生物学特性[J]. 江苏农业科学, 2012, 40(8): 234-236. Chen Li, Bai Xiaosong, Zhang Xiaojun, et al. Biological characteristics of Psychrobacter sp. as the brown spot pathogen on Porphyrae yezoensis[J]. Jiangsu Agricultural Sciences, 2012, 40(8): 234-236. DOI:10.3969/j.issn.1002-1302.2012.08.093 |
| [14] |
María O A J, Miguel S C J, Fabiola G A, et al. Fatal Psychrobacter sp. infection in a pediatric patient with meningitis identified by metagenomics next-generation sequencing in cerebrospinal fluid[J]. Archives of Microbiology, 2015, 198(2): 1-7. |
| [15] |
Bowman J P. The Prokaryotes:The Genus Psychrobacter[M]. New York: Springer New York, 2006: 920-930.
|
| [16] |
陈兴麟, 翁金樑, 曾润颖, 等. 极地产适冷脂肪酶菌株的酶学性质研究[J]. 台湾海峡, 2010, 29(1): 12-19. Chen Xinglin, Weng Jinliang, Zeng Runying, et al. Research of enzyme characters of cold-adapted lipase-produced bacteria from the polar[J]. Journal of Oceanography in Taiwan Strait, 2010, 29(1): 12-19. |
| [17] |
徐国英, 林学政, 王能飞, 等. 产低温蛋白酶极地菌株的筛选及Pseudoalteromonas sp.QI-1产蛋白酶粗酶性质[J]. 生物加工过程, 2010, 8(2): 55-60. Xu Guoying, Lin Xuezheng, Wang Nengfei, et al. Screening of cold-active protease strains from polar microorganism and partial characteristics of crude protease excreted by Pseudoalteromonas sp.QI-1[J]. Chinese Journal of Bioprocess Engineering, 2010, 8(2): 55-60. DOI:10.3969/j.issn.1672-3678.2010.02.011 |
| [18] |
张锋, 黄硕芩, 张金伟. 一株南极嗜冷杆菌产丙酮酸脱氢酶的性质研究及基因克隆[J]. 现代生物医学进展, 2006, 6(12): 40-43. Zhang Feng, Huang Suochen, Zhang Jinwei. Characterization and gene cloning of the pyruvate dehydrogenase E1(PDHE1) from an Antarctic Psychrobacter Strain[J]. Progress in Modern Biomedicine, 2006, 6(12): 40-43. |
| [19] |
Prado S, Romalde J L, Montes J, et al. Pathogenic bacteria isolated from disease outbreaks in shellfish hatcheries.First description of Vibrio neptunius as an oyster pathogen[J]. Diseases of Aquatic Organisms, 2005, 67(3): 209. |
| [20] |
Austin B, Austin D, Sutherland R, et al. Pathogenicity of vibrios to rainbow trout (Oncorhynchus mykiss, Walbaum) and Artemia nauplii[J]. Environmental Microbiology, 2010, 7(9): 1488-1495. |
| [21] |
马爱敏, 闫茂仓, 胡利华, 等. 患溃烂症的眼斑拟石首鱼分离的灿烂弧菌的鉴定[J]. 微生物学报, 2010, 50(4): 542-547. Ma Aimin, Yan Maocang, Hu Lihua, et al. Identification of a Vibrio splendidus strain from ill Sciaenops ocellatus[J]. Acta Microbiologica Sinica, 2010, 50(4): 542-547. |
| [22] |
Rackmil M, Messbauer A, Morgano M, et al. Investigations into the nutritional composition of moon jellyfish Auerlia aurita[J]. Drum and Croaker, 2009, 40: 33-47. |
| [23] |
刘全永, 胡江春, 马成新, 等. 渤海水母体细菌的微生态分布及弧菌生物学特性[J]. 应用生态学报, 2001, 12(6): 900-902. Liu Quanyong, Hu Jiangchun, Ma Chengxin, et al. Bacteria distribution and biological characteristics of vibrios in jellyfish from Bohai Sea[J]. Chinese Journal of Applied Ecology, 2001, 12(6): 900-902. DOI:10.3321/j.issn:1001-9332.2001.06.024 |
| [24] |
曲长凤, 宋金明, 李宁. 水母消亡对海洋生态环境的影响[J]. 生态学报, 2015, 35(18): 6224-6232. Qu Changfeng, Song Jinming, Li Ning. The effects of jellyfish decomposition on marine ecological environments[J]. Acta Ecologica Sinica, 2015, 35(18): 6224-6232. |
| [25] |
Mills C E. Natural mortality in NE Pacific coastal hydromedusae:grazing predation, wound healing and senescence[J]. Bulletin of Marine Science, 1993, 53(1): 194-203. |
| [26] |
Sexton M A, Hood R R, Sarkodeeadoo J, et al. Response of Chrysaora quinquecirrha medusae to low temperature[J]. Hydrobiologia, 2010, 645(1): 125-133. DOI:10.1007/s10750-010-0222-y |
| [27] |
Ausubel J H, Trew C D, Waggoner P E. First census of marine life 2010:highlights of a decade of discovery[J]. Census of Marine Life, 2010. |
| [28] |
Sugahara T, Ueno M, Goto Y, et al. Immunostimulation effect of jellyfish collagen[J]. Bioscience, Biotechnology, and Biochemistry, 2006, 70(9): 2131-2137. DOI:10.1271/bbb.60076 |
| [29] |
Ovchinnikova T V, Balandin S V, Aleshina G M, et al. Aurelin, a novel antimicrobial peptide from jellyfish Aurelia aurita with structural features of defensins and channel-blocking toxins[J]. Biochemical and Biophysical Research Communications, 2006, 348(2): 514-523. DOI:10.1016/j.bbrc.2006.07.078 |
| [30] |
Imamichi Y, Yokoyama Y. Purification, characterization and cDNA cloning of a novel lectin from the jellyfish Nemopilema nomurai[J]. Comparative Biochemistry and Physiology, Part B, Biochemistry & Molecular Biology, 2010, 156(1): 12-18. |
| [31] |
Liu G Y, Zhou Y H, Liu D, et al. Global transcriptome analysis of the tentacle of the jellyfish Cyanea capillata using deep sequencing and expressed sequence tags:insight into the toxin-and degenerative disease-related transcripts[J]. Plos One, 2015, 10(11): e0142680. DOI:10.1371/journal.pone.0142680 |
| [32] |
Zeng Y, Huang X, Huang B, et al. Relationship between bacteria and phytoplankton during the giant jellyfish Nemopilema nomurai bloom in an oligotrophic temperate marine ecosystem[J]. Acta Oceanologica Sinica, 2016, 35(10): 107-113. DOI:10.1007/s13131-016-0894-x |
| [33] |
Condon R H, Steinberg D K. Development, biological regulation, and fate of ctenophore blooms in the York River estuary, Chesapeake Bay[J]. Marine Ecology Progress, 2008, 69(1): 153-168. |
| [34] |
Condon R H, Steinberg D K, Giorgio P A D, et al. Jellyfish blooms result in a Major microbial respiratory sink of carbon in marine systems[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(25): 10225-10230. DOI:10.1073/pnas.1015782108 |
| [35] |
Hansson L J, Norrman B. Release of dissolved organic carbon (DOC) by the scyphozoan jellyfish Aurelia aurita and its potential influence on the production of planktic bacteria[J]. Marine Biology, 1995, 121(3): 527-532. DOI:10.1007/BF00349462 |
| [36] |
Wang L, Xu K D. Spatiotemporal distribution of protozooplankton and copepod nauplii in relation to the occurrence of giant jellyfish in the Yellow Sea[J]. Chinese Journal of Oceanology and Limnology, 2013, 31(6): 1226-1240. DOI:10.1007/s00343-014-3095-5 |
| [37] |
张喆, 孟祥红, 肖慧, 等. 青岛近岸水体夏冬季浮游病毒、细菌分布特征及其与环境因子的关系[J]. 武汉大学学报(理学版), 2008, 54(2): 209-214. Zhang Zhe, Meng Xianghong, Xiao Hui, et al. Distribution characteristics of planktonic virus, bacteria and their relationship with environmental factors in coastal waters of Qingdao in summer and winter[J]. Journal of Wuhan University (Science Edition), 2008, 54(2): 209-214. DOI:10.3321/j.issn:1671-8836.2008.02.017 |
| [38] |
Zhao S J, Xiao T, Lu R H, et al. Spatial variability in biomass and production of heterotrophic bacteria in the East China Sea and the Yellow Sea[J]. Deep Sea Research Part Ⅱ:Topical Studies in Oceanography, 2010, 57(11): 1071-1078. |
 2018, Vol. 42
2018, Vol. 42

