文章信息
- 刘小卓, 张晓明, 董波. 2018.
- LIU Xiao-zhuo, ZHANG Xiao-ming, DONG Bo. 2018.
- 海洋动物microRNAs研究进展与展望
- Progress and perspective of microRNA research in marine animals
- 海洋科学, 42(3): 157-168
- Marine Sciences, 42(3): 157-168.
- http://dx.doi.org/10.11759/hykx20171211001
-
文章历史
- 收稿日期:2017-12-11
- 修回日期:2018-01-16
2. 青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室, 山东 青岛 266237
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266237, China
海洋动物由于其生存环境的特殊性造就了其独特的生理过程, 包括渗透压调控、免疫和幼虫附着变态等, 但目前调控这些生理过程的分子机制还知之甚少。最近几年, 随着组学等技术的发展推进, 在海洋动物中已经获得了大量miRNAs, 其中部分miRNAs的功能得到了验证。microRNA (miRNA)是一类长约18—25个核苷酸的非编码单链RNA分子, 通过碱基互补配对结合靶基因3’非翻译区, 降解靶基因mRNA或抑制其翻译[1-2]。miRNA首先在秀丽隐杆线虫(Caenorhabditis elegans)中被发现: Ambros等[3-5]通过定位克隆和定点突变的方法发现秀丽隐杆线虫lin-4基因并不编码蛋白, 而是在转录后形成22和61个核苷酸的转录产物, 这些转录产物结合到另外一个基因lin-14的3’非翻译区, 抑制lin-14的翻译。Reinhart[6]等也在秀丽隐杆线虫中发现let-7可以通过结合lin-41的3’非翻译区, 从而抑制其表达。后来, 对其他物种的研究发现miRNA普遍存在于真核动物中, 物种间具有高度的保守性, 表达具有时序性和组织特异性, miRNA参与调控动物生长发育、免疫、应激反应及附着变态等多种生物学过程。作者将对近年来发现的海洋动物miRNA及其涉及的功能研究进行综述, 并对此领域未来研究提出展望。
1 miRNA的合成和调控机制miRNA是一类非编码短序列RNA, 在RNA聚合酶Ⅱ作用下, 1个miRNA基因被转录形成一段长度超过1kb的miRNA初级转录产物(pri-miRNA)[7]; 或者几个距离较近的miRNA构成一个多顺反子转录单元, 共同起始转录[8]。动物体内典型的pri-miRNA包含1个长约33~35个碱基的茎环双链结构, 在5’和3’末端各存在一段单链RNA。pri-miRNA被RNA Ⅲ型核酸内切酶Drosha识别并剪切, 释放出一段65个碱基左右的发夹状片段, 形成miRNA前体(precursor miRNA, pre-miRNA)[9]。miRNA前体和转运蛋白Exportin-5以及GTP结合蛋白RanGTP形成转运复合体被转运到细胞质[10], RNA Ⅲ型核酸内切酶Dicer对其进行加工形成miRNA双链复合体。随后双链miRNA在解旋酶的作用下解链, 产生两条具有生物学活性的成熟miRNA[11](图 1)。
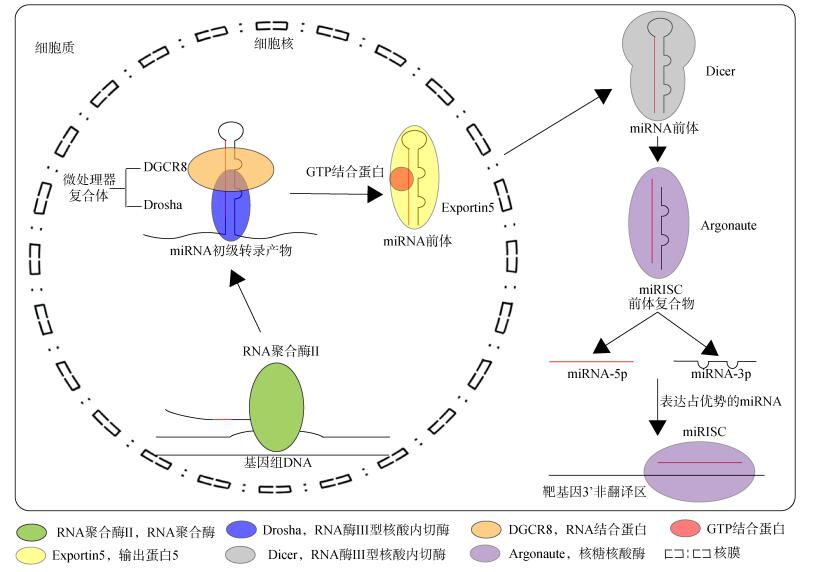 |
| 图 1 动物miRNAs合成过程和调控机制示意图 Fig. 1 Biogenesis and regulatory mechanisms of animal miRNAs |
miRNA主要通过抑制靶基因翻译[12], 或者降低靶基因稳定性[13], 甚至是激活基因的表达来调控靶基因的功能[14]。新近研究发现菟丝子(Cuscuta campestris)的miRNA能够跨物种靶向寄主靶基因, 从而调控寄主基因的表达, 展示了一种miRNA跨物种调控基因表达的新模式[15]。因为只有极少数动物miRNA与靶基因以完全互补的形式结合, 大部分miRNA可以不完全与靶基因序列配对结合, 因此一个miRNA可以调控多个靶基因[16]。
2 海洋动物miRNAs海洋占据了总地球总面积的71%, 拥有着数量庞大的生物资源, 截至2017年5月, 海洋生物种类多达243 000种[17]。海洋动物miRNA研究起步相对较晚, 截至2017年12月, miRBase(http://www.mirbase.org)中已收录2191条海洋动物成熟miRNA, 它们在生殖、生长、发育、免疫调控和代谢等方面具有重要的功能。除了海鞘纲(Ascidiacea)和海胆纲(Echinoidea)等海洋模式动物以及部分鱼类, 大部分海洋物种的miRNA研究还仅限于miRNA的筛选及鉴定, 对于miRNA功能的研究报道还比较有限(表 1)。
| 物种 | 发现miRNA数量/个 | 数据来源 | 表达情况 | 具有调控功能的miRNAs | 功能 | 参考文献 | |
| 海洋鱼类 | 牙鲆 | 140 | 孵化后13, 17, 19, 21, 23, 29, 31, 33, 36及42 d个体 | 12个miRNA在29 dpf表达上调;54个miRNA在29 dpf表达下调 | let-7 | 参与变态期细胞增殖与分化 | [18] |
| miR-731 | 诱导病毒在侵染阶段的复制 | [19] | |||||
| 孵化后17与29 d个体 | 孵化后17 d表达量显著低于29 d表达量 | miR-1, miR-206b | 调掉牙鲆变本过程 | [20] | |||
| 孵化后17 d表达量显著高于29 d表达量 | miR-10d, miR-22a | ||||||
 鱼 鱼 |
6 | 鳗弧菌侵染后 鱼个体 鱼个体 |
侵染后表达量上升 | miR-8195 | 抑制TLR13负向调节 鱼免疫应答 鱼免疫应答 |
[21] | |
| 侵染后表达量下降 | miR-122 | 参与到Toll样受体调节的免疫反应 | [22] | ||||
哈维氏弧菌侵染后 鱼个体 鱼个体 |
侵染后表达量上调 | miR-148 | 抑制炎症细胞因子表达量,炎症反应 | [23] | |||
| 大西洋庸鲽 | 42 | 5龄个体 | 在脑部表达量显著高于其他组织 | miR-3588, miR-34b, miR-574 | 脑部发育和再生 | [24] | |
| 在性腺高表达 | miR-24 | 调节性成熟 | |||||
| 未发现特异性表达区域 | miR-301b | 抑制6-磷酸葡萄糖酸脱氢酶调控代谢途径 | |||||
| 头索动物 | 厦门文昌鱼 | 155 | 经细菌侵染6, 12, 18, 24, 32, 40, 48 h后个体及正常个体 | 侵染后表达量下调 | miR-92d | 调节急性免疫反应抵抗细菌侵染 | [25] |
| 侵染后表达量上调 | miR-7, miR-4868a, miR-2065, miR-34b | 抑制病毒转录 | |||||
| 棘皮动物 | 刺参 | 40 | 正常及皮肤溃疡综合征个体 | 被侵染后表达量下调 | miR-137 | 调控体腔细胞的凋亡过程 | [26] |
| 经灿烂弧菌侵染后的个体及正常个体 | 被侵染后表达量下调 | miR-210 | 调控TLR通路抵御细菌侵染 | ||||
| 被侵染后表达量上调 | miR-31 | [27] | |||||
| miR-200 | 作用到AjTollip上增强体腔细胞抗菌能力 | [28] | |||||
| 光棘球海胆 | 345 | 正常个体 | 表达量最高 | miR-184 | 物种进化 | [29] | |
| 紫海胆 | 49 | 卵巢,受精卵,32细胞期,囊胚期,原肠胚,早期幼虫 | 在早期胚胎高表达 | miR-1, miR-71, miR-31 miR-2012 | 调控早期胚胎发育 | [30] | |
| 点状分布于早期胚胎中 | miR-31 | 影响PMC基因调控网络,抑制海胆骨架形成 | [31] | ||||
| 甲壳动物 | 日本囊对虾 | 199 | 经MSSV病毒侵染个体 | 分布于被MSSV侵染的组织中 | miR-7 | 参与机体免疫反应,抑制WSSV病毒表达 | [32] |
| miR-12 | 抵御病毒的吞噬,启动抗病毒细胞凋亡机制 | [33] | |||||
| miR-100 | 抑制细胞凋亡 | [34] | |||||
| 大西洋无眼裂缝虾 | 193 | 肌肉组织 | 表达量最高的miRNA | miR-100 | 消化器官生成 | [35] | |
| 梭子蟹 | 67 | 鳃部 | 低盐胁迫处理个体 | miR-317, miR-2788, miR-305 | 抵抗低盐胁迫对机体生理机能的影响 | [36] | |
| 拟穴青蟹 | 421 | 肠,心脏,肝脏,腮,脑,肌肉,血 | 在被副溶血弧菌侵染的个体表达存在差异 | miR-146, miR-125 | 调控先天免疫 | [37] | |
| 软体动物 | 太平洋牡蛎 | 199 | 分别经染菌、加热染菌共处理个体的血细胞 | 4个miRNA在染菌、加热染菌共处理个体中表达量显著上升 | miR-2a, miR-307, miR-745b, miR-1984 | 维持逆境血细胞功能 | [38] |
| 3个miRNA在染菌、加热染菌共处理个体中表达量显著降低 | miR-10a, miR-10b, miR-182 | ||||||
| 过表达miRNA | 血细胞迁移率显著降低 | scaffold42648 _ 5080 | 通过降低血细胞迁移率,调控免疫响应 | [39] | |||
| 经暴露过空气及未经处理 | 暴露过空气的个体miRNA表达量显著提升 | miR-365 | 抵抗潮间带的缺水环境 | [40] | |||
| 香港巨牡蛎 | 87 | 正常个体及低盐胁迫个体 | 经低盐胁迫后高表达 | miR-3205 | 应对胁迫环境 | [41] | |
| 经低盐胁迫后低表达 | miR-2353 | ||||||
| 栉孔扇贝 | 68 | 正常个体及经AVNV侵染个体 | 经侵染后表达量显著上升 | miR-92, miR-745b, miR-745a, miR-279 | 抵御AVNV病毒侵染 | [42] | |
| 泥蚶 | 254 | 正常及经重金属镉处理个体 | 经重金属处理后表达量上升 | miR-21 | 维持体内稳态及耐受性 | [43] | |
| miR-8, miR-10, miR-67 | 与重金属转运相关 | ||||||
| 马氏珠母贝 | 258 | 高通量测序 | 过表达后珍珠层生长模式紊乱 | miR-2305, miR-29a, miR-183 | 调控珍珠层生长模式 | [44-47] | |
| 在马氏珠母贝中特有 | miR-0046 | 参与生物矿化调控 | [44] | ||||
海洋动物miRNAs具有进化保守性。学者[20]在牙鲆(Paralichthys olivaceus)中发现了140个在后口动物中保守的miRNAs, 在头索动物(包括佛罗里达文昌鱼(Branchiostoma floridae)[48-49]和厦门文昌鱼(Branchiostoma belcheri)[50])中鉴定出54个脊椎动物保守的miRNAs家族、18个无脊椎动物中保守的miRNAs, 在光棘球海胆(Strongylocentrotus nudus)中发现了345个后口动物保守的miRNAs[29]。
除了进化上保守的miRNAs, 海洋动物在进化过程中还丢失了一些miRNAs或获得了新的特异性miRNAs。通过与已知的miRNAs比对, 发现佛罗里达文昌鱼和厦门文昌鱼都存在一些物种特异的miRNAs[25]。从深海热液中存活的优势种——大西洋无眼裂缝虾(Rimicaris exoculata)的肌肉组织中鉴定出159个已知的miRNAs和34个新miRNAs, 其中miR-275、miR-276、miR-iab4-3p和miR-iab4-5被推测是在节肢动物出现以后, 添加到后口动物基因组中的; miR-2001和miR-277系首次在甲壳纲(Crustacean)中发现[35]。Xu等[51]获得19个太平洋牡蛎(Crassostrea gigas)的新pre-miRNAs, 这些miRNAs的表达具有明显的组织特异性。
3 miRNA调控海洋动物重要生理过程miRNA广泛参与各种不同生理过程, 包括发育、生长、细胞增殖、细胞凋亡和应激反应等。由于海洋环境的特殊性, 海洋动物具有许多独特的生理机能, 如感知水流、渗透压调节、鳃呼吸、附着变态、无限生长发育等, 多种miRNA已被鉴定出参与其中(图 2)。
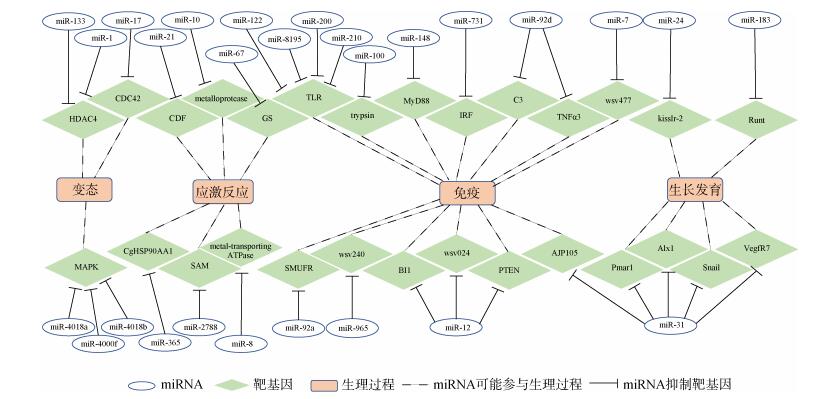 |
| 图 2 miRNAs与靶基因相互作用调控海洋动物重要生理过程 Fig. 2 miRNAs regulate the physiological processes of marine animals through their targeted genes |
研究发现miRNAs对紫海胆(Strongylocentrotus purpuratus)早期细胞命运决定起了重要的调控作用, 通过抑制Dicer和Drosha基因功能从而影响海胆miRNAs的形成可导致胚胎无法正常发育成原肠胚, 同时也影响调控胚层分化相关基因的表达[30]。Stepicheva等[31]发现miR-31在海胆不同发育时期不同组织中的表达没有显著差异, 但敲除miR-31后, 影响初级间充质细胞正常功能, 海胆胚胎不能正常发育。研究证实miR-31通过抑制Pmar1、Alx1、Snail和VegfR7基因表达, 从而最终抑制海胆骨架形成。总之, miRNA在海胆发育过程中起到了至关重要的作用。
性腺的发育情况及生殖能力直接关系到海水养殖物种所带来的经济价值。研究发现miRNAs let-7、let-7c、let-7f、mir-2、mir-184和mir-276在梭子蟹(Portunus trituberculatus)卵巢和精巢高表达[52]。已有研究表明miR-184调控果蝇(Drosophila melanogaster)雌性生殖细胞的发育, miR-184的缺失导致卵子形成的异常[53], miR-184还可以促进雄性生殖细胞的增殖[54], 据此推测性腺中高表达的这些miRNAs在梭子蟹性腺发育中发挥重要作用。在大西洋庸鲽中, miR-24主要表达在精巢中, 实验证实kiss1r-2是miR-24的靶基因, 而kiss1r-2是脊椎动物调控性腺发育的关键基因, 这也暗示了miR-24通过调控kiss1r-2表达, 进而调控大西洋庸鲽的性腺发育[24]。
miRNA还参与调控贝类珍珠层的形成及生物矿化过程。在马氏珠母贝中发现miR-2305和miR-0046的靶基因包括多个生物矿化相关基因(biominerali za tion-related genes)[44], 其中将miR-2305注射入马氏珠母贝的肌肉后, 珍珠层的生长模式紊乱[45]。过表达miR-29a的表型与过表达miR-2305的表型一致, 也会引起珍珠层生长模式的紊乱[46]。另外, miR-183被发现可以显著降低其靶基因Runt转录因子的表达, 从而调控珍珠层的形成[47]。
3.2 免疫鱼类免疫系统包括先天性或非特异性防御系统以及获得性或特异性防御系统, miRNAs在鱼类免疫系统的作用机制接近于哺乳动物。细菌或病毒感染的半滑舌鳎(Cynoglossus semilaevis)[55]和牙鲆[19]的不同组织中均检测到表达量明显变化的miRNAs, 预测结果显示这些miRNAs的靶基因参与多种免疫相关的分子通路。Toll样受体(Toll-like receptors, TLR)是参与非特异性免疫(天然免疫)的一类重要蛋白质。当病原入侵时, TLRs被活化并识别病原体相关分子模式, 其后通过结合髓样分化因子(Myeloid differentiation factor 88, MyD88), 诱导炎症细胞因子的转录, 激活下游信号通路, 产生免疫反应。TLR信号通路基因TLR13、TLR14、IRF7和MyD88等都已发现是参与鱼类炎症反应及免疫应答miRNAs的靶基因[19, 21-23], 一方面TLRs可以诱导miRNAs的表达, 同时miRNAs也调控TLRs通路。在
在高等脊椎动物中已有研究表明病毒可以利用寄主的miRNA为病毒自身复制提供更良好的条件[57], 在鱼类中也发现病毒可以诱导寄主miRNA表达, 使机体失去抗病毒的功能[19]。当细胞肿大病毒(megalo cytivirus)侵染牙鲆时, miR-731的表达量明显上升, miR-731通过调控干扰素调节因子7(interferon regulatory factor 7, IRF7)和抑癌基因p53表达, 增加病毒在早期感染时的复制, 也抑制了p53介导的细胞凋亡[58], 这与哺乳动物miR-146a和miR-155的功能类似。IRF7是Ⅰ型干扰素(type Ⅰ IFN)响应的主要效应因子, miRNA通过抑制Ⅰ型IFN通路, 负调控Ⅰ型IFN免疫响应, 维持免疫响应稳态[59-60]。上述研究成果丰富了鱼类miRNA在免疫应答中的功能, 勾画了一个由miRNAs构成的免疫调控网络。
无脊椎动物对病原体的防御是非特异性的, 它们缺乏获得性免疫功能, 仅具有先天性免疫, 主要通过细胞免疫和体液免疫来发挥免疫应答功能。现有研究已经在刺参[61]、太平洋牡蛎[41]、栉孔扇贝[42], 拟穴青蟹[37]、日本囊对虾[32]等海洋无脊椎动物中鉴定出上百个参与免疫反应的miRNAs。当灿烂弧菌侵染刺参后, miR-200通过正向调节Toll作用蛋白(Toll-interacting protein, AjTollip)的表达参与刺参的免疫响应[28]。miR-31的靶基因Ajp105属于一类重要的转录因子家族成员, 其可正向调控NF-κB基因的表达, 过表达miR-31后显著提高了机体的氧化应激反应[27], 说明miR-31在免疫响应中发挥作用。Aj14- 3-3ζ基因是miR-137的靶基因之一, 分别敲降miR- 137或者Aj14-3-3ζ都会促进体腔细胞凋亡, 据此推测miR-137正向调节Aj14-3-3ζ的表达从而调控体腔细胞的凋亡过程[26]。研究发现灿烂弧菌侵染太平洋牡蛎后, miR-92d的表达量显著降低, 而其靶基因肿瘤坏死因子α效应因子3(Tumor necrosis factor α factor 3, TNFα factor 3, LITAF3)的表达量显著提高, LITAF3可以通过TLR-2/4调节的信号通路激活肿瘤坏死因子(Tumor necrosis factor a, TNFa)的表达, 参与调控免疫响应[62]。对太平洋牡蛎进行热激处理后, 共有65个miRNAs表达量发生变化, 其中42个miRNAs在细菌侵染时也发生了显著变化, 说明机体通过调节与免疫相关miRNA的表达应对外界热激刺激[38]。血细胞中也包含了大量的免疫细胞, 在免疫系统中扮演了重要的角色, 血细胞迁移被认为是免疫响应最主要的特点[39]。scaffold42648_5080是在太平洋牡蛎中特异表达的miRNA, 由牡蛎编码基因的内含子区域转录而来。经灿烂弧菌或内毒素处理后, scaffold42648_5080的表达量均显著下调, 因此推断scaffold42648_5080对于革兰氏阴性菌普的免疫响应具有普适性; 过表达scaffold42648_5080, 血细胞细胞吞噬、细胞凋亡及迁移率都与对照组存在显著差别; 敲降scaffold42648_5080的靶基因整合素连接激酶(integrin-linked kinase, ILK), 血细胞迁移率也显著降低, 因此推测scaffold42648_5080参与调控太平洋牡蛎免疫应答过程[39]。
多项研究证实miRNA在海洋动物抵抗外界病毒侵染过程中起到了重要的作用。Yang等[34]通过分析日本囊对虾miRNA微阵列数据发现了199个miRNA与WSSV感染引起的细胞凋亡有关, 其中, miR-100通过作用于胰蛋白酶(trypsin)信号通路, 抑制血细胞凋亡, 进而抑制WSSV病毒的侵染。miR-100可以通过负调控靶基因的表达诱导细胞凋亡已经在多种哺乳动物细胞系中被证实[63-64]。对虾白斑综合症病毒侵染对虾后, miRNA可以通过作用于病毒基因或宿主基因, 诱导机体释放多种激活因子, 激活抗病毒信号通路抑制病毒增殖[32-33]。日本囊对虾被WSSV侵染后, miR-7可以作用于对虾白斑综合征病毒wsv477基因, 抑制WSSV病毒复制, 参与机体的免疫反应[65]。Shu[33]等在日本囊对虾中发现miR-12的靶基因包括wsv024、PTEN和BI1。miR-12可以靶向结合wsv024的3’UTR, 抵御WSSV病毒的感染; miR-12与PTEN相互作用, PTEN抑制免疫受体酪氨酸活化基序的信号转导, 并控制RAC的活性, 从而抑制细胞自噬; miR-12还可抑制BI1表达, 启动抗病毒的细胞凋亡机制。在栉孔扇贝的研究中Chen[42]等构建了经急性病毒性坏死病毒侵染和未侵染的miRNA文库, 在病毒侵染组中获得了37个表达量变化明显的miRNAs, 这些miRNA靶基因主要是与免疫或胁迫相关的蛋白, 因此推断栉孔扇贝miRNA在抵御病原体侵染中有着十分重要的作用。
3.3 附着变态附着变态是很多海洋动物类群生活史中的关键环节, 涉及底质选择、幼体器官退化和成体器官发生等过程, 期间幼体细胞经历形变、迁移、生长、分化和凋亡等复杂细胞学过程。这些细胞学变化通过系列级联信号传导通路和基因网络调控, 并且在幼体附着变态过程中高度协调有序发生。鲽形目(Pleuro nectiformes)鱼类发育过程中, 经历了一个非常奇妙的变态过程, 右眼经过背部迁移到左眼上方位于身体同一侧, 身体偏转90度, 在变态过程中, 随着组织结构及器官功能的改变, 鱼的生活习性由浮游型生活方式转变为底栖型生活方式[66-67]。继Inui等[68]首次发现甲状腺素T4在牙鲆变态过程中显著上升以来, 大量关于牙鲆变态的研究围绕着甲状腺素展开, 但甲状腺素调节牙鲆变态的分子机制仍不清楚。全基因组和转录组数据分析揭示, 褐牙鲆幼体变态过程受甲状腺激素、视黄酸信号通路以及光传导等通路调控[69]。可能参与鱼类变态过程的miRNA也已经被报道, 通过miRNA微阵列技术(miRNA-microarray)发现了66个miRNA在牙鲆变态时期的表达存在显著差异, 其中包括3个调控成肌细胞增殖分化的miR-1、miR-133和miR-206、调控色素沉积的miR-25及在皮肤特异表达的miR-203[20]。变态前期let-7[18]和miR-17[70]表达量显著提高, 用硫脲抑制let-7的表达, 牙鲆变态过程停滞, 说明let-7参与调控牙鲆变态过程; 过表达miR-17, 其靶基因CDC42的表达量显著降低, miR-17通过抑制CDC42表达调控软骨形成、增殖及骨的重吸收过程, 最终调控牙鲆体轴不对称的发育和组织器官的重排。
海鞘属于尾索动物, 具有浮游生活和固着生活两个生活阶段, 附着变态是衔接这两个阶段的重要生理过程。海鞘幼体附着变态包含了一系列细胞学改变, 由多种分子机制进行调节。MAPK信号通路参与调控玻璃海鞘(Ciona intestinalis)的附着变态过程, 浮游幼体后期, 表皮细胞生长因子(Epidermal Growth Factor, EGF)和ERK信号通路被激活, 细胞外信号促使浮游幼体通过附着乳突附着在基底, 然后位于中枢神经系统的JNK和β肾上腺素信号传导通路被激活, 从而触发海鞘蝌蚪幼体尾部不同类型细胞的凋亡退化[71]。作者已经筛选出海鞘附着变态期表达量明显变化的诸多miRNAs, 并鉴定出3个miRNAs (miR-4000f、miR-4018a和miR-4018b)特异性表达在海鞘幼体的间充质细胞中, 幼虫变态后, 其表达水平显著降低, 这意味着其可能在海鞘幼体变态中通过释放抑制表达的靶基因而调控成体组织的新生过程[72]。
研究人员应用差异显示PCR、基因芯片和转录组学等方法鉴别了大量鲍[73-75]和牡蛎[76-77]等附着变态的相关基因; 应用蛋白质组学研究方法, 识别了牡蛎附着变态相关的多种蛋白[78]。参与调控藤壶(Amphibalanus amphitrite)附着变态和基质选择的重要基因和蛋白也相继被发现[79]。但海洋无脊椎动中与附着变态相关的miRNAs至今报道不多。太平洋牡蛎中, 已经识别了包括cgi-miR-33-5p在内的多个miRNA在面盘幼虫时期高表达, 由于miR-33-5p的保守性及其人类同源miRNAs, miR-33a-5p和miR-33b- 5p对胰岛素信号通路和脂肪酸代谢的调控功能[80-81], 推测miR-33-5p可能参与幼虫变态时期的代谢调控[51]。
3.4 应激反应盐度是水生动物生理代谢的重要影响因子, 水环境中盐度的变化会引起动物各项生理指标的变化, 海洋动物对盐度的反应主要靠渗透压调节来完成。miRNAs对海洋动物渗透压的调控机制尚不明确[41], 这方面的研究报道也非常有限。在受低盐胁迫的情况下, 太平洋牡蛎miR-1984、miR-92-3p高表达, 而miR-183、miR-2353和miR-184-3p表达明显降低[40], miR-183通过激活丝裂原活化蛋白激酶(MAPK)通路调节细胞凋亡[82], 参与环境胁迫调控; 香港巨牡蛎miR-3205表达量增加而miR-2353显著降低[41]; 梭子蟹miR-2788可以调控腺苷甲硫氨酸合成酶(S-adenosylmethionine synthetase, SAM)的表达, SAM对植物抵御高盐、高碱环境有着重要的作用, 暗示了miR-2788在梭子蟹渗透压调控中有着十分重要的作用[36]。上述表达变化显著miRNA的靶基因涉及了很多渗透压调节过程, 如细胞内蛋白运输, 氨基酸代谢, 几丁质代谢和离子转运等[36]。鱼类可以通过肾脏、鳃保持体内水分和盐的平衡, 控制体表细胞膜的通透性来调节渗透压, 渗透胁迫转录因子1(osmotic stress transcription factor 1, OSTF1)在渗透压调节过程中起着重要作用, miR-8家族[83]和miR-30c[84]被报道参与淡水鱼类渗透压的调节, 但调控海水鱼类渗透压的miRNA至今未见报道。
研究证明去甲肾上腺素(Norepinephrine, NE)在牡蛎处于干燥环境时会显著上升, 在太平洋牡蛎中, miR-365被去甲肾上腺素调控, 从而进一步上调CgHSP90AA1的表达, 抵抗潮间带的缺水环境; 抑制去甲肾上腺素的分泌, miR-365的表达也会受到抑制[40], 因此在缺水条件下, 机体可以通过NE调节miRNA的表达抵御干燥环境。Bao[43]等构建了重金属镉处理泥蚶(Tegillarca granosa)及未处理泥蚶的miRNA库, 获得了重金属镉处理后表达量显著变化的miRNA, 其中, miR-21的靶基因是阳离子转运和扩散蛋白(cation transport and diffusion facilitators, CDF), 说明miR-21可能通过抑制CDF, 维持体内稳态及耐受性; 同时他们还发现了一些与重金属转运相关的靶基因, 如重金属转运ATP酶, 解聚素和金属蛋白酶, 谷氨酰胺合成酶和GTP酶激活蛋白, 它们分别是miR-8、miR-10、miR-67的靶基因, 因此他们推测miRNA在软体动物环境胁迫中扮演了极为重要的角色。
4 海洋动物miRNA研究存在的问题及展望通过建立不同组织、不同发育时期或不同生长条件下的miRNA文库, 已经在海洋动物中筛选并鉴定出上千个miRNA。研究结果表明miRNA在海洋动物免疫、发育、生长等过程中起着重要作用。虽然miRNA数量较多, 但对其表达定位及功能分析的研究还比较少, 经实验证实参与调控海洋动物发育、生长和免疫等过程的miRNA更是鲜有报道。
海洋动物具有在脊椎动物中功能保守的miRNA, 还包括一部分海洋动物自身特有的miRNA, 研究海洋动物特异miRNA将有利于了解miRNA的起源和进化机制, 发现miRNA的新功能。miRNA通过调控靶基因的表达行使生物学功能, 生物信息学预测靶基因数量增长迅速, 靶基因的鉴定就成为miRNA功能研究的关键。开发高通量特异的, 灵敏度高的miRNA靶基因筛选与鉴定方法对miRNA功能研究变得十分重要。大多数海洋动物是重要的经济物种, 发现并验证与经济性状相关的miRNA, 将会提高养殖动物的繁殖, 生长及抗病能力。
总之, 海洋动物的各项生理过程都通过复杂的信号传导通路和基因通路调控, miRNA是这一调控网络中的重要因子, 深入研究miRNA在海洋动物中的调控作用, 将促进对重要功能基因转录后调控机制的认识, 对于深入揭示海洋动物重要生理过程的分子机制具有重要意义。
| [1] |
Carthew R W, Sontheimer E J. Origins and mechanisms of miRNAs and siRNAs[J]. Cell, 2009, 136(4): 642-655. DOI:10.1016/j.cell.2009.01.035 |
| [2] |
Guo H, Ingolia N T, Weissman J S, et al. Mammalian microRNAs predominantly act to decrease target mRNA levels[J]. Nature, 2010, 466(7308): 835-840. DOI:10.1038/nature09267 |
| [3] |
Ambros V. A hierarchy of regulatory genes controls a larva-to-adult developmental switch in C. elegans[J]. Cell, 1989, 57(1): 49-57. DOI:10.1016/0092-8674(89)90171-2 |
| [4] |
Horvitz H R, Sulston J E. Isolation and genetic characterization of cell-lineage mutants of the nematode Caenorhabditis elegans[J]. Genetics, 1980, 96(2): 435-454. |
| [5] |
Lee R C, Feinbaum R L, Ambros V. The C. elegans heterochronic gene lin-4 encodes small RNAs with antisense complementarity to lin-14[J]. Cell, 1993, 5(5): 843-854. |
| [6] |
Reinhart B J, Slack F J, Basson M, et al. The 21-nucleotide let-7 RNA regulates developmental timing in Caenorhabditis elegans[J]. Nature, 2000, 403(6772): 901-906. DOI:10.1038/35002607 |
| [7] |
Lee Y, Kim M, Han J J, et al. MicroRNA genes are transcribed by RNA polymerase Ⅱ[J]. Embo Journal, 2004, 23(20): 4051-4060. DOI:10.1038/sj.emboj.7600385 |
| [8] |
Lee Y, Jeon K, Lee J T, et al. MicroRNA maturation:stepwise processing and subcellular localization[J]. EMBO J, 2002, 21(17): 4663-4670. DOI:10.1093/emboj/cdf476 |
| [9] |
Lee Y, Ahn C, Han J, et al. The nuclear RNase Ⅲ Drosha initiates microRNA processing[J]. Nature, 2003, 425(6956): 415-419. DOI:10.1038/nature01957 |
| [10] |
Bohnsack M T, Czaplinski K, Gorlich D. Exportin 5 is a RanGTP-dependent dsRNA-binding protein that mediates nuclear export of pre-miRNAs[J]. Rna-a Publication of the Rna Society, 2004, 10(2): 185-191. DOI:10.1261/rna.5167604 |
| [11] |
Griffiths-Jones S, Hui J H, Marco A, et al. MicroRNA evolution by arm switching[J]. EMBO Rep, 2011, 12(2): 172-177. DOI:10.1038/embor.2010.191 |
| [12] |
Petersen C P, Bordeleau M E, Pelletier J, et al. Short RNAs repress translation after initiation in mammalian cells[J]. Mol Cell, 2006, 21(4): 533-542. DOI:10.1016/j.molcel.2006.01.031 |
| [13] |
Bagga S, Bracht J, Hunter S, et al. Regulation by let-7 and lin-4 miRNAs results in target mRNA degradation[J]. Cell, 2005, 122(4): 553-563. DOI:10.1016/j.cell.2005.07.031 |
| [14] |
Mortensen R D, Serra M, Steitz J A, et al. Posttranscriptional activation of gene expression in Xenopus laevis oocytes by microRNA-protein complexes (microRNPs)[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(20): 8281. DOI:10.1073/pnas.1105401108 |
| [15] |
Shahid S, Kim G, Johnson N R, et al. MicroRNAs from the parasitic plant Cuscuta campestris target host messenger RNAs[J]. Nature, 2018, 553(7686): 82-85. DOI:10.1038/nature25027 |
| [16] |
Betel D, Wilson M, Gabow A, et al. The microRNA.org resource:targets and expression[J]. Nucleic Acids Res, 2008, 36: 149-153. |
| [17] |
Costello M J, Chaudhary C. Marine biodiversity, biogeography, deep-sea gradients, and conservation[J]. Curr Biol, 2017, 27(11): R511-R527. DOI:10.1016/j.cub.2017.04.060 |
| [18] |
Fu Y S, Shi Z Y, Wang G Y, et al. Expression of let-7 microRNAs that are involved in Japanese flounder (Paralichthys olivaceus) metamorphosis[J]. Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology, 2013, 165(2): 106-113. |
| [19] |
Zhang B C, Zhou Z J, Sun L. Pol-miR-731, a teleost miRNA upregulated by megalocytivirus, negatively regulates virus-induced type Ⅰ interferon response, apoptosis, and cell cycle arrest[J]. Scientific Reports, 2016, 6: 28354. DOI:10.1038/srep28354 |
| [20] |
Fu Y, Shi Z, Wu M, et al. Identification and differential expression of microRNAs during metamorphosis of the Japanese flounder (Paralichthys olivaceus)[J]. PLoS One, 2011, 6(7): e22957. DOI:10.1371/journal.pone.0022957 |
| [21] |
Cui J X, Gao Y H, Chu Q, et al. MiRNA-8159 is involved in TLR signaling pathway regulation after pathogen infection by direct targeting TLR13 in miiuy croaker[J]. Fish & Shellfish Immunology, 2017, 66: 531-539. |
| [22] |
Cui J X, Chu Q, Xu T J. MiR-122 involved in the regulation of toll-like receptor signaling pathway after Vibrio anguillarum infection by targeting TLR14 in miiuy croaker[J]. Fish & Shellfish Immunology, 2016, 58: 67-72. |
| [23] |
Chu Q, Gao Y H, Bi D K, et al. MicroRNA-148 as a negative regulator of the common TLR adaptor mediates inflammatory response in teleost fish[J]. Scientific Reports, 2017, 7(1): 4124-4133. DOI:10.1038/s41598-017-04354-9 |
| [24] |
Bizuayehu T T, Fernandes J M O, Johansen S D, et al. Characterization of novel precursor miRNAs using next generation sequencing and prediction of miRNA targets in Atlantic halibut[J]. Plos One, 2013, 8(4): e61378. DOI:10.1371/journal.pone.0061378 |
| [25] |
Yang R, Zheng T, Cai X, et al. Genome-wide analyses of amphioxus microRNAs reveal an immune regulation via miR-92d targeting C3[J]. J Immunol, 2013, 190(4): 1491-1500. DOI:10.4049/jimmunol.1200801 |
| [26] |
Lv M, Chen H, Shao Y, et al. miR-137 modulates coelomocyte apoptosis by targeting 14-3-3zeta in the sea cucumber Apostichopus japonicus[J]. Dev Comp Immunol, 2017, 67: 86-96. DOI:10.1016/j.dci.2016.11.008 |
| [27] |
Lu M, Zhang P, Li C, et al. MiR-31 modulates coelomocytes ROS production via targeting p105 in Vibrio splendidus challenged sea cucumber Apostichopus japonicus in vitro and in vivo[J]. Fish Shellfish Immunol, 2015, 45(2): 293-299. DOI:10.1016/j.fsi.2015.04.024 |
| [28] |
Lv Z, Li C, Zhang P, et al. MiR-200 modulates coelomocytes antibacterial activities and LPS priming via targeting Tollip in Apostichopus japonicus[J]. Fish Shellfish Immunol, 2015, 45(2): 431-436. DOI:10.1016/j.fsi.2015.04.014 |
| [29] |
Wei Z, Liu X, Feng T, et al. Novel and conserved micrornas in Dalian purple urchin (Strongylocentrotus nudus) identified by next generation sequencing[J]. Int J Biol Sci, 2011, 7(2): 180-192. DOI:10.7150/ijbs.7.180 |
| [30] |
Song J L, Stoeckius M, Maaskola J, et al. Select microRNAs are essential for early development in the sea urchin[J]. Dev Biol, 2012, 362(1): 104-113. DOI:10.1016/j.ydbio.2011.11.015 |
| [31] |
Stepicheva N A, Song J L. MicroRNA-31 modulates skeletal patterning in the sea urchin embryo[J]. Development, 2015, 142(21): 3769-3780. DOI:10.1242/dev.127969 |
| [32] |
Huang T, Zhang X. Functional analysis of a crustacean microRNA in host-virus interactions[J]. J Virol, 2012, 86(23): 12997-13004. DOI:10.1128/JVI.01702-12 |
| [33] |
Shu L, Zhang X. Shrimp miR-12 suppresses white spot syndrome virus infection by synchronously triggering antiviral phagocytosis and apoptosis pathways[J]. Front Immunol, 2017, 8: 855-867. DOI:10.3389/fimmu.2017.00855 |
| [34] |
Yang L, Yang G, Zhang X B. The miR-100-mediated pathway regulates apoptosis against virus infection in shrimp[J]. Fish & Shellfish Immunology, 2014, 40(1): 146-153. |
| [35] |
Zhou Y, He Y, Wang C, et al. Characterization of miRNAs from hydrothermal vent shrimp Rimicaris exoculata[J]. Mar Genomics, 2015, 24(3): 371-378. |
| [36] |
Lv J, Liu P, Gao B, et al. The identification and characteristics of salinity-related microRNAs in gills of Portunus trituberculatus[J]. Cell Stress & Chaperones, 2015, 21(1): 1-12. |
| [37] |
Li S, Zhu S, Li C, et al. Characterization of microRNAs in mud crab Scylla paramamosain under Vibrio parahaemolyticus infection[J]. PLoS One, 2013, 8(8): e73392. DOI:10.1371/journal.pone.0073392 |
| [38] |
Zhou Z, Wang L L, Song L S, et al. The identification and characteristics of immune-related microRNAs in haemocytes of oyster Crassostrea gigas[J]. Plos One, 2014, 9(2): e88397. DOI:10.1371/journal.pone.0088397 |
| [39] |
Chen H, Wang H, Jiang S, et al. An oyster species-specific miRNA scaffold42648_5080 modulates haemocyte migration by targeting integrin pathway[J]. Fish & Shellfish Immunology, 2016, 57: 160-169. |
| [40] |
Chen H, Xin L, Song X, et al. A norepinephrine-responsive miRNA directly promotes CgHSP90AA1 expression in oyster haemocytes during desiccation[J]. Fish Shellfish Immunol, 2017, 64: 297-307. DOI:10.1016/j.fsi.2017.03.020 |
| [41] |
Zhao X, Yu H, Kong L, et al. High throughput sequencing of small RNAs transcriptomes in two Crassostrea oysters identifies microRNAs involved in osmotic stress response[J]. Sci Rep, 2016, 6: 22687. DOI:10.1038/srep22687 |
| [42] |
Chen G F, Zhang C Y, Jiang F J, et al. Bioinformatics analysis of hemocyte miRNAs of scallop Chlamys farreri against acute viral necrobiotic virus (AVNV)[J]. Fish & Shellfish Immunology, 2014, 37(1): 75-86. |
| [43] |
Bao Y B, Zhang L L, Dong Y H, et al. Identification and comparative analysis of the Tegillarca granosa haemocytes microRNA transcriptome in response to Cd using a deep sequencing approach[J]. Plos One, 2014, 9(4): e93619. DOI:10.1371/journal.pone.0093619 |
| [44] |
Jiao Y, Zheng Z, Du X, et al. Identification and characterization of microRNAs in pearl oyster Pinctada martensii by Solexa deep sequencing[J]. Mar Biotechnol (NY), 2014, 16(1): 54-62. DOI:10.1007/s10126-013-9528-x |
| [45] |
Jiao Y, Zheng Z, Tian R, et al. MicroRNA, Pm-miR-2305, participates in nacre formation by targeting pearlin in pearl oyster Pinctada martensii[J]. Int J Mol Sci, 2015, 16(9): 21442-21453. DOI:10.3390/ijms160921442 |
| [46] |
Tian R R, Zheng Z, Huang R L, et al. MiR-29a participated in nacre formation and immune response by targeting Y2R in Pinctada martensii[J]. International Journal of Molecular Sciences, 2015, 16(12): 29436-29445. DOI:10.3390/ijms161226182 |
| [47] |
Zheng Z, Du X D, Xiong X W, et al. PmRunt regulated by Pm-miR-183 participates in nacre formation possibly through promoting the expression of collagen Ⅵ-like and Nacrein in pearl oyster Pinctada martensii[J]. Plos One, 2017, 12(6): e0178561. DOI:10.1371/journal.pone.0178561 |
| [48] |
Campo-Paysaa F, Semon M, Cameron R A, et al. MicroRNA complements in deuterostomes:origin and evolution of microRNAs[J]. Evolution & Development, 2011, 13(1): 15-27. |
| [49] |
Dai Z H, Chen Z Z, Ye H, et al. Characterization of microRNAs in cephalochordates reveals a correlation between microRNA repertoire homology and morphological similarity in chordate evolution[J]. Evolution & Development, 2009, 11(1): 41-49. |
| [50] |
Wheeler B M, Heimberg A M, Moy V N, et al. The deep evolution of metazoan microRNAs[J]. Evol Dev, 2009, 11(1): 50-68. DOI:10.1111/j.1525-142X.2008.00302.x |
| [51] |
Xu F, Wang X, Feng Y, et al. Identification of conserved and novel microRNAs in the Pacific oyster Crassostrea gigas by deep sequencing[J]. PLoS One, 2014, 9(8): e104371. DOI:10.1371/journal.pone.0104371 |
| [52] |
Meng X, Zhang X, Li J, et al. Identification and comparative profiling of ovarian and testicular microRNAs in the swimming crab Portunus trituberculatus[J]. Gene, 2018, 640: 6-13. DOI:10.1016/j.gene.2017.10.026 |
| [53] |
Iovino N, Pane A, Gaul U. MiR-184 has multiple roles in drosophila female germline development[J]. Developmental Cell, 2009, 17(1): 123-133. DOI:10.1016/j.devcel.2009.06.008 |
| [54] |
Wu J W, Bao J Q, Wang L, et al. MicroRNA-184 downregulates nuclear receptor corepressor 2 in mouse spermatogenesis[J]. Bmc Developmental Biology, 2011, 11(1): 1-10. DOI:10.1186/1471-213X-11-1 |
| [55] |
Sha Z, Gong G, Wang S, et al. Identification and characterization of Cynoglossus semilaevis microRNA response to Vibrio anguillarum infection through high-throughput sequencing[J]. Dev Comp Immunol, 2014, 44(1): 59-69. DOI:10.1016/j.dci.2013.11.014 |
| [56] |
Purcell M K, Smith K D, Hood L, et al. Conservation of toll-like receptor signaling pathways in teleost fish[J]. Comp Biochem Physiol Part D Genomics Proteomics, 2006, 1(1): 77-88. DOI:10.1016/j.cbd.2005.07.003 |
| [57] |
Cullen B R. MicroRNAs as mediators of viral evasion of the immune system[J]. Nature Immunology, 2013, 14(3): 205-210. DOI:10.1038/ni.2537 |
| [58] |
Zhang B C, Zhou Z J, Sun L. Pol-miR-731, a teleost miRNA upregulated by megalocytivirus, negatively regulates virus-induced type Ⅰ interferon response, apoptosis, and cell cycle arrest[J]. Sci Rep, 2016, 6: 28354. DOI:10.1038/srep28354 |
| [59] |
Hou J, Wang P, Lin L, et al. MicroRNA-146a feedback inhibits RIG-I-dependent Type Ⅰ IFN production in macrophages by targeting TRAF6, IRAK1, and IRAK2[J]. J Immunol, 2009, 183(3): 2150-2158. DOI:10.4049/jimmunol.0900707 |
| [60] |
Zhou H, Huang X, Cui H, et al. MiR-155 and its star-form partner miR-155* cooperatively regulate type Ⅰ interferon production by human plasmacytoid dendritic cells[J]. Blood, 2010, 116(26): 5885-5894. DOI:10.1182/blood-2010-04-280156 |
| [61] |
Li C, Feng W, Qiu L, et al. Characterization of skin ulceration syndrome associated microRNAs in sea cucumber Apostichopus japonicus by deep sequencing[J]. Fish Shellfish Immunol, 2012, 33(2): 436-441. DOI:10.1016/j.fsi.2012.04.013 |
| [62] |
Chen H, Jiang S, Wang L, et al. Cgi-miR-92d indirectly regulates TNF expression by targeting CDS region of lipopolysaccharide-induced TNF-alpha factor 3(CgLITAF3) in oyster Crassostrea gigas[J]. Fish Shellfish Immunol, 2016, 55: 577-584. DOI:10.1016/j.fsi.2016.06.036 |
| [63] |
Ng W L, Yan D, Zhang X, et al. Over-expression of miR-100 is responsible for the low-expression of ATM in the human glioma cell line:M059J[J]. Dna Repair, 2010, 9(11): 1170-1175. DOI:10.1016/j.dnarep.2010.08.007 |
| [64] |
Sun J, Chen Z, Tan X, et al. MicroRNA-99a/100 promotes apoptosis by targeting mTOR in human esophageal squamous cell carcinoma[J]. Medical Oncology, 2013, 30(1): 411-419. DOI:10.1007/s12032-012-0411-9 |
| [65] |
Huang T, Zhang X. Functional analysis of a crustacean microRNA in host-virus interactions[J]. J Virol, 2012, 86(23): 12997-13004. |
| [66] |
雷霁霖, 马爱军, 刘新富, 等. 大菱鲆(Scophthalmus maximus L.)胚胎及仔稚幼鱼发育研究[J]. 海洋与湖沼, 2003, 34(1): 9-18. Lei Jilin, Ma Aijun, Liu Xinfu, et al. Study on the development of embryo, larval and juvenile of turbot Scophthalmus maximus L.[J]. Oceanologia Et Limnologia Sinica, 2003, 34(1): 9-18. DOI:10.11693/hyhz200301002002 |
| [67] |
张俊玲, 施志仪. 牙鲆早期阶段的变态发育及其机制[J]. 上海海洋大学学报, 2003, 12(4): 348-352. Zhang Junling, Shi Zhiyi. Metamorphosis and mechanism during early development of Japanese flounder, Paralichthys olivaceus[J]. Journal of Shanghai Fisheries University, 2003, 12(4): 348-352. |
| [68] |
Inui Y, Miwa S. Thyroid hormone induces metamorphosis of flounder larvae[J]. General & Comparative Endocrinology, 1985, 60(3): 450. |
| [69] |
Shao C, Bao B, Xie Z, et al. The genome and transcriptome of Japanese flounder provide insights into flatfish asymmetry[J]. Nature Genetics, 2017, 49(1): 119-124. DOI:10.1038/ng.3732 |
| [70] |
Zhang H, Fu Y, Shi Z, et al. MiR-17 is involved in Japanese flounder (Paralichthys olivaceus) development by targeting the Cdc42 mRNA[J]. Comp Biochem Physiol B Biochem Mol Biol, 2016, 191: 163-170. DOI:10.1016/j.cbpb.2015.10.005 |
| [71] |
Chambon J P, Nakayama A, Takamura K, et al. ERK- and JNK-signalling regulate gene networks that stimulate metamorphosis and apoptosis in tail tissues of ascidian tadpoles[J]. Development, 2007, 134(6): 1203-1219. DOI:10.1242/dev.002220 |
| [72] |
Zhang X, Liu X, Liu C, et al. Identification and characterization of microRNAs involved in ascidian larval metamorphosis[J]. BMC Genomics, 2018, 19(1): 168. DOI:10.1186/s12864-018-4566-4 |
| [73] |
He T, Chen J, Zhang J, et al. SARP19 and vdg3 gene families are functionally related during abalone metamorphosis[J]. Development Genes and Evolution, 2014, 224(4-6): 197-207. DOI:10.1007/s00427-014-0478-8 |
| [74] |
Huang Z, Chen Z, Ke C, et al. Pyrosequencing of haliotis diversicolor transcriptomes:insights into early developmental molluscan gene expression[J]. PloS One, 2012, 7(12): e51279. DOI:10.1371/journal.pone.0051279 |
| [75] |
Williams E A, Degnan S M. Carry-over effect of larval settlement cue on postlarval gene expression in the marine gastropod Haliotis asinina[J]. Molecular Ecology, 2009, 18(21): 4434-4449. DOI:10.1111/mec.2009.18.issue-21 |
| [76] |
Qin J, Huang Z, Chen J, et al. Sequencing and de novo analysis of Crassostrea angulata (Fujian oyster) from 8 different developing phases using 454 GSFlx[J]. PloS One, 2012, 7(8): e43653. DOI:10.1371/journal.pone.0043653 |
| [77] |
Yang B, Li L, Pu F, et al. Molecular cloning of two molluscan caspases and gene functional analysis during Crassostrea angulata (Fujian oyster) larval metamorphosis[J]. Molecular Biology Reports, 2015, 42(5): 963-975. DOI:10.1007/s11033-014-3833-y |
| [78] |
Huan P, Wang H, Liu B. A label-free proteomic analysis on competent larvae and juveniles of the Pacific oyster Crassostrea gigas[J]. PloS One, 2015, 10(8): e0135008. DOI:10.1371/journal.pone.0135008 |
| [79] |
Zhang G, He L, Wong Y, et al. p38 MAPK regulates PKAα and CUB-serine protease in Amphibalanus amphitrite cyprids[J]. Scientific Reports, 2015, 5(1): 14767. |
| [80] |
Davalos A, Goedeke L, Smibert P, et al. MiR-33a/b contribute to the regulation of fatty acid metabolism and insulin signaling[J]. Proc Natl Acad Sci U S A, 2011, 108(22): 9232-9237. DOI:10.1073/pnas.1102281108 |
| [81] |
Najafi-Shoushtari S H, Kristo F, Li Y, et al. MicroRNA-33 and the SREBP host genes cooperate to control cholesterol homeostasis[J]. Science, 2010, 328(5985): 1566-1569. DOI:10.1126/science.1189123 |
| [82] |
Kovalchuk O, Zemp F, Filkowski J N, et al. MicroRNAome changes in bystander three-dimensional human tissue models suggest priming of apoptotic pathways[J]. Carcinogenesis, 2010, 31(10): 1882-1888. DOI:10.1093/carcin/bgq119 |
| [83] |
Flynt A S, Thatcher E J, Burkewitz K, et al. MiR-8 microRNAs regulate the response to osmotic stress in zebrafish embryos[J]. J Cell Biol, 2009, 185(1): 115-127. DOI:10.1083/jcb.200807026 |
| [84] |
Yan B, Guo J T, Zhao L H, et al. MiR-30c:a novel regulator of salt tolerance in tilapia[J]. Biochem Biophys Res Commun, 2012, 425(2): 315-320. DOI:10.1016/j.bbrc.2012.07.088 |
 2018, Vol. 42
2018, Vol. 42


