文章信息
- 陈招弟, 李健, 翟倩倩, 常志强, 王佳佳, 葛倩倩. 2018.
- CHEN Zhao-di, LI Jian, ZHAI Qian-qian, CHANG Zhi-qiangp>, WANG Jia-jia, GE Qian-qian. 2018.
- 水产用微生态制剂耐药性评估及耐药相关遗传元件检测
- Assessment of drug resistance and detection of resistance genes and mobile genetic elements of aquatic microecologic agents
- 海洋科学, 42(6): 132-140
- Marine Sciences, 42(6): 132-140.
- http://dx.doi.org/10.11759/hykx20180408001
-
文章历史
- 收稿日期:2018-04-08
- 修回日期:2018-06-09
2. 青岛海洋科学与技术试点国家实验室 海洋渔业科学与食物产出过程功能实验室, 山东 青岛 266071;
3. 中国水产科学研究院 黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室, 山东 青岛 266071
2. Pilot National Laboratory for Marine Science and Technology, Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao 266071, China;
3. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Maricultural Organism Disease Control, Ministry of Agriculture and Rural Affairs, Qingdao 266071, China
我国水产养殖业发展迅速, 从1990年到2016年水产品养殖年产量从9.1×106 t升至5.1×107 t [1, 2], 成为最大的水产养殖大国。但高密度集约化养殖导致环境恶化、疾病频发, 严重制约了水产养殖业的可持续健康发展。使用抗菌类药物如抗生素等传统防治措施对治疗水产动物疾病具有一定的效果, 但是随着药物的大量使用以及滥用, 致使病原菌产生耐药性, 这不仅会降低抗菌药物的药效, 而且细菌耐药基因的传播, 也会给水产养殖业和人类健康造成威胁。
为缓解以上问题, 微生态制剂(Microbial ecological agent, MEA)作为抗生素的替代品被研制并应用于水产养殖。微生态制剂是具有维持养殖动物肠道健康、提高养殖动物免疫机能、抑制有害菌生长、提高养殖动物成活率等多种功能的活菌制剂[3]。我国微生态制剂产业发展迅速, 据不完全统计, 2018年微生态制剂的年使用量已达20万~30万t。在微生态制剂大面积应用的同时, 抗生素作为辅助防治手段也会少量应用于水产养殖中, 这就存在益生菌耐药的可能。现在我国微生态制剂厂家的生产技术良莠不齐, 在筛选益生菌菌种的过程中没有统一的质量标准和相应的管理机制, 致使微生态制剂的质量安全难以保证。国内外对细菌耐药性的报道大部分集中在致病菌株上, 而对益生菌耐药性的研究仅见零星报道。
本实验采用琼脂稀释法筛选市场上购买的9种微生态制剂中的耐药菌株, 微量肉汤稀释法检测菌株对万古霉素、庆大霉素、四环素、土霉素、恩诺沙星、诺氟沙星、氟苯尼考、磺胺嘧啶、磺胺甲基嘧啶9种水产养殖常用抗生素的耐药性, 同时检测耐药基因和可移动遗传元件, 为微生态制剂的安全生产和使用提供参考。
1 材料与方法 1.1 实验材料 1.1.1 实验菌株选取市场上常见的三个厂家(简称B、G、W)的9种微生态制剂产品, 分别为枯草芽孢杆菌(简称BK)、地衣芽胞杆菌(简称BD); 解淀粉芽孢杆菌(简称GJ)、短小芽孢菌(简称GD)、粪肠球菌1(简称GF)、200亿枯草(简称GK)、水产用枯草芽孢杆菌(简称WK)、粪肠球菌2(简称WF)、乳杆菌(简称WR)。
大肠杆菌ATCC25922、金黄色葡萄球菌ATC C 29 213、粪肠球菌ATCC29212三种标准质控菌株购自于北纳创联生物技术有限公司。
1.1.2 实验药品9种抗生素分别购自北京索莱宝科技有限公司(诺氟沙星(100%)、四环素(≥98%)、土霉素(≥99%)、庆大霉素(≥590 μg/mg))、大连美仑生物技术有限公司(万古霉素(> 90%))、上海阿拉丁生化科技股份有限公司(磺胺甲基嘧啶(> 99%)、恩诺沙星(100%)、氟苯尼考(> 98%)、磺胺嘧啶(> 98%))。
1.2 实验方法 1.2.1 耐药菌株的分离、纯化微生态制剂中耐药菌株的分离遵照CALVA等[4]提供的方法, 筛选微生态制剂中对万古霉素、庆大霉素和四环素的抗性菌株, 将抗性平板上生长的菌株挑取单菌落于液体培养基中, 培养(37℃, 200 r/min)16~20 h划线纯化后富集培养, 保存菌种备用。
1.2.2 DNA和质粒提取分别按细菌基因组DNA提取试剂盒和质粒小提试剂盒的说明书提取基因组DNA和质粒。
1.2.3 菌株鉴定以基因组DNA为模板, 进行16S rRNA的PCR扩增, 扩增产物经琼脂糖凝胶电泳20 min后用凝胶成像仪成像分析, 选取条带明亮的样品送样至青岛睿博兴科进行测序, 测序结果在NCBI(http://www.ncbi.nlm.nih.gov)上进行同源比对, 结合生化鉴定确定其种类。所有引物序列及反应条件见表 1。
| 引物 | 引物序列(5′-3′) | 长度/bp | 反应条件 |
| 16S rRNA-F 16S rRNA-R |
AGAGTTTGATCCTGGCTCAG GGCTACCTTGTTACGACTT |
1500 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| 16S-23S rRNA-F 16S-23S rRNA-R |
CAAGGCATCCACCGT GTGAAGTCGTAACAAGG |
300/400 | 94℃ 5 min, (94℃ 45 s, 54℃ 30 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| tetA-F tetA-R |
GCTACATCCTGCTTGCCTTC GCATAGATCGCCGTGAAGAG |
211 | 94℃ 5 min, (94℃ 45 s, 60℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| tetB-F tetB-R |
TACGTGAATTTATTGCTTCGG ATACAGCATCCAAAGCGCAC |
206 | 94℃ 5 min, (94℃ 45 s, 60℃ 1 min, 72℃ 45 s) 35个循环, 72℃ 10 min |
| tetC-F tetC-R |
CTGCTCGCTTCGCTACTTG GCCTACAATCCATGCCAACC |
897 | 94℃ 5 min, (94℃ 45 s, 60℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| tetD-F tetD-R |
TGTGCTGTGGATGTTGTATCTC CAGTGCCGTGCCAATCAG |
844 | 94℃ 5 min, (94℃ 45 s, 58℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| tetE-F tetE-R |
ATGAACCGCACTGTGATGATG ACCGACCATTACGCCATCC |
744 | 94℃ 5 min, (94℃ 45 s, 58℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| tetM-F tetM-R |
ACAGAAAGCTTATTATATAAC TGGCGTGTCTATGATGTTCAC |
171 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| vanA-F vanA-R |
GCTATTCAGCTGTACTC CAGCGGCCATCATACGG |
783 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| vanB-F vanB-R |
CATCGCCGTCCCCGAATTTCAAA GATGCGGAAGATACCGTGGCT |
667 | 94℃ 5 min, (94℃ 45 s, 55℃ 35 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| VanC1-F VanC1-R |
GGTATCAAGGAAACCTC CTTCCGCCATCATAGCT |
822 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| qnrA-F qnrA-R |
TTCAGCAAGAGGATTTCTCA GGCAGCACTATTACTCCCAA |
500 | 94℃ 5 min, (94℃ 45 s, 55℃ 35 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| qnrB-F qnrB-R |
CCTGAGCGGCACTGAATTTAT GTTTGCTGCTCGCCAGTCGA |
617 | 94℃ 5 min, (94℃ 45 s, 55℃ 35 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| qnrS-F qnrS-R |
ACATAAAGACTTAAGTGATC CAATTAGTCAGGATAAAC |
619 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| qepA-F qep A-R |
CCAGCTCGGCAACTTGATAC ATGCTCGCCTTCCAGAAAA |
570 | 94℃ 5 min, (94℃ 45 s, 60℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| aac(6’)-Ib-F aac(6’)-Ib-R |
TTGCGATGCTCTATGAGTGGCTA CTCGAATGCCTGGCGTGTTT |
482 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| ant(3’)-Ia(aadA)-F ant(3’)-Ia(aadA)-R |
ATCTGGCTATCTTGCTGACA TTGGTGATCTCGCCTTTC |
388 | 94℃ 5 min, (94℃ 45 s, 57℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| aph(6’)-Id(strB)-F aph(6’)-Id(strB)-R |
ATGTTCATGCCGCCTGTTTTT CTAGTATGACGTCTGTCGC |
837 | 94℃ 5 min, (94℃ 45 s, 54℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| floR-F floR-R |
CTGCTGATGGCTCCTTTC GCCGTGGCGTAACAGAT |
650 | 94℃ 5 min, (94℃ 45 s, 57℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| cfr-F cfr-R |
TGAAGTATAAAGCAGGTTGGGAGTCA ACCATATAATTGACCACAAGCAGC |
746 | 94℃ 5 min, (94℃ 45 s, 59℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| Sul1-F Sul1-R |
GTGACGGTGTTCGGCATTCT TCCGAGAAGGTGATTGCGCT |
800 | 94℃ 5 min, (94℃ 45 s, 58℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| Sul2-F Sul2-R |
CATCATTTTCGGCATCGTC TCTTGCGGTTTCTTTCAGC |
793 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| Sul3-F Sul3-R |
GCAACAGTTGGTGCTAAACGAGA AGCAGATGTGATTGATTTGGGAG |
578 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| int1-F int1-R |
GTTCGGTCAAGGTTCTGG CGTAGAGACGTCGGAATG |
890 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| int2-F int2-R |
CAAGCATCTCTAGGCGTA AGTAGCATCAGTCAATCC |
1100 | 94℃ 5 min, (94℃ 45 s, 53℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| int3-F int3-R |
CATCAAGCTGCTCGATCA ACAACTCTTGCACCGTTC |
900 | 94℃ 5 min, (94℃ 45 s, 53℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| SXT-F SXT-R |
CTGTGGCCAATCATCAACTC CGACCGAGATGGGCTAAGTG |
1030 | 94℃ 5 min, (94℃ 45 s, 57℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| qacEΔ1-F qacEΔ1-R |
ATCGCAATAGTTGGCGAAGT CAAGCTTTTGCCCATGAAGC |
200 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| Hep58 Hep59 |
TCATGGCTTGTTATGACTGT GTAGGGCTTATTATGCACGC |
未知 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 1 min) 35个循环, 72℃ 10 min |
| ISCR-VF ISCR-VR |
CGAACACTGCGGCGGTCA TTCAGAATGCCGAACACC |
1400 | 94℃ 5 min, (94℃ 45 s, 60℃ 45 s, 72℃ 1 min) 35个循环, 72℃ 10 min |
| intTn916/Tn1514-F intTn916/Tn1514-R |
CATCAGACGCTAAAGAATGG GGCTTCTTCAACCATAGGAA |
452 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| Tnp513-F Tnp513-R |
ATGTCGCTGGCAAGGAACGC GGGTTCGCTGCGAGGATTGT |
240 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| ISaba1-F ISaba1-R |
AATGATTGGTGACAATGAAG ATGCAGCGCTTCTTTGCAGG |
372 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| IS26-F IS26-R |
ATGAACCCATTCAAAGGCCGGCAT CCGTCGTAACAGCAAAGCTGCATA |
387 | 94℃ 5 min, (94℃ 45 s, 55℃ 45 s, 72℃ 45 s) 35个循环, 72℃ 10 min |
| TraA-F TraA-R |
AAGTGTTCAGGGTGCTTCTGCGC GTCATGTACATGATGACCATTT |
272 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 45 s) 35个循环, 72℃ 10 min |
| TrbC-F TrbC-R |
CGGYATWCCGSCSACRCTGCG GCCACCTGYSBGCAGTCMCC |
255 | 94℃ 5 min, (94℃ 45 s, 55℃ 1 min, 72℃ 45 s) 35个循环, 72℃ 10 min |
按照美国临床与实验室标准协会(CLSI)规定的微量肉汤稀释法的操作方法[5], 以大肠杆菌ATCC25922、金黄色葡萄球菌ATCC29213和粪肠球菌ATCC29212为标准菌株, 对分离菌株进行9种抗生素的药敏试验, 庆大霉素、万古霉素、四环素、土霉素、诺氟沙星、恩诺沙星、氟苯尼考、磺胺嘧啶和磺胺甲基嘧啶的最高浓度分别为512、128、64、256、64、64、512、4096、4096 μg/mL。每组设置3个重复。结果跟据CLSI2014版中的判定标准进行统计, 对于缺乏敏感、中介、耐药范围的菌株参考其标准质控菌株。判定标准如表 2所示。
| 抗菌药物 | 敏感(S) | 中介(I) | 耐药(R) |
| 庆大霉素 | ≤4 | 8 | ≥16 |
| 万古霉素 | ≤4/≤2 | 8~16/4~8 | ≥32/≥16 |
| 四环素 | ≤4 | 8 | ≥16 |
| 氟苯尼考 | ≤2 | 4 | ≥8 |
| 土霉素 | ≤4 | 8 | ≥16 |
| 恩诺沙星 | ≤4 | 8 | ≥16 |
| 诺氟沙星 | ≤4 | 8 | ≥16 |
| 磺胺嘧啶 | ≤256 | - | ≥512 |
| 磺胺甲基嘧啶 | ≤256 | - | ≥512 |
| 注: “-”代表CLSI上无相应的判定标准; “/”前代表肠球菌、乳杆菌、芽孢杆菌对万古霉素的敏感、中介和耐药值; “/”后代表其他菌株对万古霉素的敏感、中介和耐药值。 | |||
以提取的细菌基因组DNA为模板, 采用PCR方法对氨基糖苷类(ant(3’)-Ia(aadA)、aph(6’)-Id(strB))、氟喹诺酮类(qnrA、qnrB、qnrS、qepA、aac(6’)-Ib-cr)、酰胺醇类(floR、cfr)、磺胺类(Sul1、Sul2、Sul3)、四环素类(tetA、tetB、tetC、tetD、tetE、tet M)和糖肽类(vanA、vanB、vanC1)耐药基因进行检测。
1.2.6 可转移原件检测以质粒DNA为模板扩增接合性质粒遗传标记traA和trbC基因; 以提取的细菌基因组DNA为模板, 对转座子intTn916/Tn1514、Tnp513和插入序列ISaba1、IS26、ISCR进行扩增, 同时检测5′CS保守区(5′-conserved segment, 5′-CS)的1-4类整合子包含的整合酶基因: int1、int2、int3和SXT, 3′CS保守区(3′-conserved segment, 3′-CS)的Sul1和qacEΔ1基因, 以及1类整合子的可变区(引物为Hep58、Hep59)。
2 结果 2.1 微生态制剂耐药菌株分离鉴定实验发现大肠杆菌ATCC25922、金黄色葡萄球菌ATCC29213、粪肠球菌ATTCC29212在庆大霉素、万古霉素和四环素抗性平板上不生长的最小浓度分别为8、256、16 μg/mL; 2、1、0.5 μg/mL; 32、8、64 μg/mL。分别以上述浓度筛选乳杆菌、芽孢杆菌和肠球菌对三种抗生素的耐药菌株, 共分离纯化出100株耐药菌(表 3), 16S rRNA PCR均扩增到1500 bp左右的目的条带(图 1)。比对分析时发现有12株菌与粪肠球菌(Enterococcus faecalis)和屎肠球菌(Enterococcus faecium)同源性相同, 此时采用16S-23S rRNA间区序列多态性分析技术[6]将其继续区分。
| 菌株种类 | B | G | W | 总计 | |
| 芽孢杆菌属 | 蜡样芽孢杆菌Bacillus cereus | 4 | 10 | 0 | 14 |
| 地衣芽胞杆菌Bacillus licheniformis | 4 | 4 | 2 | 10 | |
| 枯草芽孢杆菌Bacillus subtilis | 2 | 4 | 4 | 10 | |
| 甲基营养性芽孢杆菌Bacillus methylotrophicus | 1 | 1 | 1 | 3 | |
| 贝莱斯芽孢杆菌Bacillus velezensis | 8 | 8 | 0 | 16 | |
| Bacillus tequilensis | 1 | 2 | 1 | 4 | |
| 肠球菌属 | 粪肠球菌Enterococcus faecalis | 3 | 0 | 2 | 5 |
| 屎肠球菌Enterococcus faecium | 3 | 3 | 11 | 17 | |
| 鹑鸡肠球菌Enterococcus gallinarum | 0 | 0 | 1 | 1 | |
| 乳杆菌属 | 鼠李糖乳杆菌Lactobacillus rhamnosus | 0 | 0 | 3 | 3 |
| 副干酪乳杆菌Lactobacillus paracasei | 0 | 0 | 2 | 2 | |
| 葡萄球菌属 | 金黄色葡萄球菌Staphylococcus aureus | 1 | 1 | 0 | 2 |
| 埃希氏菌属 | 弗格森埃希菌Escherichia fergusonii | 0 | 0 | 5 | 5 |
| 志贺菌属 | 福氏志贺菌Shigella flexneri | 0 | 7 | 0 | 7 |
| 不动菌属 | 抗辐射不动杆菌Acinetobacter radioresistens | 0 | 0 | 1 | 1 |
| 总计 | 27 | 40 | 33 | 100 | |
 |
| 图 1 16S rRNA(左)和16S-23S rRNA(右)基因扩增的产物 Fig. 1 The PCR product of 16S rRNA(left) and 16S-23S rRNA(right) M: Maker; 1-6:部分菌株的16S rRNA和16S-23S rRNA M: Maker; 1-6: 16s rRNA and 16S-23S rRNA of some strains |
100株菌株对氟苯尼考(100%)、磺胺嘧啶(99%)、磺胺甲基嘧啶(99%)、土霉素(79%)、四环素(38%)、庆大霉素(38%)、万古霉素(18%)和诺氟沙星(15%)都存在不同程度的耐药; 但对恩诺沙星的耐药率为0, 其中敏感菌株占94%, 处于中介的菌株占6%(表 4)。100株耐药菌株中多重耐药菌株占99%, 最低的为3重耐药, 最高的为8重耐药, 4重以上耐药率达86%(图 2)。
| 抗菌药物 | 敏感(S) | 中介(I) | 耐药(R) | |||||
| 菌株数量/株 | 比率/% | 菌株数量/株 | 比率/% | 菌株数量/株 | 比率/% | |||
| 庆大霉素 | 20 | 20 | 42 | 42 | 38 | 38 | ||
| 万古霉素 | 81 | 81 | 1 | 1 | 18 | 18 | ||
| 四环素 | 58 | 58 | 4 | 4 | 38 | 38 | ||
| 诺氟沙星 | 81 | 81 | 4 | 4 | 15 | 15 | ||
| 氟苯尼考 | 0 | 0 | 0 | 0 | 100 | 100 | ||
| 土霉素 | 11 | 11 | 10 | 10 | 79 | 79 | ||
| 恩诺沙星 | 94 | 94 | 6 | 6 | 0 | 0 | ||
| 磺胺甲基嘧啶 | 1 | 1 | 0 | 0 | 99 | 99 | ||
| 磺胺嘧啶 | 1 | 1 | 0 | 0 | 99 | 99 | ||
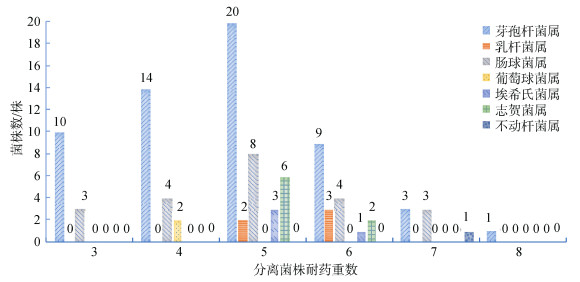 |
| 图 2 不同种类菌株的耐药重数 Fig. 2 The multi-drug resistance of different bacterial species |
对100株分离菌株进行耐药基因扩增发现, qepA、aac(6’)-Ib-cr、cfr、sul1、tetA、tetC、tetM、ant(3’)- Ia(aadA)、aph(6’)-Id(strB)、vanC1的阳性率分别9%、90%、98%、89%、81%、36%、98%、23%、68%、75%, 其他基因检测阴性。46%的株菌同时携带氟喹诺酮类、酰胺醇类、磺胺类、四环素类、氨基糖胺类和糖肽类耐药基因。
2.4 可转移原件检测100株菌中整合子int1的阳性率为91%, 未检测到int2、int3和SXT等整合子, 有83株菌同携带int1、qacEΔ1和Sul1基因, 具有完整的整合子保守区(表 5)。具有完整整合子的菌株中有78株携带二氢叶酸还原酶(dihydrofolate reductase, dfrA15)和/或氨基糖苷类-3’-磷酸转移酶(aminoglycoside-3′-ade nyly ltra nsferase, aadA1)耐药基因盒, 分别介导甲氧苄啶和氨基糖苷类耐药。
| int1 | int2 | int3 | qacEΔ1 | Sul1 | SXT | |
| 数量 | 91 | 0 | 0 | 100 | 89 | 0 |
| 阳性率/% | 91 | 0 | 0 | 100 | 89 | 0 |
此外研究发现细菌接合性质粒遗传标记基因traA的阳性率为23%, 未检测到trbC基因; 转座子intTn916/Tn1514的阳性率为100%, 插入序列ISaba1的阳性率为78%, Tnp513、IS26和ISCR基因全部为阴性。分离菌株中有15株菌同时携带整合子-基因盒、质粒、转座子和插入序列, 携带两种及两种以上可移动遗传原件的比例为95%。
3 讨论 3.1 微生态制耐药菌株分离鉴定以标准质控菌株在抗生素抗性平板上不生长的最小浓度, 作为微生态制剂抗性菌株的筛选浓度, 用该种方法筛选得到的菌株是疑似耐药的菌株, 经过后续的药敏试验和耐药基因检测进一步确定耐药性。实验从3个微生态制剂厂家的9种微生态制剂产品中都分离到了耐药菌株。耐药菌中芽孢杆菌所占比例最大为57%, 其次为肠球菌, 占23%。值得注意的是100株分离菌中益生菌有85株, 而在我国农业部2013年规定的饲用微生物菌种名单[7]之内的仅有44株, 不足分离株的50%, 此外还分离到15株致病菌株, 分别为金黄色葡萄球菌、弗格森埃希菌、福氏志贺菌和抗辐射不动杆菌, 致病菌所占的比例为15%。金黄色葡萄球菌可引起人和动物的化脓感染, 美国食品药物管理局对进口水产品中的金黄色葡萄球菌有一定的限量指标, 欧盟(EC)466-2001指令更是要求水产品中禁止检出金黄色葡萄球菌[8]; 弗格森埃希菌可引起人畜共患菌血症、腹泻、伤口及尿道感染等; 福氏志贺菌可引起肠道感染, 也可使人畜共患细菌性痢疾; 抗辐射不动杆菌为一种条件致病菌, 不容易被杀死, 可引起多种组织损伤。耐药益生菌可能成为抗生素抗性基因的储藏库, 使耐药基因在益生菌和致病菌之间传播[9]; 致病菌的存在也会影响养殖动物的健康, 水产品携带的人畜共患菌更会给人类的健康造成巨大威胁。
3.2 微生态制剂耐药性水产用微生态制剂大都分离自健康动物的肠道和养殖水体, 养殖过程中益生菌和抗生素的联合应用, 致使耐药菌的数量逐渐增多, 分离益生菌技术不规范或者缺乏耐药菌的筛选等环节, 就会大大降低微生态制剂的产品质量。药敏实验发现耐药菌株对除恩诺沙星外的8种抗生素都有不同程度的耐药表型, 且有46%的菌株同时存在六大类抗生素抗性基因, 恩诺沙星耐药表型低可能与恩诺沙星原代药与其代谢产物环丙沙星在生物体内共同发挥作用有关[10]。Cataloluk等[11]从不同来源的乳制品中分离到195株乳杆菌, 发现有91株菌分别携带或同时携带红霉素耐药基因ermB和四环素耐药基因tetM; Temmerman等[12]从欧洲55种益生菌产品中分离到187株益生菌, 发现其对四环素、万古霉素、卡那霉素等多种抗生素都有耐药性, 且多重耐药率为68.4%。以上研究结果与本实验结果相似, 但是针对食品和保健领域微生态制剂耐药性的研究, 目前在水产养殖领域未见相关性报道。水产养殖环境中益生菌耐药性的研究仅见零星报道, 但耐药性结果略低于本实验结果。贺刚等[13]从草鱼肠道分离到一株枯草芽孢杆菌, 发现其对磺胺类和土霉素药物具有较强的耐药性, 但对氟苯尼考、诺氟沙星、四环素和庆大霉素高度敏感; 高盼盼等[14]从天津6个养殖场的底泥和水样中采集样品, 用抗性平板筛选磺胺甲恶唑耐药菌株, 发现筛选的耐药菌株中芽孢杆菌属所占比例最大, 为63.27%, 肠球菌属占2.04%。水产养殖领域存在耐药益生菌, 耐药益生菌如若成为微生态制剂菌种资源, 势必会给水产养殖业带来巨大威胁。在微生态制剂广泛大量应用于水产、畜禽、医药、食品等领域的今天, 加强益生菌耐药性监测对微生态制剂产业的发展以及人类的健康具有重要意义。
3.3 微生态制剂可移动遗传元件检测获得性耐药又叫做耐药基因的水平转移, 是细菌产生耐药的主要方式。细菌利用该种方式借助质粒、整合子、转座子和插入序列等遗传元件实现耐药基因在同种细菌乃至不同种细菌之间的传播。traA和trbC接合性质粒遗传标记基因是细菌性毛的编码基因, 具备该基因说明细菌具备接合的能力。国内外已有从肺炎克雷伯菌(Klebsiella pneumoniae)和大肠杆菌(Escherichia coli)中检出traA和trbC的报道[15-16], 本实验traA的检出率为23%, 全部来源于芽孢杆菌属和肠球菌属。转座子是一种可携带多种耐药基因且极易改变自身座位的DNA片段, 插入序列为一种简单的转座子。陈文标等[17]从临床中分离到45株肺炎链球菌(Streptococcus pneumoniae), 发现其对多种抗生素都存在抗性, 且intTn916/Tn1514转座酶的检出率为91%;苏兆亮等[18]检测了19株多药耐药鲍氏不动杆菌(Acinetobacter baumannii), 其中ISaba1、IS26、tnp513都有不同程度的检出; Ruzin等[19]研究发现ISaba1插入鲍氏不动杆菌adeABC外排泵编码基因之前可致外排泵高表达而耐多种药。本实验转座子intTn916/Tn1514和插入序列ISaba1的阳性率分别为100%和78%, 且菌株4重以上耐药率为85.3%, 与之前研究结果相似。整合子极易捕获和切除耐药基因盒, 在介导细菌多重耐药的同时使耐药基因易发生传播转移。Ndi等[20]研究发现从鳟鱼养殖环境中分离到Ⅰ类整合子阳性菌株与鱼源Ⅰ类整合子同源性较高; 冯永永等[21]从猪-鱼复合养殖场分离了317株气单胞菌(Aeromonas), 发现携带Ⅰ类整合子的50株菌中存在16种耐药基因盒, 携带耐药基因盒的菌株多重耐药性较高。本实验携带耐药基因盒的菌株中五重及五重以上耐药菌株占65.8%, 与冯永永等研究结果相似。微生态制剂中耐药菌株及可转移遗传元件的检出, 提示我们益生菌耐药性问题不容忽视, 相关部门在检测致病菌耐药性及可转移遗传元件的同时要加强对微生态制剂益生菌的检测, 促进微生态制市场的健康发展。
| [1] |
FAO. The state of world fisheries and aquaculture: Opportunities and challenges[J]. State of World Fisheries & Aquaculture, 2014, 4(78): 40-41. |
| [2] |
郭云峰, 赵文武, 高宏泉, 等. 中国渔业统计年鉴[M]. 北京: 中国农业出版社, 2017: 1-143. Guo Yunfeng, Zhao Wenwu, Gao Hongquan, et al. China Fishery Statistics Yearbook[M]. Beijing: China Agriculture Press, 2017: 1-143. |
| [3] |
夏蒙, 李朋富. 微生态制剂作用机理及其在水产养殖中的应用[J]. 江西水产科技, 2013, 2: 42-44. Xia Meng, Li Pengfu. The mechanism of microbial ecological agent and its application in aquaculture[J]. Jiangxi Fishery Sciences and Technology, 2013, 2: 42-44. DOI:10.3969/j.issn.1006-3188.2013.02.016 |
| [4] |
Calva J J, Sifuentes-osornio J, Ceron C. Antimicrobial Resistance in Fecal Flora: Longitudinal Community-Based Surveillance of Children from Urban Mexico[J]. Antimicrobial Agents & Chemotherapy, 1996, 40(7): 1699-702. |
| [5] |
Clinical and Laboratory Standards Institute. Reference method for broth dilution antifungal susceptibility testing Of yeasts; approved standard M27-A3, 3rd ed[S]. Clinical and Laboratory Standards Institute, Wayne, PA, 2007.
|
| [6] |
李梅花, 王芳, 孙婷, 等. 自然发酵乳中粪肠球菌和屎肠球菌的4种鉴定方法的比较[J]. 食品与发酵工业, 2011, 37(9): 98-103. Li Meihua, Wang Fang, Sun Ting, et al. Comparison of four methods for differentiation of Enterococcus faecalis and Enterococcus faecium[J]. Food and Fermentation Industries, 2011, 37(9): 98-103. |
| [7] |
吕永辉, 李明爽. 我国水产养殖用微生态制剂行业现状与发展策略[J]. 中国水产, 2015, 8: 34-36. Lü Yonghui, Li Mingshuang. Current situation and development strategy of microbial ecological agent industry for aquaculture in China[J]. China Fisheries, 2015, 8: 34-36. |
| [8] |
范韩生. 世界水产品市场竞争激烈中国产品路在何方[J]. 中国检验检疫, 2003, 5: 45-47. Fan Hansheng. The world's aquatic product market is fiercely competitive and where is the road of Chinese products[J]. China Inspection and Quarantine, 2003, 5: 45-47. |
| [9] |
Salyers A A, Gupta A, Wang Y P. Human intestinal bacteria as reservoirs for antibiotic resistance genes[J]. Trends In Microbiology, 2004, 12(9): 412-416. DOI:10.1016/j.tim.2004.07.004 |
| [10] |
郭娇娇, 宫智勇. 恩诺沙星在水产养殖中的研究进展[J]. 江苏农业科学, 2011, 39(4): 290-292. Guo Jiaojiao, Gong Zhiyong. Research progress of enrofloxacin in aquaculture[J]. Jiangsu Agricultural Sciences, 2011, 39(4): 290-292. DOI:10.3969/j.issn.1002-1302.2011.04.119 |
| [11] |
Cataloluk O, Gogebaken B. Presence of drug resistance in intestinal lactobacilli of dairy and human origin in Turkey[J]. FEM Smicrobiology letters, 2004, 236(1): 7-12. DOI:10.1111/fml.2004.236.issue-1 |
| [12] |
Temmerman R, Pot B, Huys G, et al. Identification andantibiotic susceptibility of bacterial isolates from probiotic products[J]. International Journal of Food Microbiology, 2003, 81(1): 1-10. DOI:10.1016/S0168-1605(02)00162-9 |
| [13] |
贺刚, 何力, 谢从新, 等. 草鱼肠道枯草芽孢杆菌的耐药性分析[J]. 现代农业科技, 2008, 22: 219-222. He Gang, He Li, Xie Congxin, et al. Analysis of drug resistance of Bacillus subtilis in the intestinal tract of grass carp[J]. Modern Agricultural Science and Technology, 2008, 22: 219-222. |
| [14] |
高盼盼, 罗义, 毛大庆. 天津水产业磺胺类耐药细菌及其分布[J]. 生态毒理学报, 2011, 6(1): 74-79. Gao Panpan, Luo Yi, Mao Daqing. Sulfonamide resistant bacteria in tianjin's aquaculture and their distribution[J]. Asian Journal of Ecotoxicology, 2011, 6(1): 74-79. |
| [15] |
高慧, 蔡培泉, 郁震. 泛耐药肺炎克雷伯菌β-内酰胺类、氨基糖苷类、喹诺酮类耐药基因研究分析[J]. 中华医院感染学杂志, 2016, 26(21): 4805-4808. Gao Hui, Cai Peiquan, Yu Zhen. Drug resistance genes in pandrug-resistant Klebsiella pneumoniae to β-lacta ms, aminoglycosides, and quinolones[J]. Chinese Jo urnal of Nosocomiology, 2016, 26(21): 4805-4808. |
| [16] |
Fricke W F, Wright M S, Lindell A H, et al. Insights into the Environmental Resistance Gene Pool from the Genome Sequence of the Multidrug-Resistant Environmental Isolate Escherichia coli SMS-3-5[J]. Journal of Bacteriology, 2008, 190(20): 6779-6794. DOI:10.1128/JB.00661-08 |
| [17] |
陈文标, 朱焱, 黄东红, 等. 临床分离的肺炎链球菌耐药性分析及多位点序列分型[J]. 中国抗生素杂志, 2016, 41(1): 60-63. Chen Wenbiao, Zhu Yan, Huang Donghong, et al. Antibiotic resistance analysis and multi-locus sequence type of Streptococcus pneumoniae isolated from clinic[J]. Chinese Journal of Antibiotics, 2016, 41(1): 60-63. DOI:10.3969/j.issn.1001-8689.2016.01.009 |
| [18] |
苏兆亮, 糜祖煌, 孙光明, 等. 多药耐药鲍氏不动杆菌耐药性与转座子及插入序列遗传标记研究[J]. 中华医院感染学杂志, 2010, 20(20): 3085-3087. Su Zhaoliang, Mi Zuhuang, Sun Guangming, et al. Drug resistant and transposon and insertion sequence of multi-drug resistent Acinetobacter baumannii[J]. Chinese Journal of Nosocomiology, 2010, 20(20): 3085-3087. |
| [19] |
Ruzin A, Keeney D, Bradford. AdeABC multidrug efflux pumpis associated with decreased susceptibility to tigecycline in Acinetobacter calcoaceticus-Acine toba cter baumannii complex[J]. J Antimicrobial Chemother, 2007, 59(5): 1001-1004. DOI:10.1093/jac/dkm058 |
| [20] |
Ndi O L, Barton M D. Incidence of class 1 integron and other antibiotic resistance determinants in Aeromonas spp[J]. Journal of Fish Diseases, 2011, 34(8): 589-599. DOI:10.1111/jfd.2011.34.issue-8 |
| [21] |
冯永永, 姜兰, 邓玉婷, 等. 猪-鱼复合养殖模式中气单胞菌类整合子的流行情况及其耐药特征[J]. 水产学报, 2016, 40(1): 92-99. Feng Yongyong, Jiang Lan, Deng Yuting, et al. Prevalence and characterization of class Ⅰ integron in Aeromonas from pig-fish integrated farm[J]. Journal of Fisheries of China, 2016, 40(1): 92-99. |
 2018, Vol. 42
2018, Vol. 42


