
文章信息
- 刘昊昕, Sadaf Riaz, 刘源, Anam Khurshid, 张巍, 隋正红. 2019.
- LIU Hao-xin, Sadaf RIAZ, LIU Yuan, Anam KHURSHID, ZHANG Wei, SUI Zheng-hong. 2019.
- 链状亚历山大藻组蛋白H3基因克隆与生长过程中的表达分析
- Cloning and expression analysis of histone H3 in Alexandriumcatenella during the growth process
- 海洋科学, 43(11): 19-27
- Marine Sciences, 43(11): 19-27.
- http://dx.doi.org/10.11759/hykx20190401001
-
文章历史
- 收稿日期:2019-04-01
- 修回日期:2019-05-05
链状亚历山大藻(Alexandriumcatenella)是亚历山大藻属的代表物种之一, 能够产生麻痹性贝毒, 曾在世界各地引发赤潮, 是一种典型的产毒赤潮甲藻[1]。赤潮甲藻在爆发性生长过程中的分子调控机制一直是研究的热点。
大多数真核生物中, 组蛋白与基因组DNA动态结合构成染色质, 核小体是其基本单位, 由H2A、H2B、H3和H4四种组蛋白二聚体形成八聚体结构。不同于此, 甲藻拥有十分独特的核结构, 其基因组巨大, DNA呈液晶态, 染色体永久浓缩[2], 其DNA与蛋白的比例约为10︰1, 显著高于其他真核生物接近1︰1的比例[3-4]。早期研究中, 甲藻一度被认为没有组蛋白, 或在进化过程中丢失了组蛋白[5]。目前已在多种甲藻中检测到全部四个核心组蛋白的转录[6-10], 但多种证据显示组蛋白并不像在其他生物中一样主要参与核小体组装[11], 对于组蛋白在甲藻中的表达模式与具体功能还缺乏深入的研究。
核心组蛋白可进行乙酰化、甲基化、磷酸化以及泛素化等组蛋白翻译后修饰(HPTM)。核心组蛋白中, 组蛋白H3序列的种间保守性最强, 其N端序列包含最多的共价修饰位点, 与H4一同作为蛋白酶修饰的主要作用蛋白[12]。作为一类重要的表观遗传调控机制, HPTM可以灵活标记染色质, 通过招募效应蛋白、引起染色质重组、改变染色质结构等方式, 最终激活或抑制转录过程, 在动植物生长发育、响应逆境胁迫等生理过程中发挥调控功能[13-17]。除了作为包装DNA的结构蛋白, 组蛋白也可演化为多种变体, 可产生具有表观遗传学效果的染色质分化, 目前已知的组蛋白变体主要发生在组蛋白H3和H2A上。H3变体主要包括CENP-A和H3.1、H3.2、H3.3等, CENP-A是组蛋白H3在染色体着丝粒部位的特征变体[18]; H3.1的沉积与基因沉默有关, 而H3.3存在于转录活跃区域。组蛋白加以替换的同时, 也会导致翻译后修饰的擦除或改变[13], 例如携带H3乙酰化和H3K4甲基化的H3.3沉积在转录活性基因上, 形成稳定的标记[19]。
鉴于组蛋白H3对生物生长调控的重要作用, 本文报道了链状亚历山大藻组蛋白H3变体基因的克隆与序列分析; 并在链状亚历山大藻不同生长时期取样, 对比分析链状亚历山大藻爆发性生长至衰亡过程中组蛋白H3基因在转录水平的表达, 探讨组蛋白H3在链状亚历山大藻生长过程中的表达模式及可能的功能。
1 材料与方法 1.1 材料培养与收集链状亚历山大藻(Alexandrium catenella)藻株来源于中国海洋大学海洋生物遗传与育种教育部重点实验室。具体培养条件如下:培养基: f/2培养基(不加硅); 光照强度: 30 μmol m–2 s–1; 温度: 20±1℃; 光暗周期: 12h︰12h。
链状亚历山大藻于f/2培养基中培养至对数生长期, 以2×106个/L的浓度将藻接种于新的f/2培养基, 共设4个实验组, 分别于链状亚历山大藻生长延滞期(第3天)、对数期前期(第9天)、对数期末期(第15天)和衰亡期(第30天)取样, 每组设置三个生物学平行; 另设对照组以相同条件培养, 隔天计数。藻细胞收集方法:在20℃条件下, 用经过无菌处理的10 μm孔径筛绢过滤藻液, 收集至1.5 mL离心管中, 保存于–80℃冰箱。
1.2 RNA提取与cDNA合成使用RNAiso Plus (Trizol) (TaKaRa)方法提取RNA, 琼脂糖凝胶电泳检测条带情况, Nano Drop 2 000(Thermo)测定RNA浓度与纯度;
取1 μg高质量的RNA(OD 260/280, 1.9-2.1)用于cDNA合成(Takara PrimeScriptTM RT reagent Kit with gDNA Eraser), 存储于–20℃冰箱备用。
1.3 目的基因克隆及生物信息学分析根据实验室已有的链状亚历山大藻转录组数据(SRX368254)的注释结果, 从中挑选注释为组蛋白H3及其变体的基因序列, 使用Primer Premier 5.0软件设计引物, 详细信息如表 1所示。PCR反应体系20μL, 包含正向和反向引物(10 μM, 各0.5 μL), 模板cDNA(10 ng), PCR预混液(UniqueTMTaq Master Mix, 10μL)。按照以下程序运行: 94℃ 10 min; 94℃ 30s; 65℃ 30 s; 72℃ 2 min; 返回至第二步循环30次; 72℃ 10 min。回收目的条带后, 以PMD19-T为连接载体进行克隆, 挑取阳性菌落进行PCR验证后, 送青岛擎科梓熙生物技术有限公司测序。
| 基因名 | 转录组数据库(SRX368254)中的名称 | 引物序列(5′-3′) | 产物(bp) |
| H3.c | Algae_064-2_Unigene_BMK.1001 | TGGAGGAGGCAGCCTCCCGATG/ CGCCCCTCAGACGCGCTCAGAT | 446 |
使用DNAMAN软件进行不同物种组蛋白H3及变体的氨基酸序列比对。使用在线工具Protparam预测蛋白的理化性质; 使用Predictprotein预测蛋白的二级结构; 使用MEGA 5.0软件, 采用clustalW法对不同物种的氨基酸序列进行同源比对, 以最大似然法(ML), Bootstrap重复次数1 200, 构建系统进化树。
1.4 实时荧光定量PCR及数据分析目前, 在转录组数据中注释为组蛋白H3及其变体的基因中, 有三个基因经mRNA水平克隆验证了表达, 即H3.a(Algae_064-1_Unigene_BMK.42014)、H3.b(Algae_064-1_Unigene_BMK.10241)和H3.c(Algae_ 064-2_Unigene_BMK.1001)。其中, H3.a和H3.b在前期研究中被验证能够强烈响应营养胁迫[20], 故挑选这两个具有代表性的基因进行不同生长时期的表达分析, 进一步探究其功能。
使用Primer Premier 5.0软件设计引物, 详细信息如表 2所示。
| 基因名 | 转录组数据库(SRX368254)中的名称/基因名 | 引物序列正向/反向(5′-3′) | 产物(bp) |
| H3.a | Algae_064-1_Unigene_BMK.42014 | CACGGACCTTCTCATTCGCAAAC/ CAAGCACAGCCTGGGATTGGAAC | 101 |
| H3.b | Algae_064-1_Unigene_BMK.10241 | CTTCGAGGACGCCAACCTCTGC/ TGCGACGACACCCCCTTTACCC | 125 |
| Control 1 | GAPDH | GACGGTGACTCCCTGGTGATTG/ TTCAAGTGCGGCTGCACCTTTT | 152 |
| Control 2 | β-actin | TCGCGTTGTATTCTTGTTGTAGCC/ TCGTCGCCGATGTAACTGTCCT | 197 |
使用Roche LightCycler 480系统进行实时荧光定量PCR实验, 反应体系为20 μL: SYBR GreenⅠPremix(10 μL), 正向和反向引物(10 μm, 各0.5 μL), 模板cDNA(10 ng)。程序设置: 94℃ 5 min; 94℃ 10s; 60℃退火1 min, 循环40次。
针对培养至第3天、第9天、第15天和第30天的链状亚历山大藻, 进行基因表达分析。以甘油醛-3-磷酸脱氢酶(GAPDH)和肌动蛋白(β-actin)基因作为内参, 以两者平均Ct值以此作为参照数据进行计算; 以培养3天的材料作为对照条件, 用比较阈值(2–ΔΔCt)方法计算倍数变化[21]; 若表达变化超过2倍, 则认定基因表达发生显著改变[22]。
1.5 TCA法提取组蛋白(酸溶解蛋白)使用三氯乙酸(TCA)沉淀法[9]提取链状亚历山大藻组蛋白。收集细胞并悬浮于1mL冰冷的酸提取缓冲液中, 缓冲液配方: 1.5 mmol/L MgCl2, 10 mmol/L HEPES, 0.5 mmol/L DTT(pH 7.9), 10 mmol/LKCl, 1.5 mmol/L苯甲基磺酰氟和0.25 mol/L HCl; 加入石英砂, 匀浆仪破碎; 冰上混匀孵育1小时; 所得细胞裂解物4℃以11 000×g离心10分钟, 收集上清液, 加入100%TCA使之终浓度为33%(w/v); 将该溶液在4℃下孵育7小时以上; 4℃以16 000×g离心15分钟获得酸溶性蛋白质(ASP); 保留沉淀, 用冷丙酮洗涤两次, 并且溶于100 μL去离子水中。Bradford测定法进行定量[23]。
1.6 蛋白免疫印迹(Western Blot)及数据分析根据氨基酸序列同源性比对结果, 选用人源组蛋白的兔单克隆商业抗体(ab176842)对不同生长时期的链状亚历山大藻组蛋白H3进行蛋白质印迹(Western Blot)分析。
配制15%的聚丙烯酰胺凝胶进行电泳(SDS- PAGE), 上样量300 μg; 取用0.22 μm孔径的PVDF膜, 使用半干电转移系统(Bio-Rad, USA)转膜; 采用人源组蛋白的兔单克隆商业抗体(abcam, ab176842)为一抗, HRP标记的山羊抗兔IgG(Sangon D110058)为二抗进行免疫反应; 化学发光底物(PierceTM ECL Western Blotting Substrate, Thermo)用于显影反应, 将膜置于化学发光成像系统(Tanon 5200, China)检测拍照; 使用ImageJ软件对显影图中的条带进行灰度分析。
2 结果与分析 2.1 链状亚历山大藻生长曲线链状亚历山大藻生长曲线如图 1所示。可见在前三天, 藻生长缓慢, 处于生长的延滞期; 藻细胞数目第4天之后开始激增, 进入对数生长期; 第10天之后藻的增长速率趋于稳定, 进入对数末期; 直到25天之后生长逐渐停滞。
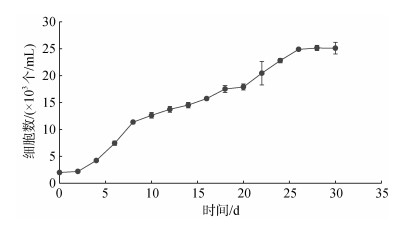 |
| 图 1 链状亚历山大藻生长曲线 Fig. 1 Growth curve of A. catenella |
ProtParam软件预测显示链状亚历山大藻组蛋白H3分子式为C680H1146N212O186S4, 蛋白分子量15.4 kDa, 理论等电点11.04; Predictprotein预测蛋白质二级结构显示H3.c由α螺旋(51.47%)、无规则卷曲(46.23%)、β折叠(2.21%)构成。
将链状亚历山大藻另外两个经过验证的组蛋白H3基因H3.a(Algae_064-1_Unigene_BMK.42014)、H3.b(Algae_064-1_Unigene_BMK.10241)[20]与本实验中的H3.c(Algae_064-2_Unigene_BMK.1001)一同进行序列比对, 如图 2。分析发现, 链状亚历山大藻与其他物种H3的组蛋白核心折叠区序列相似性较高, H3.b序列与其他物种的序列相似性较低, 尤其是N末端序列差异较大, 为链状亚历山大藻所特有的组蛋白H3变体, 甚至在通常保守的组蛋白折叠结构域都有很多变异位点; H3.c与H3.a序列相似性非常高, 并且与原生动物、绿藻的序列更为相近。组蛋白H3第87-90位氨基酸的变异被认为是H3.1/3.2与H3.3变体产生功能差异的因素之一[24], 在这里, H3.c、H3.a与团藻、莱茵衣藻同为QAVL, 而H3.b为QALL, 区别于其他各物种。
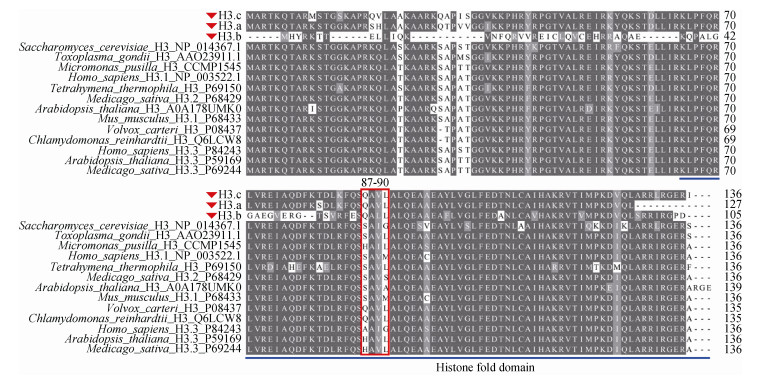 |
| 图 2 链状亚历山大藻与其他物种组蛋白H3序列比对结果 Fig. 2 Alignment result of histone H3 sequences between A. catenella and other species 注:图中红色三角标注了三种链状亚历山大藻组蛋白H3变体, 红色方框圈出的是变体特异性的氨基酸序列, 蓝色线条显示了组蛋白折叠结构域比对物种包括:酿酒酵母(Saccharomyces cerevisiae), 弓形虫(Toxoplasma gondii), 细小微胞藻(Micromonas pusilla), 智人(Homo sapiens), 四膜虫(Tetrahymena thermophila), 紫花苜蓿(Medicago sativa), 拟南芥(Arabidopsis thaliana), 小鼠(Mus musculus), 团藻(Volvox carteri), 莱茵衣藻(Chlamydomonas reinhardtii) |
将链状亚历山大藻H3.a、H3.b、H3.c基因进行系统进化分析, 进化树如图 3所示, 发现H3.b与大多真核生物组蛋白H3未能聚为一支, 具有较古老的起源; 而另外两个组蛋白基因H3.a、H3.b与四豆藻、莱茵衣藻等绿藻同源基因具有较近的起源。
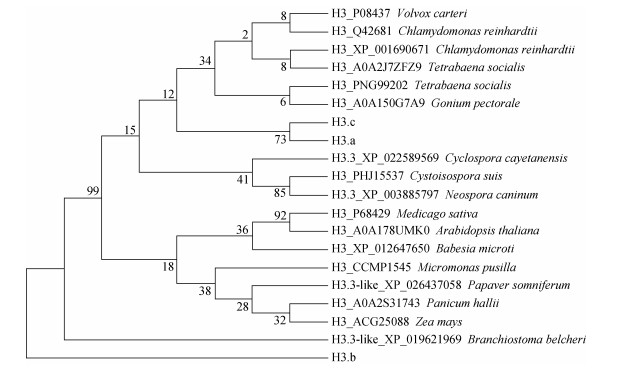 |
| 图 3 组蛋白H3及其变体ML系统进化树 Fig. 3 Maximum likelihood phylogenetic tree of H3 proteins 注: Bootstrap重复次数为1200;其中H3.a、H3.b和H3.c为链状亚历山大藻组蛋白H3。对比物种包括:团藻(Volvox carteri), 莱茵衣藻(Chlamydomonas reinhardtii), 四豆藻(Tetrabaena socialis), 胸状盘藻(Gonium pectorale), 环孢子虫(Cyclospora cayetanensis), 等孢子虫(Cystoisospora suis), 新孢子虫(Neospora caninum), 紫花苜蓿(Medicago sativa), 拟南芥(Arabidopsis thaliana), 微小巴贝虫(Babesia microti), 细小微胞藻(Micromonas pusilla), 罂粟(Papaver somniferum), 黍(Panicum hallii), 玉米(Zea mays), 文昌鱼(Branchiostoma belcheri) |
组蛋白翻译后修饰位点分析见图 4, 包括乙酰化、泛素化、甲基化、磷酸化位点[25]。结果表明, H3.a和H3.c两种变体的修饰位点与人组蛋白非常相似, 除了二者的H3K18、H3S28以及H3.c的H3K9之外, 两者都保留了组蛋白密码相关的所有关键残基。此外, H3.b仅在H3K56和H3K64处保守。
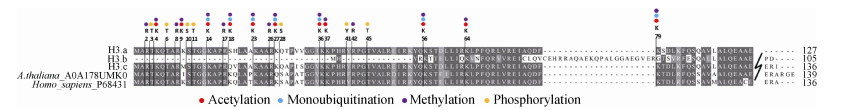 |
| 图 4 链状亚历山大藻与其他物种组蛋白H3及其变体的修饰位点对比 Fig. 4 Comparison of modification sites of H3 and its variants between A. catenella and other species |
链状亚历山大藻生长第3天、第9天、第15天和第30天, 即生长的延迟期、对数前期、对数后期和衰亡期, H3基因表达水平如图 5所示。可见组蛋白H3.a在藻生长过程中稳定表达, 而组蛋白H3.b在生长对数期后期(培养至第15天)表达上调, 衰亡期又下调至正常水平。
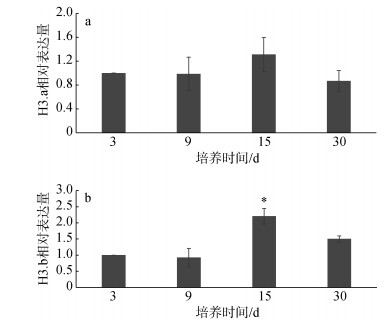 |
| 图 5 链状亚历山大组蛋白H3基因在不同生长时期的基因表达差异 Fig. 5 Relative gene expression of the H3 gene of A. catenella at different growth stages 注:图a和图b分别表示H3.a基因和H3.b基因在链状亚历山大藻不同生长时期的表达差异; 星号表示此时期基因表达呈现显著差异 |
实验所用商业抗体其免疫原系人组蛋白H3第100位氨基酸至碳端的保守序列。结果显示链状亚历山大藻组蛋白H3在对数期培养期间虽然表达量有波动, 但未达到显著差异的水平, 见图 6。当甲藻生长进入衰亡期(培养至第30天)组蛋白H3的表达呈现明显下调, 表明在衰亡期组蛋白H3存在衰减现象。
 |
| 图 6 链状亚历山大藻组蛋白H3 Western Blot结果 Fig. 6 Immuno detection of histone H3 in A. catenella during different growth stages 注:图a表示各时期H3显影灰度值; 图b所示为不同生长时期组蛋白H3 Western Blot显影图 |
核心组蛋白不仅可作为包装DNA的结构蛋白, 也可演化为多种变体, 产生具有表观遗传学效果的染色质分化。后生动物中, H3变体可通过偶联DNA复制(replication-dependent, RD)或复制非依赖性(replication-independent, RI)方式组装核小体, RI型变体H3.3虽与常规组蛋白H3仅相差4个氨基酸序列, 但区别于常规组蛋白H3或某些H3.1、H3.2变体, 它能够灵活替换常规组蛋白并发挥调控功能, 普遍存在于活跃的染色质区域[18, 26]。
甲藻中组蛋白H3的演化十分多样, 除尚未明确发现演化迅速的着丝粒H3(CENP), 部分甲藻中已经发现多种H3变体[25]; 大多数真核生物常规组蛋白H3.1在细胞周期S期集中表达, 包装新合成的DNA, 而H3.3变体虽然仅与常规H3相差少数几个残基, 最关键的差别在于它属于复制非依赖(RI)亚型, 能够灵活地在其他细胞时期替代常规H3构成染色质, 参与染色质重塑, 进而发挥调控或表观遗传功能[27]。常规RD组蛋白3’端含有保守的茎环而不是polyA尾[28-29], 而基于mRNA 3’polyA序列分析的高通量转录组测序数据中, 缺乏RD组蛋白等相关信息。此前Riaz对链状亚历山大藻组蛋白在细胞周期中的表达研究中发现, 各个组蛋白在G期和S期都未呈现差异表达[20], 推测本文研究的三种H3均为RI组蛋白; 而H3.b的序列同源性较其他真核生物差别较大, 推测其为甲藻特有的组蛋白H3变体。
同时, Riaz的研究说明链状亚历山大藻组蛋白的表达并不像经典组蛋白一样, 受细胞周期的调控而跟随分裂进程大量表达。本研究发现在分裂旺盛的对数期(培养第3天至第9天), 链状亚历山大藻组蛋白H3基因保持相对稳定的表达, 这为前人的观点提供了新的证据。事实上, 许多藻类研究发现, 甲藻通过转录过程对mRNA表达水平的改变十分有限, 例如通过RNA-Seq检测多边舌甲藻(Lingulodinium polyedrum)的转录组, 发现在12︰12光暗周期或24小时恒定光照期间没有检测到任何转录物的表达发生显著变化[30], 对甲藻转录模型的研究表明, 其大部分转录发生在非凝缩的染色体外的染色质环上, 这些环突出于永久浓缩的普通染色质团块之外[11], Georgi等认为如果这一模型成立, 那么核小体的组装则可能发生在这一临时形成的环结构中, 一系列调控进程都应与活跃的转录水平相关联, 而不是依赖于细胞周期进程[25]。
培养至第15天时, 藻的增长速率放缓并趋于平稳, 这说明随着种群密度逐渐升高、水体环境的改变, 一些藻细胞的生长代谢受到种群压力或某些环境因素的诱导而开始减缓。营养盐结构及浓度、光照等环境变化都会极大影响甲藻的生长繁殖与代谢水平, 与赤潮的爆发生消息息相关[31-32]; 前期研究发现, 在氮、磷营养过剩或胁迫的条件下, 链状亚历山大藻组蛋白H3基因表达均能够响应这些生长因素的骤变[20]; 本研究中, H3.b基因在对数末期(培养第15天)出现上调, 因此推测部分藻细胞组蛋白H3在逐渐加剧且复杂化的种群压力、营养竞争中参与了一些基因的表达调控。
而到了培养第30天, 各组蛋白的表达量较第15天均有所下调, 这很可能因为进入衰亡期的链状亚历山大藻生长状态变差, 蛋白质的合成受阻或降解[33], 组蛋白的表达也随之降低。
在蛋白水平上, 由于实验所用商业抗体的免疫原为人组蛋白H3第100位氨基酸至碳端序列, 由序列比对结果可知这段序列在不同物种乃至不同H3变体之间都十分保守, 目前暂没有能识别组蛋白H3.3等变体的特异性抗体在售。因此, 免疫反应结果可代表总体H3蛋白表达水平。对数生长期的组蛋白H3虽然有部分变体在第15天表达量提高, 但并未呈现显著变化, 而培养至30天的H3已显示出明显衰减, 可能是因为进入衰亡期的链状亚历山大藻细胞中蛋白质合成受阻或降解[33], 推测在衰亡期基于H3的表观遗传调控受到抑制。此外对于甲藻基因与蛋白表达水平差异的研究发现, 许多甲藻蛋白水平和RNA水平之间的相关性不是很好, 例如, 58种不同浓度的共生甲藻蛋白中, 没有一种表达水平与它们的转录本一致[34]。
组蛋白及其变体具有表观遗传调控功能, 组蛋白翻译后修饰(HPTM)作为一类重要的表观遗传调控机制, 可以灵活标记染色质; 复制非依赖型(RI)的组蛋白变体发生替换的同时, 也会导致原有翻译后修饰的擦除或改变, 例如哺乳动物细胞中, 携带着H3乙酰化和H3K4甲基化标记的H3.3沉积在转录活性基因上[19]; 在RNA酶Ⅰ和Ⅱ转录的基因座, 带有H3K9甲基化修饰的H3被RI置换成加标签的H3.3[28]; 因此, 组蛋白的共价修饰可能来自组蛋白变体被组装时其上既有的修饰, 修饰酶会跟随组装装置, 并可能促进这一进程[35]。对链状亚历山大藻组蛋白H3序列修饰位点的预测表明, H3.a和H3.c保留了绝大多数组蛋白修饰位点, 具备形成H3翻译后修饰的条件; H3.b独特的N端尾巴序列虽然与其他H3差异较大, 但不排除发生某些翻译后修饰的可能, 例如H3K64, 乙酰化的H3K64富集在活性基因的转录起始位点, 用于判定活性染色质[36]; 而与之相对的H3K64me3则富集于异染色质, 与重复序列和转录不活跃的基因组区域相关[37]。
此外, 对赤潮甲藻的研究表明, 细胞生理状态突遭强烈水流或细胞密度过高等机械冲击时, 失去鞭毛形成的不动细胞可以通过形成暂时性孢囊缓解种群生存压力, 且在环境适宜时能迅速恢复成有游动能力的营养细胞[38-40]。甲藻孢囊处于停止分裂的静止状态, 本实验中多次镜检观察到链状亚历山大藻形成的暂时性孢囊。有研究表明, 变体H3.3多在休眠细胞和分化细胞中表达[41], 休眠期基因表达受到抑制, 为维持染色质活性状态的表观遗传记忆, H3.3大量表达, 参与组蛋白替换, 从而有利于休眠解除后转录调控元件的结合, 影响染色质的活性。Gurdon课题组证实在没有转录的情况下, 活性基因表观遗传的记忆依赖于H3.3嵌入染色质[41]; 高等植物中也发现了类似现象, 如油桃PpH3.3在休眠期表达水平显著高于休眠诱导期和休眠解除期[42]。因此不排除这种猜想, 即链状亚历山大藻生长至平稳期, 部分细胞在逐渐加剧的种群压力下形成暂时性孢囊, 组蛋白H3变体表达, 替换相关基因的结合蛋白, 可能为休眠期的藻细胞提供染色质标记, 参与其萌发过程相关基因的转录调控。
4 小结本研究克隆了链状亚历山大藻组蛋白H3变体之一H3.c的全长ORF序列; 根据链状亚历山大藻生长曲线, 对比分析在链状亚历山大藻爆发性生长至衰亡过程中组蛋白H3基因在转录和蛋白水平的表达。综上所述, 无论基因或蛋白水平上, 组蛋白H3在链状亚历山大藻生长过程中, 特别是对数生长期均活跃稳定表达, 但表达量不会随着藻爆发性增长而上调或下调; 一些组蛋白H3变体可响应伴随藻不断生长而逐渐增强的细胞密度和缺营养带来的胁迫, 推测其参与某些表观遗传修饰, 调控链状亚历山大藻的生长进程, 而生长至衰亡期基于H3的表观遗传调控受到抑制。
| [1] |
Hallegraeff G. A review of harmful algal blooms and their apparent global increase[J]. Phycologia, 1993, 32(2): 79-99. DOI:10.2216/i0031-8884-32-2-79.1 |
| [2] |
Bouligand Y, Norris V. Chromosome separation and segregation in dinoflagellates and bacteria may depend on liquid crystalline states[J]. Biochimie, 2001, 83(2): 187-192. |
| [3] |
Lin S. Genomic understanding of dinoflagellates[J]. Research in Microbiology, 2011, 162(6): 551-569. DOI:10.1016/j.resmic.2011.04.006 |
| [4] |
Hou Y, Li nS. Distinct gene number-genome size relationships for eukaryotes and non-eukaryotes: gene content estimation for dinoflagellate genomes[J]. PLOS ONE, 2009, 4(9): e6978. DOI:10.1371/journal.pone.0006978 |
| [5] |
Chan Y H, Wong J T Y. Concentration-dependent organization of DNA by the dinoflagellate histone-like protein HCc3[J]. Nucleic Acids Research, 2007, 35(8): 2573-2583. DOI:10.1093/nar/gkm165 |
| [6] |
Kohli G S, John U, Figueroa R I, et al. Polyketide synthesis genes associated with toxin production in two species of Gambierdiscus (Dinophyceae)[J]. BMC Genomics, 2015, 16(1): 410. DOI:10.1186/s12864-015-1625-y |
| [7] |
Zhang S, Sui Z, Chang L, et al. Transcriptome de novo assembly sequencing and analysis of the toxic dinoflagellate Alexandrium catenella using the Illumina platform[J]. Gene, 2014, 537(2): 285-293. DOI:10.1016/j.gene.2013.12.041 |
| [8] |
Shoguchi E, Shinzato C, Kawashima T, et al. Draft assembly of the Symbiodinium minutum nuclear genome reveals dinoflagellate gene structure[J]. Current biology, 2013, 23(15): 1399-1408. DOI:10.1016/j.cub.2013.05.062 |
| [9] |
Roy S, Morse D. A full suite of histone and histone modifying genes are transcribed in the dinoflagellate Lingulodinium[J]. PLoS One, 2012, 7(4): e34340. DOI:10.1371/journal.pone.0034340 |
| [10] |
Bayer T, Aranda M, Sunagawa S, et al. Symbiodinium transcriptomes: genome insights into the dinoflagellate symbionts of reef-building corals[J]. PloS one, 2012, 7(4): e35269. DOI:10.1371/journal.pone.0035269 |
| [11] |
Wisecaver J H, Hackett J D. Dinoflagellate genome evolution[J]. Annual review of microbiology, 2011, 65: 369-387. DOI:10.1146/annurev-micro-090110-102841 |
| [12] |
Seto E. Histone modifications[J]. New Comprehensive Biochemistry, 2003, 31(1): 1-2. |
| [13] |
Allis C D, Jenuwein T, Reinberg D. Epigenetics[M]. New York: Cold Spring Harbor Laboratory Press, 2007.
|
| [14] |
Meaney M J, Ferguson-Smith A C. Epigenetic regulation of the neural transcriptome: the meaning of the marks[J]. Nature neuroscience, 2010, 13(11): 1313-1318. DOI:10.1038/nn1110-1313 |
| [15] |
Portela A, Esteller M. Epigenetic modifications and human disease[J]. Nature biotechnology, 2010, 28(10): 1057. DOI:10.1038/nbt.1685 |
| [16] |
王迪, 傅彬英, 张立军. 植物表观遗传变化与环境压力研究进展[J]. 分子植物育种, 2008, 6(3): 569-573. Wang Di, Fu Binying, Zhang Lijun. The Plant's epigenetic variation and response to environmental stress[J]. Molecular Plant Breeding, 2008, 6(3): 569-573. DOI:10.3969/j.issn.1672-416X.2008.03.026 |
| [17] |
Berr A, Shafiq S, Shen W H. Histone modifications in transcriptional activation during plant development[J]. Biochimicaet Biophysica Acta (BBA)-Gene Regulatory Mechanisms, 2011, 1809(10): 567-576. DOI:10.1016/j.bbagrm.2011.07.001 |
| [18] |
Chow C M, Georgiou A, Szutorisz H, et al. Variant histone H3.3 marks promoters of transcriptionally active genes during mammalian cell division[J]. Embo Reports, 2005, 6(4): 354-360. DOI:10.1038/sj.embor.7400366 |
| [19] |
Hake S B, Allis C D. Histone H3 variants and their potential role in indexing mammalian genomes: the "H3 barcode hypothesis"[J]. Proceedings of the National Academy of Sciences, 2006, 103(17): 6428-6435. DOI:10.1073/pnas.0600803103 |
| [20] |
RiazS, Sui Z. Molecular cloning, transcript profiling, and characterization of histone genes in the dinoflagellate Alexandriumpacificum[J]. Journal of Microbiology and Biotechnology, 2018, 28(7): 1185-1198. DOI:10.4014/jmb.1802.01075 |
| [21] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCtmethod[J]. Methods, 2001, 25: 402-408. DOI:10.1006/meth.2001.1262 |
| [22] |
Lupke M, Frahm J, Lantow M, et al. Gene expression analysis of ELF-MF exposed human monocytes indicating the involvement of the alternative activation pathway[J]. Biochimicaet Biophysica Acta, 2006, 1763(4): 402-412. DOI:10.1016/j.bbamcr.2006.03.003 |
| [23] |
Kruger N. The Bradford method for protein quantitation[J]. Methods in Molecular Biology, 1988, 32(32): 9-15. |
| [24] |
Ahmad K, Henikoff S. The histone variant H3.3 marks active chromatin by replication-independent nucleosome assembly[J]. Molecular cell, 2002, 9(6): 1191-1200. DOI:10.1016/S1097-2765(02)00542-7 |
| [25] |
Marinov G K, Lynch M. Diversity and divergence of dinoflagellate histone proteins[J]. G3: Genes, Genomes, Genetics, 2016, 6(2): 397-422. |
| [26] |
Talbert P B, Ahmad K, Almouzni G, et al. A unified phylogeny-based nomenclature for histone variants[J]. Epigenetics & Chromatin, 2012, 5(1): 7. |
| [27] |
Desvoyes B, Fernándezmarcos M, Sequeiramendes J, et al. Looking at plant cell cycle from the chromatin window[J]. Front Plant Science, 2014, 5(5): 369. |
| [28] |
Lyons S M, Cunningham C H, Welch J D, et al. A subset of replication-dependent histone mRNAs are expressed as poly adenylated RNAs in terminally differentiated tissues[J]. Nucleic Acids Research, 2016, 44(19): 9190-9205. |
| [29] |
Marzluff W F, Wagner E J, Duronio R J. Metabolism and regulation of canonical histone mRNAs: life without a poly (A) tail[J]. Nature Reviews Genetics, 2008, 9(11): 843. DOI:10.1038/nrg2438 |
| [30] |
Roy S, Beauchemin M, Dagenaisbellefeuille S, et al. The Lingulodinium circadian system lacks rhythmic changes in transcript abundance[J]. BMC Biology, 2014, 12(1): 107. DOI:10.1186/s12915-014-0107-z |
| [31] |
张淑雯, 欧林坚, 吕颂辉, 等. 光照及营养盐对3种赤潮甲藻吞噬营养行为的影响[J]. 海洋科学, 2011, 35(4): 94-99. Zhang Shuwen, Ou Linjian, Lv Songhui, et al. Effects of light and nutrients on the phagotrophic behaviors of three harmful dinoflagellates[J]. Marine Sciences, 2011, 35(4): 94-99. |
| [32] |
张青田, 董双林, 胡桂坤, 等. 不同氮源对微藻增殖的影响[J]. 海洋科学, 2005, 29(2): 8-11. Zhang Qingtian, Dong Shuanglin, Hu Guikun, et al. Effects of different nitrogen on growth of micro algae[J]. Marine Sciences, 2005, 29(2): 8-11. DOI:10.3969/j.issn.1000-3096.2005.02.003 |
| [33] |
马金华, 孟希, 张淑, 等. 链状亚历山大藻赤潮衰亡的生理调控[J]. 生态学报, 2013, 33(13): 3978-3986. Ma Jinhua, Meng Xi, Zhang Shu, et al. Physiological regulation related to the decline of Alexandrium catenella[J]. Acta Ecologica Sinica, 2013, 33(13): 3978-3986. |
| [34] |
Mayfield A B, Wang Y B, Chen C S, et al. Dual-compartmental transcriptomic+proteomic analysis of a marine endosymbiosis exposed to environmental change[J]. Molecular Ecology, 2016, 25(23): 5944-5958. DOI:10.1111/mec.13896 |
| [35] |
Steven H, Kami A. Assembly of variant histones into chromatin[J]. Annual Review of Cell & Developmental Biology, 2005, 21: 133-153. |
| [36] |
Di Cerbo V, Mohn F, Ryan D P, et al. Acetylation of histone H3 at lysine 64 regulates nucleosome dynamics and facilitates transcription[J]. Elife, 2014, 3: e01632. DOI:10.7554/eLife.01632 |
| [37] |
Daujat S, Weiss T, Mohn F, et al. H3K64 trimethylation marks heterochromatin and is dynamically remodeled during developmental reprogramming[J]. Nature structural & molecular biology, 2009, 16(7): 777-781. |
| [38] |
肖咏之.中国沿海甲藻孢囊分类、分布和孢囊形成动力学研究[D].广州: 暨南大学, 2001. Xiao Yongzhi. Taxonomy, bio-distribution and production dynamics of dinoflagellate cyst from Recent Chinese marine sediments[D]. Guangzhou: Jinan University, 2001. http://cdmd.cnki.com.cn/Article/CDMD-10559-2001001770.htm |
| [39] |
Brosnahan M L, Ralston D K, Fischer A D, et al. Bloom termination of the toxic dinoflagellate Alexandrium catenella: Vertical migration behavior, sediment infiltration, and benthic cyst yield[J]. Limnology & Oceanography, 2017, 62(6): 2829-2849. |
| [40] |
康伟, 王朝晖. 桂山岛海域甲藻孢囊的分布与浮游植物休眠体萌发研究[J]. 海洋科学, 2016, 40(7): 33-40. Kang Wei, Wang Zhaohui. Distribution of dinocysts and the germination of phytoplankton resting spores in surface sediments from the Guishan Island sea area in the Pearl River Estuary of the South China Sea[J]. Marine Sciences, 2016, 40(7): 33-40. |
| [41] |
Ng R, Gurdon J. Epigenetic memory of an active gene state depends on histone H3.3 incorporation into chromatin in the absence of transcription[J]. Nature Cell Biology, 2008, 10(1): 102-109. DOI:10.1038/ncb1674 |
| [42] |
冷传远, 李玲, 谭钺, 等. 油桃组蛋白基因PpH3.3的克隆和表达分析[J]. 植物生理学报, 2013, 49(8): 764-770. Leng Chuanyuan, Li Ling, Tan Yue, et al. Clone and expression analysis of a cDNA encoding histone H3.3 in prunuspersica var. nectarina maxim[J]. Plant Physiology Journal, 2013, 49(8): 764-770. |
 2019, Vol. 43
2019, Vol. 43


