
文章信息
- 付新华, 刘国宁, 孙凤祥, 辛鹿, 陈倩倩. 2019.
- FU Xin-hua, LIU Guo-ning, SUN Feng-xiang, XIN Lu, CHEN Qian-qian. 2019.
- 基于线粒体COI序列莱州湾单环刺螠遗传多样性分析
- Analysis of the genetic diversity of Urechis unicinctus in Laizhou Bay based on the mitochondrial COI gene sequence
- 海洋科学, 43(11): 56-61
- Marine Sciences, 43(11): 56-61.
- http://dx.doi.org/10.11759/hykx20190510001
-
文章历史
- 收稿日期:2019-05-10
- 修回日期:2019-08-11
2. 潍坊市环境监控中心, 山东 潍坊 261041
2. Weifang Environmental Monitoring Center, 261041, China
单环刺螠(Urechis unicinctus)是螠虫动物门的重要经济海洋动物, 同时也是无管螠目(Xenopneusta)在中国海域分布的唯一物种[1]。该螠虫形似海参, 体壁中蛋白和营养必需氨基酸含量高, 内脏中也存在大量多糖、酶等生理活性物质, 具有巨大的经济价值。单环刺螠可以存活于硫化物含量较高的海洋环境中, 并对其环境中的硫化物具有一定的解毒能力[2], 对人类治理海洋硫化物污染给予了新的启示。基于单环刺螠的独特分类学地位、优于其他海洋生物对硫化物的耐受解毒能力以及广阔经济前景, 针对单环刺螠的研究已经在国内外成为新的热点之一[3-5]。在中国, 单环刺螠主要分布于渤海三大海湾之一的莱州湾地区, 近年来该海域不仅遭受时有发生的海上漏油事件的影响, 而且遭受陆源污染物的大量排放, 海洋污染问题较为突出。环境污染可影响地理种群的多样性, 使生物资源衰退[6]。进行莱州湾单环刺螠遗传多样性研究, 对该种群的进一步针对性保护和合理开发均有深刻意义。
近年来, 包括简单重复序列、简单重复序列中间区域以及单核苷酸多态性分析等在内的多种分子标记在生物遗传多样性的分析中广泛应用[7-9]。在当前所有分子标记中, 单核苷酸多态性(SNP)标记是涵盖生物体全部DNA多态的惟一标记方法[10], 基于COI基因序列的SNP已用于多种海洋生物的遗传多样性分析[11-13]。生物体线粒体DNA是探究群体遗传学的有效标记, 而COI基因是线粒体DNA的标准目标区域, 并在所有物种中进行编码[14], 现已进行的大量工作已证明COI基因序列用于物种鉴定、遗传多样性分析和进化分析具有可行性、有效性和准确性[15-17]。本研究对莱州湾单环刺螠多个群体进行COI基因分析, 以期为该地理种群的保护和合理利用提供基础依据。
1 材料与方法 1.1 样本采集实验共采集单环刺螠3个群体84个样本, 其中群体1的21个样本(编号1-1~1-21)采自潍坊莱州湾单环刺螠保护区, 群体2的30个样本(编号2-1~2-30)采自莱州浅滩保护区, 群体3的33个样本(编号3-1~3-33)采自昌邑海区。样本来源见表 1。单环刺螠活体运输至实验室。
| 群体 | 样本编号 | 采样地 | 经纬度 |
| 1 | 1-1~1-21 | 潍坊莱州湾单环刺螠保护区 | N 37°25′; E 119°15′ |
| 2 | 2-1~2-30 | 莱州浅滩保护区 | N 37°14′; E 119°38′ |
| 3 | 3-1~3-34 | 昌邑海区 | N 37°20′; E 119°48′ |
解剖单环刺螠活体, 去除其内脏组织, 取其体壁, 采用Omega公司的动物组织DNA提取试剂盒提取单环刺螠基因组DNA。
1.3 基于COI基因序列的遗传多样性分析 1.3.1 COI基因的PCR扩增根据NCBI上搜索到的单环刺螠COI序列(登录号: EF656365), 利用Primer Premier软件设计扩增该序列所需的特异引物, 并由华大科技有限公司合成, 引物序列(CXF、CXR)见表 2; 同时合成后生动物通用引物, 引物序列(LCO、HCO)见表 2。
| 引物 | 序列(5′→3′) |
| CXF | GTGCGATGATTATACTCAACAAACCACAAAGAC |
| CXR | TTATTGGGTAATTACTCCTGTTTCGTCTGC |
| LCO | GGTCAACAAATCATAAAGATATTGG |
| HCO | TAAACTTCAGGGTGACCAAAAAATCA |
分别以CXF、CXR以及LCO、HCO作为上下游引物, 提取的单环刺螠DNA为模板进行PCR扩增。反应总体系50 μL, 其中上下游引物(10 μmol/L)各2 μL, DNA模板2 μL。反应程序: 94℃预变性5 min; 95℃变性30 s, 57℃退火30 s, 72℃延伸45 s, 30个循环; 最后72℃延伸10 min。纯化后的COI扩增产物由华大科技有限公司进行双向测序。
1.3.2 COI数据分析利用MEGA5软件中的ClustalW对得到的COI序列进行比对[18], 去掉两端有误差的部分, 最终得到一个用于分析的碱基序列阵式文件。应用DnaSP软件计算各个核苷酸的多样性[19], 并分析平均核苷酸差异。利用MEGA5软件进行简约信息位点和突变位点分析[18], 计算简约信息位点的比率以及各碱基的含量; 利用Kimura 2-parameater模型构建NJ分子系统树[20, 21]。
2 结果与分析 2.1 基因组DNA提取结果单环刺螠DNA的提取对于整个实验过程至关重要, 使用试剂盒提取的单环刺螠DNA检测后表明, 提取结果较好。
在提取过程中, 单环刺螠的保鲜和细胞裂解条件至关重要。活体单环刺螠内的基因组降解酶活性小, 而机体失活后, 其内的DNA酶迅速活跃, 可使大量DNA损失, 导致所提量甚微。在液氮中快速研磨组织块, 加入蛋白酶水浴保温裂解细胞, 裂解时间一般3 h左右, 同时避免剧烈振荡等造成DNA的断裂。提取后的DNA应即时贮存于–20℃冰箱中, 以减缓其降解的速度。
2.2 莱州湾3个群体COI序列分析实验结果单环刺螠COI序列扩增特异引物(CXF、CXR)扩增效果较好(图 1), 后生动物通用引物(LCO、HCO)扩增效果较差。扩增后单环刺螠COI序列全长1 533 bp, 去除去掉两端有误差的序列, 最终得到用于分析的1 309 bp碱基序列, 84个样本中, 除去测序失败和序列过短的14个样本, 用于最终分析的样本为70个。将单环刺螠COI序列70个样本的全部序列作为一个共同数据矩阵, 用DnaSP软件进行处理。结果发现:单环刺螠COI序列有变异位点数125个, 其中43个单一变异位点, 82个简约信息位点; 48个单倍型, 单倍型多样性0.977;单倍型多样性方差0.00079, 单倍型多样性标准差为0.028;核苷酸多样性指数为0.01048;平均核苷酸差异数量为13.725。平均碱基
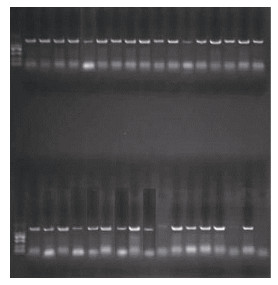 |
| 图 1 单环刺螠COI序列特异引物(CXF、CXR)扩增图 Fig. 1 Diagram of amplification of COI sequence of Urechis unicinctus using specific primers (CXF, CXR) |
组成: A, 26.3%; G, 17.6%; C, 24.2%; T, 31.8%; A+ T(58.1%)含量高于G+C(41.9%)含量。不同群体的遗传变异参数见表 3。莱州湾单环刺螠70个体的聚类图见图 2, 其中样品1-1~1-21为潍坊莱州湾单环刺螠保护区地理种群(群体1);样品2-1~2-30为莱州浅滩保护区地理种群(群体2);样品3-1~3-34为昌邑海区地理种群(群体3)。从图 2中可见, 70个个体聚类并没有体现出和地理位置相关。
| 群体 | 变异位点数 | 单倍型 | 单倍型多样性 | 核苷酸多样度 | 平均核苷酸差异 |
| 1 | 79 | 18 | 0.984±0.024 | 0.01273 | 16.705 |
| 2 | 32 | 19 | 0.945±0.027 | 0.01010 | 8.8182 |
| 3 | 14 | 22 | 0.978±0.021 | 0.01326 | 13.422 |
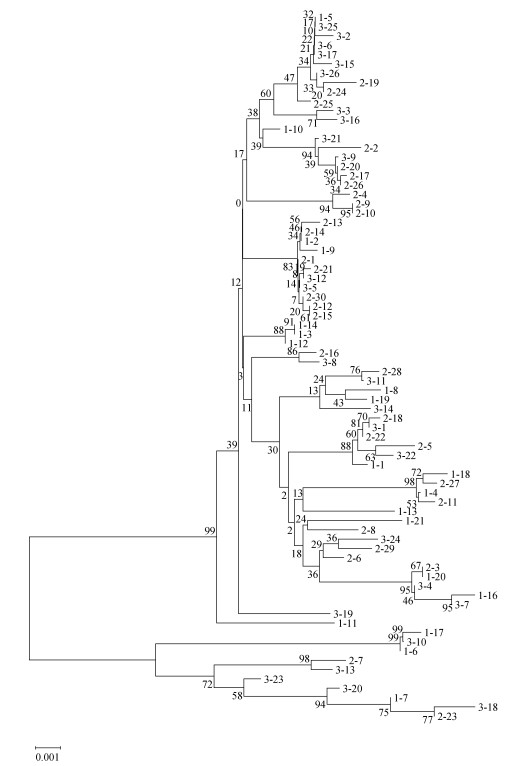 |
| 图 2 莱州湾3个群体单环刺螠系统发育树 Fig. 2 Phylogenetic tree of Urechis unicinctus of three populations in Laizhou Bay |
作为生物多样性的重要层次, 遗传多样性是评价生物资源的重要依据, 种群遗传多样性的度量和保护已成为对其生物资源进行评价的核心问题[22, 23]。不了解重要种群的遗传多样性, 就无法实现有针对性的科学保护和合理开发。
物种的遗传多样性是否丰富与单倍型多样性和核苷酸多样性指数高低密切相关[24], 数值越高, 多样性越丰富, 该物种的遗传多样性越高。莱州湾单环刺螠COI序列的单倍型多样性和核苷酸多样性明显高于中国海文昌鱼(Branchiostoma sp.)、前鳞鮻(Liza affinis)、丽文蛤(Meretrix lusoria)等其他海洋生物[12, 25, 26], 这可能由于在这些海洋动物中大多数种类已进行了多年人工增养殖, 近亲交配或遗传渐渗使其遗传多样性水平降低。未来在开展单环刺螠大规模人工增养殖活动时, 避免近亲交配, 减少遗传渐渗的出现, 将会对维持该地理种群的较高遗传多样性有重要作用。增殖放流时, 可增加放流单环刺螠的遗传多样性检测, 以保持该种群的较高遗传多样性。本研究结果还显示, 莱州湾3个单环刺螠群体的个体聚类并无地理位置相关性, 1个群体退化时可从其他群体引种, 以维系该地理种群较高的遗传多样性。
作者开展了莱州湾单环刺螠遗传多样性研究, 分析了其变异的大小, 并根据遗传多样性指数, 确定其遗传状况, 从而为该地理种群的长期保护和可持续性利用提供重要基础依据。
| [1] |
李诺, 宋淑莲, 唐永政. 单环刺螠[J]. 生物学通报, 1998, 33(8): 12-14. Li Nuo, Song Shulian, Tang Yongzheng. Urechis unicinctus[J]. Bulletin of Biology, 1998, 33(8): 12-14. |
| [2] |
张立涛.单环刺螠硫化物应激数字基因表达谱分析及硫双加氧酶功能研究[D].青岛: 中国海洋大学, 2014: 1-10. Zhang Litao. Single-incinctus sulfide stress digital gene expression profile analysis and research on the function of sulfide dioxygenase[D]. Qingdao: Ocean University of China, 2014: 1-10. http://cdmd.cnki.com.cn/Article/CDMD-10423-1014369016.htm |
| [3] |
许秀秀, 高壹, 叶晓通. 重组单环刺螠纤溶酶对大鼠急性心肌缺血的保护作用[J]. 中国现代应用药学, 2019, 36(8): 910-915. Xu Xiuxiu, Gao Yi, Ye Xiaotong. Protective effect of recombinant Urechis Unicinctus fibrinolytic enzyme on rats with acute myocarchal ischemia[J]. Chin J Mod Appl Pharm, 2019, 36(8): 910-915. |
| [4] |
牛荣丽, 叶桂华, 程珞瑶, 等. 单环刺螠重组血红蛋白酶解组分hemocidins的初步抗菌活性分析[J]. 中国新药杂志, 2019, 28(2): 184-189. Niu Rongli, Ye Guihua, Cheng Luoyao, et al. Antibacterial activities of different hydrolyzed peptides of recombinant hemoglobin from Urechis unicinctus[J]. Chinese Journal of New Drugs, 2019, 28(2): 184-189. |
| [5] |
Niu R, Chen X. Full-length cDNA, prokaryotic expression, and antimicrobial activity of Uu Hb-F-I from Urechis unicinctus[J]. Bio Med Res Inter, 2016, 2: 1-8. |
| [6] |
申丹, 焦琳琳, 常禹, 等. 黄海和渤海沿海地区生态系统健康评价[J]. 生态学杂志, 2015, 34(8): 2362-2372. Shen Dan, Jiao Linlin, Chang Yu, et al. Ecosystem health assessment of Yellow Sea and Bohai coastal areas[J]. Chinese Journal of Ecology, 2015, 34(8): 2362-2372. |
| [7] |
Banerjee S, Das M, Mir R R, et al. Assessment of genetic diversity and population structure in a selected germplasm collection of 292 Jute genotypes by microsatellite (SSR) markers[J]. Molecular Plant Breeding, 2012, 3(2): 11-26. |
| [8] |
Wu J Y, Deng Y Y, Ding Y L. RAPD analysis of the varieties of Phyllostachys nidularia[J]. Journal of Changzhou Institute of Technology, 2008, 21(1): 59-63. |
| [9] |
Lou Y F, Yang H Y, Zhang Y Z, et al. Analysis of genetic variation of some bamboo species by AFLP, ISSR, and SRAP[J]. Journal of Fujian College of Forestry, 2011, 31(1): 38-43. |
| [10] |
Zeng Q D, Wu J H, Huang S, et al. SNP-based linkage mapping for validation of adult plant stripe rust resistance QTL in common wheat cultivar Chakwal 86[J]. T he Crop Journal, 2019, 7(2): 176-186. |
| [11] |
李玉龙, 王彬, 王文波, 等. 基于COI基因分析辽东湾海蜇群体遗传多样性[J]. 水产科学, 2016, 35(4): 404-409. Li Yulong, Wang Bin, Wang Wenbo, et al. Genetic diversity of Jellyfish Rhopilema esculentum in Liaodong Bay based on COI sequence[J]. Fisheries Science, 2016, 35(4): 404-409. |
| [12] |
吴玲, 宋娜, 高天翔. 中国沿海2种文昌鱼遗传多样性研究[J]. 海洋湖沼通报, 2013, 3: 80-88. Wu Ling, Song Na, Gao Tianxiang. Molecular dversity of two Branchiostoma species of China coast[J]. Transactions of Oceanology and Limnology, 2013, 3: 80-88. |
| [13] |
徐田军, 孙悦娜, 廖智, 等. 日本囊对虾4个地理群体线粒体16S rRNA序列及遗传结构分析[J]. 动物学杂志, 2010, 45(2): 93-100. Xu Tianjun, Sun Yuena, Liao Zhi, et al. Genetic structure and sequence analysis of four stocks of Marsupenaeus japonicus using mitochondrial 16S rRNA[J]. Chinese Journal of Zoology, 2010, 45(2): 93-100. |
| [14] |
张正青.花绒寄甲线粒体基因组与合成调控基因及其COXI基因的种群遗传结构研究[D].杨凌: 西北农林科技大学, 2017: 22-30. Zhang Zhengqing. Mitochondrial genome, population genetic structure and mitochondrial synthesis regulation genes of Aastarcus helophoroides[D]. Yangling: North West Agriculture and Forestry University, 2017: 22-30. http://cdmd.cnki.com.cn/Article/CDMD-10712-1017100359.htm |
| [15] |
石林波, 张小燕, 汪雁, 等. 福建华溪蟹线粒体DNA COI和16S rRNA基因序列的遗传多样性[J]. 中国人兽共患病学报, 2013, 29(7): 671-675. Shi Linbo, Zhang Xiaoyan, Wang Yan, et al. Genetic diversity of Sinopotamon fukienense based on mitochondrial DNA COI & 16S rRNA[J]. Chinese Journal of Zoonoses, 2013, 29(7): 671-675. |
| [16] |
郑文娟, 朱世华, 沈锡权, 等. 基于线粒体COI基因序列探讨泥蚶的遗传分化[J]. 动物学研究, 2009, 30(1): 17-23. Zheng Wenjuan, Zhu Shihua, Shen Xiquan, et al. Genetic differentiation of Tegillarca granosa based on mitochondrial COI gene sequences[J]. Zoological Research, 2009, 30(1): 17-23. |
| [17] |
张玲玲, 姚立农, 陈华良, 等. 输入性卵形疟原虫Cytb、CoxI和LDH基因单核苷酸多态性分析[J]. 中国寄生虫学与寄生虫病杂志, 2017, 35(4): 1-5. Zhang Lingling, Yao Linong, Chen Hualiang, et al. Single nucleotide polymorphism analysis of Cytb, Cox I and LDH genes of imported Plasmodium ovale[J]. Chinese Journal of Parasitology and Parasitic Diseases, 2017, 35(4): 1-5. |
| [18] |
Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011, 28(10): 2731-2739. |
| [19] |
Librado P, Rozas J. DnaSP V5: Asoftware for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452. |
| [20] |
谭严芳, 金人超. 一种基于NJ的高效构建系统进化树算法[J]. 计算机工程与应用, 2004, 40(21): 84-85, 97. Tan Yanfang, Jin Renchao. The efficient algorithm for reconstructing phylogenetic tree based on Neibor-joining method[J]. Computer Engineering and Applications, 2004, 40(21): 84-85, 97. |
| [21] |
高凯. NJ进化树构建方法的改进及其应用[D].北京: 北京工业大学, 2008: 10-25. Gao Kai. Improvement and application of NJ evolutionary tree construction method[D]. Beijing: Beijing University of Technology, 2008: 10-25. |
| [22] |
Naeem S, Iii F S C, Costanza R, et al. Biodiversity and ecosystem functioning: Maintaining natural life support processes[J]. Issues in Ecology, 1999, 4: 2-12. |
| [23] |
徐伟, 车静. 从隐存种到我国生物多样性保护研究:现状与展望[J]. 中国科学:生命科学, 2019, 49(4): 519-530. Xu Wei, Che Jing. Cryptic species serve as a window into biodiversity conservation in China (in Chinese)[J]. Sci Sin Vitae, 2019, 49(4): 519-530. |
| [24] |
Bonin A, Nicole F, Pompanon F, et al. Population adaptive index: A new method to help measure intraspecific genetic diversity and prioritize populations for conservation[J]. Conservation Biology, 2010, 21(3): 697-708. |
| [25] |
黄镇宇, 章群, 卢丽锋, 等. 基于线粒体COI序列的中国前鳞鮻遗传多样性分析[J]. 渔业科学进展, 2019, 40(1): 46-52. Huang Zhenyu, Zhang Qun, Lu Llifeng, et al. Genetic diversity of Liza affinis in coastal waters of China deduced from mitochondrial COI sequences[J]. Progress in Fishery Sciences, 2019, 40(1): 46-52. |
| [26] |
田云方, 叶莹莹, 吴常文, 等. 基于线粒体COI基因序列的丽文蛤群体遗传多样性和遗传结构分析[J]. 浙江海洋学院学报(自然科学版), 2017, 36(4): 320-325, 347. Tian Yunfang, Ye Yingying, Wu Changwen, et al. Genetic diversity and genetic structure of Meretrix lusoria population based on mitochondrial COI gene sequences[J]. Journal of Zhejiang Ocean University(Natural Science), 2017, 36(4): 320-325, 347. |
 2019, Vol. 43
2019, Vol. 43

