文章信息
- 张文燕, 潘红苗, 董逸, 杜海舰, 陈一然, 吴龙飞, 肖天. 2019.
- ZHANG Wen-yan, PAN Hong-miao, DONG Yi, DU Hai-jian, CHEN Yi-ran, WU Long-Fei, XIAO Tian. 2019.
- 基于高通量测序方法分析荣成月湖潮间带趋磁细菌多样性
- Diversity of magnetotactic bacteria in intertidal sediments of Lake Yuehu, Rongcheng
- 海洋科学, 43(3): 1-10
- Marine Sciences, 43(3): 1-10.
- http://dx.doi.org/10.11759/hykx20180925001
-
文章历史
- 收稿日期:2018-09-25
- 修回日期:2019-05-08
2. 青岛海洋科学与技术国家实验室海洋生态与环境科学功能实验室, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 中国科学院海洋大科学研究中心, 山东 青岛 266071;
5. 中法趋磁多细胞生物进化与发育联合实验室, 山东 青岛 266071;
6. 法国科研中心地中海微生物研究所, 法国 马赛 13009
2. Laboratory for Marine Ecology and Environmental Science, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071;
5. International Associated Laboratory of Evolution and Development of Magnetotactic Multicellular Organisms(LIA-MagMC), CNRS-CAS;
6. Aix Marseille University, CNRS, LCB, Marseille, France
趋磁细菌(Magnetotactic bacteria, MTB)是一类能够沿着磁力线运动的特殊细菌, 最早由Bellini和Blakemore独立发现[1-4]。趋磁细菌并不具有分类学意义, 在形态、生理代谢以及系统发育等方面具有多样性[5-9]。趋磁细菌形态多样, 常见有球形、杆状、弧形、螺旋形及多细胞聚集体形式(即多细胞趋磁原核生物)等。趋磁细菌生理代谢类型多样, 包括化能自养、化能异养等营养型及专性微好氧、专性或者兼性厌氧; 趋磁细菌系统发育地位也具有多样性, 目前, 已知的趋磁细菌隶属于变形菌门的α-变形菌纲(Alphaproteobacteria)、γ-变形菌纲(Gammaproteobacteria)、δ-变形菌纲(Deltaproteobacteria)以及硝化螺菌门(Nitrospirae)和暂定分类单元Omnitrophica[6, 9-10]。最近林巍等通过宏基因组分析的方法在暂定分类单元Latescibacteria和浮霉菌门(Planctomycetes)细菌的基因组中发现了趋磁细菌的磁小体岛基因, 证明这两个门中也可能存在趋磁细菌[11-12]。
趋磁细菌最重要的特征是体内可以生物成矿合成生物膜包裹的磁性纳米颗粒-磁小体(magnetosome)。趋磁细菌通过磁小体的导向作用, 借助鞭毛进行趋磁运动, 使其更有效地找到最适生存环境, 即有氧-无氧过渡区(oxic-anoxic transition zone, OATZ)[13-15]。趋磁细菌趋磁细菌在自然界分布极为广泛, 主要存在于淡水湖泊、海洋潮间带、潟湖、盐湖等环境, 在河流、土壤、大洋等环境中也有趋磁细菌的发现。趋磁细菌在自然环境中可能扮演着重要的生态角色, 参与铁、氮、硫、碳等元素的生物地球化学循环[6, 9, 12, 16]。由于趋磁细菌对营养的苛刻要求及微好氧等特性, 趋磁细菌的实验室分离纯化比较困难, 趋磁细菌纯培养菌株较少, 环境中的趋磁细菌多通过分析16S rRNA基因来确定它们的系统进化位置[12, 17-18]。
荣成月湖紧邻黄海, 是一个典型的天然潟湖, 是亚洲最大的天鹅冬季栖息地。该环境环境因子稳定变化, 沉积物界面稳定[19]。前期调查发现, 该区域潮间带沉积物中趋磁细菌丰度高, 种类丰富, 但克隆文库方法仅得到很少的遗传信息, 远远低于预期[20]。故本研究采用罗氏454高通量测序的方法, 对月湖沉积物和磁收样品中趋磁细菌的多样性进行研究, 以细菌群落结构作为背景, 预测和发现新的趋磁细菌种类。
1 材料与方法 1.1 样品采集及趋磁细菌形态观察 1.1.1 采样点及样品采集荣成月湖(36°43′~37°27′N, 122°09′~122°42′E)是一个半封闭天然潟湖, 面积约4.94 km2, 其北、西、南三侧均被陆地包围, 东部由沙坝与外海隔开, 有一条与外海相通的潮汐汊道。月湖是亚洲最大的天鹅冬季栖息地, 受潮汐、天鹅迁徙、人工养殖、淡水输入等活动影响[19]。
于2009年5月—2010年8月在荣成月湖北部潮间带(37°21′33.857″N, 122°34′41.571″E)进行样品采集, 采样底质均为砂质。利用自制工具采集黄黑交界面处的沉积物, 部分沉积物采集后放入–20℃保存备用(B_S); 部分沉积物装入采样瓶, 添加原位海水后带回实验室进行下一步研究。
1.1.2 趋磁细菌收集将沉积物样品分装500 mL塑料瓶中, 每瓶装入约150 mL泥样, 加入约150 mL水样, 共分装10瓶。用胶带将磁铁粘在瓶壁上(S极面向瓶壁), 30 min后, 用吸管吸取磁铁附近的样品1 mL左右, 混合加入20 mL的小玻璃瓶中, 用磁铁再次富集约30 min, 再用吸管吸取磁铁附近的液体1 mL左右[21]。显微镜观察后, 部分液体经8 000 r/min离心5min, 制备成为磁收样品(B_M), –20℃保存; 部分液体进行T-T收集用于电镜观察。
1.1.3 趋磁细菌形态观察使用Olympus BX51显微镜的微分干涉(Differential Interference Contrast, DIC)模式, 悬滴法观察磁收样品中趋磁细菌的有无。将磁收集的菌液后采用“T-T”法进一步富集趋磁细菌[22], 取富集的菌液滴铜网(北京中镜科仪技术有限公司)后, 用透射电子显微镜(HITACHI H8100, 中国海洋大学电镜室)对趋磁细菌形态进行观察。
1.2 细菌和趋磁细菌遗传多样性分析 1.2.1 沉积物总DNA提取利用土壤DNA提取试剂盒(MP, FastDNA Spin Kit for Soil)和核酸裂解仪(Thermo, Fast Prep TM FP120), 按说明书对沉积物样品(B_S)0.5 g和磁收样品(B_M)进行总DNA的提取。DNA抽提后, 利用分光光度计(GE, Nano Vue Plus)检测DNA浓度, 同时利用1%琼脂糖凝胶电泳进行DNA质量检测。
1.2.2 细菌16S rRNA基因的PCR扩增及测序采用细菌通用引物8F(5′-AGAGTTTGATCCTGG CTCAG-3′)[23]和533R(5′-TTACCGCGGCTGCTGGCA C-3′)[24]扩增16S rRNA基因的V1-V3高变区。PCR反应条件: 95℃预变性3 min, 30个循环(95℃变性30 s, 55℃退火30 s, 72℃延伸45 s), 最后72℃延伸10 min。PCR反应体系: 5×PCR buffer 4 μL; 2.5 mmol/L dNTP 2 μL; 5 μmol/L P1 (8F) 0.8 μL; 5 μmol/L P2 (533R) 0.8 μL; 5 U/μL Taq酶0.4 μL; DNA模板2 μL; ddH2O 10 μL。每个样品进行3个重复, 将同一样品PCR产物混合后经电泳检测。PCR产物利用2%琼脂糖凝胶回收和纯化, 经检测定量后构建文库, 利用Roche 454平台进行测序(上海美吉生物医药科技有限公司)。
1.3 数据分析 1.3.1 原始测序序列质量控制对原始数据进行处理得到优化序列[25], 包括以下4个步骤: (1)去除测序接头、标记、前引物等序列; (2)利用特异性引物信息去除扩增过程中产生的非特异性扩增片段序列(引物碱基含2个以上错配); (3)去除长度过短和过长(< 150 bp或 > 600 bp)的序列; (4)去除含有模糊碱基、单碱基高重复(单碱基重复超过6个)的序列。对优化序列进行数据及长度分布统计后进行下一步分析。
1.3.2 OTU聚类分析利用Mothur软件对优化序列以97%相似性水平做为阈值进行OTU(Operational taxonomic units)聚类[26], 生成OTU表格, 并得到每个OTU代表序列, 并进行信息统计分析, 使用UCHIME软件剔除嵌合体[27]。
1.3.3 稀释曲线和多样性指数利用Mothur做稀释曲线分析, 利用R语言工具制作曲线图, 用以检验样本的测序数据量是否合理[28]。使用Mothur软件进行丰富度和多样性指数计算分析, 分析微生物群落的丰富度和多样性[26]。丰富度指数包含ACE(http://www.mothur.org/wiki/Ace)[29]和Chao(http:// www.mothur.org/wiki/Chao)[30]; 多样性指数包括Simpson指数(http://www.mothur.org/wiki/Simpson)[31]和Shannon指数(http://www.mothur.org/wiki/Shannon)[32], 此外还有测序深度指数Coverage(http://www.mothur.org/wiki/Coverage)。所有分析在OTU相似水平97%(0.03)进行。
1.3.4 OTU分类学分析及趋磁细菌鉴定OTU代表序列与SILVA数据库(121版本)进行比对, 找出其最相近且可信度达80%以上的种属信息, 根据SILVA库中的参考序列对OTU进行种属鉴定, 将得到的结果记录在OTU表格文件中[33]。对得到的物种分类信息进行人工校正, 对各OTU的代表序列进行Local BLAST比对[34], 数据库选择GenBank核酸数据库(nt/nr), 从输出结果中筛选趋磁细菌相关序列。
1.3.5 系统进化树构建利用NCBI Blast search在线比对, 找出趋磁细菌相关序列在GenBank数据库中最相似的序列[34]。选择参照序列, 与本研究得到的序列一起用Clustal W软件进行比对, 并进行手工校正[35]。利用MEGA 6软件中的邻接法(Neighbor-Joining)构建系统发育树, 用Bootstrap法检验系统发育树, 自展系数为1 000[36-37]。
2 研究结果 2.1 趋磁细菌的形态多样性光学显微镜下用悬滴法观察, 可见对磁场敏感的趋磁细菌, 经换算, 沉积物中丰度可达103~104 cells/cm3。通过光学和透射电子显微镜观察菌体形态, 绝大多数趋磁细菌为球形与卵圆形, 也存在杆菌、弧菌、螺菌及多细胞趋磁原核生物(图 1)。趋磁细菌体内均发现磁小体, 其中, 球菌所含磁小体形状多为棱柱状, 可呈单链、双链或者不规则排列, 同时菌体内含多个致密颗粒。
 |
| 图 1 月湖潮间带沉积物中趋磁细菌形态 Fig. 1 TEM micrographs of individual MTB cells in intertidal sediments from Lake Yuehu 注:标尺: C和G, 500 nm; J, 2 μm, 其余1 μm |
表 1为2个沉积物样品的测序的统计数据, B_M和B_S样品测序的优化序列分别为27 991和21 794条, 序列平均长度接近, 均为454 bp左右。从样品的Coverage来看, 2个样品的覆盖率分别达到92.6%和87.7%, 能较好地反映样品菌群的整体情况。稀释曲线(Rarefaction curve)反映了样品的取样深度, 可以用来评价测序量是否足够。从图 2可知, 两样品在97%相似性水平下的稀释曲线逐渐趋于平缓, 随机测序数据可以覆盖绝大多数细菌的多样性, 能够较真实地反映该环境的细菌群落结构。此外, B_S样品相较B_M样品而言有一定的上升趋势, 表明在B_S比B_M样品可能存在更多的未知序列。
| 样品名称 | 优化序列数 | 序列平均长度 | OTUs数 | ACE | Chao | Shannon | Simpson | Coverage |
| B_M | 27 991 | 454.1 | 3 199 | 8 290 | 7 904 | 4.62 | 0.389 3 | 0.926 2 |
| B_S | 21 794 | 453.6 | 5 074 | 9 946 | 9 477 | 10.49 | 0.003 6 | 0.877 3 |
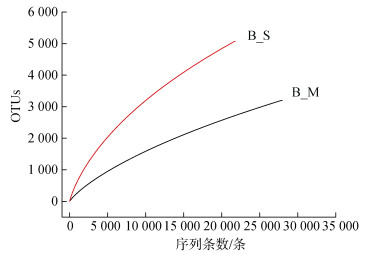 |
| 图 2 月湖潮间带沉积物样品稀释曲线(97%相似性水平) Fig. 2 Rarefaction curve of samples from intertidal sediments of Lake Yuehu (97% similarity level) |
对2个样品进行序列的OTU聚类, 其中B_S样品共得到5 074个细菌的OTUs, 而B_M样品含OTUs数量更少, 仅有3 199个OTUs; B_M的Shannon指数明显低于B_S, 分别为4.62和10.49, 而B_M样品的Simpson指数明显高于B_S(表 1), 说明B_S样品细菌多样性明显高于B_M样品; 同时, 丰富度指数ACE和Chao, 均为B_S样品高于B_M样品, 同样说明B_S样品有着更高的物种丰富度。磁收样品(B_M)的多样性和丰富度较沉积物样品(B_S)明显降低。
2.3 OTU分类地位及细菌群落多样性分析对2个样品的OTU与Silva数据库(128版本)对比进行分类学分析, B_S样品的5 074个OTUs属于30门、76纲、135目、152科、和241属; 而B_M样品的3 199个OTUs属于32门、66纲、116目、127科和197属。除门水平外, B_M样品含有的各分类阶元的总数均低于B_S样品。
在门和纲水平上对2个样品进行细菌群落结构分析, 2个样品的细菌主要类群比较一致, 但相对比例差别很大。如图 3所示, 在门水平细菌分类的统计分析发现, B_M样品中变形菌门(Proteobacteria)占绝对优势, 相对比例达88.0%, 其余门类比例均小于4%。B_S的优势度最高的也是变形菌门, 相对比例为51.5%。其次是拟杆菌门(Bacteroidetes)和浮霉菌门(Planctomycetes) (所占比例分别是10.2%和9.6%)。在纲水平, B_M样品中α-变形菌纲(Alphaproteobacteria)占绝对优势, 相对比例达72.6%, 其次是δ-变形菌纲(Deltaproteobacteria), 比例为4.4%。而B_S样品δ-变形菌纲、γ-变形菌纲(Gammaproteobacteria)和α-变形菌纲较多, 比例分别是26.4%、9.9%和8.8%。而在属水平上, B_M和B_S两个样品均为未分类的细菌比例最高, 且B_M样品远高于B_S样品(58.0%), 达到了90.7%。
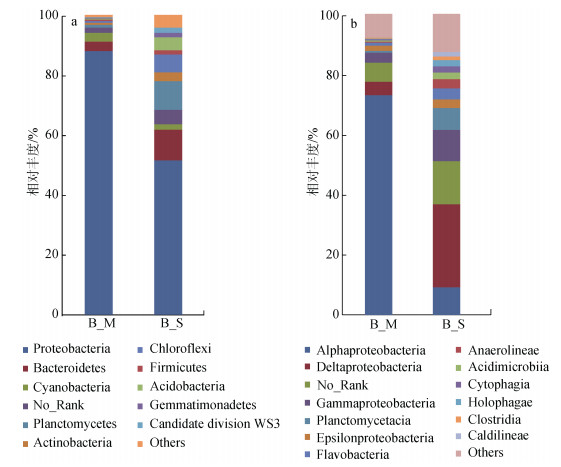 |
| 图 3 月湖潮间带沉积物细菌群落结构 Fig. 3 Community structures of bacteria in intertidal sediments from Lake Yuehu 注: a:门水平; b:纲水平 |
对所有OTU代表序列的分类信息进行手工校正, 筛选趋磁细菌相关序列。以相似性水平90%为阈值, 在样品B_M中发现242个与趋磁细菌相关的OTUs, 包含1 612条reads, 分别占细菌总OTUs和reads总数的7.56%和5.76%。而B_S中有40个OTUs和186条reads序列与趋磁细菌相关, 占细菌总OTUs数和reads数的0.79%和0.85%。B_M样品中的趋磁细菌reads数占总细菌reads数的比例, 即趋磁细菌相对丰度, 远高于B_S样品, 约为B_S样品的6.8倍, 大大提高了趋磁细菌的检出率。
对这些趋磁细菌相关序列进行分类地位统计(图 4)和系统发育分析(图 5), 这些序列多数属于变形菌门的α-变形菌纲, 分别占趋磁细菌总reads数的99.2%(B_M)和87.5%(B_S), 此外, 还有少数属于δ-变形菌纲的多细胞趋磁原核生物。趋磁细菌序列在属水平上的分类地位显示, B_M和B_S样品中趋磁球菌属(Magnetococcus)均占绝对优势, 比例分别达到了88.8%和77.5%。趋磁弧菌(Magnetovibrio)是B_M样品中第二优势类群(7.4%), 而多细胞趋磁原核生物(Magnetoglobus)和趋磁螺菌属(Magnetospirillum)在B_S样品中优势度较高, 比例分别为12.5%和7.5%, 海洋趋磁螺菌属(Magnetospira)仅在B_M样品中出现。可见, 两样品趋磁细菌的优势类群相同, 非优势类群的群落结构略有差别。
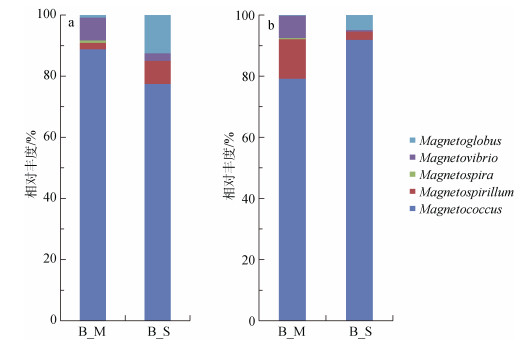 |
| 图 4 月湖潮间带沉积物趋磁细菌群落结构 Fig. 4 Community structures of magnetotactic bacteria in intertidal sediments from Lake Yuehu |
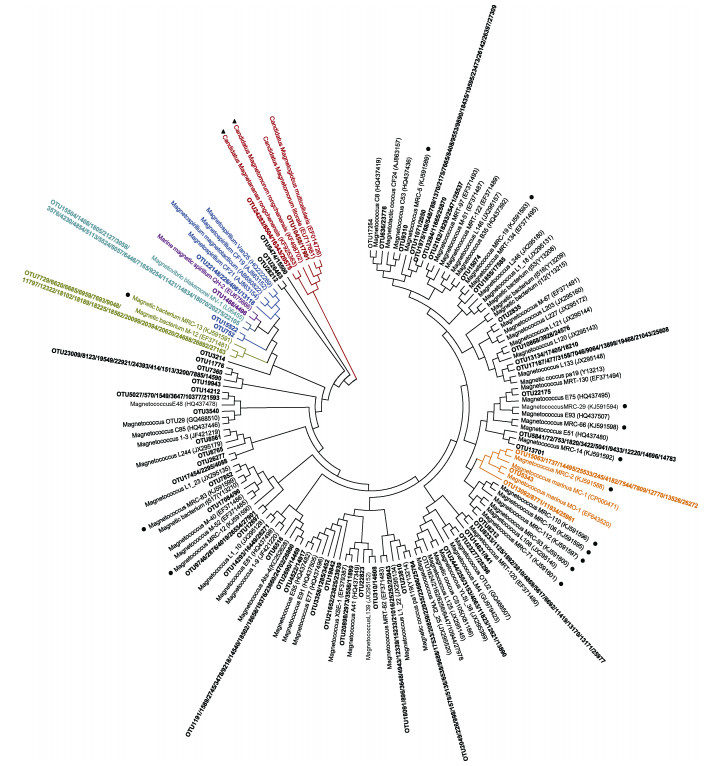 |
| 图 5 月湖潮间带沉积物中趋磁细菌的系统发育树 Fig. 5 Phylogenetic tree of magnetotactic bacteria in intertidal sediments from Lake Yuehu 注: ●月湖潮间带传统克隆文库测序得到的趋磁细菌序列; ▲显微操作调取获得的趋磁多细胞原核生物的序列 |
本文通过Roche 454高通量测序平台对荣成月湖潮间带沉积物进行了细菌与趋磁细菌多样性分析。沉积物样品(B_S)和磁收样品(B_M)两个样品分别获得了21 794和27 991条细菌优化序列, 分别归属于5 074个和3 199个OTUs。多样性指数分析显示, 磁收样品(B_M)的多样性和丰富度较沉积物样品(B_S)明显降低。通过对Silva数据库的对比, B_S中主要的细菌类群为δ-变形菌纲, 占总细菌数的26.4%, 其次是γ-变形菌纲和α-变形菌纲; 而B_M以α-变形菌纲占绝对优势, 相对比例达72.6%, 其次是δ-变形菌纲。与沉积物样品B_S相比较, 磁收样品B_M中细菌种类明显降低, 与多样性和丰富度指数结果也一致, 磁收通过磁场的作用对沉积物中的细菌进行选择性富集, 趋磁细菌和磁性颗粒附着细菌在此过程中比例会明显增加。通过与NCBI nt/nr数据库的比对, 在磁收样品(B_M)和沉积物样品(B_S)分别发现了1 612条和186条趋磁细菌相关序列, 分别占细菌总数的5.76%和0.85%, B_M样品中趋磁细菌数远高于B_S样品(6.8倍), 相较于原始的沉积物样品, 磁收样品中趋磁细菌的相对丰度明显增加。
对得到的趋磁细菌OTU进行系统分类和进化分析, 结果显示这些趋磁细菌序列多数属于变形菌门的α-变形菌纲, 两个样品中趋磁球菌属均占绝对优势, 还存在海洋趋磁螺菌属、趋磁弧菌属和趋磁螺菌属。此外, 还有少数属于δ-变形菌纲的多细胞趋磁原核生物。而在相同采样地点, 通过对沉积物中趋磁细菌的富集, 利用传统克隆文库和Sanger测序的方法研究趋磁细菌多样性, 共获得了29条趋磁细菌16S rDNA序列, 经聚类得到了14个OTUs, 其中13个OTUs属于α-变形菌纲的趋磁球菌, 1个OTU属于γ-变形菌纲(图 5, 黑色圆圈代表序列)[20]。而在此处较常见的多细胞趋磁原核生物由于丰度较低没有检出, 其16S rDNA序列和系统进化地位分析是通过显微操作挑取单细胞, 经基因组扩增后进行16S rDNA的二次扩增和文库测序得到的(图 5, 黑色三角代表序列)[38-39]。将本研究得到的趋磁细菌序列与克隆文库到的序列进行相似性分析, 属于α-变形菌纲的趋磁球菌的10个OTUs在本研究中检出。综上分析, 与克隆文库方法相比, 高通量测序方法得到的优势趋磁细菌类群一致, 可以较好地覆盖克隆文库的结果, 证明高通量测序方法可靠; 同时, 高通量测序方法得到了更多的趋磁细菌序列, 包含受关注的多细胞趋磁原核生物和其他趋磁细菌类群, 在趋磁细菌检出和多样性分析方面具有明显的优势。但两个样品中非优势趋磁细菌的群落结构略有不同, 可能是磁收对不同种类趋磁细菌的收集效率不一致所导致, 例如对轴向运动的趋磁螺菌和乒乓运动的多细胞趋磁原核生物, 磁收效率有待于进一步提高, 探索效率更高的方法收集趋磁细菌有待于提高。本研究中, 高通量测序(Roche 454)得到的序列片段较短, 只有500 bp左右, 只能覆盖16S rDNA的V1-V3高变区, 目前其他二代测序方法也难以获得16S rDNA序列的全长[40-41]。后期可以考虑利用PacBio等三代测序方法替代二代测序方法[42], 可以实现16S rDNA全长序列测序, 将会得到更多的趋磁细菌序列信息。
由于本实验所用的比对方法以已知的趋磁细菌为参照, 对新的趋磁细菌类型确定有限, 因此可能会大大低估趋磁细菌的多样性。我们尝试找到了磁收样品B_M和沉积物样品B_S的差异OTUs, 同时也对B_M样品中相对丰度显著增加的OTUs进行了分析, 以此预测新的趋磁细菌类群。共有和差异分析显示, 磁收样品B_M和沉积物样品B_S共有的OTUs仅为1 177个, 分别占B_M和B_S样品的36.8%和23.2%, 磁收样品中特有的OTUs有2 022个, 比例达到了63.2%, 磁收处理是收集趋磁细菌的有效方法, 该处理明显改变了样品中细菌的群落结构。OTU系统进化地位分析显示, 磁收样品中特有OTUs主要属于变形菌门的α-变形菌纲、δ-变形菌纲和γ-变形菌纲。为排除扩增子测序偏差的影响, 我们对这些OTUs中丰度相对较高(reads数大于10)的35个OTUs进行深入分析, 这些OTUs除一个分类地位未定外, 其余均属于变形菌门。其中, 19个OTUs与趋磁细菌相关, 16个未发现与趋磁细菌相关, 这些差异OTUs有可能是潜在的趋磁细菌新类群。同时, 我们找到了B_M样品中比例显著增加(> 3倍)的OTUs共58个, 系统分类和进化地位分析显示, 这些序列多数属于变形菌门的α-变形菌纲、γ-变形菌纲和δ-变形菌纲, 还有部分OTUs属于拟杆菌门、浮霉菌门和蓝细菌门; 与数据库中的已知趋磁细菌进行对比分析, 发现其中仅有4个OTUs与已知的趋磁细菌序列相关, 多数OTUs与未培养的细菌相似性最高。例如, OTU4, 其reads数在在B_S样品中仅为3.3%, 而B_M样品中为显著优势菌, 占总reads数的61.7%, 是B_S样品中相对丰度的19倍, 如此之高的差异极有可能是磁收造成的, 所以我们推测, OTU4及其他相对比例显著增加的OTUs也可能是新的趋磁细菌种类。目前在变形菌门的α-变形菌纲、γ-变形菌纲和δ-变形菌纲已发现了趋磁细菌, 也发现了浮霉菌门中存在趋磁细菌的新证据[12, 43], 而在拟杆菌门和蓝细菌门中是否存在新的趋磁细菌种类, 值得期待, 需要进行下一步的研究。
趋磁细菌体内磁小体的合成是受基因即磁小体岛基因控制的, 磁小体岛基因是趋磁细菌的特异功能基因, 研究表明, 趋磁细菌的磁小体岛可能是通过基因的横向转移得到的[8-9, 12, 43]。本论文方法主要基于趋磁细菌16S rRNA基因的分析, 由于16S rRNA基因和基因组尤其是磁小体岛的进化并不是完全平行的, 与趋磁细菌16S rRNA基因相似性很高的细菌有的可能不具有趋磁性, 并非趋磁细菌。相对于磁小体岛上的功能基因, 单纯以16S rRNA基因作为判断是否为趋磁细菌可能会不准确。另外, 磁收富集样品中也含有许多非趋磁细菌, 例如磁性颗粒附着细菌, 磁收样品B_M中特有的OTUs和比例显著增加的OTUs可能是新的趋磁细菌种类, 但我们目前无法排除磁性颗粒附着细菌的可能性。下一步, 我们将综合16S rRNA基因的扩增子测序和宏基因组测序中磁小体岛基因进行分析, 尝试发现或验证这些新的细菌种类。
综上所述, 本研究通过高通量测序平台对潮间带沉积物和磁收集样品进行测序, 了解沉积物细菌类群, 对趋磁细菌进行多样性和系统进化研究, 并预测新的趋磁细菌种类, 这些结果将为下一步发现和开发新的趋磁细菌类群提供参考结果。通过与传统方法比较, 在趋磁细菌多样性分析方面, 高通量测序方法效果优于传统方法, 具有明显的优势。
致谢: 本研究的样品由中国科学院海洋研究所徐剑虹老师协助采集, 电子显微镜的使用得到中国海洋大学姜明老师的帮助和支持, 在此向两位老师表示感谢。
| [1] |
Bellini S. On a unique behavior of freshwater bacteria[J]. Chinese Journal of Oceanology and Limnology, 2009, 27(1): 3-5. DOI:10.1007/s00343-009-0003-5 |
| [2] |
Bellini S. Further studies on "magnetosensitive bacteria"[J]. Chinese Journal of Oceanology and Limnology, 2009, 27(1): 6-12. DOI:10.1007/s00343-009-0006-2 |
| [3] |
Blakemore R.P. Magnetotactic bacteria[J]. Science, 1975, 190(4212): 377-379. DOI:10.1126/science.170679 |
| [4] |
Frankel R B. The discovery of magnetotactic/magnetosensitive bacteria[J]. Chinese Journal of Oceanology and Limnology, 2009, 27(1): 1-2. DOI:10.1007/s00343-009-0001-7 |
| [5] |
Spring S, Bazylinski D A. Magnetotactic Bacteria, in Dworkin M, Falkow S, Rosenberg E, Schleifer K-H, Stackebrandt E (Ed), The Prokaryotes (Vol. 2:Ecophysiology and Biochemistry)[J]. New York, NY, USA:Springer, 2006, 842-862. |
| [6] |
Amann R, Peplies J, Schüler D. Diversity and Taxonomy of Magnetotactic Bacteria, in Schüler D (Ed), Magnetoreception and Magnetosomes in Bacteria[M]. Berlin: Springer, 2006: 25-36.
|
| [7] |
Faivre D, Schüler D. Magnetotactic Bacteria and Magnetosomes[J]. Chemical Reviews, 2008, 108(11): 4875-4898. DOI:10.1021/cr078258w |
| [8] |
Jogler C, Schüler D. Genomics, genetics, and cell biology of magnetosome formation[J]. Annual Review of Microbiology, 2009, 63: 501-521. DOI:10.1146/annurev.micro.62.081307.162908 |
| [9] |
Lefevre C T, Bazylinski D A. Ecology, diversity, and evolution of magnetotactic bacteria[J]. Microbiology and Molecular Biology Reviews, 2013, 77(3): 497-526. DOI:10.1128/MMBR.00021-13 |
| [10] |
Kolinko S, Jogler C, Katzmann E, et al. Single-cell analysis reveals a novel uncultivated magnetotactic bacterium within the candidate division OP3[J]. Environmental Microbiology, 2012, 14(7): 1709-1721. DOI:10.1111/j.1462-2920.2011.02609.x |
| [11] |
Lin W, Pan Y. A putative greigite-type magnetosome gene cluster from the candidate phylum Latescibacteria[J]. Environmental Microbiology Reports, 2015, 7(2): 237-242. DOI:10.1111/1758-2229.12234 |
| [12] |
Lin W, Pan Y, Bazylinski D A. Diversity and ecology of and biomineralization by magnetotactic bacteria[J]. Environmental Microbiology Reports, 2017, 9(4): 345-356. DOI:10.1111/emi4.2017.9.issue-4 |
| [13] |
Uebe R, Schüler D. Magnetosome biogenesis in magnetotactic bacteria[J]. Nature Reviews Microbiology, 2016, 14(10): 621-637. DOI:10.1038/nrmicro.2016.99 |
| [14] |
Schüler D. Genetics and cell biology of magnetosome formation in magnetotactic bacteria[J]. FEMS Microbiology Reviews, 2008, 32(4): 654-672. DOI:10.1111/j.1574-6976.2008.00116.x |
| [15] |
Bazylinski D A, Frankel R B. Magnetosome formation in prokaryotes[J]. Nature Reviews Microbiology, 2004, 2(3): 217-230. DOI:10.1038/nrmicro842 |
| [16] |
Zhou K, Zhang W Y, Yu-Zhang K, et al. A novel genus of multicellular magnetotactic prokaryotes from the Yellow Sea[J]. Environmental Microbiology, 2012, 14(2): 405-413. DOI:10.1111/j.1462-2920.2011.02590.x |
| [17] |
Pradel N, Fuduche M, Ollivier B. Magnetotactic bacteria population in a pristine French Atlantic lagoon[J]. Environmental Science and Pollution Research, 2016, 23(1): 691-697. DOI:10.1007/s11356-015-5322-9 |
| [18] |
Dong Y, Li J, Zhang W, et al. The detection of magnetotactic bacteria in deep sea sediments from the east Pacific Manganese Nodule Province[J]. Environmental Microbiology Reports, 2016, 8(2): 239-249. DOI:10.1111/1758-2229.12374 |
| [19] |
牟晓燕, 杨永亮, 李春雁, 等. 山东半岛荣成湾月湖环境特征研究[J]. 青岛大学学报, 1999, 14: 56-59. Mu Xiaoyan, Yang Yongliang, Li Chunyan, et al. Environmental characteristics of seawater in the Yuehu inlet, Rongcheng Bay, China[J]. Journal of Qingdao University, 1999, 14: 56-59. |
| [20] |
Du H J, Chen Y R, Zhang R, et al. Temporal distributions and environmental adaptations of two types of multicellular magnetotactic prokaryote in the sediments of Lake Yuehu, China[J]. Environmental Microbiology Reports, 2015, 7(3): 538-546. DOI:10.1111/emi4.2015.7.issue-3 |
| [21] |
Wolfe R S, Thauer R K, Pfennig N. A 'capillary racetrack' method for isolation of magnetotactic bacteria[J]. FEMS Microbiology Ecology, 1987, 45: 31-35. DOI:10.1111/fml.1987.45.issue-1 |
| [22] |
周克.黄海沉积物多细胞趋磁原核生物的特性研究[D].北京: 中国科学院研究生院, 2010. Zhou Ke. Characterization of multicellular magnetotactic prokaryotes from coastal sediments of Yellow Sea[D]. Beijing: Graduate University of Chinese Academy of Sciences, 2010. |
| [23] |
Turner S, Pryer K M, Miao V P, et al. Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis[J]. The Journal of Eukaryotic Microbiology, 1999, 46(4): 327-338. DOI:10.1111/jeu.1999.46.issue-4 |
| [24] |
Huse S M, Dethlefsen L, Huber J A, et al. Exploring microbial diversity and taxonomy using SSU rRNA hypervariable tag sequencing[J]. PLoS Genetics, 2008, 4(11): e1000255. DOI:10.1371/journal.pgen.1000255 |
| [25] |
Bolger A.M., Lohse M., Usadel B. Trimmomatic:a flexible trimmer for Illumina sequence data[J]. Bioinformatics, 2014, 30(15): 2114-2120. DOI:10.1093/bioinformatics/btu170 |
| [26] |
Schloss P D, Westcott S L, Ryabin T, et al. Introducing mothur:open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [27] |
Edgar R C, Haas B J, Clemente J C, et al. UCHIME improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-200. DOI:10.1093/bioinformatics/btr381 |
| [28] |
Amato K R, Yeoman C J, Kent A, et al. Habitat degradation impacts black howler monkey (Alouatta pigra) gastrointestinal microbiomes[J]. The ISME Journal, 2013, 7(7): 1344-1353. DOI:10.1038/ismej.2013.16 |
| [29] |
Chao A, Lee S M. Estimating the number of classes via sample coverage[J]. Publications of the American Statistical Association, 1992, 87: 210-217. DOI:10.1080/01621459.1992.10475194 |
| [30] |
Chao A. Nonparametric Estimation of the Number of Classes in a Population[J]. Scandinavian Journal of Statistics, 1984, 11(4): 265-270. |
| [31] |
Simpson J M, Kocherginskaya S A, Aminov R I, et al. Comparative microbial diversity in the gastrointestinal tracts of food animal species[J]. Integrative and Comparative Biology, 2002, 42(2): 327-331. DOI:10.1093/icb/42.2.327 |
| [32] |
Magurran A E. Ecological diversity and its measurement[M]. Princeton, N.J., USA: Princeton University Press, 1988: 81-89.
|
| [33] |
Quast C, Pruesse E, Yilmaz P, et al. The SILVA ribosomal RNA gene database project:improved data processing and web-based tools[J]. Nucleic Acids Research, 2013, 41: 590-596. |
| [34] |
Altschul S F, Gish W, Miller W, et al. Basic local alignment search tool[J]. Journal of Molecular Biology, 1990, 215(3): 403-410. DOI:10.1016/S0022-2836(05)80360-2 |
| [35] |
Thompson J D, Higgins D G, and Gibson T J. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice[J]. Nucleic Acids Research, 1994, 22(22): 4673-4680. DOI:10.1093/nar/22.22.4673 |
| [36] |
Kumar S, Nei M, Dudley J, et al. MEGA:A biologist-centric software for evolutionary analysis of DNA and protein sequences[J]. Briefings in Bioinformatics, 2008, 9(4): 299-306. DOI:10.1093/bib/bbn017 |
| [37] |
Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular Evolutionary Genetics Analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197 |
| [38] |
Zhang R, Chen Y R, Du H J, et al. Characterization and phylogenetic identification of a species of spherical multicellular magnetotactic prokaryotes that produces both magnetite and greigite crystals[J]. Research in Microbiology, 2014, 165(7): 481-489. DOI:10.1016/j.resmic.2014.07.012 |
| [39] |
Chen Y R, Zhang R, Du H J, et al. A novel species of ellipsoidal multicellular magnetotactic prokaryotes from Lake Yuehu in China[J]. Environmental Microbiology, 2015, 17(3): 637-647. DOI:10.1111/emi.2015.17.issue-3 |
| [40] |
Shokralla S, Spall J L, Gibson J F, et al. Next-generation sequencing technologies for environmental DNA research[J]. Molecular Ecology, 2012, 21(8): 1794-1805. DOI:10.1111/j.1365-294X.2012.05538.x |
| [41] |
Metzker M L. Sequencing technologies-the next generation[J]. Nature Reviews Genetics, 2010, 11(1): 31-46. DOI:10.1038/nrg2626 |
| [42] |
Wagner J, Coupland P, Browne H P, et al. Evaluation of PacBio sequencing for full-length bacterial 16S rRNA gene classification[J]. BMC Microbiology, 2016, 16(1): 274. DOI:10.1186/s12866-016-0891-4 |
| [43] |
Lin W, Zhang W, Zhao X, et al. Genomic expansion of magnetotactic bacteria reveals an early common origin of magnetotaxis with lineage-specific evolution[J]. The ISME Journal, 2018, 12(6): 1508-1519. DOI:10.1038/s41396-018-0098-9 |
 2019, Vol. 43
2019, Vol. 43


