文章信息
- 信业宏, 商晓梅, 孙军. 2019.
- XIN Ye-hong, SHANG Xiao-mei, SUN Jun. 2019.
- 硅藻硅质化蛋白——亲硅蛋白(silaffins)
- State of the art of the diatom silica-forming protein silaffins
- 海洋科学, 43(3): 98-105
- Marine Sciences, 43(3): 98-105.
- http://dx.doi.org/10.11759/hykx20181224002
-
文章历史
- 收稿日期:2018-12-24
- 修回日期:2019-02-16
2. 天津科技大学 印度洋生态系统研究中心, 天津 300457
2. Research Centre for Indian Ocean Ecosystem, Tianjin University of Science and Technology, Tianjin 300457, China
硅藻是一类生活于海洋、淡水以及潮湿泥土中的单细胞真核藻类[1]。硅藻出现于距今至少1亿8千万年的侏罗纪早期, 它们不仅在白垩纪的物种大灭绝中存活了下来, 而且在新生代时期发生大量分化使其多样性更加丰富[2]。如今, 硅藻已经成为海洋浮游植物的主要群体, 占全球年碳固定量的20%[3], 是海洋生物硅的最大贡献者[3], 其在水生食物网、硅循环等全球生物地球化学循环以及海洋环境中都发挥至关重要的作用[3-6]。硅藻具有独特且多样化的硅质壁, 这被认为是其生存并大量繁殖进化的关键。
很多生物(水稻, 放射虫, 领鞭虫、海绵等)中都有关于硅的生物矿化的报道, 但硅藻中的生物矿化独一无二, 很多硅藻中的硅质化相关蛋白并没有在硅藻以外的物种中发现[7]。硅藻硅质壁是一种有机—无机复合材料[8], 其亚结构的合成需要很多不同的蛋白调节[9]。目前分离到的与硅质壁形成相关的蛋白有:硅体蛋白(frustulins)[10]、侧壁蛋白(pleuralins)[11]、微环蛋白(cingulins)[12]、酸性沉硅蛋白(silacidins)[13]以及亲硅蛋白[14]等。Shrestha和Smith等[15-16]在研究中也发现了很多与上述基因共表达的类似基因, 例如类亲硅蛋白响应基因(Silaffin-Like Response Gene, SLRG)。
目前硅藻硅质壁的特殊组成及其形态的多样性是受关注与研究的热点, 在硅藻硅质壁中分离到的第一类可以在体外沉淀SiO2的蛋白是亲硅蛋白, 它是在硅藻硅质壁中鉴定到的除长链多胺(long chain polyamines, LCPAs)以外的第二大类生物分子[15], 其多样性与硅藻硅质壁形态的多样性密不可分。亲硅蛋白在硅藻硅质壁的形成过程中具有很大的作用[17]。每种硅藻都具有一套特异的多胺和亲硅蛋白。不同链长的长链多胺与亲硅蛋白或类似亲硅蛋白的多肽相互作用可以产生不同形态的SiO2沉淀[18]。人们通过仿生的方法将亲硅蛋白应用到了生物技术及医学领域, 其在固定化酶以及靶向药物设计方面的应用有很大的前景[15], 下面本综述将从分离鉴定、功能以及其在生物技术和生物医药方面的应用等方面介绍亲硅蛋白。
1 亲硅蛋白的分离亲硅蛋白是一类定位于硅沉积囊泡(silica deposition vesicles, SDVs)中的可溶性蛋白, 它最终会和沉积的SiO2一起嵌入到形成的硅质壁中[19]。亲硅蛋白最初由Kröger等[20]分离于纺锤柱鞘藻(Cylindrotheca fusiformis)的硅质壁中。氧化氢(HF)可以溶解硅藻硅质壁中的SiO2而对肽键等化学键无影响[10]。用无水HF处理硅藻的硅质壁可以释放与硅质壁结合紧密的有机物, 其中就包括侧壁蛋白[21]以及亲硅蛋白[20], 利用SDS-PAGE电泳可以将这些蛋白分离开来。最初在纺锤柱鞘藻硅质壁中分离到的亲硅蛋白有三种, 分别为silaffin-1A、silaffin-1B和silaffin-2, 其相对分子质量分别为4 kDa、8 kDa、17 kDa[17]。
silaffin-1A和silaffin-1B都是亲硅蛋白前体蛋白sil1p的水解产物。sil1p由265个氨基酸残基组成, 其中N末端的19个氨基酸残基组成用于转移到内质网上的信号肽; 第20-107个氨基酸残基为功能未知的N端结构域; 第108-265个氨基酸残基为强碱性且具有高度重复性的C末端(图 1)[20]。sil1p中含有较多的RXL基序, 是仅在硅质化相关蛋白中发现的水解位点[19], 这种基序还广泛存在于硅体蛋白、微环蛋白以及酸性沉硅蛋白的前体蛋白中[13, 22]。
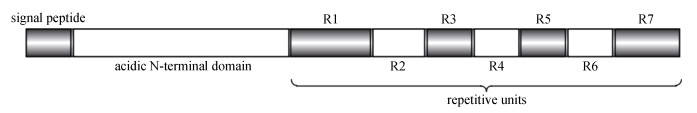 |
| 图 1 纺锤柱鞘藻中亲硅蛋白前体蛋白sil1p的示意图, 包括N端信号肽(signal peptide)、N端酸性结构域(acidic N-terminal domain)、7个重复度较高的多肽单位(repetitive units R1~R7) Fig. 1 The map shows sil1p (the precursor protein of silaffin in Cylindrotheca fusiformis), which includes the N-terminal signal peptide, the acidic N-terminal domain, and seven repetitive units (R1~R7) |
目前已经在包括假微型海链藻(Thalassiosira pseudonana)、纤细角毛藻(Chaetoceros gracilis)等至少4种硅藻种分离得到了亲硅蛋白[20, 23-25]。Poulsen等[21]用Kröger等[20]的研究方法在假微型海链藻的硅质壁中分离得到称为“HF处理后亲硅蛋白(HF-treated silaffins)”的蛋白, 对其进行了化学分析, 并通过克隆得到的cDNA序列推测出亲硅蛋白的前体多肽: tpSilp1p、tpSilp2p、tpSilp3p和tpSilp4p。其中tpSil1p和tpSil2p会合成一个称为tpSil1/2的多肽, tpSil1/2后被蛋白水解酶水解为H和L两种蛋白[23]。Poulsen等[21]的研究中没有将这种蛋白直接称为亲硅蛋白可能是由于很难说明利用HF溶解硅质壁中的二氧化硅来释放有机成分的方法分离得到的蛋白质为原位蛋白。Syah等[24]在纤细角毛藻硅质壁中分离亲硅蛋白时使用到了氟化铵(ammonium fluoride, NH4F)替代HF, 将分离得到的亲硅蛋白称为“原位亲硅蛋白(native silaffins, natSil)”。
亲硅蛋白分化差异导致其同源保守性较差[9]。其中在纺锤柱鞘藻与假微型海链藻中就发现了4种表达亲硅蛋白的基因, 它们共可以表达9种不同的亲硅蛋白[20, 23]。虽然这些蛋白都富含丝氨酸与赖氨酸, 且具有相似的翻译后修饰, 但它们的性质却不尽相同, 在体外情况下形成的SiO2沉淀也形态各异。这为固定化酶及靶向药物设计特定形状的SiO2牢笼提供了理论基础。
2 亲硅蛋白的翻译后修饰及序列特征很多硅质化相关蛋白的成熟都要经过翻译后修饰[8], 亲硅蛋白也不例外, 需要经过磷酸化、甲基化、多胺附着、糖基化、硫化、蛋白水解等修饰后才可以发挥其功能[20, 23, 25-26]。亲硅蛋白沉淀SiO2的能力与它复杂的翻译后修饰有关, 其前体蛋白N末端的信号肽可以引导它转移至内质网和高尔基体中进行磷酸化和糖基化等修饰。
纺锤柱鞘藻中, silaffin-1A会被水解为silaffin- 1A1和silaffin-1A2。silaffin-1A1具有完整的翻译后修饰, 可以在体外pH=5.5的硅酸溶液中沉淀SiO2[25]。silaffin-1A具有赖氨酸修饰但不具有磷酸化的丝氨酸, 不能独自在体外沉淀SiO2, 但如果在硅酸溶液中加入磷酸阴离子, 它沉淀SiO2的活性就会被激活, 实验证明在pH=5条件下它具有最高的活性[26]。
根据纺锤柱鞘藻亲硅蛋白前体蛋白sil1p人工合成的R5肽没有进行任何翻译后修饰, 在pH < 6的硅酸溶液以及SDVs中没有沉淀SiO2的活性[27]。因为SDVs中环境为酸性, 而且亲硅蛋白是在SDVs内开始聚合SiO2的[28], 由此可以推测亲硅蛋白上丝氨酸的修饰至关重要。值得注意的是, 在体外pH > 6的条件下R5肽能以可控的方式结合沉积SiO2[29], 这使得R5肽能够广泛应用于固定化酶以及靶向药物的研制中。
提取硅藻硅质壁中的有机物时用NH4F代替HF处理硅质壁[30], 根据分离得到的natSil翻译后修饰保持不变发现, 亲硅蛋白上所有的丝氨酸羟基都发生了磷酸化[25]。磷酸化的丝氨酸修饰赋予了亲硅蛋白不依赖磷酸根就可以在体外沉淀SiO2的能力。
相较于丝氨酸, 赖氨酸位点上的翻译后修饰会更加复杂, 包括了甲基化、羟基化和磷酸化[26, 31], 人们对于这些翻译后修饰的功能研究得并不透彻, 因此目前并不能准确得知这些修饰的功能。
假微型海链藻中, 相较于tpSil1/2L, tpSil1/2H富含羟基氨基酸, 具有硫酸基团的修饰且碳水化合物的含量较高[23]。而tpSil1/2L具有较多的磷酸化修饰[23]。通过实验发现, 当硅酸溶液中的tpSil1/2L浓度增加时, 其沉淀SiO2的能力会随之一直增加。而当硅酸溶液中的tpSil1/2H到达一定的浓度后它沉淀二氧化硅的能力则会受到抑制[23]。由此可以推测, 亲硅蛋白上的磷酸化修饰会促进蛋白对SiO2的聚合, 硫酸基团的修饰和糖基化则会抑制这种活性[23]。这一推测在对纺锤柱鞘藻中natSil-2与natSil-1A的研究中得到了验证[30], 硅藻的这种翻译后修饰的机制可以起到调节自身SiO2聚合的作用。
Polsen等[22]通过对假微型海链藻中亲硅蛋白的研究发现了一个戊赖氨酸基序, 在12-14个氨基酸残基中存在五个不连续的赖氨酸, 这是一种硅靶向基序[9]。
假微型海链藻tpSil3中的赖氨酸会以KXXK样式的四肽排列[32], 这种KXXK基序在纺锤柱鞘藻的亲硅蛋白和微环蛋白中也有发现, 可能是翻译后修饰相关酶类识别的基序[14, 25]。
虽然, 氨基酸序列比较是评估蛋白质能力的一种重要方法, 但在硅藻硅质壁的形成中, 蛋白质的翻译后修饰也起到了很大的作用。
3 不同亲硅蛋白对SiO2的沉积SiO2最初形成于SDVs中[28], LCPAs和亲硅蛋白可以直接参与SiO2沉淀的形成。体外实验中, 不仅是多胺的结构和磷酸盐比例决定了SiO2微粒的最终尺寸, 相同条件下, 不同的亲硅蛋白也可以形成形态各异的SiO2微粒[33]。总之多种分子的相互协调才形成了硅藻精细的硅质壁结构。
Kröger等[17]对silaffin-1A及亲硅蛋白的混合物在硅酸溶液中沉淀SiO2的现象进行了研究[20]。silaffin-1A在硅酸溶液中形成的SiO2颗粒的直径为500~700 nm(图 2 a), 亲硅蛋白混合物所形成的颗粒为20 nm(图 2 b)[20]。silaffin-2无法独自在硅酸溶液中沉淀SiO2, 但通过调节它与silaffin-1A在硅酸溶液的浓度及比例可以形成形态各异的SiO2沉淀, 这说明silaffin-2可能通过与其他硅质化相关蛋白相互作用参与到硅藻硅质壁的形成中, 它可能在硅质化的过程中扮演着支架的角色[30]。
 |
| 图 2 不同亲硅蛋白体外情况下沉积SiO2的电镜照片 Fig. 2 Electron microscopic photographs of SiO2 deposited by different silaffins in vitro 注: a: silaffin-1A在硅酸溶液中沉积的SiO2微粒[20]; b:纺锤柱鞘藻中亲硅蛋白的混合物对SiO2的沉积[20]; c:假微型海链藻中20 μmol/L tpSil1/2L和LCPAs沉积的SiO2[23]; d: 3.5 μmol/L tpSil1/2H和LCPAs沉积的SiO2[23]; e: 20 μmol/L tpSil3和LCPAs沉积的SiO2[23]; f: 30 μmol/L tpSil3和LCPAs沉积的SiO2[23]; g:纤细角毛藻中的亲硅蛋白在TEOS溶液中沉积的SiO2微粒[24]) |
在假微型海链藻中分离得到的亲硅蛋白都没有独自在体外硅酸溶液中沉淀SiO2的能力, 但当在硅酸溶液中加入LCPAs后, 它们沉淀SiO2的活性会被激活[23]。这种现象被称为多胺诱导的SiO2沉析(polyamine-induced silica precipitation)[18]。LCPAs本身具有在体外沉淀SiO2的能力, 但亲硅蛋白的存在可以使其形成的SiO2沉淀形态更具多样性[33]。tpSil1/2L形成规则的球形SiO2微粒, 在蛋白浓度为10 μmol/L时形成直径为230±25 nm的SiO2微粒, 当蛋白浓度增加至100 μmol/L时, 它所形成微粒的直径会增加到2.6±1.7 μm[23]。tpSil1/2H与tpSil3都会在硅酸溶液中形成形状不规则的SiO2沉淀(图 2 e, f)。并且到达一定浓度界限后, tpSil1/2H和tpSil3对SiO2聚合的能力会随着浓度的增加受到抑制[23]。
在纤细角毛藻中分离得到的亲硅蛋白可以以正硅酸乙酯(Tetraethyl orthosilicate, TEOS)为底物沉淀SiO2, 形成球状的SiO2微粒(图 2 g)[24]。
随着更多种类的亲硅蛋白被发现, 多肽在硅藻SiO2形成中的作用机理也被慢慢揭示。通过对这种机理的理解, 人们逐渐将仿生SiO2材料应用到了越来越多的领域[7]。
4 仿生SiO2材料和亲硅蛋白在生物技术及医学领域的应用SiO2具有稳定的化学及物理特性, 人工合成的SiO2材料可以具有明确的结构及特性, 因此其应用范围十分广泛[16]。人工合成SiO2需要非常苛刻的反应条件, 仿生SiO2的发展解决了这一问题, 所以近年来仿生SiO2材料越来越多地被应用在生物技术及医学领域[16]。
仿生SiO2材料出现之前, 一种名为MCM-41 (Mobile Crystalline Material-41)的有序介孔SiO2材料(Mesoporous Silica Nanoparticles, MSNs)已经在生物技术及医药方面的应用取得了突破性的进展[34-40]。作为古代硅藻的遗骸, 硅藻土也被广泛应用于吸附剂、天然杀虫剂、绝缘材料、废水处理中的过滤助剂以及光催化反应中的催化剂载体[41-45]。存活硅藻硅质壁也具有和MSNs一样的特性, 在生物传感、生物分离、显示设备、电化学检测等方面有着很好的应用前景[16, 46-49]。
亲硅蛋白和LCPAs可以在体外情况下进行SiO2的聚合, 通过对它们结构及其在SiO2聚合中作用的研究, 人们开始了对仿生SiO2材料的探索[50]。将硅藻硅质壁形成机理的理解转移到体外工艺中有助于开发出具有明确结构的新型SiO2材料。在室温下根据不同的条件、人工多肽及添加剂可以形成很多不同形状的SiO2[33, 51-59], 例如:利用聚丙烯亚胺(PPI)与一些胺端的树枝状分子可以形成仿生二氧化硅微粒, 通过改变聚丙烯亚胺与树枝状分子在溶液中的比例形成大小不同的二氧化硅纳米球[53]; 通过亲硅蛋白、R5肽以及线性或者环状的多胺可以获得具有优秀热力学结构的球形二氧化硅[32, 53]; 静态条件下, 聚-L-赖氨酸(PLL)可以触发球形或者六边形二氧化硅的形成[54-55], 当存在一定的反应或者静电场干扰时, 二氧化硅的形状会有所改变, 变为纤维状、树突状或者具有周期性孔隙的梯形[56]; 最近的研究表明, 利用大分子量的PLL可以形成与MCM-41媲美的介孔二氧化硅材料, 并且这种方法不需要苛刻的反应条件[57]; 人工合成的八肽——兰瑞肽(Lanreotide)可以自组装成纳米管, 能够用来生产双壁二氧化硅纳米管[58]; 利用聚环乙亚胺(PEI)可以在磷酸缓冲液中形成完美球形的二氧化硅颗粒, 添加一定量的甲醛溶液可以形成弥散的二氧化硅微粒[59]。
仿生SiO2在生物技术方面应用最突出的例子是酶类等敏感生物分子的固定化[60-62]。将生物分子固定在具有机械稳定性和化学惰性的SiO2中可以延长生物分子的寿命, 以便这些分子在生物传感、生物催化或药物传递中重复使用。例如利用PEI对多亚基酶苯丙氨酸解胺酶的固定化, 可以有效地防止酶蛋白亚基的脱落[61]。除了保持内部分子的活性, 还可以用固态核磁共振(Soild State NMR)对包裹在规则结构中分子的结构进行研究[62]。
仿生SiO2对酶类固定化的方法的主要缺陷是随机性, 不能保证高效且均匀的装载。通过二硫键将货物分子与亲硅蛋白中的R5肽共价结合, 并通过pH或者氧化还原反应控制货物分子的结合与释放, 同样是一种高效的酶固定化手段[63]。利用基因融合的手段, 将R5肽与靶酶类等蛋白质共价结合, 形成靶蛋白-亲硅蛋白嵌合体, 从而将自己嵌入到形成的SiO2颗粒中, 可以克服普通的固定化方法固定不均匀的缺点[64]。这一理论已经被应用到了生物传感器等方面[65-66]。
5 展望亲硅蛋白沉积二氧化硅主要依靠其特异的结构域和富有特色的翻译后修饰共同调节[9, 20, 23, 25-26, 30], 但其作用机制仍然未知或处于推测阶段, 需要对亲硅蛋白的基因特征、同源序列和保守性结构域等进行深入研究。不同硅藻中大量亲硅蛋白的分离及鉴定将有利于探究其遗传进化关系, 能够更好地理解硅藻硅质壁多样性的成因, 加快揭开硅藻硅质壁形成这一神秘面纱。亲硅蛋白存在于硅藻的硅质壁和SDVs中[19], 分离得到研究SDVs中的亲硅蛋白, 将有助于我们更清晰、更全面地认识其功能。发现和鉴定更多亲硅蛋白将帮助我们更好的实现亲硅蛋白在仿生SiO2材料中的应用。
| [1] |
金德祥, 黄凯歌, 陈金环, 等. 中国海洋浮游硅藻类[M]. 上海: 上海科学技术出版社, 1965. Jin Dexiang, Huang Kaige, Chen Jinhuan, et al. Marine Planktonic Diatoms in China[M]. Shanghai: Shanghai Science Press, 1965. |
| [2] |
Tirichine L, Rastogi A, Bowler C. Recent Progress in Diatom Genomics and Epigenomics[J]. Current Opinion in Plant Biology, 2017(36): 46-55. |
| [3] |
Nelson D M, Tréguer P, Brzezinski M A, et al. Production and Dissolution of Biogenic Silica in the Ocean:Revised Global Estimates, Comparison with Regional Data and Relationship to Biogenic Sedimentation[J]. Global Biogeochemical Cycles, 1995, 9(3): 359-400. DOI:10.1029/95GB01070 |
| [4] |
刘凤娇, 李顺兴, 郑凤英, 等. 近海污染物对海洋浮游植物生长及生化组成影响的比较研究[J]. 海洋科学, 2014, 38(5): 66-71. Liu Fengjiao, Li Shunxing, Zheng Fengying, et al. Comparison of the effects of coastal pollutants on the growth and biochemical composition of marine phytoplankton[J]. Marine Sciences, 2014, 38(5): 66-71. |
| [5] |
张丽君, 臧家业, 马永星, 等. 钱塘江河口溶解硅的变化与影响研究[J]. 海洋科学, 2015, 39(8): 51-57. Zhang Lijun, Zang Jiaye, Ma Yongxing, et al. Variability and influence of dissolved silica in the Qiangtangjiang Estuary[J]. Marine Sciences, 2015, 39(8): 51-57. |
| [6] |
黄玥. 广西钦州湾外湾表层沉积硅藻分布特征[J]. 海洋科学, 2017, 41(1): 96-103. Huang Yue. Distribution of the surface sediment diatoms in the outer bay of Qinzhou bay of Guangxi[J]. Marine Sciences, 2017, 41(1): 96-103. DOI:10.3969/j.issn.1671-6647.2017.01.010 |
| [7] |
Makoto O, Shungo S, Yoshio H, et al. Peptides for Silica Precipitation:Amino Acid Sequences for Directing Mineralization[J]. Protein & Peptide Letters, 2018, 25(1): 15-24. |
| [8] |
Rodríguez M. Molecular mechanism for biomineralization in a eukaryotic single-celled organism:pentalysine clusters mediate silica targeting of silaffins in Thalassiosira pseudonana[J]. Journal of Biological Chemistry, 2013, 288(28): 20110-20110. DOI:10.1074/jbc.P113.469379 |
| [9] |
Brembu T, Chauton M S, Winge P, et al. Dynamic responses to silicon in Thalasiossira pseudonana-identification, characterisation and classification of signature genes and their corresponding protein motifs[J]. Scientific Reports, 2017, 7(1): 4865. DOI:10.1038/s41598-017-04921-0 |
| [10] |
梁君荣, 陈丹丹, 高亚辉, 等. 海洋硅藻硅质细胞壁结构的形成机理研究概述[J]. 海洋学报, 2010, 32(5): 1-8. Liang Junrong, Chen Dandan, Gao Yahui, et al. A review on the process and mechanism of marine diatom silica structure formation[J]. Acta Oceanologica Sinca, 2010, 32(5): 1-8. DOI:10.3969/j.issn.1009-5470.2010.05.001 |
| [11] |
Sanctis S D, Wenzler M, Kröger N, et al. PSCD domains of pleuralin-1 from the diatom cylindrotheca fusiformis:NMR structures and interactions with other biosilica-associated proteins[J]. Structure, 2016, 24(7): 1178-1191. DOI:10.1016/j.str.2016.04.021 |
| [12] |
Scheffel A, Poulsen N, Shian S, et al. Nanopatterned protein microrings from a diatom that direct silica morphogenesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(8): 3175-3180. DOI:10.1073/pnas.1012842108 |
| [13] |
Kirkham A R, Richthammer P, Schmidt K, et al. A role for the cell-wall protein silacidin in cell size of the diatom Thalassiosira pseudonana[J]. Isme Journal, 2017, 11(11). |
| [14] |
Carolin L, Christian B. Silaffins in silica biomineralization and biomimetic, silica precipitation[J]. Marine Drugs, 2015, 13(8): 5297-5333. DOI:10.3390/md13085297 |
| [15] |
Shrestha R, Tesson B, Nordenkrichmar T, et al. Whole transcriptome analysis of the silicon response of the diatom Thalassiosira pseudonana[J]. Bmc Genomics, 2012, 13(1): 499-499. DOI:10.1186/1471-2164-13-499 |
| [16] |
Smith S R, Glé C, Abbriano R M, et al. Transcript level coordination of carbon pathways during silicon starvation-induced lipid accumulation in the diatom Thalassiosira pseudonana[J]. New Phytologist, 2016, 210(3): 890-904. DOI:10.1111/nph.13843 |
| [17] |
Igor P, Kirill G. Silaffins of diatoms:from applied biotechnology, to biomedicine[J]. Marine Drugs, 2013, 11(9): 3155-3167. DOI:10.3390/md11093155 |
| [18] |
Kröger N, Deutzmann R, Bergsdorf C, et al. Species-specific polyamines from diatoms control silica morphology[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(26): 14133-14138. DOI:10.1073/pnas.260496497 |
| [19] |
Tesson B, Lerch S J L, Hildebrand M. Characterization of a new protein family associated with the silica deposition vesicle membrane enables genetic manipulation of diatom silica[J]. Scientific Reports, 2017, 7(1): 13457. DOI:10.1038/s41598-017-13613-8 |
| [20] |
Kröger N. Polycationic peptides from diatom biosilica that direct silica nanosphere formation[J]. Science, 1999, 286(5442): 1129-1132. DOI:10.1126/science.286.5442.1129 |
| [21] |
Kröger N, Lehmann G, Rachel R, et al. Characterization of a 200-kDa diatom protein that is specifically associated with a silica-based substructure of the cell wall[J]. European journal of biochemistry/FEBS, 1997, 250(1): 99-105. DOI:10.1111/ejb.1997.250.issue-1 |
| [22] |
Wenzl S, Hett R, Richthammer P, et al. Silacidins:highly acidic phosphopeptides from diatom shells assist in silica precipitation in vitro[J]. Angewandte Chemie International Edition, 2008, 47: 1729-1732. DOI:10.1002/(ISSN)1521-3773 |
| [23] |
Poulsen N, Kröger N. Silica Morphogenesis by alternative processing of silaffins in the diatom Thalassiosira pseudonana[J]. Journal of Biological Chemistry, 2004, 279(41): 42993-42999. DOI:10.1074/jbc.M407734200 |
| [24] |
Manurung A I, Pratiwi A R, Syah D, et al. Isolation and characterization of silaffin that catalyze biosilica formation from marine diatom Chaetoceros gracilis[J]. Hayati Journal of Biosciences, 2007, 14(3): 119-122. DOI:10.4308/hjb.14.3.119 |
| [25] |
Kröger N, Deutzmann R, Sumper M, et al. Silica-precipitating peptides from diatoms the chemical structure of silaffin-1A from Cylindrotheca Fusiformis[J]. Journal of Biological Chemistry, 2001, 276(28): 26066-26070. DOI:10.1074/jbc.M102093200 |
| [26] |
Kröger N, Lorenz S, Brunner E, et al. Self-assembly of highly phosphorylated silaffins and their function in biosilica morphogenesis[J]. Science, 2002, 298(5593): 584-586. DOI:10.1126/science.1076221 |
| [27] |
Senior L, Crump M P, Williams C, et al. Structure and function of the silicifying peptide R5[J]. Journal of Materials Chemistry B, 2015, 3(13): 2607-2614. DOI:10.1039/C4TB01679C |
| [28] |
Li C W, Chu S, Lee M, et al. Characterizing the silica deposition vesicle of diatoms[J]. Protoplasma, 1989, 158-163. |
| [29] |
Deptuch T, Damskozlowska H. Silk materials functionalized via genetic engineering for biomedical applications[J]. Materials, 2017, 10(12). |
| [30] |
Poulsen N, Sumper M, Kröger N, et al. Biosilica formation in diatoms:characterization of native silaffin-2 and its role in silica morphogenesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(21): 12075-12080. DOI:10.1073/pnas.2035131100 |
| [31] |
Nakajima T, Volcani B E. Epsilon-N-trimethyl-L-delta-hydroxysine phosphate and its nonphosphorylated compound in diatom cell walls[J]. Biochemical & Biophysical Research Communications, 1970, 39(1): 28-33. |
| [32] |
Sumper M, Hett R, Lehmann G, et al. A code for lysine modifications of a silica biomineralizing silaffin protein[J]. Angewandte Chemie, 2007, 46(44): 8405-8408. DOI:10.1002/(ISSN)1521-3773 |
| [33] |
Sumper M, Kroger N. Silica formation in diatoms:the function of long-chain polyamines and silaffins[J]. Journal of Materials Chemistry, 2004, 14(14): 2059-2065. DOI:10.1039/B401028K |
| [34] |
张金花.介孔二氧化硅MCM-41的吸附性能与生物安全性研究[D].天津: 天津工业大学, 2011. Zhang Jinhua. Adsorption and biosafety of mesoporous silica MCM-41[D]. Tianjin: Tianjin Polytechnic University, 2011. http://d.wanfangdata.com.cn/Thesis/Y1879463 |
| [35] |
Muñoz B, Rámila A, Perez-Pariente J P, et al. MCM-41 organic modification as drug delivery rate regulator[J]. Chemistry of Materials, 2012, 15(2): 500-503. |
| [36] |
Tournepeteilh C, Brunel D, Begu S, et al. Synthesis and characterisation of ibuprofen-anchored MCM-41 silica and silica Gel[J]. New Journal of Chemistry, 2003, 27(10): 1415-1418. DOI:10.1039/B307046H |
| [37] |
赵岚, 朱广山, 张大粱, 等. 孔壁部分有序化的介孔二氧化硅材料的合成与表征[J]. 高等学校化学学报, 2006, 27(2): 208-211. Zhao Lan, Zhu Guangshan, Zhang Daliang, et al. Synthesis and characterization of mesoporous silica material with partly-ordered pore walls[J]. Chemical Journal of Chinese University, 2006, 27(2): 208-211. DOI:10.3321/j.issn:0251-0790.2006.02.003 |
| [38] |
Li H, He J, Zhao Y, et al. Immobilization of glucose oxidase and platinum on mesoporous silica nanoparticles for the fabrication of glucose biosensor[J]. Electrochimica Acta, 2011, 56(7): 2960-2965. DOI:10.1016/j.electacta.2010.12.098 |
| [39] |
Aghayan M, Mahmoudi A, Sazegar M R, et al. Enzymatic activity of Fe-grafted mesoporous silica nanoparticles:an insight into H2O2 and glucose detection[J]. New Journal of Chemistry, 2018, 42(19): 16060-16068. DOI:10.1039/C8NJ03534B |
| [40] |
Soleymani J. Advanced materials for optical sensing and biosensing of neurotransmitters[J]. Trends in Analytical Chemistry, 2015, 27-44. |
| [41] |
刘洁, 赵东风. 硅藻土的研究现状及进展[J]. 环境科学与管理, 2009, 34(5): 104-106. Liu Jie, Zhao Dongfeng. The present situation and development of diatomite[J]. Environmental Science and Management, 2009, 34(5): 104-106. DOI:10.3969/j.issn.1673-1212.2009.05.028 |
| [42] |
Authority E F S. Conclusion on the peer review of the pesticide risk assessment of the active substance kieselgur (diatomaceous earth)[J]. Efsa Journal, 2012, 10(7): 1-35. |
| [43] |
Chang B S, Zhong L, Akinc M, et al. Low cost composites for vacuum insulation core material[J]. Vacuum, 2016, 120-126. |
| [44] |
Bello O S, Adegoke K A, Oyewole R O, et al. Insights into the adsorption of heavy metals from wastewater using diatomaceous Earth[J]. Separation Science and Technology, 2014, 49(12): 1787-1806. DOI:10.1080/01496395.2014.910223 |
| [45] |
Nascimento C C, Andrade G R, Santos O S, et al. Biosilica from diatomaceous earth as support to CdS-mediated photocatalysis in dry and aqueous phase[J]. Materials & Design, 2017, 8-14. |
| [46] |
Payne E K, Rosi N L, Xue C, et al. Sacrificial biological templates for the formation of nanostructured metallic microshells[J]. Angewandte Chemie, 2005, 44(32): 5064-5067. DOI:10.1002/(ISSN)1521-3773 |
| [47] |
Jeffryes C, Campbell J, Li H, et al. The potential of diatom nanobiotechnology for applications in solar cells, batteries, and electroluminescent devices[J]. Energy and Environmental Science, 2011, 4(10): 3930-3941. DOI:10.1039/c0ee00306a |
| [48] |
Townley H E, Parker A R, Whitecooper H, et al. Exploitation of diatom frustules for nanotechnology:tethering active biomolecules[J]. Advanced Functional Materials, 2008, 18(2): 369-374. DOI:10.1002/(ISSN)1616-3028 |
| [49] |
Lin K C, Kunduru V, Bothara M, et al. Biogenic nanoporous silica-based sensor for enhanced electrochemical detection of cardiovascular biomarkers proteins[J]. Biosensors and Bioelectronics, 2010, 25(10): 2336-2342. DOI:10.1016/j.bios.2010.03.032 |
| [50] |
Lutz K, Groger C, Sumper M, et al. Biomimetic silica formation:analysis of the phosphate-induced self-assembly of polyamines[J]. Physical Chemistry Chemical Physics, 2005, 7(14): 2812-2815. DOI:10.1039/b505945c |
| [51] |
Patwardhan S V, Clarson S J. Silicification and biosilicification[J]. Silicon Chemistry, 2002, 1(3): 207-214. DOI:10.1023/A:1021243810915 |
| [52] |
Knecht M R, Wright D W. Amine-terminated dendrimers as biomimetic templates for silica nanosphere formation[J]. Langmuir, 2004, 20(11): 4728-4732. DOI:10.1021/la0494019 |
| [53] |
Knecht M R, Wright D W. Functional analysis of the biomimetic silica precipitating activity of the R5 peptide from Cylindrotheca fusiformis[J]. Chemical Communications, 2003, 9(24): 3038-3039. |
| [54] |
Xia L, Li Z. Poly(L-lysine)-mediated biomimetic silica synthesis:effects of mixing sequences and counterion concentrations[J]. Langmuir, 2011, 27(3): 1116-1122. DOI:10.1021/la104030f |
| [55] |
Patwardhan S V, Mukherjee N, Clarson S J, et al. The use of poly-L-lysine to form novel silica morphologies and the role of polypeptides in biosilicification[J]. Journal of Inorganic and Organometallic Polymers and Materials, 2001, 11(3): 193-198. DOI:10.1023/A:1015293712319 |
| [56] |
Patwardhan S V, Mukherjee N, Steinitzkannan M, et al. Bioinspired synthesis of new silica structures[J]. chemical communications (London), 2003, 1122-1123. |
| [57] |
Pires J, Fernandes A C, Avo R, et al. Bio-inspired synthesis of mesoporous sílicas using large molecular weight poly-l-lysine at neutral pH[J]. Journal of Materials Science, 2014, 49(17): 6087-6092. DOI:10.1007/s10853-014-8335-8 |
| [58] |
Pouget E, Dujardin E, Cavalier A, et al. Hierarchical architectures by synergy between dynamical template self-assembly and biomineralization[J]. Nature Materials, 2007, 6(6): 434-439. DOI:10.1038/nmat1912 |
| [59] |
Jin R, Yuan J. One-pot and rapid synthesis of uniformed silica spheres via mediation of linear poly(ethyleneimine)s and dyes[J]. Polymer Journal, 2007, 39(8): 822-827. DOI:10.1295/polymj.PJ2006277 |
| [60] |
周冉, 常明, 王飞, 等. 仿生硅化-磁性Fe3O4固定化木瓜蛋白酶的性质[J]. 食品工业科技, 2014, 35(12): 180-183. Zhou Ran, Chang Ming, Wang Fei, et al. Property of papain immobilized on magnetic ferroferric oxide by biosilicification[J]. Science and Technology of Food Industry, 2014, 35(12): 180-183. |
| [61] |
Cui J, Liang L, Han C, et al. Stabilization of phenylalanine ammonia lyase from Rhodotorula glutinis by encapsulation in polyethyleneimine-mediated biomimetic silica[J]. Applied Biochemistry and Biotechnology, 2015, 176(4): 999-1011. DOI:10.1007/s12010-015-1624-0 |
| [62] |
周冉, 宋玉品, 刘晓妹, 等. 仿生硅化固定化葡萄糖氧化酶与辣根过氧化酶研究[J]. 食品工业科技, 2017(02): 184-188. Zhou Ran, Song Yupin, Liu Xiaomei, et al. Immobilization of glucose oxidase and horseradish peroxidase in biomimetic silica particles[J]. Science and Technology of Food Industry, 2017(02): 184-188. |
| [63] |
Lechner C C, Becker C F. Modified silaffin R5 peptides enable encapsulation and release of cargo molecules from biomimetic silica particles[J]. Bioorganic & Medicinal Chemistry, 2013, 21(12): 3533-3541. |
| [64] |
Forsyth C, Yip T W, Patwardhan S V. CO2 sequestration by enzyme immobilized onto bioinspired silica[J]. Chemical Communications, 2013, 49(31): 3191-3193. DOI:10.1039/C2CC38225C |
| [65] |
Choi O, Kim B, An J H, et al. A biosensor based on the self-entrapment of glucose oxidase within biomimetic silica nanoparticles induced by a fusion enzyme[J]. Enzyme and Microbial Technology, 2011, 49(5): 441-445. DOI:10.1016/j.enzmictec.2011.07.005 |
| [66] |
毛争争, 柳永, 孙燕, 等. 新型Resilin-R5融合蛋白的大肠杆菌表达和纯化[J]. 浙江农业学报, 2014(1): 141-147. Mao Zhengzheng, Liu Yong, Sun Yan, et al. Expression and purification of the novel Resilin-R5 fusion protein in Escherichia coli[J]. Acta Agriculturae Zhejiangensis, 2014(1): 141-147. DOI:10.3969/j.issn.1004-1524.2014.01.26 |
 2019, Vol. 43
2019, Vol. 43


