
文章信息
- 李璐, 张朋艳, 姚建亭, 段德麟. 2019.
- LI Lu, ZHANG Peng-yan, YAO Jian-ting, DUAN De-lin. 2019.
- 海带CRY-DASH基因的克隆与转录表达分析
- Cloning and transcriptional analysis of CRY-DASH from Saccharina japonica (Laminariales, Phaeophyceae)
- 海洋科学, 43(4): 1-7
- Marine Sciences, 43(4): 1-7.
- http://dx.doi.org/10.11759/hykx20170222003
-
文章历史
- 收稿日期:2017-02-22
- 修回日期:2017-08-06
2. 中国科学院实验海洋生物学重点实验室, 山东 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室实验海洋生物学与生物技术实验室, 山东 青岛 266071;
4. 中国科学院大学, 北京 100049
2. CAS Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
3. Marine Biology and Biotechnology Laboratory, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
蓝光(350~500 nm)易穿透海水[1], 其对生活在潮下带的褐黄藻类有更明显的诱导效应。海带(Sac charina japonica)是生活在潮下带弱光环境中的大型褐藻, 蓝光为其吸收的主要光谱。以往研究报道, 蓝光能调控和诱导海带的生长及繁殖[2-3], 但其调控的分子机制鲜有报道。
隐花色素/光裂解酶家族(cryptochrome/photol yase family, CPF)是一类广泛存在的能被蓝光诱导激活的蛋白家族。光裂解酶能修复由紫外线引起的DNA损伤, 其催化主要依赖于350~500 nm的蓝光诱导[4-5]。隐花色素是与光裂解酶同源的蓝光受体, 尽管与光裂解酶结构相似, 但其丧失了修复损伤DNA的能力, 而在调控生物钟[6]、引发植物光形态建成[7]等方面有重要作用。
CRY-DASH(Cryptochrome-Drosophila, Arabidopsis, Synechocystis, Human)是CPF家族的新成员。目前, 人们已经明确CRY-DASH具有修复损伤DNA中环嘧啶酮二聚体(cyclobutane pyrimidine dimer, CPD)的功能。拟南芥中属于CRY-DASH家族的CRY3晶体结构已被解析, 晶体结构和生化分析均表明CRY- DASH能修复ssDNA和dsDNA环形结构中的CPD[8-9]。真菌(Phycomyces blakesleeanus)中已证明CRY-DASH修复ssDNA和dsDNA中的CPD效率一致[10]。
尽管在高等植物中人们对CRY-DASH光修复功能有所了解, 但还缺乏证实其具有调控功能的直接证据。Brudler和Kleine等通过生化方法证实这类蛋白可与未损伤的DNA结合, 由于在体外均未发现CRY-DASH的光裂解酶活性, 其认为CRY-DASH与DNA的结合是与信号转导有关[11-12]。
本研究针对海带中CRY-DASH基因进行了克隆与分析, 通过对获得的序列与其它藻类等的系统比较分析, 并探究不同光质诱导下CRY-DASH的响应, 为解析CRY-DASH是否具备光受体功能, 探讨其是否受蓝光介导对海带孢子体早期发育起作用提供理论依据。
1 材料与方法 1.1 材料处理海带孢子体采自山东高绿水产有限公司俚岛海域的海带养殖筏架上, 选择海带“中科2号”幼孢子体(长15~25 cm)数棵。采集后的幼孢子体运回实验室, 用灭菌冷却海水冲洗几次除去附生杂藻, 置于10℃黑暗条件下过夜预培养。将海带幼孢子体于黑暗条件下培养60 h后, 再分别置于蓝光[25 μmol/(m2·s)]、白光[70 μmol/(m2·s)]和红光[40 μmol/(m2·s)]条件下照射60 min。在照射前、照射10 min、30 min和60 min时分别取3棵海带, 锡箔纸包好后置于液氮内速冻, 用于后续RNA的提取。
1.2 实验方法 1.2.1 cDNA合成采用OMEGA公司的RNA提取试剂盒(Plant RNA Kit), 按照有关说明, 进行海带总RNA的提取。分光光度计(Denovix DS-11)检测RNA质量, 将高纯度的RNA(OD260/280=1.8~2.2)用于后续cDNA的合成。用于实时定量PCR实验的cDNA合成使用TaKaRa公司cDNA合成试剂盒(PrimeScriptTM Ⅱ 1st strand cDNA synthesis kit), 用于5′-cDNA末端快速克隆(5′-RACE)和3′- cDNA末端快速克隆(3′-RACE)实验的cDNA合成分别按照相应试剂盒说明书的要求进行。其中, 5′-RACE试剂盒(SMARTer RACE cDNA Amplication Kit)购自Clontech公司, 3′-RACE试剂盒(3′-Full RACE Core Set Ver. 2.0)购自TaKaRa公司。
1.2.2 海带CRY-DASH基因(SjCRY-DASH)扩增根据本研究组已注册海带受蓝光诱导的转录组信息(注册号GSE33853)[13], 选取候选目的序列进行同源性比对。为进一步获得SjCRY-DASH序列全长, 用Primer Premier 5软件设计5′-RACE和3′-RACE特异引物(表 1)。根据RACE扩增反应体系的要求, 分别扩增得到5′末端和3′末端, 将两段有重叠部分的序列拼接在一起。根据拼接的序列信息, 用高保真DNA聚合酶扩增SjCRY-DASH的开放阅读框(open reading frame, ORF), 引物使用CRY-DASH-F和CRY- DASH-R(表 1)。将所获目标片段进行胶回收, 然后连接于pMD19-T载体(TaKaRa公司), 经过转化到感受态细胞DH5α, 通过引物和PCR检测分析, 获得的阳性克隆送至上海生物工程有限公司测序。
| 引物名称 | 序列(5′-3′) | 描述 |
| CRY-DASH-5O | CCCCAAGTGGAATATGGAGTTGCCATTC | 5′RACE |
| CRY-DASH-5I | CCCGCCGTCTCGCCCCCCTTGAA | 5′RACE |
| CRY-DASH-3O | CAAGGAGGTCTGGGGCAATG | 3′RACE |
| CRY-DASH-3I | CTGCCTTCCGACTTGCCGTT | 3′RACE |
| CRY-DASH-F | CATATGATGGCGAGCTTTTCATCTGA | ORF扩增 |
| CRY-DASH-R | GAATTCCTAGGAAAACTGCCGAAACG | ORF扩增 |
| qCRY-DASH-F | AATGGAGGTCGGGAAGTAC | 扩增SjCRY-DASH的定量PCR引物 |
| qCRY-DASH-R | CCCCCAAGTGGAATATGG | 扩增SjCRY-DASH的定量PCR引物 |
| Actin-F | GACGGGTAAGGAAGAACGG | 扩增β-actin的定量PCR引物 |
| Actin-R | GGGACAACCAAAACAAGGGCAGGAT | 扩增β-actin的定量PCR引物 |
使用ORF finder软件预测所获cDNA序列的ORF, 并将ORF序列翻译为氨基酸序列。氨基酸序列的分子量与等电点预测利用ProtParam[14]软件完成。使用DNAman 6.0软件将SjCRY-DASH分别与已知的5种藻类和高等植物CRY-DASH氨基酸序列进行多序列比对, 预测SjCRY-DASH功能。为比较与其他物种中CPF家族成员的进化关系, 从GenBank数据库中获得不同物种的21个CPF家族成员的氨基酸序列, 利用MEGA 6.0[15]软件将其与SjCRY- DASH序列进行比对, 选用邻接法(NJ)设置重复1000次, 构建SjCRY-DASH及相关的系统发育树。使用SWISS-MODEL软件, 以拟南芥中已解析的CRY-DASH晶体结构为参照, 预测海带中CRY-DASH三维结构。
1.2.4 实时定量PCR分析为探究SjCRY-DASH对不同光质诱导的响应, 利用实时定量PCR方法, 分析了其在不同光质诱导下的转录情况。目的基因扩增使用特异性引物qCRY-DASH-F和qCRY-DASH-R(表 1), 内参基因选用β-actin。扩增酶使用TaKaRa公司的SYBR Premix Ex Taq Ⅱ, 扩增程序如下: 95℃预变性30 s; 95℃变性5 s, 60℃退火延伸30 s, 40个循环。用2–ΔΔCt法[16]计算目的基因的相对表达量。
2 实验结果 2.1 SjCRY-DASH基因克隆基于转录组数据中注释为隐花色素/光裂解酶的unigene, 以海带幼孢子体提取RNA反转成的cDNA为模版, 从低丰度转录本中快速扩增cDNA的3′和5′末端, 最后从两个有相互重叠序列的3′和5′-RACE产物中获得全长cDNA。根据Blastx比对, 得到SjCRY-DASH cDNA全长。用高保真酶扩增ORF区进行进一步验证, 凝胶电泳检测显示清晰条带(图 1)。
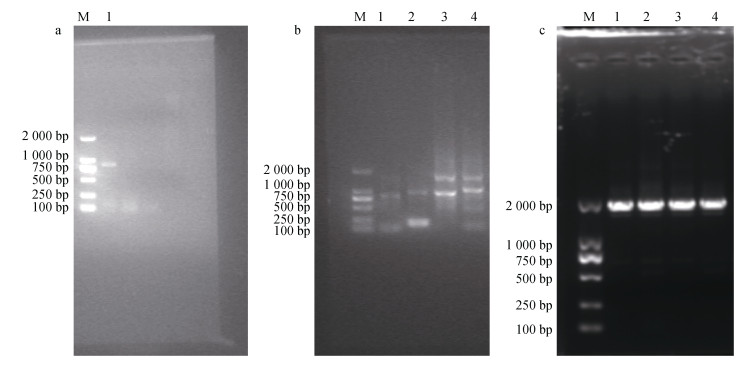 |
| 图 1 PCR扩增产物电泳检测 Fig. 1 Electrophoresis of PCR products a: 3′-RACE扩增结果, 1是扩增得到的3′末端; b: 5′-RACE扩增结果, 1—4分别是用不同的引物组合扩增得到的5′末端; c: ORF扩增结果 |
通过序列拼接获得SjCRY-DASH的cDNA序列, 其中ORF长1779 bp, 编码592个氨基酸, 5′-非翻译区(5′-UTR)和3′-非翻译区(3′-UTR)分别长122 bp和459 bp。经预测, 其编码的氨基酸序列分子量约为63.8 u, 等电点为9.30。将该蛋白序列与其他藻类及高等植物的CRY-DASH进行BLAST比对, 其与三角褐脂藻(Phaeodactylum tricornutum)、颤藻(Oscilla toriales cyanobacterium)、聚球藻(Synechococcus sp.)、长囊水云(Ectocarpus siliculosus)和拟南芥(Arabido psis thaliana)的同源性达到37%~68%。尽管CRY- DASH蛋白序列在不同物种中的同源性不高, 但其结构相似度很高。海带中与捕光因子MTHF作用的氨基酸残基和其他物种中基本一致, 包括Glu-144, Glu-145, Phe-374, Glu-448, Tyr-454(图 2)。海带中与FAD作用的残基同其他物种亦具有很强的保守性, 这些残基分别是Trp-389、Trp-441和Trp-464(图 2)。
 |
| 图 2 SjCRY-DASH多序列比对 Fig. 2 Multiple alignment of SjCRY-DASH with other CRY-DASH sequences ▲与MTHF作用的残基; ◆与FAD作用的残基 |
对SjCRY-DASH和其他物种的隐花色素/光裂解酶成员进行系统进化分析(图 3), 结果显示, 所获序列归于CRY-DASH家族, 但其与脊椎动物CRY- DASH的亲缘性比光合作用生物的亲缘性更近。
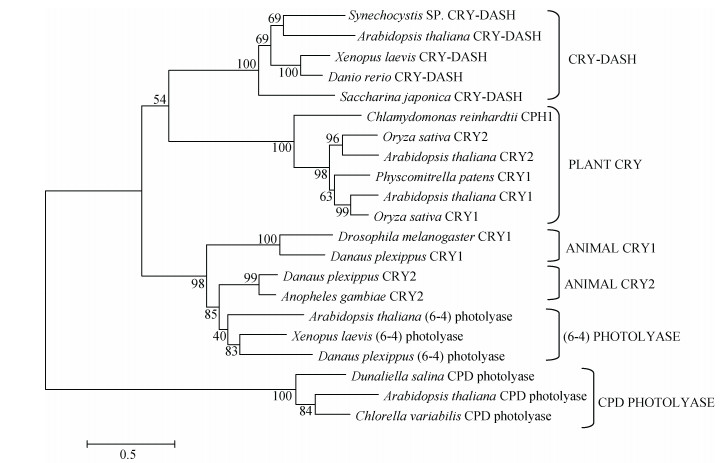 |
| 图 3 基于CRY-DASH氨基酸序列的系统进化分析 Fig. 3 Phylogenetic analysis of the amino acid sequence of CRY-DASH |
三维结构预测表明, SjCRY-DASH与已解析的拟南芥CRY-DASH晶体结构[17]极为相似, N末端均为α/β domain, C末端均为α domain(图 4)。
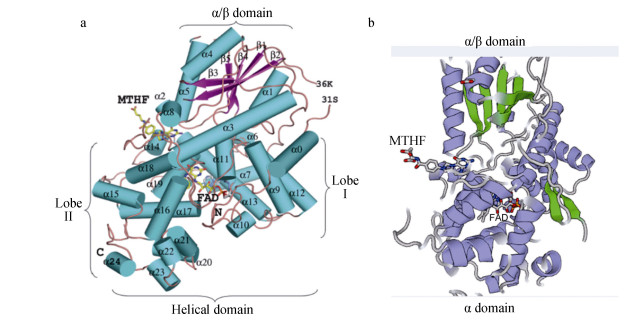 |
| 图 4 海带CRY-DASH三维结构预测 Fig. 4 Prediction of the spatial structure of CRY-DASH from S. japonica a:拟南芥CRY-DASH三维结构; b: SjCRY-DASH三维结构预测 |
利用不同光质(蓝光、白光和红光)诱导海带幼孢子体, 发现SjCRY-DASH在蓝光和白光诱导1 h内表达水平明显升高。SjCRY-DASH对于蓝光诱导有很强的响应, 在10, 30和60 min时表达水平分别是起始的3.68倍, 5.89倍和6.10倍。白光诱导10, 30和60 min时, 转录水平分别增加至起始的1.58倍, 2.12倍和6.54倍。尽管二者在60 min时转录水平相近, 但10 min和30 min时蓝光诱导下转录本上升更显著, 这说明SjCRY-DASH对蓝光的响应更为明显。而红光诱导不同时间下的转录水平基本一致, 表明其对SjCRY- DASH诱导无明显作用(图 5)。
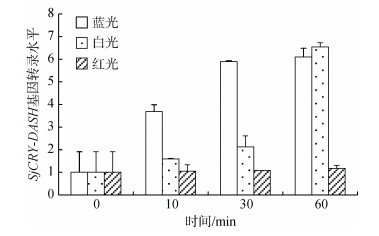 |
| 图 5 不同光质对SjCRY-DASH转录的影响 Fig. 5 Effects of blue, white, and red light on the transcription of SjCRY-DASH |
光质对于海带生长和繁殖的影响已有报道, 低温和蓝光均能促进海带配子体排卵[2]。Lüning[18]发现糖海带受精过程需要蓝光, 且蓝光含量要大于17%。Shi等[19]也认为海带雌、雄配子的结合依赖于一定光照强度的蓝光, 且随着蓝光强度的增加而增强。这说明蓝光对于海带的生殖意义重大。蓝光也影响海带的生长。Wang等[20]发现, 蓝光能促进海带的光形态建成和光合作用, 从而促进海带孢子体的早期发育。在蓝光下海带生长8周, 其细胞数目和细胞层数明显多于红光下培养的孢子体。隋等[21]探究了不同光质对海带配子体生长、发育的影响, 发现蓝光对生长、发育的促进作用最大。
本研究通过比较不同光质对海带中SjCRY-DASH的影响, 我们发现该基因的转录与蓝光密切相关。蓝光诱导下, SjCRY-DASH在1 h内转录水平明显升高, 这说明SjCRY-DASH属于能被蓝光诱导激活的基因家族。白光诱导1 h, SjCRY-DASH转录水平亦升高, 但升高趋势比蓝光诱导下的缓慢。由于白光中包含多种光质, 因此白光影响要弱于蓝光。SjCRY-DASH转录水平在红光处理下维持稳定, 表明红光对SjCRY- DASH无诱导效应。
CRY-DASH是否具备信号转导功能尚待探索。Brudler等[11]最早认为CRY-DASH具有光受体调控功能, 其通过对聚球藻的突变株进行分析, 发现与野生型相比, 一些基因的转录上升, 由此得出CRY- DASH对转录起抑制作用。另外, Brunelle等[22]证实甲藻(Karenia brevis)在蓝光介导下可提前进入细胞分裂的S期, 而红光则无明显作用, 且构建的EST数据库中找到了蓝光受体中的唯一一位成员CRY-DASH, 据此推断其有可能参与细胞周期调控。
本研究从转录水平印证了海带中CRY-DASH在短时间内对蓝光有响应, 这为探讨大型褐藻-海带中CRY-DASH受光诱导调控功能提供理论依据。
| [1] |
Wolken J J. Photobehavior of marine invertebrates: extraocular photoreception[J]. Comparative Biochemistry & Physiology Part C Comparative Pharmacology & Toxicology, 1988, 91: 145-149. |
| [2] |
Lüning K, Dring M J. Reproduction induced by blue light in female gametophytes of Laminaria saccharina[J]. Planta, 1972, 104(3): 252-256. DOI:10.1007/BF00387080 |
| [3] |
Lüning K, Dring M J. Reproduction, growth and photosynthesis of gametophytes of Laminaria saccharina grown in blue and red light[J]. Marine Biology, 1975, 29(3): 195-200. DOI:10.1007/BF00391846 |
| [4] |
Sancar A. Structure and function of DNA photolyase and cryptochrome blue-light photoreceptors[J]. Chemical Reviews, 2003, 103: 2203-2237. DOI:10.1021/cr0204348 |
| [5] |
Brettel K, Byrdin M. Reaction mechanisms of DNA photolyase[J]. Current Opinion in Structural Biology, 2010, 20: 693-701. DOI:10.1016/j.sbi.2010.07.003 |
| [6] |
Cashmore A R. Cryptochromes: Enabling plants and animals to determine circadian time[J]. Cell, 2003, 114: 537-543. DOI:10.1016/j.cell.2003.08.004 |
| [7] |
Cashmore A R, Jarillo J A, Wu Yingjie, et al. Cryptochromes: Blue light receptors for plants and animals[J]. Science, 1999, 284: 760-765. DOI:10.1126/science.284.5415.760 |
| [8] |
Selby C P, Sancar A. A cryptochrome/photolyase class of enzymes with single-stranded DNA-specific photolyase activity[J]. Proceedings of the National Academy of Sciences, 2006, 103: 17696-17700. DOI:10.1073/pnas.0607993103 |
| [9] |
Pokorny R, Klar T, Hennecke U, et al. Recognition and repair of UV lesions in loop structures of duplex DNA by DASH-type cryptochrome[J]. Proceedings of the National Academy of Sciences, 2008, 105: 21023-21027. DOI:10.1073/pnas.0805830106 |
| [10] |
Tagua V G, Pausch M, Eckel M, et al. Fungal cryptochrome with DNA repair activity reveals an early stage in cryptochrome evolution[J]. Proceedings of the National Academy of Sciences, 2015, 112: 1-6. DOI:10.1073/iti0115112 |
| [11] |
Brudler R, Hitomi K, Daiyasu H, et al. Identification of a new cryptochrome class: Structure, function, and evolution[J]. Molecular Cell, 2003, 11: 59-67. DOI:10.1016/S1097-2765(03)00008-X |
| [12] |
Kleine T, Lockhart P, Batschauer A. An arabidopsis protein closely related to synechocystis cryptochrome is targeted to organelles[J]. The Plant Journal, 2003, 35: 93-103. DOI:10.1046/j.1365-313X.2003.01787.x |
| [13] |
Deng Yunyan, Yao Jianting, Wang Xiuliang, et al. Transcriptome sequencing and comparative analysis of Saccharina japonica (Laminariales, Phaeophyceae) under blue light induction[J]. Plos One, 2012, 7(6): e39704. DOI:10.1371/journal.pone.0039704 |
| [14] |
Gasteiger E, Hoogland C, Gattiker A, et al. Protein identification and analysis tools on the ExPASy Server[C]//Walker J. The Proteomics Protocols Handbook. Totowa, NJ: Humana Press, 2005: 571-607.
|
| [15] |
Tamura K, Dudley J, Nei M, et al. MEGA4: Molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Molecular Biology and Evolution, 2007, 24: 1596-1599. DOI:10.1093/molbev/msm092 |
| [16] |
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(–ΔΔC(T)) method[J]. Methods, 2001, 25: 402-408. DOI:10.1006/meth.2001.1262 |
| [17] |
Huang Yihua, Baxter R, Smith B S, et al. Crystal structure of cryptochrome 3 from Arabidopsis thaliana and its implications for photolyase activity[J]. Proceedings of the National Academy of Sciences, 2006, 103: 17701-17706. DOI:10.1073/pnas.0608554103 |
| [18] |
Lüning K. Critical levels of light and temperature regulating the gametogenesis of three laminaria species (Phaeophyceae)[J]. Journal of Phycology, 1980, 16(1): 1-15. DOI:10.1111/j.1529-8817.1980.tb02992.x |
| [19] |
Shi Cuijuan, Kataoka H, Duan D L. Effects of blue light on gametophyte development of Laminaria japonica (Laminariales, Phaeophyta)[J]. Chinese Journal of Oceanology and Limnology, 2005, 23(3): 323-329. DOI:10.1007/BF02847155 |
| [20] |
Wang Wenjun, Sun Xiutao, Wang Feijiu. Effect of blue light on early sporophyte development of Saccharina japonica (Phaeophyta)[J]. Marine Biology, 2010, 157: 1811-1817. DOI:10.1007/s00227-010-1453-1 |
| [21] |
隋晓伟, 任伟, 闫文华, 等. 光质对海带配子体生长发育影响的研究[J]. 海洋科学, 2011, 35(4): 33-36. Sui Xiaowei, Ren Wei, Yan Wenhua, et al. Effects of light qualities on growth and development of Saccharina japonica gametophytes[J]. Marine Sciences, 2011, 35(4): 33-36. |
| [22] |
Brunelle S A, Hazard E S, Sotka E E, et al. Characterization of a dinoflagellate cryptochrome blue-light receptor with a possible role in circadian control of the cell cycle[J]. Journal of Phycology, 2007, 43: 509-518. DOI:10.1111/j.1529-8817.2007.00339.x |
 2019, Vol. 43
2019, Vol. 43


