文章信息
- 刘顺, 胡自民, 张全胜, 段德麟. 2019.
- LIU Shun, HU Zi-min, ZHANG Quan-sheng, DUAN De-lin. 2019.
- 海萝抗氧化酶系与失水响应因子表达模式分析
- Expression patterns of antioxidant enzymes and desiccation-responsive factors in Gloiopeltis furcata (Gigartinales, Rhodophyta)
- 海洋科学, 43(5): 1-10
- Marine Sciences, 43(5): 1-10.
- http://dx.doi.org/10.11759/hykx20190404002
-
文章历史
- 收稿日期:2019-02-04
- 修回日期:2019-03-27
2. 青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 烟台大学 海洋学院, 山东 烟台 264005
2. Laboratory for Marine Biology and Biotechnology, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Ocean School, Yantai University, Yantai 264005, China
潮间带大型海藻固着生长于礁石上, 其每天需经受节律性的干出与浸没[1]。退潮时, 潮间带海藻直接暴露于空气中, 其生长状态受失水、温度、高光强等多种环境因子的影响, 而失水是最为重要的一项非生物胁迫[2]。失水会损伤藻体组织和细胞结构[3], 同时也会影响海藻光合、呼吸等生理过程[1]。此外, 失水会使藻体产生过量的活性氧(reactive oxygen species, ROS), 造成脂质过氧化, DNA与蛋白质损伤, 甚至引起藻体细胞死亡[4]。因此, 失水耐受能力对潮间带海藻十分重要, 是影响其垂直分布的主要因素之一[1, 2]。
目前, 对潮间带红藻失水响应机制的转录组学研究主要集中于紫菜属。Im等[5]对不同失水程度的甘紫菜(Pyropia tenera)进行了转录组测序, 其鉴定到了一条叶绿体蛋白编码基因PtDRG2, 过表达该基因可增强衣藻对渗透胁迫的耐受能力。Wang等[6]对坛紫菜(Porphyra haitanensis)不同发育阶段和失水处理下的转录组数据进行分析发现, 不饱和脂肪酸合成相关基因、转录因子、分子伴侣共同参与了坛紫菜失水与复水过程。C4和C3通路、海藻糖生物合成、卟啉和叶绿素代谢、细胞凋亡、生殖发育、碳水化合物代谢等过程的相关基因参与了失水胁迫。而水通道蛋白和ABC转运蛋白可能参与了坛紫菜复水初期反应。Yang等[7]对条斑紫菜(Porphyra yezoensis)进行转录组测序发现, 大量的Unigenes与条斑紫菜胁迫耐受性相关, 主要包括失水耐受相关基因, 高光耐受相关基因, 类黄酮生物合成相关基因以及活性氧清除相关基因。Sun等[8]分析了条斑紫菜渗透胁迫下的转录组数据发现, 其光合作用相关的Unigenes在渗透胁迫时显著下调以防止光合副产物的损伤, 抗氧化酶相关Unigenes则上调表达以防止活性氧的损伤。但目前, 对周期性连续失水—复水条件下, 潮间带红藻的转录组研究仍未见报道。
潮间带海藻含有大量抗氧化酶和非酶类抗氧化剂, 可清除失水产生的过量ROS和有毒代谢物[9]。Flores-Molina等[3]认为, 藻类抗氧化能力与其所处潮位相关。Fierro等[10]对红藻Pyropia orbicularis的研究发现, 过氧化物酶(peroxiredoxin, PRX)、过氧化氢酶(catalase, CAT)和硫氧还蛋白(thioredoxin, TRX)在有效消除ROS方面发挥重要的作用。此外, 潮间带红藻可产生红藻糖苷(floridoside), 其具有渗透压调节、细胞壁修复以及细胞抗氧化等重要功能, 可能显著响应失水胁迫[11-13]。红藻糖苷是一种多糖, 其合成需要甘油-3-磷酸和UDP-半乳糖作为前体物质。UDP-葡萄糖焦磷酸化酶(UDP-glucose pyrophosphorylase, UGPase)、甘油激酶(glycerol kinase, GK)和3-磷酸甘油脱氢酶(glycerol 3-phosphate dehydrogenase, GPDH)是红藻糖苷合成过程中关键性的酶[11, 12]。Li等[13]证实红藻糖苷可直接作为合成细胞壁多糖的前体物质, 参与受损细胞壁合成。Qian等[11]发现, 坛紫菜(Porphyra haitanensis)在失水胁迫时会快速积累红藻糖苷。Chen等[12]发现, 海萝(Gloiopeltis furcata)中的红藻糖苷含量远高于其他潮位的潮间带红藻, 他认为, 红藻糖苷含量与海藻所处潮位高度呈正相关。
烟台潮间带典型红藻—海萝(Gloiopeltis furcata)失水耐受力强[14], 经济价值较高[15], 是研究潮间带红藻适应性机制的良好材料。Liu等[14]研究发现, 海萝藻体虽不具备明显的保水能力, 但却可以耐受超过72 h的失水, 当海萝藻体含水量低至6%时仍能够在复水后恢复光合活性。此外, Takei等[16]证实, 海萝还具有较高的氧自由基(O2–)清除能力。
本研究选择海萝为实验材料, 在24 h内设计两次连续的失水-复水循环处理, 模拟烟台海域半日潮特征, 通过测定并分析海萝抗氧化酶活力与转录组数据, 研究其失水响应相关因子, 为探索潮间带红藻失水响应分子机制提供理论参考。
1 材料与方法 1.1 材料海萝采自山东烟台小黑山高潮带岩石上(37°27′45.23N, 121°26′34.28E), 于退潮时进行样品采集。采集后的海萝加冰运至实验室。采用过滤海水清洗海萝藻体表面, 人工选择大小、粗细、颜色、形状均基本一致的幼年期健康藻体(生长期约3~4个月, 长约3 cm, 宽约1.5 mm)作为实验材料。正式实验前, 将海萝样品置于光照培养箱(GZP-250N, 上海森信股份有限公司, 中国)中预培养3 d, 使各藻体达到相对一致的生理状态。海萝预培养条件为:温度11℃, 光照50 μmol photons/(m2·s), 光周期12 L︰12 D, 光照由培养箱中白色冷光源提供。
1.2 失水-复水循环处理本实验对海萝采取两次连续的失水-复水循环处理方式。用滤纸吸干海萝藻体表面的水分后, 取部分健康藻体作为实验对照组(CG)。使用木架固定藻体, 将其分别悬挂在通风光照培养箱中进行初次失水处理(first dehydration, FD), 总时长6 h。分别在脱水处理4.5 h(FD4.5)、6 h(FD6.0)时取样, 取样后立即放入液氮中, 并储存在–80℃。之后进行初次复水处理(first rehydration, FR)。将样品浸没到过滤海水中, 初次复水总时长6 h, 分别在复水5.5 h(FR5.5)和6 h (FR6.0)时取样。接着进行二次失水-复水循环处理, 二次失水(second dehydration, SD)处理方法与初次失水处理方法相同, 二次复水(second rehydration, SR)处理方法与初次复水处理方法相同, 在二次失水4.5 h (SD4.5)和6 h(SD6.0), 二次复水6 h(SR6.0)时取样。实验过程中, 温度控制在11℃, 相对湿度为77%。在两次失水-复水循环过程中共取8个时间点的海萝藻体作为实验样本, 缩写为CG, FD4.5, FD6.0, FR5.5, FR6.0, SD4.5, SD6.0, SR6.0。每个样本包含3个以上生物学重复, 每个重复包含4~6枝藻体。
1.3 抗氧化酶活力的测定粗酶液的制备:取0.1 g海萝藻体, 经液氮研磨成粉末后转移至2 mL离心管中, 加入2 mL预冷PBS缓冲液(PBS缓冲液为科铭生物CAT酶活力测定试剂盒中自带提取液), 涡旋混匀后, 8000 g离心10 min, 去除沉淀, 所得上清液为待测粗酶液, 于4℃冰箱内暂存, 所有酶活力指标在1 d内测完。
可溶性蛋白浓度测定按照索莱宝公司BCA蛋白浓度测定试剂盒说明书进行测定, 总抗氧化能力(T-AOC)、硫氧还蛋白还原酶(TrxR)、CAT酶活力测定试剂盒均购自科铭生物技术有限公司, 超氧化物歧化酶(SOD)活力测定试剂盒购自南京建成生物工程研究所。按照各试剂盒说明书进行测定操作。
利用Excel对酶活力测定数据进行单因素方差分析(ANOVA), 并应用基迪奥云平台T-test检验(http://www.omicshare.com/tools/Home/Soft/t_test)将处理组与对照组进行比较, P < 0.05为差异显著, P < 0.01为差异极显著。
1.4 海萝总RNA的提取取0.05 g海萝藻体, 液氮预冷后研磨成粉, 用植物RNA提取试剂盒(Omega Plant RNA Kit, USA)进行海萝样品总RNA提取, 操作流程参照试剂盒说明书。通过1%琼脂糖凝胶电泳和微量型分光光度计Nanodrop 2000检测RNA质量, 判断其浓度、完整性及核酸和蛋白质的污染情况。
1.5 海萝转录组测序在广州基迪奥生物科技有限公司开展文库制备工作, 构建好的文库通过Illumina HiSeqTM进行测序。去除原始数据中包含有接头、未知碱基占比 > 10%、低质量的(Q < 20%的碱基占整条read的40%以上)的reads, 得到High quality clean reads, 用于Uni genes拼接。利用BLAST数据库进行Unigenes注释和通路注释, 所用数据库包括KEGG、KOG、Nr、Swissport。利用edgeR进行差异基因分析, 若P < 0.05, 且|log2FC| > 1则为显著差异表达基因。
1.6 实时荧光定量PCR实验以海萝延伸因子2基因(Elongation factor 2, EF2)作为内参, 在GeneBank中获得其碱基序列(EF033553.1), 按照荧光定量引物设计原则, 通过BLAST在线引物设计工具和Primer Premier 5软件进行引物设计, 所设计引物序列均由青岛瑞博兴科生物技术有限公司进行合成, 引物浓度为10 µmol/L。
选用Takara PrimeScriptTM RT regent Kit with gDNA Eraser对海萝RNA进行反转录实验, 合成cDNA模板。以10× cDNA稀释液为模板, 进行荧光定量PCR(qRT-PCR)扩增。qRT-PCR反应操作流程参照SYBR® Premix Ex TaqTM II(Tli RNaseH Plus, Takara)说明书进行。20 µL反应体系包括: 2 µL稀释后cDNA模板, 正向反向引物各0.8 µL, 10 µL SYBR® Premix Ex TaqTM(2×), 6.4 µL ddH2O。使用三步法进行扩增, 扩增程序为: 95℃30 s变性; 95℃5 s, 55℃10 s, 72℃20 s, 共40个循环。循环结束后通过95℃15 s, 60℃30 s, 95℃15 s绘制溶解曲线。qRT-PCR扩增反应在Takara TP800 Cycler Dice上进行。每个反应设置3次技术重复, 使用2–ΔΔCt法计算目的基因相对表达量, 应用Excel处理数据并绘图。所用引物见表 1。
| 引物名称 | 引物序列 |
| GfEF2F | ACTCTGTTGAGGGTGTCTGCG |
| GfEF2R | AAAGAGCGGTCCAGCTTGTTA |
| GfPRXF | CCGTCATCGACAAGGTAGCA |
| GfPRXR | GCTGTCGGTGCTAACGGTAA |
| GfGSTF | CGATTCTTCGATACGCCGGA |
| GfGSTR | CCGCTTGCAGCTTCTCCTTA |
| GfUGPaseF | AAGCTGCGAGACATGAACGA |
| GfUGpaseR | CCCACCATGTAACGCGAGTA |
| GfGPDHF | CTGCATGACGACGAGCTTTG |
| GfGPDHR | CGGCAGAGGGGGTTAAAGTT |
| GfGKF | GCCTCCCACTTGGCTGTTT |
| GfGKR | TGCTGTAGGTTTGTACCCGTCT |
| GfCAF | ACGTGCCAATTACAGCGAGA |
| GfCAR | ATCTTCACTTCCATGCCCCG |
| GfMYBF | GCCATGCTTGCGAATAGGTG |
| GfMYBR | ACAACCACCGCTCTTTCCAT |
| GfHSP70F | GGTGTTAAGCGGGTTGGAAG |
| GfHSP70R | TGGAGAATGGCGTTGTTGAG |
| GfMAPK3F | TGCTCTCCCGTCAAACTGTC |
| GfMAPK3R | CAAAGTTTCGTCGTGCTGGG |
| GfGPDHF | CTGCATGACGACGAGCTTTG |
| GfGPDHR | CGGCAGAGGGGGTTAAAGTT |
| GfMPK3F | TGCTCTCCCGTCAAACTGTC |
| GfMPK3R | CAAAGTTTCGTCGTGCTGGG |
两次连续失水-复水循环处理过程中海萝T-AOC、SOD、CAT、TrxR的测定结果如图 1所示。海萝T-AOC在两次失水过程中均有显著上调趋势(P < 0.05), 在失水4.5 h时(FD4.5、SD4.5), T-AOC上调幅度较小, 约为对照组的1.2倍, 在失水6 h时(FD6.0、SD6.0)可升高至对照组的1.5倍, 复水过程中T-AOC下调至正常水平。海萝SOD酶活力较高, 除FR5.5、FR6.0外, 各处理组SOD酶活力均大于200 U/mg prot。在本实验中, SOD酶活力在初次失水过程中并没有出现上调, 初次复水(FR5.5、FR6.0)时, SOD酶活力下调, 低于正常水平。在二次失水(SD4.5、SD6.0)时SOD酶活力出现显著上升趋势(P < 0.05), 但上升倍数较小, 在SD6.0时达到正常水平的1.2倍。与对照组相比, 海萝CAT酶活力在两次失水过程中(FD.5、FD6.0、SD4.5、SD6.0)均有极显著(P < 0.01)上调。FD4.5、FD6.0时的CAT酶活力约为对照组1.8倍, SD4.5时CAT酶活力升高至对照组2倍, 在SD6.0时其酶活力达到最大值91.17 U/mg prot, 约为对照组4.1倍。在复水过程中(FR5.5、FR6.0、SR6.0), CAT酶活力出现下调, 但仍略高于对照组。TrxR酶活力在FD4.5即出现极显著上调(P < 0.01), 达到峰值3.7 U/mg prot, 约为对照组2.5倍, FD6.0时略有下调, 但仍保持在对照组1.9倍左右, 在初次复水时(FR5.5、FR6.0)TrxR酶活力下调至正常水平, SD6.0时TrxR酶活力再次上调, 达到对照组1.9倍, SR6.0时再次下调至正常水平。
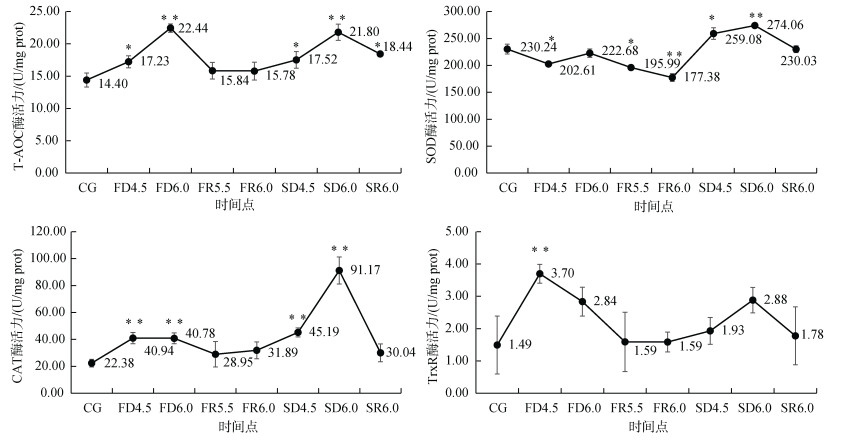 |
| 图 1 周期性失水-复水条件下海萝抗氧化酶活力变化情况 Fig. 1 Changes in antioxidant enzymes activities during periodic dehydration-rehydration cycles *P < 0.05, **P < 0.01 |
对海萝测序得到的clean reads进行过滤后发现, 除T7-e外, 所有样本的HQ quality clean reads占比均大于98%。将得到clean reads进行序列组装后, 得到32 681条基因, N50为1 238 bp, 平均GC含量为55.32%, 组装得到的Unigenes平均长度为799 bp(表 2), 数据组装效果理想。将拼接获得的Unigenes进行功能注释后发现, 共有12 813条Unigenes获得注释信息, 19 868条基因暂无注释信息(表 2)。利用BLASTX将Unigenes序列与Nr数据库进行比对后, 确定其同源序列所属物种, 统计比对到各个物种的同源序列数量发现(图 2), 海萝转录组组装得到的Unigenes其同源数目最多的物种是皱波角叉菜(Chondrus crispus), 其次是另外一种可在极端环境中生存的红藻(Galdieria sulphuraria)。
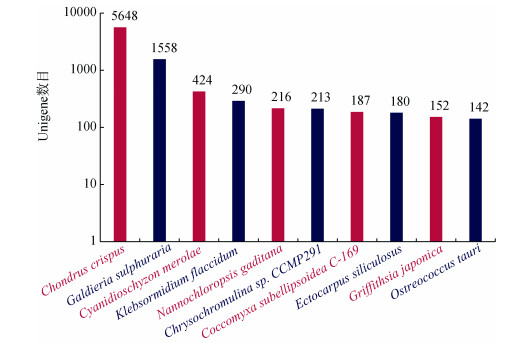 |
| 图 2 Unigenes物种分布统计图 Fig. 2 Cartogram of unigene distribution in different species |
| 海萝转录组测序整体情况 | 数据统计 |
| 组装到Unigenes总体数目 | 32 681 |
| Unigenes平均长度/bp | 799 |
| GC含量/% | 55 |
| N50长度/bp | 1 238 |
| 有注释基因数目 | 12 813 |
| 无注释基因数目 | 19 868 |
不同实验组间PCA分析显示(图 3a), PC1可解释所有变量(所有基因表达量)总体方差的38.1%, PC2可解释总体方差的16.7%。本研究中所用实验样本重复性较好, 同一处理组中的三个生物学重复样本可较好地聚集在一起, 仅FR5.5组内e与d相关系数相对较小, 分析可能是海萝藻体个体间差异造成的。另外, 除FR5.5-e与FR5.5-d外, 初次失水-复水处理的样本更倾向于聚为一枝, 而二次失水-复水处理的样本倾向于聚为另一枝。
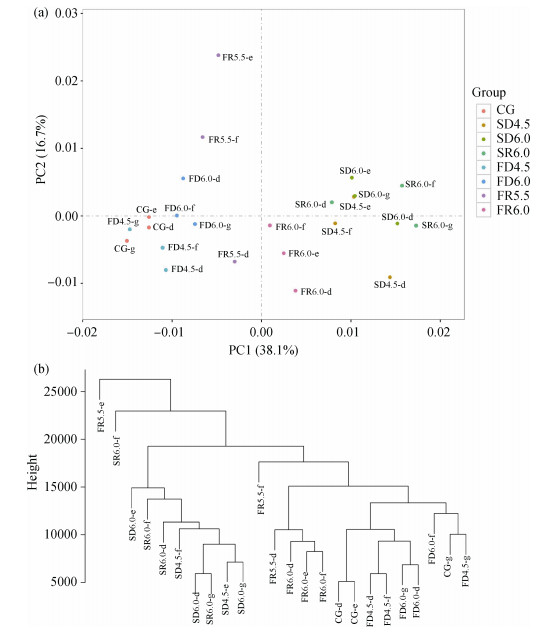 |
| 图 3 转录组重复性检验与样本聚类 Fig. 3 Transcriptome repetition test and sample clustering 注: a:样本PCA分析结果; b:所有样本聚类分析结果 |
海萝转录组共表达了7161条差异表达基因(differentially expressed genes, DEGs), 不同处理组与对照组之间的上调、下调DEGs数目统计结果见图 4a。初次失水-复水循环中, 各处理组分别获得了222(FD4.5), 689(FD6.0), 2930(FR5.5), 3552(FR6.0)条DEGs。二次失水-复水循环中, 各处理组分别获得了4164(SD4.5), 1739(SD6.0), 1907(SR6.0)条DEG。总体来看, 二次失水处理DEGs数目多于初次失水处理。对所有差异进行KEGG富集分析显示(图 4b), 差异基因分布在代谢、环境信息加工、有机体系统、遗传信息加工、细胞进程五大方面。其中, 参与代谢相关的DEGs数目最多, 达到1003条, 另有302条DEGs与遗传信息加工相关, 65条DEGs与细胞进程相关, 23条DEGs与有机系统相关, 16条DEGs与环境信息加工相关。
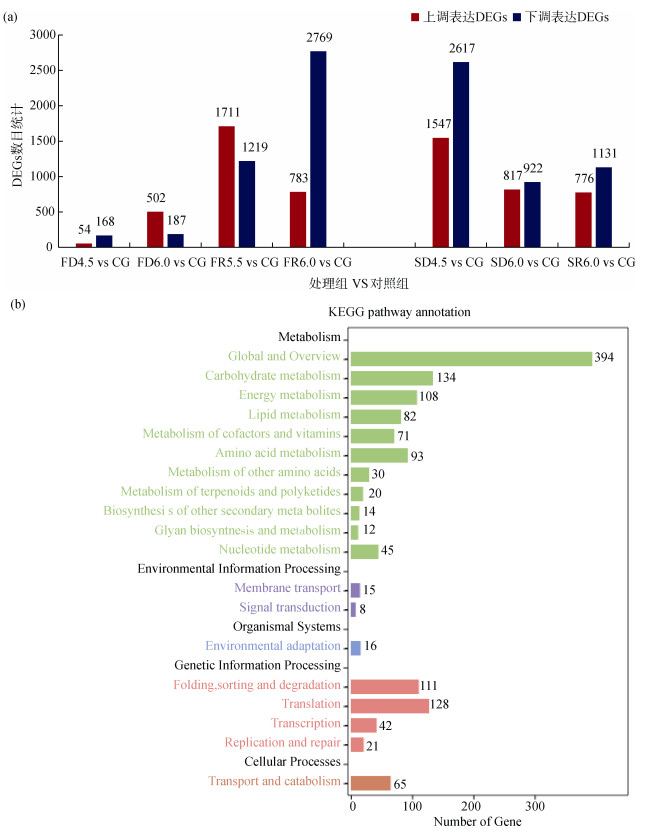 |
| 图 4 差异表达基因的数目统计与分析 Fig. 4 Statistical analysis of differentially expressed genes 注: a:组间上、下调差异基因数目统计; b:差异基因KEGG分析 |
通过海萝转录组注释情况以及基因表达情况, 我们选择了与海萝失水耐受可能相关的9条基因(表 3)进行qRT-PCR验证。分析其表达模式(图 5)发现:
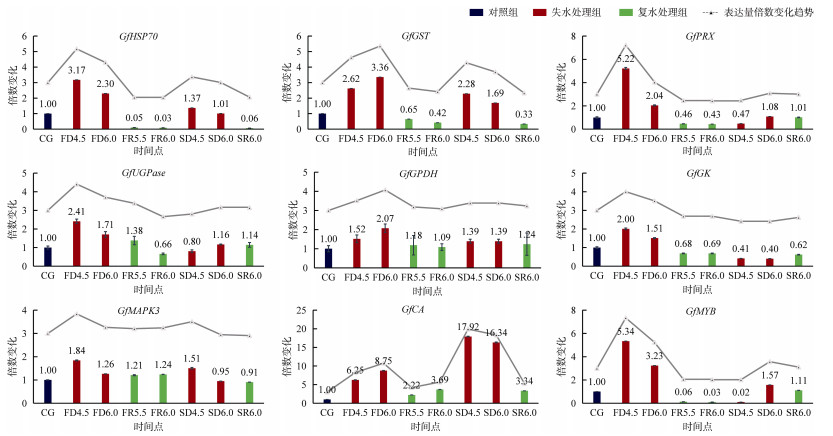 |
| 图 5 候选失水响应基因转录水平表达模式 Fig. 5 Transcription-level expression patterns of candidate dehydration-responsive genes |
| 失水响应候选基因简称 | 基因编码蛋白 | 中文名称 | 参与过程及功能 |
| GfPRX | Peroxiredoxin | 过氧化物酶 | 抗氧化与细胞解毒 |
| GfGST | Glutathione S-transferase | 谷胱甘肽S-转移酶 | 抗氧化与细胞解毒 |
| GfUGPase | UDP-glucose pyrophosphorylase | UDP-葡萄糖焦磷酸化酶 | 红藻糖苷合成相关 |
| GfGPDH | Glycerol-3-phosphate dehydrogenase | 甘油-3-磷酸脱氢酶 | 红藻糖苷合成相关 |
| GfGK | Glycerol kinase | 甘油激酶 | 红藻糖苷合成相关 |
| GfCA | Carbonic anhydrase | 碳酸酐酶 | 二氧化碳浓缩机制 |
| GfMYB | MYB domain-containing protein | MYB蛋白 | 转录调控相关 |
| GfHSP70 | Heat shock protein 70 | 热激蛋白70 | 分子伴侣 |
| GfMAPK | Mitogen-activated protein kinase 3 | 促分裂原活化蛋白激酶 | 信号转导 |
海萝中, 热激蛋白70基因(GfHSP70)、碳酸酐酶基因(GfCA)、MYB蛋白基因(GfMYB)以及编码谷胱甘肽S转移酶(Glutathione S-transferase, GST)的基因(GfGST)的转录表达随海萝失水-复水处理表现出两次明显上下波动变化。其中, GfHSP70在FD4.5时达到峰值, 约为对照组3.2倍, 在FD6.0时略有下降, 为对照组2.3倍, 初次复水时(FR5.5、FR6.0)GfHSP70转录表达几乎下降为0, 在SD4.5和SD6.0时再次上调至正常水平。GfGST在初次失水时上调至对照组2.6倍(FD4.5)和3.4倍(FD6.0), 在二次失水时(SD4.5、SD6.0)其表达水平均在对照组1.5倍以上, 在两次复水处理中(FR5.5、FR6.0、SR6.0)GfGST的表达量均低于对照组。GfCA在二次失水中的上调幅度大于初次失水, 其在FD4.5、FD6.0时分别达到对照组的6.3倍和8.3倍, 而在二次失水中(SD4.5、SD6.0)升高至对照组16倍以上。复水时GfCA转录水平有明显下调, 但仍高于对照组。GfMYB在初次失水过程中上调表达, 在FD4.5时达到峰值, 约为对照组5.3倍, 在FD6.0时约为对照组3.2倍, 在初次复水过程中(FR5.5、FR6.0)其转录表达几乎下降为0, SD6.0时再次上升至略高于对照组的水平。另外, 海萝中编码PRX酶的基因(GfPRX)的转录水平只在初次失水时(FD4.5、FD6.0)有上调表达, 初次复水过程中其表达量降低至对照组以下, 在SD6.0时基本恢复至正常水平。与信号转导相关的促分裂原活化蛋白激酶基因(GfMAPK)只在FD4.5和SD4.5时上调表达, 分别达到对照组1.8倍和1.5倍。
海萝中参与红藻糖苷合成的关键基因GfUGPase、GfGK和GfGPDH的表达模式相似, 均在FD4.5和FD6.0时上调表达, 而在二次失水处理中则变化不明显。其中, GfUGPase与GfGK在FD4.5时均达到对照组2倍以上, 在FD6.0时稍有下降但仍保持在对照组1.5倍左右, 而GfGPDH则在FD6.0时达到表达量峰值, 约为对照组2倍。
3 讨论O2−、过氧化氢(H2O2)和羟基自由基(-OH)等具有强氧化能力的含氧化合物统称ROS, 正常情况下, 细胞内ROS维持在相对稳定的水平, 其产生和清除之间保持动态平衡[17]。潮间带海藻的抗氧化系统对清除失水胁迫所产生的过量ROS十分重要[4, 9]。海萝总体抗氧化能力T-AOC在两次失水-复水过程中的表达趋势基本一致, 失水时T-AOC表现出显著升高趋势(P < 0.05), 但上升幅度较小, 在复水时降低至正常水平(图 1)。这种升高趋势可及时清除海萝失水过程中产生的过量ROS, 起到保护海萝藻体的作用, T-AOC上升幅度较小可能是因其ROS清除系统复杂, 总抗氧化能力的轻微上调即会对ROS含量产生重大影响, 且ROS的清除不能无限制的进行, 需使其含量维持在一定水平, 以起到信号分子等作用[18]。本实验中, 不同的抗氧化酶在连续的失水-复水循环过程中的表达趋势不尽相同。其中, 海萝CAT的酶活力在两次失水过程中的变化趋势最为明显(图 1)。因此, 我们认为CAT可能在海萝抵抗失水胁迫时发挥主要所用。CAT可通过催化H2O2还原为水, 消除其负面影响[10]。Fierro等[10]也发现, 失水胁迫下, 红藻P. orbicularis的CAT酶活力上调表达, 且其数值明显高于其它失水不耐受海藻。此外, SOD主要负责清除细胞中多余的O2–, 催化O2–发生歧化反应生成氧和H2O2[19], TrxR可通过增强酶活力使还原态Trx增加[20], 以达到抗氧化防御作用。它们在海萝清除过量ROS过程中也起到一定的作用。
Chen等[12]研究表明, 海萝在失水时会积累红藻糖苷及其异构体异红藻糖苷。在海萝转录组中, 本研究发现了参与红藻糖苷合成过程的关键酶UGPase, GPDH和GK的编码基因(GfUGPase、GfGK、GfGPDH)。於辰佳等[21]研究发现, 坛紫菜失水过程中, 编码UGPase的基因(Phugp)在转录水平上调表达, 并在复水后迅速恢复。Qian等[11]认为, 坛紫菜GK基因(PhNHO1)和GPDH基因(PhGPDH)的转录表达也与失水胁迫密切相关。本实验中, 海萝GfUGPase、GfGK、GfGPDH基因的转录水仅在初次失水时有上调趋势, 可达到对照组1.5倍至2.4倍, 但三者在二次失水过程中的表达量变化却不明显(图 5)。
此外, 海萝中还存在参与CO2浓缩机制(CO2 concentrating mechanism)的碳酸酐酶基因(GfCA), 参与蛋白质折叠的热激蛋白70基因(GfHsp70), 编码MYB蛋白的基因(GfMYB), 以及参与细胞解毒过程的GST编码基因(GfGST)。它们在两次失水胁迫中均表现出明显的上调趋势(图 5)。Chen等对坛紫菜CA的研究也发现, 坛紫菜的5条CA基因均在失水时显著上调[22]。Sun等[8]也报道了条斑紫菜HSP基因在其失水时出现上调表达。MYB蛋白可能通过转录调节作用[23], 参与非生物胁迫[24]。GST具有细胞解毒作用与过氧化物清除作用[25], 可减轻藻体损伤。
在周期性失水-复水循环过程中, CAT、TrxR、SOD参与海萝抗氧化过程, 其中CAT酶活力对海萝抵抗失水胁迫尤为重要; 海萝红藻糖苷合成相关基因(GfUGPase、GfGK、GfGPDH)仅在初次失水处理中表现出上调趋势。此外, 海萝GfHSP70、GfCA、GfMYB、GfGST基因的转录水平在两次失水处理时均表现出升高趋势, 它们可能也参与了海萝失水响应过程。本研究结果对分析海萝失水耐受机制有重要的参考意义, 也为研究潮间带红藻适应性分子机制提供了理论依据。
| [1] |
Davison I R, Pearson G A. Stress tolerance in intertidal seaweeds[J]. Journal of Phycology, 1996, 32(2): 197-211. DOI:10.1111/j.0022-3646.1996.00197.x |
| [2] |
Zaneveld J S. Factors controlling the delimitation of littoral benthic marine algal zonation[J]. American Zoologist, 1969, 9(2): 367-391. DOI:10.1093/icb/9.2.367 |
| [3] |
Flores-Molina M R, Thomas D, Lovazzano C, et al. Desiccation stress in intertidal seaweeds:Effects on morphology, antioxidant responses and photosynthetic performance[J]. Aquatic Botany, 2014, 113: 90-99. DOI:10.1016/j.aquabot.2013.11.004 |
| [4] |
Contreras-Porcia L, Thomas D, Flores V, et al. Tolerance to oxidative stress induced by desiccation in Porphyra columbina (Bangiales, Rhodophyta)[J]. Journal of Experimental Botany, 2011, 62(6): 1815-1829. DOI:10.1093/jxb/erq364 |
| [5] |
Im S, Lee H N, Jung H S, et al. Transcriptome-based identification of the desiccation response genes in marine red algae Pyropia tenera (Rhodophyta) and enhancement of abiotic stress tolerance by PtDRG2 in Chlamydomonas[J]. Marine Biotechnology, 2017, 19(3): 232-245. DOI:10.1007/s10126-017-9744-x |
| [6] |
Wang Li, Mao Yunxiang, Kong Fanna, et al. Genome-wide expression profiles of Pyropia haitanensis in response to osmotic stress by using deep sequencing technology[J]. BMC Genomics, 2015, 16: 1012. DOI:10.1186/s12864-015-2226-5 |
| [7] |
Yang Hui, Mao Yunxiang, Kong Fanna, et al. Profiling of the transcriptome of Porphyra yezoensis with Solexa sequencing technology[J]. Chinese Science Bulletin, 2011, 56(20): 2119-2130. DOI:10.1007/s11434-011-4546-4 |
| [8] |
Sun Peipei, Tang Xianghai, Bi Guiqi, et al. Gene expression profiles of Pyropia yezoensis in response to dehydration and rehydration stresses[J]. Marine Genomics, 2019, 43: 43-49. DOI:10.1016/j.margen.2018.09.005 |
| [9] |
Kumar M, Gupta V, Trivedi N, et al. Desiccation induced oxidative stress and its biochemical responses in intertidal red alga Gracilaria corticata (Gracilariales, Rhodophyta)[J]. Environmental and Experimental Botany, 2011, 72(2): 194-201. DOI:10.1016/j.envexpbot.2011.03.007 |
| [10] |
Fierro C, López-Cristoffanini C, Meynard A, et al. Expression profile of desiccation tolerance factors in intertidal seaweed species during the tidal cycle[J]. Planta, 2017, 245(6): 1149-1164. DOI:10.1007/s00425-017-2673-0 |
| [11] |
Qian Feijian, Luo Qijun, Yang Rui, et al. The littoral red alga Pyropia haitanensis uses rapid accumulation of floridoside as the desiccation acclimation strategy[J]. Journal of Applied Phycology, 2015, 27(1): 621-632. DOI:10.1007/s10811-014-0336-0 |
| [12] |
Chen Juanjuan, Song Dandan, Luo Qiyun, et al. Determination of floridoside and isofloridoside in red algae by high-performance liquid chromatography-tandem mass spectrometry[J]. Analytical Letters, 2014, 47(14): 2307-2316. DOI:10.1080/00032719.2014.905950 |
| [13] |
Li Shiyan, Shabtai Y, Arad S. Floridoside as a carbon precursor for the synthesis of cell-wall polysaccharide in the red microalga Porphyridium sp.(Rhodophyta)[J]. Journal of phycology, 2002, 38(5): 931-938. DOI:10.1046/j.1529-8817.2002.01143.x |
| [14] |
Liu Hongliang, Li Xuemeng, Nan Guoning, et al. Photosynthetic activity of Gloiopeltis furcata (intertidal red macroalga) in response to desiccation[J]. The journal of applied ecology, 2014, 25(5): 1491-1498. |
| [15] |
郭永坚, 张文文, 李俊伟, 等. 海萝属藻类繁殖生物学及增养殖研究进展[J]. 生态学杂志, 2015, 8: 2339-2343. Guo Yongjian, Zhang Wenwen, Li Junwei, et al. Breeding, restocking and culture of Gloiopeltis[J]. Chinese Journol of Ecology, 2015, 8: 2339-2343. |
| [16] |
Takei M, Kuda T, Eda M, et al. Antioxidant and fermentation properties of aqueous solutions of dried algal products from the Boso Peninsula[J]. Japan. Food Bioscience, 2017, 19: 85-91. DOI:10.1016/j.fbio.2017.06.006 |
| [17] |
杜秀敏, 殷文璇, 赵彦修, 等. 植物中活性氧的产生及清除机制[J]. 生物工程学报, 2001, 17(2): 121-125. Du Xiumin, Yin Wenxuan, Zhao Yanxiu, et al. The production and scavenging of reactive oxygen species in plants[J]. Chinese Journal of Biotechnology, 2001, 17(2): 121-125. DOI:10.3321/j.issn:1000-3061.2001.02.003 |
| [18] |
Mullineaux P M, Exposito-Rodriguez M, Laissue P P, et al. ROS-dependent signaling pathways in plants and algae exposed to high light:comparisons with other eukaryotes[J]. Free Radical Biology and Medicine, 2018, 122: 52-64. DOI:10.1016/j.freeradbiomed.2018.01.033 |
| [19] |
Miura C, Sugawara K, Neriya Y, et al. Functional characterization and gene expression profiling of superoxide dismutase from plant pathogenic phytoplasma[J]. Gene, 2012, 510(2): 107-112. DOI:10.1016/j.gene.2012.09.001 |
| [20] |
孙文艳. 硫氧还蛋白还原酶研究进展[J]. 国外医学(医学地理分册), 2012, 26(4): 148-151. Sun Wenyan. The progress in studies of thioredoxin reductase[J]. Foreign Medical Sciences (Section of Medgeography), 2012, 26(4): 148-151. |
| [21] |
於辰佳, 张晗晗, 徐燕, 等. 坛紫菜UDP-葡萄糖焦磷酸化酶基因(Phugp)的克隆及表达分析[J]. 集美大学学报(自然科学版), 2017, 5: 4-12. Yu Chenjia, Zhang Hanhan, Xu Yan, et al. Cloning and expression analysis of UDP-Glucose Pyrophosphorylase (UGPase) gene from Pyropia haitanensis[J]. Journal of Jimei University(Natural Science), 2017, 5: 4-12. |
| [22] |
Chen Changsheng, Dai Zhenzhen, Xu Yan, et al. Cloning, expression, and characterization of carbonic anhydrase genes from Pyropia haitanensis (Bangiales, Rhodophyta)[J]. Journal of Applied Phycology, 2016, 28(2): 1403-1417. DOI:10.1007/s10811-015-0646-x |
| [23] |
万小荣, 李玲. 植物的MYB蛋白[J]. 植物生理学通讯, 2016, 38(2): 165-170. Wan Xiaorong, Li Ling. MYB proteins in plants[J]. Plant Physiology Communications, 2016, 38(2): 165-170. |
| [24] |
刘蕾, 杜海, 唐晓凤, 等. MYB转录因子在植物抗逆胁迫中的作用及其分子机理[J]. 遗传, 2008, 30(10): 1265-1271. Liu Lei, Du Hai, Tang Xiaofeng, et al. The roles of MYB transcription factors on plant defense responses and its molecular mechanism[J]. Hereditas, 2008, 30(10): 1265-1271. DOI:10.3321/j.issn:0253-9772.2008.10.005 |
| [25] |
周向红, 易乐飞, 李信书, 等. 条斑紫菜谷胱甘肽S-转移酶基因的克隆与表达分析[J]. 水产学报, 2011, 35(9): 1354-1361. Zhou xianghong, Yi Lefei, Li Xinshu, et al. Molecular cloning and expression pattern of glutathione S-transferase gene in Porphyra yezoensis Ueda (Bangiales, Rhodophyta)[J]. Journal of Fisheries of China, 2011, 35(9): 1354-1361. |
 2019, Vol. 43
2019, Vol. 43


