文章信息
- 秦乐宁, 黄鑫洁, 朱大玲, 侍亚敏, 唐娜, 王学魁. 2019.
- QIN Le-ning, HUANG Xin-jie, ZHU Da-ling, SHI Ya-min, TANG Na, WANG Xue-kui. 2019.
- 淡化浓海水日晒盐田原核微生物多样性分析
- Diversity analysis of prokaryotic microorganisms in the solar saltern of concentrated seawater from desalination
- 海洋科学, 43(5): 27-35
- Marine Sciences, 43(5): 27-35.
- http://dx.doi.org/10.11759/hykx20190115003
-
文章历史
- 收稿日期:2019-01-15
- 修回日期:2019-03-10
日晒盐田由一系列盐度不同的盐池组成, 海水逐渐蒸发, 使得卤水浓度逐渐提高, 特殊的盐度条件使得日晒盐田存在着独特的生态系统。日晒盐田生态系统中主要的生物群落包括浮游植物、浮游动物和原核微生物等。其中, 藻类等浮游植物和一些光合细菌作为生产者, 卤虫等浮游动物作为消费者, 而嗜盐微生物则作为分解者[1]。良好且稳定的生态系统, 对原盐品质及盐田的可持续发展具有深远的影响。
在低盐度(< 7%)盐池中, 浮游植物群落的种类最为丰富, 数量也最多。浮游植物为卤虫等浮游动物提供饵料。当盐池中的氮、磷等营养盐质充足时, 藻类会在池底形成垫状结构, 有效防止盐池渗漏。中盐度(< 15%)盐池中, 卤虫及卤蝇幼虫等嗜盐动物数量增加。卤虫既可以净化卤水, 又可为高盐区的嗜盐微生物提供营养物质。低、中盐度盐池卤水中的微生物可以分解卤水中的有机质, 且有助于碳酸钙和硫酸钙的析出, 可提高原盐中氯化钠产量和质量[2-3]。高盐度(> 20%)盐池中, 几乎仅有嗜盐微生物生存下来。含有类胡萝卜素的红色嗜盐微生物对卤水着色, 有效提高卤水对阳光的吸收率, 增加卤水蒸发量提高蒸发效率, 可提高原盐的产量达9.14%[1]。此外, 结晶池底层的结晶盐使得反光率增加, 卤水蒸气压处于最低值, 如果没有红色嗜盐菌的着色, 蒸发过程很难进行[4-6]。
近年来, 海水淡化与制盐相结合技术迅速发展[7-9]。天津汉沽日晒盐田为淡化浓海水盐田, 其海水淡化工艺为低温多效法(Multi-Effect Distillation, MED)[10]。淡化浓海水的理化性质较正常海水有所不同, 其盐度约为正常海水的两倍[11]。低温多效法产生的淡化浓海水温度比海水高5~15℃, 这会对卤水的溶氧造成影响。此外, 由于海水淡化过程要经过脱氧、加氯、添加阻垢剂、还原剂等预处理过程, 使得淡化浓海水pH值降低到6.5左右, 且铜、镍、锌、铬、钼、铁等金属离子含量高于天然海水平均值。经海水淡化预处理和淡化过程, 海水中原有的浮游动植物及原核微生物等都难以存活下来。因此, 利用海水淡化浓海水直接排入日晒盐田制盐对原有盐田生态系统造成较大冲击, 其较难自动恢复到正常平衡状态。
日晒盐田生态系统中原核微生物作为主要的生物群落, 发挥着重要的作用。随着盐度的逐渐变化, 各盐池中的生物群落组成也在逐渐演变[12-15]。在盐湖、日晒盐场、南极湖泊等高盐环境中, 都有原核微生物存在[16]。高盐环境中原核微生物可分为轻度嗜盐微生物、中度嗜盐微生物、极端嗜盐微生物和耐盐微生物[17], 其中, 极端嗜盐微生物包括极端嗜盐细菌和极端嗜盐古菌两大类。虽然如今是焦磷酸测序技术的时代, 但PCR-指纹图谱技术因所需样品量少等特点仍被广泛应用于环境微生物多样性的研究中, 常被用来分析不同空间和时间中微生物群落的变化[18-22]。因此, 本文采用温度梯度凝胶电泳(TGGE)技术, 对天津汉沽淡化浓海水日晒盐田原核微生物多样性进行研究, 分析不同盐度盐池在不同月份中的细菌和古菌多样性, 对不同季节和不同盐度盐池中原核微生物群落结构变化进行分析, 以期为评估淡化浓海水对盐田原核微生物群落影响提供理论依据。
1 材料与方法 1.1 样品采集2016年10月至11月和2017年4月至9月期间, 每月1次, 从天津汉沽日晒盐场(118°3′11″E, 39°15′43″N)采集淡化水、溴后水及七个不同盐度的盐池卤水。因盐场每天走水, 各盐池盐度几乎不变, 各盐池盐度如表 1所示。盐池水面下10 cm处取卤水200 mL, 经孔径为200目的滤网滤除浮游动物和浮游植物后, 装入无菌的聚乙烯瓶, 快速运回实验室进行后续试验。
将200 mL水样经0.22 μm孔径的醋酸纤维膜过滤, 收集水样中的原核微生物。DNA提取采用酚氯仿抽提法[23], 具体步骤如下:将收集的菌体溶于200 μL TE buffer (10 mmol/L Tris-HCl, 1 mmol/L EDTA, pH 8.0), 加入3 μL溶菌酶(50 mg/mL), 65℃水浴15 min。水浴结束后加入5 μL蛋白酶K(20 mg/mL), 以及25 μL 10% SDS溶液, 37-56℃水浴30 min。之后加入2 μL RNAse, 37℃水浴30 min。加入等体积的酚︰氯仿︰异戊醇(V︰V︰V=25︰24︰1), 上下颠倒数次后, 10000 r/min离心10 min。吸取上层水相, 再加入等体积氯仿︰异戊醇(V︰V=24︰1), 上下颠倒数次, 10000 r/min离心10 min, 吸取上层水相。加入1/40体积的乙酸钠(3 mol/L, pH 5.2)及2.5倍体积预冷的无水乙醇, 轻轻混匀, –20℃放置过夜。10000 r/min离心15 min, 弃上清。经70%乙醇洗涤, 乙醇蒸干后, 溶于30 μL TE buffer, –20℃保存备用。
1.3 PCR扩增利用PCR技术分别对细菌和古菌16S rDNA进行扩增。本实验采用的细菌通用引物为GC-357F (5′-CG-clamp-CCTACGGGAGGCAGCAG-3′)和518R (5′-ATTACCGCGGCTGCTGG-3′)。上游引物的5′端连接专为TGGE设计的CG-clamp(CGCCCGCCGCGCC CCGCGCCCGGCCCGCCGCCCCCGCCCC)[24]。反应体系25 μL:上游引物(10 μmol/L) 1 μL, 下游引物(10 μmol/L) 1 μL, Premix TaqTM 12.5 μL, ddH2O 10 μL, DNA模板1 μL。扩增条件为: 94℃预变性5 min; 94℃变性45 s, 57℃退火45 s, 72℃延伸1 min, 循环35次; 72℃延伸10 min。古菌通用引物采用Arc-GC-344F(5′-ACGGGG YGCASAGKCGVGA-3′)和Arc958R(5′-YCCGGCGTT GAVTCCAATT-3′)。上游引物的5′端连接专为TGGE设计的CG-clamp(CGCCCGGGGCGCGCCCCGGGCG GGGCGGGGGCACGGGGG)[25]。反应体系25 μL:上游引物(10 μmol/L) 1 μL, 下游引物(10 μmol/L) 1 μL, Premix TaqTM (TaKaRa TaqTM Version 2.0 plus dye) 12.5 μL, ddH2O 10 μL, DNA模板1 μL。扩增条件为: 94℃预变性5 min; 94℃变性45 s, 52℃退火45 s, 72℃延伸1 min, 循环35次; 72℃延伸10 min。
PCR扩增产物经琼脂糖凝胶电泳, 凝胶成像系统检测, 条带单一且与预期片段大小相近的PCR产物用于下一步TGGE分析。
1.4 TGGE分析及测序分析PCR扩增产物进行TGGE分析。采用Bio-Rad TGGE分析系统, 实验条件如下:细菌PCR扩增产物采用10%(w/v)聚丙烯酰胺(30︰1)变性胶, 古菌PCR产物采用6%(W/V)的聚丙烯酰胺(30︰1)变性胶, 变性温度范围为54~66℃, 变温速率3℃/h, 1×TAE电泳缓冲液(Tris Acetate-EDTA Buffer)中进行电泳, 电压130 V, 电泳时间4 h, 具体操作按照仪器使用手册说明进行。进样量: 10 μL PCR产物+1 μL 10× DNA Loading Buffer。电泳结束后, TGGE凝胶经0.5 μg/mL溴化乙锭(EB)染色10 min, 经TAE缓冲液漂洗后, 凝胶成像系统拍照记录进行分析。
对有代表性优势菌的条带进行切取, 采用聚丙烯酰胺凝胶回收试剂盒进行回收, 0.5 μL的回收产物作为模板进行PCR扩增。使用无CG-clamp细菌(357F, 518R)和古菌(Arc-344F, Arc-958R)通用引物, PCR扩增条件如上文所示。扩增产物经1.2%琼脂糖凝胶电泳检测后, 送专业测序公司(北京奥科鼎盛生物科技有限公司)进行测序。测序结果通过NCBI数据库Blast比对分析, 使用MEGA 7.0.14软件, 采用NJ法构建系统进化树, Bootstrap置信值估算重复次数1 000次[26]。
1.5 多样性指数分析使用Quantity one 4.6.2软件对凝胶成像系统获得的照片进行数字化分析, 可得各泳道的条带数以及各条带的相对光密度、相对迁移率, 用于多样性指数计算及不同样本中原核微生物群落结构的比较。
利用香农-威纳指数(Shannon-Weiner index, H)、加权丰富度指数(Range-weighted richness index, Rr)和均匀度指数(Pielou evenness index, E)分析微生物群落多样性、丰富度和均匀度。香农-威指纳数(H)、丰富度指数(Rr)和均匀度系数(E)计算公式[27-28]如下:
H=–∑(ni/N)log(ni/N),
Rr=(S2×Tg),
E=H/logS,
其中, ni指各条带的相对光密度, S为样本中的条带总数, N为样本中所有条带相对光密度之和。
2 实验结果与分析 2.1 PCR-TGGE图谱分析本研究在2016—2017年间近一年的时间里连续监测了天津汉沽日晒盐场不同盐度盐池卤水中的原核微生物变化。其中, 12月—3月处于严冬季节, 气温低导致蒸发量非常低, 部分盐池结冰且每年这个时期盐池不走水, 所以这四个月的样品没有采集。本研究共8批次, 每批次9个样品进行了细菌和古菌16S rRNA基因的PCR-TGGE分析。
据已有报道, 海岸带水体中细菌的数量为1.0× 1026, 平均丰度可达5×104 cells/cm3 [29]。而在用低温多效法进行海水淡化之前, 需对海水进行加氯除菌、过滤等预处理[30]。这就导致原本可随海水进入日晒盐田的微生物在海水淡化过程中难以存活。采用细菌和古菌的通用引物分别对淡化水及溴后水样品进行PCR扩增, 结果未出现任何扩增条带, 表明在经过海水淡化前预处理和淡化过程、吹溴过程之后, 淡化浓海水中的原核微生物数量非常少, 难以被检测到。该结果与理论相符, 淡化浓海水经引水渠引入日晒盐场后, 微生物需适应淡化浓海水的性质在卤水中逐渐繁殖。
不同盐度的7个盐池卤水中的细菌16S rDNA V3区的PCR扩增产物经1.2%的琼脂糖凝胶电泳检测, 均获得了片段大小约200 bp的阳性结果。对7个盐池各样品中古菌16S rDNA V3区的PCR扩增结果发现盐度最高的Ⅶ号池PCR扩增呈阳性结果, 获得约600 bp的片段, 其他盐池PCR扩增呈阴性结果, 表明仅高盐度Ⅶ号池卤水中有古菌存在。
不同盐池在4—11月卤水样品的细菌16S rDNA V3区PCR-TGGE结果如图 1a所示。从图上可看出每条泳道分别有1~8个条带不等, 其中, 较为优势的条带有3条, 分别为1号、2号和8号, 在盐田微生物群落演替中占据主要地位。4月的7个盐池样品中均有1号和2号条带; 5月的1~7号盐池样品中1号条带和2号条带均有存在, 其中Ⅰ号盐池中2号条带是优势条带, 8号条带出现在Ⅱ、Ⅳ~Ⅵ号等4个盐池样品中; 6—7月的7个盐池中, 1号和2条带均有出现, 8号条带出现在Ⅰ~Ⅲ号盐池样品中, 3号和4号条带出现在7月的Ⅰ号盐池样品中; 8月各盐池样品的条带数较其他月份有了较为明显的变化, 1号条带出现在除Ⅲ号盐池外的其他盐池样品中, 2号条带仅出现在Ⅰ、Ⅵ、Ⅶ号盐池样品中, 8号条带出现在Ⅱ、Ⅲ号盐池样品中, 9~13号条带出现在Ⅵ号盐池样品中; 9—11月各盐池样品中1号条带均有出现, 除9月的Ⅳ、Ⅵ、Ⅶ号盐池样品和10月的Ⅰ、Ⅱ号盐池样品, 2号条带在其他盐池样品中均有出现。14号和15号条带, 是为了验证出现在相同位置的条带即代表同一个菌种, 经切胶测序, 14号和15号条带序列分别与1号、2号序列相同。TGGE图谱中, 每一个条带即代表一个优势菌种或一个操作单元(Operational taxonomic unit, OTU), 一个泳道中的条带数目越多, 则说明这个样本中生物多样性越丰富, 条带信号越亮, 则说明该种属的丰度越高[30-31]。根据所得到的TGGE图谱中各条带的相对亮度, 可直观地看出不同月份各盐池中的优势菌种的相对多寡。
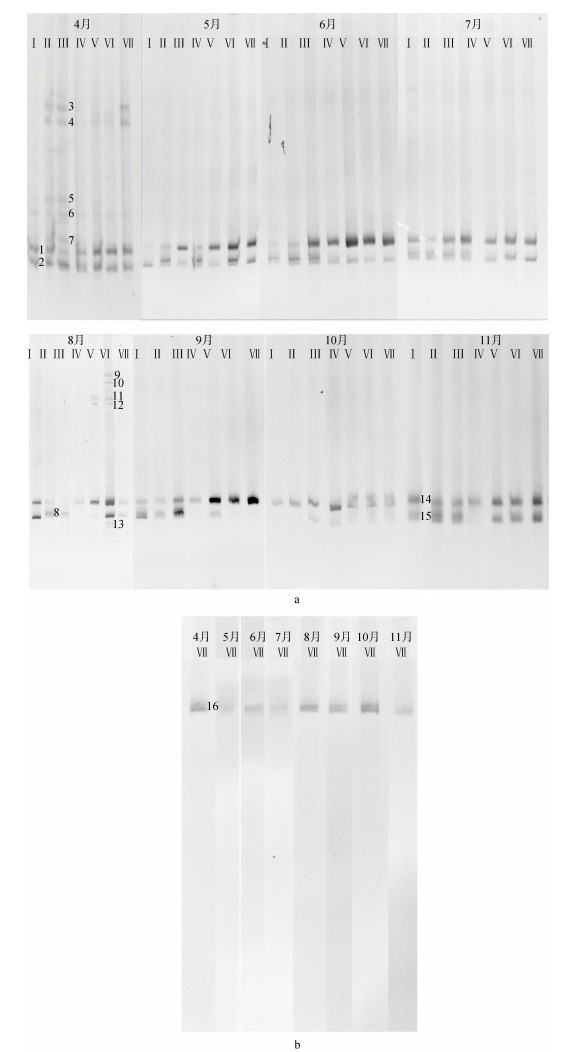 |
| 图 1 天津汉沽日晒盐场不同盐池细菌(a)和古菌(b)16S rDNA的TGGE图谱 Fig. 1 TGGE profile of bacteria (a) and Archaea (b) based on 16S rDNA genes of different ponds in the solar saltern 注:不同盐池编号标示在凝胶上方, 不同条带在相应位置进行标示并编号 |
使用Quantity One 4.6.2对TGGE图谱中条带的相对亮度进行定量分析。针对在相同月份中不同盐度样品的分析发现, 1号条带在低盐度盐池中亮度较低, 随着盐度升高亮度增大。2号条带在较低盐度盐池样本中亮度较大, 随盐度升高, 亮度逐渐降低。8号条带几乎仅出现在中低盐度(< 15°Be’)的盐池中。此外, 比较TGGE图谱发现, 4月各盐池中的条带数相对较多, 但相对亮度较其他月份较低; 8月的Ⅵ号盐池(盐度21°Be’), 条带数明显高于其他样品, 其中11、12号条带仅出现在该样本中。Ⅵ号盐池在4—11月卤水样品的古菌16S rDNA V3区PCR-TGGE结果如图 1b所示, 古菌TGGE图谱中仅有一个条带, 表明在高盐度条件下菌群中古菌的多样性低。
2.2 TGGE图谱中条带的鉴定TGGE图谱中16个条带(15个细菌条带和1个古菌条带)进行了切胶、纯化、PCR扩增及测序, 结果12个条带测序成功。本研究中所得的序列均已提交至GenBank数据库, 登录号为MK182943-MK182954。获得的序列分别在GeneBank数据库中进行BLAST同源性比对, 比对结果见表 2。结果表明所有的条带序列与GeneBank中已公布的菌种序列的相似性为90%~100%, 相似性的菌株都来源于海水、含盐废水、海滩、盐湖等高盐环境。细菌主要为γ-Proteo bacteria、α-Proteobacteria和Actinobacteria三大类, 其中优势菌为γ-Proteobacteria, 1号和14号条带的优势菌皆为Spiribacter salinus, 2号和15号条带的优势菌皆为Pseudoalteromonas sp., 16号条带的优势古菌为Halogeometricum sp., 相似性高达99%。采用NJ法构建条带序列的系统进化树如图 2所示, 表明多数条带序列属于变形菌门, 主要包括α-Proteobac teria(5个条带)和γ-Proteobacteria(4个条带), 少数条带序列属于放线菌门Actinomycetales(1个条带)。1个古菌条带鉴定为广古菌门(Euryarchaeota)几何菌属(Halogeometricum sp.)菌株。
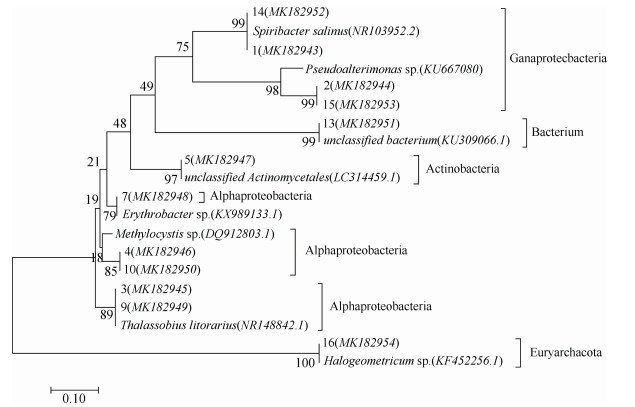 |
| 图 2 NJ法构建的条带基因序列系统进化树 Fig. 2 Phylogenetic tree constructed using the neighbor-joining method based on the 16S rDNA gene sequence of bands |
| 条带编号 | GenBank登录号 | 聚类组 | 相似序列 | 同源性 | 相似序列来源 |
| 1 | MK182943 | γ-Proteobacteria | Spiribacter salinus (NR103952.2) | 96% | Aquatic hypersaline environment |
| 2 | MK182944 | γ-Proteobacteria | Pseudoalteromonas sp. (KU667080.1) | 97% | Coastal water |
| 3 | MK182945 | α-Proteobacteria | Thalassobius Titorarius (NR148842.1) | 100% | Sea-tidal flat sediment |
| 4 | MK182946 | α-Proteobacteria | Methylocystis sp. (DQ912803.1) | 90% | salinity wastewater |
| 5 | MK182947 | Actinobacteria | unclassified Actinomycetales (LC314459.1) | 97% | Coastal fish-farming area |
| 7 | MK182948 | α-Proteobacteria | Erythrobacter sp. (KX989133.1) | 97% | Shallow hyperthermal system |
| 9 | MK182949 | α-Proteobacteria | Thalassobius Titorarius (NR148842.1) | 99% | Sea-tidal flat sediment |
| 10 | MK182950 | α-Proteobacteria | Methylocystis sp. (DQ912803.1) | 90% | salinity wastewater |
| 13 | MK182951 | Bacterium | Unclassified bacterium (KU309066.1) | 99% | Saline microbial mat |
| 14 | MK182952 | γ-Proteobacteria | Spiribacter salinus (NR103952.2) | 96% | Aquatic hypersaline environment |
| 15 | MK182953 | γ-Proteobacteria | Pseudoalteromonas sp. (KU667080) | 97% | Coastal water |
| 16 | MK182954 | Euryarchaeota | Halogeometricum sp. (KF452256.1) | 99% | Hypersaline lake |
本研究采用Quantity one 4.6.2软件将TGGE图谱中的条带信息数字化, 分别从香农-威纳指数(H)、加权丰富度指数(Rr)和均匀度系数(E)三个多样性指标对天津汉沽淡化浓海水盐田原核微生物的多样性、丰富度和均匀度进行分析, 结果如图 3所示。
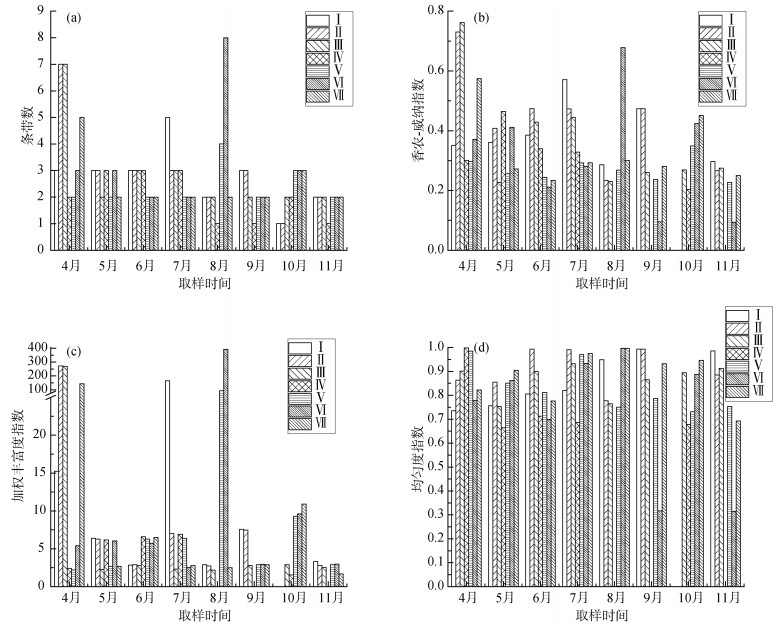 |
| 图 3 基于TGGE图谱不同样品细菌多样性的相关指标 Fig. 3 Correlative indexes of bacterial diversity for different samples based on TGGE patterns 注: a:条带数; b:香农-威纳指数; c:加权丰富度指数; d:均匀度指数 |
本研究采用香农-威纳指数表示原核微生物的多样性, 香农-威纳指数越高, 则说明该样本生物多样性越高。针对同一盐度的样本比较, 4月的香农-威纳指数(H)明显高于其他月份, 推测原因可能为水温较为适中, 约为15~22℃, 适宜在较低温度环境中生存的菌种得以繁殖, 因此菌种多样性较高。在中盐度盐池(Ⅱ~Ⅳ号盐池)中, 5—7月的香农-威纳指数(H)要高于8-11月, 这说明在春季到初夏, 水温适合中盐度盐池中细菌的生长繁殖。盐度较高的盐池, 在夏季香农-威纳指数(H)较低, 而在温度相对较低的春季和秋季升高。推测原因为在夏季由于气温高, 光照时间长, 日间高盐度盐池中卤水温度最高可达到50℃, 高温不利于细菌的生长与繁殖。
加权丰富度指数(Rr)小于10, 则说明该环境不适宜微生物生存; 丰富度指数大于30, 则说明该环境非常适宜微生物生存[27]。本实验中4月的Ⅱ、Ⅲ、Ⅶ号样本, 7月的Ⅰ号样本, 八月的Ⅴ、Ⅵ号样本的丰富度指数较高外, 大多数样本的丰富度指数(Rr)都低于10, 这说明盐田环境不适宜微生物的生存繁殖, 只有耐受性强的极端嗜盐和耐盐微生物的生物量较高。马灌楠[32]等2012年采用DGGE法对普通日晒盐田的微生物群落进行了分析, 所有样本的加权丰富度指数在12.15~20.08之间。与普通日晒盐田相比, 淡化浓海水盐田的微生物的丰富度指数较低, 且普通日晒盐田中的优势种群与本文中的优势种群不同, 这都表明淡化浓海水引入盐田对盐池中原有的微生物群落造成了一定的影响。
针对同一月份内均匀度系数(E)分析发现, 4—8月间各样本的均匀度系数相差不大, 说明在这几个月中, 同一盐度盐池中细菌群落结构比较稳定, 变化不显著。且同一个月份不同盐度盐池中的细菌群落结构也比较稳定, 变化不显著。9—11月间各样本的均匀度系数变化较大, 说明在温度逐渐降低的秋冬季节, 盐田中各盐池的细菌群落结构发生了变化。盐度不同的盐池中细菌群落结构变化较大, 随着盐度逐渐升高随着盐度逐渐升高, Spiribacter salinus逐渐取代Pseudoalteromonas sp.成为占主要地位的优势菌种。
3 结论本研究采用PCR-TGGE和测序方法, 分析了天津汉沽淡化浓海水盐田原核微生物多样性现状, 了解了一年中不同盐度盐池的原核微生物群落结构变化。结果表明淡化浓海水和溴后水中, 原核微生物量非常少, 经PCR扩增未被检测到。淡化浓海水排入日晒盐田后, 微生物逐渐在卤水中繁殖。
在天津汉沽淡化浓海水日晒盐田中, 优势细菌主要为γ变形菌门(γ-Proteobacteria)中的Spiribacter salinus和 Pseudoalteromonas sp.。优势古菌主要为广古菌门(Euryarchaaeota)中的Halogeometricum sp.仅出现在盐度最高的Ⅶ号盐池中。
香农-威纳指数分析结果表明中盐度盐池中原核微生物的多样性春季高于夏季。丰富度指数较低表明盐田环境不利于微生物的生长和繁殖。均匀度系数分析结果表明春夏季节各盐池中细菌群落结构较稳定, 秋冬季节盐度不同的盐池中细菌群落结构变化较大, 随着盐度逐渐升高, Spiribacter salinus逐渐取代Pseudoalteromonas sp.成为占主要地位的优势菌种。
| [1] |
李爱民, 王威, 张志香, 等. 盐田良性生态系统中嗜盐菌的研究[J]. 南开大学学报:自然科学版, 1994(4): 68-72. Li Aimin, Wang Wei, Zhang Zhixiang, et al. Studies on halophilic bacteria in a balanced salt pan ecosystem[J]. Acta Scientiarum Naturalium Universitatis Nankaiensis, 1994(4): 68-72. |
| [2] |
Moral A D, Roldan E, Navarro J, et al. Formation of calcium carbonate crystals by moderately halophilic bacteria[J]. Geomicrobiology Journal, 1987, 5(1): 79-87. DOI:10.1080/01490458709385958 |
| [3] |
Rivadeneyra M A, Delgado R, Párraga J, et al. Precipitation of minerals by 22 species of moderately halophilic bacteria in artificial marine salts media:influence of salt concentration[J]. Folia Microbiologica, 2006, 51(5): 445-453. DOI:10.1007/BF02931589 |
| [4] |
李岿然, 白洁. 盐田生态系统结构分析[J]. 海洋科学, 1998, 22(5): 36-39. Li Kuiran, Bai Jie. Analysis of structure on salt field ecosystem[J]. Marine Sciences, 1998, 22(5): 36-39. |
| [5] |
李娜.浓海水理化生态特性及对盐业生产影响的研究[D].天津: 河北工业大学, 2006. Li Na. Study on physico-chemical zoology characteristic of desalinated seawater and effect in salt technics[D]. Tianjin: Hebei University of Technology, 2006. |
| [6] |
Dieter G, Jacob S, Luise G, et al. Microbial mats and physicochemistry in a saltern in the Bretagne (France) and in a laboratory scale saltern model[J]. Fems Microbiology Letters, 1989, 62(3): 151-161. DOI:10.1111/j.1574-6968.1989.tb03689.x |
| [7] |
朱梁, 柴子华. 海水淡化与制盐相关性研究初探[J]. 中国盐业, 2016(24): 30-34. Zhu Liang, Chai Zihua. Preliminary study on correlation between seawater desalination and salt making[J]. China Salt Industry, 2016(24): 30-34. |
| [8] |
邢立谦, 陈延辉. "发电-海水淡化-制盐及盐化工"技术发展展望[J]. 盐业与化工, 2012, 41(3): 1-3. Xing Liqian, Chen Yanhui. Forecast of technology development for electricity generation-sea water desalination-salt and salt chemical engineering[J]. Journal of Salt & Chemical Industry, 2012, 41(3): 1-3. |
| [9] |
欧阳琼梅. 海水淡化及制盐循环经济项目分析[J]. 中国井矿盐, 2014(4): 40-43. Ouyang Qiongmei. Desalination of sea water and analysis on the recycling economy project of salt production[J]. China Well & Rock Salt, 2014(4): 40-43. DOI:10.3969/j.issn.1001-0335.2014.04.015 |
| [10] |
于海淼.天津北疆发电厂循环经济项目可行性研究[D].天津: 天津大学, 2012. Yu Haimiao. Research on recycling economy project feasibility in Tianjin Beijing power plant[D]. Tianjin: Tianjin University, 2012. |
| [11] |
王涛, 王亮, 张亚南, 等. 淡化副产浓海水对滩晒制盐影响研究[J]. 盐业与化工, 2014, 43(11): 20-24. Wang Tao, Wang Liang, Zhang Yanan, et al. Study on the influence of concentrated seawater after desalination on saltern production[J]. Journal of Salt and Chemical Industry, 2014, 43(11): 20-24. |
| [12] |
Pedrós-Alió C, Calderón-Paz J, Maclean MH, et al. The microbial food web along salinity gradients[J]. Fems Microbiology Ecology, 2000, 32(2): 143-155. DOI:10.1111/j.1574-6941.2000.tb00708.x |
| [13] |
Benlloch S. Prokaryotic genetic diversity throughout the salinity gradient of a coastal solar saltern[J]. Environmental Microbiology, 2002, 4(6): 349-360. DOI:10.1046/j.1462-2920.2002.00306.x |
| [14] |
Ruth AS, Evy FS, Gunnar B. Virioplankton community structure along a salinity gradient in a solar saltern[J]. Extremophiles, 2003, 7(5): 347-351. DOI:10.1007/s00792-003-0328-5 |
| [15] |
Jesse D, Mark C, Abraham G, et al. Patterns of microbial diversity along a salinity gradient in the Guerrero Negro solar saltern, Baja CA Sur, Mexico[J]. Frontiers in Microbiology, 2013, 4(399): 399. |
| [16] |
Oren A. Halophilic microbial communities and their environments[J]. Current Opinion in Biotechnology, 2015, 33: 119-124. DOI:10.1016/j.copbio.2015.02.005 |
| [17] |
Kushner DJ. The Halobacteriaceae[A]. Sokatch J R, Ornston L N, Ed. The Bacteria, a Treatise on Structure and Function, Vol Ⅷ[M]. San Diego: Academic Press, 1985: 171-206.
|
| [18] |
Kabilan M, Sivaraman C, Bhakti BS, et al. Comparison of bacterial diversity from solar salterns and a simulated laboratory study[J]. Annals of Microbiology, 2015, 65(2): 995-1005. DOI:10.1007/s13213-014-0944-6 |
| [19] |
Manel BA, Fatma K, Najla M, et al. Prokaryotic diversity in a Tunisian hypersaline lake, Chott El Jerid[J]. Extremophiles Life Under Extreme Conditions, 2016, 20(2): 1-14. |
| [20] |
Meglio LD, Santos F, Gomariz M, et al. Seasonal dynamics of extremely halophilic microbial communities in three Argentinian salterns[J]. Fems Microbiology Ecology, 2016, 92(12). |
| [21] |
Zhang JJ, Ma GN, Deng YG, et al. Bacterial Diversity in Bohai Bay Solar Saltworks, China[J]. Current Microbiology, 2016, 72(1): 1-9. |
| [22] |
Emilio C, Ramon M, Susana B, et al. Changes in archaeal, bacterial and eukaryal assemblages along a salinity gradient by comparison of genetic fingerprinting methods in a multipond solar saltern[J]. Environmental Microbiology, 2002, 4(6): 338-348. DOI:10.1046/j.1462-2920.2002.00297.x |
| [23] |
朱大玲, 唐啸龙, 张宝玉, 等. 一株海藻多糖降解菌的分离鉴定及产酶条件优化[J]. 海洋科学, 2017, 41(8): 99-107. Zhu Daling, Tang Xiaolong, Zhang Baoyu, et al. Isolation identification and enzyme-producing conditions analysis of a seawater polysaccharides-degrading bacteria[J]. Marine Sciences, 2017, 41(8): 99-107. |
| [24] |
Zhang D, Li W, Zhang S, et al. Evaluation of the impact of DNA extraction methods on BAC bacterial community composition measured by denaturing gradient gel electrophoresis[J]. Letters in Applied Microbiology, 2011, 53(1): 44-49. DOI:10.1111/j.1472-765X.2011.03064.x |
| [25] |
Pires A, Cleary D, Almeida A, et al. Denaturing gradient gel electrophoresis and barcoded pyrosequencing reveal unprecedented archaeal diversity in mangrove sediment and rhizosphere samples[J]. Applied & Environmental Microbiology, 2012, 78(16): 5520. |
| [26] |
Tamura K, Peterson D, Peterson N, et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology & Evolution, 2011, 28(10): 2731. |
| [27] |
Marzorati M, Wittebolle L, Boon N, et al. How to get more out of molecular fingerprints:practical tools for microbial ecology[J]. Environmental Microbiology, 2010, 10(6): 1571-1581. |
| [28] |
Kabilan M, Sivaraman C, Bhakti S, et al. Comparison of bacterial diversity from solar salterns and a simulated laboratory study[J]. Annals of Microbiology, 2015, 65(2): 995-1005. DOI:10.1007/s13213-014-0944-6 |
| [29] |
王彩霞.渤海海域微生物群落结构的时空变化及其对环境压力的响应[D].山东烟台: 中国科学院烟台海岸带研究所, 2018. Wang Caixia. Spatial and temporal variation of microbial community structure in the Bohai sea and its response to environmental pressure[D]. Yantai, Shandong Province: Yantai Institute of Coastal Zone Research, Chinese Academy of Sciences, 2018. http://cdmd.cnki.com.cn/Article/CDMD-80180-1018978066.htm |
| [30] |
Gerard M, Kornelia S. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis (TGGE) in microbial ecology[J]. Antonie Van Leeuwenhoek, 1998, 73(1): 127-141. DOI:10.1023/A:1000669317571 |
| [31] |
冯佩英, 陆春, 朱国兴. DGGE/TGGE技术在微生物基因分类鉴定中的应用[J]. 国际检验医学杂志, 2005, 26(2): 95-97. Feng Peiying, Lu Chun, Zhu Guoxing. Application of DGGE/TGGE technology in the identification of microbial genes[J]. Foreign Medical Sciences, 2005, 26(2): 95-97. DOI:10.3969/j.issn.1673-4130.2005.02.011 |
| [32] |
马灌楠.高盐环境下微生物群落结构与多样性研究[D].天津: 天津科技大学, 2015. Ma Guannan. Study on microbial community structure and diversity in hypersaline conditions[D]. Tianjin: Tianjin University of Science & Technology, 2015. |
 2019, Vol. 43
2019, Vol. 43


