
文章信息
- 李妍, 李培海, 刘长水, 马庆军. 2019.
- LI Yan, LI Pei-hai, LIU Chang-shui, MA Qing-jun. 2019.
- 一株副溶血弧菌D3112致病性和抗药性的表征
- Characterization of pathogenicity and drug resistance in Vibrio parahaemolyticus D3112
- 海洋科学, 43(6): 25-33
- Marine Sciences, 43(6): 25-33.
- http://dx.doi.org/10.11759/hykx20190324001
-
文章历史
- 收稿日期:2019-03-24
- 修回日期:2019-04-24
2. 青岛海洋科学与技术试点国家实验室海洋生物与生物技术功能实验室, 青岛 266273;
3. 中国科学院大学, 北京 100049;
4. 中国科学院海洋大科学研究中心, 青岛 266071
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao, 266273;
3. University of Chinese Academy of Sciences, Beijing 100049;
4. Centre for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071
副溶血弧菌(Vibrio parahaemolyticus)是许多水产养殖动物如鱼类、贝类和螃蟹等的重要流行病原菌之一, 可导致高死亡率的弧菌病, 给水产养殖业带来巨大的经济损失[1]。更为严重的是, 副溶血弧菌能通过生鲜或不熟的海产品, 引发人类急性肠胃炎[2]。据美国疾病控制和预防中心统计, 弧菌在美国每年可引起80 000多例疾病, 其中超过50%的病例是由副溶血弧菌引起的(https://www.cdc.gov/vibrio/faq.html)。
副溶血弧菌主要分布在大洋和沿海环境中。据报道, 随着全球海水温度的不断升高, 弧菌加速增殖, 其致病性的传播范围也有所扩大[3-4]。尽管多数环境菌株尚未证明有临床致病性, 但它们常常携有毒力相关基因, 因而具备潜在的致病力。监测环境菌的致病性或有助于预防弧菌病的暴发。目前对于环境菌的致病性研究, 常常关注部分关键毒力基因。溶血素基因被认为是副溶血弧菌形成潜在毒力的主要因素, 已成为区分毒力与非毒力菌株的关键性标志物[5]。溶血素主要包括TDH(thermostable direct haemolysin)和TRH(thermostable-related haemolysin)。通过PCR技术检测tdh和trh基因已广泛用于评估环境菌株是否具有潜在致病性[6-7]。然而据报道, 部分临床致病菌株并不含有tdh和trh基因[8], 对有些菌株, Δtdh/Δtrh突变株和野生株的致病性并没有显著差异[9], 表明tdh和trh基因并非不可或缺的致病基因。因此, 即使菌株缺少tdh/trh基因, 仍无法排除其具有致病的可能, 而仅仅检测这些基因是否存在, 可能低估环境菌的致病风险。所以, 其他毒力相关基因, 如Ⅲ型分泌系统基因、ureR、orf8和toxRS, 也用作辅助致病性标志物[10-12]。然而, 目前由于尚未建立副溶血弧菌致病性的确切遗传基础, 所以找到一套合适的分子标志物来准确评估环境菌的潜在致病性并非易事。对副溶血弧菌的致病性进行遗传学和表型信息研究, 将有助于我们进一步理解环境中副溶血弧菌基因型与致病性之间关系。
为了更好地了解副溶血弧菌在海洋环境中的潜在致病性, 我们从海水样品中分离出副溶血弧菌菌株D3112, 并分析其基因组、致病性特征及耐药谱。本文为准确评价环境菌的潜在致病性提供了有用的数据信息, 对副溶血弧菌引起的弧菌病的防治具有重要的价值和意义。
1 材料与方法 1.1 实验材料 1.1.1 主要试剂细菌基因组提取试剂盒购于天根生化科技公司; PrimeSTAR Max Premix(2×)高保真DNA聚合酶和DL2000 DNA Ladder购于TaKaRa生物技术有限公司; 细菌微量生化鉴定试剂盒购于青岛海博; 药敏试纸购于杭州微生物; 脱脂奶粉、Tween 80、明胶、可溶性淀粉购于北京索莱宝科技有限公司。
1.1.2 培养基2216E培养基; TCBS培养基(北京陆桥)[13]。
1.2 实验方法 1.2.1 细菌的分离和纯化从黄海海域(36°07′N, 120°38′E, 山东青岛)收集的海水样品, 将样品用无菌蒸馏水稀释至10–1~10–3, 各浓度取100 μL用无菌涂布棒涂至TCBS固体平板上, 26℃培养24 h。挑选蓝绿色、圆形菌落用划线法进一步纯化, 直至得到纯种菌株。
1.2.2 鉴定菌株以保守的16S rRNA基因、热激蛋白基因hsp60 (GenBank序列号: AAK69016.1)和副溶血弧菌特有的铁调节型毒力调节蛋白基因irgB(Genbank序列号: vp2603)三者为目的基因, 鉴定此菌株, 命名为D3112。引物见表 1。所有的PCR扩增产物经测序后, 分别与GenBank核酸数据库进行Blast序列比对(https://blast.ncbi.nlm.nih.gov/Blast.cgi)。此外, 还使用了细菌微量生化鉴定试剂盒对菌株加以鉴定。
| 目标基因 | 引物序列 | 来源 |
| 16s-27F | 5′-AGAGTTTGATCCTGGCTCAG-3′ | |
| 16s-1492R | 5′-GGTTACCTTGTTACGACTT-3′ | |
| hsp60-F | 5′-ACAACAGCAACGGTACTAGC-3′ | [14-15] |
| hsp60-R | 5′-CAACTTTCACGATGCCAC-3′ | |
| irgB-F | 5′-CGATACACACCACGATCCAG-3′ | [16] |
| irgB-R | 5′-ATACGGCCGGGGTGATGTTTCT-3′ | |
| tlh-F | 5′-AAAGCGGATTATGCAGAAGCACTG-3′ | [17] |
| tlh-R | 5′-GCTACTTTCTAGCATTTTCTCTGC-3′ | |
| tdh-F | 5′-GTAAAGGTCTCTGACTTTTGGAC-3′ | [18] |
| tdh-R | 5′-TGGAATAGAACCTTCATCTTCACC-3′ | |
| trh-F | 5′-TTGGCTTCGATATTTTCAGTATCT-3′ | |
| trh-R | 5′-CATAACAAACATATGCCCATTTCCG-3′ |
将提取的基因组送到天津生物芯片技术有限公司进行全基因组测序。本研究采用单分子测序方法进行测序, 通过PacBio RS Ⅱ平台的测序数据[19], 使用PacBio SMRT Analysis ver. 2.3.0软件的HGAP模块进行基因序列的重新组装。采用Prodigal软件预测开放式阅读框(ORFs)。使用Barrnap和tRNAscan预测包括rRNA和tRNA在内的非编码序列。使用BLAST进行功能预测和注释。根据基因组序列, 构建系统进化树。利用毒力因子数据库(VFDB)和综合抗生素研究数据库(CRGD)对副溶血性弧菌D3112的潜在毒力基因和耐药基因进行比对分析。
1.2.4 溶血活性检测采用PCR技术检测副溶血弧菌中常见的三种溶血素基因tdh、trh和tlh(thermolabile hemolysin)是否存在于菌株D3112中, 引物见表 1。此外, 用改良的Wagatsuma琼脂平板检测神奈川现象[20], 即将5 μL菌液滴加至含有5%脱纤维绵羊血的琼脂平板上, 检测菌株是否具有溶血活性, 如果在培养板上的液滴周围出现水解圈, 说明细菌具有溶血活性。
1.2.5 胞外酶活性检测通过改良的琼脂平板法, 检测几种胞外酶的活性[21]。首先分别制备内含10%脱脂奶粉、1% Tween 80、10%明胶、10%蛋黄液或1%可溶性淀粉的2216E固体平板。将5 μL处于对数生长期的菌液滴加在上述平板中, 晾干, 于26℃培养24 h后观察。如果在含有特定物质的平板上出现透明圈(平板含脱脂奶粉、明胶或可溶性淀粉)、晕圈(平板含Tween 80)或浑浊圈(平板含蛋黄液), 则表明测试结果为阳性。
1.2.6 人工感染实验用不同剂量的D3112菌液对体型相当的健康蓝斑马鱼进行人工感染, 以检测菌株致病性。在感染之前, 蓝斑马鱼禁食一周以适应环境。感染实验中, 菌株D3112于26℃活化后, 转接至2216E液体培养基, 培养至对数生长中后期时进行收集, 菌体使用0.85%的无菌生理盐水悬洗, 并梯度稀释至1×109, 1×108, 1×107, 1×106和1×105 CFU/mL 5个终浓度。取50 μL各浓度的菌液, 注射至斑马鱼的腹腔内。设置5个实验组, 一个对照组(注射等体积的无菌生理盐水), 每组均设置3个平行, 每个平行设置8尾鱼。实验重复两次。鱼体注射后, 每天记录斑马鱼的死亡情况。将感染致死的斑马鱼在超净工作台中解剖, 并将鱼腹腔内的物质溶于2216E中, 稀释100倍后涂布至TCBS固体培养基, 于26℃培养。挑取单克隆菌落并活化后, 提取基因组, 用16S rRNA基因和hsp60基因引物做PCR扩增, 将扩增产物测序。测序结果与GenBank核酸数据库进行比对, 以确定菌株是否为D3112。
1.2.7 药物敏感实验本实验采用美国临床实验室标准化研究所(CLSI)推荐的标准纸片扩散法, 检测D3112对30种抗生素的耐药性。将2216E固体培养基灭菌, 冷却至35℃左右后, 1/100的比例加入对数生长期的菌液, 轻轻摇晃均匀后倒平板。待平板晾干后, 将药敏试纸放至平板中央, 每种抗生素各做3组平行。平板于26℃放置24 h后, 观察是否有抑菌圈形成, 对形成抑菌圈的直径大小加以测量和记录。按照CLSI标准判断菌株对不同抗生素的敏感和耐受程度。本实验同时使用大肠杆菌BW25113做质控菌株。
2 结果 2.1 分离并鉴定副溶血弧菌从海水分离的菌株D3112在TCBS培养基上呈现出蓝绿色圆形克隆, 为典型的副溶血弧菌形态特征。生化鉴定试剂盒检测结果如表 2所示, 这些特征与《伯杰氏细菌鉴定手册》上描述的副溶血弧菌的相同, 所以初步鉴定为副溶血弧菌。此外, 由于16S rRNA、hsp60和irgB基因的核苷酸序列经常被用于副溶血弧菌的分类学定位[14, 22-23], 因此我们以提取的菌株基因组为模板, 对这3个基因进行扩增和测序, 并将测序结果在GenBank数据库中比对。比对结果显示三个基因序列均与副溶血弧菌最为接近, 其中16S rRNA基因序列与副溶血弧菌的相似度高达99%, hsp60和irgB基因的序列相似度也分别达97%和98%。因此, 我们初步确认分离的菌株为副溶血弧菌。
| 特性 | 结果 |
| 氧化酶 | + |
| 赖氨酸脱酸酶 | + |
| β半乳糖苷酶 | - |
| 运动性 | + |
| Voges–Proskauer | - |
| 无盐蛋白胨水 | - |
| 6% NaCl蛋白胨水 | + |
| 8% NaCl蛋白胨水 | + |
| 10% NaCl蛋白胨水 | - |
| 42℃ | + |
| O/F | F |
| 甘露醇 | + |
| 蔗糖 | - |
| 注: “+”, 阳性, “-”, 阴性; “O”, 氧化型, “F”, 发酵型。 | |
我们对菌株D3112进行了全基因组测序。该菌株具有2个染色体, 大小分别为3 239 077 bps(染色体Ⅰ)和1 886 547 bps(染色体Ⅱ), G+C含量分别为45.40%和45.32%。染色体Ⅰ含有2891个开放阅读框(ORFs)、34个rRNA基因和118个tRNA基因, 染色体Ⅱ含有1 672个ORFs, 3个rRNA基因和14个tRNA基因。此外, 该菌株还携有大小分别为66 230 bp和94 438 bp的两个质粒。具体基因组信息见表 3。
| 染色体Ⅰ | 染色体Ⅱ | 质粒1 | 质粒2 | |
| 大小 | 3 239 077 bp | 1 886 547 bp | 66 230 bp | 94 438 bp |
| C+G (%) | 45.40% | 45.32% | 45.005% | 44.448% |
| CDS个数 | 2 891 | 1 672 | 81 | 105 |
| 编码百分比 | 85.29% | 86.60% | 78.43% | 23.81% |
| COGs基因 | 79.49% | 72.41% | 24.69% | 76.76% |
| 平均CDS长度 | 956 bp | 977 bp | 641 bp | 690 bp |
| rRNA个数 | 34 | 3 | 0 | 0 |
| tRNA个数 | 118 | 14 | 0 | 0 |
基于全基因组序列, 我们利用CVTree3软件构建了该菌株的进化树(图 1)。结果表明, 菌株D3112最接近于从拉丁美洲南美白对虾中分离的副溶血弧菌菌株R13。D3112的全基因组序列已经提交到GenBank数据库中, 序列号为CP034565.1-CP034568.1。
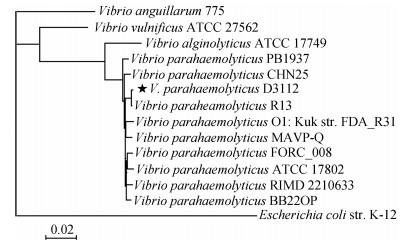 |
| 图 1 全基因组进化树 Fig. 1 Phylogenetic tree based on whole genome sequences |
VFDB收集了包括30个属(74个病原菌)的细菌毒力基因序列信息, 提供了毒力基因的核苷酸和蛋白序列信息[24]。副溶血弧菌毒力因子主要包括粘附素、铁获取系统、运动性、分泌系统、溶血素和毒素[25]。溶血素TDH和TRH与副溶血性弧菌的致病性直接相关[26]。将副溶血弧菌D3112的基因组序列与VFDB数据库中的毒力基因进行比对, 发现D3112并不具有溶血素基因tdh和trh基因, 但却含有几个编码其他溶血素的基因, 如不耐热溶血素基因(D3112_g2_0773)、溶血素VllY基因(D3112_g1_ 1175), 以及一些潜在的溶血素基因。
铁获取系统在病原菌产生毒力的过程中至关重要[27]。D3112含有很多与铁获取相关的基因(表 4), 如铁载体vibrioferrin合成蛋白基因和转运蛋白基因。D3112还能编码利用肠菌素(enterobactin)等异源铁载体蛋白和转运铁-铁载体的Fhu系统。因此, 该菌株具有从环境和潜在宿主中摄取和捕获铁的能力。分泌系统, 尤其是Ⅲ型分泌系统, 与副溶血弧菌的致病性息息相关, 在D3112基因组中存在30多个编码Ⅲ型分泌系统相关蛋白的基因。此外, D3112还能编码胞外金属蛋白酶(D3112_g1_1338)、粘附素和运动性相关蛋白(表 4)。总之, D3112基因组含有一系列假定的毒力基因, 这表明该菌株具有潜在的致病性。
| 基因名称 | 序列号 | 基因位置 |
| 铁载体合成和运输相关基因 | ||
| pvsA-pvsE | D3112_g2_0458-0462 | Chr2 |
| pvuEDCBA | D3112_g2_0452-0456 | Chr2 |
| vctPDGC、vctA | D3112_g2_1152-1155、1158 | Chr2 |
| fhuACDB | D3112_g2_0244-0247 | Chr2 |
| Ⅲ型分泌系统 | ||
| yscW、yscCD、yscF-yscL、yscUTSROPQN、yscXYV | D3112_g1_1270、1274-1275、1277-1283、1296-1303、1307-1309 | Chr1 |
| cesT | D3112_g1_1284、1286 | Chr1 |
| yopSRQNBD | D3112_g1_1285、1287、1289、1304、1314、1315 | Chr1 |
| lcrR、lcrG、lcrH | D3112_g1_1310、1311、1313 | Chr1 |
| vecA | D3112_g1_1288 | Chr1 |
| tyeA | D3112_g1_1305 | Chr1 |
| Ⅳ型菌毛 | ||
| pilM- pilQ、pilU、pilDCBA、pilZWE | D3112_g1_0270-0274、0403、0487-0490、0581、2245、2247 | Chr1 |
| tadBC | D3112_g1_0599、0600 | Chr1 |
| MSHA type 4 | ||
| mshH-mshN、mshEGFA、mshO-mshQ | D3112_g1_0309-0316、0317-0320、0324-0326 | Chr1 |
| cpaBC | D3112_g1_0591、0592 | Chr1 |
| fimT | D3112_g1_2246 | |
| 毒素 | ||
| tcpN/toxT | D3112_g1_0587 | Chr1 |
| tcpI | D3112_g1_0826 | Chr1 |
| 运动性和趋化性 | ||
| chpE | D3112_g1_0042 | Chr1 |
| fliL、fliY、fliA、fliR-fliE | D3112_g1_0095、0858、2002、2007-2020 | Chr1 |
| fliJ-fliE | D3112_g2_0337-0342 | Chr2 |
| motY、motAB | D3112_g2_0344、0361-0362 | Chr2 |
| motX | D3112_g1_0215 | Chr1 |
| cheV3、cheV | D3112_g2_0489、0178 | Chr2 |
| cheM、cheY | D3112_g1_0905、1456 | Chr1 |
TDH或TRH依赖型溶血活性被认为是副溶血弧菌致病性的典型标志。然而PCR检测发现, D3112缺少了tdh和trh基因(图 2), 这与基因组分析结果一致。尽管如此, 绵羊血琼脂平板上却依然出现了明显的溶血圈, 显示了很强的溶血活性。因而我们推测, 基因组中存在的其他溶血素基因的表达或导致了溶血现象。
 |
| 图 2 副溶血弧菌D3112溶血活性检测 Fig. 2 Hemolysis detection of V. parahaemolyticus D3112 注: (a)M为DL2000 DNA Ladder, 第1、2、3、4泳道分别是以副溶血弧菌特有的irgB基因(对照)、溶血素tlh、trh和tdh基因设计引物, 以菌株D3112基因组为模板进行PCR扩增的产物。(b)M为DL2000 DNA Ladder, 第1、2、3、4泳道分别是以副溶血弧菌ATCC 17802标准株的基因组为模板, 以irgB(对照)、tlh、trh和tdh基因设计引物进行PCR扩增的产物。(c)菌株D3112在绵羊血琼脂平板上出现溶血圈, 显示出溶血活性 |
蛋白酶、脂酶、卵磷脂酶、淀粉酶和明胶酶等胞外酶通常被认为是弧菌重要的毒力因子[28-29], 这些酶已广泛用于检测弧菌潜在的致病性[30]。将菌株D3112滴加至脱脂奶粉平板、Tween 80平板和明胶平板后出现了明显的水解圈或晕圈, 而蛋黄液平板却未出现显著的浑浊圈, 这表明该菌株呈现出蛋白酶、脂酶和明胶酶活性, 但不具有卵磷脂酶活性。另外, 在该菌株生长的淀粉平板上滴加1%碘液后, 出现了透明圈, 显示出菌株具有淀粉酶活性。结果见表 5。以上结果证明, 菌株D3112能表达多种胞外酶, 可能会引发潜在的致病性。
| 胞外酶 | 结果 |
| 蛋白酶 | + |
| 脂酶 | + |
| 卵磷脂酶 | – |
| 淀粉酶 | + |
| 明胶酶 | + |
| 注: “+”, 阳性, “–”, 阴性 | |
为了评价菌株的潜在毒力, 我们用斑马鱼进行了人工感染实验。结果显示, 在注射剂量不小于5×105 CFU/尾时, 注射12 h后, 实验鱼体均表现出明显的出血症状, 甚至死亡, 表明该菌株具有发病快的特点。从实验组鱼的出血部位分离菌株, 在TCBS培养基中分离培养, 菌落均呈现副溶血弧菌标志性的绿色。随机挑选5个单克隆菌落, 通过PCR扩增16S rRNA基因和hsp60基因并测序, 测序结果均与D3112相应基因序列一致。
人工感染实验测得D3112对斑马鱼的半数致死量为5×105 CFU/尾, 5×107 CFU/尾的剂量可使死亡率达到100%, 而对照组的鱼全部存活(见表 6)。当注射剂量超过半致死量LD50时, 在斑马鱼鳃部和消化腺都观察到了溶血现象, 患病症状与报道的致病性副溶血弧菌感染海洋鱼类的症状相似[31]。但是当注射更高剂量的菌液时, 这种现象遍布鱼体。感染现象见图 3。
| 剂量(CFU) | 死亡(尾) | 总数(尾) | 死亡率(%) | |
| 实验组 | 5×107 | 24 | 24 | 100 |
| 5×106 | 21 | 24 | 87.5 | |
| 5×105 | 12 | 24 | 50 | |
| 5×104 | 3 | 24 | 12.5 | |
| 5×103 | 0 | 24 | 0 | |
| 对照组 | 生理盐水 | 0 | 24 | 0 |
 |
| 图 3 副溶血弧菌D3112感染后的斑马鱼症状 Fig. 3 Symptom of the zebrafishes infected with V. parahaemolyticus 注:上方为对照组的鱼, 下方为实验组的鱼 |
鉴于该菌株是潜在的致病菌, 我们同时分析了菌株对不同抗生素的敏感性。结果显示, 按照美国CLSI标准中不同抗生素对细菌抑菌圈的直径范围, 该菌株对多西环素、呋喃唑酮、复方新诺明、多粘菌素B和氯霉素高度敏感, 而对头孢类、氨基糖苷类和米诺环素中度敏感。然而, 该菌株还表现出多重耐药性, 特别是青霉素类抗生素。而对青霉素类、喹诺酮类以及四环素、克林霉素、万古霉素、红霉素表现出多重耐药性。具体结果见表 7。D3112基因组中发现的多重耐药基因从分子水平上解释了菌株耐药的原因(表 8)。
| 抗生素种类 | 名称 | 含量(μg/片) | 抑菌圈(mm) | 敏感性 |
| 青霉素类 | 氨苄西林 | 10 | 11 | R |
| 羧苄西林 | 100 | 7 | R | |
| 苯唑西林 | 1 | 7 | R | |
| 阿莫西林 | 20 | 7 | R | |
| 青霉素 | 10U | 7 | R | |
| 哌拉西林 | 100 | 17 | R | |
| 头孢菌素类 | 头孢呋辛 | 30 | 18 | I |
| 头孢拉定 | 30 | 16 | I | |
| 头孢唑林 | 30 | 19 | S | |
| 头孢氨苄 | 30 | 18 | S | |
| 头孢他啶 | 30 | 24 | S | |
| 头孢哌酮 | 75 | 16 | I | |
| 头孢曲松 | 30 | 20 | S | |
| 喹诺酮类 | 环丙沙星 | 5 | 13 | R |
| 氧氟沙星 | 5 | 12 | R | |
| 诺氟沙星 | 10 | 12 | R | |
| 素环素类 | 米诺环素 | 30 | 18 | I |
| 多西环素 | 30 | 15 | S | |
| 四环素 | 30 | 12 | R | |
| 氨基糖苷类 | 丁胺卡那 | 30 | 18 | I |
| 卡那霉素 | 30 | 14 | I | |
| 新霉素 | 30 | 16 | I | |
| 庆大霉素 | 10 | 14 | I | |
| 呋喃类 | 呋喃唑啉 | 300 | 32 | S |
| 磺胺类 | 复方新诺明 | 25 | 20 | S |
| 多粘菌素类 | 多粘菌素B | 300 IU | 16 | S |
| 氯霉素类 | 氯霉素 | 30 | 26 | S |
| 作用于G+细菌 | 克林霉素 | 2 | 7 | R |
| 多肽类 | 万古霉素 | 30 | 8 | R |
| 大环内酯类 | 红霉素 | 150 | 9 | R |
| 注:药敏纸片直径为7 mm。“S”:敏感, “I”:中度敏感, “R”:耐受 | ||||
| 基因作用 | 基因名称 | 基因序列号 | 基因位置 |
| β-内酰胺酶抗性 | bla | D3112_g2_0146、0609 | Chr2 |
| 四环素修饰酶 | tet 34 | D3112_g1_2231 | Chr1 |
| 多药抗性 | mdtL | D3112_g1_0034 | Chr1 |
| 多药抗性 | mdtL | D3112_g2_0105、0377 | Chr2 |
| 多药抗性 | mdtA | D3112_g2_0905、1391 | Chr2 |
| 多药抗性 | mdtG | D3112_g2_0460 | Chr2 |
| 多药抗性 | emrA | D3112_g2_1017 | Chr2 |
| 多药抗性 | vmeE、vmeG | D3112_g1_0806、1010 | Chr1 |
| 多药抗性 | norM | D3112_g1_1460 | Chr1 |
| 多药抗性 | mdlA、mdlB | D3112_g1_1503、1504 | Chr1 |
环境细菌是毒力基因和耐药基因的重要载体, 因此监测环境细菌的致病性可以防控潜在疾病的暴发。副溶血弧菌作为人畜共患菌, 可引发宿主伤口感染、败血症和急性肠炎, 对水产养殖业和人类健康带来巨大的危害。本研究对野外环境中分离的副溶血弧菌进行了全基因组测序和致病性描述, 发现该菌株对斑马鱼具有致病毒力, 并且不依赖于TDH和TRH。先前广泛认为, TDH和TRH是致病性副溶血弧菌的典型标志物[17], 然而, Chung等从鱼缸水中分离的副溶血弧菌虽然没有TDH和TRH, 但是基因组中含有其他的致病基因, 体外细胞实验也表现了明显的细胞毒性[32]。以上综合本研究可知, 仅通过检测tdh/trh基因的存在来判定副溶血弧菌的致病性仍尚有欠缺。我们可能需要选择tdh和trh基因以外的更多标志物对弧菌的致病性进行更加全面准确的评估, 并在必要时通过活体感染实验进一步加以佐证。
本研究分离的菌株编码了一系列假定的毒力因子, 均有望成为菌株致病性诊断的潜在标志。目前, 来自不同环境的副溶血弧菌的全基因序列信息量仍不充足, 尚无法找到更为可靠的标志物来判断菌株的致病性。随着此类基因组序列的报道不断增多, 副溶血弧菌致病性的基因基础将愈加清晰, 而通过基因和致病性的相关性分析, 我们将有望找到更好的标志基因, 从而更加准确的诊断环境细菌的致病潜力, 对建设健康的海洋环境具有重要的价值和意义。
| [1] |
Joseph S W, Colwell R R, Kaper J B. Vibrio parahaemolyticus and related halophilic Vibrios[J]. Critical Reviews in Microbiology, 1982, 10(1): 77-124. DOI:10.3109/10408418209113506 |
| [2] |
Wang C C, Krantz E, Klarquist J, et al. Acute hepatitis C in a contemporary US cohort: modes of acquisition and factors influencing viral clearance[J]. Journal of Infectious Disease, 2007, 196(10): 1474-1482. DOI:10.1086/522608 |
| [3] |
Yeung P S, Boor K J. Epidemiology, pathogenesis, and prevention of foodborne Vibrio parahaemolyticus infections[J]. Foodborne Pathogens and Disease, 2004, 1(2): 74-88. DOI:10.1089/153531404323143594 |
| [4] |
Vezzulli L, Colwell R R, Pruzzo C. Ocean warming and spread of pathogenic vibrios in the aquatic environment[J]. Microbial Ecology, 2013, 65(4): 817-825. DOI:10.1007/s00248-012-0163-2 |
| [5] |
Broberg C A, Calder T J, Orth K. Vibrio parahaemolyticus cell biology and pathogenicity determinants[J]. Microbes & Infection, 2011, 13(12): 992-1001. |
| [6] |
Alaboudi A R, Ababneh M, Osaili T M, et al. Detection, identification and prevalence of pathogenic Vibrio parahaemolyticus in fish and coastal environment in Jordan[J]. Journal of Food Science, 2016, 81(1): M130-M134. DOI:10.1111/1750-3841.13151 |
| [7] |
Cabrera Garcia M E, Vazquez Salinas C, Quinones Ramirez E I. Serologic and molecular characterization of Vibrio parahaemolyticus strains isolated from seawater and fish products of the Gulf of Mexico[J]. Applied & Environmental Microbiology, 2004, 70(11): 6401-6406. |
| [8] |
Raghunath P. Roles of thermostable direct hemolysin (TDH) and TDH-related hemolysin (TRH) in Vibrio parahaemolyticus[J]. Frontiers in Microbiology, 2015, 5. |
| [9] |
Bhowmik S K, Pazhani G P, Ramamurthy T. Phylogenetic and in silico functional analyses of thermostable- direct hemolysin and tdh-related encoding genes in Vibrio parahaemolyticus and other Gram-negative bacteria[J]. Biomed Research International, 2014, 2014: 576528. |
| [10] |
Kaysner C A, Abeyta C, Trost P A, et al. Urea hydrolysis can predict the potential pathogenicity of Vibrio parahaemolyticus strains isolated in the Pacific Northwest[J]. Applied and Environmental Microbiology, 1994, 60(8): 3020-3022. |
| [11] |
Paranjpye R, Hamel O S, Stojanovski A, et al. Genetic diversity of clinical and environmental Vibrio parahaemolyticus strains from the Pacific Northwest[J]. Applied and Environmental Microbiology, 2012, 78(24): 8631-8638. DOI:10.1128/AEM.01531-12 |
| [12] |
Kewen C, Daodong P, Jun T, et al. Colorimetric integrated PCR protocol for rapid detection of Vibrio parahaemolyticus[J]. Sensors, 2016, 16(10): 1600. DOI:10.3390/s16101600 |
| [13] |
Kobayashi T, Enomoto S, Sakazaki R. A new selective isolation medium for vibrio group (modified Nakanishi's medium-TCBS agar)[J]. Japanese Journal of Bacteriology, 1963, 18(10-11): 387-392. DOI:10.3412/jsb.18.387 |
| [14] |
Kwok A Y C, Wilson J T, Michael C, et al. Phylogenetic study and identification of human pathogenic Vibrio species based on partial hsp60 gene sequences[J]. Canadian Journal of Microbiology, 2002, 48(10): 903-910. DOI:10.1139/w02-089 |
| [15] |
Woods C W, Karpati A M, Grein T, et al. An outbreak of Rift Valley Fever in Northeastern Kenya, 1997-98[J]. Emerging Infectious Diseases, 2002, 8(2): 138-144. DOI:10.3201/eid0802.010023 |
| [16] |
Wang D, Yu S, Chen W, et al. Enumeration of Vibrio parahaemolyticus in oyster tissues following artificial contamination and depuration[J]. Letters in Applied Microbiology, 2010, 51(1): 104-108. |
| [17] |
Taniguchi H, Ohta H, Ogawa M, et al. Cloning and expression in Escherichia coli of Vibrio parahaemolyticus thermostable direct hemolysin and thermolabile hemolysin genes[J]. Journal of Bacteriology, 1985, 162(2): 510-515. |
| [18] |
Bej A K, Patterson D P, Brasher C W, et al. Detection of total and hemolysin-producing Vibrio parahaemolyticus in shellfish using multiplex PCR amplification of tl, tdh and trh[J]. Journal of Microbiological Methods, 1999, 36(3): 215-225. DOI:10.1016/S0167-7012(99)00037-8 |
| [19] |
Rhoads A, Au K F. PacBio sequencing and its applications[J]. Genomics, Proteomics & Bioinformatics, 2015, 13(5): 278-289. |
| [20] |
Chun D, Chung J K, Tak R, et al. Nature of the Kanagawa phenomenon of Vibrio parahaemolyticus[J]. Infection & Immunity, 1975, 12(1): 81-87. |
| [21] |
Amaro C, Biosca E G, Esteve C, et al. Comparative study of phenotypic and virulence properties in Vibrio vulnificus biotype1 and Biotype 2 obtained from a European eel farm experiencing mortalities[J]. Diseases of Aquatic Organisms, 1992, 13(1): 29-35. |
| [22] |
Turenne C Y, Witwicki E, Hoban D J, et al. Rapid identification of bacteria from positive blood cultures by fluorescence based PCR single strand conformation polymorphism analysis of the 16S rRNA gene[J]. Journal of Clinical Microbiology, 2000, 38(2): 513-520. |
| [23] |
Yu S, Chen W, Wang D, et al. Species-specific PCR detection of the food-borne pathogen Vibrio parahaemolyticus using the irgB gene identified by comparative genomic analysis[J]. FEMS Microbiology Letters, 2010, 307(1): 65-71. DOI:10.1111/j.1574-6968.2010.01952.x |
| [24] |
Chen L. VFDB: a reference database for bacterial virulence factors[J]. Nucleic Acids Research, 2004, 33(Database issue): D325-D328. DOI:10.1093/nar/gki008 |
| [25] |
Perez Acosta J A, Martinez Porchas M, Elizalde Contreras J M, et al. Proteomic profiling of integral membrane proteins associated to pathogenicity in Vibrio parahaemolyticus strains[J]. Microbiology and Immunology, 2018, 62(1): 14-23. DOI:10.1111/1348-0421.12556 |
| [26] |
Miyamoto Y, Kato T, Obara Y, et al. In vitro Hemolytic characteristic of Vibrio parahaemolyticus: its close correlation with human pathogenicity[J]. Journal of Bacteriology, 1969, 100(2): 1147-1149. |
| [27] |
Kim S, Chung H Y, Lee D H, et al. Complete genome sequence of Vibrio parahaemolyticus strain FORC_008, a foodborne pathogen from a flounder fish in South Korea[J]. Pathogens Disease, 2016, 74(5): ftw044. DOI:10.1093/femspd/ftw044 |
| [28] |
Ahmed H A, El Bayomi R M, Hussein M A, et al. Molecular characterization, antibiotic resistance pattern and biofilm formation of Vibrio parahaemolyticus and V. cholerae isolated from crustaceans and humans[J]. International Journal of Food Microbiology, 2018, 274: 31-37. DOI:10.1016/j.ijfoodmicro.2018.03.013 |
| [29] |
Hiyoshi H, Kodama T, Iida T, et al. Contribution of Vibrio parahaemolyticus virulence factors to cytotoxicity, enterotoxicity, and lethality in mice[J]. Infection & Immunity, 2010, 78(4): 1772-1780. |
| [30] |
Rodrigues D P, Ribeiro R V, Alves R M, et al. Evaluation of virulence factors in environmental isolates of Vibrio species[J]. Memórias do Instituto Oswaldo Cruz, 1993, 88(4): 589-592. DOI:10.1590/S0074-02761993000400016 |
| [31] |
Wang D, Yu S, Chen W, et al. Enumeration of Vibrio parahaemolyticus in oyster tissues following artificial contamination and depuration[J]. Letters in Applied Microbiology, 2010, 51(1): 104-108. |
| [32] |
Chung H Y, Na E J, Lee K H, et al. Complete genome sequence of Vibrio parahaemolyticus FORC_023 isolated from raw fish storage water[J]. Pathogens and Disease, 2016, 74(4): ftw032. DOI:10.1093/femspd/ftw032 |
 2019, Vol. 43
2019, Vol. 43


