文章信息
- 宋小含, 孙忠民, 胡自民, 段德麟. 2020.
- SONG Xiao-han, SUN Zhong-min, HU Zi-min, DUAN De-lin. 2020.
- 中国近海外来囊藻(Colpomenia peregrina)种群遗传多样性研究
- Population genetic diversity of Colpomenia peregrina along the coast of China
- 海洋科学, 44(1): 89-96
- Marine Sciences, 44(1): 89-96.
- http://dx.doi.org/10.11759/hykx20190501001
-
文章历史
- 收稿日期:2019-05-01
- 修回日期:2019-05-10
2. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 中国科学院海洋研究所 海洋生物分类与系统演化实验室, 山东 青岛 266071
2. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Department of Marine Organism Taxonomy and Phylogeny, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China
外来囊藻(Colpomenia peregrina Sauvageau)隶属于Ochrophyta门, 褐藻纲(Phaeophyceae), 墨角藻亚纲(Fucophycidae), 水云目(Ectocarpales), 萱藻科(Scytosiphonaceae), 囊藻属(Colpomenia), 在温带近海潮间带分布广泛, 主要生长在坡度较缓的岩石上或附生在其他大型海藻上。
外来囊藻在形态上呈棕褐色的中空球形或不规则囊形, 表面随着藻体的生长发育出现深浅不一的褶皱。外来囊藻多为一年生, 生活史中有异型世代交替, 生殖期可产生单室孢子囊和多室孢子囊[1]。
由于外形相似, 长期以来囊藻属物种之间的区分鉴定始终是一个难题[2]。20世纪初, 法国科学家Sauvageau发现法国西海岸的一种囊藻在形态上与囊藻属的模式种囊藻(Colpomenia sinuosa)有所不同, 它比囊藻更厚且表面光滑, 缺少褶皱和塌陷, 芽孢囊群和褐藻鞭毛起始部位也不同。Sauvageau认为这个形态种是囊藻的一个变种(Variety), 并将其命名为C. sinuosa var. peregrina。1937年, Hamel将该变种提升至独立物种水平, 命名为C. peregrine[3]。Clayton通过混合指数分析(Hybrid Index Analysis)和方差分析(ANOVA), 首次提出区分外来囊藻和囊藻的3个主要特征:藻体表面芽孢囊群的形态呈点状不连续或广泛连续; 孢子囊表面是否有角质层覆盖; 髓层细胞层数不同[4]。
外来囊藻呈中空状, 在藻体完整未受损伤时内部会因光合作用而充满氧气。当藻体附生在牡蛎壳上时, 会因气体的浮力而使牡蛎浮在水面并随水漂走, 在20世纪曾给法国牡蛎养殖业带来损失, 外来囊藻也因此被称为“牡蛎小偷” [3, 5]。除漂浮扩散外, 外来囊藻也可随牡蛎养殖的跨海区转运而被引入新的栖息地, 建立新种群[6]。这一特征使外来囊藻成为研究海洋生物多样性变动、群体遗传变异和生物地理格局形成的良好模型。
近年来, 已有研究逐步涉及囊藻属物种的种群分布变动、入侵路径及隐存多样性。Cho等利用核糖体内转录间隔区(internal transcribed spacer, ITS)和叶绿体核酮糖1, 5-二磷酸羧化酶大亚基(the large subunit of ribulose 1, 5-bisphosphate carboxylase/oxygenase, rbcL)研究了全球范围内囊藻的遗传多样性, 发现囊藻的ITS标记中存在独特的串联重复序列, 其重复数可将囊藻分为南半球和北半球两个不同的地理分布区[7]。Lee等对19个国家和地区的囊藻细胞色素氧化酶第Ⅲ亚基(Cytochrome c oxidase subunit 3, cox3)和rbcL序列进行系统发育分析, 发现囊藻种内一个分支在印度洋和大西洋重叠分布, 另一分支仅分布在热带地区, 剩余则集中分布在红海和地中海西部, 这样的模式可能源于历史遗存和现代人类干扰[8]。Lee等最近报道西北太平洋地区可能是外来囊藻的发源地, 其遗传多样性中心可能位于韩国西南海岸, 在距今约2万年前扩散到西南太平洋和东北太平洋地区, 在最近一个世纪受人类活动干扰入侵到北大西洋[6]。
外来囊藻在中国近海潮间带分布广泛, 然而关于其在中国境内种群遗传多样性和分化的研究尚为空白。本研究采集了来自中国近海13个地点的外来囊藻种群样本, 应用线粒体cox3标记研究其种群遗传结构和多样性分布模式。
1 材料与方法 1.1 样品采集2013—2018年间, 我们采集了辽宁、山东、浙江等地13个地点(27.45—39.02°N, 120.96—122.73°E)的外来囊藻样本, 详细信息见表 1。采集时, 每个采样点内样品均间隔10米。样品冲洗干净并阴干处理, 24 h后加入变色硅胶干燥保存。
| 序号 | 简称 | 样本量 | 采集地点 | 经纬度 | 采集时间 | 采集人 |
| 1 | ZZ | 26 | 辽宁省大连市獐子岛 | 39.02°N, 122.73°E | 2014.7.13 | 李晶晶 |
| 2 | HN | 30 | 辽宁省大连市黄泥川 | 38.86°N, 121.45°E | 2015.4.20 | 胡自民 |
| 3 | CD | 17 | 山东省长岛县 | 37.91°N, 120.73°E | 2014.7.10 | 胡自民 |
| 4 | PL | 23 | 山东省蓬莱市 | 37.80°N, 120.75°E | 2014.7.10 | 胡自民 |
| 5 | GL | 30 | 山东省荣成市俚岛镇高绿水产公司 | 37.16°N, 122.48°E | 2015.2.6 | 刘若愚, 孙忠民 |
| 6 | SY | 30 | 山东省青岛市第三海水浴场 | 36.07°N, 120.33°E | 2013.4.26 | 胡自民, 刘若愚 |
| 7 | DL | 22 | 浙江省平阳县南麂列岛大檑岛 | 27.49°N, 121.09°E | 2018.5.15 | 宋小含, 孙忠民 |
| 8 | LC | 30 | 浙江省平阳县南麂列岛龙船礁 | 27.49°N, 121.09°E | 2015.5.16 | 刘若愚, 孙忠民 |
| 9 | HJ | 30 | 浙江省平阳县南麂列岛后麂岛 | 27.47°N, 121.13°E | 2018.5.15 | 宋小含, 孙忠民 |
| 10 | XH | 30 | 浙江省平阳县南麂列岛小虎屿 | 27.47°N, 121.07°E | 2014.4.18 | 刘若愚, 孙忠民 |
| 11 | HK | 32 | 浙江省平阳县南麂列岛火焜岙 | 27.46°N, 121.08°E | 2015.5.18 | 刘若愚, 孙忠民 |
| 12 | DS | 31 | 浙江省平阳县南麂列岛大沙岙 | 27.46°N, 121.06°E | 2018.5.16 | 宋小含, 孙忠民 |
| 13 | SM | 30 | 浙江省平阳县南麂列岛上马鞍 | 27.45°N, 120.96°E | 2015.5.17 | 刘若愚, 孙忠民 |
取0.03 g外来囊藻样品, 在液氮中预冷后研磨成藻粉, 采用植物基因组DNA提取试剂盒DP305(天根, 北京)进行DNA提取, 操作步骤参照说明书。提取的基因组DNA采用1%琼脂糖凝胶电泳和微量型分光光度计Nanodrop 2000检测其浓度和纯度, 选择条带明亮、A260/280为1.8~2.0的基因组DNA溶液编号并置于–20℃保存。
1.3 线粒体cox3序列扩增、目的条带检测与测序采用褐藻通用引物对F49(5′-CATTTAGTNGAY CCWAGYCCTTGGC-3′)和R20(5′-AACAAARTGCC AATACCAKG-3′)[6], 选用2×EasyTaq MasterMix(康为世纪, 北京), 以提取的基因组DNA为模板扩增外来囊藻线粒体cox3片段。PCR反应体系为50 μL, 包括25 μL 2 × EasyTaq MasterMix, 1 μL DNA溶液(浓度20 mg/L), 1 μL正向引物F49(浓度10 μmol/L), 1 μL反向引物R20(浓度10 μmol/L), 22 μL RNase free Water。反应程序如下: 94℃预变性4 min; 94℃变性1 min, 53℃退火30 sec, 72℃延伸1 min, 共进行35个循环; 72℃延伸10 min。
采用1%琼脂糖凝胶电泳检测PCR目的条带, 条带单一明亮的PCR产物纯化后在ABI3730测序平台进行正向测序。
1.4 数据处理采用BioEdit v7.1.4[9]检查cox3序列峰图, 选择峰形良好的序列作为可用数据。以GenBank中外来囊藻cox3序列集(JX027338-75)[6]作为参考序列, 采用MEGA v7.0[10]的MUSCLE模型进行序列比对, 确定序列起点和终点, 人工校正去除错误碱基与空隙。
1.5 系统进化基于贝叶斯信息准则(Bayesian Information Criterion, BIC), 采用JModelTest v2.0[11]测得最佳核苷酸替代模型HKY+I(I=0.79)。以同属物种囊藻(C. sinuosa)和C. claytoniae为外类群, 采用PhyML v3.1[12]进行基于最大似然法(Maximum Likelihood, ML)的系统发育树构建, 分支节点的置信度采用自展检验方法(Bootstrap method)估算, 检验重复1000次。采用MrBayes v3.2[13]进行基于贝叶斯法(Bayesian Inference, BI)的系统进化树构建, 运行4条马尔科夫链, 迭代107次, 每100代保留一次, 前104次用作“burnin”处理, 计算保留树的拓扑学和后验概率值。
1.6 种群遗传结构采用DNAsp v5.10软件[14]计算cox3序列的变异位点(Variable sites)和简约信息位点(Parsimony informative sites), 计算各种群的单倍型数量、单倍型多样性(h)和核苷酸多样性(π)。采用MEGA v7.0的p-distance模型计算所有样本间平均遗传距离(genetic distance)。使用Arlequin 3.5.1.2软件[15]计算群体间两两分化系数(Fst), 1 000次重复抽样检验显著性。使用Network 4.6软件[16]中的Median-joining算法构建外来囊藻cox3序列单倍型网络图, 分析单倍型间遗传结构。
2 结果 2.1 外来囊藻cox3序列与系统进化本研究共获得301条cox3序列(长度为620 bp), 序列中无碱基插入和缺失, 序列中共检测到25个变异位点(Variable sites)和19个简约信息位点(Parsimony informative sites)。Blastn比对显示每条cox3序列与GenBank中发表的外来囊藻序列相似度均超过99%。
ML和BI树(图 1)显示本研究的cox3序列聚为一个大分支, 与外类群C. sinuosa和C. claytoniae明显分开。在中国近海, 来自辽宁大连的2个种群(HN、ZZ)和来自山东烟台、青岛、威海的4个种群(CD、GL、PL、SY)分化为两个分支, 来自浙江南麂列岛的7个种群聚为第三个分支。这表明中国近海外来囊藻种内可能存在3个隐存的遗传世系。
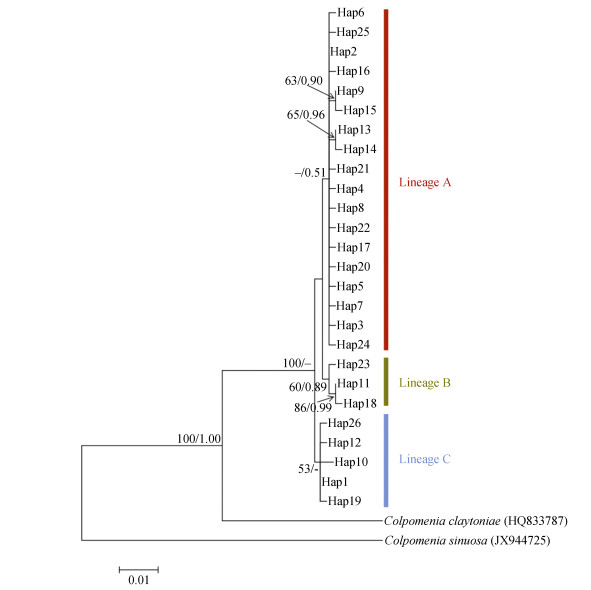 |
| 图 1 基于线粒体cox3序列的系统进化树 Fig. 1 Phylogenetics trees derived from mitochondrial cox3 sequences of C. peregrina 注:分支上的数值为ML自展值(左)/BI后验概率(右), 小于50%和0.5的值被去除 |
在301条cox3序列中共产生26个单倍型, 其中山东荣成高绿(GL)种群和青岛第三海水浴场(SY)种群的遗传多样性较高(h=0.5221~0.6615, π=0.0032~ 0.0050), 浙江南麂列岛的龙船礁(LC)、后麂岛(HJ)、小虎屿(XH)、大沙岙(DS)种群遗传多样性也较高(h=0.6184~0.8016, π=0.0012~0.0039)(表 2)。基于全部序列的单倍型多样性平均值(Hd)为0.775, 核苷酸多样性(π)平均值为0.364×10–2, 遗传距离平均值为0.4%。
| 种群地理位置 | 简称 | 经纬度 | cox3 | |||
| n | Haplotypes | h | π (×10–2) | |||
| 辽宁大连獐子岛 | ZZ | 39.02°N, 122.73°E | 11 | c1(9), c19(1), c26(1) | 0.3455 | 0.0587 |
| 辽宁大连黄泥川 | HN | 38.86°N, 121.45°E | 26 | c1(1), c11(22), c18(2), c19(1) | 0.2862 | 0.1315 |
| 山东长岛 | CD | 37.91°N, 120.73°E | 14 | c1(14) | 0 | 0 |
| 山东蓬莱 | PL | 37.80°N, 120.75°E | 18 | c1(18) | 0 | 0 |
| 山东荣成俚岛高绿水产公司 | GL | 37.16°N, 122.48°E | 26 | c1(3), c10(8), c11(13), c12(2) | 0.6615 | 0.5022 |
| 山东青岛三浴 | SY | 36.07°N, 120.33°E | 17 | c1(11), c11(5), c23(1) | 0.5221 | 0.3250 |
| 浙江南麂列岛大檑岛 | DL | 27.49°N, 121.09°E | 22 | c2(22) | 0 | 0 |
| 浙江南麂列岛龙船礁 | LC | 27.49°N, 121.09°E | 30 | c2(17), c3(1), c4(8), c7(2), c20(2) | 0.6184 | 0.1175 |
| 浙江南麂列岛后麂岛 | HJ | 27.47°N, 121.13°E | 29 | c2(16), c7(1), c9(3), c13(7), c14(1), c15(1) | 0.6453 | 0.1398 |
| 浙江南麂列岛小虎屿 | XH | 27.47°N, 121.07°E | 28 | c2(15), c4(2), c7(8), c16(1), c24(1), c25(1) | 0.6700 | 0.3917 |
| 浙江南麂列岛火焜岙 | HK | 27.46°N, 121.08°E | 22 | c2(17), c16(1), c17(4) | 0.3853 | 0.0649 |
| 浙江南麂列岛大沙岙 | DS | 27.46°N, 121.06°E | 28 | c2(9), c3(1), c4(8), c5(2), c6(1), c7(5), c8(1), c9(1) | 0.8016 | 0.1856 |
| 浙江南麂列岛上马鞍 | SM | 27.45°N, 120.96°E | 30 | c2(27), c21(2), c22(1) | 0.1908 | 0.0315 |
| 注: n, 样本量; Haplotypes, 单倍型; h, 单倍型多样性; π, 核苷酸多样性 | ||||||
基于cox3构建的单倍型网状图(图 2)和地理分布图(图 3)显示26个单倍型明显分化为3个遗传世系, 与进化树分支结构相互印证, 且3个遗传世系间没有共享单倍型。遗传世系A包含18个单倍型, 其中单倍型Hap2为核心单倍型, 它们全部分布于浙江南麂列岛地区; 遗传世系B包含3个单倍型, 其中Hap11为核心单倍型, 它们主要分布在辽宁大连黄泥川及山东荣成、青岛地区; 遗传世系C包含5个单倍型, 其中Hap1为核心单倍型, 它们主要分布于辽宁大连及山东蓬莱、长岛、荣成和青岛。
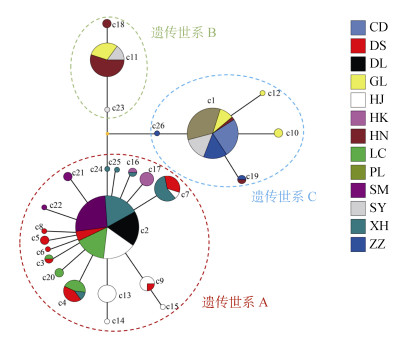 |
| 图 2 基于线粒体cox3序列的单倍型网络图 Fig. 2 Mitochondrial cox3 haplotype network of C. peregrina 注:圆形大小表示单倍型中包含序列的数量, 颜色表示地理位置 |
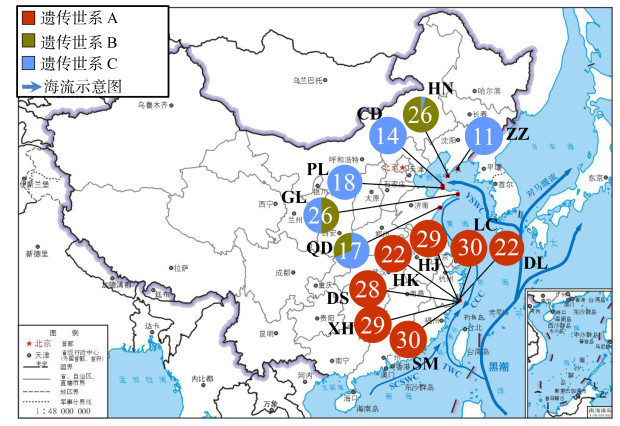 |
| 图 3 基于单倍型的外来囊藻遗传世系地理分布图(底图审图号: GS(2019)1824号) Fig. 3 Geographical distribution of cox3 genetic lineages in C. peregrina 注:圆圈内数字表示种群序列数量, 颜色表示不同遗传世系。 |
基于线粒体cox3序列的Fst值(表 3)显示, 13个外来囊藻种群间存在较大的遗传分化(平均Fst= 0.504), 其中山东、辽宁6个种群与浙江南麂列岛7个种群间存在显著的遗传分化(平均Fst=0.748), 而山东和辽宁6个种群间存在中等程度的遗传分化(平均Fst=0.354), 浙江南麂列岛7个种群间的遗传分化程度较低(平均Fst=0.099)。
| ZZ | HN | CD | PL | GL | SY | DL | LC | HJ | XH | HK | DS | |
| HN | 0.831 | |||||||||||
| CD | 0.023 | 0.863 | ||||||||||
| PL | 0.047 | 0.875 | 0.000 | |||||||||
| GL | 0.313 | 0.300 | 0.346 | 0.374 | ||||||||
| SY | 0.210 | 0.507 | 0.260 | 0.295 | 0.061 | |||||||
| DL | 0.962 | 0.859 | 1.000 | 1.000 | 0.557 | 0.697 | ||||||
| LC | 0.828 | 0.783 | 0.859 | 0.870 | 0.546 | 0.637 | 0.132 | |||||
| HJ | 0.807 | 0.768 | 0.838 | 0.851 | 0.536 | 0.619 | 0.137 | 0.157 | ||||
| XH | 0.616 | 0.627 | 0.648 | 0.670 | 0.446 | 0.477 | 0.056 | 0.045 | 0.088 | |||
| HK | 0.886 | 0.813 | 0.924 | 0.931 | 0.531 | 0.649 | 0.114 | 0.136 | 0.138 | 0.057 | ||
| DS | 0.767 | 0.740 | 0.799 | 0.814 | 0.514 | 0.589 | 0.131 | 0.006 | 0.141 | 0.017 | 0.132 | |
| SM | 0.927 | 0.849 | 0.957 | 0.961 | 0.581 | 0.706 | 0.010 | 0.134 | 0.141 | 0.072 | 0.095 | 0.140 |
| 注:加黑Fst系数表示群体间存在明显遗传分化。 | ||||||||||||
线粒体cox3因其保守型强、进化速率快等特点, 成为囊藻属新种鉴定、种内种间遗传变异及谱系结构探究的常用分子标记[6, 8, 17-18]。本研究利用cox3标记重建了中国近海外来囊藻种内单倍型间的进化关系, 发现其种内存在明显的谱系分化, 13个地理种群共分化出3个遗传世系。种群间两两分化Fst系数显示13个地理种群间的平均值达0.504, 表明外来囊藻种内遗传分化显著, 其中遗传世系A与遗传世系B、C间平均Fst值达0.748, 因此我们推测外来囊藻遗传世系间可能存在过长期的地理隔离。
近海海洋环境变动和气候变化是影响西太平洋海藻多谱系遗传结构的重要原因[19-21], 其中第四纪末期海洋环境变化可能与外来囊藻种群分化有关。作为太平洋西部的边缘海, 黄渤海和东海在第四纪更新世晚期形成, 并在末次盛冰期(Last Glacial Maximum, LGM)期间经历了剧烈的气候和环境变迁。在LGM期间东海的海平面比当前低约150 m, 而黄渤海边缘海则消失成为陆地[22-23]。海洋、陆地面积的大幅度变化和冰期海岸线的快速移动产生了深刻的环境影响, 海岸线的重新配置和边缘海冰期避难所的形成使某些海洋生物多样性得以存留, 随着时间的推移, 种群间产生地理隔离并导致遗传谱系的分化[24-25]。在本研究中, 黄渤海的外来囊藻种群和东海种群间分化显著, 而这种遗传分化同样出现在西北太平洋潮间带其他海藻物种中。譬如, 羊栖菜(Sargassum fusiforme)的线粒体cox1和cox3单倍型在黄海、东海、南海种群的分布显示出显著差异[21-22]; 鼠尾藻(Sargassum thunbergii)的ITS和cox3标记在黄渤海和东海种群中也分化出独特的单倍型[26]。因此, 我们推测外来囊藻的3个遗传世系可能具有共同的祖先, 因历史气候的变化在边缘海的不同避难所中分隔开来, 并逐渐产生种内的遗传分化。
其次, 海流系统也是驱使海藻迁移和空间分布的重要因素[27]。黑潮是当前影响中国海域海洋生物地理分布的主要海流系统, 它对生物多样性和种群结构产生重大影响[22, 28-29]。因此, 黑潮驱动的水体交换可能会促进外来囊藻在不同边缘海之间的扩散, 并影响种群的遗传分化。
外来囊藻在中国主要分布于黄渤海和东海地区, 单倍型分析发现分布于浙江南麂列岛的遗传世系A具有最多的特有单倍型, 表明此处外来囊藻的遗传多样性最高。这可能与南麂列岛独特的地理位置和潮间带环境有关。南麂列岛位于浙江省南部海域外侧, 与陆地最近距离超过30英里。远离陆地的地理位置使得南麂列岛受河流和淡水团的影响较小。因此, 该地区的海水盐度、温度和光照等环境因素比较稳定, 有利于海洋生物多样性的形成[30]。此外, 南麂列岛岛屿众多, 既有陡峭的悬崖, 也有平坦的沙滩, 潮间带范围广泛, 附生的牡蛎等贝类为海洋生物的生长提供了独特的生长环境, 十分有利于物种多样性和遗传多样性的形成[31-33]。最后, 南麂列岛海域受黑潮和江浙沿岸流两大水系的交互影响, 北上的南海暖流和台湾暖流与南下的黄海冷水团和江浙沿岸流在不同的季节影响着南麂列岛的水环境[34]。浙南沿岸上升流将深水区的营养盐带到表层, 为该海域提供了丰富的营养物质和适宜的水文环境, 这对促进海藻等海洋生物生长发育起到重要作用[35]。
Lee等分析了12个国家的外来囊藻cox3序列, 计算得种内平均遗传分歧为3.1%, 囊藻属平均遗传分歧为11.2%[6]。本研究中外来囊藻cox3序列数据与Lee等的报道具有可比性, 计算得序列间平均遗传分歧为0.4%, 表明我国近海外来囊藻的遗传变异程度在世界范围内处于较低水平。同时, Lee等认为外来囊藻可能是最具入侵性的褐藻之一, 发源于西北太平洋地区, 其遗传多样性中心位于韩国西南海岸, 且根据其遗传分歧大小推断外来囊藻在更新世前就已发生进化, 最早可能分化于晚中新世托尔顿阶(7.2-11.6 Ma) [6]。而中国近海外来囊藻是否自韩国引入, 何时开始进化, 又存在着怎样的种群时空变动格局, 这些问题都有待后续分析和解答。
本研究利用线粒体cox3序列分析了中国近海外来囊藻种群多样性和遗传分化, 对重新认知中国近海囊藻属物种分布和多样性提供了重要参考, 但外来囊藻在中国近海的具体分布界限仍不明确, 种群时空变动格局也尚不清楚, 这需要在后续研究中进行广泛的连续采样和深入的数据分析。
4 结论本研究通过扩增外来囊藻线粒体cox3序列、分析种群系统进化关系及遗传结构发现, 中国近海外来囊藻种内存在明显的谱系分化, 13个地理种群分化为3个遗传世系, 可能因历史上黄渤海和东海边缘海环境变化、黑潮等海流影响而导致; 南麂列岛种群的遗传多样性高, 这可能与南麂列岛独特的地理位置、复杂的潮间带环境以及黑潮和江浙沿岸流的影响有关。
| [1] |
Kogame K, Yamagishi Y. The life history and phenology of Colpomenia peregrina (Scytosiphonales, Phaeophyceae) from Japan[J]. Phycologia, 1997, 36(5): 337-344. DOI:10.2216/i0031-8884-36-5-337.1 |
| [2] |
Parsons M J. Colpomenia (Endlicher) Derbès et Solier (Phaeophyta) in New Zealand[J]. New Zealand Journal of Botany, 1982, 20(3): 289-301. DOI:10.1080/0028825X.1982.10428496 |
| [3] |
Vandermeulen H. The taxonomy and autecology of Colpomenia peregrina (Sauv.) Hamel (Phaeophyceae)[D]. The University of British Columbia, 1984.
|
| [4] |
Clayton M N. A study of variation in Australian species of Colpomenia (Phaeophyta, Scytosiphonales)[J]. Phycologia, 1975, 14(4): 187-195. DOI:10.2216/i0031-8884-14-4-187.1 |
| [5] |
Blackler H. The occurrence of Colpomenia peregrina (Sauv.) Hamel in the Mediterranean (Phaeophyta, Scytosiphonales)[J]. Blumea, 1967, 15: 5-8. |
| [6] |
Lee K M, Boo G H, Coyer J A, et al. Distribution patterns and introduction pathways of the cosmopolitan brown alga Colpomenia peregrina, using mt cox3 and atp6 sequences[J]. Journal of Applied Phycology, 2014, 26(1): 491-504. DOI:10.1007/s10811-013-0052-1 |
| [7] |
Cho G Y, Choi D W, Kim M S, et al. Sequence repeats enlarge the internal transcribed spacer 1 region of the brown alga Colpomenia sinuosa (Scytosiphonaceae, Phaeophyceae)[J]. Phycological Research, 2009, 57(3): 242-250. DOI:10.1111/j.1440-1835.2009.00543.x |
| [8] |
Lee K M, Boo S M, Kain J M, et al. Cryptic diversity and biogeography of the widespread brown alga Colpomenia sinuosa (Ectocarpales, Phaeophyceae)[J]. Botanica Marina, 2013, 56(1): 15-25. |
| [9] |
Hall T. BioEdit:a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41: 95-98. |
| [10] |
Kumar S, Stecher G, Tamura K. MEGA7:Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33: 1870-1874. DOI:10.1093/molbev/msw054 |
| [11] |
Darriba D, Taboada G L, Ramón D, et al. Jmodeltest 2:more models, new heuristics and parallel computing[J]. Nature Methods, 2012, 9(8): 772. |
| [12] |
Guindon S, Gascuel O. A simple, fast and accurate algorithm to estimate large phylogenies by maximum likelihood[J]. Systematic Biology, 2003, 52(5): 696-704. DOI:10.1080/10635150390235520 |
| [13] |
Ronquist F, Teslenko M, Mark P V D. MrBayes 3.2:Efficient Bayesian Phylogenetic Inference and Model Choice Across a Large Model Space[J]. Systematic Biology, 2012, 61(3): 539-542. DOI:10.1093/sysbio/sys029 |
| [14] |
Librado P, Rozas J. DnaSP v5:A software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25: 1451-1452. DOI:10.1093/bioinformatics/btp187 |
| [15] |
Laval E L G, Schneider S. Arlequin ver. 3.0:An integrated software package for population genetics data analysis[J]. Evolutionary Bioinformatics Online, 2005, 1: 47-50. |
| [16] |
Bandelt H-J, Forster P, Röhl A. Median-joining networks for inferring intraspecific phylogenies[J]. Molecular Biology and Evolution, 1999, 16: 37-48. DOI:10.1093/oxfordjournals.molbev.a026036 |
| [17] |
Boo S M, Lee K M, Cho G Y, et al. Colpomenia claytonii sp. nov. (Scytosiphonaceae, Phaeophyceae) based on morphology and mitochondrial cox3 sequences[J]. Botanica Marina, 2011, 54: 159-167. |
| [18] |
Green L A, Mathieson A C, Neefus C D, et al. Southern expansion of the brown alga Colpomenia peregrina Sauvageau (Scytosiphonales) in the Northwest Atlantic Ocean[J]. Botanica Marina, 2012, 55(6): 643-647. |
| [19] |
刘若愚, 孙忠民, 姚建亭, 等. 中国近海重要生态建群红藻真江蓠的群体遗传多样性[J]. 生物多样性, 2016, 4(7): 781-790. Liu Ruoyu, Sun Zhongmin, Yao Jianting, et al. Genetic diversity of the habitat-forming red alga Gracilaria vermiculophylla along Chinese coasts[J]. Biodiversity Science, 2016, 4(7): 781-790. |
| [20] |
Hu Z M, Li J J, Sun Z M, et al. Phylogeographic structure and deep lineage diversification of the red alga Chondrus ocellatus Holmes in the Northwest Pacific[J]. Molecular Ecology, 2015, 24(19): 5020-5033. DOI:10.1111/mec.13367 |
| [21] |
Hu Z M, Li J J, Sun Z M, et al. Hidden diversity and phylogeographic history provide conservation insights for the edible seaweed Sargassum fusiforme in the Northwest Pacific[J]. Evolutionary Applications, 2017, 10(4): 366-378. DOI:10.1111/eva.12455 |
| [22] |
刘敬圃. 末次盛冰期时西太平洋边缘海区古海洋学特征[J]. 海洋科学, 1995, 19(3): 14-16. Liu Jingpu. Paleoceanographic characteristics during the LGM period in the west Pacific marginal seas[J]. Marine Sciences, 1995, 19(3): 14-16. |
| [23] |
Wang P X. Response of western Pacific marginal seas to glacial cycles:paleoceanographic and sedimentological features[J]. Marine Geology, 1999, 156: 5-39. DOI:10.1016/S0025-3227(98)00172-8 |
| [24] |
Cheang C C, Chu K H, Put A O. Phylogeography of the marine macroalga Sargassum hemiphyllum (Phaeophyceae, Heterokontophyta) in the northwestern Pacific[J]. Molecular Ecology, 2010, 19: 2933-2948. DOI:10.1111/j.1365-294X.2010.04685.x |
| [25] |
Hu Z M, Zhang J, Lopez-Bautista J, et al. Asymmetric genetic exchange in the brown seaweed Sargassum fusiforme (Phaeophyceae) driven by oceanic currents[J]. Marine Biology, 2013, 160(6): 1407-1414. DOI:10.1007/s00227-013-2192-x |
| [26] |
Li J J, Hu Z M, Sun Z M, et al. Historical isolation and contemporary gene flow drive population diversity of the brown alga Sargassum thunbergii along the coast of China[J]. BMC Evolutionary Biology, 2017, 17(1): 246. DOI:10.1186/s12862-017-1089-6 |
| [27] |
Tseng C K, Chang J F. On the regional division of the marine algal flora of the western north Pacific[J]. Oceanologia et Limnologia Sinica, 1959, 2(4): 244-265. |
| [28] |
Barkley R A. The Kuroshio current[J]. Science Journal, 1970, 6: 54-60. |
| [29] |
Lin S M, Tseng L C, Ang P O, et al. Long-term study on seasonal changes in floristic composition and structure of marine macroalgal communities along the coast of Northern Taiwan, southern East China Sea[J]. Marine Biology, 2018, 165(5): 83. DOI:10.1007/s00227-018-3344-9 |
| [30] |
蔡厚才, 彭欣. 走进贝藻王国[M]. 上海: 上海人民美术出版社, 2011. Cai Houcai, Peng Xin. Welcome to the Kingdom of Shellfish and Algae[M]. Shanghai: Shanghai People's Fine Arts Publishing House, 2011. |
| [31] |
徐芝敏, 蒋加伦, 孙建璋. 南麂列岛潮间带海藻资源与生态[J]. 东海海洋, 1994, 12(2): 29-43. Xu Zhimin, Jiang Jialun, Sun Jianzhang. Study on ecology and resources of marine algae in the intertidal zone, Nanji Archipelago[J]. Donghai Marine Science, 1994, 12(2): 29-43. |
| [32] |
朱根海. 南麂列岛自然保护区药用海藻资源及其应用[J]. 东海海洋, 1998(2): 63-68. Zhu Genhai. Study on the resources of marine pharmaceutical algae and its applications in Nanji Islands Marine Nature Conservation Area[J]. Donghai Marine Science, 1998(2): 63-68. |
| [33] |
姚瑞.基于高通量DNA测序的南麂列岛海洋底栖环节动物多样性分析[D].浙江舟山: 浙江海洋大学, 2018. Yao Rui. Analysis of marine benthic annelid diversity in Nanji Islands based on high-throughput DNA sequencing[D]. Zhoushan, Zhejiang Province: Zhejiang Ocean University, 2018. |
| [34] |
孙建璋, 陈万东, 庄定根, 等. 中国南麂列岛铜藻Sargassum horneri实地生态学的初步研究[J]. 南方水产, 2008(3): 58-63. Sun Jianzhang, Chen Wandong, Zhuang Dinggen, et al. In situ ecological studies of the subtidal brown alga Sargasssum horneri at Nanji Island of China[J]. South China Fisheries Science, 2008(3): 58-63. |
| [35] |
谢旭, 俞存根, 蔡厚才, 等. 南麂列岛浅海区鱼种组成、分布与环境因子的关系[J]. 广东海洋大学学报, 2017, 37(4): 46-54. Xie Xu, Yu Cungen, Cai Houcai, et al. The Composition and distribution of fish assemblage in the shallow waters around NANJI Islands and its relationship with environmental factors[J]. Journal of Guangdong Ocean University, 2017, 37(4): 46-54. |
 2020, Vol. 44
2020, Vol. 44


