文章信息
- 张一超, 吴韦韦, 李奥, 刘宝华, 庞秋香. 2020.
- ZHANG Yi-chao, WU Wei-wei, LI Ao, LIU Bao-hua, PANG Qiu-xiang. 2020.
- 蛋白质精氨酸甲基转移酶1调控DNA损伤修复和细胞凋亡
- Arginine methyltransferase 1 regulates DNA damage repair and apoptosis
- 海洋科学, 44(3): 146-152
- Marine Sciences, 44(3): 146-152.
- http://dx.doi.org/10.11759/hykx20191223004
-
文章历史
- 收稿日期:2019-12-23
- 修回日期:2019-12-30
2. 深圳大学 基础医学院, 广东 深圳 518060
2. School of Basic Medicine, Shenzhen University, Shenzhen 518060, China
蛋白质是执行细胞功能的基本功能单元, 其表达和功能的发挥受到多种因素的影响, 例如基因组、表观遗传修饰和翻译后修饰等。至今已知, 在真核生物中存在300多种蛋白质翻译后修饰(post-translational modifications, PTMs), PTMs通过共价或者非共价的方式修饰特定的氨基酸[1]。
蛋白质的精氨酸甲基化是细胞质和细胞核中普遍存在的翻译后修饰。1967年, Paik[2]等使用14C-S-腺苷甲硫氨酸(SAM or AdoMet)标签实验, 从小牛胸腺的核心组蛋白中成功证实精氨酸甲基化。随着科学技术的进步, 目前已知总精氨酸残基中有0.7%~ 1%被甲基化[3], 催化这种反应的酶称为蛋白质精氨酸甲基转移酶(protein arginine methyltransferases, PRMTs)。
真核生物中有3种精氨酸甲基化类型:单甲基化精氨酸(monomethylated arginine, MMA), 不对称性二甲基化精氨酸(asymmetric dimethylated arginine, aDMA)和对称性二甲基化精氨酸(symmetric dimethylated arginine, sDMA)。根据底物分子精氨酸甲基化类型, PRMTs被分为4类(Ⅰ, Ⅱ, Ⅲ和Ⅳ)。Ⅰ型PRMTs (PRMT1, 3, 4, 6, 8)催化形成aDMA; Ⅱ型PRMTs (5, 7, 9)催化形成sDMA; 其中PRMT7还表现出Ⅲ型PRMT的酶活, 催化形成MMA; Ⅳ型PRMTs在酵母中被发现[4]。PRMT2的酶活至今还未被发现。精氨酸二甲基化是发挥功能的主要形式, 对称性二甲基化和不对称性二甲基化是两种不同的状态, 这意味着不同的生物学意义和识别机制。
PRMT1是第一个被克隆出来的蛋白质精氨酸甲基转移酶, 属于Ⅰ型PRMTs, 是PRMTs家族主要成员, 主要定位在细胞核, 调节组蛋白等的甲基化, 在细胞质中也有表达并调节多种蛋白底物的甲基化[5, 6], 参与多种细胞过程。同时, 有许多疾病由错误的精氨酸甲基化造成的。
1 PRMT1结构特点与底物PRMT1位于人染色体19q13.3, 跨越11.2 kb基因组长度。人类PRMT1包含12个外显子和11个内含子[7], 导致许多可变的PRMT1亚型的形成。前体mRNA的选择性剪接可生成7种N-末端不同的主要亚型, 分子大小约40 kDa, 细胞内广泛分布表达, 有十分广谱的底物特异性[8]。近些年, 有研究发现一种新的PRMT1亚型, 称为PRMT1Δarm, 缺失形成二聚化手臂的外显子8和9, 从而导致其酶活性的缺失。PRMT1Δarm能作为PRMT1竞争性抑制剂, 调控肿瘤细胞的生物学活性[9]。PRMT1是一类SAM依赖型甲基转移酶, 其SAM结合口袋处有一组十分保守的基序(motif Ⅰ, post-Ⅰ, motif Ⅱ, motif Ⅲ)和一个典型的Thr-His-Trp (THW) loop[10, 11], 在整个生物演化过程中都十分保守。
精氨酸是碱性最强的氨基酸, 因此其带正电荷, 能形成氢键。PRMT1作用于底物分子先形成单甲基化的状态, 随后该酶继续催化底物形成二甲基化。甲基化修饰不会改变精氨酸的带电性, 但是可以通过减少氢键的形成, 使其空间位阻和疏水性加强以及改变蛋白的三级结构, 从而影响其与底物相互作用[12]。
PRMT1拥有十分丰富的底物分子, 包括组蛋白和非组蛋白(图 1)。PRMT1主要参与修饰组蛋白H4第3位精氨酸(H4R3), H4R3的不对称性二甲基化会抑制基因的表达, 而当该位点修饰转变为对称性二甲基化时, 则表现出完全相反的功能[13, 14]。
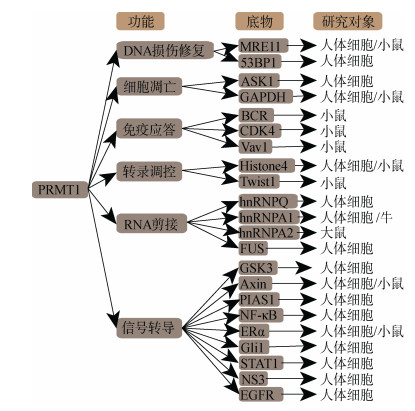 |
| 图 1 PRMT1的底物 Fig. 1 The substrates of PRMT1 |
PRMT1的底物还包括众多非组蛋白。B细胞抗原受体(B cell antigen receptor, BCR)胞内段免疫球蛋白的α链第198位保守的精氨酸对B细胞分化起决定性作用。PRMT1识别并甲基化BCR, 甲基化的BCR激活脂质激酶PI(3)K, 调节细胞因子IL-7, 促进B细胞的分化。当Lys代替同位点的Arg时, 会抑制细胞因子IL-7表达, 从而抑制B细胞的分化[15]。PRMT1能结合Ⅰ型干扰素受体胞内部分, 通过甲基化与去甲基化作用调节NGF(nerve growth factor)受体信号[16]。PRMT1又对Wnt信号通路起关键调控。普遍的甲基供体甲硫氨酸是细胞内吞作用和Wnt信号通路必须分子[17]。在Wnt3α触发的Wnt信号转导过程中, 伴随细胞大胞饮会形成一系列多囊泡体, 这个过程被称为微自噬[18]。微自噬转移甲基化的GSK3进入多囊体内, 这过程又是受典型的Wnt信号通路调控[19]。Axin是β-catenin破坏复合物的关键性结构蛋白, 其378位精氨酸的甲基化能加强其与GSK3β的结合, 封闭GSK3β泛素化, 从而起抑制降解的作用, 最终导致Wnt/β-catenin信号的调节受抑制[20]。
2 PRMT1对DNA损伤修复DNA是遗传的物质基础, 基因是具有特定生物功能的DNA序列, 通过基因的表达, 能够使上一代的性状准确地在下一代表现出来。由此可见, 维持DNA的稳定尤为重要。但是, 在整个生物体的生命周期中, DNA一直遭受外源性损伤和内源性损伤的影响, 比如ionizing radiation(IR), ultraviolet radiation (UV), 化学试剂, DNA的脱嘌呤作用, 生物体内的氧化应激反应等, 从而造成DNA的损伤[21]。DNA双链的断裂(DNA double-strand breaks, DSBs)会对真核生物引起严重的后果, 包括细胞自噬, 衰老, 甚至引发癌症[22]。
基因组的稳定性依赖于DNA复制过程中对自然产生的DNA损伤进行精确修复, 也依赖于对外源性DNA损伤的治疗。为了应对不同类型的DNA损伤, 生物体在漫长的进化过程中形成多种对应的修复机制。错配修复(mismatch repair, MMR)将错误的碱基替换回正确的[23], DNA碱基的化学改变通过损伤碱基的切除修复(base excision repair, BER)[24]。例如嘧啶二聚体、链内交联等复杂的损伤就需要核苷酸切除修复(nucleotide excision repair, NER), 通过切除一段包含受损碱基约30 bp的寡核苷酸片段来修复[25]。还有DNA单链断裂修复(single-strand break repair, SSBR)[26], 人类细胞中的DSBs可以通过末端的非同源连接(NHEJ)或者同源重组(HR)[26]。NHEJ对DSBs的修复并不准确, HR则是以姐妹染色体为模板, 能精确修复DSBs。PRMT1介导的DNA损伤修复通过甲基化DSBs修复相关的蛋白MRE11, 53BP1等修复损伤DNA(图 2)。
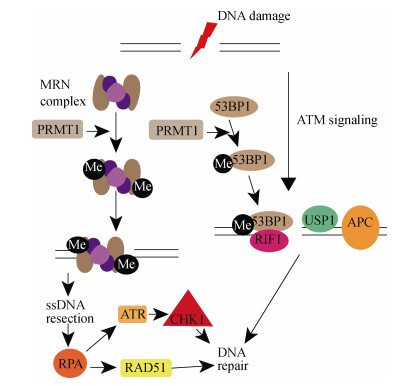 |
| 图 2 PRMT1调控DNA损伤修复 Fig. 2 PRMT1 regulates DNA damage repair |
MRE11在DSBs修复过程中起关键作用[27]。它与Rad50, Nbs1形成的复合物MRN在真核生物中十分的保守。MRN复合物作为一个传感器识别DSBs[28], 随后通过同源重组修复DSBs。研究发现, MRE11含有广泛的转录后修饰, 包括磷酸化、乙酰化、甲基化等, 这些修饰对蛋白功能起至关重要的作用[29]。有研究表明, 在HeLa细胞中MRE11的C末端的Glycine-Arginine-Rich motif (GAR)被PRMT1甲基化[30, 31]。在正常细胞中, PRMT1通过调节MRE11甲基化, 首先调控MRE11对DSBs的定位和结合能力, 随后还影响MRE11对损伤DNA切除的核酸酶活性。而在增殖细胞中, PRMT1对MRE11甲基化又是G2/M检验点激活所必需的。
电离辐射造成严重的DSBs, 感应器MRN迅速形成并转移至细胞核DNA损伤区域, 进而引起一系列DNA损伤修复应答。MRE11的C末端GAR结构域的甲基化在该过程中起到决定性的作用。细胞实验中发现, 使用甲基转移酶抑制剂会使MRE11定位到DSBs的量减少; 随后把GAR结构域中的精氨酸突变成甘氨酸, MRE11定位到DSBs的量同样减少; 缺失GAR结构域直接导致MRE11不能定位到DSBs区域[29]。体外实验证明, GAR结构域甲基化调控MRE11与DNA结合, 缺失GAR结构域MRE11完全不能结合DNA, 非甲基化的GAR结构域与甲基化的GAR结构域相比较对DNA结合能力远远减弱[29]。表示MRE11的GAR结构域甲基化首先通过影响MRE11的定位和与DNA的结合能力来影响其发挥DNA损伤修复功能。MRE11具有核酸酶的功能, 切除损伤处的DNA片段, 进而形成RAP-ssDNA, 招募ATR-ATRIP, 激活CHK1, 修复DNA损伤[29, 32]。突变GAR结构域中的精氨酸, 其核酸酶功能严重受损。已有报道显示, MRE11能调控ATM通路参与DNA损伤修复。然而这个过程并不受MRE11的GAR结构域的甲基化调控, 将GAR结构域中的精氨酸突变成赖氨酸使之不能被甲基化, 检测ATM及其下游底物CHK2的表达量和磷酸化水平均没有明显变化[32]。
当细胞遭受DNA损伤刺激, 会激活细胞周期检验点, 阻止细胞进程, 给予细胞修复DNA损伤的时间[33]。实验取正常的小鼠胚胎成纤维细胞(mouse embryonic fibroblasts, MEFs)和MRE11的GAR结构域中精氨酸突变成赖氨酸的突变型MEFs, 同时使用10Gy强度的γ-irradiation(IR)处理, 90min后使用组蛋白H3pS10抗体测量大量进入有丝分裂的细胞。统计发现, 只有大约10%正常的MEFs经过G2/M检验点, 而突变的MEFs中大约有30%的细胞经过G2/M检验点。结果表明, MRE11的GAR结构域的非甲基化会导致G2/M检验点的缺失[32]。有趣的是, 已经有报道MRE11敲除的果蝇中, 经过低剂量的电离辐射处理会诱导G2/M检验点的缺失。但是, 近期有科学家解剖低剂量的电离辐射处理MRE11敲除的果蝇中发现, 其wing disc和eye disc细胞中G2/M检验点并未受到影响。
2.2 PRMT1调控53BP1介导的DNA损伤修复53BP1是一个大蛋白, N端含有28SQ/TQ位点, 中间串联两个Tudor结构域和一个泛素依赖型招募结构域, C端是BRCA1羧基端重复。如其名, 53BP1首次发现是作为肿瘤抑制蛋白P53的结合蛋白[34]。经过数十年研究, 发现其在DSBs修复过程中起十分重要的作用。在应答DNA损伤时, 53BP1被ATM磷酸化, 对依赖ATM通路的NHEJ进行DNA损伤位点的预处理[35]。PRMT1通过调控53BP1对DSBs识别和结合, 从而影响53BP1修复DSBs。
体内实验发现, 53BP1能被PRMT1甲基化, 突变着丝粒结合结构域中GAR基序的精氨酸, 突变蛋白检测甲基化明显减弱[36]。已有报道该区域与dsDNA或者ssDNA的结合有关[34]。检测突变蛋白的DNA结合能力发现其对dsDNA和ssDNA的吸引力都很弱, 因此甲基化GAR基序还调控53BP1的DNA结合能力[36]。使用甲基转移酶抑制剂过夜处理细胞, 随后进行DNA损伤诱导一小时, 结果显示53BP1和γ-H2AX在DNA损伤处聚集减少。表明53BP1甲基化影响其定位至DNA损伤处。但是, 有研究表明组蛋白H3的79位赖氨酸甲基化对募集53BP1至DNA损伤处是必须的, 而该区域与Tudor结构域直接相互作用[37]。实验通过分别缺失GAR结构域和Tudor结构域, 发现Tudor结构域和GAR结构域在DNA损伤定位过程中都是必须的, 缺失任何一个结构域都不能使其成功定位到DNA损伤区域[36]。
3 PRMT1对细胞凋亡的调控细胞凋亡又称细胞程序性死亡, 是由生理刺激引起的细胞死亡, 对于控制组织、器官内稳态至关重要。其通过不引起炎症反应, 消除有缺陷的细胞, 维持机体的稳定。细胞凋亡受机体一系列严密调控, 从而维持机体各个组织器官之间的稳定。当细胞凋亡调控异常, 会引发多种疾病, 例如癌症, 神经衰弱等。PRMT1在氧化应激条件下对细胞凋亡进行调控(图 3)。
 |
| 图 3 PRMT1调节细胞凋亡 Fig. 3 PRMT1 regulates apoptosis |
凋亡信号调控激酶1(ASK1), 是MAP3K家族中的一员, 能选择性激活JNK和p38 MAP激酶信号通路, 调控各种类型的细胞应激[38]。在未激活的状态下, ASK1通过其末端的氨基区域与硫氧还蛋白结合, 随后又通过羧基端结构域形成同源二聚体。当遇到各种细胞压力刺激, 硫氧还蛋白从ASK1上脱离, 募集TRAF2或者TRAF6到ASK1, 形成ASK1持续激活的复合体, 诱导细胞凋亡。PRMT1能介导ASK1的78和80位精氨酸甲基化。甲基化的ASK1在压力刺激下不能释放硫氧还蛋白, 因此无法结合TRAF2或者TRAF6, 导致ASK1不能被激活, 从而致使细胞凋亡异常[39]。Tripartite motif 48 (TRIM48)是一种E3泛素连接酶, 能调控ASK1激活。TRIM48在氧化应激产生的时候, 使PRMT1被泛素化并被降解。促使ASK1甲基化减弱, ASK1和硫氧还蛋白的相互作用减弱, 激活细胞凋亡, 抑制肿瘤发育[40]。
甘油醛-3-磷酸脱氢酶(Glyceraldehyde-3-phosphate dehydrogenase, GAPDH)除了参与糖酵解作用, 还参与多种细胞功能, 例如基因表达调控、维持端粒结构、DNA损伤修复、膜泡运输、细胞凋亡等[41]。巨噬细胞在LPS/IFNγ刺激下, 会持续表达促炎症因子并通过一氧化氮合酶合成NO, 而促炎症因子的持续分泌和过量的NO可能导致组织损伤和败血症休克。活化的巨噬细胞可以通过诱导细胞凋亡来消除这种影响[42]。LPS/IFNγ刺激下, 一氧化氮合酶合成NO, NO能使GAPDH S-亚硝酸化, S-亚硝酸化的GAPDH与SIAH1形成复合物并转移到细胞核, 促使部分核蛋白S-亚硝酸化, 进而引起细胞凋亡。但是在NO产生的过程中, 会促进PRMT1的表达, PRMT1介导GAPDH甲基化, 从而抑制GAPDH的S-亚硝酸化, 导致GAPDH不能与SIAH1结合, 抑制巨噬细胞凋亡[43]。
糖尿病性肾病是慢性肾病的主要病因之一, 有30%~40%糖尿病患者患有肾病, 是糖尿病最常见的并发症[44]。脂毒性诱导的肾小球系膜细胞凋亡与糖尿病性肾病恶化有关。在糖尿病患者的肾小球上皮细胞中, PRMT1的表达量增加。使用棕榈酸酯处理肾小球系膜细胞, 模拟细胞脂毒性刺激, 通过PERK和ATF6介导内质网应激, 诱导肾小球系膜细胞凋亡[45]。棕榈酸酯处理后, 肾小球系膜细胞中PRMT1表达量上升。敲除PRMT1导致棕榈酸酯介导的细胞凋亡减弱。小鼠实验中发现, PRMT1杂合型的小鼠在高脂饮食诱导下, 肾小球细胞凋亡情况明显减弱[45]。这也为我们提供了一种策略, 可以通过调控PRMT1的表达来抑制糖尿病患者肾病恶化。
4 展望在过去二十几年中, 精氨酸甲基化的重要性越来越被关注。借助不同的高通量筛选尤其是基于质谱的蛋白组学技术, 成功鉴定出成千上万的精氨酸甲基化蛋白, 包括组蛋白和非组蛋白。蛋白质精氨酸甲基化异常调控会造成许多疾病, 包括心脑血管疾病、肝炎、癌症的发生等。因此, 我们可以通过以下两个方面来进行进一步研究。首先, 随着PRMT1晶体结构解析的深入及计算机模拟药物分子与蛋白质对接方法的成熟, 直接作用于PRMT1的抑制剂和激动剂的筛选, 将成为治疗多种疾病良好的药物选择。其次, 我们仍需探索PRMT1调控疾病发生的机制及其因不同剪切产生亚型而改变的结构功能。PRMT1的mRNA能通过自身不同剪切形成PRMT1Δarm, 能与自身产生竞争性抑制, 调控底物甲基化。去甲基化酶家族, 如Jumonji C domain-containing protein 6 (JMJD6), 是机体平衡自身蛋白质甲基化的重要途径之一, 通过调节蛋白质甲基化水平从而引导蛋白正确的功能发挥。研究PRMT1间接调控, 这将有助于更好地展现精氨酸甲基化在细胞和疾病中的多重作用, 并有助于合成新的、更有效的药物用于癌症和疾病的治疗。
| [1] |
Walsh C T, Garneau-Tsodikova S, Gatto G J. Protein posttranslational modifications:the chemistry of proteome diversifications[J]. Angewandte Chemie International Ed in English, 2005, 44(45): 7342-7372. DOI:10.1002/anie.200501023 |
| [2] |
Paik W K, Kim S. Enzymatic methylation of protein fractions from calf thymus nuclei[J]. Biochemical and Biophysical Research Communications, 1967, 29(1): 14-20. DOI:10.1016/0006-291X(67)90533-5 |
| [3] |
Uhlmann T, Geoghegan V L, Thomas B, et al. A method for large-scale identification of protein arginine methyllation[J]. Molecular & Cellular Proteomics, 2012, 11(11): 1489-1499. |
| [4] |
Bedford M T. Arginine methylation at a glance[J]. Journal of Cell Science, 2007, 120(Pt 24): 4243-4246. |
| [5] |
Pawlak M R, Scherer C A, Chen J, et al. Arginine N-methyltransferase 1 is required for early postimplantation mouse development, but cells deficient in the enzyme are viable[J]. Molecular and Cellular Biology, 2000, 20(13): 4859-4869. DOI:10.1128/MCB.20.13.4859-4869.2000 |
| [6] |
Tang J, Frankel A, Cook R J, et al. PRMT1 is the predominant type Ⅰ protein arginine methyltransferase in mammalian cells[J]. Journal of Biological Chemistry, 2000, 275(11): 7723-7730. DOI:10.1074/jbc.275.11.7723 |
| [7] |
Scorilas A, Black M H, Talieri M, et al. Genomic organization, physical mapping, and expression analysis of the human protein arginine methyltransferase 1 gene[J]. Biochemical and Biophysical Research Communications, 2000, 278(2): 349-359. DOI:10.1006/bbrc.2000.3807 |
| [8] |
Goulet I, Gauvin G, Boisvenue S, et al. Alternative splicing yields protein arginine methyltransferase 1 isoforms with distinct activity, substrate specificity, and subcellular localization[J]. Journal of Biological Chemistry, 2007, 282(45): 33009-33021. DOI:10.1074/jbc.M704349200 |
| [9] |
Patounas O, Papacharalampous I, Eckerich C, et al. A novel splicing isoform of protein arginine methyltransferase 1(PRMT1) that lacks the dimerization arm and correlates with cellular malignancy[J]. Journal of Cellular Biochemistry, 2018, 119(2): 2110-2123. DOI:10.1002/jcb.26373 |
| [10] |
Lin W J, Gary J D, Yang M C, et al. The mammalian immediate-early TIS21 protein and the leukemia-associated BTG1 protein interact with a protein-arginine N-methyltransferase[J]. Journal of Biological Chemistry, 1996, 271(25): 15034-15044. DOI:10.1074/jbc.271.25.15034 |
| [11] |
Peng C, Wong C C. The story of protein arginine methylation:characterization, regulation, and function[J]. Expert Review of Proteomics, 2017, 14(2): 157-170. DOI:10.1080/14789450.2017.1275573 |
| [12] |
Sun L, Wang M, Lv Z, et al. Structural insights into protein arginine symmetric dimethylation by PRMT5[J]. Proc Natl Acad Sci U S A, 2011, 108(51): 20538-20543. DOI:10.1073/pnas.1106946108 |
| [13] |
Huang S, Litt M, Felsenfeld G. Methylation of histone H4 by arginine methyltransferase PRMT1 is essential in vivo for many subsequent histone modifications[J]. Genes & Development, 2005, 19(16): 1885-1893. |
| [14] |
Raman B, Guarnaccia C, Nadassy K, et al. N(omega)-arginine dimethylation modulates the interaction between a Gly/Arg-rich peptide from human nucleolin and nucleic acids[J]. Nucleic Acids Res, 2001, 29(16): 3377-3384. DOI:10.1093/nar/29.16.3377 |
| [15] |
Infantino S, Benz B, Waldmann T, et al. Arginine methylation of the B cell antigen receptor promotes differentiation[J]. The Journal of Experimental Medicine, 2010, 207(4): 711-719. DOI:10.1084/jem.20091303 |
| [16] |
Bedford M T, Richard S. Arginine methylation an emerging regulator of protein function[J]. Molecular Cell, 2005, 18(3): 263-272. DOI:10.1016/j.molcel.2005.04.003 |
| [17] |
Albrecht L V, Bui M H, De Robertis E M. Canonical Wnt is inhibited by targeting one-carbon metabolism through methotrexate or methionine deprivation[J]. Proc Natl Acad Sci USA, 2019, 116(8): 2987-2995. DOI:10.1073/pnas.1820161116 |
| [18] |
Tejeda-Munoz N, Albrecht L V, Bui M H, et al. Wnt canonical pathway activates macropinocytosis and lysosomal degradation of extracellular proteins[J]. Proc Natl Acad Sci USA, 2019, 116(21): 10402-10411. DOI:10.1073/pnas.1903506116 |
| [19] |
Albrecht L V, Ploper D, Tejeda-Munoz N, et al. Arginine methylation is required for canonical Wnt signaling and endolysosomal trafficking[J]. Proc Natl Acad Sci USA, 2018, 115(23): 5317-5325. DOI:10.1073/pnas.1804091115 |
| [20] |
Cha B, Kim W, Kim Y K, et al. Methylation by protein arginine methyltransferase 1 increases stability of Axin, a negative regulator of Wnt signaling[J]. Oncogene, 2011, 30(20): 2379-2389. DOI:10.1038/onc.2010.610 |
| [21] |
Ciccia A, Elledge S J. The DNA damage response:making it safe to play with knives[J]. Molecular Cell, 2010, 40(2): 179-204. DOI:10.1016/j.molcel.2010.09.019 |
| [22] |
Wei S, Li C, Yin Z, et al. Histone methylation in DNA repair and clinical practice:new findings during the past 5-years[J]. Journal of Cancer, 2018, 9(12): 2072-2081. DOI:10.7150/jca.23427 |
| [23] |
Jiricny J. The multifaceted mismatch-repair system[J]. Nature Reviews Molecular Cell Biology, 2006, 7(5): 335-346. DOI:10.1038/nrm1907 |
| [24] |
Lindahl T, Barnes D E. Repair of endogenous DNA damage[J]. Cold Spring Harb Symp Quant Biol, 2000, 65: 127-133. DOI:10.1101/sqb.2000.65.127 |
| [25] |
Caldecott K W. Single-strand break repair and genetic disease[J]. Nature Reviews Genetics, 2008, 9(8): 619-631. DOI:10.1038/nrg2380 |
| [26] |
West S C. Molecular views of recombination proteins and their control[J]. Nature Reviews Molecular Cell Biology, 2003, 4(6): 435-445. DOI:10.1038/nrm1127 |
| [27] |
Haber J E. The many interfaces of Mre11[J]. Cell, 1998, 95(5): 583-586. DOI:10.1016/S0092-8674(00)81626-8 |
| [28] |
Petrini J H, Stracker T H. The cellular response to DNA double-strand breaks:defining the sensors and mediators[J]. Trends in Cell Biology, 2003, 13(9): 458-462. DOI:10.1016/S0962-8924(03)00170-3 |
| [29] |
Blanc R S, Richard S. Arginine methylation:the coming of age[J]. Molecular Cell, 2017, 65(1): 8-24. DOI:10.1016/j.molcel.2016.11.003 |
| [30] |
Boisvert F M, Dery U, Masson J Y, et al. Arginine methylation of MRE11 by PRMT1 is required for DNA damage checkpoint control[J]. Genes & Development, 2005, 19(6): 671-676. |
| [31] |
Dery U, Coulombe Y, Rodrigue A, et al. A glycine-arginine domain in control of the human MRE11 DNA repair protein[J]. Molecular and Cellular Biology, 2008, 28(9): 3058-3069. DOI:10.1128/MCB.02025-07 |
| [32] |
Yu Z, Vogel G, Coulombe Y, et al. The MRE11 GAR motif regulates DNA double-strand break processing and ATR activation[J]. Cell Research, 2012, 22(2): 305-320. DOI:10.1038/cr.2011.128 |
| [33] |
Zhou B B, Elledge S J. The DNA damage response:putting checkpoints in perspective[J]. Nature, 2000, 408(6811): 433-439. DOI:10.1038/35044005 |
| [34] |
Iwabuchi K, Bartel P L, Li B, et al. Two cellular proteins that bind to wild-type but not mutant p53[J]. Proc Natl Acad Sci U S A, 1994, 91(13): 6098-6102. DOI:10.1073/pnas.91.13.6098 |
| [35] |
Ditullio R A, Mochan T A, Venere M, et al. 53BP1 functions in an ATM-dependent checkpoint pathway that is constitutively activated in human cancer[J]. Nature Cell Biology, 2002, 4(12): 998-1002. DOI:10.1038/ncb892 |
| [36] |
Boisvert F M, Rhie A, Richard S, et al. The GAR motif of 53BP1 is arginine methylated by PRMT1 and is necessary for 53BP1 DNA binding activity[J]. Cell Cycle, 2005, 4(12): 1834-1841. DOI:10.4161/cc.4.12.2250 |
| [37] |
Huyen Y, Zgheib O, Ditullio R A, et al. Methylated lysine 79 of histone H3 targets 53BP1 to DNA double-strand breaks[J]. Nature, 2004, 432(7015): 406-411. DOI:10.1038/nature03114 |
| [38] |
Nishitoh H, Kadowaki H, Nagai A, et al. ALS-linked mutant SOD1 induces ER stress- and ASK1-dependent motor neuron death by targeting Derlin-1[J]. Genes & Development, 2008, 22(11): 1451-1464. |
| [39] |
Cho J H, Lee M K, Yoon K W, et al. Arginine methylation-dependent regulation of ASK1 signaling by PRMT1[J]. Cell Death & Differentiation, 2012, 19(5): 859-870. |
| [40] |
Hirata Y, Katagiri K, Nagaoka K, et al. TRIM48 promotes ASK1 activation and cell death through ubiquitination-dependent degradation of the ASK1-negative regulator PRMT1[J]. Cell Reports, 2017, 21(9): 2447-2457. DOI:10.1016/j.celrep.2017.11.007 |
| [41] |
Tristan C, Shahani N, Sedlak T W, et al. The diverse functions of GAPDH:views from different subcellular compartments[J]. Cellular Signalling, 2011, 23(2): 317-323. DOI:10.1016/j.cellsig.2010.08.003 |
| [42] |
Munn D H, Beall A C, Song D, et al. Activation-induced apoptosis in human macrophages:developmental regulation of a novel cell death pathway by macrophage colony-stimulating factor and interferon gamma[J]. The Journal of Experimental Medicine, 1995, 181(1): 127-136. DOI:10.1084/jem.181.1.127 |
| [43] |
Cho J H, Lee R, Kim E, et al. PRMT1 negatively regulates activation-induced cell death in macrophages by arginine methylation of GAPDH[J]. Experimental Cell Research, 2018, 368(1): 50-58. DOI:10.1016/j.yexcr.2018.04.012 |
| [44] |
Rossing K, Christensen P K, Hovind P, et al. Progression of nephropathy in type 2 diabetic patients[J]. Kidney International, 2004, 66(4): 1596-1605. DOI:10.1111/j.1523-1755.2004.00925.x |
| [45] |
Park M J, Han H J, Kim D I. Lipotoxicity-induced PRMT1 exacerbates mesangial cell apoptosis via endoplasmic reticulum stress[J]. International Journal of Molecular Sciences, 2017, 18(7): 1421. DOI:10.3390/ijms18071421 |
 2020, Vol. 44
2020, Vol. 44


