文章信息
- 刘凯旋, 岳欣, 刘保忠. 2020.
- LIU Kai-xuan, YUE Xin, LIU Bao-zhong. 2020.
- 文蛤鳃组织内共生菌分离鉴定和多样性分析
- Isolation, identification, and diversity analysis of endosymbiotic bacteria in gill tissue of Meretrix petechialis
- 海洋科学, 44(8): 178-185
- Marine Sciences, 44(8): 178-185.
- http://dx.doi.org/10.11759/hykx20200319002
-
文章历史
- 收稿日期:2020-03-19
- 修回日期:2020-04-01
2. 中国科学院实验海洋生物学重点实验室, 山东 青岛 266071;
3. 青岛海洋科学与技术试点国家实验室海洋生物学与生物技术功能实验室, 山东 青岛 266237;
4. 中国科学院大学, 北京 100049
2. CAS Key Laboratory of Experimental Marine Biology, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
3. Marine Biology and Biotechnology Laboratory, Pilot National Laboratory for Marine Science and Technology(Qingdao), Qingdao 266237, China;
4. University of Chinese Academy of Sciences, Beijing 100049, China
内共生菌是指生活在另一个有机体(宿主)体内或细胞内的微生物[1]。宿主的微生物群落处于动态变化之中, 其与宿主的生长发育、信息交流、免疫及健康密切相关。植物内共生菌的研究已有大量报道, 并且有研究发现感染内共生菌的宿主比未感染植株具有生长快速、抗逆境等优势[2]。动物内共生菌目前在珊瑚、海绵等海洋生物中已有较多研究, 发现了大量具有抗菌活性的有益细菌及真菌[3]; 在牡蛎、贻贝等双壳贝类体内也发现了多种共生细菌[4], 其中多种细菌具有抗菌活性[5]。
文蛤(Meretrix petechialis)是一种广温、广盐性的贝类, 属于双壳纲(Bivalvia)、帘蛤科(Veneridae)、文蛤属(Meretrix), 是广泛分布于我国沿海的一种重要的经济贝类[6]。文蛤在夏季高温时期易发生爆发性死亡, 病原菌感染是主要原因之一[4]。已有的研究指出, 水产动物体内的天然微生物群体对水产动物抵御病原体入侵的能力和机体的抗逆性有一定的影响[7]。文蛤等双壳类的鳃作为其与水体直接接触的器官, 也是病原菌攻击的目标。因此, 鳃组织在文蛤防御和免疫系统中的作用就显得尤为重要, 其中的微生物群落也值得我们探究。目前, 文蛤内共生细菌的分离与鉴定尚无相关报道。开展文蛤鳃组织内共生菌的筛选和分析, 有助于发现贝类潜在的病原菌以及有益的微生物, 对于文蛤的生态养殖和病害防控具有重要的科学意义。
1 材料与方法 1.1 实验材料本实验所使用的多批次文蛤分别于2019年3—6月在山东省青岛市南山水产市场以及浙江省温州市乐清市翁垟镇购买, 于室温海水暂养两天后进行解剖取样。暂养过程中水体保持充气, 早、晚各换水1次, 期间不投喂饵料。
1.2 细菌菌株的分离与纯化取健康文蛤的鳃, 用无菌海水清洗3次后, 使用磁珠组织匀浆仪打碎匀浆, 磁珠及离心管均为无菌状态。匀浆液用灭菌海水稀释成10–1、10–2、10–3、10–4、10–5、10–6共6个梯度。在超净工作台中, 分别吸取原液及6个稀释梯度的样品各200 μL加到2216E、BG-11、ASN-III、放线菌培养基共4种分离培养基上, 每个梯度设3个平行, 将培养基表面的样品用无菌玻璃涂布棒涂布均匀。将平板放置于25℃恒温培养箱倒置培养, 至少培养120 h后挑取形态、颜色各异的单个菌落, 在新的固体培养基上采用四区划线分离法对挑取菌落进行分离纯化, 3次划线纯化后挑取单菌落保种于添加了20%(W/V)甘油的各液体培养基中, 冻存于–80℃冰箱待用。
1.3 菌株16S rRNA鉴定对分离、纯化过后的细菌进行16S rRNA鉴定。挑取单菌落到已加入20 μL灭菌超纯水的200 μL小管中, 100℃金属浴加热15 min后离心, –20℃过夜, 溶解后上清液作为模板在PCR仪中进行扩增。扩增引物在上海生工生物有限公司订购, 为16S-27F (5’-AGAGTTTGATCMTGGCTCAG-3’)和16S-1492R (5’-GGTTACCTTGTTACGACTT-3’)。PCR扩增的反应体系为: LA Taq mix 25 μL, 16S-27F 1.25 μL, 16S-1492R 1.25 μL, DNA模板2.0 μL, ddH2O 20.5 μL。扩增的条件为95℃预变性4 min, 95℃变性30 s, 55℃退火30 s, 72℃延伸60 s, 进行35个循环, 4℃保存。PCR产物经1%琼脂糖凝胶电泳检测后, 送至上海生工生物有限公司进行Sanger测序。
1.4 数据分析及系统发育树构建将测序所得的近1 500 bp的基因序列在NCBI (https://blast.ncbi.nlm.nih.gov/Blast.cgi)及EzBioCloud (https://www.ezbiocloud.net/identify)网站上进行相似性比对, 分析判断分离到的细菌的种类, 以相似度低于98%作为潜在新种的筛选标准[8]。将选取的代表性序列及其同源序列使用CLUSTAL_X 2.1软件进行多序列比对, 使用MEGA X软件, 根据Kimura模型估算系统进化矩阵, 采用邻接法进行聚类分析, 设置自举法(Boostrap method)1 000次构建系统发育树。
2 结果通过对文蛤鳃组织的内共生菌的多批次筛选, 共筛选到215株细菌, 无潜在新种。对这些细菌进行16S rRNA基因序列比对, 结果显示所筛选到的全部菌株分布在变形菌门、厚壁菌门、拟杆菌门和放线菌门4个门级类群中(图 1)。其中, 变形菌门为本次分离获得的可培养细菌的主要门级类群, 占全部纯培养细菌的76%。其次为厚壁菌门, 占本次分离到的可培养细菌的13%, 拟杆菌门和放线菌门仅占此次分离到的可培养细菌的6%及5%。
 |
| 图 1 4个门级类群中菌株数量所占比例分布 Fig. 1 Proportion of the number of strains in four phyla |
实现纯培养的变形菌门细菌分布于α-变形菌纲、γ-变形菌纲和ε-变形菌纲的10个目的30个属, 共163株, 是文蛤鳃组织中获得的可纯培养菌株数量最多的类群。其中包括α-变形菌纲15个属的59株细菌, γ-变形菌纲14个属的96株细菌, 以及ε-变形菌纲弓形杆菌属的8株细菌。α-变形菌纲及γ-变形菌纲中属级类群的菌株分布如图 2所示。
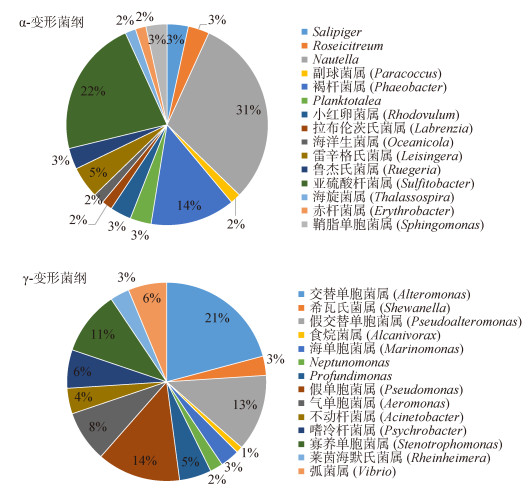 |
| 图 2 α-变形菌纲和γ-变形菌纲中属级类群的菌株比例分布 Fig. 2 Proportion of the numbers of strains in 29 genera of α-Proteobacteria and γ-Proteobacteria |
在α-变形菌纲中, 分离菌株最多的属级类群为Nautella属, 共分离到相关菌株18株; 其次为红杆菌科的亚硫酸杆菌属, 共分离到13株细菌。另外, 该纲中筛选得到的菌株数量较多的类群还包括褐杆菌属(8株), 其余属级类群分离到的菌株数均小于等于3株。
在γ-变形菌纲中, 共分离到交替单胞菌属菌株20株, 是分离菌株最多的属级类群; 假单胞菌属也是本研究中出现频率较高的类群之一, 共分离到13株细菌; 其次为假交替单胞菌属, 共分离到12株细菌。另外, 筛选得到菌株数量较多的类群还包括寡养单胞菌属(10株)、气单胞菌属(8株)和弧菌属(6株), 其余属级类群分离到的菌株数均小于等于6株。
2.2 厚壁菌门本研究中实现纯培养的厚壁菌门细菌分布于芽孢杆菌纲的5个属, 共27株, 各属级类群的菌株分布见图 3。在属的水平上, 厚壁菌门分离菌株最多的类群为葡萄球菌属, 共分离到15株, 是本研究中出现频率最高的种类之一; 其次为芽孢杆菌属, 共分离到7株; 此外, 类芽孢杆菌属分离到3株细菌, 大洋芽胞杆菌属及赖氨酸芽胞杆菌属均只分离到1株细菌。
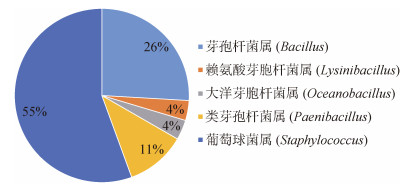 |
| 图 3 厚壁菌门中属级类群的菌株数量所占比例分布 Fig. 3 Proportion of the numbers of strains in five genera of Firmicutes |
拟杆菌门共有黄杆菌纲的5个属共14株细菌实现了纯培养, 每个属的代表菌株及其16S rRNA序列相似性如见表 1。分离菌株最多的属级类群为Bizionia属, 共分离到9株; 其次为Euzebyella属, 分离到2株; 此外Aequorivita属、Tenacibaculum属及Winogradskyella属均只分离到1株。
| 属 | 代表菌株 | 最相似菌株 | 序列号 | 相似度/% |
| Bizionia | MP21 | Bizionia hallyeonensis strain T-y7 | NR_109525.1 | 100 |
| Euzebyella | MP226 | Euzebyella saccharophila strain 7SM30 | NR_117007.1 | 99.86 |
| Aequorivita | MP32 | Aequorivita sp. strain 609 | MK347067.1 | 98.43 |
| Tenacibaculum | MP102 | Tenacibaculum litoreum strain KNUSC3025 | KP342148.1 | 100 |
| Winogradskyella | MP18 | Winogradskyella thalassocola isolate S4-6 | AY771730.1 | 98.85 |
本研究中共有分布于放线菌纲的6个属、11株放线菌门细菌实现了纯培养, 每个属的代表菌株及其16S rRNA序列相似性见表 2。其中微球菌属是实现纯培养菌株最多的属级类群, 共分离到4株; 其次为链霉菌属, 分离到3株; 此外考克氏菌属、壤霉菌属、小单胞菌属以及罗氏菌属均只分离到1株。
| 属 | 代表菌株 | 最相似菌株 | 序列号 | 相似度/% |
| Kocuria | MP157 | Kocuriasp. 13-3-7 | KM886142.1 | 99.93 |
| Micrococcus | MP2 | Micrococcus aloeverae strain SGP3 | MK426769.1 | 99.93 |
| Agromyces | MP214 | Agromycessp. AK1 | FN868445.1 | 99.57 |
| Micromonospora | MP206 | Micromonospora sp. 2802GPT1-4 | JQ836677.1 | 99.42 |
| Rothia | MP176 | Rothia amarae strain R-36507 | FR682692.1 | 99.54 |
| Streptomyces | MP210 | Streptomyces chungwhensis strain AA-98 | AY382292.2 | 99.79 |
由于本研究中分离到的变形菌门细菌种类及数量较多, 而放线菌门、拟杆菌门及厚壁菌门所包含菌株的纲级和目级类群比较单一, 故选取变形菌门的代表菌株进行系统发育树的构建。将测序得到的文蛤鳃组织中变形菌门30个属的共30株代表菌株的16S rRNA序列进行系统发育分析, 获得的系统发育树如图 4所示。文蛤鳃组织内共生变形菌门聚类成3个大的分支, 分别为α-变形菌纲、γ-变形菌纲和ε-变形菌纲。建树所选取的代表菌株之间没有发生聚集, 且和其同源菌株均聚集形成了独立的小分支。从发育树中可以看出, α-变形菌纲可分为3个主要分支, 其中红杆菌科12个属的代表菌株以及其同源菌株聚集在一个较大的分支内, 鞘脂单胞菌科的Erythrobacter属和Sphingomonas属的代表菌株及同源菌株在较小的分支内, 红螺菌科的Thalassospira属代表菌株及其同源菌株聚集在一个独立的小分支里。γ-变形菌纲中科级类群众多, 其中海洋螺菌科以及莫拉菌科的代表菌株及其同源菌株分别聚集在两个独立分支中。莫拉氏菌科中的Acinetobacter属与Psychrobacter属相关菌株聚成独立的一支, 海洋螺菌科中的Marinomonas属、Neptunomonas属及Profundimonas属的代表菌株及同源菌株聚集成一个较大的独立分支。
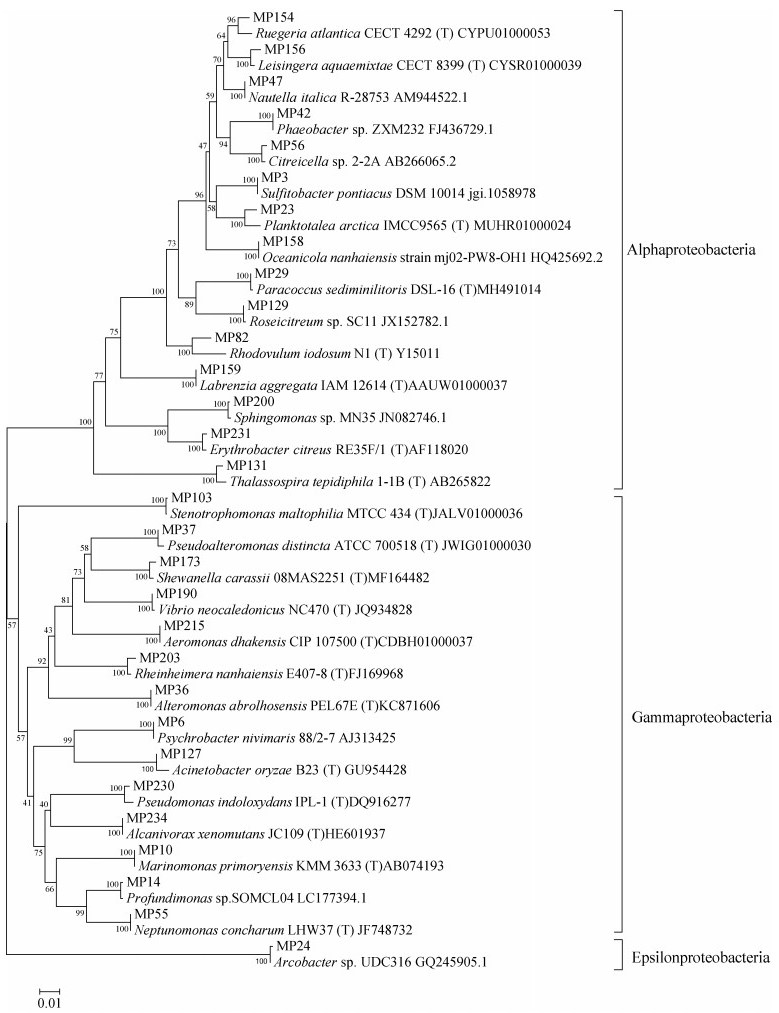 |
| 图 4 基于16S rRNA基因序列构建的文蛤鳃组织分离到的变形菌门代表菌株的系统发育树 Fig. 4 Neighbor-joining phylogenetic trees of the selected references strains in Proteobacteria and their related strains isolated from the gill of Meretrix petechialis |
鳃组织作为文蛤的滤食器官, 直接与水体接触, 相比其他器官更易受到病原菌的侵袭。越来越多的证据表明, 在贝类等无脊椎动物中, 鳃是参与病原体侵袭免疫应答的主要组织[9]。本研究通过纯培养方式, 对文蛤鳃组织内共生微生物进行了分析。对筛选到的细菌进行16S rRNA序列测序及比对分析, 共得到了215株细菌。分析显示, 其中很多优势种群具有分泌活性化合物抑制病原菌的潜能, 同时也发现了多个潜在的致病菌株。
内共生微生物在多细胞生物体中与宿主共同进化并对宿主具有重要影响[10]。在胚胎发育初始阶段, 长臂虾Palaemon macrodactylus胚胎表面的共生细菌可分泌化合物保护胚胎免受真菌侵害[11]; 夏威夷短尾鱿鱼Euprymna scolopes在胚胎发生过程中会形成一个复杂的器官, 可促进随后的共生细菌Vibrio fischeri定植[12]。另外, 共生菌可以合成昆虫所需的各种营养物质, 如甲虫和吸血性昆虫的共生菌可提供B族维生素, 而蟑螂和同翅目昆虫体内的共生菌则可提供必需氨基酸[13]。在斑马鱼中, 恶唑酮引起的小肠结肠炎的浸润及其严重程度受到微生物群组成的影响, 万古霉素处理后梭杆菌门成为肠道菌群的优势菌门, 且对恶唑酮的炎症反应明显降低[14]。本研究中获得的α-变形菌纲中的鞘脂单胞菌属菌株, 有文献报道其在水生动物中的分离株具有抗菌能力。蚶(Anadara broughtonii)中分离得到的鞘脂单胞菌, 对金黄色葡萄球菌、枯草杆菌、粪肠杆菌等多种病原菌均有抗菌活性[5]。亚硫酸杆菌属是本次分离过程中出现频率最高的类群之一, 已有文献表明从海草和浮游植物中分离的亚硫酸杆菌, 对真菌和菌鳗弧菌表现出很强的抑菌能力[15-16]。本研究中γ-变形菌纲中分离菌株最多的交替单胞菌属, 也有多篇文献证实其分泌物具有抗菌活性。从海水中分离得到的Alteromonas rava sp. nov. SANK73390可产生一种抗生素, 对革兰氏阴性菌和阳性菌均有很强的抑制活性[17]; 而菌株Alteromonas strain P-31则可以分泌一种大分子耐热糖蛋白, 对几种重要的鱼类致病菌均有抑制作用[18]。此外, 从海藻中分离得到的交替单胞菌也被证实具有抑菌活性[19]。厚壁菌门中分离菌株数量最多的葡萄杆菌属, 已有大量文献报道其能够产生抗菌的代谢产物, 在海草中分离得到的该属菌株对多种病原真菌有很强的抑制活性[16]。放线菌作为一类具有高G+C含量的革兰氏阳性细菌, 因其分布广泛并能产生丰富的活性次生代谢产物而著称。例如, 从丛生盔形珊瑚中分离得到的白刺放线菌的分泌物具有广谱抑菌活性, 对多种病原菌均有抑菌活性[20]。本次分离得到的放线菌中较为优势的类群链霉菌, 有报道其对副溶血弧菌、鳗弧菌和哈维氏弧菌等海水养殖病原菌均有较强的抑菌活性[21-22]。
本研究也从文蛤鳃组织也分离到多株潜在的病原菌。α-变形菌纲中分离菌株数量最多的Nautella属, 曾被广泛报道为藻类的致病菌, 如Nautella italica R11是红藻的病原[23]。有文献报道, 该属细菌在发病的凡纳滨对虾体内含量很高, 猜想其可能与虾的健康有密切关系[24]。本研究中分离到的ε-变形菌纲中的弓形杆菌属, 在濒死的牡蛎、饥饿的鲍鱼和坏死的海绵中都观察到Arcobacter sp.菌株的大量存在, 当这些菌株的浓度足够高时, 它们有可能成为条件致病菌[7]。在文蛤体内, 我们也发现了弧菌感染后文蛤肝胰腺中Arcobacter丰度的显著上升[25]。拟杆菌门Winogradskyella属细菌, 在许多海洋生物中包括牡蛎等十分常见, 据报道高浓度的Winogradskyella菌株与鱼类阿米巴鳃病密切相关[26], 此外, 黄杆菌属细菌也曾报道为鱼类的病原菌[27]。
已有研究表明, 水产动物的内共生菌群与其自身的免疫能力与抗病能力密切相关[28]。本研究通过纯培养方式对文蛤鳃组织的内共生细菌进行了分离、鉴定, 对获得的主要类群进行了分析, 并对其中具有潜在致病性或者抑菌活性的共生细菌进行了探讨, 为后续文蛤内共生菌群的深入研究提供了重要参考。
| [1] |
Brinker P, Fontaine M C, Beukeboom L W, et al. Host, symbionts, and the microbiome: The missing tripartite interaction[J]. Trends in Microbiology, 2019, 27(6): 480-488. |
| [2] |
宋薇薇, 朱辉, 余凤玉, 等. 植物内生菌及其对植物病害的防治作用综述[J]. 江苏农业科学, 2018, 46(6): 12-16. Song Weiwei, Zhu Hui, Yu Fengyu, et al. A review of endophytes and their effects on plant disease control[J]. Jiangsu Agricultural Sciences, 2018, 46(6): 12-16. |
| [3] |
汪福源, 顾觉奋, 高向东, 等. 动物内生真菌研究进展[J]. 国外医药(抗生素分册), 2017, 38(2): 58-61. Wang Fuyuan, Gu Juefen, Gao Xiangdong, et al. Research progress of animal endophytic Fungi[J]. World Notes on Antibiotics, 2017, 38(2): 58-61. |
| [4] |
Kueh C S W, Chan K-Y. Bacteria in bivalve shellfish with special reference to the oyster[J]. Journal of Applied Microbiology, 1985, 59(1): 41-47. |
| [5] |
Romanenko L A, Uchino M, Frolova G M, et al. Sphingomonas molluscorum sp. nov., a novel marine isolate with antimicrobial activity[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(Pt 2): 358-363. |
| [6] |
宋文涛. 文蛤的养殖和研究现状[J]. 山东工业技术, 2013(11): 246. Song Wentao. Current situation of culture and research of Meretrix[J]. Shandong Industrial Technology, 2013(11): 246. |
| [7] |
Lokmer A, Wegner M K. Hemolymph microbiome of Pacific oysters in response to temperature, temperature stress and infection[J]. The International Society for Microbial Ecology Journal, 2015, 9(3): 670-682. |
| [8] |
Stackebrandt E, Ebers J. Taxonomic parameters revisited: Tarnished gold standards[J]. Microbiology Today, 2006, 6(4): 152-155. |
| [9] |
Li Y, Song X, Wang W, et al. The hematopoiesis in gill and its role in the immune response of Pacific oyster Crassostrea gigas against secondary challenge with Vibrio splendidus[J]. Developmental and Comparative Immunology, 2017, 71: 59-69. |
| [10] |
Mcfall-Ngai M, Hadfield M G, Bosch T C, et al. Animals in a bacterial world, a new imperative for the life sciences[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(9): 3229-3236. |
| [11] |
Gil-Turnes M S, Hay M E, Fenical W. Symbiotic marine bacteria chemically defend crustacean embryos from a pathogenic fungus[J]. Science, 1989, 246(4926): 116. |
| [12] |
Montgomery M K, Mcfall-Ngai M. Bacterial symbionts induce host organ morphogenesis during early postembryonic development of the squid Euprymna scolopes[J]. Development, 1994, 120(7): 1719-1729. |
| [13] |
Douglas A E. Mycetocyte symbiosis in insects[J]. Biological reviews of the Cambridge Philosophical Society, 1989, 64(4): 409-434. |
| [14] |
Brugman S, Liu K Y, Lindenbergh-Kortleve D, et al. Oxazolone-induced enterocolitis in zebrafish depends on the composition of the intestinal microbiota[J]. Gastroenterology, 2009, 137(5): 1757-1767. |
| [15] |
Sharifah E N, Eguchi M. The phytoplankton Nannochloropsis oculata enhances the ability of Roseobacter clade bacteria to inhibit the growth of fish pathogen Vibrio anguillarum[J]. The Public Library of Science One, 2011, 6(10): e26756. |
| [16] |
Bibi F, Naseer M I, Hassan A M, et al. Diversity and antagonistic potential of bacteria isolated from marine grass Halodule uninervis[J]. 3 Biotech, 2018, 8(1): 48. |
| [17] |
Shiozawa H, Kagasaki T, Kinoshita T, et al. Thiomarinol, a new hybrid antimicrobial antibiotic produced by a marine bacterium. Fermentation, isolation, structure, and antimicrobial activity[J]. Journal of Antibiotics, 1993, 46(12): 1834-1842. |
| [18] |
Barja J L, Lemos M L, Toranzo A E. Purification and characterization of an antibacterial substance produced by a marine Alteromonas species[J]. Antimicrobial Agents and Chemotherapy, 1989, 33(10): 1674-1679. |
| [19] |
Lemos M L, Toranzo A E, Barja J L. Antibiotic activity of epiphytic bacteria isolated from intertidal seaweeds[J]. Microbial Ecology, 1985, 11(2): 149-163. |
| [20] |
张斯颖, 陈宇丰, 骆焱平, 等. 丛生盔形珊瑚共附生放线菌NLG-3的抑菌活性及鉴定[J]. 西北农林科技大学学报(自然科学版), 2019, 47(10): 97-105. Zhang Siying, Chen Yufeng, Luo Yanping, et al. Antibacterial activity and identification of epiphytic actinomycete NLG-3 from Galaxea facicularis[J]. Journal of Northwest A & F University(Natural Science Edition), 2019, 47(10): 97-105. |
| [21] |
杜英侠, 任浩杰, 张菊林, 等. 海绵放线菌的分离与抑菌活性筛选研究[J]. 现代农业科技, 2017(7): 229-231, 233. Du Yingxia, Ren Haojie, Zhang Julin, et al. Isolation of sponge-associated actinobacteria and its antibacterial activity screening[J]. Modern Agricultural Science and Technology, 2017(7): 229-231, 233. |
| [22] |
杜英侠, 李晶莹, 傅敏, 等. 一株海绵放线菌的分离培养、发酵条件优化及其抑制弧菌效果[J]. 大连海洋大学学报, 2017, 32(4): 457-464. Du Yingxia, Li Jingying, Fu Min, et al. Isolation, optimization of fermentation conditions and inhibition of Vibrio in a sponge-associated actinobacterium FH[J]. Journal of Dalian Ocean University, 2017, 32(4): 457-464. |
| [23] |
Hudson J, Gardiner M, Deshpande N, et al. Transcriptional response of Nautella italica R11 towards its macroalgal host uncovers new mechanisms of host- pathogen interaction[J]. Molecular Ecology, 2018, 27(8): 1820-1832. |
| [24] |
Zheng Y, Yu M, Liu J, et al. Bacterial community associated with healthy and diseased pacific white shrimp (Litopenaeus vannamei) larvae and rearing water across different growth stages[J]. Frontiers in Microbiology, 2017, 8: 1362. |
| [25] |
王瑞.文蛤在弧菌感染过程中载菌量及免疫相关因子变化的初步研究[D].北京: 中国科学院大学; 青岛: 中国科学院海洋研究所, 2019. Wang Rui. Study on bacterial load and the changes of immune-related factors of Meretrix petechialis in Vibrio infection[D]. Beijing: University of Chinese Academy of Sciences; Qingdao: Institute of Oceanology, Chinese Academy of Sciences, 2019. |
| [26] |
Embar-Gopinath S, Butler R, Nowak B. Influence of salmonid gill bacteria on development and severity of amoebic gill disease[J]. Diseases of Aquatic Organisms, 2005, 67(1-2): 55-60. |
| [27] |
Fernandez-Alvarez C, Santos Y. Identification and typing of fish pathogenic species of the genus Tenacibaculum[J]. Applied Microbiology and Biotechnology, 2018, 102(23): 9973-9989. |
| [28] |
Lokmer A, Kuenzel S, Baines J F, et al. The role of tissue-specific microbiota in initial establishment success of Pacific oysters[J]. Environmental Microbiology, 2016, 18(3): 970-987. |
 2020, Vol. 44
2020, Vol. 44


