文章信息
- 赵珍, 王宝杰, 刘梅, 蒋克勇, 王雷. 2021.
- ZHAO Zhen, WANG Bao-jie, LIU Mei, JIANG Ke-yong, WANG Lei. 2021.
- 过硫酸氢钾复合物对对虾养殖底泥硝化作用、氨氧化微生物丰度和群落结构的影响
- Effects of potassium monopersulfate on the nitrification, abundance, and community structure of ammonia-oxidizing microorganisms in shrimp culture sediments
- 海洋科学, 45(11): 54-61
- Marine Sciences, 45(11): 54-61.
- http://dx.doi.org/10.11759/hykx20210228002
-
文章历史
- 收稿日期:2021-02-28
- 修回日期:2021-03-23
2. 中国科学院 海洋大科学中心, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 青岛海洋科学与技术国家实验室海洋生物学与生物技术功能实验室, 山东 青岛 266237
2. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Laboratory for Marine Biology and Biotechnology, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
对虾产业不仅为人类提供优质的蛋白质来源, 而且还是全球水产养殖产值增长的主要推动力。随着水产养殖业的集约化发展, 越来越多的养殖污染问题随着而来, 其中一个主要的问题就是养殖过程中含氮化合物的积累。这些含氮化合物包括有机氮和无机氮(氨氮、亚硝酸盐和硝酸盐), 主要来源于虾类排泄物和未被食用的饲料[1]。在集约化养殖中被大量使用的肥料和补充饲料都是含氮量很高的有机物质, 但这些物质中只有15%~30% 能够被虾吸收利用, 其余未被吸收和利用的部分会沉积在养殖池塘底部的水-泥界面。沉积物中过量的氮不仅会破坏水产养殖环境, 对养殖生物产生毒害, 而且排放还会影响周围水体[2]。因此去除沉积物中多余的氮是非常重要的。
硝化作用是生态系统氮素循环过程中的一个重要过程, 整个过程包括两个步骤, 氨氧化和亚硝酸盐氧化。其中氨氧化是硝化反应的第一步也是限速步骤, 该步骤由氨氧化微生物-氨氧化古菌(AOA)和氨氧化细菌(AOB)共同介导进行。该步骤中起重要作用的酶——氨单加氧酶(ammonia monooxygenase, AMO)由A、B、C三个亚基组成, 其中编码A亚基的amoA基因具有一定序列保守性, 为所有氨氧化微生物共有的基因, 因此常被作为分子标记应用于氨氧化微生物的研究[3-5]。氮素浓度、温度、盐度等环境因子的变化以及施肥和化学物质干扰等都可能引起AOA和AOB的数量和群落结构的变化[6-8]。
长期以来围绕着氮素污染防治进行了大量的研究, 过硫酸氢钾复合物2KHSO5·KHSO4·K2SO4(KMPS)溶于水后发生链式反应, 产生活性氧, 羟基自由基、硫酸自由基等多种活性成分[9], 广泛的用作水产中的消毒剂使用。KMPS可以降解多种有机物, 并且可以将NH4+氧化为亚硝酸, 因此可用于降低池塘中氨氮、亚硝酸盐、硫化氢等有害物质的浓度, 用于养殖水体的治理和底质改造[10]。目前KMPS在水产养殖中的研究多集中作为消毒剂对水产致病菌的抑菌作用[11], 而其对底质氮素污染防治的研究有所欠缺。本研究通过对表层底泥中氨氮、亚硝态氮水平变化进行检测, 分析KMPS对氮素含量的影响, 并通过qPCR, 16s高通量测序等分子生物学技术对底泥中AOA和AOB数量和群落结构进行分析, 以期揭示KMPS对硝化作用的影响。
1 材料与方法 1.1 实验设计及养殖管理实验在中国山东省东营市黄河三角洲海洋科技有限公司研发实验室进行, 从8月3日至9月14日共进行6周, 为模拟封闭池塘的养殖环境, 准备9个柱形养殖桶(500 L), 桶内铺设10 cm新鲜底泥, 覆入盐度为34的海水静置24 h后使用。实验所用的凡纳滨对虾养殖至重量0.3 g, 体长1.5 cm后被随机分配至每桶300只对虾。暂养1周之后将9个养殖桶分成3个组, 分别为: 处理组A为高频率低剂量组: 以5 g/m3的剂量, 每2周投放3次KMPS; 处理组B为低频率高剂量组: 以15 g/m3的剂量, 每2周投放1次KMPS; 对照组C: 除日常养殖管理外不进行其他处理。养殖期间, 每日按体重的8%, 分别于6: 00 am、12: 00 am、6: 00 pm、12: 00 pm投喂商品饲料。每周少量换水10 cm, 本研究使用的KMPS为片剂, 由山东嘉源环保科技有限公司提供, 纯度为100%。
1.2 样品的采集及前处理使用自制的采集器采集0.5~2 cm的表层底泥[12], 分别于实验开始时和实验结束时采集1次, 实验期间每周采集1次。采集的底泥样品一部分用于氨氮、亚硝氮检测, 另一部分迅速置于液氮中保存, 之后送回实验室保存在-80 ℃冰箱待之后的分子生物学实验分析使用。
1.3 氨氮、亚硝态氮的测定取5 g新鲜样品置于50 mL离心管中, 加入25 mL, 2 mol/L的KCl溶液, 25 ℃摇床浸提1 h, 之后泥浆经3 000 r离心10 min, 取上清液测定营养盐含量。按照海洋监测规范的标准采用次溴酸盐氧化法、萘乙二胺分光光度法分别检测氨氮和亚硝酸盐浓度[13]。
1.4 DNA的提取和PCR扩增采用Fast DNA Spin Kit For soil(MPbio), 美国试剂盒进行底泥样品中微生物DNA提取, 具体操作步骤按照说明书进行。在下一步分析前将提取的基因组DNA储存在-20 ℃。提取后的DNA的浓度和质量分别使用NanoDrop ND-1000分光光度计(Thermo Fisher Scientific, Waltham, MA, USA)和琼脂糖凝胶电泳进行检测。选用引物Arch-amoA26F (5′-GACTACATMTTCTAYACWGAYTGGGC-3′)和Arch-amoA417R(5′-GGKGTCATRTATGGWGGYAAYGTTGG-3′)扩增AOA-amoA基因; 引物amoA1F (5′-GGGGTTTCTACTGGTGGT-3′)和amoA2R(5′- CCCCTCKGSAAAGCCTTCTTC-3′)扩增AOB-amoA基因。PCR反应体系为: 5×反应buffer 5 μL, 5×GC buffer 5 μL, dNTP(2.5mmol·L-1)2 μL, 上游和下游引物各(10 μmol·L-1)1 μL, DNA模板2 μL, ddH2O 8.75 μL, Q5 DNA聚合酶0.25 μL; 反应程序: 98 ℃ 2 min, 98 ℃ 15 s, 55 ℃ 30 s, 72 ℃ 30 s, 72 ℃ 5 min, 共进行35个循环, 琼脂糖凝胶电泳进行检测。
1.5 荧光定量PCR将上述PCR产物回收, 连接至pMD18-T载体, 转化至大肠杆菌DH5α感受态中, 用含有Amp+的LB琼脂平板培养基筛选阳性克隆, 并对阳性克隆测序分析, 分别提取含AOA-amoA和AOB-amoA基因的质粒, 用NanoDrop2000(Thermo) 测定质粒浓度, 计算AOA和AOB amoA基因拷贝数, 将标准质粒进行一系列10倍浓度稀释(标准质粒范围AOA-amoA 102~108, AOA-amoA 10~106)用于构建qPCR标准曲线。各标准曲线R2均超过0.99, 在本研究使用的浓度范围内呈良好的线性关系。使用TransStar Top Green qRT-PCR Supermix(北京全式金生物科技有限公司)试剂盒进行AOA和AOB amoA基因丰度测定, 具体操作按说明书进行。
1.6 Illumina MiSeq高通量测序及分析采用Illumina平台对群落DNA片段进行双端(paired-end)测序。利用QIIME(QIIME, v1.8.0)软件进行DADA2对测序数据进行质量过滤、降噪和去除嵌合体获得高质量序列[14]。高质量序列通过UCLUST软件按照97%的序列同源性聚集成操作分类单元(OTUs)[15]。序列数据分析主要使用QIIME和R包(v3.2.0)基于OUT表计算alpha多样性指数, 以Chao1[16]和Observed species指数表征丰富度, 以Shannon[17]和Simpson[18]指数表征多样性。通过OUT表计算不同样本在各分类水平所含有的分类单元的数目, 实现所有样本在各分类水平上组成分布的可视化, 以堆叠柱状图呈现分析结果。
1.7 统计分析所有数据以“平均值±标准差”表示。用SPSS 25.0(IBM Corp, Armonk, NY, USA)统计软件中的单因素变量方差分析方法(One-Way ANOVA)对数据进行统计学分析。以P < 0.05代表差异显著。
2 结果与分析 2.1 底泥中氨氮及亚硝态氮含量变化对照组和实验组的氨氮及亚硝态氮变化如图 1a和图 1b所示, 整个实验期间各组的氨氮和亚硝氮均呈现上升的趋势。处理组B中的氨氮和亚硝氮在整个实验期间处于较低水平并且低于其他2组。实验开始的前2周, 对照组C中的氨氮水平高于2个处理组, 随着实验的进行, 对照组和实验组B中的氨氮含量先上升后下降, 而实验组A中的氨氮含量持续上升。对于亚硝氮, 前2周各组的水平与氨氮相似, 在对照组中最高。从第3周开始处理组A中的亚硝氮浓度快速上升, 迅速超过对照组C和处理组B。
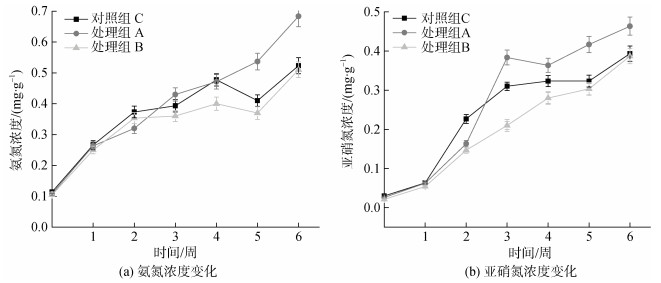 |
| 图 1 不同处理组中氨氮(a)亚硝氮(b)浓度变化 Fig. 1 Variations in ammonia (a) and nitrite (b) concentrations under different treatments 注: C: 对照组; A: 高频率低剂量组; B: 低频率高剂量组 |
在实验刚开始的前两周, 与对照组相比两个处理组的氨氮和亚硝氮的含量均下降, 证明KMPS的投放能够降低底泥中氨氮和亚硝氮的含量, 一方面可能是因为KMPS对含氮有机物的降解, 使进入硝化作用的含氮有机物减少, 从而使氨氮和亚硝氮的含量降低, 另一方面KMPS的强氧化作用改善了底泥中的氧化还原状态, 并且还能够释放氧气, 进一步促进了硝化作用。但是处理组A以少量多次进行KMPS的持续性的投放, 却没有使氨氮和亚硝氮降低, 反而高于对照组中氨氮和亚硝氮的含量。推测持续性的KMPS的投放可能对硝化作用微生物存在持续的抑制。硝化作用无法顺利进行, 造成氨氮和亚硝氮的积累。
2.2 AOA和AOB微生物丰度变化氨单加氧酶amoA基因的丰度可以用来表征环境中氨氧化微生物的数量, AOA-amoA基因拷贝数为2.3×104~3.31×105 copies·g-1, AOB-amoA基因拷贝数为1.48×103~2.68×104 copies·g-1。同期的AOA-amoA比AOB-amoA基因拷贝数高1~2个数量级, 此时环境中的AOA数量高于AOB。养殖的前2周处理组和对照组之间AOA-amoA基因拷贝数没有显著性差异(图 2a)(P>0.05), 第3周处理组B和对照组C中拷贝数上升, 显著高于同期处理组A中该基因的拷贝数(P < 0.05)。之后各组的AOA-amoA基因拷贝数均持续上升, 且处理组B和对照组C中基因拷贝数显著高于同期处理组A中基因的拷贝数。除第4周对照组C中AOA-amoA基因拷贝数显著高于处理组B中外, 其余时期该基因拷贝数在2个组之间没有显著差异(P>0.05)。各组AOB-amoA基因拷贝数逐渐上升(图 2b), 前四周各组间没有显著差异(P>0.05); 后两周处理组B和对照组C中该基因的拷贝数显著高于处理组A, 实验结束时的处理组B中基因拷贝数最高, 随后是对照组C(P < 0.05)。
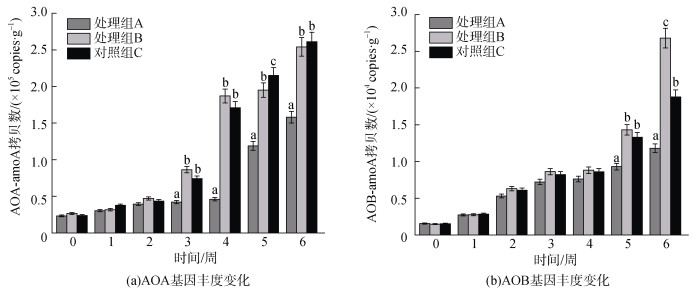 |
| 图 2 不同处理组中AOA(a)和AOB(b) amoA基因丰度变化 Fig. 2 Variations in AOA and AOB amoA gene abundance under different treatments |
对实验结束时的3个组的9个样品进行高通量测序分析, 平均每个样品获得高质量的AOA amoA基因序列78 344条, AOB amoA基因序列52 568条。将以上获得的序列按照97%的相似度进行聚类, 分别输出OTU代表序列。从表 1的结果可以看出处理组A的AOA和AOB的OUT数均显著低于其他2个组(P < 0.05)。通过alpha多样性指数对不同处理组的AOA和AOB微生物群落丰富度和多样性进行评估。对照组C的AOA微生物群落的丰富度指数(Chao1和ACE指数)显著高于两个处理组(P < 0.05), 但各组的多样性指数(Shannon和Simpson指数)没有显著的差异(P>0.05)。AOB微生物群落结果显示处理组A的丰富度指数显著低于对照组和处理组B, 多样性指数各组间没有显著的差异(P>0.05)。
| 处理 | 序列数 | 操作分类单元 | ACE指数 | Chao1指数 | Shannon指数 | Simpson指数 |
| 氨氧化古菌群落 | ||||||
| MA | 79 851 | 136a | 68.43±1.59a | 83.29±5.35a | 3.15±0.56 | 0.86±0.15 |
| MB | 65 322 | 212b | 105.30±1.85a | 111.01±1.73a | 2.51±0.67 | 0.74±0.11 |
| MC | 89 860 | 222b | 150.43±6.27b | 181.68±2.95b | 2.79±0.79 | 0.72±0.18 |
| 氨氧化细菌群落 | ||||||
| MA | 65 545 | 59a | 24.70±2.36a | 37.93±1.47a | 1.99±0.43 | 0.69±0.19 |
| MB | 56 694 | 165b | 62.26±1.93b | 81.07±1.51b | 1.59±0.82 | 0.49±0.16 |
| MC | 35 467 | 129b | 55.96±1.33b | 64.92±2.43b | 2.62±1.13 | 0.73±0.23 |
| 注: 同一列内标有不同字母表示差异显著(P < 0.05) | ||||||
为了了解样本中AOA和AOB群落的具体组成, 对抽平后的ASV/OTU表格进行统计计算之后获得每个样本在属水平组成和丰度分布表, 并以柱状图呈现分析结果。AOA群落组成如图 3a所示, 共有3个属组成该群落, 分别为Nitrosopumilus属、Nitrososphaera属和Candidatus Nitrosocosmicus属, 其中的优势属(相对丰度>1%)是Nitrosopumilus属和Nitrososphaera属, 分别占AOA amoA基因总序列的64.02%~91.05%和8.1%~27.4%。对照组C中Nitrosopumilus属的相对丰度显著高于处理组A和B (P < 0.05), 在两个处理组之间没有显著差异(P>0.05)。而对照组C中的Nitrososphaera属的相对丰度显著低于处理组A和B, 同样在2个处理组中没有显著差异。Candidatus Nitrosocosmicus属占AOA amoA基因总序列的比例较低且在各组的相对丰度没有显著差异(P>0.05)。AOB微生物群落共由6个属组成(图 3b), 其中Nitrosospira属和Nitrosomonas属为主要优势属, 分别占AOB amoA基因总序列的39.5%~67.6%和10.7%~50.1%。与对照组相比处理组的Nitrosospira属的相对丰度显著上升, 而处理组A中Nitrosomonas属的相对丰度显著下降(P < 0.05)。在2个处理组之间处理组B中Nitrosomonas属和Nitrosospira属的相对丰度均显著高于处理组A中(P < 0.05)。另外的4个属占AOB amoA基因总序列的比例较低且在各组的相对丰度没有显著差异(P>0.05)。
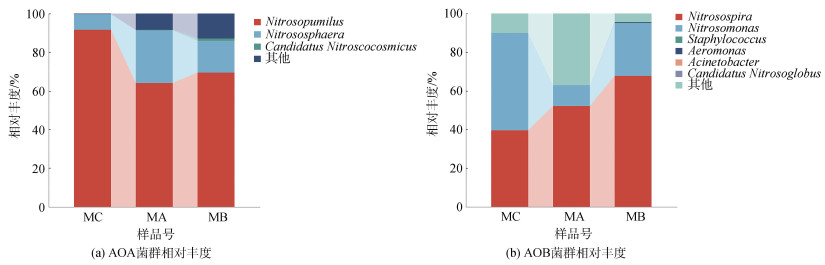 |
| 图 3 不同处理组AOA(a)和AOB(b)在属水平的菌群相对丰度 Fig. 3 Relative abundance of the AOA (a) and AOB (b) microbiota under different treatments at the genus level 注: MC对照组; MA: 高频率低剂量组; MB: 低频率高剂量组 |
养殖水体和底泥中的氨氮和亚硝氮是微生物利用含氮有机物进行硝化作用过程中产生的重要中间产物, 可以作为判断养殖环境好坏的重要指标[12]。对虾的排泄物、残余的饵料等的腐烂性分解会沉积在水-底泥界面, 容易造成界面的缺氧状态。硝化作用是需氧的氧化还原反应, 一定的氧含量是介导硝化作用的微生物进行硝化作用所必须的[19]。沉积在水-泥界面的对虾粪便、未利用的饵料等都是高含氮量的有机物, 致使底泥中氨氮和亚硝氮比水中高几个数量级[20]。随着养殖的进行各组的氨氮和亚硝氮含量均有不同程度的上升, 其中处理组A中氨氮和亚硝氮含量上升速度最快, 随后是对照组C; 第3周开始处理组A中的氨氮和亚硝氮含量持续高于对照组C和处理组B, 而且在处理组B中含量最低。这表明单次高剂量KMPS的投放, 可以有效的降低底泥中氨氮和亚硝氮的含量。这可能是因为KMPS片剂在水-泥界面分解释放氧气, 促进了硝化作用, 同理于过碳酸钠的施用下分解释放氧气, 溶解氧的增加促进氨氮的硝化作用, 进而促进了氮的循环[21]; 而且研究表明, KMPS可以直接与氨氮发生反应, 生成亚硝酸盐; 另外KMPS在污泥处理中可以降解多种复杂的有机物[22], 对底泥中的有机物也可能存在同样的作用。但是高频率低剂量KMPS的投放反而不利于硝化作用, 可能是连续的投放对硝化作用微生物产生持续性的抑制作用, 导致中间产物氨氮和亚硝氮的积累。
氨氧化微生物介导的有机物氨氧化过程作为限速步骤, 在整个硝化作用中起到决定性的作用。驱动氨氧化作用的微生物AOA和AOB广泛分布于几乎所有土壤、淡水、湖泊底泥和海洋环境中; 并且在许多环境中AOA比AOB更加丰富[23-26]。在我们实验环境中, AOA数量明显高于AOB, 推测AOA在此环境下的硝化作用中发挥着主导作用。随着养殖地进行, 介导氨氧化作用的AOA和AOB丰度均增长, 但同期高频率低剂量KMPS投放组的AOA和AOB丰度显著低于对照组, 表明高频率低剂量KMPS的投放对二者存在部分抑制作用。养殖开始的前两周KMPS的投放没有对AOA数量造成影响, 从第3周开始至实验结束时, 期间处理组A中的AOA数量始终低于其他两组, 表明高频率低剂量的KMPS投放持续对AOA造成影响。KMPS在水中能够在短时间内快速分解, 对很多微生物产生抑制作用[11]。除第5周外, 对照组C和处理组B中AOA数量没有显著差异, 推测单次高剂量KMPS投放对AOA的抑制作用不持续存在, 随着抑制的解除AOA可以恢复生长和繁殖。与AOA相比AOB在养殖的前中期的较长时间内对KMPS的投放并不敏感, 但从第4周开始在投放频率与投放量积累的双重作用下, 处理组A中AOB数量显著低于其余两组。
与基因丰度检测结果相似, alpha分析结果显示, 处理组A中的AOA和AOB相对丰度显著低于对照组C和处理组B, 表明高频率低剂量的KMPS投放对AOA和AOB起到抑制的作用。AOA和AOB的多样性在各组间没有显著差异, 表明KMPS投放与否以及投放的方式对氨氧化微生物的多样性不产生影响。KMPS对不同种微生物可产生不同作用, 可对某些微生物产生抑制而对其他一些微生物无影响[27]。当前的养殖环境中检测到AOA的种类较少, 优势属分别为Nitrosopumilus属、Nitrososphaera属, 这与之前的研究中表明Nitrosopumilus属和Nitrososphaera属是组成AOA群落主要的属一致[28-30]。本研究结果显示KMPS对二者的影响不同, 与对照组相比KMPS的投放降低了底泥中Nitrosopumilus属的丰度, 提升了Nitrososphaera属的丰度; 但在2个处理组之间2个优势属的相对丰度没有显著差异, 表明KMPS对AOA的影响与投放的方式关系不大。AOB微生物群落共由6个属组成, 其中Nitrosospira属和Nitrosomonas属为主要优势属, 与之前对水产养殖池塘沉积物[31]和淡水湖沉积物[32]的研究类似。与对照组相比, KMPS的投放提升了Nitrosospira属的相对丰度, 降低了处理组A中Nitrosomonas属的相对丰度。在2个处理组中, 处理组B中Nitrosomonas属和Nitrosospira属的相对丰度均显著高于处理组A中, 表明不仅是KMPS的投放与否, 投放的方式也会对AOB产生不同的影响, 低频率高剂量KMPS投放对AOB具有促进的作用。
4 结论本实验首次探究了KMPS投放对养殖底泥中氮素循环及氨氧化作用的影响。实验结果表明, 不同的投放方式产生的影响有明显的差别, 高频率低剂量KMPS投放对底泥中AOA和AOB的生长产生部分抑制作用, 使其不能够满足氮素转化的需求, 而低频率高剂量KMPS投放对AOA丰度没有明显影响, 对AOB群落生长表现出积极的作用, 因此促进硝化作用顺利进行。以上结果为KMPS可作用于底质中氮素的降解提供了理论依据, 同时为虾类养殖环境改善提供了更多的可选择办法。
| [1] |
SRITHEP P, KHINTHONG B, CHODANON T, et al. Communities of ammonia-oxidizing bacteria, ammonia-oxidizing archaea and nitrite-oxidizing bacteria in shrimp ponds[J]. Annals of Microbiology, 2014, 65(1): 267-278. |
| [2] |
DAI L, LIU C, YU L, et al. Organic matter regulates ammonia-oxidizing bacterial and archaeal communities in the surface sediments of ctenopharyngodon idellus aquaculture ponds[J]. Frontiers in Microbiology, 2018, 9: 2290. DOI:10.3389/fmicb.2018.02290 |
| [3] |
VENTER J C, REMINGTON K, HEIDELBERG J F, et al. Environmental genome shotgun sequencing of the Sargasso Sea[J]. Science, 2004, 304: 66-74. DOI:10.1126/science.1093857 |
| [4] |
TREUSCH A H, LEININGER S, KLETZIN A, et al. Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling[J]. Environmental Microbiology, 2005, 7(12): 1985-1995. DOI:10.1111/j.1462-2920.2005.00906.x |
| [5] |
WU R N, MENG H, WANG Y F, et al. A more comprehensive community of ammonia-oxidizing archaea (AOA) revealed by genomic DNA and RNA analyses of amoA gene in subtropical acidic forest soils[J]. Microbial Ecology, 2017, 74(4): 910-922. DOI:10.1007/s00248-017-1045-4 |
| [6] |
LIU H, LI J, ZHAO Y, et al. Ammonia oxidizers and nitrite-oxidizing bacteria respond differently to longterm manure application in four paddy soils of south of China[J]. Science of the Total Environment, 2018, 633: 641-648. DOI:10.1016/j.scitotenv.2018.03.108 |
| [7] |
LIMPIYAKORN T, SHINOHARA Y, KURISU F, et al. Distribution of ammonia-oxidizing bacteria in sewage activated sludge: analysis based on 16S rDNA sequence[J]. Water Science & Technology, 2004, 50(8): 9-14. |
| [8] |
LIMPIYAKORN T, KURISU F, SAKAMOTO Y, et al. Effects of ammonium and nitrite on communities and populations of ammonia-oxidizing bacteria in laboratory-scale continuous-flow reactors[J]. Fems Microbiology Ecology, 2007, 60(3): 501-512. DOI:10.1111/j.1574-6941.2007.00307.x |
| [9] |
ERMER O, RÖBKE C. Crystal structure and chemical stabilization of the triple salt (KHSO5)2·KHSO4·K2SO4[J]. Helvetica Chimica Acta, 2003, 86(8): 2908-2913. DOI:10.1002/hlca.200390238 |
| [10] |
唐兴刚, 魏文康, 罗胜军, 等. 过硫酸氢钾复合物在畜牧水产中的应用研究进展[J]. 中国兽药杂志, 2020, 54(8): 1002-1280. TANG Xingang, WEI Wenkang, LUO Shengjun, et al. Advances in research of potassium peroxomonosulfate compound on animal husbandry and aquatic[J]. Chinese Journal of Veterinary Drug, 2020, 54(8): 1002-1280. |
| [11] |
曾晓丹, 曹阳, 张盈娇, 等. 单过硫酸氢钾粉对5种水产常见致病菌的体内外抑菌作用[J]. 渔业科学进展, 2014, 35(2): 104-109. ZENG Xiaodan, CAO Yang, ZHANG Yingjiao, et al. Bacteriostasis in vitro and in vivo of potassium monopersulfate powder on five strains of common pathogenic bacteria in aquaculture[J]. Progress in Fishery Sciences, 2014, 35(2): 104-109. DOI:10.3969/j.issn.1000-7075.2014.02.015 |
| [12] |
邵长清, 高勤峰, 董双林, 等. 过碳酸钠对刺参养殖底质改良和生长效果的研究[J]. 海洋湖沼通报, 2018(4): 126-132. SHAO Changqing, GAO Qinfeng, DONG Shuanglin, et al. Effect of sodium percarbonate on growth of sea cucumber (Apostichopus japonicus) and sediment environment[J]. Transactions of Oceanology and Limnoligy, 2018(4): 126-132. |
| [13] |
SOA of China. Specification for Marine Monitoring GB 17378, 4-2007[S]. 2007a.
|
| [14] |
CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al. DADA2:High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [15] |
EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [16] |
CHAO A. Nonparametric estimation of the number of classes in a population[J]. Scandinavian Journal of Statistics, 1984, 11: 265-270. |
| [17] |
SHANNON C E. A mathematical theory of communication[J]. Bell System Technical Journal, 1948, 27(3): 379-423. DOI:10.1002/j.1538-7305.1948.tb01338.x |
| [18] |
SIMPSON E H. Measurement of Diversity[J]. Nature, 1949, 163: 688-688. DOI:10.1038/163688a0 |
| [19] |
LU S, LIAO M, XIE C, et al. Removing ammonium from aquaculture ponds using suspended biocarrierimmobilized ammonia-oxidizing microorganisms[J]. Annals of Microbiology, 2015, 65(4): 2041-2046. DOI:10.1007/s13213-015-1042-0 |
| [20] |
AVNIMELECH Y. Shrimp and fish pond soils: processses and management[J]. Aquaculture, 2003, 220(1/4): 549-567. |
| [21] |
熊代群, 杜晓明, 唐文浩, 等. 海河天津段与河口海域水体氮素分布特征及其与溶解氧的[J]. 环境科学研究, 2005, 18(3): 1-4, 19. XIONG Daiqun, DU Xiaoming, TANG Wenhao, et al. Nitrogen distribution in the water of the Haihe River mainstream and estuary seawater and its relationship with water dissolved oxygen levels[J]. Research of Environmental Sciences, 2005, 18(3): 1-4, 19. DOI:10.3321/j.issn:1001-6929.2005.03.001 |
| [22] |
KHAN N E, ADEWUYI Y G. Absorption and oxidation of nitric oxide (NO) by aqueous solutions of sodium persulfate in a bubble column reactor[J]. Industrial & Engineering Chemistry Research, 2010, 49: 8749-8760. |
| [23] |
BEMAN J M, FRANCIS C A. Diversity of ammonia-oxidizing archaea and bacteria in the sediments of a hypernutrified subtropical estuary: Bahia del Tobari, Mexico[J]. Applied Environmental Microbiology, 2006, 72(12): 7767-7777. DOI:10.1128/AEM.00946-06 |
| [24] |
SAHAN E, MUYZER G. Diversity and spatio-temporal distribution of ammonia-oxidizing Archaea and Bacteria in sediments of the Westerschelde estuary[J]. Fems Microbiology Ecology, 2008, 64(2): 175-186. DOI:10.1111/j.1574-6941.2008.00462.x |
| [25] |
SUN W, XIA C, XU M, et al. Distribution and abundance of archaeal and bacterial ammonia oxidizers in the sediments of the Dongjiang River, a drinking water supply for Hong Kong[J]. Microbes Environments, 2013, 28(4): 457-465. DOI:10.1264/jsme2.ME13066 |
| [26] |
LIU Y, ZHANG J, ZHAO L, et al. Distribution of sediment ammonia-oxidizing microorganisms in plateau freshwater lakes[J]. Applied Microbiology and Biotechnology, 2015, 99: 4435-4444. DOI:10.1007/s00253-014-6341-z |
| [27] |
MAINOUS M E, SMITH S A, KUHN D D. Effect of common aquaculture chemicals against Edwardsiella ictaluri and E. tarda[J]. Journal of Aquatic Animal Health, 2010, 22(4): 224-228. DOI:10.1577/H10-020.1 |
| [28] |
PESTER M, RATTEI T, FLECHL S, et al. amoA-based consensus phylogeny of ammonia-oxidizing archaea and deep sequencing of amoA genes from soils of four different geographic regions[J]. Environmental Microbiology, 2012, 14(2): 525-539. DOI:10.1111/j.1462-2920.2011.02666.x |
| [29] |
ZHENG Y, HOU L, LIU M, et al. Diversity, abundance, and activity of ammonia-oxidizing bacteria and archaea in Chongming eastern intertidal sediments[J]. Applied Microbiology and Biotechnology, 2013, 97: 8351-8363. DOI:10.1007/s00253-012-4512-3 |
| [30] |
YANG Y, ZHANG J, ZHAO Q, et al. Sediment ammonia-oxidizing microorganisms in two plateau freshwater lakes at different trophic states[J]. Microbiology Ecology, 2016, 71(2): 257-265. DOI:10.1007/s00248-015-0642-3 |
| [31] |
LU S, LIU X, MA Z, et al. Vertical segregation and phylogenetic characterization of ammonia-oxidizing bacteria and archaea in the sediment of a freshwater aquaculture pond[J]. Frontiers in Microbiology, 2015, 6: 1539. |
| [32] |
BAO Linlin, WANG Xiaoyan, CHEN Yongjuan. Abundance and distribution of ammonia-oxidizing microorganisms in the sediments of Beiyun River, China[J]. Annals of Microbiology, 2016, 66(3): 1075-1086. DOI:10.1007/s13213-016-1191-9 |
 2021, Vol. 45
2021, Vol. 45


