文章信息
- 马蕊, 阿尔祖古丽·买买提吐尔逊, 张卫红. 2022.
- MA Rui, AERZUGULI Maimaitituerxun, ZHANG Wei-hong. 2022.
- 新疆一种琥珀螺线粒体基因组测序分析
- Complete mitochondrial genome of a succineid species from Xinjiang
- 海洋科学, 46(10): 159-166
- Marine Sciences, 46(10): 159-166.
- http://dx.doi.org/10.11759/hykx20211022001
-
文章历史
- 收稿日期:2021-10-22
- 修回日期:2022-03-07
琥珀螺科Succineidae广泛分布于世界各地, 是陆生软体动物中较原始的类群。该科共分为3个亚科, 琥珀螺亚科Succineinae、原生琥珀螺亚科Catinellinae和尖缘螺亚科Oxylomatinae[1-2]。由于贝壳特征趋同明显、许多属种的解剖特征未知, 该科的种数和分属(亚属)情况目前仍不确定[3]。中国已报道该科2亚科3属27种[4-6], 因为研究基础不足, 一些属种的分类有待厘定。目前, 该科仅有腐败琥珀螺Succinea putris (Linneaus, 1758)、五家渠尖缘螺Oxyloma wujiaquensis Li, Guo & Zhang, 2017和Omalonyx unguis (d’Orbigny, 1836)的线粒体全基因组序列被报道[7-9]。本研究对采自新疆昭苏一种琥珀螺的线粒体全基因组进行了测序分析, 研究结果可以丰富琥珀螺科分子数据, 也可为该种琥珀螺的分类以及我国琥珀螺科系统分类研究提供基础数据。
1 材料与方法 1.1 材料标本(标本号: XJU S13035)于2013年8月14日采自新疆伊犁昭苏县夏特柯尔克孜自治乡东图沟山沟公路边岩石壁上(42°37.096′N, 80°34.880′E), 无水乙醇浸泡, –20 ℃保存于新疆大学生命科学与技术学院陆生贝类实验室。
标本贝壳呈卵圆形, 壳厚, 不透明, 黄棕色, 壳面大部由于角质层脱落呈灰白色, 有3.00~3.25个螺层; 壳高18.00~23.14 mm, 壳宽11.78~17.06 mm; 壳口高13.64~18.64 mm, 壳口宽9.84~12.80 mm。壳顶钝圆。胚螺层光滑, 无螺纹。体螺层增长迅速, 壳面粗糙, 螺纹粗、稀疏。缝合线深。壳口卵圆形, 口缘完整、连续。无脐孔(图 1)。
 |
| 图 1 实验标本生态照片 Fig. 1 An experimental specimen |
剪取腹足肌肉约30 mg, 用动物DNA组织提取试剂盒[天根生化科技(北京)有限公司]提取基因组DNA。DNA样品送至上海天昊生物有限公司进行质量检测和文库构建, 质检合格的文库片段在Illumina Hiseq平台, 用2×150双端测序策略进行序列测定。
1.2.2 序列处理及分析用FASTP软件去除原始序列中的低质量序列, 包括长度低于100 bp, N超过3, 3′端碱基质量低以及高质量碱基(Phred score≥20)占比小于60%的序列。质控后的序列以腐败琥珀螺(Genbank No. JN627206)的线粒体基因组作为参考, 用MetaSPAdes软件输入双端数据进行基因组组装。使用Geneious R11.0软件修正组装结果, 分析碱基组成情况; 使用MITOS Webserver在线平台注释线粒体基因组, 预测tRNA二级结构[10]; 使用CondonW软件分析密码子使用情况; 使用OGDRAW在线平台绘制基因结构图[11]。
从GenBank中选取腹足纲9个物种的线粒体全基因组序列(表 1), 用MEGA X软件进行多序列比对, 选用最佳模型GTR(general time reversible model), 步长检验设定为1 000次, 以新腹足目Neogastropoda, 塔螺科Turridae的Lophinotoma cerithiformis作为外群[7], 基于13个蛋白质编码基因构建最大似然法(maximum likeli-hood, ML)系统发育树[12]。
| 科 | 种 | GenBank登录号 |
| Pleurobranchidae | Berthellina ilisima | DQ991929 |
| Chromodoridae | Chromodoris magnifica | DQ991931 |
| Acteonidae | Pupa strigosa | AB028237 |
| Ellobiidae | Myosotella myosotis | AY345053 |
| Onchidiidae | Onchidlla celtica | AY345048 |
| Succineidae | Succinea putris | JN627206 |
| Succineidae | Oxyloma wujiaquensis | MT670402 |
| Succineidae | Omalonyx unguis | MT449229 |
| Turridae | Lophiotoma cerithiformis | DQ284754 |
实验样本经Illumina Hiseq 2 500测序共获得25 406 852条原始序列, 质控后获得23 358 662条序列, 对其进行拼接获得昭苏琥珀螺一未知种线粒体全基因组(GenBank No. OL681899), 长度14 219 bp, 为一闭合双链DNA分子(图 2)。全基因组由37个基因组成, 包括13个蛋白质编码基因(protein coding genes, PCGs), 22个tRNA基因, 2个rRNA基因和1个控制区。昭苏琥珀螺未知种线粒体全基因组A、T、G、C碱基的含量分别为33.79%、42.64%、12.75%、10.82%, 与琥珀螺科已知3种腐败琥珀螺、五家渠尖缘螺和Omalonyx unguis相似, 均具有明显的AT偏向性(腐败琥珀螺线粒体全基因组长度14 092 bp, AT含量为77.1%、GC含量22.9%; 五家渠尖缘螺线粒体全基因组长度14 086 bp, AT含量为75.6%、GC含量为24.3%; Omalonyx unguis线粒体全基因组长度13 984 bp, AT含量为77.5%、GC含量为22.5%)(表 2)。
 |
| 图 2 昭苏琥珀螺未知种线粒体基因组结构 Fig. 2 Circular map of the mitochondrial genome of the succineid species from Zhaosu |
| 琥珀螺科基因序列 | 全基因组序列 | 蛋白质编码基因 | tRNA基因 | rRNA基因 | H链编码基因 | J链编码基因 | |
| 昭苏琥珀螺未知种 | 长度/bp | 14 219 | 10 415 | 1 373 | 1 759 | 3 279 | 10 498 |
| AT% | 76.43 | 75.13 | 77.64 | 78.59 | 76.54 | 74.86 | |
| GC% | 23.57 | 24.97 | 22.36 | 21.41 | 23.45 | 25.14 | |
| AT偏移 | –0.12 | –0.21 | –0.06 | –0.03 | –0.01 | –0.05 | |
| GC偏移 | 0.08 | 0.08 | 0.12 | 0.09 | 0.11 | 0.03 | |
| 腐败琥珀螺 | 长度/bp | 14 092 | 10 830 | 1 406 | 1 775 | 3 307 | 10 593 |
| AT% | 77.00 | 78.04 | 78.74 | 76.58 | 77.06 | 76.94 | |
| GC% | 23.00 | 21.96 | 21.26 | 23.42 | 22.94 | 23.06 | |
| AT偏移 | –0.11 | –0.21 | –0.04 | –0.04 | –0.11 | –0.19 | |
| GC偏移 | 0.06 | 0.04 | 0.15 | 0.05 | 0.03 | 0.08 | |
| 五家渠尖缘螺 | 长度/bp | 14 086 | 10 713 | 1 554 | 1 819 | 3 242 | 10 526 |
| AT% | 75.64 | 75.41 | 75.96 | 76.75 | 73.97 | 75.90 | |
| GC% | 24.36 | 24.59 | 24.04 | 26.25 | 26.03 | 24.10 | |
| AT偏移 | –0.11 | –0.19 | 0.03 | 0.01 | –0.07 | –0.17 | |
| GC偏移 | 0.04 | 0.04 | 0.05 | 0.13 | 0.04 | 0.06 | |
| Omalonyx unguis | 长度/bp | 13 984 | 10 786 | 1 414 | 1 784 | 3 332 | 10 652 |
| AT% | 77.45 | 76.7 | 78.6 | 80.3 | 76.6 | 77.6 | |
| GC% | 22.55 | 23.3 | 21.4 | 19.7 | 23.4 | 22.4 | |
| AT偏移 | –0.14 | 0.04 | –0.04 | –0.08 | 0.09 | –0.20 | |
| GC偏移 | 0.06 | 0.05 | 0.01 | 0.05 | –0.004 | 0.08 | |
| 注: AT偏移= (A–T) /(A + T); GC偏移= (G–C) /(G + C) | |||||||
昭苏琥珀螺未知种的基因排列顺序与同科的腐败琥珀螺、五家渠尖缘螺和Omalonyx unguis存在差异。腐败琥珀螺、五家渠尖缘螺和Omalonyx unguis的tRNA-Tyr和tRNA-Trp基因位于tRNA-Ser2和tRNA-Ser1之间, 而昭苏琥珀螺未知种的上述两个基因位于COX2和tRNA-Gly之间。
2.2 基因组组成昭苏琥珀螺未知种线粒体基因组序列中存在21处基因间隔, 最长的位于tRNA-Leu2与ATP8之间, 长度141 bp; 存在12处基因重叠, 最长的位于NAD5与NAD1之间, 长度16 bp。基因组中有24个基因位于正链, 13个基因位于负链(表 3)。
| 基因 | 位置 | 长度/bp | 方向 | 起始密码子 | 终止密码子 | 反密码子 | 间隔或重叠/bp |
| COX1 | 33-1 560 | 1 528 | + | TTG | TAA | 0 | |
| tRNA-Val | 1 561-1 631 | 71 | + | TAC | 67 | ||
| 16S rRNA | 1 699-2 679 | 981 | + | –5 | |||
| tRNA-Pro | 2 675-2 738 | 64 | + | TGG | 2 | ||
| tRNA-Leu | 2 741-2 806 | 66 | + | TAG | 9 | ||
| tRNA-Ala | 2 816-2 885 | 70 | + | TGC | 5 | ||
| NAD6 | 2 891-3 343 | 493 | + | ATT | TAG | 10 | |
| NAD5 | 3 354-5 015 | 1 662 | + | ATT | TAA | –16 | |
| NAD1 | 5 000-5 899 | 900 | + | TTA | TAA | 2 | |
| NAD4L | 5 902-6 178 | 277 | + | ATA | TAA | 0 | |
| CYTB | 6 179-7 276 | 1 098 | + | ATG | TAA | 0 | |
| tRNA-Phe | 7 277-7 345 | 69 | + | GAA | 4 | ||
| tRNA-Asp | 7 350-7 410 | 60 | + | GTC | 0 | ||
| tRNA-Cys | 7 411-7 476 | 66 | + | GCA | 8 | ||
| COX2 | 7 485-8 133 | 649 | + | ATG | TAA | 0 | |
| tRNA-Tyr | 8 134-8 199 | 66 | + | GTA | –7 | ||
| tRNA-Trp | 8 193-8 265 | 73 | + | TCA | 3 | ||
| tRNA-Gly | 8 269-8 334 | 66 | + | TCC | –13 | ||
| tRNA-His | 8 322-8 385 | 64 | + | GTG | 19 | ||
| tRNA-Gln | 8 405-8 466 | 62 | – | TTG | –9 | ||
| tRNA-Leu2 | 8 458-8 522 | 65 | – | TAA | 13 | ||
| ATP8 | 8 536-8 658 | 123 | – | ATG | TAA | 4 | |
| tRNA-Asn | 8 663-8 728 | 66 | – | TCG | 19 | ||
| ATP6 | 8 748-9 404 | 657 | – | ATG | TAA | 20 | |
| tRNA-Arg | 9 425-9 488 | 64 | – | TCG | 5 | ||
| tRNA-Glu | 9 494-9 559 | 66 | – | TTC | 0 | ||
| 12SrRNA | 9 560-10 339 | 780 | – | 4 | |||
| tRNA-Met | 10 344-10 413 | 70 | – | CAT | 2 | ||
| NAD3 | 10 416-10 775 | 360 | – | ATA | TAA | 12 | |
| tRNA-Ser2 | 10 788-10 850 | 63 | – | TGA | 18 | ||
| tRNA-Ser1 | 10 869-10 928 | 60 | + | GCT | 14 | ||
| NAD4 | 10 943-12 254 | 1 312 | + | ATG | TAA | –4 | |
| tRNA-Thr | 12 251-12 323 | 73 | – | TGT | 2 | ||
| COX3 | 12 326-13 105 | 780 | – | ATA | T(AA) | 54 | |
| tRNA-Ile | 13 160-13 230 | 71 | + | GAT | 18 | ||
| NAD2 | 13 249-14 175 | 927 | + | ATT | TAA | 15 | |
| tRNA-Lys | 14 191-32 | 61 | + | TTT | 0 |
昭苏琥珀螺未知种13个蛋白质编码基因长度在132 bp到1 635 bp之间, 总长10 415 bp, 其中AT含量为75.13%, AT偏移值为–0.21, GC值偏移值为0.08; 腐败琥珀螺13个蛋白质编码基因全长10 830 bp, AT含量为78.04%, AT偏移值为–0.21, GC偏移值为0.04; 五家渠尖缘螺13个蛋白质编码基因全长10 678 bp, AT含量为75.64%, AT偏移值为–0.11, GC偏移值为0.04; 三者蛋白质编码基因长度和AT含量差异不大, 但五家渠尖缘螺AT偏移值较小。
昭苏琥珀螺未知种13个蛋白质编码基因中除了NAD1和COX1分别以TAA、TTG作为起始密码子, 其余11个基因的为典型起始密码子ATN; 除了NAD6和COX3分别以TAG、T作为终止密码子, 其余11个基因的终止密码子为TAA。昭苏琥珀螺未知种起始密码子的使用情况除CYTB和NAD1之外, 其余11个基因与腐败琥珀螺和五家渠尖缘螺的相似; 终止密码子的使用也与二者存在一定差异, 主要出现在NAD6、NAD1、NAD4L和COX3上。
昭苏琥珀螺未知种共编码3 741个密码子, 其中密码子AGA(Arg)、UUA(Leu)、AGU(Ser)等的使用次数较高(图 3)。腐败琥珀螺共编码3 583个密码子, 五家渠尖缘螺共编码3 571个密码子, Omalonyx unguis共编码3 607个密码子, 三者使用次数较高的密码子均为UUA (Leu)、UUU(Phe)、AUU(Ile), 与昭苏琥珀螺未知种有所不同。编码密码子数均不含终止密码子。
 |
| 图 3 昭苏琥珀螺未知种蛋白编码基因同义密码子使用频率 Fig. 3 The relative synonymous codon usage of the PCGs for the succineid species from Zhaosu |
昭苏琥珀螺未知种线粒体基因组中22个tRNA基因总长为1 373 bp, 各基因长度在60 bp到73 bp之间, 其中AT含量为77.64%, GC含量为22.36%。tRNA基因二级结构中, tRNA-Ser1和tRNA-Ser2基因缺少DHU臂, tRNA-Ser1基因在缺失的DHU臂处有一小茎环结构, 其他基因均形成经典的三叶草结构。大多数后生动物线粒体基因组中tRNA-Ser1基因缺DHU臂[13-14]。本文研究的4种琥珀螺中, 昭苏琥珀螺未知种、腐败琥珀螺和五家渠尖缘螺的tRNA-Ser1和tRNA-Ser2基因均缺少DHU臂, 而Omalonyx unguis仅tRNA-Ser2缺少DHU臂(图 4), 与大多数后生动物有所不同。
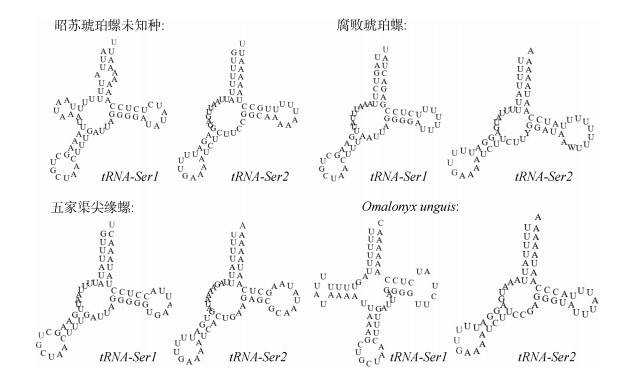 |
| 图 4 四种琥珀螺线粒体基因组tRNA-Ser二级结构预测图 Fig. 4 Predicted secondary structures of tRNA-Ser genes in the mitochondrial genome of four species of Succineidea |
昭苏琥珀螺未知种线粒体基因组中rRNA基因总长为1 761 bp, AT含量为78.59%。16S rRNA(981 bp)位于tRNA-Val和tRNA-Pro之间, 12S rRNA(780 bp)位于tRNA-Glu和tRNA-Met之间, 基因位置与同科3个已知种一致。昭苏琥珀螺未知种线粒体基因组中有一段长度为54 bp的控制区, 位于COX3与tRNA-Ile之间; 腐败琥珀螺中控制区长度50 bp位于NAD3与tRNA-Ser之间; 五家渠尖缘螺中控制区长度42 bp, 位于COX1和tRNA-Val之间, 三者有所不同。
2.5 系统发育树系统发生树(图 5)结果显示, 后鳃亚纲的种类聚为一支, 为单系群; 肺螺亚纲为非单系群, 与White等获得的系统发育分析结果一致[7]。琥珀螺科的四个种聚为一支, 显示共同的起源, 其中腐败琥珀螺与Omalonyx unguis的亲缘关系最近, 而昭苏琥珀螺未知种位于琥珀螺科所在分支的最基部, 关于它们的系统演化关系有待加入琥珀螺科更多种类的线粒体基因组数据进行分析。同源性比对结果显示, 昭苏琥珀螺未知种与五家渠尖缘螺线粒体基因组的相似性为75.8%, 与腐败琥珀螺的相似性为77.4%, 与Omalonyx unguis的相似性为78.7%, Omalonyx unguis与腐败琥珀螺的相似性为80.8%, 腐败琥珀螺与Omalonyx unguis的相似性最高, 与系统发育树结果一致。
 |
| 图 5 基于线粒体基因组蛋白质编码基因构建的最大似然树 Fig. 5 Maximum likelihood tree based on the protein-coding gene sequences of the mitochondrial genome |
昭苏琥珀螺未知种目前已知仅在新疆昭苏县夏特乡有分布, 生境为较干燥的山地环境。琥珀螺科的种类贝壳一般较小, 壳高大多1 cm左右。昭苏琥珀螺未知种贝壳较大, 壳高2 cm左右。杨吕娟在其硕士论文中对本文研究的昭苏琥珀螺未知种的形态解剖结构进行了描述, 认为其阴茎结构与琥珀螺科已知属种存在明显差异, 将其定为一新属新种Rosuccinea zhaosuensis[15]。阿尔祖古丽·买买提吐尔逊在其硕士论文中进一步依据COI、ITS1和ITS2基因的分析, 支持杨吕娟的结果[16]。
琥珀螺科目前已有3个种的线粒体全基因组被报道。本研究昭苏琥珀螺未知种的线粒体全基因组与三者相比差异较大。主要差异在于, 第一, 线粒体基因组基因排列顺序不同, 昭苏琥珀螺未知种的tRNA-Tyr和tRNA-Trp基因位于COX2和tRNA-Gly之间, 而腐败琥珀螺、五家渠尖缘螺和Omalonyx unguis的tRNA-Tyr和tRNA-Trp基因位于tRNA-Ser2和tRNA-Ser1之间; 第二, tRNA-Ser1基因二级结构存在差异, 昭苏琥珀螺未知种在tRNA-Ser1基因缺失的DHU臂处有一小茎环结构。本研究系统发育树结果也显示, 昭苏琥珀螺未知种位于琥珀螺科所在分支的最基部, 未与其他三种琥珀螺中的任何一种单独聚为一支, 应当不属于这三种所在的属, 即不属于Oxyloma Westerlund, 1885、Succinea Draparnaud, 1801和Omalonyx d’Orbigny, 1838(该属仅分布于美洲和加勒比群岛)。我国琥珀螺科共报道三个属, Neosuccinea Matekin, 1956、Oxyloma和Succinea, 其中Neosuccinea在新疆没有分布[4-6]。结合形态学数据, 昭苏琥珀螺未知种也不属于Neosuccinea属, 应为中国一未报道的属种。因为杨吕娟和阿尔祖古丽·买买提吐尔逊的研究结果尚未正式发表, 目前琥珀螺科线粒体全基因以及其他分子数据有限, 本文仍将所研究采自昭苏的蜗牛定为琥珀螺科一未知种。
4 结论本研究获得新疆昭苏琥珀螺未知种线粒体全基因组序列, 对其碱基组成、基因排列顺序、各类基因的结构特征等进行了分析, 并与琥珀螺科已知3个种的线粒体全基因组序列进行了比较。结果显示, 四者基因组长度、碱基含量相似; 基因位置、蛋白质编码基因密码子使用情况、tRNA基因二级结构、控制区的位置等存在一定差异, 其中昭苏琥珀螺未知种与其他三种的差异较大。昭苏琥珀螺未知种应为中国一未报道的属种, 但要确定其是否为一新属新种尚需更多的分子和形态数据。
| [1] |
PATTERSON C M. Cytogenetics of gastropod mollusks[J]. Malacological Review, 1973, 6: 141-150. |
| [2] |
SCHIEYKO A A, LIKHAREV I M. Fauna, sistematika i filogeniya bespozvonochnykh zhivotnykh[M]. Moscow: Moskovskogo universiteta, 1986: 197-239.
|
| [3] |
SCHILEYKO A A. Treatise on recent terrestrial pulmonate mollusks[M]. Moscow: Ruthenica, 2007: 2095-2099.
|
| [4] |
陈德牛, 高家祥. 中国琥珀螺属Succinea的研究[J]. 四川动物, 1985, 4(1): 34-37. CHEN Deniu, GAO Jiaxiang. Studies on the genus of Chinese succinea[J]. Sichuan Journal of Zoology, 1985, 4(1): 34-37. |
| [5] |
ZHANG W H. Towards a revision of Chinese Succineidae: Comparative morphology two Succinea species from Xinjiang, northwestern China[J]. Molluscan Research, 2015, 35(2): 102-111. DOI:10.1080/13235818.2015.1023172 |
| [6] |
李成有, 郭阳阳, 张卫红. 中国尖缘螺属一新种记述(柄眼目, 琥珀螺科)[J]. 海洋科学, 2017, 41(11): 130-132. LI Chengyou, GUO Yangyang, ZHANG Weihong. A new species of Oxyloma from Xinjiang (Stylommatophora: Succineidae)[J]. Marine Sciences, 2017, 41(11): 130-132. |
| [7] |
WHITE T R, CONRAD M M, TSENG R, et al. Ten new complete mitochondrial genomes of pulmonates (Mollusca: Gastropoda) and their impact on phylogenetic relationships[J]. BMC Evolutionary Biology, 2011, 11(1): 1-15. DOI:10.1186/1471-2148-11-1 |
| [8] |
阿尔祖古丽·买买提吐尔逊, 张子俊, 张卫红. 五家渠尖缘螺线粒体基因组测序分析[J]. 华北农学报, 2021, 36(3): 230-238. AERZUGULI Maimaitituerxun, ZHANG Zijun, ZHANG Weihong. Complete mitochondrial genome sequence of Oxyloma wujiaquensis Li, Guo & Zhang, 2017(Gastropoda: Succineidae)[J]. Acta Agriculturae Boreali-Sinica, 2021, 36(3): 230-238. |
| [9] |
GUZMAN L B, VOGLER R E, BELTRAMINO A A. The mitochondrial genome of the semi-slug Omalonyx unguis (Gastropoda: Succineidae) and the phylogenetic relationships within Stylommatophora[J]. PloS One, 2021, 16(6): 1-22. |
| [10] |
MATTHIAS B, ALEXANDER D, FRANK J, et al. MITOS: Improved de novo metazoan mitochondrial genome annotation[J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 313-319. DOI:10.1016/j.ympev.2012.08.023 |
| [11] |
MARC L, OLIVER D, RALPH B. OrganellarGenomeDRAW (OGDRAW): a tool for the easy generation of high-quality custom graphical maps of plastid and mitochondrial genomes[J]. Current Genetics, 2007, 52(5/6): 267-274. |
| [12] |
KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular BIOLOGY and Evolution, 2018, 35(6): 1547-1549. |
| [13] |
WANG Y Z, JIN G H, ZHU J Y, et al. The mitochondrial genome of the garden pea leafminer Chromatomyia horticola (Goureau, 1851) (Diptera: Agromyzidae)[J]. Mitochondrial DNA Part A, DNA Mapping, Sequencing, and Analysis, 2016, 27(4): 2653-2655. |
| [14] |
LAVROV D V, BROWN W M, BOORE J L. A novel type of RNA editing occurs in the mitochondrial tRNAs of the centipede Lithobius forficatus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(25): 13738-13742. |
| [15] |
杨吕娟. 新疆山地环境琥珀螺比较形态解剖学研究[D]. 乌鲁木齐: 新疆大学, 2019. YANG lvjuan. Comparative morphology and anatomy on Succineidae in mountain environment from Xinjiang[D]. Urumqi: Xinjiang University, 2019. |
| [16] |
阿尔祖古丽·买买提吐尔逊. 新疆琥珀螺科分子系统学研究[D]. 乌鲁木齐: 新疆大学, 2020. AERZUGULI Maimaitituerxun. The molecular phylogeny of Succineidae from Xinjiang[D]. Urumqi: Xinjiang University, 2020. |
 2022, Vol. 46
2022, Vol. 46


