
文章信息
- 刘英, 王全超, 李海汇, 纪莹璐, 陈琳琳, 李宝泉. 2022.
- LIU Ying, WANG Quan-chao, LI Hai-hui, JI Ying-lu, CHEN Lin-lin, LI Bao-quan. 2022.
- 小清河河口邻近海域口虾蛄(Oratosquilla oratoria)COI序列特征及遗传多样性分析
- Analysis of the COI sequence characteristics and genetic diversity of Oratosquilla oratoria in the Xiaoqing River Estuary and its adjacent sea
- 海洋科学, 46(11): 38-46
- Marine Sciences, 46(11): 38-46.
- http://dx.doi.org/10.11759/hykx20210618001
-
文章历史
- 收稿日期:2021-06-18
- 修回日期:2021-07-06
2. 中国科学院大学, 北京 100049;
3. 寿光市渔业发展中心, 山东 寿光 262700;
4. 国家海洋局北海预报中心, 山东 青岛 266061
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Shouguang Marine Fishery Development Center, Shouguang 262700, China;
4. North China Sea Marine Forecasting Center of State Oceanic Administration, Qingdao 266061, China
口虾蛄(Oratosquilla oratoria), 俗称爬虾、虾爬子, 隶属于节肢动物门(Arthropoda)、甲壳纲(Crustacea)、口足目(Stomatopoda)、虾蛄科(Squillidae), 自然分布于西太平洋温热带浅水海区, 在中国沿海均广泛分布, 其中以黄渤海产量最大[1]。口虾蛄栖息于潮下带泥沙质海底, 其食性主要以贝类及甲壳类为主[2]。口虾蛄具有个体大、产量高、繁殖周期短、易捕捞等优点, 使其具有很高的经济价值, 是中国沿海重要的海产经济种类, 同时其肉质鲜美、营养价值高, 沿海民众喜食。因此, 口虾蛄多年来均作为近海渔业主要捕捞对象之一[3]。特别在渤海, 口虾蛄已成为支柱渔业之一[4]。但近年来, 由于全球气候变化、环境污染、养殖疾病危害以及过度捕捞等导致口虾蛄野生资源急剧减少[5], 并进而影响或降低口虾蛄野生种群的遗传多样性。
鉴于口虾蛄重要的经济价值, 国内对该物种已开展了较多研究, 涉及到生物学特征[6-8]、生理生态[9]、渔业资源[10-11]和人工繁育[12-13]等方面。随着分子生物学技术发展以及口虾蛄资源管理和种质资源保护的需要, 对口虾蛄种群遗传多样性的研究也日渐增多。刘海映等[14]基于随机扩增多态性DNA标记(random amplified polymorphic DNA, RAPD)分析了大连海域口虾蛄遗传多样性; Liu等[15]报道了口虾蛄线粒体全长序列, 自此关于利用线粒体基因作为遗传标记研究其种群遗传多样性的报道也日渐增多; Zhang等[16]使用线粒体ND5基因作为遗传标记分析了口虾蛄在中国海域的地理分布、扩散与遗传差异; Du等[17]以16S rRNA作为遗传标记分析口虾蛄在渤海、黄海和东海的遗传多样性; 隋宥珍等[18]基于细胞色素b(cytochrome b, Cyt b)基因分析了口虾蛄黄渤海种群、东海种群、南海种群的遗传多样性; 隋宥珍等[19]基于细胞色素氧化酶亚基I(cytochrome oxidase subunit I, COI)基因分析了东海海域口虾蛄遗传多样性。上述研究基本摸清了中国不同海域口虾蛄种群的遗传多样性现状和不同地理种群的遗传分化。但对个别区域, 尤其是具有重要生态意义的河口区如小清河河口, 口虾蛄种群的遗传多样性研究迄今还尚未详细开展。
小清河河口及邻近海域曾经是渤海莱州湾重要的渔业区, 是渤海生态系统中渔业生物的产卵场与索饵场。小清河是典型受污染河流之一, 沿河污染物随河流入海, 使河口及邻近海域污染加重[20-22], 且导致河口内出现低氧区[23]。小清河河口及邻近海域的污染加重以及不断增大的捕捞强度, 必然导致该区口虾蛄种群数量的维持和生存受到影响。因此有必要对口虾蛄开展遗传多样性研究, 摸清该海域口虾蛄的遗传分化潜力, 以维持资源生产力。线粒体DNA(mitochondrial DNA, mtDNA)为母系遗传, 基因结构简单, 且容易提取扩增, 是研究海洋生物遗传多样性的重要手段。COI基因是线粒体基因的重要组成部分, 具有结构简单、进化速率适中、具有较高的多态性以及易被通用引物扩增的优势, 已被广泛用于种群遗传多样性研究。本研究采集了小清河河口及邻近海域口虾蛄生物样本, 基于口虾蛄种群COI基因标记研究其遗传多样性, 评估小清河河口及邻近海域口虾蛄种质资源现状, 为该种群健康维持、资源合理开发利用以及相关部门管理决策的制定提供理论性参考。
1 材料与方法 1.1 材料2020年6—10月于小清河河口及其邻近海域(表 1), 采用渔业拖网方式, 采集口虾蛄生物样品。随机选取部分样品放于体积分数95%的乙醇中暂存并转移至实验室备用。
| 站位号 | 经度/°E | 纬度/°N |
| 1 | 119.018 2 | 37.352 0 |
| 2 | 119.071 4 | 37.310 3 |
| 3 | 119.119 0 | 37.276 2 |
| 4 | 119.161 3 | 37.243 4 |
| 5 | 119.092 5 | 37.366 1 |
| 6 | 119.148 1 | 37.329 2 |
| 7 | 119.194 4 | 37.295 3 |
| 8 | 119.241 2 | 37.263 7 |
| 9 | 119.168 7 | 37.379 9 |
| 10 | 119.224 4 | 37.348 6 |
| 11 | 119.266 4 | 37.324 6 |
| 12 | 119.293 6 | 37.401 8 |
取口虾蛄背部肌肉, 使用海洋动物组织基因组DNA提取试剂盒[天根生化科技(北京)有限公司]按照操作说明书提取每个口虾蛄个体总DNA。用NanoDrop 1000分光光度计(NanoDrop, Wilmington, DE, USA)测定提取DNA的纯度和完整性, 并进行1%琼脂糖凝胶电泳。合格DNA放于–20 ℃暂存备用。
COI基因片段扩增引物为通用引物, 引物序列: LCO1490: 5′-GGTCAACAAATCATAAAGATATTGG-3′; HCO2198: 5′-TAAACTTCAGGGTGACCAAAAAATCA-3′[24]。PCR扩增在50 μL反应体系中进行。50 μL反应体系包括: 25 μL含DNA聚合酶反应混合物[天根生化科技(北京)有限公司], 17 μL ddH2O, 上游引物和下游引物各2 μL(100 μmol/L)及模板DNA 4 μL。PCR扩增按照以下热循环程序进行: 94 ℃初步变性5 min; 随后94 ℃变性45 s, 40 ℃退火45 s, 72 ℃延伸30 s, 进行35个循环; 最后在72 ℃下延伸10 min, 10 ℃保存。PCR产物经1%琼脂糖凝胶电泳检验合格后, 送测序公司[生工生物工程(上海)股份有限公司]进行双向测序。
1.3 数据分析通过BioEdit软件对获得的序列进行可视化、修剪、编辑、拼接以及人工校对, 获得完整无误的序列, 并在NCBI(https://www.ncbi.nlm.nih.gov/)上进行复检。在MEGA 7.0.26进行CLUSTAL比对和序列碱基组成及变异分析。采用DAMEB 7软件分析COI基因序列的碱基替换饱和度。使用DNAsp v6.12.03计算多态位点(S)、总变异位点(η)、单倍型数量(h)、单倍型多样性指数(Hd)、遗传多样性指数(π)、平均核苷酸差异数(K)等遗传多样性参数, 分析种群遗传多样性。
为明确小清河河口及邻近海域口虾蛄种群遗传结构现状, 采用从GenBank上额外下载的唐山、青岛、舟山和广州口虾蛄种群的COI序列, 进行系统发育树构建和遗传结构分析。COI序列包括唐山种群(GenBank登录号: MF173560-MF173566)、青岛种群(GenBank登录号: MF173526-MF173534)、舟山种群(GenBank登录号: MF173576-MF173598)[25]以及广州种群(GenBank登录号: HQ621811-HQ621829)[26]。分子系统发育树的重建是在MEGA 7.0.26中使用基于Kimura双参数法模型(Kimura 2-parameter)遗传距离的非加权分组平均法(unweighted pair group method using arithmetic average, UPGMA)进行, 并选用bootstrap法进行1 000次重复自举检验, 评估系统发育树。遗传结构分析是利用Arlequin 3.5.2.2软件开展分子方差分析(AMOVA)和遗传分化系数FST计算。
2 结果 2.1 小清河河口及邻近海域口虾蛄COI序列获得序列后经剪切、人工校正及NCBI复检后, 共获得216条长度为655 bp的口虾蛄COI基因部分序列片段。序列片段无插入和缺失。所有序列A、G、C、T的平均含量分别为36.6%、17.1%、18.4%和27.9%, A+T含量明显高于G+C含量。4种碱基在密码子第1位上分布较为均匀(A=26.48%, G=32.96%, C=16.90%, T=24.65%), 第2位上T的含量较高(A=13.30%, G=18.81%, C=25.23%, T=42.66%), 第3位上呈现G和C的含量较低的偏倚现象(A=43.83%, G=4.32%, C=9.22%, T=42.63%)。
所有序列共发现66个变异位点, 其中63个多态位点, 由30个单一突变位点和33个简约信息位点组成。63个多态位点中包括59个转换位点, 1个颠换位点和3个转换与颠换同时存在的位点。4个变异位点发生在密码子的第1位, 59个变异位点发生在第3位, 无变异位点发生在第2位。655 bp的COI基因序列共定义218个氨基酸, 且碱基的替换并未导致氨基酸序列发生变化。
碱基的替换饱和分析见图 1, 成对碱基的转换率、颠换率都与遗传距离成线性相关, 其中, 94.18%的碱基转换和5.84%的碱基颠换能分别得到相关线性分析。COI序列碱基替换饱和度分析结果也表明, 标准碱基替换饱和值(Iss.c)显著大于实际检测到的COI基因碱基替换值(Iss)(P < 0.05)。
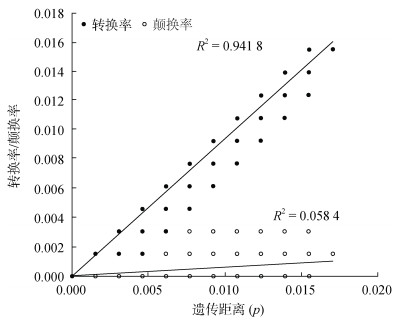 |
| 图 1 小清河河口口虾蛄种群COI基因碱基替换饱和分析 Fig. 1 Substitution saturation analysis of the COI fragments in the O. oratoria population at Xiaoqing River Estuary |
该海域口虾蛄种群Hd为0.970 0, π为0.005 1, K为3.330 0。所有序列共定义90个单倍型, 其中, 单倍型Hap-1、Hap-16、和Hap-27分别为20、19和15个个体所有, 是优势单倍型。
本研究口虾蛄种群单倍型多样性与粤东海种群、东海种群相当, 略高于渤海湾种群、皮口种群、绥中种群和青岛种群; 与Zhu等[25]研究的唐山种群、龙口种群、青岛种群、连云港种群以及七星列岛种群单倍型多样性相当, 但略高于东营种群、小洋山种群、舟山种群和三门种群; 核苷酸多样性低于粤东海种群, 东海种群, 高于渤海湾种群、辽东湾种群和青岛种群, 低于Zhu等[25]研究中的青岛种群、连云港种群和七星列岛种群, 高于唐山种群、小洋山种群和三门种群(表 2)。
| 海域 | 片段长度 | 单倍型多样性指数 | 核苷酸多样性指数 | 参考文献 |
| 皮口 | 585 | 0.878 3 | 0.003 2 | 董鑫等[27] |
| 绥中 | 585 | 0.896 8 | 0.003 9 | |
| 青岛 | 585 | 0.906 0 | 0.003 9 | |
| 广州 | 585 | 0.933 8 | 0.004 9 | |
| 东海 | 658 | 0.963 0 | 0.005 9 | 隋宥珍等[19] |
| 粤东海 | 592 | 0.971 0 | 0.007 6 | 黄映萍等[26] |
| 渤海湾 | 633 | 0.947 0 | 0.004 9 | 张代臻等[28] |
| 唐山 | 658 | 0.963 8 | 0.004 1 | Zhu等[25] |
| 东营 | 658 | 0.926 4 | 0.005 2 | |
| 龙口 | 658 | 0.978 0 | 0.005 1 | |
| 青岛 | 658 | 0.980 4 | 0.006 0 | |
| 连云港 | 658 | 0.978 4 | 0.005 8 | |
| 小洋山 | 658 | 0.934 6 | 0.004 7 | |
| 舟山 | 658 | 0.935 7 | 0.005 4 | |
| 三门 | 658 | 0.866 7 | 0.004 4 | |
| 七星列岛 | 658 | 0.971 4 | 0.006 0 |
小清河河口口虾蛄种群与唐山种群、青岛种群、舟山种群以及广州种群之间的亲缘关系和遗传结构见图 2。分子系统发育树显示所有单倍型聚为两支: 小清河河口种群首先与青岛种群聚集在一起, 然后再与唐山种群汇合, 组成一支, 广州种群和舟山种群组成则组成另一支。
 |
| 图 2 基于5个口虾蛄种群COI单倍型序列遗传距离构建的UPGMA系统发育树 Fig. 2 Unweighted pair group method using arithmetic average (UPGMA) tree based on the genetic distance of COI haplotypes in 5 O. oratoria populations |
AMOVA分析结果显示, 5个口虾蛄种群遗传变异主要来自于种群间, 且5个口虾蛄种群间具有显著的遗传分化水平(FST=0.857 38, P < 0.05)(表 3)。不同种群间成对FST范围在–0.020 62~0.900 91之间(表 4)。结果表明, 唐山种群、小清河河口种群及青岛种群与舟山种群和广州种群间存在显著遗传分化(P < 0.05), 但唐山种群、小清河河口种群和青岛种群间不存在显著遗传分化(P > 0.05), 广州种群和舟山种群间不存在显著遗传分化(P > 0.05)。
| 变异来源 | 自由度 | 平方和 | 变异组分 | 变异百分数 | 遗传分化系数 |
| 种群间 | 4 | 927.577 | 11.273 90 | 85.74 | 0.857 38(P<0.05) |
| 种群内 | 139 | 260.680 | 1.875 40 | 14.26 | |
| 总计 | 143 | 1 188.257 | 13.149 29 | ||
| 注: P<0.05差异显著 | |||||
| 广州种群 | 小清河河口种群 | 青岛种群 | 唐山种群 | |
| 小清河河口种群 | 0.901 73* | |||
| 青岛种群 | 0.903 07* | –0.020 67 | ||
| 唐山种群 | 0.887 70* | 0.003 64 | 0.038 78 | |
| 舟山种群 | –0.006 13 | 0.896 01* | 0.885 28* | 0.872 59* |
| 注: *表示种群间差异显著, P<0.05差异显著 | ||||
遗传多样性是生物多样性的基础, 开展遗传多样性的研究, 对于渔业资源管理的可持续利用以及渔业资源的管理和保护都具有重要意义[29]。一般而言, 遗传多样性水平高的物种, 具有较强的环境适应能力、生存能力和进化潜力。因此, 物种遗传多样性一旦降低, 可导致物种适应及生存能力降低、物种退化甚至灭绝[30]。
物种在基因水平上的遗传变异最直接的表现就是碱基组成的差异。目前已观测到的无脊椎动物线粒体基因组中碱基组成基本规律均为A+T含量显著高于G+C含量[31]。本研究中口虾蛄种群的COI序列碱基组成A+T含量高于G+C含量, 该结果与三疣梭子蟹(Portunus trituberculatus)[32]、脊尾白虾(Exopalaemon carinicauda)[33]、芋螺[34]的COI基因片段研究结果相似。本研究中, 口虾蛄的COI序列碱基转换与颠换之比为9, 远大于基因变异达到饱和状态的临界值2[35], 说明小清河河口的COI序列变异还未达到饱和状态。碱基转换数大于颠换数, 符合在动物线粒体进化过程中碱基的替换主要以转换为主的结论[36]。并且一般在线粒体DNA进化过程中, 亲缘关系较近物种间, 转换更容易频繁发生, 而颠换则在亲缘关系较远的物种间更容易发生。同一物种内部, 转换数量高于颠换数量[37]。同时, COI基因片段的碱基饱和性分析显示小清河河口口虾蛄种群实际碱基替换值显著低于期望碱基替换饱和值, 表明COI基因碱基替换未达到饱和状态。基于COI基因未校正的转换、颠换与遗传距离的散点分布图得到的线性关系也表明小清河河口口虾蛄种群的COI基因序列未达到饱和状态, 因此, 线粒体COI基因适合用作研究口虾蛄群体遗传多样性的分子标记。
在所有碱基变异中, 有59个变异位点发生在密码子的第3位上, 仅有4个变异位点发生在密码子的第1位上, 无变异位点发生在密码子的第2位上。可见, 密码子第3位的变异率明显高于第1位和第2位, 这与密码子第1位相对保守、第2位非常保守以及第3位具有摇摆性的特点相符, 这与某些鱼类研究结果一致[38-39]。同时本结果检测到的所有核苷酸变异均为同义突变。这进一步说明密码子简并性大多发生在密码子第3位上且在物种稳定上起着重要作用。
Grant和Bowen[40]根据线粒体基因序列的单倍型多样性和核苷酸多样性, 将海洋生物的遗传多样性分为4种类型: 第1类, 低单倍型多样性(Hd > 0.5), 低核苷酸多样性(π < 0.005)型, 近期种群瓶颈或是由单一、少数种群所产生的建立者效应; 第2类, 高单倍型多样性(Hd > 0.5), 低核苷酸多样性(π < 0.005)型, 种群瓶颈, 随后是种群快速增长和突变积累; 第3类, 低单倍型多样性(Hd < 0.5), 高核苷酸多样性(π > 0.005)型, 种群差异, 这种差异可能来自孤立种群间的二次接触或者是一个大而稳定的种群中发生过瓶颈效应; 第4类, 高单倍型多样性(Hd > 0.5), 高核苷酸多样性(π > 0.005)型, 种群由一个大而稳定的种群经过长时间演化所产生, 或是两个不同系群的种群二次接触产生。本研究口虾蛄种群总的单倍型多样性Hd为0.970 0, 核苷酸多样性π为0.005 1, 属于第4类种群状态, 即种群属于高单倍型多样性和高核苷酸多样性模式, 种群遗传多样性较高, 处于长期稳定状态。本研究口虾蛄种群单倍型多样性和核苷酸多样性在中国沿海多数海域中处于相对较高水平, 这说明小清河河口口虾蛄种群的遗传多样性在我国沿海处于相对较高的水平, 可考虑作为种质资源选育及建立种质资源库的选择之一。
种群遗传结构受地理隔离、洋流、生境、人类活动、迁移和历史因素等多种因素的影响[41-42]。唐山种群、小清河河口种群和青岛种群位于黄渤海, 这3个种群间两两相互聚为姐妹群, AMOVA和FST分析也显示3个种群间没有显著遗传分化, 表明3个种群间极可能存在基因交流, 而且小清河河口种群与青岛种群交流更为频繁。一方面在黄渤海水交换过程中, 黄海沿岸流流动方向基本终年不变, 沿山东半岛北岸从渤海进入北黄海, 然后南下到达南黄海[43], 另一方面口虾蛄具有大约2个月的幼虫浮游期。因此, 口虾蛄幼体可在海洋环流的驱动下发生扩散, 从而维持口虾蛄种群连通性[44]。此外, 本研究中AOMVA、FST和分子系统发育树也均显示以长江为界, 长江口以北口虾蛄种群和长江口以南的种群间存在显著的遗传分化。但目前, 关于中国沿海口虾蛄种群遗传结构尚未得到统一。Cheng和Sha[45]通过线粒体COI基因标记和核基因ITS标记分析认为, 在长江入海口两侧存在两个口虾蛄亚种。一些学者分别通过对16S rRNA和COI分析, 认为东海和黄海的口虾蛄种群的遗传分化的原因可能是由长江淡水的注入形成的物理屏障和局部适应造成[17, 25]。也有学者通过以线粒体ND5基因和核基因H3为遗传标记, 认为口虾蛄在冰川时期受台湾海峡分隔, 长期隔离形成了南海种群孤立于其他海域种群的一个显著的南北种群结构[16]。因此未来对中国沿海口虾蛄种群的遗传研究中, 在选择遗传标记时, 应综合考虑遗传标记的进化速度、多态性、遗传信息的全面性等。
4 结论与展望口虾蛄作为小清河河口及邻近海域常年优势种之一, 面临着巨大的捕捞和生存压力。研究结果表明小清河河口及邻近海域口虾蛄种群遗传多样性目前还处于较高水平, 为以后口虾蛄的苗种选育、野生种质资源保护和合理开发利用提供了新的依据。为了维持该海域口虾蛄种群的遗传多样性水平, 建议加强口虾蛄资源保护, 基于现有资源量的科学评估, 进行合理采捕, 如控制网具网目大小, 减小网目尺寸, 以利于口虾蛄幼体的存活和自然种群的延续, 保障口虾蛄野生资源可持续利用。同时, 由于口虾蛄在4月下旬便进入繁殖期, 而目前休渔期在5月1日至9月1日, 有必要考虑适当延长休渔时间至4月下旬。
| [1] |
吴强, 陈瑞盛, 黄经献, 等. 莱州湾口虾蛄的生物学特征与时空分布[J]. 水产学报, 2015, 39(8): 1166-1177. WU Qiang, CHEN Ruisheng, HUANG Jingxian, et al. Fishery biology characteristics, temporal and spatial distribution of Oratosquilla oratoria in Laizhou Bay, Bohai Sea[J]. Journal of Fisheries of China, 2015, 39(8): 1166-1177. |
| [2] |
宁加佳, 杜飞雁, 王雪辉, 等. 基于稳定同位素的口虾蛄食性分析[J]. 水产学报, 2016, 40(6): 903-910. NING Jiajia, DU Feiyan, WANG Xuehui, et al. Feeding habits of mantis shrimp based on stable isotope analysis[J]. Journal of Fisheries of China, 2016, 40(6): 903-910. |
| [3] |
刘修泽, 郭栋, 王爱勇, 等. 辽东湾海域口虾蛄的资源特征及变化[J]. 水生生物学报, 2014, 38(3): 602-608. LIU Xiuze, GUO Dong, WANG Aiyong, et al. The resource characteristics and their variation of Oratosquilla oratoria in Liaodong Bay[J]. Acta Hydrobiolo gica Sincia, 2014, 38(3): 602-608. |
| [4] |
李显森, 吴亚飞, 尤宗博, 等. 渤海口虾蛄三重刺网渔获组成及其捕捞性能分析[J]. 海洋渔业, 2016, 38(5): 516-524. LI Xiansen, WU Yafei, YOU Zongbo, et al. Analysis on the catch composition by trammel net and its fishing performance for Oratosquilla oratoria in the Bohai Sea[J]. Marine Fisheries, 2016, 38(5): 516-524. DOI:10.3969/j.issn.1004-2490.2016.05.008 |
| [5] |
谷德贤, 刘茂利. 天津海域口虾蛄群体结构及资源量分析[J]. 河北渔业, 2011, 212(8): 24-26. GU Dexian, LIU Maoli. Analysis on the Population structure and abundance of in Tianjin Sea Area[J]. Hebei Fisheries, 2011, 212(8): 24-26. DOI:10.3969/j.issn.1004-6755.2011.08.008 |
| [6] |
王春琳, 徐善良, 梅文骧, 等. 口虾蛄的生物学基本特征[J]. 浙江水产学院学报, 1996, 15(1): 60-62. WANG Chunling, XU Shanliang, MEI Wenxiang, et al. A biological basic character of Oratosquilla aoatoria[J]. Journal of Zhenjian College of Fisheries, 1996, 15(1): 60-62. |
| [7] |
林月娇, 刘海映, 徐海龙, 等. 大连近海口虾蛄形态参数关系的研究[J]. 大连水产学院学报, 2008, 23(3): 215-217. LIN Yuejiao, LIU Haiying, XU Hailong, et al. Morphometry of mantis shrimp Oratosquilla oratoria in Dalian coast[J]. Journal of Dalian Ocean University, 2008, 23(3): 215-217. DOI:10.3969/j.issn.1000-9957.2008.03.011 |
| [8] |
孙东昱, 王蕾, 邱盛尧, 等. 海阳和烟台近岸口虾蛄群体的形态比较[J]. 渔业科学进展, 2021, 42(1): 154-164. SUN Dongyu, WANG Lei, QIU Shengyao, et al. Morphological comparison of Oratosquilla oratoria stocks along the coast of Haiyang and Yantai[J]. Progress in Fishery Sciences, 2021, 42(1): 154-164. |
| [9] |
王春琳, 母昌考, 丁爱侠, 等. 口虾蛄(Oratosquilla oratoria)同工酶的组织特异性及生化遗传分析[J]. 海洋与湖沼, 2004, 35(3): 258-263. WANG Chunlin, MU Changkao, DING Aixia, et al. Tissue specificity and biochemical genetic analysis of isozyme on crustacean Oratosquilla oratoria[J]. Ocea nologia et Limnologia Sinica, 2004, 35(3): 258-263. DOI:10.3321/j.issn:0029-814X.2004.03.010 |
| [10] |
潘国良, 张龙, 朱增军, 等. 浙江南部近岸海域春季口虾蛄生物量的时空分布[J]. 海洋与湖沼, 2013, 44(2): 366-370. PAN Guoliang, ZHANG Long, ZHU Zengjun, et al. Spatial-temporal distribution of the biomass of Oratosquilla oratoria in the coastal waters of south Zhejiang during spring[J]. Oceanologia et Limnologia Sinica, 2013, 44(2): 366-370. |
| [11] |
李鹏程, 张崇良, 任一平, 等. 山东近海口虾蛄空间分布特征及其与环境因子的关系[J]. 中国水产科学, 2020, 27(12): 1515-1523. LI Pengcheng, ZHANG Chongliang, REN Yiping, et al. Relationship between Oratosquilla oratoria spatial distribution and environmental factors in coastal Shangdong[J]. Journal of Fishery Sciences of China, 2020, 27(12): 1515-1523. |
| [12] |
王春琳, 叶选怡, 丁爱侠, 等. 虾蛄繁殖生物学与繁育技术研究[J]. 海洋湖沼通报, 2002(3): 58-64. WANG Chunlin, YE Xuanyi, DING Aixia, et al. Studies on reproductive biology and breeding technology of squilla[J]. Transactions of Oceanology and Lim nology, 2002(3): 58-64. DOI:10.3969/j.issn.1003-6482.2002.03.008 |
| [13] |
张年国, 潘桂平, 周文玉, 等. 不同饵料和水温对口虾蛄成活、生长及育肥性能的影响[J]. 动物学杂志, 2019, 54(3): 425-435. ZHANG Nianguo, PAN Guiping, ZHOU Wenyu, et al. Effects of different diets and water temperatures on survival, growth, fattening performance of Oratosquilla oratoria[J]. Chinese Journal of Zoology, 2019, 54(3): 425-435. DOI:10.13859/j.cjz.201903012 |
| [14] |
刘海映, 王桂娥, 王秀利. 大连海域口虾蛄资源遗传多样性的分析[J]. 大连水产学院学报, 2009, 24(4): 350-353. LIU Haiying, WANG Guie, WANG Xiuli. Genetic diversity analysis of mantis shrimp Oratosquilla oratoria from Dalian coast[J]. Journal of Dalian Ocean Uni versity, 2009, 24(4): 350-353. DOI:10.3969/j.issn.1000-9957.2009.04.013 |
| [15] |
LIU Y, CUI Z X. The complete mitochondrial genome of the mantid shrimp Oratosquilla oratoria (Crustacea: Malacostraca: Stomatopoda): Novel non-coding regions features and phylogenetic implications of the Stomatopoda[J]. Comparative Biochemistry and Physiology Part D: Genomics and Proteomics, 2010, 5(3): 190-198. DOI:10.1016/j.cbd.2010.04.001 |
| [16] |
ZHANG D Z, DING G, GE B M, et al. Geographical distribution, dispersal and genetic divergence of the mantis shrimp Oratosquilla oratoria (Stomatopoda: Squillidae) in China Sea[J]. Biochemical Systematics and Ecology, 2016, 65: 1-8. DOI:10.1016/j.bse.2016.01.009 |
| [17] |
DU X W, CAI S S, YU C G, et al. Population genetic structure of mantis shrimps Oratosquilla oratoria: Testing the barrier effect of the Yangtze River outflow[J]. Biochemical Systematics and Ecology, 2016, 66: 12-18. DOI:10.1016/j.bse.2016.02.033 |
| [18] |
隋宥珍, 刘连为, 徐开达, 等. 基于线粒体Cyt b基因的口虾蛄种群遗传结构研究[J]. 大连海洋大学学报, 2019, 34(3): 355-361. SUI Youzhen, LIU Lianwei, XU Kaida, et al. Population genetic structure of mantis shrimp Oratosquilla oratoria based on the partial mitochondrial DNA cytochrome b gene[J]. Journal of Dalian Ocean University, 2019, 34(3): 355-361. |
| [19] |
隋宥珍, 周永东, 卢占晖, 等. 东海海域口虾蛄种群遗传多样性[J]. 动物学杂志, 2019, 51(2): 291-300. SUI Youzhen, ZHOU Yongdong, LU Zhanhui, et al. Genetic diversity of mantis shrimp (Oratosquilla oratoria) populations in the East China Sea[J]. Chinese Journal of Zoology, 2019, 51(2): 291-300. |
| [20] |
刘建强. 莱州湾海洋工程建设对小清河口环境影响数值研究[D]. 青岛: 中国海洋大学, 2012. LIU Jianqiang. Numerical study on the environment effects of marine engineering on the mouth of Xiaoqing River in Laizhou Bay[D]. Qingdao: Ocean University of China, 2012. |
| [21] |
LUO X X, ZHANG S S, YANG J Q, et al. Macrobenthic community in the Xiaoqing River Estuary in Laizhou Bay, China[J]. Journal of Ocean University of China, 2013, 12(3): 366-372. DOI:10.1007/s11802-013-1955-9 |
| [22] |
LUO X X, LI S, YANG J Q, et al. Benthic habitat quality assessment based on biological indices in Xiaoqing River estuary and its adjacent sea of Laizhou Bay, China[J]. Journal of Ocean University of China, 2017, 16(3): 537-546. DOI:10.1007/s11802-017-3038-9 |
| [23] |
孟春霞, 邓春梅, 姚鹏, 等. 小清河口及邻近海域的溶解氧[J]. 海洋环境科学, 2005, 24(3): 25-28. MENG Chunxia, DNEG Chunmei, YAO Peng, et al. Dissolved oxygen in the Xiaoqinghe Estuary and Adjacent Waters[J]. Marine Environmental Science, 2005, 24(3): 25-28. DOI:10.3969/j.issn.1007-6336.2005.03.007 |
| [24] |
FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294-299. |
| [25] |
ZHU W B, DU X W, HAN Z Q, et al. The genetic divergence of Oratosquilla oratoria between the East China Sea and Yellow Sea: physical barrier and possible local adaptation[J]. Journal of the Marine Biological Association of the United Kingdom, 2019, 99(3): 631-638. DOI:10.1017/S0025315417001461 |
| [26] |
黄映萍, 王莹, 苗素英. 粤东海域口虾蛄遗传多样性[J]. 动物学杂志, 2011, 46(2): 82-89. HUANG Yingping, WANG Ying, MIAO Suying. Genetic diversity analysis of mantis shrimp (Oratosquilla oratoria) from the eastern coast of Guangzhou province[J]. Chinese Journal of Zoology, 2011, 46(2): 82-89. |
| [27] |
董鑫, 邢坤, 隋宥珍, 等. 基于线粒体COI基因序列的4个海域口虾蛄群体的遗传多样性研究[J]. 海洋科学, 2015, 39(7): 29-36. DONG Xin, XING Kun, SUI Youzhen, et al. Genetic diversity of Oratosqilla oratoria from four dea waters based on the mitochondrial COI gene sequences analysis[J]. Marine Sciences, 2015, 39(7): 29-36. |
| [28] |
张代臻, 丁鸽, 张华彬, 等. 渤海湾口虾蛄线粒体COI基因的遗传多样性研究[J]. 南京师大学报(自然科学版), 2010, 33(4): 80-83. ZHANG Daizhen, DING Ge, ZHANG Huabin, et al. Genetic diversity of Oratosquilla oratoria from different sea area of Bohai Bay by mitchondrial COI gene[J]. Journal of Nanjing Normal University (Natural Science Edition), 2010, 33(4): 80-83. DOI:10.3969/j.issn.1001-4616.2010.04.017 |
| [29] |
RYMAN N, UTTER F, LAIKRE L. Protection of intraspecific biodiversity of exploited fishes[J]. Reviews in Fish Biology and Fisheries, 1995, 5(4): 417-446. DOI:10.1007/BF01103814 |
| [30] |
季维智, 宿兵. 遗传多样性研究的原理和方法[M]. 杭州: 浙江科学技术出版社, 1999. JI Weizhi, SU Bing. Principles and methodologies of genetic diversity studies[M]. Hangzhou: Zhejiang Science and Technology Publishing House, 1999. |
| [31] |
杨建敏, 郑小东, 王如才, 等. 3种鲍16S rRNA基因片段序列的初步研究[J]. 中国海洋大学学报(自然科学版), 2003, 33(1): 36-40. YANG Jianmin, ZHENG Xiaodong, WANG Rucai, et al. Comparative study on mtDNA 16S rRNA gene sequence of three species of abalone[J]. Periodical of Ocean University of China, 2003, 33(1): 36-40. |
| [32] |
王景, 张凤英, 蒋科技, 等. 基于线粒体COI基因序列的三疣梭子蟹东海区群体遗传多样性分析[J]. 海洋渔业, 2015, 37(2): 114-121. WANG Jing, ZHANG Fengying, JIANG Keji, et al. Genetic diversity of Portunus trituberculatus based on the mitochondrial Cytochrome Oxidase Subunit I sequence from the East China Sea[J]. Marine Fisheries, 2015, 37(2): 114-121. DOI:10.3969/j.issn.1004-2490.2015.02.003 |
| [33] |
马朋, 刘萍, 李健, 等. 脊尾白虾3个野生群体线粒体COI基因的遗传多样性及其系统发育分析[J]. 渔业科学进展, 2011, 32(6): 50-26. MA Peng, LIU Ping, LI Jian, et al. The genetic diversity and phylogenetic analysis of COI gene in mitochondrial DNA of three populations of Exopalaemon carinicauda[J]. Progress in Fishery Sciences, 2011, 32(6): 50-26. DOI:10.3969/j.issn.1000-7075.2011.06.008 |
| [34] |
陈琴, 彭超, 赵峰, 等. 基于COI序列研究南海芋螺遗传多样性[J]. 基因组学与应用生物学, 2020, 39(9): 3955-3960. CHEN Qin, PENG Chao, ZHAO Feng, et al. Study on genetic diversity of Cone snail in South China Sea based on COI sequences[J]. Genomics and Applied Biology, 2020, 39(9): 3955-3960. |
| [35] |
KNIGHT A, MINDELL D P. Substitution bias, weighting of DNA sequence evolution, and the phylogenetic position of Fea's Viper[J]. Systematic Biology, 1993, 42(1): 18-31. DOI:10.1093/sysbio/42.1.18 |
| [36] |
IRWIN D M, KOEHER T D, WILSON A C. Evolution of the cytochrome b gene of mammals[J]. Jounal of Molecular Evolution, 1991, 32(2): 128-144. DOI:10.1007/BF02515385 |
| [37] |
KOCHER T D, THOMAS W K, MEYER A, et al. Dynamics of mitochondrial DNA evolution in animals: amplification and sequencing with conserved primers[J]. Proceedings of the National Academy of Sciences, 1989, 86(16): 6196-6200. DOI:10.1073/pnas.86.16.6196 |
| [38] |
李鹏飞, 贺舟挺, 徐开达, 等. 基于COI基因和D-loop序列的东海鳓鱼种质资源遗传变异研究[J]. 浙江海洋学院学报(自然科学版), 2013, 32(2): 93-98. LI Pengfei, HE Zhouting, XU Kaida, et al. Genetic diversity of llisha elongata in the East China Sea revealed by COI and D-loop sequence[J]. Journal of Zhejiang Ocean University (Natural Science), 2013, 32(2): 93-98. DOI:10.3969/j.issn.1008-830X.2013.02.001 |
| [39] |
李鹏飞, 周永东, 徐汉祥. 大黄鱼、鮸鱼及美国红鱼线粒体DNA的Cyt b基因序列比较[J]. 南方水产, 2008, 4(3): 43-47. LI Pengfei, ZHOU Yongdong, XU Hanxiang. Sequence analysis on mitochondrial (mtDNA) Cyt b genes in Pseudosciaena crocea, Miichthy miiuy and Sciaenops ocellatus[J]. South China Fisheries Science, 2008, 4(3): 43-47. DOI:10.3969/j.issn.2095-0780.2008.03.008 |
| [40] |
GRANT W S, BOWEN B W. Shallow population histories in deep evolutionary lineages of marine fishes: Insights from sardines and anchovies and lessons for conservation[J]. Journal of Heredity, 1998, 89(5): 415-426. DOI:10.1093/jhered/89.5.415 |
| [41] |
GROSBERG R K, CUNNINGHAM C W. Genetic structure in the sea from population to community[M]. Sunderland: Marine Community Ecology, 2001.
|
| [42] |
LEHMANN T, HAWLEY W A, GREBERT H, et al. The effective population size of Anopheles gambiae in Kenya: implications for population structure[J]. Molecular Biology and Evolution, 1998, 15(3): 264-276. DOI:10.1093/oxfordjournals.molbev.a025923 |
| [43] |
GUAN B X. Patterns and structures of the currents in Bohai, Huanghai and East China Seas[M]. Dordrecht: Oceanology of China Seas, 1994.
|
| [44] |
YANG M, LI X Z. Population genetic structure of the mantis shrimp Oratosquilla oratoria (Crustacea: Squillidae) in the Yellow Sea and East China Sea[J]. Journal of Oceanology and Limnology, 2018, 36(3): 905-912. DOI:10.1007/s00343-018-7030-z |
| [45] |
CHENG J, SHA Z L. Cryptic diversity in the Japanese mantis shrimp Oratosquilla oratoria (Crustacea: Squillidae): Allopatric diversification, secondary contact and hybridization[J]. Scientific Reports, 2017, 7: 1-13. DOI:10.1038/s41598-016-0028-x |
 2022, Vol. 46
2022, Vol. 46


