文章信息
- 刘杰, 薛文萍, 王玉璟, 袁嘉琳, 王龙, 张德超. 2022.
- LIU Jie, XUE Wen-ping, WANG Yu-jing, YUAN Jia-lin, WANG Long, ZHANG De-chao. 2022.
- 一株分离自太平洋卡罗琳深海海山水域NX-5T菌株的多相分类鉴定
- Polyphasic taxonomy of strain NX-5T isolated from the Caroline Seamount waters in the Pacific Ocean
- 海洋科学, 46(11): 47-54
- Marine Sciences, 46(11): 47-54.
- http://dx.doi.org/10.11759/hykx20201229002
-
文章历史
- 收稿日期:2020-12-29
- 修回日期:2021-01-24
2. 中国科学院海洋研究所, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 中国科学院海洋大科学中心, 山东 青岛 266071
2. Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China
黄杆菌属(Flavobacterium)是拟杆菌门(Bacteroidetes)、黄杆菌科(Flavobacteriaceae)中的一个成员, 1923年建属以来, 先后由Bernardet等[1]、Dong等[2]、Kang等[3]和Kuo等[4]等人进行了校正分类和新种补充。该属细菌的特征为好氧、革兰氏阴性、杆状、不运动或滑行运动、黄色菌落; 具有甲基萘醌类、磷脂酰乙醇胺等极性脂质; DNA G+C摩尔百分比为30%~52%[5]。该属成员分布较广, 并已经从海洋(如海水[6-8]、海洋藻类[9-11]、海洋沉积物[12-13])和南极[14-18]等环境中分离出来。2017年, 我们从热带西太平洋深海海山水域中分离到一株黄杆菌XN-5T。经初步16S rRNA基因序列分析, 该菌与关系最近模式菌株Flavobacterium frigoris和Flavobacterium degerlachei的序列相似性分别为97.8%和97.5%。在此基础上, 本研究采用包括传统形态特征、生理生化表型特征、脂肪酸和极性脂等化学特征、以及全基因组信息在内的多相分类途径, 对该菌株进行了详细的种水平上的分类鉴定。
1 材料与方法 1.1 样品采集与菌株分离样品为从热带西太平洋卡罗琳深海海山(10°48′N, 140°18′E, 水深525 m)周围收集的海水(“科学号”科考船2017年海山航次)。现场取100 μL海水无菌操作涂布在海水琼脂2216E (MA, Difco)平板培养基上, 并在20 ℃孵育2周; 挑取单菌落在MA平板上划线纯化并镜检合格, 作为后续实验菌株, 命名为XN-5T菌株。XN-5T菌株可在LB上20 ℃常规培养, 并保藏于–80 ℃超低温冰箱中(含10%脱脂牛奶的菌悬液)。另外, 从CGMCC保藏中心购买模式菌株Flavobacterium frigoris CGMCC1.9174T和Flavobacterium degerlachei CGMCC 1.9173T[14]作为本实验参比菌株, 其培养、保藏条件同上。
1.2 16S rRNA基因系统发育XN-5T菌株的DNA提取与纯化参考Sambrook和Russell[19]描述。16S rRNA基因的PCR扩增、克隆和测序按照Zhang等人[20]的方案进行。采用EzTaxon-e数据库[21]对16S rRNA基因序列进行序列比对。将XN-5T的序列与下载的相关模式菌株序列, 使用MEGA version 7.0软件[22]中的Clustal W程序进行多序列比对, 然后用MEGA version 7.0的邻位连接(NJ)[23]、最大似然(ML)和最大简约(MP)算法构建系统发育树。对于NJ、ML和MP算法通过Kimura双参数模型[24]计算遗传距离。最后通过基于1 000次重复的Bootstrap分析, 对3种方法生成的系统发育树进行评估。
1.3 表型特征测定革兰氏染色检测采用试剂盒(Bio Merieux Gramstain)。按照东秀珠等[25]方法进行硝酸盐还原、吲哚生产、柠檬酸、甲基红和Voges-Proskauer实验。酪蛋白、淀粉、吐温60、吐温80、羧甲基纤维素、海藻酸、琼脂和甲壳素的降解在MA固体平板上添加适当底物进行测试。用无菌海水中制备DNAase琼脂进行DNA水解试验。厌氧性生长检测采用厌氧瓶, 并在MA中添加20 mmol/L硝酸钠(NaNO3)和10 mmol/L亚硝酸钠(NaNO2), 25 ℃孵育7 d。
使用API 20E、API 20NE和API ZYM试剂盒(法国bioMérieux), 在20 ℃条件下进行生化特性和酶活性测定。API ZYM测试条在20 ℃下接种4 h后进行分析, API 20E和API 20NE测试条在20 ℃下接种7 d后进行分析。另外, 对XN-5T菌株进行4~50 ℃的生长温度范围测试; 同时对其进行系列梯度的耐盐性(0~10% NaCl)与pH(4.0~11.0)生长范围测试。
1.4 脂肪酸、呼吸醌、极性脂分析将XN-5T菌株和两个参比菌株Flavobacterium frigoris CGMCC1.9174T和Flavobacterium degerlachei CGMCC 1.9173T分别在TSA(胰蛋白胨15.0 g, 大豆蛋白胨5.0 g, 氯化钠5.0 g, 琼脂15.0 g, 蒸馏水1 000 mL)平板上20 ℃培养4 d后, 从TSA平板刮取菌体。脂肪酸的提取和准备工作是根据全自动细菌鉴定系统中的标准方法(MIDI, version 6.1)[26], 并使用数据库TSBA40进行脂肪酸组分识别。另外, 根据Collins[27]方案提取呼吸醌, 并用高效液相色谱(HPLC)分析; 采用甲醇-氯仿法抽提总脂, 采用Tindall[28-29]方法双向薄层层析分析极性脂成分。
1.5 全基因组分析XN-5T菌株的基因组测序利用北京诺华基因生物信息学技术有限公司的Illumina NovaSeq PE150测序平台进行, 筛选每个数据集的reads, 并使用SOAP denovo组装高质量的配对末端reads。将组装好的基因组框架图序列上传到NCBI数据库。
使用全基因组序列, 在GGDC网络服务器(http://ggdc.dsmz.de/)中计算XN-5T与模式菌株的DNA-DNA杂交值isDDH(in silico DNA-DNA hybridization), 以70%的相似性作为细菌物种边界的标准阈值。在EzBioCloud(https://www.ezbiocloud.net/tools)网站, 计算XN-5T与模式菌株的平均核苷酸一致性(ANI), 建议菌种边界的95%为阈值。基因组DNA的G+C百分比从全基因组序列中测得。
2 结果与分析 2.1 16S rRNA基因测序结果与系统发育分析XN-5T菌株的16S rRNA基因序列提交到GenBank/ EMBL/DDBJ, 获得序列号为MK956925。经序列比对后, 用neighbour-joining (NJ)和maximum-likelihood (ML)法构建系统发育进化树。
从图 1进化树可以看到, 在Flavobacterium属内, XN-5T菌株与F.degerlachei LMG 21915T、F.frigoris LMG21922T、F.sinopsychrotolerans 0533T、F.urumqiense Sr25T、F. xinjiangense AS1.2749T聚为一簇, 其中与F.degerlachei LMG 21915T、F.frigoris LMG21922T关系最近, 16S rRNA基因序列相似性最高, 分别为97.5%和97.8%。
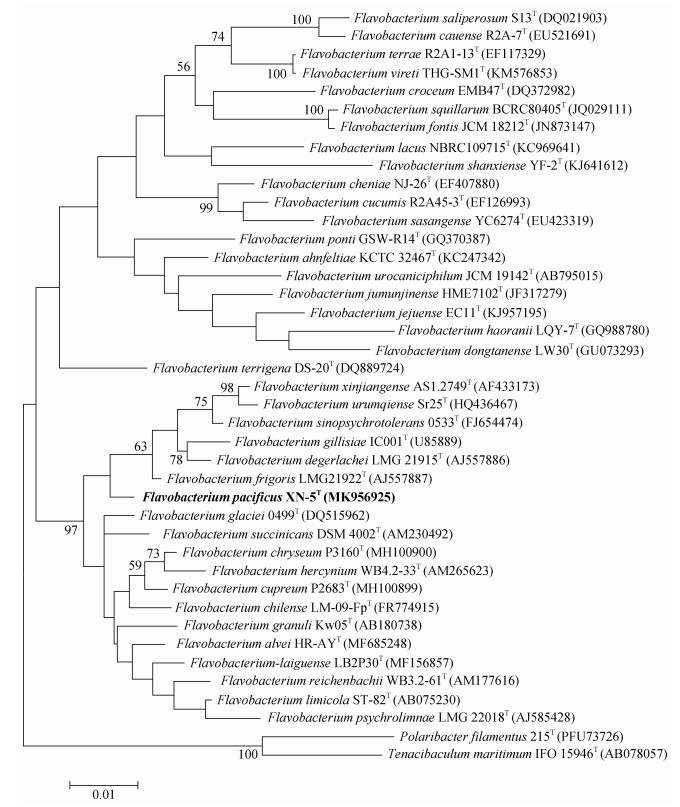 |
| 图 1 XN-5T菌株基于16SrRNA基因序列的邻接法系统进化树 Fig. 1 Neighbor -joining phylogenetic tree based on the 16S rRNA gene sequences showing the phylogenetic position of strain XN-5T |
在电镜(JEM-1400, JEOL)下观察(图 2), XN-5T菌株个体为短杆状、无鞭毛, 长0.8~1.7 µm、宽0.3~ 0.5 µm, 25 ℃在PYG培养基(蛋白胨10 g, 酵母粉5 g, 葡萄糖1 g, 琼脂18 g)上生长4 d后, 菌落呈黄色, 表面光滑, 整体凸起的圆形。
 |
| 图 2 XN-5T菌体细胞的透射电镜观察(25 ℃、LB中培养) Fig. 2 Cells of strain XN-5Tgrown at 25 ℃ on LB as visualized by transmission electron microscopy |
经测定, 菌株XN-5T为需氧型细菌, 生长不需要Na+。在PYG培养基上温度生长范围是4~30 ℃, 最适生长温度是在20~25 ℃, 在40 ℃菌株不生长。实验显示, 其耐盐范围是0~4.5% (w/v)NaCl, 最适生长范围是0~1% (w/v)NaCl。pH生长范围是6~9, 最适生长范围是7~8。
经测试, XN-5T菌株为革兰氏染色阴性、过氧化氢酶阳性且氧化酶阳性。XN-5T菌株能水解七叶苷, 但不能水解吐温60、吐温80、酪蛋白、羧甲基纤维素、几丁质、淀粉、尿素、海藻酸盐和DNA; 硝酸还原、甲基红、3-羟基丁酮实验和吲哚产生均为阴性。
在API ZYM体系中, 碱性磷酸酶、酯酶(C4)、酯酶脂肪酶(C8)、亮氨酸芳基胺酶、缬氨酸芳基胺酶、酸性磷酸酶、萘酚- AS -双磷酸水解酶、N-乙酰-D-氨基葡萄糖实验阳性; 在API 20NE和20E系统中, 胱氨酸芳基胺酶、β-半乳糖苷酶和β-葡萄糖醛酸酶弱阳性, 同化D-葡萄糖、D-甘露糖、N-乙酰氨基葡萄糖和D-麦芽糖, 蔗糖发酵实验均为阳性; 脂肪酶(C14)、胰蛋白酶、胰凝乳蛋白酶、α-葡萄糖苷酶、β-葡萄糖苷酶、α-甘露糖苷酶和α-岩藻糖苷酶检测阴性。色氨酸脱氨酶、精氨酸二水解酶、赖氨酸二水解酶、鸟氨酸二水解酶、H2S的产生、葡萄糖酸钾、癸酸、己二酸、苹果酸、柠檬酸三钠和苯乙酸的同化、肌醇、山梨醇、鼠李糖、蜜二糖和杏仁苷的发酵均为阴性。
表 1显示, XN-5T菌株在生长温度、淀粉与酪蛋白水解、硝酸盐还原, 产α-半乳糖苷酶、α-葡萄糖苷酶、β-葡萄糖苷酶, 同化L-阿拉伯糖、D-甘露醇、N-乙酰-氨基葡萄糖、葡萄糖酸钾, 以及利用蔗糖、阿拉伯糖产酸等生理生化特征方面, 均与参比菌株F. degerlachei和F. frigoris有明显差异。
| 特征 | XN-5T | F. degerlachei CGMCC 1.9173T |
F. frigoris CGMCC 1.9174T |
| 生长在30 ℃ | + | – | – |
| 水解: | |||
| 淀粉 | – | + | – |
| 酪蛋白 | – | – | + |
| 硝酸盐还原 | – | – | + |
| 酶活性(API ZYM): | |||
| α-半乳糖苷酶 | + | – | – |
| α-葡糖苷酶 | – | + | + |
| β-葡糖苷酶 | – | + | – |
| 同化: | |||
| L-阿拉伯糖 | – | – | + |
| D-甘露醇 | – | – | + |
| N-乙酰氨基葡萄糖 | + | + | – |
| 葡萄糖酸钾 | – | – | + |
| 产酸: | |||
| 蔗糖 | + | – | – |
| 阿拉伯糖 | – | + | – |
| 注: +反应阳性, –反应阴性 | |||
XN-5T菌株与参比菌株主要脂肪酸成分及摩尔百分比见表 2。
| 脂肪酸 | XN-5T | F. degerlachei CGMCC 1.9173T |
F. frigoris CGMCC 1.9174T |
| 饱和性 | |||
| C14: 0 | Tr | 1.3 | 1.1 |
| C16: 0 | 1.0 | 3.8 | 2.6 |
| 羟基化 | |||
| iso-C12: 03-OH | Tr | Tr | Tr |
| iso-C14: 03-OH | Tr | ND | ND |
| iso-C15: 03-OH | 9.3 | 3.7 | 5.1 |
| C15: 02-OH | 1.4 | 1.0 | 1.0 |
| C15: 03-OH | ND | 1.7 | 1.8 |
| iso-C16: 03-OH | 4.0 | 2.6 | 3.5 |
| C16: 02-OH | Tr | Tr | Tr |
| C16: 03-OH | 1.7 | 5.0 | 2.4 |
| iso-C17: 03-OH | 8.7 | 3.8 | 3.6 |
| C17: 02-OH | 1.6 | 1.5 | 1.4 |
| C17: 03-OH | Tr | Tr | Tr |
| 分支 | |||
| anteiso-C13: 0 | Tr | Tr | Tr |
| iso-C15: 0 | 14.5 | 7.0 | 14.2 |
| anteiso-C15: 0 | 12.4 | 17.1 | 25.0 |
| iso-C16: 0 | Tr | 1.2 | 1.5 |
| iso-C17: 0 | Tr | 0.2 | 0.3 |
| anteiso-C17: 0 | Tr | Tr | Tr |
| 不饱和性 | |||
| C14: 1ω5c | Tr | Tr | Tr |
| C15: 1ω6c | 6.3 | 3.2 | 8.9 |
| iso-C15: 1G | 3.7 | 2.8 | 3.6 |
| iso-C16: 1H | 1.5 | Tr | Tr |
| C17: 1ω8c | 1.6 | Tr | Tr |
| C17: 1ω6c | 3.4 | 3.3 | 1.7 |
| anteiso-C17: 1ω9c | 5.1 | ND | ND |
| C18: 1ω9c | Tr | Tr | Tr |
| 综合特征 | |||
| 2 | Tr | 1.1 | Tr |
| 3 | 12.1 | 30.3 | 14.6 |
| 9 | 4.8 | 1.3 | 1.5 |
| 注: 所有菌株中脂肪酸总量小于1%的脂肪酸没有显示出来。Tr, 痕迹(< 1%); ND, 没有检测到。综合特征是两种或三种脂肪酸, 在MIDI系统中不能被GLC分离。综合特征2包含iso-C16: 1-I和/或C14: 0 3-OH; 综合特征3包含C16: 1ω7c和/或C16: 1ω6 c; 综合特征9包含iso-C17: 1ω9 c和/或C16: 0 10-methyl。 | |||
表 2数据显示, 主要细胞脂肪酸为iso-C15: 0, anteiso-C15: 0和综合特征3 (包括C16: 1ω7c和/或C16: 1ω6 c)。
2.4 极性脂和醌类测试结果图 3显示的是菌株XN-5T与2个参比菌株的极性脂双向薄层层析图。从图中可以看出, XN-5T菌株的极性脂主要包含磷脂酰乙醇胺(PE), 3个未知的氨基脂(AL1-3), 1种不确定的糖脂(GL)和4种未知的极性脂(L1-4)。
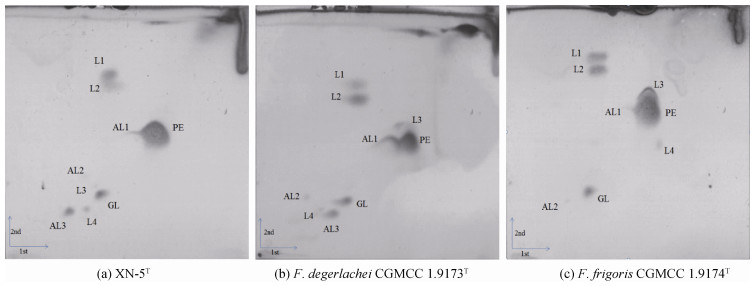 |
| 图 3 XN-5T与参比菌株的极性脂双向薄层层析图 Fig. 3 Two-dimensional TLC of polar lipids from strain XN-5T and the two reference strains 注: PE, 磷脂酰乙醇胺: AL1-3, 氨基脂; GL, 未识别的糖脂; L1-4, 未识别的极性脂 |
另外, 经测定菌株XN-5T的醌主要由甲基萘醌MK-6组成。
2.5 全基因组测试结果XN-5T菌株的基因组测试结果表明(DDBJ/ENA/ GenBank登录号为VOGZ00000000), 其DNA G+C百分比为34.5%。其与系统发育关系最近的两个模式菌株F. frigoris CGMCC 1.9174T和F. degerlachei CGMCC 1.9173T的平均核苷酸一致性(ANI)分别是83.35% 和78.77%, 远低于细菌物种边界建议的95%阈值(Yoon[30]); 另外, XN-5T菌株与上述两个模式菌株的DNA-DNA杂交值isDDH分别是30.80% 和22.40%, 也远低于细菌物种边界的建议70%阈值(Meier-Kolthoff[31])。
3 讨论黄杆菌种类比较多, 分布比较广。除一般陆地环境外, 目前已从海洋和南极等极端环境中分离出来, 但从深海海山环境中发现黄杆菌新物种的报道还没有。
此次我们针对从西太平洋卡罗琳深海海山水域分离到的XN-5T菌株, 与黄杆菌属(Flavobacterium)内系统发育关系最近的模式菌株F.degerlachei LMG 21915T、F.frigoris LMG 21922T之间的相似性分别为97.5%和97.8%。该菌为革兰氏染色阴性、氧化阳性、过氧化阳性、无鞭毛的短杆菌; 其菌落呈黄色、光滑、凸起、周边圆形; 该菌可在含0~4.5%(w/v) NaCl的培养基中生长(最适0~1%), 生长温度为4~30 ℃(最适为20~25 ℃), 生长pH为6.0~9.0(最适为pH 7.0~ 8.0); 其主要细胞脂肪酸为iso-C15: 0、anteiso-C15: 0、C16: 1ω7c和/或C16: 1ω6c, 极性脂主要有磷脂酰乙醇胺、3种未知氨基脂和一种不确定的糖脂。醌类主要组成是甲基萘醌MK-6, 该菌株DNA的G+C摩尔百分比34.5%, 与F. frigorisCGMCC 1.9174T和F. degerlachei CGMCC 1.9173T的ANI和isDDH也都远低于建议的细菌物种边界阈值。
4 结论根据以上多相分类测试数据及分析, 这株分离自西太平洋卡罗琳深海海山水的XN-5T菌株为黄杆菌属(Flavobacterium)内的一个新种, 并命名为Flavobacterium pacifica sp. nov.。现该菌株已保藏于中国普通微生物菌种保藏中心(CGMCC 1.17114T)和韩国典型菌种保藏中心(KCTC 72435T)菌种保藏中心。
| [1] |
BERNARDET J F, SEGERS P, VANCANNEYT M, et al. Cutting a Gordian Knot: emended classification and description of the genus Flavobacterium, emended description of the family Flavobacteriaceae, and proposal of Flavobacterium hydatis nom. Nov. (Basonym, Cytophagaaquatilis Strohl and Tait 1978)[J]. International Journal of Systematic Bacteriology, 1996, 46: 128-148. DOI:10.1099/00207713-46-1-128 |
| [2] |
DONG K, CHEN F, DU Y, et al. Flavobacterium enshiense sp. nov., isolated from soil, and emended descriptions of the genus Flavobacterium and Flavobacterium cauense, Flavobacterium saliperosum and Flavobacterium suncheonense[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63: 886-892. DOI:10.1099/ijs.0.039974-0 |
| [3] |
KANG J Y, CHUN J, JAHNG K Y. Flavobacterium aciduliphilum sp. nov., isolated from freshwater, and emended description of the genus Flavobacterium[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63: 1633-1638. DOI:10.1099/ijs.0.044495-0 |
| [4] |
KUO I, SAW J, KAPAN D D, et al. Flavobacterium akiainvivens sp. nov., from decaying wood of Wikstroemiaoahuensis, Hawai'i, and emended description of the genus Flavobacterium[J]. International Journal of Syste matic and Evolutionary Microbiology, 2013, 63: 3280-3286. DOI:10.1099/ijs.0.047217-0 |
| [5] |
BERNARDET J F, BOWMAN J P. BERGEY G H, et al. Genus I. Flavobacterium(1923) In Whitman W. (editor) Bergey's manual of systematic bacteriology[M]. Baltimore: Williams & Wilkins, 2011.
|
| [6] |
LI D D, LIU C, ZHANG Y Q, et al. Flavobacterium arcticum sp. nov., isolated from Arctic seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2007, 67: 1070-1074. |
| [7] |
NUPUR B V, SRINIVAS T N, KUMAR P A, Flavobacterium nitratireducens sp. nov., an amylolytic bacterium of the family Flavobacteriaceae isolated from coastal surface seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2013, 63: 2490-2496. DOI:10.1099/ijs.0.046524-0 |
| [8] |
YOON J H, PARK S, KANG S J, et al, Flavobacterium ponti sp. nov., isolated from seawater[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61: 81-85. DOI:10.1099/ijs.0.017582-0 |
| [9] |
MIYASHITA M, FUJIMURA S, NAKAGAWA Y, et al. Flavobacterium algicola sp. nov., isolated from marine algae[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60: 344-348. DOI:10.1099/ijs.0.009365-0 |
| [10] |
NEDASHKOVSKAYA O I, BALABANOVA L A, ZHUKOVA N V, et al. Flavobacterium ahnfeltiae sp. nov., a new marine polysaccharide-degrading bacterium isolated from a Pacific red alga[J]. Archives of Microbiology, 2014, 196(10): 745-752. DOI:10.1007/s00203-014-1010-2 |
| [11] |
PARK S H, KIM J Y, KIM Y J, et al. Flavobacterium jejuensis sp. nov., isolated from marine brown alga Ecklonia cava[J]. Journal of Microbiology, 2015, 53: 756-761. DOI:10.1007/s12275-015-5280-4 |
| [12] |
KAUR I, KAUR C, KHAN F, et al. Flavobacterium rakeshii sp. nov., isolated from marine sediment, and emended description of Flavobacterium beibuense[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 2897-2902. DOI:10.1099/ijs.0.035691-0 |
| [13] |
BAE S S, KIM M R, JUMG Y, et al. Flavobacterium sediminis sp. nov., a starch-degrading bacterium isolated from tidal flat sediment[J]. International Journal of Systematic and Evolutionary Microbiology, 2018, 68: 3886-3891. DOI:10.1099/ijsem.0.003081 |
| [14] |
VAN Y S, VANDECANDELAERE I, MERGAERT J, et al. Flavobacterium degerlachei sp. nov., Flavobacterium frigoris sp. nov. and Flavobacterium micromati sp. nov., novel psychrophilic bacteria isolated from microbial mats in Antarctic lakes[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54: 85-92. DOI:10.1099/ijs.0.02857-0 |
| [15] |
YI H, OH H M, LEE J H, et al. Flavobacterium antarcticum sp. nov., a novel psychrotolerant bacterium isolated from the Antarctic[J]. International Journal of Systematic and Evolutionary Microbiology, 2005, 55: 637-641. DOI:10.1099/ijs.0.63423-0 |
| [16] |
YI H, CHUN J. Flavobacterium weaverense sp. nov. and Flavobacterium segetis sp. nov., novel psychrophiles isolated from the Antarctic[J]. International Journal of Systematic and Evolutionary Microbiology, 2006, 56: 1239-1244. DOI:10.1099/ijs.0.64164-0 |
| [17] |
REN Q, YU M, LI Y, et al. Flavobacterium ovatum sp. nov., a marine bacterium isolated from an Antarctic intertidal sandy beach[J]. International Journal of Systematic and Evolutionary Microbiology, 2018, 68: 795-800. DOI:10.1099/ijsem.0.002586 |
| [18] |
KRALOVE S, SVEC P, BUSSE H J, et al. Flavobacterium chryseum sp. nov. and Flavobacterium psychroterrae sp. nov., novel environmental bacteria isolated from Antarctica[J]. International Journal of Systematic and Evolutionary Microbiology, 2018, 68: 3132-3139. DOI:10.1099/ijsem.0.002952 |
| [19] |
SAMBROOK J, RUSSELL D W. Molecular Cloning: a Laboratory Manual. Cold Spring member of the family Oxalobacteraceae isolated from alpine glacier cryoconite[J]. International Journal of Systematic and Evolutionary Microbiology, 2011, 61: 2186-2190. DOI:10.1099/ijs.0.028001-0 |
| [20] |
ZHANG D C, SCHINNER F, MARGESIN R. Pedobacter bauzanensis sp. nov., isolated from soil[J]. International Journal of Systematic and Evolutionary Microbiology, 2010, 60: 2592-2595. DOI:10.1099/ijs.0.018903-0 |
| [21] |
KIM O S, CHO Y J, LEE K, et al. Introducing EzTaxon: a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62: 716-721. DOI:10.1099/ijs.0.038075-0 |
| [22] |
KUMAR S, STECHER G, TAMURA K. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution, 2016, 33(7): 1870-1874. DOI:10.1093/molbev/msw054 |
| [23] |
SAITOU N, NEI M. The neighbor-joining method: a new method for reconstructing phylogenetic trees[J]. Molecular Biology and Evolution, 1987, 4(4): 406-425. |
| [24] |
KIMURA M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. Journal of Molecular Evolution, 1980, 16: 111-120. DOI:10.1007/BF01731581 |
| [25] |
东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001. DONG Xiuzhu, CAI Miaoying. Manual for identification of common bacterial systems[M]. Beijing: Science Press, 2001. |
| [26] |
徐丽华, 李文均, 刘志恒, 等. 放线菌系统学[M]. 北京: 科学出版社, 2007. XU Lihua, LI Wenjun, LIU Zhiheng, et al. Systematics of actinomycetes[M]. Beijing: Science Press, 2007. |
| [27] |
COLLINS M D. Distribution of menaquinones in Actinomycetes and Corynebacteria[J]. Journal of General Microbiology, 1977, 100(2): 221-230. DOI:10.1099/00221287-100-2-221 |
| [28] |
TINDALL B J. Lipid composition of Halobacterium lacusprofundi[J]. FEMS Microbiology. Letters, 1990, 66: 199-202. DOI:10.1111/j.1574-6968.1990.tb03996.x |
| [29] |
TINDALL B J. A comparative study of the lipid composition of Halobacterium saccharovorum from varioussources[J]. Systematic and Applied Microbiology, 1990, 13: 128-130. DOI:10.1016/S0723-2020(11)80158-X |
| [30] |
YOON S H, HA S M, KWON S, et al. Introducing EzBio Cloud: a taxonomically united database of 16S rRNA gene sequences and whole-genome assemblies[J]. International Journal of Systematic and Evolutionary Microbiology, 2017, 67: 1613-1617. DOI:10.1099/ijsem.0.001755 |
| [31] |
MEIER K. Genome sequence-based species delimitation with confidence intervals and improved distance functions[J]. BMC Bioinformatics, 2013, 14: 60-74. DOI:10.1186/1471-2105-14-60 |
 2022, Vol. 46
2022, Vol. 46


