文章信息
- 张越, 赵峰, 孙梦岩, 李玉全, 徐奎栋. 2022.
- ZHANG Yue, ZHAO Feng, SUN Meng-yan, LI Yu-quan, XU Kui-dong. 2022.
- 稀有OTU对海洋底栖纤毛虫群落异质性的影响研究
- Effect of rare OTUs on the community heterogeneity of marine benthic ciliates
- 海洋科学, 46(12): 20-30
- Marine Sciences, 46(12): 20-30.
- http://dx.doi.org/10.11759/hykx20201124002
-
文章历史
- 收稿日期:2020-11-24
- 修回日期:2021-02-08
2. 中国科学院海洋研究所海洋生物分类与系统演化实验室, 山东 青岛 266071;
3. 中国科学院大学, 北京 100049;
4. 中国科学院海洋大科学研究中心, 山东 青岛 266071
2. Department of Marine Organism Taxonomy & Phylogeny Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
3. University of Chinese Academy of Sciences, Beijing 100049, China;
4. Center for Ocean Mega-Science, Chinese Academy of Sciences, Qingdao 266071, China
原生生物是海洋底栖生态系统的重要成员, 是底栖微食物网中初级生产力向高营养级传递的重要媒介[1]。其中, 底栖纤毛虫作为原生生物的一个主要类群, 其形态多样, 结构复杂, 具有代表性。经典形态学的研究方法为认知底栖纤毛虫多样性奠定了基础, 例如Hausmann等通过半定量培养的方法, 在地中海东部海域沉积物中发现35种纤毛虫[2]; 代仁海采用密度梯度离心-定量蛋白银染色法(Ludox-QPS)在春季长江口和东海检获了278种底栖纤毛虫[3]。然而, 传统形态学方法的局限性仍限制着底栖纤毛虫的研究, 如形态学鉴定需要极强的分类学基础; 稀有类群个体少易被忽视, 且难以培养, 易造成多样性的低估等。
随着分子生物学技术的发展, 新一代测序技术为研究海洋底栖纤毛虫多样性提供了新的技术支撑。分子生物学手段克服了传统形态学方法的局限性, 可以有效地检获底栖纤毛虫个体稀少及培养困难的物种, 已有研究极大地扩展了对海洋底栖纤毛虫多样性的认识。如Pawlowski等在4 g沉积物中检获630余个纤毛虫OTU(operated taxonomic unit, 可操作分类单元)[4]。
分子生物学工作发现, 原生生物在百米到米级的空间范围内, 甚至是生物学重复间, 存在较高的异质性[5-7]。大型底栖生物群落也常表现出异质性的特点[8-9], 造成群落异质性的原因众说纷纭, 一种是沉积物环境存在很高的异质性[10], 小尺度下的微生境孕育独特的底栖生物群落, 进而呈现出明显的群落异质性。另一种可能则是分子手段的方法学的局限所引入的高异质性的“假象”, 虽然分子手段在检获纤毛虫物种多样性, 尤其是稀有类群, 具有极大优势, 但分子手段检获的OTU往往比形态学方法高出几个数量级[11], 而且绝大部分OTU都属于稀有类群。Zhao等在潮间带、陆架到深海平原等生境, 均检获了高比例的稀有OTU, 比例高达80%[12-13]。通用的去噪和OTU聚类方法在去除PCR扩增和测序中产生错误的效能依然不足[14], 尤其是稀有OTU部分, 因此这些OTU的真实性仍饱受怀疑[15]。
高比例的稀有OTU与生物学重复之间的群落高异质性密切相关。然而目前仍未见稀有OTU与站位间和同一站位重复间群落异质性关系的研究。如何去除稀有OTU可真实展现群落异质性仍待解答。因此, 本研究在东海选取4个具有明显环境梯度和距离梯度的站位, 每个站设置3个重复, 采用环境总DNA提取和高通量测序技术, 研究底栖纤毛虫群落结构及分布模式。并设置不同梯度, 依次去除相对丰度低于0.01%至0.1%的OTU, 探明稀有OTU与群落异质性的关系, 以期明晰陆架海区底栖纤毛虫群落站位间及同一站位重复间差异水平, 并给出去除稀有OTU阈值的科学建议。
1 材料与方法 1.1 调查站位与样品采集本研究于2019年5月搭乘“向阳红18号”科学考察船在东海海域A(29°59′53″N, 122°41′53″E), B (29°28′19″N, 123°5′20″E), C(29°10′12″N, 123°39′4″E)和D(27°27′11″N, 122°10′16″E)四个站位进行样品采集(图 1)。
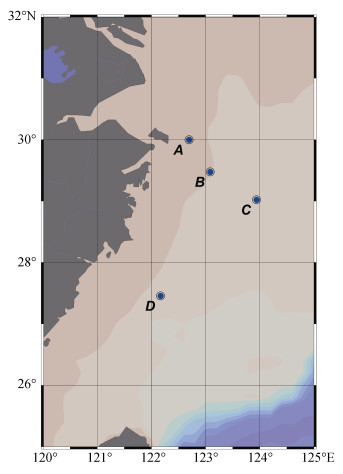 |
| 图 1 采样站位图 Fig. 1 Map of sampling sites |
利用0.1 m2改进型的Gray-Ohara箱式采泥器, 在每个站位采集1箱含有上覆水的未受扰动的沉积物, 刮取0~2 cm表层沉积物约20 g放入封口袋中, 每箱泥刮取3个重复样品。置于–80 ℃超低温冰箱中保存, 以待后续DNA提取。
1.2 DNA提取及PCR扩增采用PowerSoil DNA isolation kit(Qiagen, Germany)试剂盒, 提取沉积物DNA。每个站位的3个重复样品, 各取0.3 g沉积物, 分别提取DNA。
获得的12份DNA样品(4站位*3重复), 采用巢式PCR技术扩增纤毛虫的18S rRNA基因V4高变区[16]。使用的引物分别为纤毛虫特异性引物Cil F、Cil RⅠ、Cil RⅡ、Cil RⅢ, 以及真核微生物18S V4高变区通用引物EukF、EukR。每个样品进行3次重复PCR, 混合后送测。
PCR反应体系及程序如下: 第一步, 采用纤毛虫特异性引物(Cil F、Cil RⅠ、Cil RⅡ、Cil RⅢ)进行扩增, 反应体系如下: 正反向引物各0.5 μL, 模板1.5 μL, Q5高保真聚合酶0.25 μL, 10 mmol/L dNTPs 0.5 μL, Q5 Reaction Buffer 5 μL, High GC Enhancer 5 μL, 最后使用双蒸水补齐至25 μL。反应流程如下: 98 ℃预变性30 s; 98 ℃变性45 s, 58 ℃退火1 min, 72 ℃延伸1 min, 循环35次; 最后72 ℃延伸10 min, 4 ℃终止[17]。
第二步, 采用真核微生物18S V4高变区特异性引物(EukF、EukR), 以第一步所得PCR产物为模板, 反应体系如下: 正反向引物各1 μL, 模板1.5 μL, Q5高保真聚合酶0.5 μL, 10 mmol/L dNTPs 1 μL, Q5 Reaction Buffer 10 μL, High GC Enhancer 10 μL, 最后使用双蒸水补齐至50 μL。反应流程如下: 98 ℃预变性30 s; 98 ℃变性30 s, 57 ℃退火45 s, 72 ℃延伸1 min, 循环10次; 98 ℃变性30 s, 49 ℃退火45 s, 72 ℃延伸1 min, 循环25次; 最后72 ℃延伸5 min, 4 ℃终止[18]。
1.3 测序以及序列数据分析使用琼脂糖凝胶电泳检测PCR产物质量及片段长度, 将同一个样品的3个PCR产物合并, 最终4个站位, 每站位3重复, 共12个样品。
样品送往北京诺禾致源生物信息科技有限公司, 采用Illumina Hiseq平台进行双端测序, 使用New England Biolabs公司的NEB NextⓇ Ultra DNA Library Prep Kit (New England Biolabs, USA)试剂盒构建文库, 文库经过Qubit定量和文库检测合格后, 上机测序。
测序完成后, 下机数据根据Barcode序列和引物序列拆分出样品数据, 截掉Barcode和引物序列。原始序列使用FLASH(V1.2.7)拼接, 得到原始标签(Raw Tags)[19]。使用QIIME(V1.7.0)对上一步得到的Raw Tags进行质量控制和过滤: 1) 质量控制: 将Raw Tags从连续的低质量值碱基数(默认长度为≤19)达到设定长度(默认长度值为3)的第一个低质量碱基位点截断; 2) 过滤: 经过质量控制的Tags数据集, 将其中连续的高质量碱基长度低于Tags长度75%的Tags去掉[20]。之后使用UCHIME结合数据库进行比对以去除嵌合体序列, 得到有效数据Effective Tags[21-22]。
对有效数据使用UPARSE[23]进行处理。去冗余; 去噪; 以97%的序列相似度进行OTU聚类。对获得的OTU代表序列与Silva数据库(v.123)采用基本局部比对搜索工具(basic local alignment search tool, BLAST)进行比对, 对OTU的分类信息进行注释。相对丰度为每个类群的序列数占总序列数的比例; 丰富度为每个类群的OTU数; 相对丰富度为每个类群的OTU数占总OTU数的比例。
1.4 数据统计分析对于每个OTU, 计算其在各个站位内的相对丰度, 由该OTU在各个站位内3个重复中所检获的序列数在3个重复内总序列中所占的比例表示。规定每个站位内3个重复中总相对丰度小于0.1%的OTU为稀有OTU[24]。设置过滤梯度共计15个, 包括不去除OTU, 去除相对丰度低于0.01%、0.015%、0.02%、0.025%、0.03%、0.035%、0.04%、0.045%、0.05%、0.06%、0.07%、0.08%、0.09%和0.1%的OTU。去掉各个站位内, 相对丰度小于该梯度的OTU, 重新进行Cluster聚类分析, 并通过SIMPER分析计算各个站位间和同一站位重复间的群落相似性和差异性, 同时分析群落结构。
使用Past4.01绘制稀释性曲线。使用R语言(R3.6.3)中的“fossil”和“vegan”包进行序列的标准化处理。使用Venny2.1绘制韦恩图。使用Excel绘制条形图与折线图。使用PRIMER v6软件中的CLUSTER功能分析样品间相似度与不相似度, 原始数据进行log(x+1)转化, 其分析基于Bray-Curtis矩阵; SIMPROF和ANOSIM功能分析群落结构差异是否显著。
2 结果 2.1 基本测序数据12个测序样本共获得586 016条序列, 但是样品中检获了大量的属于舞毛亚纲和寡毛亚纲的浮游纤毛虫序列。为研究底栖纤毛虫的多样性与群落, 去除了浮游纤毛虫的OTU, 剩余208 220条序列, 平均每个样品17 352条, 最高为A1号样品25 646, 最低为D3号样品的9 237。为保证样品间可比性, 以n= 9 237对数据进行标准化, 之后分析均基于标准化后的数据。经标准化后, 12个样品共检获1 295个OTU, 其中B1最少为344个, C2最多为516个。稀释性曲线显示, 样品基本趋于饱和(图 2)。
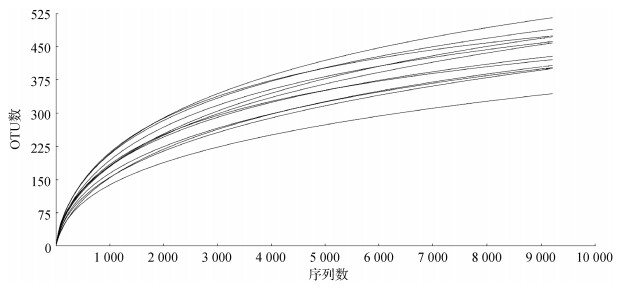 |
| 图 2 样本稀释性曲线 Fig. 2 Rarefaction curve of all the samples |
标准化后数据所检获的纤毛虫隶属于11个纲, 除篮口纲(Nassophorea)外, 其他各纲均在所有样品中都有检获。
把同一站位的重复样品合并, 四个站位群落组成差异明显, A站位裂口纲相对丰度最高, 占33.29%, 其次为核残迹纲和前口纲, 分别占22.18%和15.08%。B站位最丰富的类群则是核残迹纲, 序列相对丰度达41.14%, 其次为裂口纲和前口纲, 占19.39%和10.67%。C站位裂口纲相对丰度最高, 为31.32%, 和A站位相似, 但相对丰度第二高的类群则变成了旋唇纲, 为21.28%, 寡膜纲和前口纲则又次之, 但两者相对丰度较为接近, 分别为15.28%和13.71%。D站位最丰富的类群也为裂口纲, 相对丰度为23.84%, 寡膜纲为相对丰度第二高的类群, 占14.25%, 旋唇纲和前口纲次之, 分别为12.93%和12.29%。由于目前国际通用的Silva数据库序列信息仍不完善, D站位中未能鉴定到纲一级水平的占比为21.70% (图 3a)。
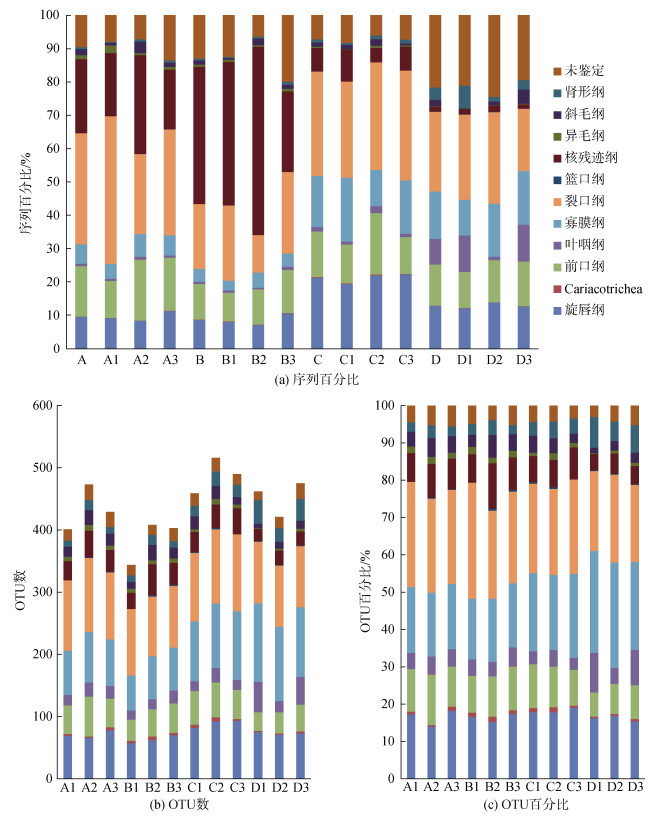 |
| 图 3 各样品纤毛虫序列百分比、OTU数和OTU百分比 Fig. 3 Proportions of sequences and OTUs, and the number of OTUs of ciliate communities in each of the 12 sediment samples |
将同一站位的重复样品分开, 12个样品各类群相对丰富度比较接近, 仅有D1和D3样品叶咽纲丰富度明显增高(图 3b), 同时这两个样品叶咽纲的相对丰度也较D2样品明显增高(图 3a)。
各站位3个重复共有OTU占各站位所检获的总OTU数的百分比最高为39.5%最低为31.9%(图 4), 但共有OTU在该站位重复中所占序列数则最高为94.79%, 最低为85.34%, 其中A、B和C站位共有OTU序列数比例均为94%以上, 只有D站位较低。每个站位的特有OTU几乎全部在该站位属于稀有OTU, 仅有叶咽纲的2个, 肾形纲的1个OTU在D站位中属丰富OTU, 均在D2号样品中未检出。
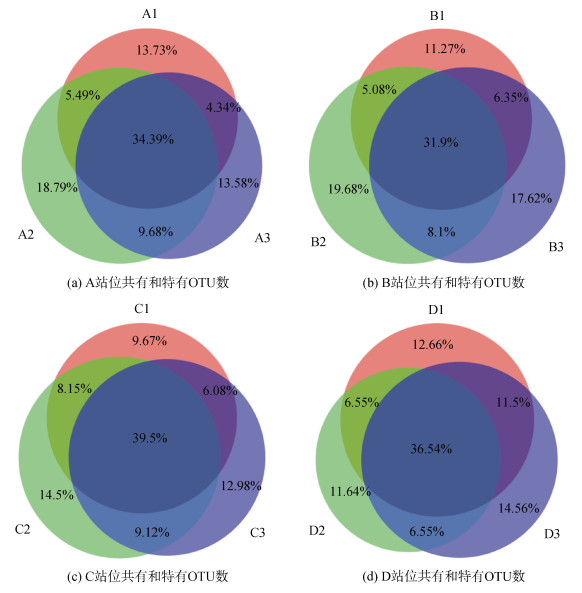 |
| 图 4 各站位共有和特有OTU数 Fig. 4 Proportion of unique and shared OTUs in three replicates of each site |
当重复合并时, 进行Cluster分析显示, 在4个站位中, A和B两站位之间差异不显著(SIMPROF: P > 0.05)。而当将每个站位的3个重复分开, 12个样品共同进行Cluster分析时, 结果显示C站位和D站位的3个重复能够各自成组, 且组内差异不显著(SIMPROF: P > 0.05)(图 5a)。但组内相似性仍不高, C、D两站位内平均相似性分别为70.23%和66.82%。A、B两站位的重复之间分组混乱, 如将A、B两站位的各自重复设为一组, 根据ANOSIM检验, A、B两组组间差异不显著(ANOSIM: R=0.333, P=0.1)。SIMPER分析显示A、B两组组内差异分别为31%和33%, 而A、B两组组间差异仅为33%。
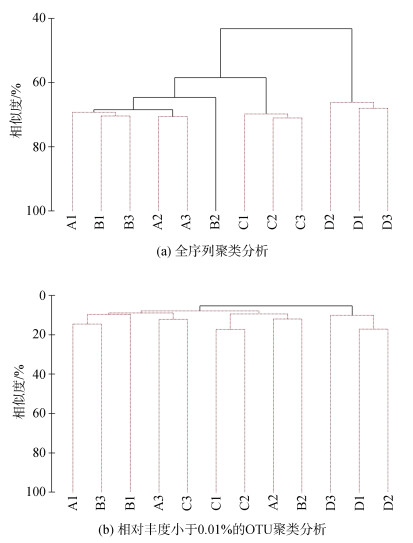 |
| 图 5 12个测序样本全序列和相对丰度小于0.01%的OTU聚类分析 Fig. 5 Clustering and SIMPROF analyses based on total OTUs and the extremely rare OTUs with relative abundance lower than 0.01%. The groups with no significant difference are marked by red bars (P > 0.05) 注: 红色显示差异不显著(P > 0.05) |
总计检获的1 297个OTU中, 有255个在全部12个样品中仅检获一条序列。稀有OTU分布极不均匀, 相对丰度小于0.01%的OTU占总OTU数的40%左右, 占稀有OTU的50%左右。但是其序列数仅占总序列数的不足2%(图 6)。相对丰度小于0.01%OTU聚类分析显示其各样品相似度均低于20%且除D站位外均不成明显分组(图 5b)。
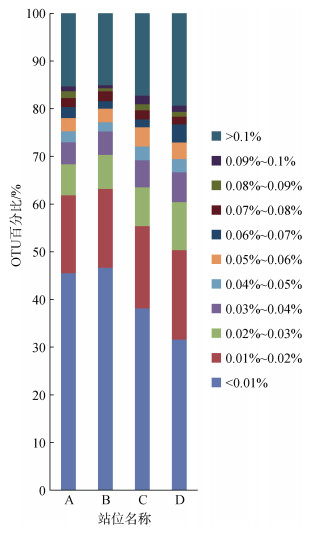 |
| 图 6 不同相对丰度OTU占总OTU百分比 Fig. 6 Proportion of OTUs with various relative abundance |
按15个梯度去掉各个站位内稀有OTU, 重新进行Cluster聚类分析, 并通过SIMPER分析计算各个站位组间相似性和组间差异性, 同时分析群落结构。
以0.01%和0.1%两梯度为例说明去除稀有OTU对群落异质性的影响。去除相对丰度低于0.01%的序列: 经标准化后每个站位3个重复总序列数为27 711, 故过滤强度为0.01%时, 仅去除了在3个重复中总序列数为1和2的OTU。平均每个站位被过滤掉103个OTU, 减少23.5%。其中最多为A2样品, OTU数由473下降到329, 减少144个, 最低为D2和D3号样品, 分别由421和475下降到339和393, 减少82个。序列数则平均仅减少125, 最高为A2样品, 减少176, 最低为D2样品, 减少98。Cluster聚类分析显示, 4个站位的各自3个重复各成一组, 分组变得明确, 组内相似度上升, A组相似度由69.21%上升到77.29%, B组相似度由67.62%上升到76.33%, C组相似度由70.23%上升到76.34%, D组相似度由66.82%上升到71.43%。组间差异受到的影响并不一致, 其中AB两站位间组间差异由33%下降到28.22%, 下降明显, 而其他组间差异下降幅度微小, 甚至B、D两组的组间差异由59.57%变为59.75%, 有微小上升。
去除相对丰度低于0.1%的序列: 稀有OTU被全部过滤。所有4个站位重复间相似性增加到80%以上, A、B两个本身相似度较高的站位组间差异由33%下降到21%, 下降明显, 而AC、BC站位间差异则有极小幅下降, AD、BD和CD间差异反而有小幅上涨。
通过记录每一次过滤后站位间和重复间异质性, 以及样品OTU数及序列数的变化, 绘制变化趋势图。
重复间相似性随过滤变化趋势可以看出, 组内相似性在过滤的一开始快速上升, 随后上升速度趋于平缓。此后随着过滤强度的增加, 降低异质性的效果逐渐降低(图 7a)。
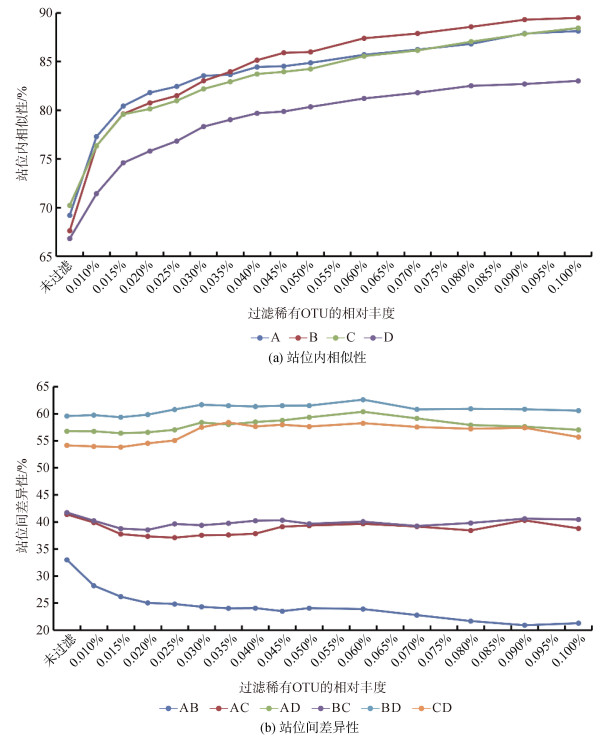 |
| 图 7 站位内相似性及站位间差异性随过滤的变化 Fig. 7 Variations in community similarity between pairwise stations and dissimilarity among replicates of each site after removing the rare OTUs |
站位间差异随过滤变化趋势可以看出, A、B两站位的差异性在过滤的一开始明显下降, 但随后趋于平缓, AC和AD之间的差异性有小幅下降, 但也随后趋于平缓, BC、BD和CD间差异性基本处于维持不变的状态, 甚至在中间几个梯度上出现了不明显的上升。但最终, 随着过滤强度的增加, 组间差异也最终趋于稳定, 而其变化趋势与其本身差异大小有关(图 7b)。
OTU数则随着过滤在一开始大幅下降, 随后下降趋势也逐渐减缓。序列数则一直保持相似的下降幅度, 没有明显拐点。相对丰度0.015%以下的OTU数普遍占该样品检获的OTU数的36.55%(最高为43.97%, 最低为30.52%)。
整体上, 去除相对丰度低于0.015%的OTU, 显著提高了重复间群落相似度水平, 又保证了站位间差异的稳定性, 同时保留了大部分序列与OTU。
2.5 以黄海某样品数据为例验证过滤处理有效性选取Huang等[25]于2015年8月采集于北黄海冷水团及渤海的4个站位的底栖纤毛虫群落结构数据, 该数据集的每个站位均具有两个生物学重复。
以这4个站位的表层沉积物数据进行相似处理, 由图可见重复间相似性和站位间差异性的变化趋势和东海样品具有一定的相似性, 组内相似性都是较快上升, 然后趋于平缓(图 8a); 组间差异性则也是差异较大的基本不受影响, 差异较低的随过滤会有一个明显的下降, 然后趋于平缓(图 8b)。去除相对丰度低于0.015%的OTU, 显著提高了重复间群落相似度水平, 并可保证站位间差异的稳定性。去除后, 各样品的OTU数平均下降32.74%(10.17%~48.72%), 但仍高于形态学所检获物种数。
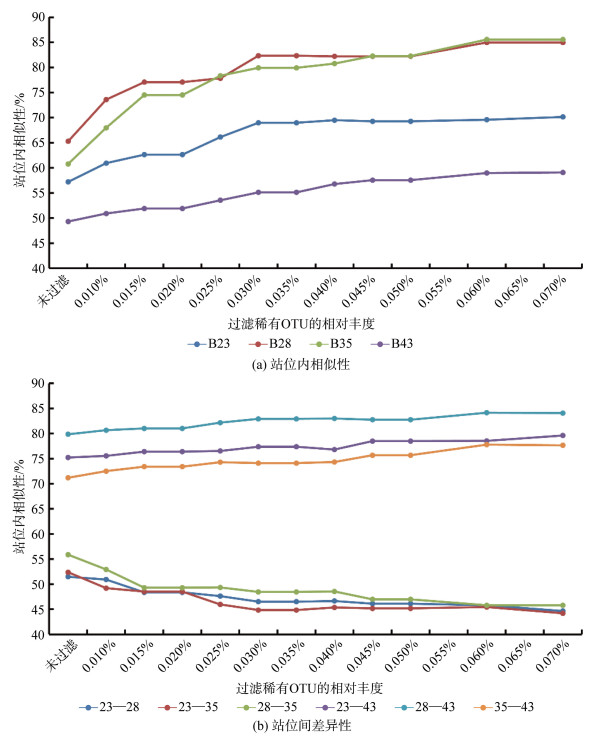 |
| 图 8 黄海站位组内相似性及组间差异性随过滤的变化 Fig. 8 Variations in community similarity between pairwise sites and dissimilarity among replicates of each site after removing the rare OTUs in the Yellow Sea |
本研究检获了沉积物中纤毛虫群落的高异质性特点, 同一站位3个重复共有OTU仅占总OTU数的不足40%。前人研究沉积物中有孔虫多样性时, 也检获了极高的群落异质性, 仅有12.25%到30%的OTU能够在同时3个重复样本中发现[7]。单一重复样本仅能检获总OTU多样性的57.9%, 该值介于14.2%至88.9%之间[7]。而在本研究中, 单个重复样品所能检获的OTU数平均为64.3%(54.6%~71.3%)。沉积物环境相比于水体, 更容易形成微生境[10], 且由于底栖类群在沉积物环境中运动受限, 也容易形成斑块化的分布模式。
但是分子手段的方法学局限也可能引入高异质性的“假象”。分子手段检获的OTU往往比形态学方法高出几个数量级[11], 而且绝大部分OTU都属于稀有类群。高比例的稀有OTU与重复之间的高异质性密切相关。PCR和测序过程中产生的错误序列, 容易表现为仅在单个重复中极少量检出。并且有研究表明包含错误的序列可能是唯一的, 而且会形成一个新的OTU[26], 这也可能是极低丰度特有OTU产生的原因之一。这也与本研究中稀有OTU分布的结果相符, 最稀有的相对丰度低于0.01%的OTU占总OTU数的40%, 而序列数仅占2%以下。绝大部分稀有OTU仅在一个重复检获, 从而导致重复间高群落异质性。
但是, 目前仍有许多研究证明过去人们对沉积物中原生生物的多样性大大低估, 如Forster等使用2.5 g表层沉积物提取RNA进行扩增测序, 根据丰富度预测, 在测序工作中未检测到的OTU的比例为66.3%[27]。高通量测序后对序列的不同处理策略会使获得的OTU总数出现很大差异, 但是其中的丰富类群则在各种方法中具有可重复性[15]。而测序产生的容易出错的序列常以低丰度出现[27-28], 随着序列数的增多, OTU的真实性也在增加。增加测序深度能够明显降低技术重复间差异性[29]。
3.2 过滤稀有OTU对重复间差异的影响过滤相对丰度较低的OTU, 能够有效降低重复间异质性, 同时不影响或极小地影响站位间差异。随着从低相对丰度到高相对丰度逐渐过滤稀有OTU, 重复之间的相似性逐渐增高, 说明去除稀有OTU确实有效降低了重复间异质性, 但是随着过滤强度的增加, 曲线逐渐平缓, 其降低异质性的效果明显降低, 拐点大致位于0.015%处。
丰富和稀有OTU对站位间差异的贡献有所不同, 相互之间差异越大的站位, 高丰度的OTU对其差异贡献越多, 而随着其差异逐渐降低, 稀有OTU对其差异的贡献则越多。本身相似性较高的A、B两站位, 其差异性快速下降, 随后趋于平缓, 这是因为被过滤掉的稀有OTU对其差异性贡献较大, 而相似性次之的AC和AD之间, 差异性则随着过滤有小幅下降, 随后仍趋于平缓。相似性最低的CB、BD和CD之间, 其差异性未受过滤的影响, 基本处于不变。而拐点则仍位于0.015%左右。
OTU数随着过滤梯度变化的拐点仍大致位于0.015%。在相对丰度小于0.015%的区间内, 聚集了大量的OTU, 其数量远高于其他区间。在本研究中, 由于标准化后每个样品的序列数为9 237, 故在站位内相对丰度小于0.015%的OTU序列数最高仅为4, 这些OTU绝大多数都是无法在该站位所有3个重复中同时检出的特有OTU。因此该区间内的OTU由测序和PCR扩增产生的错误序列可能性较高, 导致了重复间虚高的差异性。合理去除低丰度OTU, 可更清晰地体现底栖纤毛虫群落分布的特点, 获得底栖纤毛虫更加真实的分布模式。
但是, 过滤势必会降低高通量技术检获多样性的能力。而且稀有类群毫无疑问也是整个群落中的重要组成部分, 并且可能具有和丰富类群截然不同的生态功能[24], 简单地去除低丰度的OTU必然会损失稀有OTU所包含的生态信息。同时, 来自不同生境, 面向不同类群, 以及不同的预处理可能都会影响过滤的阈值。因此通过设置多个过滤梯度, 在过滤的过程中观察站位间及重复间异质性的变化, 确定合适的阈值, 可为采样量较少或者站位仅有单一样品研究提供科学指导。此外, 也有许多研究致力于从更精细的层面借助算法改进在OTU聚类前后的去噪过程[30-31], 以求更严谨地去除稀有类群中不真实的部分。
4 结论对沉积物样品进行分子多样性分析时, DNA测序中产生的重复间异质性在测序深度饱和的情况下主要是由极稀有OTU造成的。这些OTU在总OTU数中所占比例较高, 分布随机。对极稀有OTU进行过滤可以显著降低沉积物样品的异质性, 同时对站位之间的差异影响较小。而最终丰富OTU所展现的差异则能反应样品间真实的差异。但是过滤方法仍需根据采样生境、研究目标类群和测序深度等条件进一步细化, 确定适用于不同研究类型的阈值, 争取既保证减小异质性又能良好的保留稀有OTU所包含的群落信息等。
致谢: 中国科学院海洋研究所李宇航、赵荣杰协助样品采集, 谨致谢忱。
| [1] |
徐奎栋. 海洋微型底栖生物的多样性与地理分布[J]. 生物多样性, 2011, 19(6): 661-675. XU Kuidong. Biodiversity and biogeography of marine microbenthos: progress and prospect[J]. Biodiversity Science, 2011, 19(6): 661-675. |
| [2] |
HAUSMANN K, HULSMANN N, POLIANSKI I, et al. Composition of benthic protozoan communities along a depth transect in the eastern mediterranean sea[J]. Deep-Sea Research Part Ⅰ: Oceanographic Research Papers, 2002, 49(11): 1959-1970. DOI:10.1016/S0967-0637(02)00095-X |
| [3] |
代仁海. 黄东海底栖纤毛虫多样性及微型底栖生物群落结构特点[D]. 青岛: 中国科学院海洋研究所, 2012. DAI Renhai. Community structure and ecological characteristics of microbenthos with special reference to benthic ciliate diversity in the Yellow Sea and East China Sea[D]. Qingdao: Institute of Oceanology, Chinese Academy of Sciences, 2012. |
| [4] |
PAWLOWSKI J, CHRISTEN R, LECROQ B, et al. Eukaryotic richness in the abyss: Insights from pyrotag sequencing[J]. PLoS One, 2011, 6(4): e18169. DOI:10.1371/journal.pone.0018169 |
| [5] |
MCCLAIN C R, NEKOLA J C, KUHNZ L, et al. Local-scale faunal turnover on the deep pacific seafloor[J]. Marine Ecology Progress Series, 2011, 422: 193-200. DOI:10.3354/meps08924 |
| [6] |
GAEVER S V, VANREUSEL A, HUGHES J A, et al. The macro- and micro-scale patchiness of meiobenthos associated with the darwin mounds (north-east atlantic)[J]. Journal of the Marine Biological Association of the United Kingdom, 2004, 84(3): 547-556. DOI:10.1017/S0025315404009555h |
| [7] |
LEJZEROWICZ F, ESLING P, PAWLOWSKI J. Patchiness of deep-sea benthic foraminifera across the southern ocean: Insights from high-throughput DNA sequencing[J]. Deep-Sea Research Part Ⅱ-Topical Studies in Oceanography, 2014, 108: 17-26. DOI:10.1016/j.dsr2.2014.07.018 |
| [8] |
GAEVER S V, GALERON J, SIBUET M, et al. Deep-sea habitat heterogeneity influence on meiofaunal communities in the Gulf of Guinea[J]. Deep-Sea Research Part Ⅱ: Topical Studies in Oceanography, 2009, 56(23): 2259-2269. DOI:10.1016/j.dsr2.2009.04.008 |
| [9] |
DANOVARO R, CARUGATI L, CORINALDESI C, et al. Multiple spatial scale analyses provide new clues on patterns and drivers of deep-sea nematode diversity[J]. Deep-Sea Research Part Ⅱ: Topical Studies in Oceanography, 2013, 92: 97-106. DOI:10.1016/j.dsr2.2013.03.035 |
| [10] |
ORCUTT B N, SYLVAN J B, KNAB N J, et al. Microbial ecology of the dark ocean above, at, and below the seafloor[J]. Microbiology and Molecular Biology Reviews, 2011, 75(2): 361-422. DOI:10.1128/MMBR.00039-10 |
| [11] |
BACHY C, DOLAN J R, LOPEZ-GARCIA P, et al. Accuracy of protist diversity assessments: Morphology compared with cloning and direct pyrosequencing of 18s rRNA genes and its regions using the conspicuous tintinnid ciliates as a case study[J]. ISME Journal, 2013, 7(2): 244-255. DOI:10.1038/ismej.2012.106 |
| [12] |
ZHAO F, FILKER S, STOECK T, et al. Ciliate diversity and distribution patterns in the sediments of a seamount and adjacent abyssal plains in the tropical western pacific ocean[J]. BMC Microbiology, 2017, 17: 192. DOI:10.1186/s12866-017-1103-6 |
| [13] |
ZHAO F, XU K D. Molecular diversity and distribution pattern of ciliates in sediments from deep-sea hydrothermal vents in the Okinawa Trough and adjacent sea areas[J]. Deep-Sea Research Part I: Oceanographic Research Papers, 2016, 116: 22-32. DOI:10.1016/j.dsr.2016.07.007 |
| [14] |
KUNIN V, ENGELBREKTSON A, OCHMAN H, et al. Wrinkles in the rare biosphere: Pyrosequencing errors can lead to artificial inflation of diversity estimates[J]. Environmental Microbiology, 2010, 12(1): 118-123. DOI:10.1111/j.1462-2920.2009.02051.x |
| [15] |
HARDGE K, NEUHAUS S, KILIAS E S, et al. Impact of sequence processing and taxonomic classification approaches on eukaryotic community structure from environmental samples with emphasis on diatoms[J]. Molecular Ecology Resources, 2018, 18(2): 204-216. DOI:10.1111/1755-0998.12726 |
| [16] |
STOCK A, EDGCOMB V, ORSI W, et al. Evidence for isolated evolution of deep-sea ciliate communities through geological separation and environmental selection[J]. BMC Microbiology, 2013, 13: 150. DOI:10.1186/1471-2180-13-150 |
| [17] |
LARA E, BERNEY C, HARMS H, et al. Cultivation-independent analysis reveals a shift in ciliate 18S rRNA gene diversity in a polycyclic aromatic hydrocarbon-polluted soil[J]. FEMS Microbiology Ecology, 2007, 62(3): 365-373. DOI:10.1111/j.1574-6941.2007.00387.x |
| [18] |
STOECK T, BASS D, NEBEL M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water[J]. Molecular Ecology, 2010, 19: 21-31. DOI:10.1111/j.1365-294X.2009.04480.x |
| [19] |
MAGOC T, SALZBERG S L. Flash: Fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21): 2957-2963. DOI:10.1093/bioinformatics/btr507 |
| [20] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QⅡME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [21] |
HAAS B J, GEVERS D, EARL A M, et al. Chimeric 16S rRNA sequence formation and detection in sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research, 2011, 21(3): 494-504. DOI:10.1101/gr.112730.110 |
| [22] |
EDGAR R C, HAAS B J, CLEMENTE J C, et al. Uchime improves sensitivity and speed of chimera detection[J]. Bioinformatics, 2011, 27(16): 2194-2200. DOI:10.1093/bioinformatics/btr381 |
| [23] |
EDGAR R C. UPARSE: Highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [24] |
LOGARES R, AUDIC S, BASS D, et al. Patterns of rare and abundant marine microbial eukaryotes[J]. Current Biology, 2014, 24(8): 813-821. DOI:10.1016/j.cub.2014.02.050 |
| [25] |
HUANG P P, ZHAO F, XU K D. Complementary DNA sequencing (cDNA): An effective approach for assessing the diversity and distribution of marine benthic ciliates along hydrographic gradients[J]. Journal of Oceanology and Limnology, 2021, 39(1): 208-222. DOI:10.1007/s00343-020-9234-2 |
| [26] |
REEDER J, KNIGHT R. The 'rare biosphere': A reality check[J]. Nature Methods, 2009, 6(9): 636-637. DOI:10.1038/nmeth0909-636 |
| [27] |
FORSTER D, DUNTHORN M, MAHE F, et al. Benthic protists: The under-charted majority[J]. FEMS Microbiology Ecology, 2016, 92(8): fiw120. DOI:10.1093/femsec/fiw120 |
| [28] |
HUSE S M, WELCH D M, MORRISON H G, et al. Ironing out the wrinkles in the rare biosphere through improved OTU clustering[J]. Environmental Microbiology, 2010, 12(7): 1889-1898. DOI:10.1111/j.1462-2920.2010.02193.x |
| [29] |
LANZEN A, LEKANG K, JONASSEN I, et al. DNA extraction replicates improve diversity and compositional dissimilarity in metabarcoding of eukaryotes in marine sediments[J]. PLoS One, 2018, 13(1): e0192337. DOI:10.1371/journal.pone.0192337 |
| [30] |
VETROVSKY T, BALDRIAN P. Analysis of soil fungal communities by amplicon pyrosequencing: Current approaches to data analysis and the introduction of the pipeline seed[J]. Biology and Fertility of Soils, 2013, 49(8): 1027-1037. DOI:10.1007/s00374-013-0801-y |
| [31] |
QUINCE C, LANZEN A, DAVENPORT R J, et al. Removing noise from pyrosequenced amplicons[J]. BMC Bioinformatics, 2011, 12: 38. DOI:10.1186/1471-2105-12-38 |
 2022, Vol. 46
2022, Vol. 46


