文章信息
- 刘凤华, 王淏, 贾乔雅, 毕允晨. 2022.
- LIU Feng-hua, WANG Hao, JIA Qiao-ya, BI Yun-chen. 2022.
- 长牡蛎电压门控钙离子通道β亚基的无标签亲和层析纯化
- Purification of voltage-gated calcium channel β subunit from Pacific oyster with tag-free affinity chromatography
- 海洋科学, 46(2): 87-96
- Marine Sciences, 46(2): 87-96.
- http://dx.doi.org/10.11759/hykx20211231003
-
文章历史
- 收稿日期:2021-12-31
- 修回日期:2022-01-05
2. 中国科学院大学, 北京 100049;
3. 青岛海洋科学与技术试点国家实验室 海洋生物学与生物技术功能实验室, 山东 青岛 266237
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. Marine Biology and Biotechnology Laboratory, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266237, China
蛋白质是维持细胞结构和功能完整的生物大分子, 许多疾病的发病机制均与关键蛋白质的错误折叠、功能紊乱相关[1-2]。因此, 蛋白质相关研究是生物学和医学等领域的重要研究方向[3], 获得高纯度的蛋白质是首要和关键步骤。常用的蛋白质分离纯化方法有等电点沉淀[4]、离子交换层析[5]、分子排阻层析[6]、亲和层析[7]、疏水作用层析[8]、电泳[9]等。其中, 亲和层析具有特异性高、稳定性好、成本低等优点[10], 是蛋白质分离纯化的首选方法。
亲和标签是一些对特定的生物或化学配体具有高度亲和力的多肽, 选择合适的亲和标签将其融合到目的蛋白的N端或C端[11], 并与相应的载有固定化配体的亲和柱配合使用, 能在未去除核酸或其他细胞组分的情况下将目的蛋白的纯度提升数百倍甚至数千倍[12]。亲和标签与亲和柱的结合往往是可逆的, 洗脱条件也比较温和, 对纯化条件敏感的蛋白质以及蛋白质复合体尤为适用[12]。目前, 常用的亲和标签有多组氨酸(polyhistidine)标签, Flag标签, 谷胱甘肽巯基转移酶(GST)标签, 链霉亲和素结合肽(SBP)标签, 麦芽糖结合蛋白(MBP)标签等等。需要注意的是, 虽然部分亲和标签可以增加重组蛋白的溶解性、提高产量、甚至在一定条件下促进蛋白折叠[13], 但亲和标签尤其是分子量较大的亲和标签也容易给目的蛋白的结构与功能带来不利因素, 影响下游应用[14]。以GST标签为例, 它是一种天然存在于真核细胞中发挥重要作用的转移酶, 与目的蛋白融合后能提高重组蛋白溶解性, 但该标签的分子量较大(约26 kDa)且容易形成同型二聚体, 可能影响重组蛋白功能, 需要通过酶切的方式去除[15], 去除后常残留额外的氨基酸残基并伴随样品损失[16]。因此, 低分子量标签受到了一些研究者的青睐[3, 17]。最常用的低分子量标签是多组氨酸标签, 固定化的Co2+、Cu2+、Ni2+、Zn2+、Ca2+和Fe3+均可与多组氨酸标签融合蛋白结合, 其亲和作用有赖于组氨酸的咪唑基与金属离子的配位作用[18-19]。然而, 多组氨酸亲和标签也存在诸多问题, 如影响重组蛋白的热稳定性[20], 降低重组蛋白活性[21]等。
马晓川等[22]对PDB数据库中蛋白质的分析结果显示, 天冬氨酸和谷氨酸的羧基、组氨酸的咪唑基以及半胱氨酸的巯基较多地参与了同金属离子的配位。这些氨基酸多为极性氨基酸, 一般分布在蛋白质的表面, 并且上述一种或几种氨基酸可在某些蛋白质特定结构域富集[23-24]。目前, 可以利用特定氨基酸与金属离子结合能力进行纯化的蛋白通常含有极为丰富的组氨酸, histidine rich protein 2 (Plasmodium falciparum)即属此类, 该蛋白共有326个残基, 其中组氨酸残基112个, 占34.4%[25-27]。除上述报道外, 少见基于蛋白质天然序列的无标签纯化。综上, 本研究借助固定相金属离子亲和层析对C端富含组氨酸残基的长牡蛎(Crassostrea gigas)电压门控钙离子通道(VGCC) β亚基进行了无标签亲和层析纯化, 通过多个组氨酸突变体的构建进一步确定了该纯化方法的序列基础, 并利用天然β亚基的这种序列特性, 辅助了VGCC蛋白复合体的原位提取关键步骤的监测, 为具有相似特征的蛋白纯化提供了参考。
1 材料与方法 1.1 序列分析基于NCBI的非冗余蛋白序列数据库, 对长牡蛎电压门控钙离子通道β亚基的氨基酸序列进行搜索, 并下载该蛋白在部分软体动物、模式动物中的同源序列进行多序列比对分析。多序列比对借助BioEdit软件[28]完成, 然后通过EMBOSS Needle(版本6.6.0)计算各同源序列与长牡蛎电压门控钙离子通道β亚基氨基酸序列的相似性[29]。
1.2 基因序列获取以长牡蛎为实验动物对其闭壳肌进行分离, 研磨后, 使用Invitrogen的TRIzol™ Reagent提取总RNA。接着, 用Vazyme公司的反转录试剂盒HiScript Ⅲ 1st Strand cDNA Synthesis Kit (+gDNA wiper)将得到的总RNA反转录成cDNA, 并以此为模板选用引物B1-F、B1-R(表 1)进行PCR扩增。随后, 使用Vazyme公司的5 min TM TA/Blunt-Zero Cloning Kit将PCR产物连接到pCE2载体中。取适量连接产物转化大肠杆菌DH5α感受态细胞, 涂布平板后37 ℃培养过夜, 挑取单菌落进行DNA测序鉴定获得阳性克隆。
| 引物名称 | 引物序列(5'-3') |
| B1-F | GGGACGGTAGAACAAGGCAA |
| B1-R | TTGCAAATCCCTGGAAGGTT |
| B2-F | ACTTTAAGAAGGAGATATACATGGCGCAAGACAAA |
| B2-R | TGGTGGTGGTGGTGCTCGAGCTATATATCTATAGACTGTTGT |
| M1-F | GGAAGTGGGGCTGGGAGTGGGGCAGCAGGAGCTGGTGCTAGTGTTCATAGTTCATCATTTCGTGAAC |
| M1-R | TCCTGCTGCCCCACTCCCAGCCCCACTTCCTGACCCACTCCCAGGGGGCACTGTGTTATGCCTCG |
| M2-F | GAGAGGCGATGATGCTGAATCTAGTATACGTGGTGGTGATAGAGAC |
| M2-R | GTATACTAGATTCAGCATCATCGCCTCTCCGCGGTTCACGAAATG |
| M3-F | CTCTGCTAGAAAGGAGGGTGAATCCTTTGACACGAGAGAAGCC |
| M3-R | CAAAGGATTCACCCTCCTTTCTAGCAGAGTACCGGTCATCTCGC |
| 注: 下划线标注的为组氨酸突变后的氨基酸残基编码的核苷酸序列 | |
选取1.2中获得的阳性克隆为模板, 参照Ana Paula Jacobus等的同源重组方法[30], 使用表 1中引物B2-F、B2-R进行PCR扩增, 将目的片段与线性化的pET-28a载体按一定比例混合并转化大肠杆菌DH5α完成同源重组。通过对载体插入片段测序鉴定获得大肠杆菌阳性克隆。
1.4 突变型表达载体构建为探究长牡蛎VGCC β亚基中参与Ni-NTA结合的关键氨基酸残基, 本研究对多个不同位置的组氨酸残基进行分组突变。突变型β亚基分别命名为M1 (包含的突变位点是H441S, H443S, H445S, H447A, H449S, H451A, H452A, H454A, H456A), M2(在突变型M1的基础上增加突变位点H469G, H472A, H474S, H475S), M3(在突变型M2的基础上增加突变位点H533A, H537G, H539S)。表 1的引物序列中包含对相应组氨酸突变后氨基酸残基编码的核苷酸序列, 用下划线标注。突变型表达载体构建方法是, 突变型M1以1.3中的阳性克隆为模板, 使用引物M1-F、M1-R进行PCR扩增, 后续与1.3中野生型表达载体构建相同。突变型M2的构建选取M1的阳性克隆为模板, 引物使用M2-F、M2-R; 突变型M3的构建选取M2的阳性克隆为模板, 引物使用M3-F、M3-R。
1.5 表达菌株的培养及诱导表达从已完成测序鉴定的阳性克隆中提取构建成功的表达载体, 转入感受态细胞BL21(DE3), 37 ℃培养过夜并挑取单克隆, 接种至卡那抗性的LB(lysogeny broth)液体培养基中, 37 ℃, 220 r/min培养8 h后, 按照1%的比例将表达菌株转接至1 L的LB液体培养基中, 加入卡那霉素至终质量浓度50 μg/mL, 随后在37 ℃摇床中220 r/min培养至OD值为1.0~1.2, 加入0.2 mmol/L异丙基-β-D-硫代半乳糖苷(IPTG)于18 ℃诱导蛋白表达12 h[31], 6 500 r/min离心10 min收集细菌沉淀。
1.6 VGCC β亚基纯化野生型和突变型β亚基的纯化步骤完全一致。首先, 在等量野生型和各突变型菌体中加入等体积裂解缓冲液(25 mmol/L Tris-HCI、pH 8.8, 150 mmol/L NaCl, 1 mmol/L PMSF), 冰上均质重悬后, 用高压细胞破碎机进行破碎。随后在4 ℃, 15 000 r/min离心1 h去除未破碎细胞碎片, 收集上清液并将其分别与等量Ni-NTA (Invitrogen)混合。借助旋转混匀仪于4 ℃孵育120 min后, 用含10~500 mmol/L咪唑的等体积25 mmol/L Tris-HCl (pH 8.8)缓冲液梯度洗脱, 各步留样体积相同, 借此定性分析野生型和不同突变型β亚基对Ni-NTA的结合能力强弱。利用氨基三乙酸镍(Ni-NTA)、亚胺基二乙酸铜(Cu-IDA)、亚胺基二乙酸锌(Zn-IDA)纯化蛋白时, 将样品分为等体积的3份分别与等量的3种亲和柱孵育, 纯化步骤与上述相同, 各步洗脱及留样体积保持一致。
1.7 SDS-PAGE凝胶电泳及免疫印迹分析根据长牡蛎VGCC β亚基的分子量, 实验选取浓度为12.5%的SDS-PAGE对各蛋白样品进行分析鉴定, 各样品留样体积、加入的蛋白loading buffer及点样量均保持一致。鉴于长牡蛎的VGCC β亚基C端含有多个组氨酸残基, 实验选用针对多组氨酸标签的一抗His-tag (27E8) mouse mAb (Cell Signaling Technology)和二抗anti-mouse IgG, HRP-linked antibody (Cell Signaling Technology)对包括突变型长牡蛎VGCC β亚基在内的不同蛋白样品进行免疫印迹分析, 以进一步确认目的蛋白条带。蛋白量与其印迹条带的面积和深浅具有相关性[32], 通过ImageJ计算不同样品印迹条带的灰度值, 从而实现定量分析。
1.8 长牡蛎闭壳肌中VGCC蛋白复合体的原位提取分离长牡蛎闭壳肌, 研磨后加入裂解缓冲液(10 mmol/L MOPS-Na、pH 7.4, 0.3 mol/L蔗糖, 0.5 mmol/L EDTA, 复配蛋白酶抑制剂)均质重悬破碎细胞。随后4 ℃, 6 450 r/min离心5 min, 收集上清液均分为2份作为不同去垢剂实验组。超速离心(4 ℃, 41 500 r/min离心1 h)收集膜组分, 分别加入等体积含1%毛地黄皂苷(digitonin)或1% glyco-diosgenin (GDN)去垢剂的缓冲液(20 mmol/L MOPS-Na、pH 7.4, 100 mmol/L NaCl, 0.5 mmol/L CaCl2, 复配蛋白酶抑制剂)重悬后, 4 ℃过夜孵育增溶[33], 再次超速离心(73 400 r/min, 30 min)去除不溶组分, 收集含有VGCC蛋白复合体的上清液用于后续分析。
2 结果与分析 2.1 序列分析VGCC是真核细胞中钙离子内流的主要通道, 在神经和肌肉生理活动中发挥重要功能。其最多包含5个亚基: α1亚基, α2亚基, β亚基, δ亚基和γ亚基。如图 1所示, 将长牡蛎VGCC β亚基的氨基酸序列与多个同源序列进行比对, 结果表明, 长牡蛎VGCC β亚基存在两段在同源蛋白中较为保守的氨基酸序列, 分别为68~130位氨基酸组成的SH3结构域和229~409位氨基酸组成的Guanylate kinase结构域[34]。与脊椎动物相比, 其他软体动物的β亚基氨基酸序列与长牡蛎的相似性更高, 例如, 扇贝的序列相似性为67.6%, 而脊椎动物兔子的序列相似性为49.8%。长牡蛎β亚基序列C端存在3组聚集的组氨酸残基, 分别用红色、橙色、黄色标注。红色标注的(HG)7序列在真核生物CDF蛋白家族也有报道, 其loop 4含有富含组氨酸的序列(HX)n, 其中X通常是G或C, n=3~6。这些序列可能参与了生理活动中与金属离子的结合[35]。除此之外, 对比图 1中列出的脊椎动物β亚基氨基酸序列发现, 软体动物β亚基的C端普遍存在多组聚集的组氨酸序列(紫色标注), 尤其是海蜗牛β亚基C端的H3、H6序列。天冬氨酸残基也常与聚集的组氨酸残基同时出现。软体动物VGCC β亚基C端富含的组氨酸序列与功能之间的关系还有待进一步探索与研究。
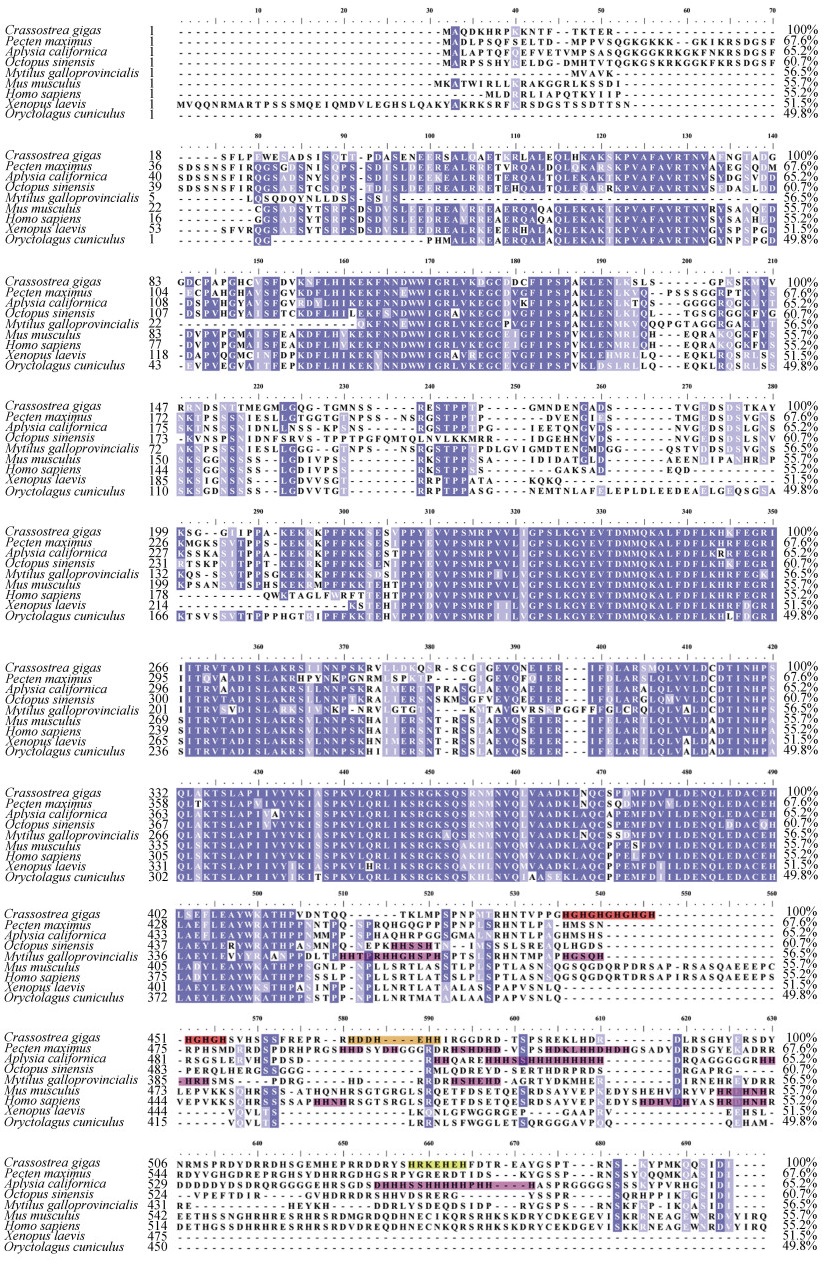 |
| 图 1 长牡蛎电压门控钙离子通道β亚基及其同源蛋白序列比对结果 Fig. 1 Sequence alignment of voltage-gated calcium channel β subunit (Crassostrea gigas) with its homologous proteins 注: 蓝色部分为一致性100%的残基, 浅蓝色部分为相似残基(Threshhold % for shading设为60%)。3组组氨酸残基分别用红、橙、黄色标记, 突变体M1对应红色区域中组氨酸残基突变, 突变体M2对应红色及橙色区域中组氨酸残基突变, 突变体M3对应红、橙、黄色区域中组氨酸残基全突变, 其他物种序列中聚集的组氨酸残基用紫色标记。氨基酸序列的Accession number如下: 长牡蛎(Crassostrea gigas) XP_011429591.2, 扇贝(Pecten maximus) XP_033728564.1, 海蜗牛(Aplysia californica) XP_035825861.1, 章鱼(Octopus sinensis) XP_029654560.1, 紫贻贝(Mytilus galloprovincialis) VDI38901.1, 小鼠(Mus musculus) XP_006497382.1, 人(Homo sapiens) XP_011517961.1, 非洲爪蟾(Xenopus laevis) XP_041435102.1, 穴兔(Oryctolagus cuniculus) NP_001075748.1 |
通过分子克隆得到的长牡蛎VGCC β亚基序列, 经DNA测序分析, 与NCBI数据库中长牡蛎VGCC β亚基X3亚型(Accession number: XP_011429591.2)完全一致。在未添加任何亲和标签的情况下, 重组表达后获得的蛋白分子量为63.7 kDa, 且该蛋白表达量较高。基于该重组蛋白的氨基酸序列, 借助C-I-TASSER On-line Server(https://zhanggroup.org//C-I-TASSER/)对长牡蛎VGCC β亚基的结构进行预测, 结果表明β亚基C端3组较为连续的组氨酸残基均位于柔性较大的区域, 有利于固定相金属亲和层析方法的运用。
在柱料体积满足目的蛋白载量要求的前提下, 未添加亲和标签的β亚基与Ni-NTA、Cu-IDA及Zn-IDA金属离子亲和柱的亲和实验结果显示(图 2), Ni-NTA亲和实验中, 在200~500 mmol/L咪唑浓度下目的蛋白洗脱量最大, 低咪唑浓度对目的蛋白洗脱能力微弱; Cu- IDA与Zn-IDA的表现类似, 在咪唑物质的量浓度为10 mmol/L时, 二者均开始出现少量β亚基洗脱, 且随着咪唑浓度的逐渐增加, β亚基洗脱量逐步加大。质谱结果显示(数据未展示), 图 2中分子量50~ 70 kDa间的条带均为目的蛋白VGCC β亚基, 其呈现的分子量大小差异原因未知。综上, 长牡蛎VGCC β亚基C端天然存在的3组较为连续的组氨酸残基可与Ni2+、Cu2+、Zn2+等几种不同的金属离子亲和柱结合, 通过分析咪唑梯度洗脱得到的目的蛋白的相对比例和洗脱趋势, 后续实验均选用更易获得的Ni-NTA进行分析。
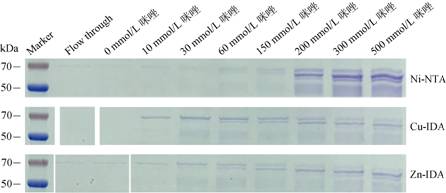 |
| 图 2 长牡蛎电压门控钙离子通道β亚基的不同金属离子亲和层析纯化 Fig. 2 Purification of voltage-gated calcium channel β subunit (Crassostrea gigas) with different metal ion affinity chromatography 注: 不同金属离子亲和层析纯化过程中, 不同浓度咪唑梯度洗脱样品的聚丙烯酰胺凝胶电泳结果, 使用考马斯亮蓝染色; Marker为蛋白分子量标准, Flow through为亲和层析流穿样品 |
为明确长牡蛎VGCC β亚基中参与Ni-NTA结合的关键氨基酸残基, 本研究对其C端3组较为连续的组氨酸残基(图 1)进行了分组突变, 并在相同的亲和层析纯化条件下, 选用不同浓度的咪唑(10~300 mmol/L)进行梯度洗脱。图 3a显示, 野生型β亚基与Ni-NTA的结合能力最强, 咪唑浓度为300 mmol/L时β亚基有峰值洗脱, 该洗脱浓度与多组氨酸标签亲和层析方法纯化蛋白通常使用的洗脱浓度相等, 显示其同Ni-NTA的结合能力与多组氨酸标签相当。相比之下, 突变型β亚基与Ni-NTA的结合能力显著下降, 且随着组氨酸残基突变数量的增多, 各突变体与Ni-NTA的结合能力逐渐变弱, 较低浓度咪唑即可洗脱大部分β亚基突变体。为了更好的对洗脱的β亚基进行量化和对比, 借助ImageJ对图 3a中的目的蛋白条带进行灰度值计算(表 2)。突变体M1在咪唑浓度为80 mmol/L时出现峰值洗脱; 突变体M2在60 mmol/L咪唑时出现峰值洗脱; 突变体M3的洗脱峰值对应的咪唑浓度为20 mmol/L。
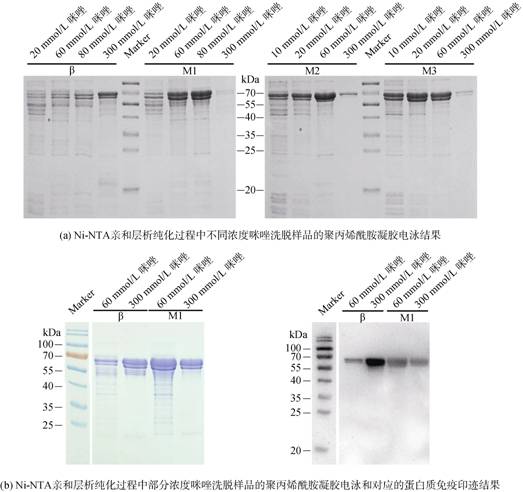 |
| 图 3 野生型及突变型长牡蛎电压门控钙离子通道β亚基的纯化 Fig. 3 Purification of wild-type and mutant voltage-gated calcium channel β subunit (Crassostrea gigas) 注: (a)为Ni-NTA亲和层析纯化过程中不同浓度咪唑洗脱样品的聚丙烯酰胺凝胶电泳结果, 使用考马斯亮蓝染色; (b)为Ni-NTA亲和层析纯化过程中部分浓度咪唑洗脱样品的聚丙烯酰胺凝胶电泳(左)和对应的蛋白质免疫印迹(右)结果, 聚丙烯酰胺凝胶电泳使用考马斯亮蓝染色, 蛋白质免疫印迹所用一抗为组氨酸标签特异性鼠源单抗, 二抗为HRP偶联的鼠IgG特异性抗体; Marker为蛋白分子量标准 |
| 样品 | 目的蛋白条带灰度值 | |||||
| 10 mmol/L咪唑 | 20 mmol/L咪唑 | 60 mmol/L咪唑 | 80 mmol/L咪唑 | 300 mmol/L咪唑 | ||
| β | — | 4.10 | 5.03 | 6.71 | 8.88 | |
| M1 | — | 5.72 | 13.80 | 15.91 | 0.70 | |
| M2 | 6.28 | 6.39 | 9.87 | — | 1.38 | |
| M3 | 8.52 | 10.73 | 8.89 | — | 0.70 | |
| 注: 上述为图 3a中目的蛋白条带灰度值的计算结果, 背景值为空白, 以“样品扣除空白后的灰度值与空白灰度值的比值”表示; “—”表示无该样品 | ||||||
选取图 3a中的部分样品进行蛋白质免疫印迹实验(图 3b)。M1虽然仍可与Ni-NTA有较好的结合, 但是其印迹信号较野生型β亚基明显减弱, 说明(HG)7序列中的组氨酸残基是多组氨酸标签抗体识别的关键位点。对剩余2组组氨酸残基进一步突变得到的M2、M3突变体难以检测到可靠免疫印迹信号(数据未展示)。
2.4 长牡蛎VGCC蛋白复合体的原位提取及免疫印迹检测长牡蛎是一种海洋软体动物, 并非模式动物, 其研究过程中, 常遇到缺少特异性抗体的情况。前述蛋白质免疫印迹结果显示, 重组表达的长牡蛎VGCC β亚基C端天然(HG)7序列能与多组氨酸标签抗体特异性结合, 因此, 在提取长牡蛎VGCC蛋白复合体时, 选用针对多组氨酸标签的一抗及相应二抗对VGCC蛋白复合体中β亚基进行免疫印迹分析, 可以监测蛋白复合体提取情况。结果显示, 在从长牡蛎闭壳肌中原位提取VGCC蛋白复合体的过程中, 天然存在的β亚基仍然具有明确的免疫印迹信号。在提取过程中, 经超速离心和去垢剂增溶后, 水溶性的β亚基主要位于膜组分中, 部分证明了VGCC蛋白复合体的完整性。作为VGCC蛋白复合体的一部分, β亚基的含量是VGCC蛋白复合体含量的合理指征, 分析β亚基的含量, 可以辅助优化VGCC蛋白复合体的提取条件。据图 4中β亚基含量分布, digitonin是更适合VGCC复合体增溶的去垢剂。
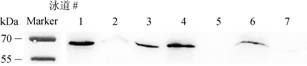 |
| 图 4 长牡蛎电压门控钙离子通道蛋白复合体的原位提取 Fig. 4 Extraction of voltage-gated calcium channel complex of Crassostrea gigas 注: 泳道1为细胞裂解后6 450 r/min离心的上清; 泳道2为细胞裂解后6 450 r/min离心的沉淀; 泳道3为41 500 r/min超速离心后的上清; 泳道4为去垢剂digitonin增溶后73 400 r/min超速离心的上清; 泳道5为去垢剂digitonin增溶后73 400 r/min超速离心的沉淀; 泳道6为去垢剂GDN增溶后73 400 r/min超速离心的上清; 泳道7为去垢剂GDN增溶后73 400 r/min超速离心的沉淀 |
蛋白质纯化是生命科学研究中重要的一环, 方法多样, 应用灵活。在实际研究中, 为满足蛋白质的纯度要求以及更好地适应下游需求, 研究者往往会选用多种纯化方法综合应用, 如先借助亲和层析去除绝大部分的杂蛋白, 然后根据蛋白质分子量的不同通过分子排阻层析对样品进一步分离, 使目的蛋白的纯度逐步提升。选用亲和层析进行蛋白纯化时, 目的蛋白与亲和柱结合的特异性以及亲和力均为重要的考虑因素。一般来说特异性越高、亲和力越强, 越有利于通过淋洗去除更多的杂质, 获得足量高纯度的蛋白。在目的蛋白的C/N端融合外源亲和标签是实现上述目标的有效方法。
当目的蛋白具有与亲和标签相似的氨基酸序列特征时, 能否利用该序列特征实现高效的无标签亲和层析纯化是本研究的重点。结果表明, 长牡蛎VGCC β亚基C端天然存在的富含组氨酸残基的序列能够满足Ni-NTA亲和层析的要求。而在该蛋白C端和N端分别添加多组氨酸标签后, 相同亲和层析条件下, 无论是纯度还是得率均无显著优势(数据未展示)。
Wu等人[36]在对兔子VGCC蛋白复合体进行纯化时, 为减少样品损失省去了分子排阻层析的步骤, 由此可见, 在样品量受限的情况下, 通过减少纯化步骤提高最终产量是必要的。无标签亲和层析法利用目的蛋白自身氨基酸序列特点, 避免了融合标签使用后对目的蛋白结构和功能的影响, 省去了酶切去除外源标签的步骤, 减少了对目的蛋白产量的影响。
蛋白复合体亚基数量多, 表达纯化复杂, 常用原位提取的方法, 无法依赖外源标签, 且非模式生物来源的蛋白质缺乏特异性抗体, 难以通过常用的免疫印迹方法对提取过程进行监测。本研究的实验结果显示, 原位提取的VGCC蛋白复合体中存在的β亚基具有明确的免疫印迹信号, 成为筛选增溶去垢剂等提取条件优化的重要参考, 可以在非模式生物蛋白复合体研究中发挥重要作用。
无标签亲和层析纯化的方法也存在一定的局限性。本研究结果显示, 长牡蛎VGCC β亚基M2、M3突变体蛋白仍可与Ni-NTA结合, 但难以获得可靠免疫印迹信号。因此, 能与Ni-NTA亲和柱结合的蛋白未必可以借助多组氨酸标签的抗体进行免疫印迹检测, 可能由于免疫印迹方法对组氨酸残基的连续性要求更高。
4 结论本研究选取C端天然富含组氨酸残基的长牡蛎电压门控钙离子通道β亚基, 开展了无标签亲和层析纯化。结果表明, 长牡蛎VGCC β亚基可与Ni2+、Cu2+、Zn2+等多种金属离子亲和柱有效结合, 其中与Ni-NTA的结合能力最强, 组氨酸残基的数量与结合能力正相关。另外, 本研究发现针对多组氨酸标签的抗体可用于(HG)7序列的蛋白质免疫印迹分析, 并将其进一步应用于VGCC蛋白复合体的提取过程中。本研究为具有相似序列特征的蛋白质纯化提供了重要参考, 也为缺少特异性抗体的蛋白复合体的原位提取过程监测提供了思路。
| [1] |
NEVONE A, MERLINI G, NUVOLONE M. Treating protein misfolding diseases: therapeutic successes against systemic amyloidoses[J]. Frontiers in Pharmacology, 2020, 11: 1024. DOI:10.3389/fphar.2020.01024 |
| [2] |
CHAUDHURI T K, PAUL S. Protein-misfolding diseases and chaperone-based therapeutic approaches[J]. FEBS Journal, 2006, 273(7): 1331-1349. DOI:10.1111/j.1742-4658.2006.05181.x |
| [3] |
GRÄSLUND S, NORDLUND P, WEIGELT J, et al. Protein production and purification[J]. Nature Methods, 2008, 5(2): 135-146. DOI:10.1038/nmeth.f.202 |
| [4] |
THEKKILAVEEDU S, KRISHNASWAMI V, MOHANAN D P, et al. Lactic acid-mediated isolation of alpha-, beta- and kappa-casein fractions by isoelectric precipitation coupled with cold extraction from defatted cow milk[J]. International Journal of Dairy Technology, 2019, 73(1): 31-39. |
| [5] |
RAJESH S, CRANDALL C, SCHNEIDERMAN S, et al. Cellulose-graft-polyethyleneamidoamine anion-exchange nanofiber membranes for simultaneous protein adsorption and virus filtration[J]. ACS Applied Nano Materials, 2018, 1(7): 3321-3330. DOI:10.1021/acsanm.8b00519 |
| [6] |
CAI W X, TUCHOLSKI T, CHEN B F, et al. Top-down proteomics of large proteins up to 223 kDa enabled by serial size exclusion chromatography strategy[J]. Analytical Chemistry, 2017, 89(10): 5467-5475. DOI:10.1021/acs.analchem.7b00380 |
| [7] |
LIU S X, LI Z H, YU B, et al. Recent advances on protein separation and purification methods[J]. Advances in Colloid and Interface Science, 2020, 284: 102254. DOI:10.1016/j.cis.2020.102254 |
| [8] |
LI B L, GUO F, HU H, et al. The characterization of column heating effect in nanoflow liquid chromatography mass spectrometry (nanoLC-MS)-based proteomics[J]. Journal of Mass Spectrometry, 2020, 55(1): e4441. |
| [9] |
XING J, WANG M H, WANG S, et al. Analysis of proteins by capillary electrophoresis with a novel diazoresin/β-Cyclodextrin covalent capillary coating method[J]. Ferroelectrics, 2020, 563(1): 45-51. DOI:10.1080/00150193.2020.1760608 |
| [10] |
AYYAR B V, ARORA S, MURPHY C, et al. Affinity chromatography as a tool for antibody purification[J]. Methods, 2012, 56(2): 116-129. DOI:10.1016/j.ymeth.2011.10.007 |
| [11] |
ZHAO X Y, LI G S, LIANG S F. Several affinity tags commonly used in chromatographic purification[J]. Journal of Analytical Methods in Chemistry, 2013, 2013: 581093. |
| [12] |
LICHTY J J, MALECKI J L, AGNEW H D, et al. Comparison of affinity tags for protein purification[J]. Protein Expression and Purification, 2005, 41(1): 98-105. DOI:10.1016/j.pep.2005.01.019 |
| [13] |
WAUGH D S. Making the most of affinity tags[J]. Trends in Biotechnology, 2005, 23(6): 316-320. DOI:10.1016/j.tibtech.2005.03.012 |
| [14] |
NOROUZI R, HOJATI Z, BADR Z. Overview of the recombinant proteins purification by affinity tags and tags exploit systems[J]. Journal of Fundamental and Applied Sciences, 2016, 8(3S): 90-104. |
| [15] |
HARPER S, SPEICHER D W. Purification of proteins fused to glutathione S-transferase[J]. Methods in Molecular Biology, 2011, 681: 259-280. |
| [16] |
WAUGH D S. An overview of enzymatic reagents for the removal of affinity tags[J]. Protein Expression and Purification, 2011, 80(2): 283-293. DOI:10.1016/j.pep.2011.08.005 |
| [17] |
UHLÉN M, FORSBERG G, MOKS T, et al. Fusion proteins in biotechnology[J]. Current Opinion in Biotechnology, 1992, 3(4): 363-369. DOI:10.1016/0958-1669(92)90164-E |
| [18] |
RIGUERO V, CLIFFORD R, DAWLEY M, et al. Immobilized metal affinity chromatography optimization for poly-histidine tagged proteins[J]. Journal of Chromatography A, 2020, 1629: 461505. DOI:10.1016/j.chroma.2020.461505 |
| [19] |
KIMPLE M E, BRILL A L, PASKER R L. Overview of affinity tags for protein purification[J]. Current Protocols in Protein Science, 2013, 73: 9.9.1-9.9.26. |
| [20] |
BOOTH W T, SCHLACHTER C R, POTE S, et al. Impact of an N-terminal polyhistidine tag on protein thermal stability[J]. ACS Omega, 2018, 3(1): 760-768. DOI:10.1021/acsomega.7b01598 |
| [21] |
MAJOREK K A, KUHN M L, CHRUSZCZ M, et al. Double trouble-buffer selection and his-tag presence may be responsible for nonreproducibility of biomedical experiments[J]. Protein Science, 2014, 23(10): 1359-1368. DOI:10.1002/pro.2520 |
| [22] |
马晓川, 费浩. 金属配位在多肽与蛋白质研究中的应用[J]. 化学进展, 2016, 28(2/3): 184-192. MA Xiaochuan, FEI Hao. The use of metal coordination in peptide and protein research[J]. Progress in Che mistry, 2016, 28(2/3): 184-192. |
| [23] |
WEISKIRCHEN R, MOSER M, WEISKIRCHEN S, et al. LIM-domain protein cysteine- and glycine-rich protein 2 (CRP2) is a novel marker of hepatic stellate cells and binding partner of the protein inhibitor of activated STAT1[J]. Biochemical Journal, 2001, 359: 485-496. DOI:10.1042/bj3590485 |
| [24] |
REN D, PENNER N A, SLENTZ B E, et al. Histidine- rich peptide selection and quantification in targeted proteomics[J]. Journal of Proteome Research, 2004, 3(1): 37-45. DOI:10.1021/pr034049q |
| [25] |
CUN S J, LI H Y, GE R G, et al. A histidine-rich and cysteine-rich metal-binding domain at the C terminus of heat shock protein A from Helicobacter pylori: implication for nickel homeostasis and bismuth susceptibility[J]. Journal of Biological Chemistry, 2008, 283(22): 15142-15151. DOI:10.1074/jbc.M800591200 |
| [26] |
SINGH B, MCCAFFERY J N, KONG A, et al. Purification of native histidine-rich protein 2 (nHRP2) from Plasmodium falciparum culture supernatant, infected RBCs, and parasite lysate[J]. Malaria Journal, 2021, 20(1): 405. DOI:10.1186/s12936-021-03946-1 |
| [27] |
PATEL K K, POON I K, TALBO G H, et al. New method for purifying histidine-rich glycoprotein from human plasma redefines its functional properties[J]. IUBMB Life, 2013, 65(6): 550-563. DOI:10.1002/iub.1168 |
| [28] |
HALL T A. BioEdit: A user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nuclc Acids Symposium Series, 1999, 41: 95-98. |
| [29] |
MADEIRA F, PARK Y M, LEE J, et al. The EMBL- EBI search and sequence analysis tools APIs in 2019[J]. Nucleic Acids Research, 2019, 47(W1): W636-W641. DOI:10.1093/nar/gkz268 |
| [30] |
JACOBUS A P, GROSS J. Optimal cloning of PCR fragments by homologous recombination in Escherichia coli[J]. PloS One, 2015, 10(3): e0119221. DOI:10.1371/journal.pone.0119221 |
| [31] |
WU J P, YAN Z, LI Z Q, et al. Structure of the voltage-gated calcium channel Cav1.1 complex[J]. Science, 2015, 350(6267): aad2395. DOI:10.1126/science.aad2395 |
| [32] |
MAHMOOD T, YANG P C. Western blot: technique, theory, and trouble shooting[J]. North American Journal of Medical Sciences, 2012, 4(9): 429-434. DOI:10.4103/1947-2714.100998 |
| [33] |
ZHAO Y Y, HUANG G X, WU J P, et al. Molecular basis for ligand modulation of a mammalian voltage-gated Ca2+ channel[J]. Cell, 2019, 177(6): 1495-1506. DOI:10.1016/j.cell.2019.04.043 |
| [34] |
CHEN Y H, LI M H, ZHANG Y, et al. Structural basis of the alpha1-beta subunit interaction of voltage-gated Ca2+ channels[J]. Nature, 2004, 429(6992): 675-680. DOI:10.1038/nature02641 |
| [35] |
PAULSEN I T, SAIER M H. A novel family of ubiquitous heavy metal ion transport proteins[J]. Journal of Membrane Biology, 1997, 156(2): 99-103. DOI:10.1007/s002329900192 |
| [36] |
WU J P, YAN Z, LI Z Q, et al. Structure of the voltage-gated calcium channel Ca(v)1.1 at 3.6 A resolution[J]. Nature, 2016, 537(7619): 191-196. DOI:10.1038/nature19321 |
 2022, Vol. 46
2022, Vol. 46


