文章信息
- 宋崇阳, 张思勉, 李永闯, 张攀, 赖晓芳, 王攀攀, 于飞, 高焕, 阎斌伦. 2022.
- SONG Chong-yang, ZHANG Si-mian, LI Yong-chuang, ZHANG Pan, LAI Xiao-fang, WANG Pan-pan, YU Fei, GAO Huan, YAN Bin-lun. 2022.
- 脊尾白虾Dnmt2启动子克隆及其转录调控分析
- Cloning and transcriptional regulation of Dnmt2 promoter in Exopalaemon carinicauda
- 海洋科学, 46(5): 95-103
- Marine Sciences, 46(5): 95-103.
- http://dx.doi.org/10.11759/hykx20211207001
-
文章历史
- 收稿日期:2021-12-07
- 修回日期:2022-02-22
2. 江苏省海洋生物产业技术协同创新中心, 江苏 连云港 222005;
3. 江苏省农业种质资源保护与利用平台, 江苏 南京 210014;
4. 连云港市海洋与渔业发展促进中心, 江苏 连云港 222005
2. Co-Innovation Center of Jiangsu Marine Bio-industry Technology, Lianyungang 222005, China;
3. The Jiangsu Provincial Infrastructure for Conservation and Utilization of Agricultural Germplasm, Nanjing 210014, China;
4. Lianyungang Marine and Fishery Development Promotion Center, Lianyungang 222005, China
DNA甲基化是由DNA甲基转移酶(DNA methyltransferase, DNMT)以活性甲基化合物S-腺苷甲硫氨酸(SAM)为甲基供体, 将甲基基团催化并转移到DNA特定碱基上的表观遗传修饰过程[1], 在基因印迹[2]、X染色体失活[3]和维持染色体结构完整性[4]等方面发挥重要作用。DNA甲基转移酶在生物机体内由甲基化转移酶基因表达而来, 已知生物基因组中含有多种DNA甲基化转移酶基因, 包括Dnmt1、Dnmt2和Dnmt3等[5], 其中DNA甲基转移酶2基因(DNA methyltransferase2, Dnmt2)在各类真核生物中广泛存在, 含有DNA甲基转移酶的所有特征性保守基序, 是DNA甲基转移酶基因家族进化过程中最保守的家族成员[6]。研究发现, DNMT2还具有催化tRNA甲基化的功能[7]。在罗氏沼虾(Macrobrachium rosenbergii)中的研究显示, Dnmt2在卵巢中的表达量显著高于其他组织, 胚胎早期发育阶段中表达量显著高于其他发育阶段, 表明其可能在罗氏沼虾早期胚胎发育中起调节作用[8]。对三疣梭子蟹(Portunus trituberculatus) Dnmt2基因于不同组织及发育阶段的表达分析中同样发现, Dnmt2在三疣梭子蟹胚胎、幼体和性腺发育调控中可能具有重要作用[9]。基因的表达受多种调控机制影响, 启动子是最主要的调控方式之一, 真核生物的启动子是一段位于结构基因5′端上游的DNA序列, 含有RNA聚合酶特异性结合和转录起始所需的保守序列, 决定基因转录起始的位置[10]。通常情况下, 转录因子通过与启动子上特定序列的结合, 将来自细胞表面的信息传递至核内基因, 促进或抑制相关蛋白的转录[11], 启动子区域的元件组成和其具有的功能对于基因的表达和转录环节的调控至关重要[12, 13]。
目前, 有关Dnmt2转录调控的研究较少, 尤其在甲壳动物中。脊尾白虾(Exopalaemon carinicauda)广泛分布于中国沿海海域, 具有繁殖能力强、生长周期短、环境适应性广的特点。近年来, 在传统虾类养殖难度增加、利润下降的情况下, 脊尾白虾养殖产量增长迅速, 已成为中国沿海地区池塘单养或混养的重要品种[14, 15]。本课题组前期克隆获得脊尾白虾Dnmt2 cDNA全长序列1 321 bp, 同源序列比对发现与罗氏沼虾Dnmt 2同源性最高为78.67%, 进一步通过构建NJ系统进化树, 显示与罗氏沼虾Dnmt 2聚为一支。在此基础上, 本研究通过染色体步移技术克隆得到脊尾白虾Dnmt2启动子序列, 运用生物信息学方法预测转录因子结合位点和CpG岛位点, 构建了相应的报告基因验证启动子活性, 为进一步揭示Dnmt2转录调控的分子机制奠定理论基础。
1 材料与方法 1.1 实验材料实验用脊尾白虾为实验室自繁而来个体, HEK293细胞为本实验室保存。
1.2 实验方法取脊尾白虾新鲜肌肉组织, 液氮研磨后用动物基因组DNA快速抽提试剂盒(生工生物工程)提取DNA。根据本课题组克隆获得的脊尾白虾Dnmt2 cDNA全长序列, 利用PrimerPremier 5.0设计步移引物(表 1), 由生工生物工程(上海)股份有限公司合成。使用Genome Walking Kit(TaKaRa)试剂盒对Dnmt2启动子序列进行克隆。
| 引物名称 | 引物序列(5′→3′) | 片段长度/bp | 退火温度/℃ | 延伸/s | 用途 |
| 1st-SP1 | CTGAGACAGTTTTTGAAGAAGATCA | 1 197 | 60.1 | 2 min | 第一次步移扩增 |
| 1st-SP2 | ATCCGGAGCAAAAGCTTCCAGATTG | 68.9 | |||
| 1st-SP3 | GTGTTGATTTCGTAGCTTCCGACGA | 65.7 | |||
| 2nd-SP1 | CGTTTCACAACATTTTCCAACGCCT | 614 | 68.4 | 2 min | 第二次步移扩增 |
| 2nd-SP2 | CCAGAAGTGTTAAAACCTGAACGAG | 61.7 | |||
| 2nd-SP3 | AGAATAGCGGTTGCCTATCACCATG | 67.8 |
利用在线分析网站BDGP(https://www.fruitfly.org/seq_tools/promoter.html)预测Dnmt2启动子序列起始位点, 使用JASPAR(https://jaspar.genereg.net/)、TFBIND(https://tfbind.hgc.jp/)和AnimalTFDB3.0(http://bioinfo.life.hust.edu.cn/AnimalTFDB/#!/)预测分析启动子转录调控位点, 通过MethPrimer(http://www.urogene.org/cgi-bin/methprimer/methprimer.cgi)预测CpG岛。
1.4 启动子活性分析 1.4.1 启动子不同截短体的构建根据克隆得到的脊尾白虾Dnmt2的启动子序列, 设计启动子区域不同长度片段扩增引物, 引物序列见表 2。上下游引物5′端分别添加MluI和SmaI酶切位点(加粗字母表示), 酶切位点前添加保护碱基, 由生工生物工程(上海)股份有限公司合成。
| 片段名称 | 引物名称 | 引物序列(5′→3′) | 片段长度/ bp | 退火温度/℃ | 延伸/s | 扩增区域/bp |
| D1 | D-F-1 | CGACGCGTGAATTTAGTCGTAAGAATTTTCTTA | 307 | 65 | 30 s | –215~+92 |
| D-R-1 | TCCCCCGGGATGGAGTGTCACAAACAGTCACTTT | |||||
| D2 | D-F-2 | CGACGCGTAGAGCGATTTACTCGTTCAGGTTTT | 533 | 68.1 | 40 s | –452~+81 |
| D-R-2 | TCCCCCGGGCAAACAGTCACTTTACTATTATCTA | |||||
| D3 | D-F-2 | CGACGCGTAGAGCGATTTACTCGTTCAGGTTTT | 346 | 68.1 | 30 s | –452~–106 |
| D-R-3 | TCCCCCGGGGTACTGTACTGTATGGTTTTCATTC | |||||
| D4 | D-F-2 | CGACGCGTAGAGCGATTTACTCGTTCAGGTTTT | 256 | 68.1 | 25 s | –452~–196 |
| D-R-4 | TCCCCCGGGAAATTCTTACGACTAAATTCACACA | |||||
| T1 | T-F-1 | CGACGCGTGAACCAACTAAACATAATCTTCCAG | 1 407 | 65.5 | 1 min30 s | –1315~+92 |
| D-R-1 | TCCCCCGGGATGGAGTGTCACAAACAGTCACTTT | |||||
| T2 | T-F-2 | CGACGCGTTTGTTCGTACATAGATACAAACCAT | 1 032 | 63.7 | 1 min15 s | –940~+92 |
| D-R-1 | TCCCCCGGGATGGAGTGTCACAAACAGTCACTTT | |||||
| T3 | T-F-3 | CGACGCGTATTGATTTTATTCTGGATAAGTATT | 732 | 64.8 | 1 min | –640~+92 |
| D-R-1 | TCCCCCGGGATGGAGTGTCACAAACAGTCACTTT | |||||
| T4 | D-F-2 | CGACGCGTAGAGCGATTTACTCGTTCAGGTTTT | 544 | 68.1 | 45 s | –452~+92 |
| D-R-1 | TCCCCCGGGATGGAGTGTCACAAACAGTCACTTT | |||||
| 注: 加粗字母表示酶切位点 | ||||||
以提取的DNA为模板, 使用Dnmt2启动子不同长度片段的扩增引物进行PCR扩增, 得到片段D1、D2、D3、D4和T1、T2、T3、T4。使用MluI和SmaI快切酶(生工生物工程, 上海)分别双酶切片段D1~D4、T1~T4和pGL3-Basic质粒, 利用T4连接酶连接构建重组质粒, 并转化至DH5α感受态细胞(Vazyme, China), 将菌液PCR筛选得到的阳性克隆送至生工生物工程(上海)股份有限公司进行测序。利用质粒小提试剂盒(生工生物工程, 上海)提取测序结果正确的阳性质粒, 使用MluI和SmaI快切酶进行双酶切验证, 将构建成功的重组质粒分别命名为pGL3-Basic-D1~D4和pGL3-Basic- T1~T4。
1.4.2 细胞培养及瞬时转染使用含有10%血清、1%双抗(青霉素和链霉素)的DMEM完全培养基(生工生物工程, 上海), 于37℃、5% CO2培养箱中培养HEK293细胞。细胞生长至指数生长期时, 接种于24孔板中, 待细胞完全贴壁生长至约80%汇合时, 使用转染试剂ExFect Transfection Reagent (Vazyme, China)进行瞬时转染, 内参质粒为pRL-TK, 分别将重组质粒和空载质粒与内参质粒以100︰1的比例共转染到细胞中, 每个样品做3次重复, 转染32 h后收集细胞, 使用Dual Luciferase Reporter Assay Kit (Vazyme, China)报告基因活性检测试剂盒进行双荧光素酶活性检测。
1.4.3 双荧光素酶活性测定待转染32 h后, 吸弃细胞培养基, 用PBS洗涤两次, 每个孔加入100 μL细胞裂解缓冲液, 室温静置裂解5 min, 吹打并吸取细胞裂解产物至1.5 mL离心管中, 12 000 g常温离心2 min后, 取上清用于后续检测。首先进行萤火虫荧光素酶反应检测: 将100 μL平衡至室温的荧光素底物加入检测管中, 小心吸取20 μL细胞裂解上清至检测管中, 迅速混匀后立即于发光检测仪(Promega GloMax 20/20)中检测萤火虫荧光素酶报告基因活性。再进行海肾荧光素酶反应检测: 在以上反应液中加入100 μL新鲜配制的海肾底物工作液, 迅速混匀后立即于发光检测仪中检测海肾荧光素酶报告基因活性, 记录结果。
1.4.4 统计分析启动子的相对活性用萤火虫荧光素酶的值/海肾荧光素酶的值表示。实验数据利用SPSS22.0进行统计学分析, 使用Origin9.0进行绘图。
2 结果与分析 2.1 脊尾白虾Dnmt2启动子序列的克隆和生物信息学分析通过染色体步移克隆获得Dnmt2启动子序列, 从翻译起始密码子(ATG)起向上游共1 551 bp。对序列进行生物信息学分析, 结果显示: 转录起始位点是位于ATG上游的第236个碱基“A”, –26 bp处发现TATA-box, 分析启动子序列发现, Dnmt2启动子含有多个转录因子结合位点, 包括STAT3、NF-κB、E2F、GATA-1和SOX等(图 1)。用在线软件MethPrimer预测启动子区域CpG岛(检索标准: Islandsize > 100.0、GCPercent > 50.0、Obs/Exp > 0.6), 结果显示本研究获得的Dnmt2启动子区域中不含CpG岛。
 |
| 图 1 脊尾白虾Dnmt2近端启动子转录因子结合位点预测 Fig. 1 Prediction of transcription factor binding sites in the proximal promoter of Dnmt2 in E. carinicauda 黑色方框标记转录起始位点(TSS); 黑色加粗字体标记起始密码子; 阴影、下划线标记转录因子 The black box marks the transcription initiation site (TSS); Black bold font marks the start codon; shaded and underlined indicate transcription factors |
以脊尾白虾基因组DNA为模板, 用引物D-F-1和D-R-1扩增片段D1(–215 bp~+92 bp), 引物D-F-2分别于D-R-2、D-R-3和D-R-4扩增片段D2(–452 bp~+81 bp), D3(–452 bp~–106 bp)和D4(–452 bp~–196 bp), 引物T-F-1, T-F-2, T-F-3和D-F-2分别与D-R-1扩增片段T1(–1315 bp~+92 bp), T2(–940 bp~+92 bp), T3(–640 bp~ +92 bp)和T4(–452 bp~+92 bp), PCR产物大小分别与预期设计的长度一致(图 2), 表明成功获得目的DNA片段。
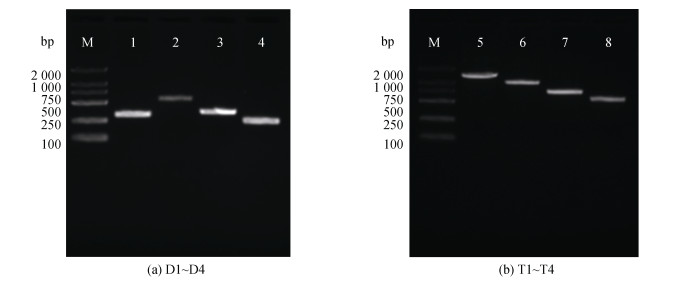 |
| 图 2 脊尾白虾Dnmt2启动子不同长度片段的扩增(M. DL2000 DNA Marker) Fig. 2 Amplification of Dnmt2 promoter deletion fragments of the E. carinicauda |
用MluI和SmaI快切酶双酶切各表达载体PGL3- Basic-D1~D4以及PGL3-Basic-T1~T4, 电泳检测结果(图 3)显示, 有相应的载体和启动子片段2条带, 说明表达载体构建成功。
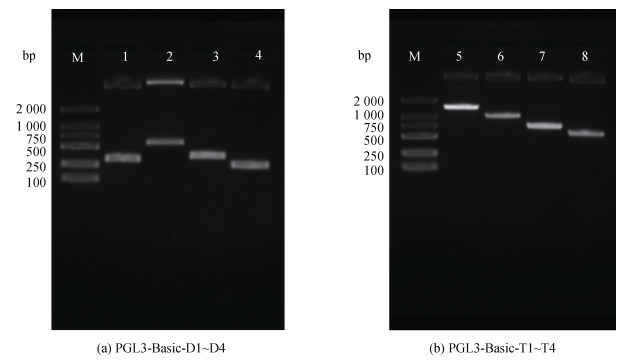 |
| 图 3 脊尾白虾Dnmt2启动子不同长度片段的扩增(M.DL2000 DNA Marker) Fig. 3 Amplification of Dnmt2 promoter recombinant plasmids of the E. carinicauda |
将构建的双荧光素酶报告基因重组质粒pGL3- Basic-D1~D4(图 4)和空载质粒pGL3-Basic分别与内参质粒pRL-TK共转染至HEK 293细胞, 转染32 h后, 收集细胞检测荧光值, 将检测的结果进行单因素方差分析。结果显示, 重组质粒pGL3-D4荧光活性与对照质粒pGL3-Basic无显著性差异(P < 0.05), pGL3-Basic-D1、pGL3-Basic-D2和pGL3-Basic-D3与对照质粒pGL3-Basic均有显著性差异(P < 0.05), 其中pGL3-Basic-D2的相对荧光活性最高, pGL3- Basic-D1和pGL3-Basic-D3活性次之(图 5)。结果表明–196bp~+81bp能够维持Dnmt2启动子基本转录活性, 为该基因核心启动子区域。
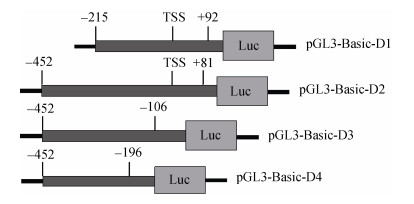 |
| 图 4 质粒pGL3-Basic-D1~D4简图 Fig. 4 Diagram of plasmids pGL3-Basic-D1~D4 |
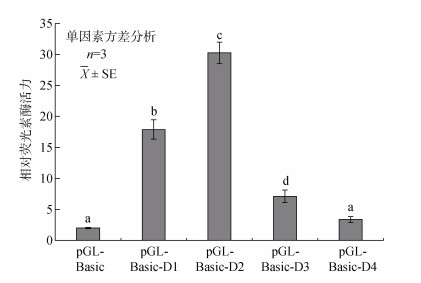 |
| 图 5 脊尾白虾Dnmt2启动子含转录起始位点和不含转录起始位点活性分析 Fig. 5 Activity analysis of the fragments containing and not containing transcription start site of the E. carinicauda Dnmt2 promoter 数据以平均值±标注误表示(每个样品3个重复); 不同小写字母表示差异显著(P < 0.05), 图 6同 Date are described as Mean±SE (n=3); Different small letters above the bars indicate significant differences (P < 0.05), the same as Fig.6 |
对Dnmt2启动子不同长度片段的双荧光素酶报告基因表达载体进行活性检测。结果显示, 重组质粒pGL3-Basic-T1、pGL3-Basic-T2、pGL3-Basic-T3、pGL3-Basic-T4转染32 h后的活性较对照质粒pGL3- Basic均显著性增强(P < 0.05)。pGL3-Basic-T3的相对荧光活性显著高于其他组(P < 0.05), pGL3- Basic-T1和pGL3-Basic-T2两组之间活性无显著性差异(P < 0.05), pGL3-Basic-T4的相对荧光活性显著低于pGL3-Basic- T3(P < 0.05)(图 6b)。结果说明, 在脊尾白虾Dnmt2启动子–640 bp~–452 bp区域可能存在促进该基因表达的转录调控元件, 在–940 bp~–640 bp区域的转录调控元件表现出抑制该基因表达的作用。
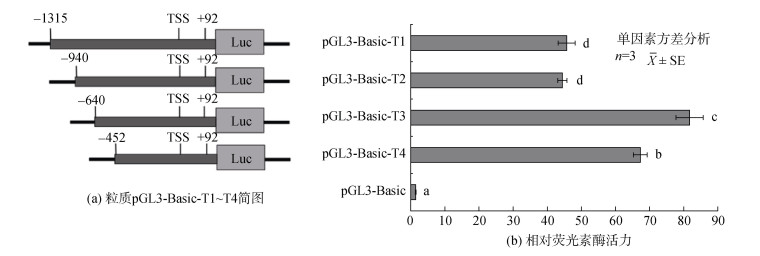 |
| 图 6 脊尾白虾Dnmt2启动子不同长度片段示意图及活性分析 Fig. 6 Diagrammatic drawing (a) and activity analysis (b) of deletion fragments of the E. carinicauda Dnmt2 promoter |
本研究获得脊尾白虾Dnmt2 5′端上游1 315 bp启动子序列, 转录起始位点为翻译起始密码子ATG上游236 bp处的碱基“A”, 在其上游26 bp处发现TATA-box转录结合位点。TATA-box为真核生物启动子序列关键的调控元件, 通常位于转录起始位点前25~30 bp区域内[16], 能与RNA聚合酶Ⅱ特异性结合形成转录前起始复合物(Transcription pre-initiation com plex, PIC), 调节转录起始。TATA-box一旦发生突变将导致转录效率降低, 甚至转录被阻止[17]。而另有研究发现, 缺少TATA-box的启动子也可以调节基因的组织特异性表达[18]。在甲壳类动物中, TATA-box调控基因表达的机制还需要进一步研究。
经在线软件预测, Dnmt2启动子区域含有STAT3、NF-кB、E2F、SOX、GATA-1等多种转录因子结合位点。其中, SOX转录因子广泛参与生物体早期胚胎及神经系统发育的调控[19], 并在性别决定和分化过程中发挥重要作用[20]。STAT3转录因子具有信号转导和转录激活的双重作用, 是JAK-STAT信号途径的重要环节[21]。同时也在胚胎早期发育的过程中扮演重要角色, 在小鼠(Mus musculus)的试验中表明, STAT3转录因子对于大鼠胚胎早期发育和存活至关重要[22]。转录因子E2F家族可以通过调节细胞DNA合成以及与细胞增殖有关的基因表达参与细胞周期的调控, 在细胞增殖、分化和凋亡的过程中发挥作用[23]。以上转录因子的存在, 提示Dnmt2启动子有可能参与脊尾白虾早期胚胎发育及性腺发育过程。
CpG岛通常位于基因的启动子和第一个外显子区域[24], 这些区域的DNA甲基化与转录激活密切相关[25]。将本研究获得的启动子进行软件分析, 发现其不含CpG岛。统计显示, 大约有40%的哺乳动物基因在其启动子和外显子区域中包含CpG岛[26], 提示脊尾白虾Dnmt2启动子可能不含CpG岛, 也另有可能是因目前获得的脊尾白虾Dnmt2启动子太短, 需进一步向上游扩增获得更长的序列再进行CpG岛检测。
3.2 核心启动子区分析核心启动子区是指在RNA聚合酶及转录因子共同作用下精确指导转录起始的最小一段DNA序列, 包含TATA-box、TFIIB识别元件和以转录起始位点为中心的起始子等结构, 是基因准确有效转录的核心区域[27]。本研究的核心启动子区验证结果显示–196~ +81 bp能够维持Dnmt2启动子基本转录活性, 为该基因核心启动子区域。在DNA甲基转移酶基因家族成员Dnmt1启动子的研究中, 小鼠Dnmt1核心启动子被发现位于–1 866~–1 734 bp区域内[28]。因此, 不同基因核心启动子在结构和功能上会存在差异[29]。
3.3 启动子调控原件分析为检测Dnmt2启动子区域活性, 本研究同时构建了4个启动子逐渐缺失片段表达载体, 用萤火虫荧光素酶和海肾荧光素酶双报告基因检测各缺失片段表达载体的转录活性。结果发现, 在脊尾白虾Dnmt2启动子–640b~–452 bp区域可能存在促进该基因表达的转录调控原件, 经在线软件分析, 在–640~–452 bp内发现GATA-1转录因子结合位点, 表明GATA-1转录因子可能正调控Dnmt2表达, 这与GATA-1转录因子在中国明对虾(Fenneropenaeus chinensis) βGBP-HDL基因启动子中表现出的正调控作用一致[30]。在胚胎发育过程中, 组织器官的形成依赖于干细胞正确的定向分化以及中间细胞的凋亡, 在这一精确调控过程中, GATA转录因子家族扮演了不可或缺的角色[31-32]。另有研究表明, Dnmt2在斑马鱼(Danio rerio)胚胎发育期敲除后, 导致斑马鱼视网膜、肝脏和大脑出现分化上的缺陷[33]。以上结果进一步提示Dnmt2可能参与调控脊尾白虾早期胚胎发育过程。本研究中, Dnmt2启动子活性并没有随序列长度的增加而上升, 长片段pGL3-Basic-T1和pGL3-Basic-T2与较短片段pGL3-Basic-T3的转录活性相比显著性下降(P < 0.05)。在小鼠Dnmt1、Dnmt3a和Dnmt3b启动子研究结果中[34], 转录因子对小鼠每种DNA甲基转移酶基因启动子片段的调节作用各不相同; 但从整体上看, 不同转录因子对各DNA甲基转移酶基因启动子区域的长启动子片段都表现出抑制作用, 这与我们的实验结果一致。接下来可利用点突变、EMSA和ChIP等方法进一步研究Dnmt2启动子中起关键调控作用的转录因子。以上结果为探明脊尾白虾Dnmt2启动子的表达调控网络奠定理论基础。
| [1] |
LYKO F. The DNA methyltransferase family: a versatile toolkit for epigenetic regulation[J]. Nature Reviews Genetics, 2018, 19(2): 81-92. DOI:10.1038/nrg.2017.80 |
| [2] |
SAZHENOVA E A, LEBEDEY I N. Evolutionary aspects of genomic imprinting[J]. Molecular Biology, 2021, 55(1): 1-15. DOI:10.1134/S0026893320060102 |
| [3] |
DEOBAGKAR D. Epigenetics with special reference to the human X chromosome inactivation and the enigma of Drosophila DNA methylation[J]. Journal of Genetics, 2018, 97(2): 371-378. DOI:10.1007/s12041-018-0937-5 |
| [4] |
KIM S, KAANG B K. Epigenetic regulation and chromatin remodeling in learning and memory[J]. Experimental & Molecular Medicine, 2017, 49(1): e281. |
| [5] |
EDWARDS J R, YARYCHKIVSKA O, BOULARD M, et al. DNA methylation and DNA methyltransferases[J]. Epigenetics & Chromatin, 2017, 10(1): 23. |
| [6] |
ASHAPKIN V V, KUTUEVA L I, VANYUSHIN B F. Dnmt2 is the most evolutionary conserved and enigmatic cytosine DNA methyltransferase in eukaryotes[J]. Russian Journal of Genetics, 2016, 52(3): 237-248. DOI:10.1134/S1022795416030029 |
| [7] |
JELTSCH A, EHRENHOFER-MURRAY A, JURKOWSKI T, et al. Mechanism and biological role of Dnmt2 in Nucleic acid methylation[J]. RNA Biology, 2017, 14(9): 1108-1123. DOI:10.1080/15476286.2016.1191737 |
| [8] |
FENG C Z, DING X, LI Y, et al. The DNA methyltransferase-2 gene in the prawn Macrobrachium rosenbergii: characteristics and expression patterns during ovarian and embryonic development[J]. Zoological Science, 2007, 24(11): 1059-1065. DOI:10.2108/zsj.24.1059 |
| [9] |
蔡影, 孟宪亮, 刘萍, 等. 三疣梭子蟹甲基转移酶2基因克隆及在胚胎、幼体和性腺发育过程中的表达分析[J]. 中国水产科学, 2018, 25(5): 928-935. CAI Ying, MENG Xianliang, LIU Ping, et al. Molecular cloning of the DNA methyltransferase-2 gene and its expression during embryonic, larval, and gonadal development in the swimming crab Portunus trituberculatus[J]. Journal of Fishery Sciences of China, 2018, 25(5): 928-935. |
| [10] |
FITZ E, WANKA F, SEIBOTH B. The promoter toolbox for recombinant gene expression in trichoderma reesei[J]. Frontiers in Bioengineering and Biotechnology, 2018, 6: 135. DOI:10.3389/fbioe.2018.00135 |
| [11] |
BANSAL M, KUMAR A, YELLA V R. Role of DNA sequence based structural features of promoters in transcription initiation and gene expression[J]. Current Opinion in Structural Biology, 2014, 25: 77-85. DOI:10.1016/j.sbi.2014.01.007 |
| [12] |
YELLA V R, BANSAL M. DNA structural features of eukaryotic TATA-containing and TATA-less promoters[J]. FEBS Open Bio, 2017, 7(3): 324-334. DOI:10.1002/2211-5463.12166 |
| [13] |
SEBASTIAN G, STEVEN H. Structural insights into transcription initiation by RNA polymerase II[J]. Trends in Biochemical Sciences, 2013, 38(12): 603-611. DOI:10.1016/j.tibs.2013.09.002 |
| [14] |
梁俊平, 李健, 李吉涛, 等. 脊尾白虾全人工繁育技术初步研究[J]. 水产科学, 2017, 36(3): 290-295. LIANG Junping, LI Jian, LI Jitao, et al. Controlled propagation of ridgetail white prawn Exopalaemon carinicauda[J]. Fisheries Science, 2017, 36(3): 290-295. |
| [15] |
裴倩倩, 张涛, 杨刚, 等. 长江口脊尾白虾繁殖生物学[J]. 生态学杂志, 2017, 36(3): 702-706. PEI Qianqian, ZHANG Tao, YANG Gang, et al. Reproductive biology of Exopalaemon carinicauda in Yangtze River Estuary[J]. Chinese Journal of Ecology, 2017, 36(3): 702-706. |
| [16] |
EMOTO M, MIKI M, SARKER A H, et al. Structure and transcription promoter activity of mouse flap endonuclease 1 gene: alternative splicing and bidirectional promoter.[J]. Gene, 2005, 357(1): 47-54. DOI:10.1016/j.gene.2005.05.009 |
| [17] |
SAVINKOVA L, DRACHKOVA I, ARSHINOVA T, et al. An experimental verification of the predicted effects of promoter TATA-Box polymorphisms associated with human diseases on interactions between the TATA boxes and TATA-Binding protein[J]. PloS One, 2013, 8(2): e54626. DOI:10.1371/journal.pone.0054626 |
| [18] |
HOCHHEIMER A, TJIAN R. Diversified transcription initiation complexes expand promoter selectivity and tissue-specific gene expression[J]. Genes & Development, 2003, 17(11): 1309-1320. |
| [19] |
SHE Z Y, YANG W X. SOX family transcription factors involved in diverse cellular events during development[J]. European Journal of Cell Biology, 2015, 94(12): 547-563. DOI:10.1016/j.ejcb.2015.08.002 |
| [20] |
JULIAN L M, MCDONALD A C, STANFORD W L. Direct reprogramming with SOX factors: masters of cell fate[J]. Current Opinion in Genetics & Development, 2017, 46: 24-36. |
| [21] |
VILLARINO A V, KANNO Y, O'SHEA J J. Mechanisms and consequences of Jak–STAT signaling in the immune system[J]. Nature Immunology, 2017, 18(4): 374-384. DOI:10.1038/ni.3691 |
| [22] |
TAKEDA K, NOGUCHI K, SHI W, et al. Targeted disruption of the mouse Stat3 gene leads to earlyembryoniclethality[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(8): 3801-3810. DOI:10.1073/pnas.94.8.3801 |
| [23] |
JULIAN L M, LIU Y, PAKENHAM C A, et al. Tissuespecific targeting of cell fate regulatory genes by E2f factors[J]. Cell Death & Differentiation, 2015, 23(4): 565-575. |
| [24] |
YAO W, LI Y X, LI B J, et al. Epigenetic regulation of bovine spermatogenic cell-specific gene Boule[J]. PloS One, 2015, 10(6): e0128250. DOI:10.1371/journal.pone.0128250 |
| [25] |
LONG H K, KING H W, PATIENT R K, et al. Protection of CpG islands from DNA methylation is DNA-encoded and evolutionarily conserved[J]. Nucleic Acids Research, 2016, 44(14): 6693-6706. DOI:10.1093/nar/gkw258 |
| [26] |
FATEMI M, PAO M M, JEONG S, et al. Footprinting of mammalian promoters: use of a CpG DNA methyltransferase revealing nucleosome positions at a single molecule level[J]. Nucleic Acids Res, 2005, 33(20): e176. DOI:10.1093/nar/gni180 |
| [27] |
VO N L, KASSAVETIS G A, KADONAGA J T. The RNA polymerase II core promoter in Drosophila[J]. Genetics, 2019, 212(1): 13-24. DOI:10.1534/genetics.119.302021 |
| [28] |
张远, 杨旬旬, 吴风瑞, 等. 小鼠Dnmt1启动子荧光素酶报告基因的构建及活性分析[J]. 生物技术通报, 2013, 10: 109-112. ZHANG Yuan, YANG Xunxun, WU Fengrui, et al. Construction and analysis of luciferase reporter vectors of mouse Dnmt1 gene promoter[J]. Biotechnology Bulletin, 2013, 10: 109-112. |
| [29] |
IL'ICHEVA I A, KHODIKOV M V, POPTSOVA M S, et al. Structural features of DNA that determine RNA polymerase II core promoter[J]. BMC Genomics, 2016, 17: 973. DOI:10.1186/s12864-016-3292-z |
| [30] |
赖晓芳, 高焕, 孔杰, 等. 中国明对虾FcβGBP-HDL基因和启动子的克隆及序列特征分析[J]. 水产学报, 2013, 37(2): 175-183. LAI Xiaofang, GAO Huan, KONG Jie, et al. Analysis of genomic structure and the promoter characterization of FcβGBP-HDL from Fenneropenaeus chinensi[J]. Journal of Fisheries of China, 2013, 37(2): 175-183. |
| [31] |
LENTJES M H, NIESSEN H E, AKIYAMA Y, et al. The emerging role of GATA transcription factors in development and disease[J]. Expert Reviews in Molecular Me di cine, 2016, 18: e3. DOI:10.1017/erm.2016.2 |
| [32] |
TREMBLAY M, SANCHEZ-FERRAS O, BOUCHARD M. GATA transcription factors in development and disease[J]. Development, 2018, 145(20): dev164384. DOI:10.1242/dev.164384 |
| [33] |
RAI K, CHIDESTER S, ZAVALA C V, et al. Dnmt2 functions in the cytoplasm to promote liver, brain, and retina development in zebrafish[J]. Genes & Development, 2007, 21(3): 261-266. |
| [34] |
杨旬旬. 小鼠植入前胚胎DNA甲基化模式比较及Dnmts启动子功能分析[D]. 合肥: 安徽大学, 2012. YANG Xunxun. Comparison of genomic-wide methylation patterns preimplantation embryos and functional analysis of Dnmts promoter of mouse[D]. Hefei: Anhui University, 2012. |
 2022, Vol. 46
2022, Vol. 46


