文章信息
- 葛建龙, 陈晓辉, 陈四清, 刘长琳, 陈昱辰, 边力, 张盛农. 2022.
- GE Jian-long, CHEN Xiao-hui, CHEN Si-qing, LIU Chang-lin, CHEN Yu-chen, BIAN Li, ZHANG Sheng-nong. 2022.
- 海蜇dmrta2基因的克隆及其在横裂生殖中的表达分析
- Cloning of dmrta2 from jellyfish Rhopilema esculentum and its expression analysis during strobilation
- 海洋科学, 46(6): 42-50
- Marine Sciences, 46(6): 42-50.
- http://dx.doi.org/10.11759/hykx20210308002
-
文章历史
- 收稿日期:2021-03-08
- 修回日期:2021-07-20
2. 盐城市华亿水产有限公司, 江苏 盐城 224300
2. Huayi aquatic breeding limited company, Yancheng 224300, China
Dmrt(double-sex and mab-3 relatated transcription factor) 是指与果蝇dsx基因和线虫mab-3基因同源的一类转录调控因子, 主要特征是所编码的多肽链中包含保守的锌指样DNA结合基序—DM结构域[1]。目前, 除海绵动物外所有后生动物中均报道了dmrt基因的存在[2], 而不同物种中dmrt基因的种类不尽相同[1], 功能涉及性别决定与分化、组织和器官的形成以及相关功能的维持等多方面[3]。
海蜇(Rhopilema esculentum)主要分布于西北太平洋, 其生长迅速、胶质层厚实, 深受消费者喜爱, 是我国重要的增养殖品种, 同时是我国开展钵水母研究的代表种类[4]。海蜇具有世代交替的生活史, 包括营栖息生活的无性阶段(即钵口幼体或水螅体)和营浮游生活的有性阶段(即水母体)[4]。在这样的生命周期中, 从水螅体转变为水母体是通过一个有序变态的过程实现的, 即“横裂生殖”。横裂生殖是海蜇等钵水母特有的一种无性繁殖方式, 首先, 横向收缩将水螅体从近口端向远口端依次分割为裂节, 其次, 两个收缩之间的裂节部分经过变态发育成碟状体, 随后, 它们与水螅体母体脱离开始其浮游生活, 并在几周内生长为幼水母[5]。随着一次横裂生殖的完成, 一些水螅体可以再生一个新的口柄和触手, 并恢复他们以前的生活方式[5]。生活史各环节中, 横裂生殖是钵水母从水螅体向水母体转变的重要阶段, 是影响水母体种群数量的重要环节。
横裂生殖作为钵水母特有的无性生殖方式, 其显著地变态发育过程一直吸引着国内外学者的关注[6-7]。我们先前的研究发现dmrt家族基因等11个转录调控因子可能与海蜇横裂生殖相关[8], 与该研究结果相似, 在海月水母(Aurelia aurita)中也鉴定了一个可能与横裂生殖相关的dmrt家族基因[9], 然而dmrt家族基因在钵水母横裂生殖中的具体作用尚无研究报道。因此, 本研究基于转录组测序获得的海蜇dmrta2基因的部分序列, 通过RACE(rapid-amplification of cDNA ends)技术获得了该基因的全长序列, 并分析了其在海蜇横裂生殖过程的表达模式, 旨在为阐明海蜇横裂生殖分子调控机制提供一定基础资料。
1 材料与方法 1.1 实验材料海蜇螅状体来自江苏省盐城市金洋水产良种场, 运回实验室暂养于光照培养箱中, 螅状体为当年繁育获得。采用升温法(培育水温按2 ℃/d, 由10 ℃升高至18 ℃)诱导其横裂生殖, 分别获取螅状体、横裂初期、横裂后期和碟状体时期的样品, 用于RNA提取的样品浸入液氮速冻后保存于–80 ℃超低温冰箱。
1.2 总RNA提取以及基因全长的克隆采用Trizol法提取各样品总RNA, 具体步骤参照Trizol试剂(Invitrogen)使用说明书。RNA质量及浓度分别通过1%琼脂糖凝胶电泳和Nanodrop 2000紫外分光光度计检测合格后, 利用Prime Script™ RT reagent Kit试剂盒(TaKaRa), 反转录获得cDNA第一链。
首先, 以先前转录组测序中获得的一段dmrta2同源序列为核心序列, 据此设计核心片段扩增引物(表 1), 通过PCR扩增和Sanger测序确认核心序列。然后, 以确认后的核心序列为模板分别设计5′RACE和3′RACE的特异性引物, 按照SMARTTM RACE cDNA Amplification Kit试剂盒(Clontech)操作说明扩增基因5′末端和3′末端。PCR产物经切胶回收目的片段, 连接至pMD19-T载体后转入E. coli DH5α细胞中, LB液体培养基培养后均匀涂布于LA(含氨苄青霉素)固体平板, 挑取阳性克隆样品送至上海生工生物公司测序。最后, 利用SeqMan将核心片段及5′RACE和3′RACE片段的测序结果进行拼接, 获得cDNA序列全长。
| 引物编号 | 引物序列 | 用途 |
| Redmrt-1F | TCAGTTGGTCATTTGTCGTTTC | 核心片段扩增 |
| Redmrt-1R | ATCTGAAGCAGGTCTGTTGGA | 核心片段扩增 |
| Redmrt-5′-OUTER | GCTTGAGGTATCCATTTGTGTCC | 5′RACE |
| Redmrt-5′-INNER | ATGCGGTTGTGGTCGGCTTC | 5′RACE |
| Redmrt-3′-OUTER | GCGAAACAGAGAAAGAAGACGAG | 3′RACE |
| Redmrt-3′-INNER | ACATCACATCCCGCAGCAGA | 3′RACE |
| GAPDH-F | ACTTGTTTCCTGGTATGACAAT | 内参基因 |
| GAPDH-R | TGAATCATCAAGTCGCAAACAC | 内参基因 |
| Redmrt-RT-F | CTGTCTCTCCTTGTGTTCCCTA | RT-PCR |
| Redmrt-RT-R | GTTTGAGGCAGTGCATTTATTC | RT-PCR |
| Hy5-17F | TCTGTTCAGCCTGTGGGTGTAT | 探针扩增 |
| Hy5-17R | TAATACGACTCACTATAGGG | 探针扩增 |
利用ORF Finder (http://www.ncbi.nlm.nih.gov/gorf/gorf.html)分析基因ORF并推导氨基酸序列; BlastP (http://blast.ncbi.nih.gov/)预测氨基酸序列的保守结构域及同源序列检索; ClusterX2将海蜇Dmrt与其他物种相应的氨基酸序列进行多重序列比对分析; Mega 7构建N-J系统进化树; SignalP 5.1(http://www.cbs.dtu.dk/services/SignalP/)和TMHMM Server v.2.0 (http://www.cbs.dtu.dk/services/TMHMM-2.0/)分别预测是否具有信号肽和跨膜结构域; ProtParam(https://web.expasy.org/protparam/)和Protscale(http://web.expasy.org/protscale/)分析氨基酸序列的理化性质及蛋白质的疏水性; PSORT II Prediction(https://psort.hgc.jp/form2.html)预测蛋白亚细胞定位。
1.4 RT-PCR定量分析根据基因全长序列设计实时定量PCR引物(表 1), 以gapdh为内参基因, 以cDNA为模板, 采用SYBR Green试剂盒(Takara), 在ABI StepOneTM Plus荧光定量PCR仪上进行定量PCR分析。反应体系如下: 2×Green PCR Master Mix 5 μL、引物(10 μmol/L)各0.2 μL和cDNA模板1 μL, 加PCR水至10 μL。扩增程序: 95 ℃、10 s; 95 ℃、15 s, 60 ℃、60 s, 40个循环, 每个循环最后一步时采集荧光信号。每个时期样品设3个生物学重复和技术重复, 相对定量的结果采用2–ΔΔCt法进行计算, 实验数据在Excel中进行数据分析和图表绘制, 运用SPSS 16.0软件进行ANOVA差异显著性分析。
1.5 整体原位杂交用于整体原位杂交的样品, 先在2%乌来糖中(海水配置)麻醉10 min, 然后用新鲜配制的4%多聚甲醛-海水固定20 min, 按25%、50%、75%、100%梯度替换为4%多聚甲醛-PBS, 每次10 min, 4 ℃固定过夜, 最后, 按25%、50%、75%、100%梯度替换为100%甲醇, –20 ℃保存。
采用体外转录法制备地高辛标记RNA探针, 方法参考T7/SP6转录试剂盒(Invitrogen)说明书。首先, 设计正义和反义探针引物(表 1), 并分别在正向引物或者反向引物5′端加T7启动子序列, 进行PCR扩增, 产物经纯化后作为探针合成的模板, 纯化试剂盒为Takara。其次, 采用上述纯化后的模板, 进行RNA体外转录合成, 反应体系: 10×transcription buffer 2 μL、10 mmol/L ATP 1 μL、10 mmol/L CTP 1 μL、10 mmol/L、GTP 1μL、10 m mmol/L UTP 0.6 μL, 3.5 mmol/L UTP-11-DIG 1 μL, RNA polymerase mix (T7/SP6) 2 μL, 模板DNA 11.4 μL, 轻弹混匀离心后, 37 ℃水浴1.5 h; 反应完成后, 加入1 μL DNase I, 于37 ℃水浴20 min, 以去除残留的DNA模板。最后, 转录反应产物经Mega RNA纯化试剂盒回收纯化, 取1μL探针进行1%琼脂糖凝胶电泳检测, Nanodrop 2000测定探针浓度, –80 ℃保存备用。
整体原位杂交方法参照先前报道的方法[9], 略有改动。其中, 蛋白酶K处理浓度为10 g/L, 处理时间20 min。杂交液: 50% Formamid, 5x SSC, 0, 1% Tween-20, 0, 1% CHAPS, 1x Denhardt’s solution, 100 µg/mL Heparin, (t-RNA 20 µg/mL, 现用现加)。采用地高辛检测试剂盒的NBT/BCIP显色, 每5 min解剖镜下观察一次, PBST漂洗10 min停止染色, 70%乙醇浸洗3次, 每次10 min, 最后替换为70%甘油, 4℃冰箱保存并于解剖镜下观察拍照。
2 结果 2.1 海蜇dmrta2基因的序列特征海蜇dmrta2基因的cDNA全长为1 804 bp, 包含169 bp的3′端与624 bp的5′端UTR, 开放阅读框(ORF)为1 011 bp, 编码336个氨基酸(图 1)。海蜇dmrta2基因序列已上传NCBI数据库, Genbank登录号为MT878494。经与参考基因组(http://gigadb.org/dataset/view/id/100720/File_page/2)比对分析发现, 海蜇dmrta2位于Scaffold SWAQ01000558.1, 含有3个外显子和2个内含子。Protein blast显示, 海蜇DMRTA2蛋白拥有DM和CUE两个特征结构域, 分别位于第33~79和231~271位氨基酸。
 |
| 图 1 海蜇dmrta2基因的核苷酸序列与推测的氨基酸序列 Fig. 1 Nucleotide and deduced amino acid sequences of Rhopilema esculentum dmrta2 注: 起始密码子(ATG)和终止密码子(TAA)采用红色标记; polyA尾巴用蓝色标记 |
通过在线软件预测了DMRTA2蛋白的理化性质, 结果显示其分子量为37 252.23 Da, 带负电荷的氨基酸残基数(Asp+Glu)为36个, 带正电荷氨基酸残基数(Arg+Lys)为48个, 理论等电点(pI)为9.11, 偏碱性。预测不稳定系数为52.3, 表明其编码蛋白理化性质不稳定, 摩尔消光系数为25 285, 理论半衰期30 h, 脂溶系数为61.52, 平均亲水系数(GRAVY)为–0.649, 表明该基因编码蛋白属于亲水性蛋白。组成海蜇DMRTA2蛋白的20种氨基酸中, 丝氨酸(Ser)最多, 所占比例为11.6%, 色氨酸(Trp)最少, 所占比例为0.6%。TMHMM Server v.2.0分析显示其无跨膜结构, Signal 5.1分析显示其不具有信号肽, 表明该分子为非分泌性蛋白, 蛋白亚细胞定位预测显示其分布在细胞核的可能性最高, 为73.9%。
2.2 氨基酸序列比对和系统进化分析从NCBI参考序列数据库中下载代表性物种的DMRT家族蛋白序列进行多序列比对, 结果显示海蜇DMRTA2蛋白的DM结构域序列与其他物种DMRTA2、DMRT5和DMRT99B序列相似性很高, 含有DM结构域最为保守的CCHHCCCC基序, 形成锌指结构位点(CCHC的Site I和HCCC的Site II), 与之相比CUE结构域保守性较差, 而结构域之外的序列变异程度很高(图 2)。
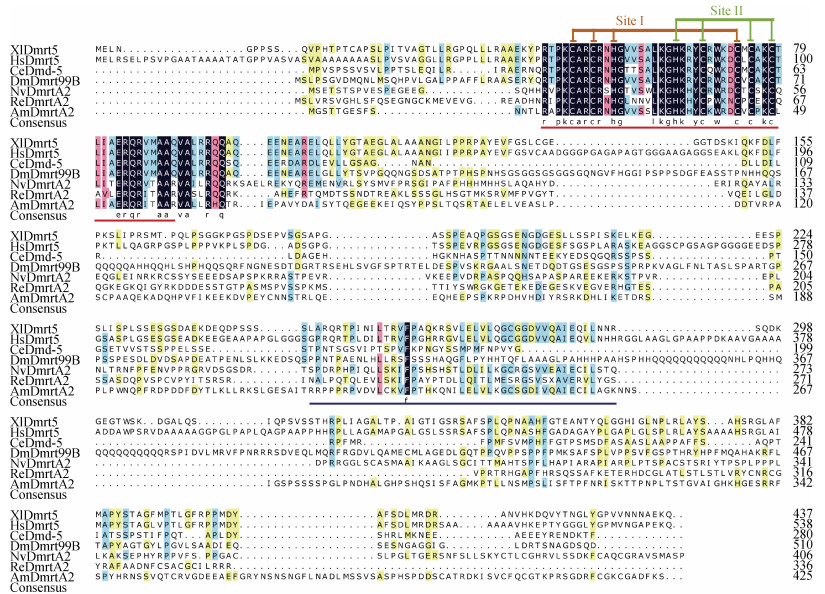 |
| 图 2 海蜇DMRTA2与同源蛋白的序列比对 Fig. 2 Sequence alignment of Rhopilema esculentum DMRTA2 and homology DMRT proteins of different species 注: 黑色背景表示完全一致的氨基酸; 粉色背景表示相似性大于75%; 浅蓝色背景表示相似性大于50%; 黄色背景表示相似性大于33%; 红色下划线指示保守的DM结构域, 蓝色下划线指示CUE结构域; 保守的锌指结构用棕色框线(Site I)和绿色框线(Site II)指示; 非洲爪蟾(Xenopus laevis, XlDmrt5: NP_001089148.1); 智人(Homo sapiens, HsDmrt5: AAI36283.1); 秀丽隐杆线虫(Caenorhabditis elegans, CeDmd-5: NP_495138.2); 果蝇(Drosophila melanogaster, DmDmrt99B: NP_524549.1); 海葵(Nematostella vectensis, NvDmrta2: XP_032231562.1); 鹿角珊瑚(Acropora millepora, AmDmrta2: XP_029187503.1) |
本研究基于7个物种22个DMRT蛋白的DM结构域构建了系统进化树, 结果显示海蜇DMRTA2与鹿角珊瑚DMRTA2、鹿角珊瑚DMRT3以及海葵DMRTA2的亲缘关系最近, 其次与线虫MAB-3、线虫DMD-4和果蝇DMRT93B聚为一支, 亲缘关系较近, 与智人DMRT5、非洲爪蟾DMRT5以及果蝇DMRT99B的亲缘关系最远。特别的是海葵DMRTA1和DMRT3单独聚为一支, 与海蜇DMRTA2等腔肠动物DMRT蛋白的亲缘关系较远(图 3)。
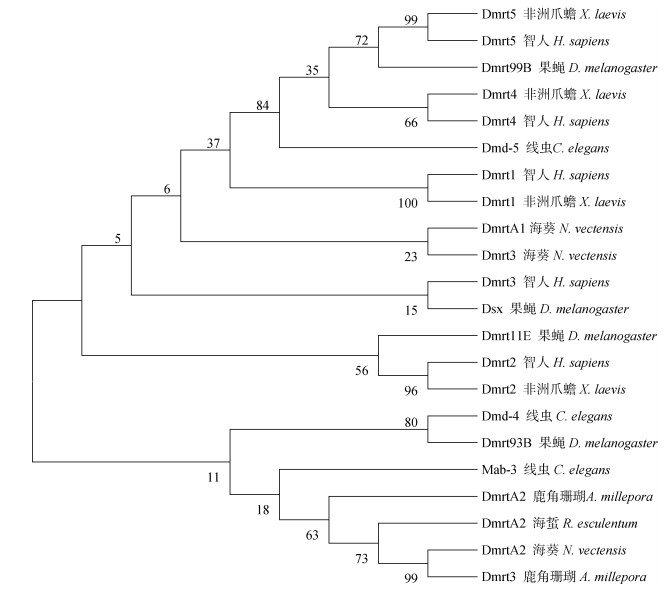 |
| 图 3 DMRT家族蛋白的系统进化分析 Fig. 3 Phylogenetic analysis of DMRT proteins among different specie 注: 鹿角珊瑚(A. millepora, Dmrta2: XP_029187503.1, Dmrt3: XP_029183089.1); 海葵(N. vectensis, Dmrta1: XP_032232660.1, Dmrta2: XP_032231562.1, Dmrt3: XP_001628137.2); 秀丽隐杆线虫(C. elegans, Mab-3: NP_001022464.1, Dmd-4: NP_510466.1, Dmd-5: NP_495138.2); 果蝇(D. melanogaster, Dmrt93B: NP_524428.1, Dmrt99B: NP_524549.1, Dmrt11E: NP_511146.2, Dsx: NP_731197.1); 非洲爪蟾(X. laevis, Dmrt1: NP_001089969.1, Dmrt2: NP_001089725.1, Dmrt4: NP_001084923.1, Dmrt5: NP_001089148.1), 智人(H. sapiens, Dmrt1: AAR89619.1, Dmrt2: NP_006548.1, Dmrt3: NP_067063.1, Dmrt4: NP_071443.2, Dmrt5: AAI36283.1) |
荧光定量PCR结果显示, 相对于螅状体时期, dmrta2基因在横裂生殖初期和碟状体时期有显著上调表达, 在横裂生殖后期表达量最高, 显著高于其他各时期(图 4)。整体原位杂交显示, dmrta2在螅状体中基本无表达信号, 横裂生殖初期的触手基部有明显表达, 横裂生殖后期显著表达于感觉缘叶原基部位, 碟状体时期主要表达于感觉棍相应区域, 此外, 横裂生殖后期以及碟状体时期在口唇位置有一定表达(图 5)。
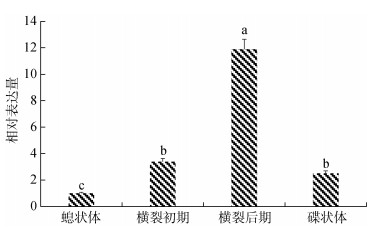 |
| 图 4 Dmrta2在海蜇横裂生殖过程的相对表达量 Fig. 4 Relative expression level of dmrta2 in the strobilation of Rhopilema esculentum 注: 在螅状体时期的相对表达量设为1, 其他时期的表达量均以螅状体表达量的倍数表示; 不同字母表示各组数据之间存在显著性差异(P < 0.01) |
 |
| 图 5 Dmrta2在海蜇横裂生殖过程的整体原位表达 Fig. 5 Whole-mount in situ expressions of dmrta2 in the strobilation of Rhopilema esculentum 注: A. 螅状体时期; B. 横裂生殖初期; C. 横裂生殖后期; D. 碟状体时期; E. 碟状体局部e的放大; bc. 体柱; bd. 基盘; gc. 消化腔管; mn. 口唇; prl. 感觉缘叶原基; pe. 碟状体原; rl. 感觉缘叶; rh. 感觉棍; sg. 横向裂节; te. 触手; 蓝色为表达阳性信号 |
腔肠动物是已知最原始的具有dmrt家族基因的后生动物[3], 因此对于dmrt基因家族起源与进化的研究有着重要意义, 目前仅在少量的腔肠动物中有dmrt基因的研究报道, 如鹿角珊瑚[10]、石珊瑚(Euphyllia ancora)[11]、海葵[12]和栉水母(Mnemiopsis leidyi)[13]。本研究克隆了钵水母纲腔肠动物的一个dmrt家族基因—海蜇dmrta2基因, 获得了该基因的cDNA全长序列, 通过NCBI序列比对发现该基因推导的氨基酸序列具有DM结构域, 属于dmrt基因家族。序列分析发现海蜇dmrta2基因的DM结构域包含保守的6个半胱氨酸(C)和2个组氨酸(H)序列, 能够形成与Zn离子结合的两个位点(CCHC和HCCC), 说明海蜇dmrta2基因功能结构完整。序列比对也显示了海蜇dmrta2符合dmrt基因家族的序列特点, 即氨基酸全长序列差异很大, 可观测到的同源性仅限于与DNA结合的DM结构域[14]。
基于DM结构域的系统进化分析结果与已有研究报道基本一致, 如脊椎动物dmrt4和dmrt5、果蝇dmrt99B以及线虫dmd-5可能起源于共同的dmrt4/5基因, 果蝇dmrt11E与脊椎动物dmrt2是直系同源基因[2], 本研究中这些基因也相应地先聚为一个分支。本研究中海蜇dmrta2与鹿角珊瑚dmrta2、鹿角珊瑚dmrt3以及海葵dmrta2的亲缘关系最近, 其次与线虫mab-3、线虫dmd-4和果蝇dmrt93b聚为一支, 符合传统的系统进化关系, 然而系统进化树中海葵dmrta1和dmrt3单独聚为一支, 与海蜇dmrta2等腔肠动物dmrt基因的亲缘关系较远, 与传统的系统进化关系不符, 一个可能原因是dmrt家族基因较短的DM结构域(约70个氨基酸)使系统进化关系的解析变得困难[2], 同时说明了dmrt家族基因在低等腔肠动物中的系统进化关系较为复杂, 有待进一步研究分析。
最初, 在果蝇和线虫中发现了含有DM结构域的基因与性别决定相关[15-16], 而后越来越多的研究发现dmrt家族基因几乎在所有动物发育的性腺组织中有特异性表达, 如珊瑚[10]、贝类[17-18]、甲壳类[19-20]、鱼类[21]、爬行类[22]、鸟类[23]和哺乳类[24], 说明了dmrt家族基因与性别决定和性腺发育的密切相关。而除了在性腺中表达外, 一些研究中发现dmrt家族成员表达于神经组织, 参与神经系统和感觉器官的分化发育。例如, 在鸡和小鼠中, dmrt3表达于前脑、脊髓和嗅板中[25]; 在斑马鱼中, dmrta2基因能够调控神经分化基因neurogenin1的表达, 参与体节形成时端脑神经细胞的分化[26]; 爪蟾dmrt4和dmrt5共同表达于发育中的嗅叶, 是爪蟾嗅觉系统神经形成的重要转录调控因子[27-28]; 海葵nvdmrtb可能是dmrt4/5同源基因, 它能够诱导爪蟾的神经形成, 而且nvdmrtb敲降处理的海葵胚胎中神经元细胞明显减少[28]。已有研究表明横裂生殖过程中一个显著的变化是逐步发育的碟状体感觉棍神经系统(rhopadial nervous system) [29], 本研究中, dmrta2基因在海蜇横裂生殖后期主要表达于感觉缘叶原基位置, 在初生碟状体中明显表达于感觉棍位置, 因此, 海蜇横裂生殖中dmrta2基因的上调表达应该与感觉棍神经系统的分化发育相关。
4 结语综上所述, 本研究成功克隆了海蜇Dmrta2基因的cDNA全长序列, 利用生物学分析软件对其编码蛋白的理化性质和系统进化关系等进行了分析, 通过原位杂交研究了其在横裂生殖中的表达模式, 根据本研究原位表达结果以及已报道的dmrt同源基因的主要功能, 我们推测该基因在海蜇横裂生殖中主要与感觉棍神经系统的分化发育相关, 研究结果为海蜇等钵水母横裂生殖调控机制的解析提供了基础数据。
| [1] |
MAWARIBUCHI S, ITO Y, ITO M. Independent evolution for sex determination and differentiation in the DMRT family in animals[J]. Biology Open, 2019, 8(8): bio041962. DOI:10.1242/bio.041962 |
| [2] |
WEXLER J R, PLACHETZKI D C, KOPP A. Pan-metazoan phylogeny of the DMRT gene family: a framework for functional studies[J]. Development Genes and Evolution, 2014, 224(3): 175-181. DOI:10.1007/s00427-014-0473-0 |
| [3] |
李法君, 付春鹏, 罗永巨. Dmrt基因在水生生物中的研究进展[J]. 水生生物学报, 2016, 40(5): 1068-1077. LI Fajun, FU Cunpeng, LUO Yongju. Research progress of Dmrt genes in hydrobiontes[J]. Acta Hydrobiologica Sinica, 2016, 40(5): 1068-1077. |
| [4] |
YOU K, MA C, GAO H, et al. Research on the jellyfish (Rhopilema esculentum Kishinouye) and associated aquaculture techniques in China: current status[J]. Aquaculture International, 2007, 15(6): 479-488. DOI:10.1007/s10499-007-9114-1 |
| [5] |
丁耕芜, 陈介康. 海蜇的生活史[J]. 水产学报, 1981, 5(2): 93-102. DING Gengwu, CHEN Jiekang. The life history of Rhopilema esculentum Kishinouye[J]. Journal of Fishe ries of China, 1981, 5(2): 93-102. |
| [6] |
HELM R R. Evolution and development of scyphozoan jellyfish[J]. Biological Reviews, 2018, 93(12): 1228-1250. |
| [7] |
林政飞, 王淑红. 钵水母水螅体横裂诱发条件及调控机制研究进展[J]. 应用生态学报, 2019, 30(3): 1057-1066. LIN Zhengfei, WANG Shuhong. Research progress in scyphozoan polyp strobilation-induced factors and regulation mechanism[J]. Chinese Journal of Applied Ecology, 2019, 30(3): 1057-1066. |
| [8] |
GE J, LIU C, TAN J, et al. Transcriptome analysis of scyphozoan jellyfish Rhopilema esculentum from polyp to medusa identifies potential genes regulating strobilation[J]. Development Genes and Evolution, 2018, 228(1): 243-254. |
| [9] |
FUCHS B, WANG W, GRASPEUNTNER S, et al. Regulation of polyp-to-jellyfish transition in Aurelia aurita[J]. Current Biology, 2014, 24(3): 263-273. DOI:10.1016/j.cub.2013.12.003 |
| [10] |
MILLER S W, HAYWARD D C, BUNCH T A, et al. A DM domain protein from a coral, Acropora millepora, homologous to proteins important for sex determination[J]. Evolution & Development, 2003, 5(3): 251-258. |
| [11] |
CHEN C J, SHIKINA S, CHEN W J, et al. A novel female-specific and sexual reproduction-associated Dmrt gene discovered in the stony coral, Euphyllia ancora[J]. Bio logy of Reproduction, 2016, 94(2): 1-13. |
| [12] |
TRAYLOR-KNOWLES N G, KANE E G, SOMBATSAPHAY V, et al. Sex-specific and developmental expression of Dmrt genes in the starlet sea anemone, Nematostella vectensis[J]. Evodevo, 2015, 6: 1-6. DOI:10.1186/2041-9139-6-1 |
| [13] |
REITZEL A M, PANG K, MARTINDALE M Q. Developmental expression of "germline"-and "sex determination"-related genes in the ctenophore Mnemiopsis leidyi[J]. Evodevo, 2016, 7: 1-16. DOI:10.1186/s13227-015-0038-y |
| [14] |
VOLFF J N, ZARKOWER D, BARDWELL V J, et al. Evolutionary dynamics of the DM domain gene family in metazoans[J]. Journal of Molecular Evolution, 2003, 57(s1): S241-S249. |
| [15] |
RAYMOND C S, SHAMU C E, SHEN M M, et al. Evidence for evolutionary conservation of sex-determining genes[J]. Nature, 1998, 391(6668): 691-695. DOI:10.1038/35618 |
| [16] |
RAYMOND C S, MURPHY M W, O'SULLIVAN M G, et al. Dmrt1, a gene related to worm and fly sexual regulators, is required for mammalian testis differentiation[J]. Genes & Development, 2000, 14(20): 2587-2595. |
| [17] |
WANG Q, SHI Y, HE M. Pf-Dmrt4, a potential factor in sexual development in the pearl oyster Pinctada fucata[J]. Journal of Oceanology and Limnology, 2019, 36(6): 2337-2350. |
| [18] |
李茜茜, 秦贞奎, 魏茂凯, 等. 栉孔扇贝Dmrt1的分子鉴定及表达模式分析[J]. 中国水产科学, 2020, 27(11): 1277-1284. LI Qianqian, QIN Zhenkui, WEI Maokai, et al. Molecular identification and expression pattern analysis of Dmrt1 in the scallop Chlamys farreri[J]. Journal of Fishery Sciences of China, 2020, 27(11): 1277-1284. |
| [19] |
KATO Y, KOBAYASHI K, WATANABE H, et al. Environmental sex determination in the branchiopod crustacean Daphnia magna: deep conservation of a doublesex gene in the sex-determining pathway[J]. PLoS Genetics, 2011, 7(3): e1001345. DOI:10.1371/journal.pgen.1001345 |
| [20] |
YU Y Q, MA W M, ZENG Q cG, et al. Molecular cloning and sexually dimorphic expression of two Dmrt genes in the giant freshwater prawn, Macrobrachium rosenbergii[J]. Agricultural Research, 2014, 3(2): 181-191. DOI:10.1007/s40003-014-0106-x |
| [21] |
HERPIN A, SCHARTL M. Dmrt1 genes at the crossroads: a widespread and central class of sexual development factors in fish[J]. The FEBS Journal, 2011, 278(7): 1010-1019. DOI:10.1111/j.1742-4658.2011.08030.x |
| [22] |
欧阳淑, 刘晓莉, 王亚坤, 等. 黄喉拟水龟Dmrt1基因克隆及其表达特征[J]. 水生生物学报, 2020, 44(4): 749-755. OUYANG Shu, LIU Xiaoli, WANG Yakun, et al. Cloning and expression characteristics of Dmrt1 gene of Mauremys mutica[J]. Acta Hydrobiologica Sinica, 2020, 44(4): 749-755. |
| [23] |
CHUE J, SMITH C A. Sex determination and sexual differentiation in the avian model[J]. The FEBS Journal, 2011, 278(7): 1027-1034. |
| [24] |
魏于栋, 申单, 程慧玲, 等. Dmrt1与哺乳动物生殖细胞发育[J]. 中国细胞生物学学报, 2015, 37(2): 278-283. WEI Yudong, SHEN Dan, CHENG Huiling, et al. Dmrt1 in mammalian germ cell development[J]. Chinese Jour nal of Cell Biology, 2015, 37(2): 278-283. |
| [25] |
SMITH C A, HURLEY T M, MCCLIVE P J, et al. Restricted expression of Dmrt3 in chicken and mouse embryos[J]. Mechanisms of Development, 2002, 119(s1): s73-s76. |
| [26] |
YOSHIZAWA A, NAKAHARA Y, IZAWA T, et al. Zebrafish Dmrta2 regulates neurogenesis in the telencephalon[J]. Genes To Cells, 2011, 16(11): 1097-1109. DOI:10.1111/j.1365-2443.2011.01555.x |
| [27] |
HUANG X, HONG C S, O'DONNELL M, et al. The doublesex-related gene, XDmrt4, is required for neurogenesis in the olfactory system[J]. Proceedings of the National Academy of Sciences, 2005, 102(32): 11349-11354. DOI:10.1073/pnas.0505106102 |
| [28] |
PARLIER D, MOERS V, CAMPENHOUT C V, et al. The Xenopus doublesex-related gene Dmrt5 is required for olfactory placode neurogenesis[J]. Developmental Biology, 2013, 373(1): 39-52. |
| [29] |
NAKANISHI N, HARTENSTEIN V, JACOBS D K. Development of the rhopalial nervous system in Aurelia sp. 1 (Cnidaria, Scyphozoa)[J]. Development Genes & Evolution, 2009, 219(6): 301-317. |
 2022, Vol. 46
2022, Vol. 46


