
文章信息
- 蒙雨, 宋一清, 谢青颖, 蔡杏伟, 张清凤, 董杨, 李芳远, 谢松光, 申志新. 2023.
- MENG Yu, SONG Yi-qing, XIE Qing-ying, CAI Xing-wei, ZHANG Qing-feng, DONG Yang, LI Fang-yuan, XIE Song-guang, SHEN Zhi-xin. 2023.
- 海南万泉河口尖鳍鲤线粒体基因组测序与结构特征分析
- Mitochondrial genome sequencing and structural characteristic analysis of Cyprinus acutidorsalis from Wanquan Estuary, Hainan
- 海洋科学, 47(11): 66-78
- Marine Sciences, 47(11): 66-78.
- http://dx.doi.org/10.11759/hykx20230720001
-
文章历史
- 收稿日期:2023-07-20
- 修回日期:2023-10-07
2. 海南大学南海海洋资源利用国家重点实验室, 海南 海口 570228;
3. 海南省海洋与渔业科学院, 海南 海口 571126;
4. 海南淡水生物资源及生态环境保护研究中心, 海南 海口 571126
2. State Key Laboratory of Marine Resources Utilization in South China Sea, Haikou 570228, China;
3. Hainan Academy of Ocean and Fisheries Sciences, Haikou 571126, China;
4. Hainan Freshwater Biological Resources and Ecological Environment Protection Research Center, Haikou 571226, China
线粒体基因组(mtDNA)是独立存在于高等动物细胞核以外的唯一遗传物质, 具有结构简单、基因排列紧凑、容易扩增、以母系遗传为主、进化率高等特点[1]。鱼类线粒体基因组是一个双链闭合环状DNA分子, 大小占整个DNA约为1%[2], 长度通常在16 kb左右, 且排列结构一般非常保守, 十分紧凑。尽管鱼类线粒体基因组整体上是高度保守的, 但某些区域仍然可以表现出高度变异性[3], 这些区域通常是非编码区域或控制区域, 例如重复序列、转座子和调节元件。除了少数物种外, 大部分动物线粒体基因组都具有相同的37个基因, 包括典型的基因互补编码13个氧化磷酸化酶的蛋白质亚单位、线粒体核糖体的两个rRNA, 以及翻译线粒体基因组编码的蛋白质所需的22个tRNAs[4]。相邻基因没有间隔或存在两种基因前端和后端碱基重叠的现象。线粒体基因组具有自己的DNA复制、转录、mRNA处理和蛋白质翻译系统, 可以作为基因组进化的模型[5]。
随着Sanger测序方法的成熟和第二代和第三代测序技术的不断改进, 线粒体基因组的测序变得简单快捷, 已成为种类鉴定、系统发育以及群体遗传分化等领域广泛应用的分子标记之一[6], 为鱼类种质资源的保护提供了有效手段[7], 进化最快的D-loop区可以用于研究某区域的种群遗传多样性水平[8]; CO I基因也是许多鱼类分类和鉴定的理想DNA条形码[9]。通过对不同物种的线粒体基因组比较分析不仅可以研究适应性进化, 也可以追踪分析特异物种的起源和进化[10]。结合形态学分析和线粒体基因组母系遗传和序列变异迅速的特征重建物种的系统发育关系, 为解决物种起源和分化问题提供了更好的遗传学证据[11]。
尖鳍鲤(Cyprinus acutidorsalis)隶属于鲤形目(Cypriniformes)、鲤科(Cyprinidae)、鲤亚科(Cyprininae)、鲤属(Cyprinus)。鲤亚属, 又名海鲤, 广西俗称“海鲫”, 其种群仅分布在海南万泉河、南渡江河口以及广西的钦江河口, 分布范围狭窄[12], 目前是中国特有的生活于河口生态系统的鲤科鱼类[13]。由于人类活动干扰等原因, 尖鳍鲤的适宜栖息地大面积缩小, 种质极度退化, 种群数量锐减, 仅有的几处河口已较难发现其踪迹, 处于极危状态[14]。
有关尖鳍鲤的研究主要集中于早期发育和养殖技术, 研究区域仅分布在广西钦江河口[15-16]。前期有少量研究基于线粒体基因的片段揭示了尖鳍鲤在鲤形目鱼类的发育地位和遗传多样性[17-18], 关于线粒体基因组的结构特征还鲜有报道。本研究利用高通量第二代测序技术对进行测序, 组装与注释尖鳍鲤线粒体全基因组序列, 分析海南和广西水域尖鳍鲤线粒体基因的相似性, 并比较了尖鳍鲤与鲤、鲫的13个PCGs碱基组成差异等线粒体基因组特征, 构建尖鳍鲤与其他16种鲤科鱼类的系统发育树。研究结果将为海南尖鳍鲤种类鉴定和系统进化提供科学依据。
1 材料与方法 1.1 实验材料及DNA的提取尖鳍鲤样本采集自海南省琼海市博鳌镇万泉河口沙美内海。剪取背部肌肉样本放入无水乙醇中保存。使用动物组织基因组试剂盒(OMEGA), 根据说明书的操作步骤提取总DNA, 通过琼脂糖凝胶电泳和核酸蛋白测定仪对其DNA纯度和浓度进行测定。
1.2 基因组测序委托上海凌恩生物科技有限公司进行尖鳍鲤的线粒体基因组测序, 具体实验步骤如下: 将提取的DNA样品以1 μg起始量构建文库, 使用Covaris M220超声打断分割DNA长度为300~500 bp的片段并在两端加接头(Illumina), 采用TBS380(Invitrogen)定量, 将文库回收(Bio-Rad), 再根据数据比例混合后上机, 在cBot固相载体上进行桥式PCR扩增生成clusters, 在Illumina NovaSeq测序平台(Truseq SBS Kit), 进行2×150 bp上机测序(300 cycles)。
1.3 基因组组装与分析用SPAdes v3.14.1(http://bioinf.spbau.ru/spades)软件对Clean Data进行拼接。挑选覆盖深度足够高且组装长度较长的序列作为候选序列, 比对NT库确认线粒体Scaffold序列, 并根据overlap连接序列。根据参考基因组确定线粒体组装序列的起始位置和方向, 获得尖鳍鲤的线粒体基因组序列。
使用MITOS(http://mitos.bioinf.uni-leipzig.de/index.py)软件对线粒体基因组进行编码蛋白、tRNA和rRNA基因的预测。然后对MITOS预测的初始基因去冗余, 并人工校正基因的起始、终止密码子位置, 获得高准确性的保守基因集。使用cusp (EMBOSS v6.6.0.0)软件获得密码子的偏好性值。利用软件CGView(http://stothard.afns.ualberta.ca/cgviewserver/)对样品基因组进行圈图展示。尖鳍鲤线粒体的基因蛋白序列与已知的蛋白数据库进行blastp(evalue < 1e-5)比对, 进行NR、GO、Swiss-Prot注释分析。
从NCBI (https://www.ncbi.nlm.nih.gov/)上获取16种鲤科鱼类的线粒体基因组序列(表 1), 以花鳗鲡(序列号: NC_006540.1)为外类群, 通过Clustal W进行序列多重比对, 使用MEGA11.0软件采用邻接法构建Neighbor-joining系统进化树, 选择Bootstrap法检验(1 000次循环)各分支的置信度, 同时基于Kimura2-parameter计算16种鲤科鱼类间的遗传距离。
| 物种名 | NCBI序列登录号 | 总长(bp) |
| 细尾白甲鱼(Onychostoma lepturum) | NC_054158.1 | 16 601 |
| 虹彩光唇鱼(Acrossocheilus iridescens) | NC_031551.1 | 16 596 |
| 越南鱊(Acheilognathus tonkinensis) | NC_042407.1 | 16 767 |
| 东方墨头鱼(Garra orientalis Nichols) | NC_021935.1 | 17 288 |
| 麦穗鱼(Pseudorasbora parva) | NC_015614.1 | 16 600 |
| 高体鳑鲏(Rhodeus ocellatus) | NC_011211.1 | 16 680 |
| 条纹小鲃(Puntius semifasciolatus) | NC_020096.1 | 16 954 |
| 光倒刺鲃(Spinibarbus hollandi Oshima) | NC_026129.1 | 16 521 |
| 点纹银鮈(Squalidus wolterstorffi) | NC_022190.1 | 16 602 |
| 南方白甲鱼(Onychostoma gerlachi) | NC_026549.1 | 16 601 |
| 露丝塔野鲮(Labeo rohita) | NC_017608.1 | 16 626 |
| 麦瑞加拉鲮(Cirrhinus cirrhosus) | NC_033964.1 | 16 588 |
| 鲤(Cyprinus carpio) | NC_001606.1 | 16 575 |
| 鲫(Carassius auratus) | CM010491.1 | 16 579 |
| 间䱻(Hemibarbus medius Yue) | NC_024527.1 | 16 614 |
| 花鳗鲡(Anguilla marmorata) | NC_006540.1 | 16 745 |
尖鳍鲤线粒体全基因组序列为典型的环状结构, 线粒体基因组全长16 581 bp, PCGs总长度11 409 bp, 平均长度878 bp, 其长度占基因组的68.81%。碱基组成为A (31.96%)、G(15.69%)、C(27.53%)和T(24.82%); A+T含量为56.78%, 显著大于G+C含量(43.22%)(表 2), 表现为明显的AT偏好性。尖鳍鲤线粒体基因组的排列顺序和结果组成与其他鱼类一致, 共38个基因(图 1): 包括13个PCGs, 22个tRNA基因, 2个rRNA基因(12S-rRNA和16S-rRNA)以和一个非编码控制区(D-loop区)。
| 区域 | 长度/bp | (T/U)/% | C/% | A/% | G/% | A+T/% | G+C/% |
| 线粒体基因组 | 16 581 | 24.82 | 27.53 | 31.96 | 15.69 | 56.78 | 43.22 |
| 蛋白质编码基因 | 11 409 | 26.68 | 28.28 | 29.93 | 15.11 | 56.61 | 43.39 |
| 第一位点 | 3 803 | 26.14 | 27.03 | 30.84 | 15.99 | 56.98 | 43.02 |
| 第二位点 | 3 803 | 27.22 | 30.5 | 27.93 | 14.36 | 55.14 | 44.86 |
| 第三位点 | 3 803 | 26.69 | 27.29 | 31.03 | 14.99 | 57.72 | 42.28 |
| tRNA | 1 562 | 27.08 | 20.87 | 28.94 | 23.11 | 56.02 | 43.98 |
 |
| 图 1 尖鳍鲤线粒体基因组图谱 Fig. 1 Mitochondrial genome map of C. acutidorsalis |
22个tRNA的序列总长度为1 562 bp, 平均长度为71 bp。2个rRNA的序列总长度为2 632 bp。尖鳍鲤的线粒体基因组、PCGs、第一位点、第二位点、第三位点、tRNA的碱基AT含量均比GC含量高(表 2)。线粒体控制区(D-loop区)结构位于基因tRNAPhe和tRNAPro之间, 长度为927 bp, A+T含量为69.6%。尖鳍鲤12S rRNA位于tRNAPhe和tRNAVal之间, 由952个碱基组成; 16S rRNA位于tRNA-Val和tRNA-Leu之间, 由1 679个碱基组成, 12S rRNA比16S rRNA更加保守。基因tRNAGln、tRNATrp、ATP6、CO III、tRNAGly、tRNAArg、ND4、ND6、tRNAPro存在着碱基重叠的现象, 重叠碱基数1~7 bp, 最长的重叠区位于ATP8和ATP6基因、ND4L和ND4基因之间。除tRNAGln、tRNAAla、tRNAAsn、ND6、tRNACys、tRNALeu (CUN)、tRNATyr、tRNASer (UCN)、tRNAPro外, 其余基因的排列于H链上(表 3)。
| 基因 | 起始位置 | 长度/bp | 编码链 | 间隔片段长度/bp | 起始密码子 | 终止密码子 | 基因 | 起始位置 | 长度/bp | 编码链 | 间隔片段长度/bp | 起始密码子 | 终止密码子 |
| tRNAPhe | 1~69 | 69 | H | — | — | — | ATP8 | 7 954~8 118 | 165 | H | 1 | ATG | TAG |
| 12S rRNA | 70~1 022 | 952 | H | 0 | — | — | ATP6 | 8 112~8 795 | 684 | H | –7 | ATG | TAA |
| tRNAVal | 1 025~1 096 | 72 | H | 2 | — | — | CO III | 8 795~9 580 | 786 | H | 0 | ATG | TAA |
| 16S rRNA | 1 097~2 775 | 1679 | H | 0 | — | — | tRNAGly | 9 580~9 651 | 72 | H | –1 | — | — |
| tRNALeu (UUR) | 2 776~2 851 | 76 | H | 0 | — | — | ND3 | 9 652~10 002 | 351 | H | 0 | ATG | TAG |
| ND1 | 2 853~3 827 | 975 | H | 1 | ATG | TAA | tRNAArg | 10 001~10 070 | 70 | H | –2 | — | — |
| tRNAIle | 3 832~3 903 | 72 | H | 4 | — | — | ND4L | 10 071~10 367 | 297 | H | 0 | ATG | TAA |
| tRNAGln | 3 902~3 972 | 71 | L | -2 | — | — | ND4 | 10 361~11 741 | 1381 | H | –7 | ATG | T |
| tRNAMet | 3 975~4 043 | 69 | H | 2 | — | — | tRNAHis | 11 742~11 810 | 69 | H | 0 | — | — |
| ND2 | 4 044~5 090 | 1047 | H | 0 | ATG | TAG | tRNASer (AGY) | 11 811~11 879 | 69 | H | 0 | — | — |
| tRNATrp | 5 089~5 159 | 71 | H | -2 | — | — | tRNALeu | 11 881~11 953 | 73 | H | 1 | — | — |
| tRNAAla | 5 162~5 230 | 69 | L | 2 | — | — | ND5 | 11 957~13 780 | 1824 | H | 3 | ATG | TAA |
| tRNAAsn | 5 232~5 304 | 73 | L | 1 | — | — | ND6 | 13 777~14 298 | 522 | L | –4 | ATG | TAA |
| tRNACys | 5 338~5 404 | 67 | L | 33 | — | — | tRNALeu (CUN) | 14 299~14 367 | 69 | L | 0 | — | — |
| tRNATyr | 5 405~5 474 | 70 | L | 0 | — | — | Cytb | 14 373~15 513 | 1141 | H | 5 | ATG | T |
| CO I | 5 482~7 026 | 1545 | H | 7 | ATC | TAA | tRNAThr | 15 514~15 585 | 72 | H | 0 | — | — |
| tRNASer (UCN) | 7 027~7 097 | 71 | L | 0 | — | — | tRNAPro | 15 585~15 654 | 70 | L | –1 | — | — |
| tRNAAsp | 7 101~7 172 | 72 | H | 3 | — | — | tRNALys | 7 877~7 952 | 76 | H | 0 | — | — |
| CO II | 7 186~>787 | 691 | H | 13 | ATG | T | D-loop | 15 655~16 581 | 927 | H | 0 | — | — |
对13个PCGs的RSCU值统计显示(图 2), CGA(Arg)、CTA(Leu)、TCA(Ser)和CCA(Pro)是最常用的4种密码子, 其中, CGA的含量最高(3.506%), CTA含量为2.87%, 其他2种密码子的含量均在2%以上。另外, 尖鳍鲤线粒体基因组中编码同一氨基酸的密码子的CGA、CGC、AAC、TGC、CAA、CAC、ATT、CTA、AAA、ATG、TTC、CCA、AGC、TCA、TCC、ACA、ACC、TGG、TAC的RSCU均大于1, 共19个, 均为偏好性(Codon Usage Bias)密码子, 其余的密码子有较弱的使用偏性。尖鳍鲤的高频密码子中有11个是以C/G碱基结尾, 其余8个以A/T碱基结尾(表 4)。
 |
| 图 2 尖鳍鲤线粒体基因组密码子的偏好性 Fig. 2 Codon bias in the mitochondrial genome of C. acutidorsalis |
| 密码子 | 氨基酸 | RSCU | tRNA | 密码子 | 氨基酸 | RSCU | tRNA | |
| CGA | Arg | 3.506 | tRNAArg | ATA | Met | 0.904 | ||
| CGC | Arg | 1.091 | ATG | Met | 1.000 | tRNAMet | ||
| CGG | Arg | 0.623 | TTC | Phe | 1.300 | tRNAPhe | ||
| CGT | Arg | 0.701 | TTT | Phe | 0.700 | |||
| AAC | Asn | 1.267 | tRNAAsn | CCA | Pro | 2.208 | tRNAPro | |
| AAT | Asn | 0.733 | CCC | Pro | 0.981 | |||
| TGC | Cys | 1.600 | tRNACys | CCG | Pro | 0.189 | ||
| TGT | Cys | 0.400 | CCT | Pro | 0.623 | |||
| CAA | Gln | 1.960 | tRNAGln | AGC | Ser | 1.114 | tRNASer | |
| CAG | Gln | 0.040 | AGT | Ser | 0.203 | |||
| CAC | His | 1.577 | tRNAHis | TCA | Ser | 2.203 | tRNASer | |
| CAT | His | 0.423 | TCC | Ser | 1.519 | |||
| ATC | Ile | 0.988 | tRNAIle | TCG | Ser | 0.228 | ||
| ATT | Ile | 1.108 | TCT | Ser | 0.734 | |||
| CTA | Leu | 2.870 | tRNALeu | ACA | Thr | 1.967 | tRNAThr | |
| CTC | Leu | 0.970 | ACC | Thr | 1.365 | |||
| CTG | Leu | 0.346 | ACG | Thr | 0.107 | |||
| CTT | Leu | 0.758 | ACT | Thr | 0.562 | |||
| TTA | Leu | 0.941 | tRNALeu | TGG | Trp | 1.000 | ||
| TTG | Leu | 0.115 | TAC | Tyr | 1.096 | tRNATyr | ||
| AAA | Lys | 1.870 | tRNALys | TAT | Tyr | 0.904 | ||
| AAG | Lys | 0.130 |
海南和广西尖鳍鲤13个PCGs序列相似性都在95%以上(表 5), 其中ATP8和Cytb相似性超过99%, 主要差异体现在碱基重叠的长度大小。海南岛尖鳍鲤的起始密码子除了CO I是ATC, 其他线粒体PCGs的起始密码子为ATG, 而广西尖鳍鲤的CO I基因的起始密码子为GTG, 其余同为ATC。广西尖鳍鲤蛋白编码基因存在TA这类不完全终止密码子。海南尖鳍鲤的CO II、ND4、和Cytb基因的不完全终止密码子为“T”, 广西尖鳍鲤PCGs的不完全终止密码子(T、TA)出现的频率比海南尖鳍鲤高。
| 基因 | 海南尖鳍鲤 | 广西尖鳍鲤 | PCGs相似性/% | |||||||
| 起止范围/bp | 长度/bp | 起始密码子 | 终止密码子 | 起止范围/bp | 长度/bp | 起始密码子 | 终止密码子 | |||
| ND1 | 2 853~3 827 | 975 | ATG | TAA | 2 853~3 827 | 975 | ATG | TAA | 98.36 | |
| ND2 | 4 044~5 090 | 1 047 | ATG | TAG | 4 044~5 088 | 1 045 | ATG | T | 97.71 | |
| COⅠ | 5 482~7 026 | 1 545 | ATC | TAA | 5 476~7 026 | 1 551 | GTG | TAA | 98.71 | |
| CO Ⅱ | 7 186~7 876 | 691 | ATG | T | 7 186~7 876 | 691 | ATG | T | 98.99 | |
| ATP8 | 7 954~8 118 | 165 | ATG | TAG | 7 954~8 118 | 165 | ATG | TAG | 99.39 | |
| ATP6 | 8 112~8 795 | 684 | ATG | TAA | 8 112~8 795 | 684 | ATG | TAA | 96.63 | |
| COⅢ | 8 795~9 580 | 786 | ATG | TAA | 8 795~9 579 | 785 | ATG | TA | 97.96 | |
| ND3 | 9 652~10 002 | 351 | ATG | TAG | 9 652~10 000 | 349 | ATG | T | 98.58 | |
| ND4L | 10 071~10 367 | 297 | ATG | TAA | 10 071~1 0367 | 297 | ATG | TAA | 98.65 | |
| ND4 | 10 361~11 741 | 1 381 | ATG | T | 10 361~11 741 | 1 381 | ATG | T | 98.41 | |
| ND5 | 11 956~13 780 | 1 824 | ATG | TAA | 11 957~13 780 | 1 823 | ATG | TAA | 98.63 | |
| ND6 | 13 777~14 298 | 522 | ATG | TAA | 13 777~14 298 | 522 | ATG | TAA | 98.28 | |
| Cytb | 14 373~15 513 | 1 141 | ATG | T | 14 373~15 513 | 1 141 | ATG | T | 99.91 | |
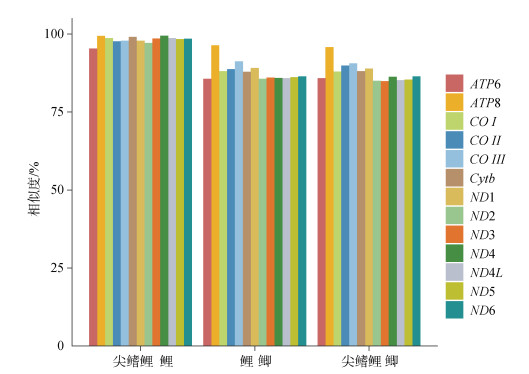
尖鳍鲤、鲤、鲫的线粒体基因排列顺序与其他大多数鱼类线粒体基因组一样均含有13个PCGs、2种rRNA基因和22种tRNA基因及非编码区的轻链复制起始区和复制和转录序列(D-loop)。3种鲤科鱼类的线粒体基因长度十分相似, 并且13个PCGs中的部分基因的起始与终止密码子相同(表 5)。3种鲤科鱼类的PCGs的AT密码子的偏好性都在50%以上, 均具有AT偏好性。3种鲤亚科鱼类的起始密码子ATG和终止密码子TAA出现频率较高, 存在着完整和不完整两类终止密码子(表 6)。对3种鲤属鱼类的线粒体全基因组13个PCGs的编码序列进行两两比较, 结果显示3种鲤属鱼类的PCGs相似性在85%~99%(图 3), 尖鳍鲤和鲤的亲缘关系较鲫更近。尖鳍鲤和鲤的基因ATP8、ND4、Cytb两两比对结果之间的相似性都在99%以上(表 7), 其中ATP8是13个PCGs中相似性最高的基因。
| 基因 | 尖鳍鲤 | 鲫 | 鲤 | 起始密码子 | 终止密码子 | ||||||||
| 起始 | 终止 | 长度/bp | 起始 | 终止 | 长度/bp | 起始 | 终止 | 长度/bp | |||||
| tRNAPhe | 1 | 69 | 69 | 924 | 992 | 69 | 929 | 997 | 69 | ||||
| 12S rRNA | 70 | 1 022 | 952 | 993 | 1 946 | 954 | 929 | 1 948 | 1 019 | ||||
| tRNAVAl | 1 025 | 1 096 | 72 | 1 947 | 2 018 | 72 | 1 49 | 2 020 | 72 | ||||
| 16S rRNA | 1 097 | 2 775 | 1 679 | 2 019 | 3 699 | 1 681 | 2 21 | 3 701 | 1 681 | ||||
| tRNALeu | 2 776 | 2 851 | 76 | 3 700 | 3 777 | 78 | 3 02 | 3 778 | 77 | ||||
| ND1 | 2 853 | 3 827 | 975 | 3 778 | 4 752 | 975 | 3 79 | 4 753 | 975 | ATG | TAA | ||
| tRNAIle | 3 832 | 3 903 | 72 | 4 756 | 4 829 | 74 | 4 757 | 4 829 | 73 | ||||
| tRNAGln | 3 902 | 3 973 | 71 | 4 827 | 4 897 | 69 | 4 827 | 4 897 | 71 | ||||
| tRNAMet | 3 975 | 4 043 | 69 | 4 899 | 4 967 | 69 | 4 900 | 4 968 | 69 | ||||
| ND2 | 4 044 | 5 090 | 1 047 | 4 968 | 6 014 | 1 047 | 4 969 | 6 015 | 1 047 | ATG | TAG | ||
| tRNATrp | 5 089 | 5 159 | 71 | 6 013 | 6 083 | 71 | 6 014 | 6 084 | 71 | ||||
| tRNAAla | 5 162 | 5 230 | 69 | 6 086 | 6 154 | 69 | 6 087 | 6 154 | 68 | ||||
| tRNAAsn | 5 232 | 5 304 | 73 | 6 156 | 6 228 | 73 | 6 156 | 6 228 | 73 | ||||
| tRNACys | 5 338 | 5 404 | 67 | 6 261 | 6 331 | 71 | 6 261 | 6 329 | 69 | ||||
| tRNATyr | 5 405 | 5 474 | 70 | 6 330 | 6 400 | 71 | 6 328 | 6 397 | 70 | ||||
| CO I | 5 482 | 7 026 | 1 545 | 6 402 | 7 952 | 1 551 | 6 399 | 7 964 | 1 566 | ATC/GTG/GTG | TAA | ||
| tRNASer | 7 027 | 7 097 | 71 | 7 953 | 8 032 | 80 | 7 950 | 8 020 | 71 | ||||
| tRNAAsp | 7 101 | 7 172 | 72 | 8 27 | 8 098 | 72 | 8 024 | 8 095 | 72 | ||||
| CO II | 7 186 | 7 876 | 691 | 8 11 | 8 801 | 691 | 8 109 | 8 799 | 691 | ATG | T | ||
| tRNALys | 7 877 | 7 952 | 76 | 8 02 | 8 877 | 76 | 8 800 | 8 875 | 76 | ||||
| ATP8 | 7 954 | 8 118 | 165 | 8 79 | 9 043 | 165 | 8 877 | 9 041 | 165 | ATG | TAG | ||
| ATP6 | 8 112 | 8 795 | 684 | 9 37 | 9 720 | 694 | 9 035 | 9 718 | 684 | ATG | TAA | ||
| CO Ⅲ | 8 795 | 9 580 | 786 | 9 20 | 10 505 | 786 | 9 718 | 10 503 | 786 | ATG | TAA | ||
| tRNAGly | 9 580 | 9 651 | 72 | 10 05 | 10 576 | 72 | 10 503 | 10 574 | 72 | ||||
| ND3 | 9 652 | 10 002 | 351 | 10 77 | 10 927 | 351 | 10 575 | 10 925 | 351 | ATG | TAG | ||
| tRNAArg | 10 001 | 10 070 | 70 | 10 26 | 10 995 | 70 | 10 924 | 10 993 | 70 | ||||
| ND4L | 10 071 | 10 367 | 297 | 10 96 | 11 292 | 297 | 10 994 | 11 290 | 297 | ATG | TAA | ||
| ND4 | 10 361 | 11 741 | 1 381 | 11 86 | 12 666 | 1 381 | 11 284 | 12 664 | 1 381 | ATG | T | ||
| tRNAHis | 11 742 | 11 810 | 69 | 12 667 | 12 735 | 69 | 12 665 | 12 733 | 69 | ||||
| tRNASer | 11 811 | 11 879 | 69 | 12 736 | 12 804 | 69 | 12 734 | 12 802 | 69 | ||||
| tRNALeu | 11 881 | 11 953 | 73 | 12 806 | 12 878 | 73 | 12 734 | 12 802 | 69 | ||||
| ND5 | 11 957 | 13 780 | 1 824 | 12 882 | 14 705 | 1824 | 12 880 | 14 703 | 1 824 | ATG | TAA | ||
| ND6 | 13 777 | 14 298 | 522 | 14 702 | 15 223 | 522 | 14 700 | 15 218 | 519 | ATG | TAA/TAG/TAA | ||
| tRNALeu | 14 299 | 14 367 | 69 | 15 224 | 15 292 | 69 | 15 219 | 15 287 | 69 | ||||
| Cytb | 14 373 | 15 513 | 1 141 | 15 298 | 16 438 | 1141 | 15 293 | 16 433 | 1 141 | ATG | T | ||
| tRNAThr | 15 514 | 15 585 | 72 | 16 439 | 16 510 | 72 | 16 434 | 16 505 | 72 | ||||
| tRNAPro | 15 585 | 15 654 | 70 | 16 509 | 16 580 | 72 | 16 504 | 16 575 | 72 | ||||
| D-loop | 15 655 | 16 581 | 927 | 1 | 923 | 923 | 1 | 928 | 928 | ||||
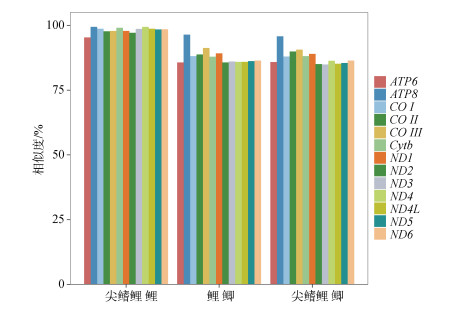 |
| 图 3 3种鲤亚科鱼类的13个蛋白编码基因之间的相似性比较 Fig. 3 Similarity of 13 protein-coding genes among the three Cyprininae species |
| 种 | 碱基 | ND1 | ND2 | CO I | CO II | ATP8 | ATP6 | CO III | ND3 | ND4L | ND4 | ND5 | ND6 | Cytb |
| 尖鳍鲤 | A+T | 54.18 | 54.29 | 53.1 | 58.85 | 64.29 | 56.46 | 56.66 | 60.43 | 56.33 | 53.43 | 56.12 | 63.57 | 58.8 |
| T | 23.27 | 20.36 | 24.05 | 28.23 | 41.43 | 22.97 | 27.14 | 23.74 | 28.74 | 23.43 | 23.47 | 40 | 27.57 | |
| C | 26.18 | 34.64 | 27.62 | 25.84 | 22.86 | 30.14 | 32.38 | 20.86 | 31.03 | 30 | 31.43 | 10.71 | 29.24 | |
| A | 30.91 | 33.93 | 29.05 | 30.62 | 25.71 | 33.49 | 29.52 | 36.69 | 27.59 | 30 | 32.65 | 13.57 | 31.23 | |
| G | 19.64 | 11.07 | 19.29 | 15.31 | 10 | 13.4 | 10.95 | 18.71 | 12.64 | 16.57 | 12.45 | 35.71 | 11.96 | |
| 鲤 | A+T | 54.91 | 53.21 | 53.1 | 58.41 | 67.14 | 56.18 | 57.14 | 43.66 | 52.83 | 53.43 | 56.53 | 51.15 | 58.74 |
| T | 24.36 | 20 | 23.81 | 27.86 | 40 | 23.71 | 27.62 | 18.31 | 28.57 | 23.43 | 23.06 | 39.29 | 28.37 | |
| C | 25.82 | 34.29 | 27.62 | 26.87 | 22.85 | 31.44 | 32.38 | 25.35 | 32.86 | 29.42 | 31.22 | 10 | 28.37 | |
| A | 30.55 | 33.21 | 29.29 | 30.85 | 27.14 | 32.47 | 29.52 | 38.03 | 24.26 | 30 | 33.47 | 12.86 | 30.37 | |
| G | 19.27 | 11.07 | 19.29 | 14.43 | 10 | 12.37 | 10.48 | 18.31 | 14.29 | 17.14 | 12.24 | 37.86 | 12.89 | |
| 鲫 | A+T | 55.2 | 55.71 | 51.43 | 61.69 | 61.4 | 61.86 | 54.29 | 52.11 | 60.92 | 59.72 | 60.61 | 50.71 | 60.47 |
| T | 25.09 | 20.71 | 23.57 | 29.35 | 17.14 | 29.9 | 25.24 | 16.9 | 34.48 | 28.29 | 26.94 | 37.14 | 30.9 | |
| C | 24.73 | 24.64 | 28.57 | 23.88 | 30 | 26.8 | 34.76 | 28.16 | 27.59 | 24.86 | 27.55 | 13.57 | 26.25 | |
| A | 30.11 | 35 | 27.86 | 32.34 | 44.26 | 31.96 | 29.05 | 35.21 | 26.44 | 31.43 | 33.67 | 13.57 | 29.57 | |
| G | 20.07 | 9.64 | 20 | 14.43 | 8.57 | 11.34 | 10.95 | 19.72 | 11.49 | 15.43 | 11.84 | 35.71 | 13.29 | |
| 相似性 | ND1 | ND2 | CO I | CO II | ATP8 | ATP6 | CO III | ND3 | ND4L | ND4 | ND5 | ND6 | Cytb | |
| 尖鳍鲤和鲤/% | 97.85 | 97.13 | 98.71 | 97.68 | 99.39 | 95.32 | 97.84 | 98.58 | 98.68 | 99.42 | 98.36 | 98.47 | 99.04 | |
| 鲤和鲫/% | 89.13 | 85.67 | 88.07 | 88.71 | 96.36 | 85.67 | 91.22 | 86.04 | 85.86 | 85.88 | 86.13 | 86.40 | 87.91 | |
| 尖鳍鲤和鲫/% | 88.92 | 85.00 | 87.94 | 89.87 | 95.76 | 85.82 | 90.59 | 84.90 | 85.19 | 86.31 | 85.42 | 86.40 | 88.08 | |
为进一步了解鲤科鱼类的系统进化关系, 以花鳗鲡为外类群, 基于邻接法构建尖鳍鲤和其他鲤科鱼类的系统进化树(图 4)。该进化树由两大进化分支构成, 尖鳍鲤和鲤聚为一支, 亲缘关系最近。由进化树可知尖鳍鲤和其他鲤鱼之间有较相近的亲缘关系, 虹彩光唇鱼、条纹小鲃、光刺倒鲃、鲫、东方墨头鱼和麦穗鱼均为单独的一支。由Kimura2-parameter模型计算出的各鲤科鱼类间的遗传距离显示(表 8), 平均遗传距离为0.184, 其中遗传距离最小为尖鳍鲤和鲤(0.046), 进一步表明二者亲缘关系十分接近。
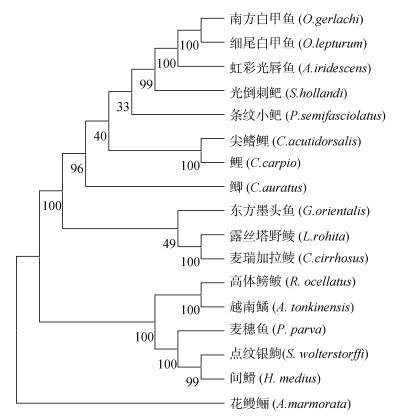 |
| 图 4 基于线粒体基因组全序列构建的NJ系统进化树 Fig. 4 NJ phylogenetic tree based on the complete mitochondrial genome sequence |
| 种名 | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 |
| 1.鲫鱼 | ||||||||||||||||
| 2.尖鳍鲤 | 0.148 | |||||||||||||||
| 3.鲤 | 0.113 | 0.046 | ||||||||||||||
| 4.高体鳑鲏 | 0.224 | 0.264 | 0.229 | |||||||||||||
| 5.麦穗鱼 | 0.197 | 0.238 | 0.203 | 0.231 | ||||||||||||
| 6.露丝塔野鲮 | 0.136 | 0.151 | 0.118 | 0.228 | 0.198 | |||||||||||
| 7.条纹小鲃 | 0.128 | 0.156 | 0.121 | 0.236 | 0.208 | 0.132 | ||||||||||
| 8.东方墨头鱼 | 0.150 | 0.180 | 0.146 | 0.249 | 0.219 | 0.143 | 0.166 | |||||||||
| 9.点纹银鮈 | 0.175 | 0.213 | 0.177 | 0.212 | 0.168 | 0.169 | 0.184 | 0.199 | ||||||||
| 10.间䱻 | 0.181 | 0.220 | 0.184 | 0.224 | 0.172 | 0.180 | 0.193 | 0.213 | 0.139 | |||||||
| 11.光倒刺鲃 | 0.139 | 0.168 | 0.133 | 0.252 | 0.222 | 0.145 | 0.151 | 0.176 | 0.201 | 0.210 | ||||||
| 12.南方白甲鱼 | 0.148 | 0.172 | 0.136 | 0.249 | 0.232 | 0.146 | 0.148 | 0.179 | 0.200 | 0.212 | 0.157 | |||||
| 13.虹彩光唇鱼 | 0.142 | 0.168 | 0.133 | 0.247 | 0.226 | 0.143 | 0.144 | 0.175 | 0.196 | 0.209 | 0.148 | 0.108 | ||||
| 14.麦瑞加拉鲮 | 0.127 | 0.150 | 0.114 | 0.237 | 0.211 | 0.091 | 0.133 | 0.152 | 0.176 | 0.188 | 0.150 | 0.149 | 0.145 | |||
| 15.越南鱊 | 0.214 | 0.257 | 0.221 | 0.197 | 0.217 | 0.216 | 0.231 | 0.242 | 0.208 | 0.214 | 0.244 | 0.240 | 0.242 | 0.231 | ||
| 16.细尾白甲鱼 | 0.148 | 0.172 | 0.137 | 0.248 | 0.234 | 0.146 | 0.151 | 0.176 | 0.201 | 0.210 | 0.155 | 0.067 | 0.108 | 0.150 | 0.241 |
尖鳍鲤线粒体基因组的基因分布组成和排列顺序与现有已知的鲤科鱼类相似, 各基因长度基本一致, 线粒体基因组中PCGs的密码子使用与其他脊椎动物相似, 可知线粒体基因组在进化上是高度保守的[19]。海南尖鳍鲤线粒体基因由37个基因组成, 排列紧凑, 存在基因重叠的情况。尖鳍鲤中基因重叠区有8处, 重叠碱基数在1~7 bp间, 共26 bp, 13个蛋白编码重复区有3处, 这种基因重叠特征同样出现在其他鱼类, 如麦穗鱼[20]、南方白甲鱼[21], 其中个别基因重叠的碱基数略有差异, 如玻璃缺鳍鲶(Kryptopterus vitreolus)[22]的ATP8和ATP6基因之间重叠为10 bp。研究表明, 由于DNA双链之间的自然突变和选择压力的差异, 碱基在整个基因组上的分布往往是不均匀的[23]。在线粒体基因组全序列分析中, 密码子AT碱基偏好性和反G现象, 且存在RSCU大于1的高频密码子, 如TCC、TCA等[24]。在19个偏好性密码子中以G或C碱基结尾的占了大约58%, 说明以G或C碱基结尾比以A或T碱基结尾出现的频率要高一些, 且尖鳍鲤线粒体基因的D-loop区A和T碱基频率较高(69.6%)。线粒体基因组中碱基偏好性的原因可能是选择压力、自然突变等因素[25]。这些特征与其他鲤科鱼类一样, 如鲤科鳈属中华鳈(Sarcocheilichthys sinensis)、小鳈(S. parvus)、黑鳍鳈(S. nigripinnis) [26]的A+T密码子富集在D-loop区, 平均含量明显大于G+C含量。海南尖鳍鲤线粒体ND6基因在L链上, 其余PCGs分布于H链上。研究表明, 分布在H链上的线粒体基因倾向于形成单链保护, 对水解和氧化更敏感[27]。在尖鳍鲤13个PCGs中的CO I、ND4、Cytb基因中存在着T不完全终止密码子, 再经过转录、加工补全成完整的终止密码子, 提高了碱基的利用率, 更加高效与节约[28]。
3.2 海南和广西水域尖鳍鲤PCGs的差异性海南尖鳍鲤线粒体基因组长度微小的差异可能是不同群体之间的非编码基因的长度多态性导致的[2]。鱼类中起始密码子的使用可能存在特定的模式和差异, 可能是地理种群隔离的结果[29]。海南尖鳍鲤的ND2、CO III和ND3蛋白编码基因略长于广西尖鳍鲤, 表现出位点多态性。生活在海南的尖鳍鲤和广西的尖鳍鲤关系十分密切, 表现出高度相似性, 但由于两者是在不同地理间的同一种群, 仍会存在一定的差异性。这与不同地理种群的高体鰤(Seriola dumerili)[30]是和暗色唇鲮(Semilabeo obscures) [31]的研究结果相似, 中国和日本海域高体鰤CO II表现出位点的多态性猜测是由地理种群隔离形成的。在海南和广西水域尖鳍鲤差异性比较中发现CO I基因的起始密码子不同, 前者为ATG, 后者为GTC。在暗色唇鲮和瓯江高体鳑鲏(Rhodeus ocellatus) [32]线粒体基因组测序研究中, 它们的CO I基因起始密码子为GTC, 而在孙利元等[33]研究的8种石首鱼科(Sciaenidae)CO I基因的起始密码子均为ATG。
3.3 尖鳍鲤、鲤和鲫线粒基因组特征的差异性根据尖鳍鲤、鲤以及鲫的13个PCGs分别两两间进行多序列对比表明尖鳍鲤和鲤有着较高的相似度, 并且这3种鱼都表现为明显的AT碱基的偏好性, 推测原因是它们均属于鲤亚科, 尖鳍鲤和鲤同属鲤亚科鲤属、而鲫属于须鲫属(Carassioides)。3种鱼ATP8基因G的含量都小于或等于10%, 存在着反G现象。尖鳍鲤的12S rRNA基因比鲤长2 bp, 比鲫长67 bp, 结合3种鲤亚科相似性分析说明尖鳍鲤和鲤的相似度更高, 两者亲缘关系最近。3种鱼类的13个PCGs中ATP8基因相似性最高, 均在95%以上。尖鳍鲤和鲤的ND4基因相对最保守, 相似度为高达99.42%, 其次是ATP8(99.39%)和Cytb(99.04%), 表明ND4基因在两种鱼类中都存在非常保守的功能和序列, 受到的选择压力和功能束缚更强。这一结果也可以用来支持分类学上对尖鳍鲤和鲤的归属问题, 进一步支持了它们可能属于同一种鱼类或者亲缘关系非常接近的亚种的假设。而在对包括玻璃缺鳍鲶在内的5种鱼类核苷酸序列比对, CO I基因相似度为78.98%~ 92.78%, 相对最保守[22]。由于海南和广西尖鳍鲤线粒体基因组的PCGs相似性都在95%以上, 差异不显著, 不适用于鉴别不同地理区域的尖鳍鲤, 但PCGs可以有效区分尖鳍鲤、鲤和鲫。在3种鲤亚科鱼类完整终止密码子有TAA、TAG, 基因CO II、ND4、Cytb的终止密码子都为不完整的T, 在同为鲤科鱼类麦穗鱼蛋白编码基因的密码子使用情况研究中, 麦穗鱼不完整终止密码子为TA或T[20], 南方白甲鱼[21]完全终止密码子为TAA和TAG, 不完全终止密码子与麦穗鱼相同。
3.4 尖鳍鲤系统发育分析鲤科鱼类是淡水中最大的科, 其形态学鉴定是长期以来在分类学研究中相对困难的工作[34]。鲤系分为野鲮亚科(Labeoninae)和鲤亚科(鲃亚科(Barbinae)、鲤亚科、裂腹鱼亚科(Schizothoracinae))两大类, 鲤在分类上隶属于鲤形目、鲤亚目、鲤科、鲤属, 分布区间东起中国和西伯利亚、西至罗马, 是淡水硬骨鱼种最重要的一类[35], 尖鳍鲤与鲤在分类学都属于鲤属。本研究的系统发育进化树中, 尖鳍鲤与鲤同属于一个进化支, 表明两者的形态分类和分子进化结果可能类似, 种间差异小, 亲缘关系最近。尖鳍鲤和鲤的遗传距离仅为0.046, 并且两者的线粒体基因组PCGs相似性均大于95%, 猜测可能具有相同或相似的基因组成, 从而使两者具有类似的形态特征和生理行为。在进化树中, 点纹银鮈(Squalidus wolterstorffi)和间䱻(Hemibarbus medius)处于系统树根部的位置, 而尖鳍鲤和鲤演化时间较晚。在姚东林等[17]的研究中, 尖鳍鲤和三角鲤(C. multitaeniata)、锦鲤(C. carpio haematopterus)亲缘关系也十分接近, 遗传距离分别为0.003和0.013, 三者都归属于鲤亚科, 说明鲤亚科鱼类在遗传和进化方面存在高度的相似背景。尖鳍鲤和鲤两者在遗传上高度相似, 但确定它们确切的进化关系, 还需借助分子标记如SSR(简单重复序列标记)、SNP(单核苷酸多态性)等手段。
4 结论本研究利用二代测序, 测定了海南尖鳍鲤的线粒体基因组全序列、基因含量、碱基组成、密码子偏好性等, 明确了尖鳍鲤线粒体基因基本结构和特征。基于DNAMAN的13个PCGs序列比较表明, 海南尖鳍鲤与广西种群基因的相似性在95%以上。分析比较3种鲤科鱼类的线粒体基因组结构特征, 海南尖鳍鲤和鲤的亲缘关系最近。研究结果为海南尖鳍鲤种群遗传结构和系统进化分析提供依据, 为解释尖鳍鲤的起源和进化以及保护生物多样性提供理论基础。
| [1] |
XIAO W H, ZHANG Y P. Genetics and evolution of mitochondrial DNA in fish[J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384-391. DOI:10.3321/j.issn:1000-3207.2000.04.014 |
| [2] |
GUO X H, LIU S J, LIU Q, et al. New progresses on mitochondrial DNA in fish[J]. Acta Genetica Sinica, 2004, 31(9): 983-1000. |
| [3] |
PROSDOCIMI F, DE CARVALHO D C, DE ALMEIDA R N, et al. The complete mitochondrial genome of two recently derived species of the fish genus Nannoperca (Perciformes, Percichthyidae)[J]. Molecular Biology Reports, 2012, 39(3): 2767-2772. DOI:10.1007/s11033-011-1034-5 |
| [4] |
CHANG Y, HUANG F, LO T. The complete nucleotide sequence and gene organization of carp (Cyprinus carpio) mitochondrial genome[J]. Journal of Molecular Evolution, 1994, 38(2): 138-155. DOI:10.1007/BF00166161 |
| [5] |
BERNT M, BRABAND A, SCHIERWATER B, et al. Genetic aspects of mitochondrial genome evolution[J]. Molecular Phylogenetics and Evolution, 2013, 69(2): 328-338. DOI:10.1016/j.ympev.2012.10.020 |
| [6] |
DONG C, XU J, WANG B, et al. Phylogeny and evolution of multiple common carp (Cyprinus carpio L.) populations clarified by phylogenetic analysis based on complete mitochondrial genomes[J]. Marine Biotechnology, 2015, 17(5): 565-575. DOI:10.1007/s10126-015-9639-7 |
| [7] |
BROUGHTON R E, MILAM J E, ROE B A. The complete sequence of the zebrafish (Danio rerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J]. Genome Research, 2001, 11(11): 1958-1967. DOI:10.1101/gr.156801 |
| [8] |
LIU J, DING X, ZENG Y, et al. Genetic diversity and phylogenetic evolution of Tibetan sheep based on mtDNA D-Loop sequences[J]. PLoS One, 2016, 11(7): e0159308. DOI:10.1371/journal.pone.0159308 |
| [9] |
BING P X, HE S L, ZHI L Z, et al. DNA barcoding for identification of fish species in the Taiwan Strait[J]. PloS One, 2018, 13(6): e0198109. DOI:10.1371/journal.pone.0198109 |
| [10] |
KOWALCZYK M, STANISZEWSKI A, KAMIñSKA K, et al. Advantages, possibilities, and limitations of mitochondrial DNA analysis in molecular identification[J]. Folia Biologica (Kraków), 2021, 69(3): 101-111. |
| [11] |
SCHROETER J C, MALOY A P, REES C B, et al. Fish mitochondrial genome sequencing: expanding genetic resources to support species detection and biodiversity monitoring using environmental DNA[J]. Conservation Genetics Resources, 2020, 12: 433-446. DOI:10.1007/s12686-019-01111-0 |
| [12] |
姚东林, 李正光, 王超等. 华南地区6个鲤亚科鱼类种群的氨基酸价值[J]. 西北农业学报, 2017, 26(7): 983-989. YAO Donglin, LI Zhengguang, WANG Chao, et al. Amino acid analysis of six kinds of genes Cyprininae fish population in southern China[J]. Acta Agriculturae Boreali-Occidentalis Sinica, 2017, 26(7): 983-989. |
| [13] |
王幼槐. 中国鲤亚科鱼类的分类、分布、起源及演化[J]. 水生生物学集刊, 1979, 6(4): 424. WANG Youhuai. On the classification, distribution, origin and evolution of the fishes referred to the subfamily cyprininae of China, with description of a new species[J]. Acta Hydrobiologica Sinica, 1979, 6(4): 424. |
| [14] |
申志新, 李高俊, 蔡杏伟等. 海南省淡水野生鱼类多样性演变及保护建议[J]. 中国水产, 2018, 516(11): 56-60. SHEN Zhixin, LI Gaojun, CAI Xingwei, et al. The evolution and protection of freshwater fish species in Hainan Province[J]. China Fisheries, 2018, 516(11): 56-60. |
| [15] |
姚东林, 王超, 吕子君等. 尖鳍鲤的肌肉基本营养成分和脂肪酸组成[J]. 广东农业科学, 2014, 41(19): 131-134. YAO Donglin, WANG Chao, LV Zijun, et al. Study on basic nutrition and fatty acid composition of muscle in Cyprinus acutidorsalis Wang[J]. Guangdong Agricultural Sciences, 2014, 41(19): 131-134. |
| [16] |
易祖盛, 王春, 陈湘粦. 尖鳍鲤的早期发育[J]. 中国水产科学, 2002, 2: 120-124, 198-199. YI Zusheng, WANG Chun, CHEN Xianglin. Embryonic and larval development of sea carp, Cyprinus acutidorsali[J]. Journal of Fishery Sciences of China, 2002, 2: 120-124, 198-199. |
| [17] |
姚东林, 王超, 周爱国, 等. 基于Cyt b标记研究尖鳍鲤在鲤形目鱼类中的系统发育地位[J]. 广东农业科学, 2014, 41(12): 114-118. YAO Donglin, WANG Chao, ZHOU Aiguo, et al. Phylogenetic analysis of Cyprinus acutidorsalis Wang in Cypriniformes based on the Cyt b marker[J]. Guangdong Agricultural Sciences, 2014, 41(12): 114-118. |
| [18] |
WANG C, CHEN J T, YAO D L, et al. The complete mitochondrial genome of the sea carp, Cyprinus acutidorsalis (Cypriniformes: Cyprinidae)[J]. Mitochondrial DNA, 2015, 26(5): 686-687. |
| [19] |
JACOBSEN M W, DA FONSECA R R, BERNATCHEZ L, et al. Comparative analysis of complete mitochondrial genomes suggests that relaxed purifying selection is driving high nonsynonymous evolutionary rate of the NADH2 gene in whitefish (Coregonus ssp.)[J]. Molecular Phylogenetics and Evolution, 2016, 95: 161-170. |
| [20] |
陈涛, 史妍茹, 尤平. 麦穗鱼线粒体基因组序列测定及分析[J]. 动物分类学报, 2012, 37(1): 10-19. CHEN Tao, SHI Yanru, YOU Ping. Sequence and analysis of complete mitochondrialgenome of pseudorasbora parva[J]. Acta Zootaxonomica Sinica, 2012, 37(1): 10-19. |
| [21] |
程琪. 南方白甲鱼线粒体基因组序列测定分析及白甲鱼属系统发育研究[D]. 武汉: 中南民族大学, 2016. CHENG Qi. Complete mitochondrial genome sequencing and analysis of Onychostoma gerlachi and phylogenetic research of genus onychostoma[D]. Wuhan: South-central University For Nationalities, 2016. |
| [22] |
孙志鹏, 吕伟华, 匡友谊, 等. 玻璃缺鳍鲶线粒体全基因组序列测定与分析[J]. 水产学杂志, 2018, 31(2): 12-19. SUN Zhipeng, LV Huawei, KUANG Youyi, et al. Sequencing and analysis of complete mitochondrial genome of catfish Kryptopterus vitreolus[J]. Chinese Journal of Fisheries, 2018, 31(2): 12-19. |
| [23] |
钟东, 赵贵军, 张振书, 等. 基因组内碱基分布整体均衡与局部不均衡的研究进展[J]. 遗传, 2002, 3: 351-355. ZHONG Dong, ZHAO Guijun, ZHANG Zhenshu, et al. Advance in the entire balance and local unbalance of base distribution in genome[J]. Hereditas(Beijing), 2002, 3: 351-355. |
| [24] |
KAMARAJ B, KUMAR A, PUROHIT R. Evolutionary reconstruction and population genetics analysis of aurora kinases[J]. Plos One, 2013, 8(9): e75763. |
| [25] |
WEI L, HE J, JIA X, et al. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution[J]. BMC Evolutionary Biology, 2014, 14: 1-12. |
| [26] |
王恒. 三种鳈属鱼类线粒体全基因组的测序与比较[D]. 合肥: 安徽农业大学, 2015. WANG Heng, Sequence and comparison of the mtDNA of three Sarcocheilichthys fishes[D]. Hefei: Anhui Agricultural University, 2015. |
| [27] |
BROWN W M, PRAGER E M, WANG A, et al. Mitochondrial DNA sequences of primates: tempo and mode of evolution[J]. Journal of Molecular Evolution, 1982, 18(4): 225-239. |
| [28] |
MONTOYA J, OJALA D, ATTARDI G. Distinctive features of the 5'-terminal sequences of the human mitochondrial mRNAs[J]. Nature, 1981, 290(5806): 465-470. |
| [29] |
DUCHAUD E, ROCHAT T, HABIB C, et al. Genomic diversity and evolution of the fish pathogen Flavobacterium psychrophilum[J]. Frontiers in Microbiology, 2018, 9: 138. |
| [30] |
王开杰, 徐永江, 崔爱君, 等. 高体鰤(Seriola dumerili)线粒体全基因组测定及结构特征分析[J]. 海洋与湖沼, 2022, 53(1): 120-132. WANG Kaijie, XU Yongjiang, CUI Aijun, et al. Complete sequence and gene organization of the mitochondrial genome of Seriola dumerili[J]. Oceanologia et Limnologia Sinica, 2022, 53(1): 120-132. |
| [31] |
韩耀全, 黄励, 朱琼华, 等. 基于Illumina Hiseq 4000高通量测序平台的暗色唇鲮线粒体基因组特征分析[J]. 福建农业学报, 2020, 35(2): 130-139. HAN Yaoquan, HUANG Li, ZHU Qionghua, et al. Characteristics of mitochondrial genomes of Semilabeo obscures analyzed using Illumina Hiseq 4000 High-throughput sequencing technique[J]. Fujian Journal of Agricultural Sciences, 2020, 35(2): 130-139. |
| [32] |
刘凯, 冯晓宇, 吴燕琴, 等. 瓯江高体鳑鲏线粒体基因组测序及结构特征分析[J]. 农业生物技术学报, 2021, 29(6): 1182-1197. LIU Kai, FENG Xiaoyu, WU Yanqin, et al. Sequencing and structural characteristics analysis of mitochondrial genome in Rhodeus ocellatus from Ou Riverv[J]. Journal of Agricultural Biotechnology, 2021, 29(6): 1182-1197. |
| [33] |
孙利元, 杨天燕, 孟玮, 等. 8种石首鱼类线粒体基因组特征及分子系统进化分析[J]. 海洋科学, 2017, 41(3): 48-54. SUN Liyuan, YANG Tianyan, MENG Wei, et al. Analysis of the mitochondrial genome characteristics and phylogenetic relationships of eight sciaenid fishes[J]. Marine Sciences, 2017, 41(3): 48-54. |
| [34] |
PALANDAČIČ A, NASEKA A, RAMLER D, et al. Contrasting morphology with molecular data: an approach to revision of species complexes based on the example of European Phoxinus (Cyprinidae)[J]. BMC Evolutionary Biology, 2017, 17(1): 1-17. |
| [35] |
HONTELA A, STACEY N. Cyprinidae[M]. Boca Raton: CRC Press, 2019: 53-78.
|
 2023, Vol. 47
2023, Vol. 47


