
文章信息
- 赵峰, 王子涵, 曹玲, 曾聪. 2023.
- ZHAO Feng, WANG Zi-han, CAO Ling, ZENG Cong. 2023.
- 黄东海间遗传连通性多物种案例分析与启示
- Analysis and enlightenment of a multispecies case study on the genetic connectivity between the Yellow and the East Seas
- 海洋科学, 47(11): 97-109
- Marine Sciences, 47(11): 97-109.
- http://dx.doi.org/10.11759/hykx20220110002
-
文章历史
- 收稿日期:2022-06-07
- 修回日期:2022-12-06
2. 厦门大学近海海洋环境科学国家重点实验室, 福建 厦门 361012;
3. 厦门大学 海洋与地球学院, 福建 厦门 361012
2. State Key Laboratory of Marine Environment Science, Xiamen 361012, China;
3. College of Ocean and Earth Sciences, Xiamen University, Xiamen 361012, China
海洋生物多样性是人类生存与可持续发展的重要物质基础与实现条件之一,但在全球气候变化及人类活动的双重胁迫下,海洋生物多样性遭受了严重威胁,其衰退与保护已成为最令人关注的全球性问题之一[1]。海洋保护区是海洋生物多样性保护的核心举措[2-5],可通过直接减少人类干扰以有效保护海洋地区的生物资源和重要栖息地[6-7]。通常,海洋保护区提供的效益随规模的增加而增加,但建立大型海洋保护区具有难度,单个保护区也很难有效维持某些海洋生物(例如海洋哺乳类)的生境需求[8],因此,由多个中小型海洋保护区组成的海洋保护区网络似乎在不损害生态效益的情况下更具成本效益[2, 9],也能提高海洋保护区的保护成效[10]。这需要多个海洋保护区之间在种群、遗传或生态系统上具有连通性[8]。联合国制定的2011~2020年生物多样性目标11中(爱知目标11)也将连通性作为海洋保护区网络设计的关键原则[11]。
黄东海是我国东部沿海地区两大边缘海,拥有丰富的海洋生物多样性资源[12]。在全球海洋生态区划分中,黄海属于西北太平洋冷温带生态区(Cold Temperate Northern Pacific Marine Ecoregion)中的黄海生态区(Yellow Sea Ecoregion),东海属于西北太平洋暖温带生态区(Warm Temperate Northern Pacific Marine Ecoregion)中的东中国海生态区(East China Sea Ecoregion)[13-14]。作为太平洋西部最大的边缘海,黄海拥有世界上面积最大的连片泥沙滩涂及湿地世界自然遗产,是由世界自然基金会认定的能代表全球生物多样性的最具优先保护价值的43个海洋生态区之一。与黄海毗邻的东海有记录的热带和亚热带物种多达12 933种,其中近一半(48%)是地方性物种[15]。在《中国国家生物多样性保护战略与行动计划(2011—2030)》中,东海是生物多样性保护的优先区域之一。然而,近年来黄、东海区域在快速发展的同时,也面临生物多样性衰退等问题[16],亟需加强保护。至2022年,东海和黄海已建海洋自然保护区48个和海洋特别保护区74个[17],这些保护区主要分散于沿岸,除江苏盐城湿地珍禽国家级自然保护区之外,其余大部分保护区面积均较小[18]。
洋流是促进保护区间连通的重要因素,在黄、东海区域有黑潮、台湾暖流、对马暖流、东海沿岸流、黄海沿岸流等洋流[19],还有长江冲淡水等水团,复杂的水动力系统可能会直接影响黄东海的保护区间连通性。这一结果也在前人研究中得到验证,例如,花笠螺[20]、蛤蜊[21]、黄鲫[22]的黄、东海群体间有较强连通性,但在栉江珧[23]、青蛤[24]、脉红螺[25]的黄、东海群体间连通性却十分有限。物种间的差异对于揭示区域间的连通性带来一定挑战,因此,需系统梳理黄、东海生物群体间是否存在连通性,为构建区域内海洋保护区网络提供科学支撑。
目前,直接评估连通性具有难度[9, 26],研究人员采用了多种方法量化空间尺度上的连通性,如遗传推断法[27]、物理标记法[28]、耳石微化学分析法及生物物理模型[29]等。其中,遗传推断法因准确性高且易于操作等特点成为目前的主流研究手段[30-31]。它可通过评估种群之间的遗传分化程度以间接推断连通性[32]。通常种群之间无显著的遗传分化意味基因流动频繁,种群之间有较高的连通性。反之,则有较低的遗传连通性[33-35]。目前,黄东海遗传连通性的研究多以单标记或单物种为主,缺乏利用多分子标记对多物种的遗传连通性和成因进行系统性的比较和分析[35]。
基于此,本文通过多物种的案例研究,探讨了黄东海之间物种遗传连通性现状及形成原因,为构建涵盖黄东海的海洋保护区网络提供理论基础,这一研究也可以验证跨生态区建立保护区网络是否可行,最终为构建涵盖沿海的海洋保护区网络提供借鉴与参考。
1 材料与方法 1.1 文献搜集与整理利用Web of Science、Google Scholar与CNKI等途径,搜索国内外已发表的期刊论文。其中,英文以(genetic OR gene OR barrier) AND ((“East Sea” AND “Yellow Sea”) OR “China Sea” OR “coastal waters of China”)为检索关键词,中文以“东海”“黄海”“遗传”为检索关键词,检索截至日期为2022年3月25日。为了保证数据的准确性和有效性,对检索到的文献进行筛选,仅保留研究物种在黄东海皆有采样点的文献,并对重复的数据予以剔除。同时按一个物种使用一种遗传标记进行研究视为一个案例进行分析。
1.2 黄东海遗传连通性现状分析若案例中研究物种的东海群体和黄海群体间存在至少一个显著的遗传分化系数值FST(Fixation index)或GST(Coefficient of gene differentiation)或φST(φ-statistics),则该案例被视为存在显著遗传分化,该研究物种的黄东海群体间具有较低的遗传连通性。反之,则该研究物种的黄东海群体间存在较高的遗传连通性。所有的研究物种分类信息查询WoRMS(World Register of Marine Species)数据库获得,统计各门存在或不存在显著遗传分化的案例数,在SPSS软件中进行卡方检验以分析物种分类与是否存在显著遗传分化的相关性。
1.3 影响遗传连通性的环境因素验证基于各案例中作者的结论确定该案例中影响黄东海群体间是否存在显著遗传分化的因素。统计提及影响因素的案例数,并计算各因素所对应的案例数占这一案例数的比值。此外,结合公海数据及搜集的研究结果进行定量分析以确定环境因素的影响效果。具体如下:
1.3.1 空间距离为了验证空间与遗传连通性的关系,筛选可获得采样点地理信息的研究进行定量分析。对于提供采样点地理坐标但未提供地理距离的案例,使用Microsoft Excel中的球面余弦定律计算大圆距离[36]。对于仅提供地图的研究,使用谷歌地图得出采样点之间的大圆距离。为了避免核基因和线粒体基因遗传分配的差异,将来自线粒体标记的遗传分化系数值(FST或GST或φST)进行转换[37]:
然后,筛选采样点大于3个的案例,在GENEPOP 4.2中计算案例的IBD(Isolation By Distance)斜率(http://genepop.curtin.edu.au/)及Mantel检验p值[36]。IBD斜率为正且Mantel检验显著,则该案例符合IBD模式。最后,仅保留黄东海群体间的空间距离与遗传分化系数值,并将空间距离取对数,在SPSS软件中进行相关性检验以分析对数空间距离与遗传分化的相关性,用R作回归散点图。
1.3.2 浮游幼体期按照浮游幼体期为15 d内,16~30 d,超过30 d将案例分为三类[38],分别统计每类中黄东海群体间存在或不存在显著遗传分化的案例数,运用SPSS软件进行卡方检验以分析浮游幼体期与是否存在显著遗传分化的相关性,以推断物种浮游幼体期与遗传连通性的关系。
1.4 采样和分析差异对遗传连通性的影响 1.4.1 样本数量统计存在或不存在显著遗传分化的各案例中总样本量及平均每个群体样本量,得到四组数据。对每组数据进行精确Kolmogorov-Smirmov检验以分析是否符合正态分布,若不符合,则用SPSS软件进行两独立样本的非参数检验Mann-Whitney U以分析总样本量、平均每个群体样本量与是否存在显著遗传分化的关系,进而推断样本数量是否会影响遗传连通性的检测。用R作散点箱线图。
1.4.2 遗传标记将所有案例按遗传标记为线粒体DNA序列(Mitochondrial DNA Sequence,MT)、扩增片段长度多态性(Amplified Fragment Length Polymorphism,AFLP)、微卫星(Simple Sequence Repeats, SSR)、单核苷酸多态性(Single Nucleotide Polymorphism,SNP)、简单重复序列间扩增(Inter Simple Sequence Repeat,ISSR)、核DNA序列(Nuclear DNA Sequence,NT)、同工酶(Enzyme)分为7类,统计每类中黄东海群体间存在或不存在显著遗传分化的案例数,在SPSS软件中采用fisher精确检验分析遗传标记与是否存在显著遗传分化的相关性,以推断遗传标记是否会影响遗传连通性的检测。
2 结果与分析 2.1 黄东海群体间有限的遗传连通性共得到137篇文献,包括英文文献93篇,中文文献44篇。有27篇文献包含多于一个案例(19.71%)。发文时间跨度为2003—2022年。发文趋势分析表明该领域的研究大致划分为2个阶段,2003—2012年为快速起步期,2013—2022年为波动发展期(图 1)。
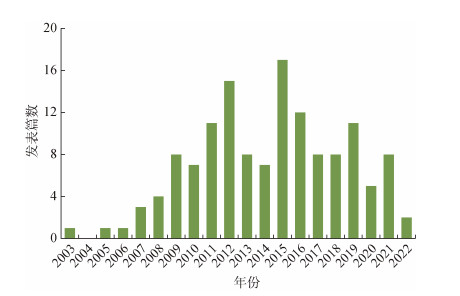 |
| 图 1 论文发表趋势分析 Fig. 1 Analysis of publication trends |
从研究物种来看,175个案例涉及脊索动物门(37个物种)、软体动物门(16个物种)、节肢动物门(13个物种)、褐藻门(3个物种)、绿藻门(1个物种)等5门共计70个物种。其中,脊索动物门的案例数最多,有101个案例(57.71%)。褐藻门的案例数最少,仅1个案例(0.57%)。50.00%的物种有超过一个的案例,其中小黄鱼的案例数最多,有22个(12.57%),口虾蛄次之,有10个(5.71%)(图 2)。
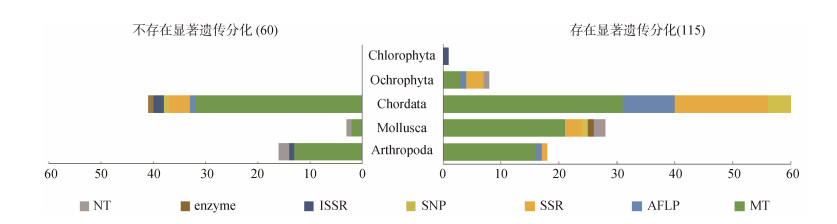 |
| 图 2 不同分类和分子标记间的遗传分化差异 Fig. 2 Differences in genetic differentiation among different taxa and molecular markers 注: MT为线粒体DNA序列,AFLP为扩增片段长度多态性,SSR为微卫星,SNP为单核苷酸多态性,ISSR为简单重复序列间扩增,NT为核DNA序列,Enzyme为同工酶。下图同上。 |
在搜集案例中,有115个案例研究物种的黄东海群体间存在显著的遗传分化,占比为65.71%,意味着黄东海群体间有限的遗传连通性。60个案例中研究物种的黄东海群体间不存在显著的遗传分化,占比为34.29%(图 2)。因褐藻门和绿藻门的案例数过少,故剔除这两个门,在剩余三门中,采用卡方检验发现是否存在显著遗传分化与物种分类有关(χ2= 12.030,P=0.002)。此外,软体动物门中存在显著遗传分化的案例数占比最高(90.32%),脊索动物门次之(59.41%),节肢动物门最低(52.94%)。
2.3 黄东海群体间遗传连通性的影响因素提及影响因素的130个案例中,81个案例提及的因素超过1个,占比为62.31%。所有因素归纳为非生物因素与生物因素两类共六种(表 1)。其中,长江冲淡水、生境异质性、空间距离和进化过程均会导致黄东海群体间的遗传分化,物种生活史及洋流既能促进又能限制基因流(图 2)。
| 影响因素 | 存在显著遗传分化(案例数,比例) | 不存在显著遗传分化(案例数,比例) | |
| 非生物因素 | 洋流 | 是(25.00%) | 是(68.00%) |
| 长江冲淡水 | 是(30.00%) | 否 | |
| 生境异质性 | 是(8.75%) | 否 | |
| 空间距离 | 是(30.00%) | 否 | |
| 生物因素 | 物种生活史 | 是(53.75%) | 是(94.00%) |
| 进化过程 | 是(38.75%) | 否 |
在115个存在显著遗传分化的案例中,有80个案例分析了潜在影响因素(表 1): 沿岸带大量小而持久的涡流带和对流带、海域的温盐锋及强烈的上升流等可能通过影响幼体扩散阻碍黄东海种群间的基因交流(25.00%);长江冲淡水汇入海域的低盐度水流对某些物种基因交流发挥屏障作用(30.00%);黄东海群体间较远的空间距离限制了群体间的基因交流(30.00%);生境异质性如海湾或东海中宁德等岛屿的存在会成为黄东海群体间基因流动的障碍(8.75%);在生物的进化过程中,历史冰期冰盖消长的反复波动导致黄东海群体间的长期隔离,促进了基因突变的积累和谱系的分化,造成了跨海域群体间显著的遗传分化(38.75%);物种的生活史如较弱的游泳能力或较短的浮游幼体期限制了跨海域群体间的基因交流(53.75%)。在65个不存在显著遗传分化的案例中,有50个案例提及了因素,可归纳为如下两点: 物种生活史如卵、幼体或成体阶段具有较强的扩散能力导致黄东海群体间产生频繁的基因交流(94.00%);黄东海的洋流促进了跨海域群体的扩散等(68.00%)。
2.4 影响因素验证 2.4.1 空间距离在可以计算出IBD斜率的117个案例中,76个案例IBD斜率为正,其中,25个案例Mantel检验值显著,表明空间距离确实是导致这些案例中黄东海群体间存在显著遗传分化的因素。对所有可计算出空间距离的案例的距离与遗传分化进行检验发现,两者之间存在较低但极显著的相关关系(r=0.190, n=1 638, P < 0.01),说明海域间群体相隔距离会影响群体间的基因交流(图 3)。
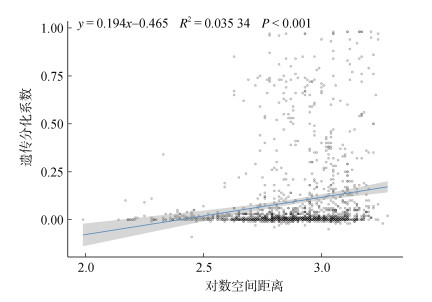 |
| 图 3 黄东海群体间对数空间距离和遗传分化的相关性 Fig. 3 Correlation between the logarithmic spatial distance and genetic differentiation of Yellow Sea and East Sea populations |
搜集资料后,可得到144个案例共48个物种的浮游幼体期。其中,浮游幼体期为15d内的案例群中存在显著遗传分化的案例数占比最高(71.05%)。浮游幼体期大于30d的占比最低(45.95%)。总体上,随着浮游幼体期增加,存在显著遗传分化的案例数比值呈下降趋势(表 2)。卡方检验结果显示浮游幼体期与是否存在显著遗传分化存在相关性(χ2=6.972,P=0.031),意味着黄东海群体间遗传连通性与浮游幼体期相关。
| 浮游幼体期/d | 存在显著遗传分化的案例数 | 不存在显著遗传分化的案例数 | 存在显著遗传分化的案例数占比/% |
| ≤15 | 27 | 11 | 71.05 |
| 16~30 | 48 | 21 | 69.57 |
| ≥31 | 17 | 20 | 45.95 |
Mann-Whitney U检验结果显示总样本量存在显著差异(Z= –2.509, P=0.012),平均每个群体样本量不存在显著差异(Z=–1.900,P=0.057),说明总样本量会影响遗传分化显著性的检测结果(图 4),平均每个群体样本量不会,推断总样本量会对遗传连通性结果产生影响。
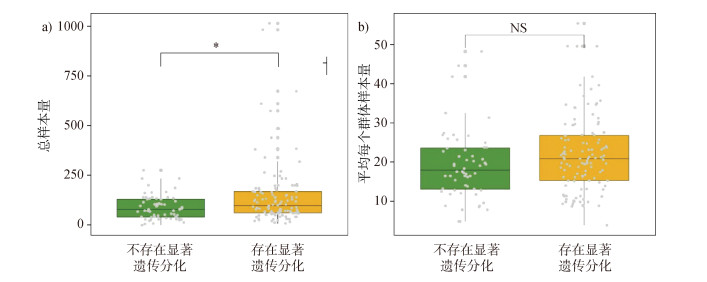 |
| 图 4 总样本量和平均每个群体样本量对黄东海群体间显著遗传分化存在与否的影响 Fig. 4 Effects of total sample size and average sample size per population on the nonsignificant or significant genetic differentiation between Yellow Sea and East Sea populations |
175个案例中,以MT、SSR、AFLP、NT、SNP、ISSR、Enzyme作为遗传标记的案例数如表 3所示,基于线粒体基因的研究仍然是主流(图 5)。fisher精确检验结果显示,遗传标记与是否存在显著遗传分化具有相关性(P < 0.05),这意味着黄东海群体间连通性可能受到遗传标记选择的影响(表 3)。
| 遗传标记 | 存在显著遗传分化的案例数 | 不存在显著遗传分化的案例数 | 存在显著遗传分化的案例数占比/% |
| MT | 71 | 46 | 60.68 |
| AFLP | 11 | 1 | 91.67 |
| SSR | 23 | 4 | 85.19 |
| SNP | 5 | 1 | 83.33 |
| ISSR | 1 | 3 | 25.00 |
| Enzyme | 1 | 1 | 50.00 |
| NT | 3 | 4 | 42.86 |
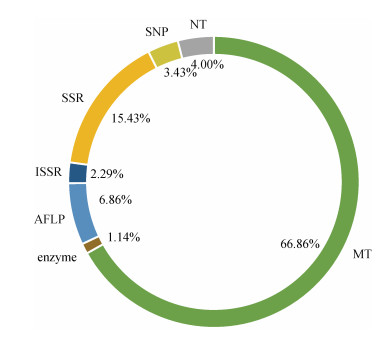 |
| 图 5 各遗传标记所对应的案例数 Fig. 5 Percentage of cases corresponding to each genetic marker |
海洋种群间的连通性对于设计大范围的海洋保护区网络和制定有效的管理制度至关重要,故有必要加强对种群间连通性模式的了解。目前,连通性分析经常集中在单一物种的样本中,再外推至群落层面,但样本的效用有限,即使在相似生态、生活史和地理范围的物种中,连通性模式也可能非常不同[39],不同生物学特性的物种也可能具有高度相似的连通性模式[40]。此外,遗传标记也会对遗传连通性高低的判定产生影响,突变速率较快的遗传标记可能会检测出较低的遗传连通性,更保守的遗传标记可能会检测出较高的遗传连通性。目前也存在对同一物种采用不同的遗传标记而得到相互矛盾结果的研究[41-45]。总样本量也会干扰物种遗传连通性高低的判定,可能与案例覆盖的地理群体较少有关。
因此单一或较少物种的连通性可能无法代表海域的总体连通性,对较多物种的连通性研究进行梳理和比较,以归纳总体连通性现状,显然更为准确。这也被现有研究采纳。例如,Gardner等人梳理了58项研究,确定了新西兰沿海的区域连通性模式[46];Crandall等人分析了来自4门27科56个物种的数据,支持了印度-太平洋海域的三个基因交流障碍的存在[47]。本研究中有65.71%的案例其物种的黄东海群体间存在显著的遗传分化,意味着黄海和东海生态区间可能存在十分有限的基因交流,即两个生态区间的种群存在潜在的连通障碍。
3.2 黄东海群体间遗传连通性的影响因素种群间的连通性通常受到自身繁殖扩散能力以及所处环境的影响,在本研究梳理的结果中也证实了这一点。在非生物因素中,长江冲淡水被认为可能是中国近海将北太平洋温水生物区与印度-西太平洋暖水生物区分开的生物地理边界[48-50],对沿海物种基因流动的阻碍作用也在鱼类[22, 51]、软体动物[20, 21, 52-53]、甲壳类[49, 54]和大型藻类[55]等多个物种中进行了研究。在其他海域,也有学者研究了主要河流冲淡水对基因流动的阻碍作用,如Tosetto等通过分析海洋生物地理信息系统(OBIS)中的物种分布数据证实了亚马逊河是热带西大西洋动物扩散的软屏障[56]。本研究发现长江冲淡水对基因交流的阻碍作用具有明显的物种偏好性,与以往研究一致[57],这一现象推测可能与物种耐盐性[57-58]、栖息地类型[53]、浮游幼体期[59]等因素有关。但长江冲淡水并不是稳定且持久的屏障,在末次盛冰期之后才起到屏障作用[57]。近年来,在气候变暖和人为因素的影响下,这一作用对某些物种而言已经减弱或丧失[60-61]。
在生物因素中,物种生活史会影响黄东海种群间的遗传连通性,不同物种的生活史不尽相同,这可能是遗传连通性与物种分类之间存在相关性的原因[62]。在不同的进化谱系中,生物体通常表现出特定于物种的生活史,如繁殖策略及浮游幼体期等,影响物种对环境因素的反应[63],进而在海洋中可能表现出相异的遗传连通性模式。同时,软体动物门具有最高比例的显著遗传分化也意味着更容易出现低遗传连通性,一项对东海海洋生物系统地理学的研究中所涉及的89%的软体动物都具有显著的遗传结构[64],这可能是因为软体动物通常不连续的栖息地分布、普遍存在的“sweepstakes”式的繁殖模式以及固着生活史阶段导致种内出现显著的遗传分化[35],也更容易受到生物地理和历史屏障的影响[65-66]。本研究还证实黄东海群体间遗传连通性与浮游幼体期相关。随着浮游幼体期的增加,具有较高遗传连通性的案例数比值呈上升趋势,这与已有研究一致[38]。浮游幼体期是物种生活史的重要部分,更长的浮游幼体期会导致物种扩散距离增加[67],促进种群间的基因流动,因此预测种群间的遗传分化应当与浮游幼体期密切相关[68]。但有多项研究得出了相反的结论[65, 69]。针对这一分歧,有研究表明遗传分化与浮游幼体期相关性的缺失并不意味着迁移率与浮游幼体期没有相关性,物种有效种群规模间的差异、错误的FST值、非平衡状态下的FST值都可能是导致浮游幼体期和遗传分化间无相关性的原因[70]。总之,浮游幼体期与遗传分化间的相关性仍存在争议,未来需更强有力的证据证实二者间的关系。
本研究也证实空间距离与遗传分化相关,发现有25个案例符合距离隔离(IBD)模式。遗传分化和空间距离间较小的相关系数也可能表明其他因素会对黄东海种群间遗传连通性造成更大影响。这也可能是因为海洋环境中IBD模式相较于陆地不太明显。能够更好地解释海洋种群遗传结构的因素包括: 自然选择、之前分离种群的再次基因渗入形成的地理渐变群以及种群间由时间遗传变异驱动的混乱遗传斑块等[71]。此外,较小的相关系数还可能源于海洋种群栖息地间的空间距离无法被欧式距离替代[72],而是与根据洋流推导出的“海洋距离”有关[73]。因此,在考虑洋流不对称的情况下,采用高分辨率海洋模型和拉格朗日粒子模拟来推导种群地理位置之间的海洋距离,以此代替欧式距离,可能会获得更高的相关系数。
在多种生物的进化过程中,冰期循环产生的栖息地收缩-扩张的变化会促进海域间谱系的分化,造成跨海域群体间较低的遗传连通性,这也与以往研究一致[24, 74-75]。在提及进化过程这一影响因素的21个物种中,52.38%的物种为潮间带物种,暗示潮间带生物可能是受海平面升降变化影响最大的一类生物[35]。海面下降时,由于冰川对海床持续的冲刷作用,它们几乎丧失了全部的栖息环境。海盆间的分隔阻碍了群体间的基因交流,退缩到不同避难所的群体间在进化过程中易形成显著的遗传分化。即使障碍消失,它们也能保持这种历史格局很长时间[76]。
除了本研究所提及的6个因素外,深度导致的强烈环境梯度也会对物种产生不同的选择机制,进而影响连通性[77, 78];气候变化导致的全球变暖[79-81]、海洋酸化[82-83]、极端气候事件[84-85]等会通过影响洋流及生物幼体存活率、扩散能力等影响连通性[79]。未来应全面考虑多个影响因素的叠加效应对黄东海种群间连通性的改变。
3.3 对构建黄东海海洋保护区网络的启示与建议为了构建有弹性的海洋保护区网络,连通性是关键的设计原则,这需要保护区之间具有较好的连通性 [86-87]。同时,在以生态系统为基础的海洋保护区管理制度中,也需依据连通性情况明晰相应生态系统的边界[88]。这些也被现有研究采纳,如一项在北美太平洋沿岸开展的研究中发现,在门多西诺角(Cape Mendocino)附近受测的6个物种中有5个出现了遗传分化,表明门多西诺角的存在可能会影响该海域保护区之间的连通性,大大降低海洋保护区网络的稳定性[65];Toonen等在夏威夷群岛27个分类和生态多样化物种的178个成对遗传分化显著性检验中发现了73个显著遗传分化,结合存在显著遗传分化的种群分布,揭示了该海域内的4个物种扩散障碍,从而建议夏威夷群岛应划分为至少五个海洋空间管理单元[88]。因此,黄、东海群体间有限的遗传连通性不利于构建涵盖黄东海的海洋保护区网络,应在黄海和东海生态区内各建立独立的海洋保护区网络,特别是针对软体动物的保护。这一结果也表明跨生态区建设海洋保护区网络可能不具有操作性,类推至中国其他海域如南海包含三个生态区[13],或许并不能构建涵盖整个南海的海洋保护区网络。
此外,本研究证实连通性与物种类群有关,这可能是因为不同的物种类群具有相异的生活史。生活史可直接或间接影响海洋保护区网络的有效性[89]。因此,在分别构建黄海和东海的海洋保护区网络时,应综合考虑不同的物种类群并整合多物种的生活史,尤其是与扩散、栖息地相关的特征[90],如浮游幼体期、成体的游泳能力等。这些与扩散相关的指标会直接影响连通性,可用来指导保护区的大小设计。如Botsford等建议保护区的大小至少为所保护物种平均扩散距离的两倍[91]。具体而言,石斑鱼、刺尾鱼等物种可以在大小为1 km的小型保护区内得到保护,隆头鹦嘴鱼等物种的保护需5~10 km的中型保护区或10~20 km的大型海洋保护区,鲷鱼等物种需10~100 km的更大海洋保护区[92]。对于金枪鱼等具有更长扩散距离的物种,无法建立足够大的保护区进行保护,需保护区网络覆盖这类物种生活史阶段的关键栖息地类型,如繁殖区、索饵场和越冬场等,以及连接各栖息地的生态廊道[93],结合管理手段进行保护,如在这些区域实施限制捕捞等政策。
海洋保护区网络设计还需考虑黄东海的水动力特征。本研究中提及影响因素的50个不存在显著遗传分化的案例中,68.00%的案例将洋流作为解释因素之一,这表明洋流可以促进遥远地理群体间的基因交流,因此,可利用洋流连接多个保护区。同时,还应明确海域内洋流的方向及强弱,当海域存在较强的单向洋流,且保护物种具有单一季节产卵的特征时,会表现出高度受洋流方向限制的扩散,此时应在洋流上游产卵点设置更多的保护区,加强对产卵点下游的幼体生长栖息地的保护[92]。黄海存在一支较强且流向终年向南的洋流,即黄海沿岸流[94],而在东海,台湾暖流终年偏北,几乎控制了东海陆架大部分区域的水文情况[94]。因此,在分别构建黄海和东海海洋保护区网络时也应将此点纳入考虑。此外,提及原因的80个存在显著遗传分化的案例中有25%的案例将洋流作为影响因素之一,这可能是环流影响了幼体的扩散。在夏季,黄海会出现一个大范围的气旋式环流,内部还存在气旋、反气旋等多个较小的环流,阻碍幼体扩散,导致幼体滞留。对于滞留程度较高的地方应设置海洋保护区予以保护。30%的案例将长江冲淡水视为阻碍某些物种基因交流的物理屏障,在网络设计时也应避开这样的区域,在屏障的两侧单独构建网络。
构建保护区网络还需考虑保护区间距。本研究也证实了空间距离与遗传连通性呈显著负相关。因此,间距不宜过大,应满足保护区间的幼体交流。现阶段,对于间距的设置有≤15km[95],15~20 km[92], ≤30 km[96], 70~100 km[97]等多种观点,但不同的推荐间距与保护物种的扩散能力有关。根据前人研究建议,保护区间距应在遗传分化系数值不超过0.2的距离以内[98]。结合本研究的IBD研究结果可知,黄东海内的保护区间距在小于320 km(即log(距离) < 2.5)时,大部分物种的遗传分化系数小于0.2(图 3)。目前,黄东海共有海洋保护区122个[17],通过Arcgis计算得到邻近保护区的间距都在200 km之内,理论上现有保护区具有形成网络的可能。但实际上,由于黄东海间存在洋流的影响以及不同生物间存在差异,这一数值仅可作为参考,在保护区网络设计时首要考虑受保护物种的扩散距离应大于保护区间距,以满足连通性需求。
最后,在管理上,国家应出台相关的政策法规以指导黄海、东海海洋保护区网络的分别构建,并在国家层面建立协调机制,统筹多部门整合不同形式的空间管理结构,指导跨省、跨部门的多层次合作,确保信息共享。此外,对构建的海洋保护区网络进行监测和评估并进行适应性管理能确保网络的可持续发展[2]。总之,黄海和东海海洋保护区网络的分别构建并不能一蹴而就,需随着时间的推移在良好的规划下不断进行。未来,这一方法也可能会为构建其他海域甚至是涵盖中国沿海的海洋保护区网络体系提供借鉴。
| [1] |
刘金立, 陈新军. 海洋生物多样性研究进展及其热点分析[J]. 渔业科学进展, 2021, 42(1): 201-213. LIU Jinli, CHEN Xinjun. Research progress and hotspots of marine biodiversity: Based on bibliometrics and knowledge mapping analysis[J]. Progress in Fishery Sciences, 2021, 42(1): 201-213. |
| [2] |
LI Y Z, FLUHARTY D L. Marine protected area networks in China: Challenges and prospects[J]. Marine Policy, 2017, 85: 8-16. DOI:10.1016/j.marpol.2017.08.001 |
| [3] |
HALPERN B S, DIAMOND J, GAINES S, et al. Near-term priorities for the science, policy and practice of Coastal and Marine Spatial Planning (CMSP)[J]. Marine Policy, 2012, 36(1): 198-205. DOI:10.1016/j.marpol.2011.05.004 |
| [4] |
银森录, 郑苗壮, 徐靖, 等. 《生物多样性公约》海洋生物多样性议题的谈判焦点、影响及我国对策[J]. 生物多样性, 2016, 24(7): 855-860. YIN Senlu, ZHENG Miaozhuang, XU Jing, et al. Negotiation focus, impacts, and China's implementation strategies of Marine and Coastal Biodiversity in the Convention on Biological Diversity[J]. Biodiversity Science, 2016, 24(7): 855-860. |
| [5] |
林金兰, 陈彬, 黄浩, 等. 海洋生物多样性保护优先区域的确定[J]. 生物多样性, 2013, 21(1): 38-46. LIN Jinlan, CHENG Bin, HUANG Hao, et al. Identification of the priority areas for marine biodiversity conservation[J]. Biodiversity Science, 2013, 21(1): 38-46. |
| [6] |
ASAAD I, LUNDQUIST C J, ERDMANN M V, et al. Delineating priority areas for marine biodiversity conservation in the Coral Triangle[J]. Biological Conservation, 2018, 222: 198-211. DOI:10.1016/j.biocon.2018.03.037 |
| [7] |
WILSON K L, TITTENSOR D P, WORM B, et al. Incorporating climate change adaptation into marine protected area planning[J]. Global Change Biology, 2020, 26(6): 3251-3267. DOI:10.1111/gcb.15094 |
| [8] |
SMITH J, METAXAS A. A decision tree that can address connectivity in the design of Marine Protected Area Networks (MPAn)[J]. Marine Policy, 2018, 88: 269-278. DOI:10.1016/j.marpol.2017.11.034 |
| [9] |
KOOL J T, NICHOL S L. Four-dimensional connectivity modelling with application to Australia's north and northwest marine environments[J]. Environmental Modelling & Software, 2015, 65: 67-78. |
| [10] |
GOETZE J S, WILSON S, RADFORD B, et al. Increased connectivity and depth improve the effectiveness of marine reserves[J]. Global Change Biology, 2021, 27(15): 3432-3447. DOI:10.1111/gcb.15635 |
| [11] |
JUINIO-MEñEZ M A. Biophysical and genetic connectivity considerations in marine biodiversity conservation and management in the South China Sea[J]. Journal of International Wildlife Law & Policy, 2015, 18(2): 110-119. |
| [12] |
HAO H, BIN C, JINLAN L. The marine spatial classification and the identification of priority conservation areas (PCAs) for marine biodiversity conservation–A case study of the offshore China[J]. Ocean & Coastal Management, 2015, 116: 224-236. |
| [13] |
SPALDING M D, FOX H E, HALPERN B S, et al. Marine ecoregions of the world: A bioregionalization of coastal and shelf areas[J]. Bioscience, 2007, 57(7): 573-583. |
| [14] |
TEH L S, CASHION T, CHEUNG W W, et al. Taking stock: a Large Marine Ecosystem perspective of socioeconomic and ecological trends in East China Sea fisheries[J]. Reviews in Fish Biology and Fisheries, 2020, 30(2): 269-292. DOI:10.1007/s11160-020-09599-8 |
| [15] |
DING H, XU H, WU J, et al. An overview of spatial management and marine protected areas in the East China Sea[J]. Coastal Management, 2008, 36(5): 443-457. DOI:10.1080/08920750802445916 |
| [16] |
张林姣. 东海陆源污染治理机制研究[D]. 宁波: 宁波大学, 2017. ZHANG Linjiao. Research on the governance mechanism of land-based pollution in the East China Sea[D]. Ningbo: Ningbo University, 2017. |
| [17] |
ZENG X, CHEN M Y, ZENG C, et al. Assessing the management effectiveness of China's marine protected areas: Challenges and recommendations[J]. Ocean & Coastal Management, 2022, 224. |
| [18] |
曲方圆, 李淑芸, 赵林林, 等. 黄海生态区保护空缺分析[J]. 生物多样性, 2021, 29(3): 385-393. QU Fangyuan, LI Shuyun, ZHAO Linlin, et al. Conservation gap analysis for the Yellow Sea ecoregion[J]. Biodiversity Science, 2021, 29(3): 385-393. |
| [19] |
MA C, ZOU J. Marine Biodiversity and its conservation in china seas[J]. Transactions of Oceanology and Limnology, 2003, 2: 41-47. DOI:10.3969/j.issn.1003-6482.2003.02.007 |
| [20] |
DONG Y W, WANG H S, HAN G D, et al. The Impact of Yangtze River Discharge, Ocean Currents and Historical Events on the Biogeographic Pattern of Cellana toreuma along the China Coast[J]. Plos One, 2012, 7(4): e36178. DOI:10.1371/journal.pone.0036178 |
| [21] |
NI G, LI Q, NI L H, et al. Population subdivision of the surf clam Mactra chinensis in the East China Sea: Changjiang River outflow is not the sole driver[J]. Peerj, 2015, 3: e1240. DOI:10.7717/peerj.1240 |
| [22] |
XU S, SONG N, LU Z, et al. Genetic variation in scaly hair-fin anchovy Setipinna tenuifilis (Engraulididae) based on the mitochondrial DNA control region[J]. Mitochondrial DNA, 2014, 25(3): 223-230. DOI:10.3109/19401736.2013.845754 |
| [23] |
LIU J, LI Q, KONG L, et al. Cryptic diversity in the pen shell Atrina pectinata (Bivalvia: Pinnidae): high divergence and hybridization revealed by molecular and morphological data[J]. Molecular Ecology, 2011, 20(20): 4332-4345. DOI:10.1111/j.1365-294X.2011.05275.x |
| [24] |
NI G, LI Q, KONG L F, et al. Phylogeography of Bivalve Cyclina sinensis: Testing the Historical Glaciations and Changjiang River Outflow Hypotheses in Northwestern Pacific[J]. Plos One, 2012, 7(11): e49487. DOI:10.1371/journal.pone.0049487 |
| [25] |
YANG J M, LI Q, ZHENG X CD, et al. Genetic diversity in populations of Rapana venosa in coastal waters of China[J]. Oceanologia et limnologia sinica, 2008, 39(3): 257-262. DOI:10.3321/j.issn:0029-814X.2008.03.011 |
| [26] |
FEUTRY P, BERRY O, KYNE P M, et al. Inferring contemporary and historical genetic connectivity from juveniles[J]. Molecular ecology, 2017, 26(2): 444-456. DOI:10.1111/mec.13929 |
| [27] |
SATO M, HONDA K, UY W H, et al. Marine protected area restricts demographic connectivity: Dissimilarity in a marine environment can function as a biological barrier[J]. Ecology and evolution, 2017, 7(19): 7859-7871. DOI:10.1002/ece3.3318 |
| [28] |
PITTMAN S J, MONACO M E, FRIEDLANDER A M, et al. Fish with chips: tracking reef fish movements to evaluate size and connectivity of Caribbean marine protected areas[J]. PLoS One, 2014, 9(5): e96028. DOI:10.1371/journal.pone.0096028 |
| [29] |
PUJOLAR J M, SCHIAVINA M, DI FRANCO A, et al. Understanding the effectiveness of marine protected areas using genetic connectivity patterns and Lagrangian simulations[J]. Diversity and Distributions, 2013, 19(12): 1531-1542. DOI:10.1111/ddi.12114 |
| [30] |
单楠, 周可新, 潘扬, 等. 生物多样性保护廊道构建方法研究进展[J]. 生态学报, 2019, 39(2): 411-420. SHAN Nan, ZHOU Kexin, PAN Yang, Research advances in design methods of biodiversity conservation corridors[J]. Acta Ecologica Sinica, 2019, 39(2): 411-420. |
| [31] |
JENKINS T L, STEVENS J R. Assessing connectivity between MPAs: Selecting taxa and translating genetic data to inform policy[J]. Marine Policy, 2018, 94: 165-173. DOI:10.1016/j.marpol.2018.04.022 |
| [32] |
EPPS C W, KEYGHOBADI N. Landscape genetics in a changing world: disentangling historical and contemporary influences and inferring change[J]. Molecular ecology, 2015, 24(24): 6021-6040. DOI:10.1111/mec.13454 |
| [33] |
TREML E A, ROBERTS J, HALPIN P N, et al. The emergent geography of biophysical dispersal barriers across the Indo-West Pacific[J]. Diversity and Distributions, 2015, 21(4): 465-476. DOI:10.1111/ddi.12307 |
| [34] |
KENDRICK G A, ORTH R J, STATTON J, et al. Demographic and genetic connectivity: the role and consequences of reproduction, dispersal and recruitment in seagrasses[J]. Biological Reviews, 2017, 92(2): 921-938. DOI:10.1111/brv.12261 |
| [35] |
倪刚. 四种广布性贝类系统地理学研究—探讨中国近海贝类遗传格局形成过程和演化机制[D]. 青岛: 中国海洋大学, 2013. NI Gang. Phylogeography of four marine bivalves along China coastline, with views into the evolutionary processes and mechanisms[D]. Qingdao: Ocean University of China, 2013. |
| [36] |
BACO A R, ETTER R J, RIBEIRO P A, et al. A synthesis of genetic connectivity in deep‐sea fauna and implications for marine reserve design[J]. Molecular Ecology, 2016, 25(14): 3276-3298. DOI:10.1111/mec.13689 |
| [37] |
KINLAN B P, GAINES S D. Propagule dispersal in marine and terrestrial environments: A community perspective[J]. Ecology, 2003, 84(8): 2007-2020. DOI:10.1890/01-0622 |
| [38] |
PASCUAL M, RIVES B, SCHUNTER C, et al. Impact of life history traits on gene flow: a multispecies systematic review across oceanographic barriers in the Mediterranean Sea[J]. PLoS One, 2017, 12(5): e0176419. DOI:10.1371/journal.pone.0176419 |
| [39] |
BIRD C E, HOLLAND B S, BOWEN B W, et al. Contrasting phylogeography in three endemic Hawaiian limpets (Cellana spp.) with similar life histories[J]. Molecular Ecology, 2007, 16(15): 3173-3186. DOI:10.1111/j.1365-294X.2007.03385.x |
| [40] |
SELKOE K A, WATSON J R, WHITE C, et al. Taking the chaos out of genetic patchiness: seascape genetics reveals ecological and oceanographic drivers of genetic patterns in three temperate reef species[J]. Molecular Ecology, 2010, 19(17): 3708-3726. DOI:10.1111/j.1365-294X.2010.04658.x |
| [41] |
程娇. 西北太平洋两种鲐属鱼类的分子系统地理学研究[D]. 青岛: 中国海洋大学, 2013. CHENG J. Molecular phylogeography of two scomber species in northwestern Pacific[D]. Qingdao: Ocean University of China, 2013. |
| [42] |
张红艳. 中国鱚(Sillago sinica)群体形态学及遗传学研究[D]. 上海: 上海海洋大学, 2013. ZHANG Hongyan. Study on morphology and genetics of Sillago sinica[D]. Shanghai: Shanghai Ocean University, 2013. |
| [43] |
徐雪娜. 中国沿海三疣梭子蟹种群的遗传结构[D]. 杭州: 杭州师范大学, 2006. XU Xuena. Genetic structure of Portunus trituberculatus miers populations in coastal China[D]. Hangzhou: Hangzhou Normal University, 2006. |
| [44] |
董志国. 中国沿海三疣梭子蟹群体形态、生化与分子遗传多样性研究[D]. 上海: 上海海洋大学, 2012. DONG Zhiguo. The study on morphology, biochemistry, molecular phylogeography and genetic diversity of swimming crab Portunus trituberculatus along China coast[D]. Shanghai: Shanghai Ocean University, 2012. |
| [45] |
FANG X, HOU X, LI X, et al. Ecological connectivity between land and sea: a review[J]. Ecological research, 2018, 33(1): 51-61. DOI:10.1007/s11284-017-1549-x |
| [46] |
GARDNER J, BELL J, CONSTABLE H, et al. Multispecies coastal marine connectivity: a literature review with recommendations for further research[J]. New Zealand aquatic environment and biodiversity report, 2010, 58. |
| [47] |
CRANDALL E D, RIGINOS C, BIRD C E, et al. The molecular biogeography of the Indo‐Pacific: Testing hypotheses with multispecies genetic patterns[J]. Global Ecology and Biogeography, 2019, 28(7): 943-960. DOI:10.1111/geb.12905 |
| [48] |
BEARDSLEY R, LIMEBURNER R, YU H, et al. Discharge of the Changjiang (Yangtze river) into the East China sea[J]. Continental Shelf Research, 1985, 4(1/2): 57-76. |
| [49] |
HAN Z, ZHU W, ZHENG W, et al. Significant genetic differentiation between the Yellow Sea and East China Sea populations of cocktail shrimp Trachypenaeus curvirostris revealed by the mitochondrial DNA COI gene[J]. Biochemical Systematics and Ecology, 2015, 59: 78-84. DOI:10.1016/j.bse.2014.12.028 |
| [50] |
LIU J. Status of marine biodiversity of the China Seas[J]. PLoS One, 2013, 8(1): e50719. |
| [51] |
XIAO Y, LI J, REN G, et al. Pronounced population genetic differentiation in the rock bream Oplegnathus fasciatus inferred from mitochondrial DNA sequences[J]. Mitochondrial DNA Part A, 2016, 27(3): 2045-2052. |
| [52] |
WANG J, TSANG L M, DONG Y W. Causations of phylogeographic barrier of some rocky shore species along the Chinese coastline[J]. Bmc Evolutionary Biology, 2015, 15(1): 1-15. |
| [53] |
GUO X, ZHAO D, JUNG D W, et al. Phylogeography of the Rock Shell Thais clavigera (Mollusca): Evidence for Long-Distance Dispersal in the Northwestern Pacific[J]. Plos One, 2015, 10(7): e0129715. |
| [54] |
DU X, CAI S, YU C, et al. Population genetic structure of mantis shrimps Oratosquilla oratoria: testing the barrier effect of the Yangtze River outflow[J]. Biochemical Systematics and Ecology, 2016, 66: 12-8. |
| [55] |
CHEANG C C, CHU K H, ANG JR P O. Phylogeography of the marine macroalga Sargassum hemiphyllum (Phaeophyceae, Heterokontophyta) in northwestern Pacific[J]. Molecular Ecology, 2010, 19(14): 2933-2948. |
| [56] |
GIACHINI TOSETTO E, BERTRAND A, NEUMANN-LEITãO S, et al. The Amazon River plume, a barrier to animal dispersal in the Western Tropical Atlantic[J]. Scientific reports, 2022, 12(1): 1-12. |
| [57] |
NI G, KERN E, DONG Y W, et al. More than meets the eye: the barrier effect of the Yangtze River outflow[Z]. Wiley Online Library, 2017.
|
| [58] |
YU S-S, WANG J, WANG Q-L, et al. DNA barcoding and phylogeographic analysis of Nipponacmea limpets (Gastropoda: Lottiidae) in China[J]. Journal of Molluscan Studies, 2014, 80(4): 420-429. |
| [59] |
CHEANG C C, TSANG L M, NG W C, et al. Phylogeography of the cold‐water barnacle Chthamalus challengeri in the north‐western Pacific: effect of past population expansion and contemporary gene flow[J]. Journal of Biogeography, 2012, 39(10): 1819-1835. |
| [60] |
WANG W, WANG J, CHOI F M P, et al. Global warming and artificial shorelines reshape seashore biogeography[J]. Global Ecology and Biogeography, 2020, 29(2): 220-231. |
| [61] |
HU L S, DONG Y W. Northward shift of a biogeographical barrier on China's coast[J]. Diversity and Distributions, 2022, 28(2): 318-330. |
| [62] |
GALLEGO A, GIBB F M, TULLET D, et al. Biophysical connectivity patterns of benthic marine species used in the designation of Scottish nature conservation marine protected areas[J]. ICES Journal of Marine Science, 2017, 74(6): 1797-1811. |
| [63] |
AYRE D, MINCHINTON T, PERRIN C. Does life history predict past and current connectivity for rocky intertidal invertebrates across a marine biogeographic barrier?[J]. Molecular ecology, 2009, 18(9): 1887-1903. |
| [64] |
NI G, LI Q, KONG L, et al. Comparative phylogeography in marginal seas of the northwestern Pacific[J]. Molecular Ecology, 2014, 23(3): 534-548. |
| [65] |
KELLY R P, PALUMBI S R. Genetic Structure Among 50 Species of the Northeastern Pacific Rocky Intertidal Community[J]. Plos One, 2010, 5(1): e8594. |
| [66] |
SAGARIN R D, GAINES S D. Geographical abundance distributions of coastal invertebrates: using one-dimensional ranges to test biogeographic hypotheses[J]. Journal of Biogeography, 2002, 29(8): 985-997. |
| [67] |
SHANKS A L. Pelagic larval duration and dispersal distance revisited[J]. The biological bulletin, 2009, 216(3): 373-385. |
| [68] |
SELKOE K, TOONEN R J. Marine connectivity: a new look at pelagic larval duration and genetic metrics of dispersal[J]. Marine Ecology Progress Series, 2011, 436: 291-305. |
| [69] |
RIGINOS C, DOUGLAS K E, JIN Y, et al. Effects of geography and life history traits on genetic differentiation in benthic marine fishes[J]. Ecography, 2011, 34(4): 566-575. |
| [70] |
FAURBY S, BARBER P H. Theoretical limits to the correlation between pelagic larval duration and population genetic structure[J]. Molecular ecology, 2012, 21(14): 3419-3432. |
| [71] |
HEDGECOCK D, BARBER P H, EDMANDS S. Genetic approaches to measuring connectivity[J]. Oceanography, 2007, 20(3): 70-79. |
| [72] |
WHITE C, SELKOE K A, WATSON J, et al. Ocean currents help explain population genetic structure[J]. Proceedings of the Royal Society B: Biological Sciences, 2010, 277(1688): 1685-1694. |
| [73] |
ALBERTO F, RAIMONDI P T, REED D C, et al. Isolation by oceanographic distance explains genetic structure for Macrocystis pyrifera in the Santa Barbara Channel[J]. Molecular Ecology, 2011, 20(12): 2543-2554. |
| [74] |
WARES J P. Community genetics in the Northwestern Atlantic intertidal[J]. Molecular Ecology, 2002, 11(7): 1131-1144. |
| [75] |
PATARNELLO T, VOLCKAERT F, CASTILHO R. Pillars of Hercules: is the Atlantic-Mediterranean transition a phylogeographical break?[J]. Molecular Ecology, 2007, 16(21): 4426-4444. |
| [76] |
HELLBERG M E, BURTON R S, NEIGEL J E, et al. Genetic assessment of connectivity among marine populations[J]. Bulletin of marine science, 2002, 70(1): 273-290. |
| [77] |
BRACCO A, LIU G, GALASKA M P, et al. Integrating physical circulation models and genetic approaches to investigate population connectivity in deep-sea corals[J]. Journal of Marine Systems, 2019, 198: 103189. |
| [78] |
ZENG C. Patterns of genetic connectivity in deep-sea vulnerable marine ecosystems and implications for conservation[D]. Wellington: Victoria University of Wellington. 2016.
|
| [79] |
LIMA L S, GHERARDI D F M, PEZZI L P, et al. Potential changes in the connectivity of marine protected areas driven by extreme ocean warming[J]. Scientific reports, 2021, 11(1): 1-12. |
| [80] |
JOHNSON J E, WELCH D J. Marine Fisheries Management in a Changing Climate: A Review of Vulnerability and Future Options[J]. Reviews in Fisheries Science, 2010, 18(1): 106-124. |
| [81] |
WERNBERG T, SMALE D A, THOMSEN M S. A decade of climate change experiments on marine organisms: procedures, patterns and problems[J]. Global Change Biology, 2012, 18(5): 1491-1498. |
| [82] |
GERBER L R, MANCHA-CISNEROS M D M, O'CONNOR M I, et al. Climate change impacts on connectivity in the ocean: Implications for conservation[J]. Ecosphere, 2014, 5(3): 1-18. |
| [83] |
PRASAD A. Coral population connectivity of Lakshadweep atolls under climate change[J]. Reef Encounter, 2020, 43. |
| [84] |
WREN J, TOONEN R. Effect of Extremes: How El Niño Events Affect Reef Fish Population Connectivity in the Hawaiian Islands[J]. American Geophysical Union, 2016, 2016: ME23A-02. |
| [85] |
CAVALCANTE G H, FEARY D A, BURT J A. The influence of extreme winds on coastal oceanography and its implications for coral population connectivity in the southern Arabian Gulf[J]. Marine pollution bulletin, 2016, 105(2): 489-497. |
| [86] |
REES S E, PITTMAN S J, FOSTER N, et al. Bridging the divide: Social–ecological coherence in Marine Protected Area network design[J]. Aquatic Conservation: Marine and Freshwater Ecosystems, 2018, 28(3): 754-763. |
| [87] |
PELC R, WARNER R, GAINES S. Geographical patterns of genetic structure in marine species with contrasting life histories[J]. Journal of Biogeography, 2009, 36(10): 1881-1890. |
| [88] |
TOONEN R J, ANDREWS K R, BAUMS I B, et al. Defining boundaries for ecosystem-based management: a multispecies case study of marine connectivity across the Hawaiian Archipelago[J]. Journal of Marine Biology, 2011, 2011. |
| [89] |
D'ALOIA C C, DAIGLE R M, CôTé I M, et al. A multiple-species framework for integrating movement processes across life stages into the design of marine protected areas[J]. Biological Conservation, 2017, 216: 93-100. |
| [90] |
CLAUDET J, OSENBERG C, DOMENICI P, et al. Marine reserves: fish life history and ecological traits matter[J]. Ecological applications, 2010, 20(3): 830-839. |
| [91] |
BOTSFORD L W, MICHELI F, HASTINGS A. Principles for the design of marine reserves[J]. Ecological applications, 2003, 13(sp1): 25-31. |
| [92] |
MCLEOD E, SALM R, GREEN A, et al. Designing marine protected area networks to address the impacts of climate change[J]. Frontiers in Ecology and the Environment, 2009, 7(7): 362-370. |
| [93] |
ALLAN J C, BEAZLEY K F, METAXAS A. Ecological criteria for designing effective MPA networks for large migratory pelagics: Assessing the consistency between IUCN best practices and scholarly literature[J]. Marine Policy, 2021, 127: 104219. |
| [94] |
陈义中. 黄海东海环流和长江冲淡水季节连续变化的数值模拟[D]. 上海: 华东师范大学, 2007. CHEN Yizhong. Numerical simulation of seasonal changes of the circulation and Changjiang diluted water in the Yellow Sea and East China Sea[D]. Shanghai: East China Normal University, 2007. |
| [95] |
AGOSTINI V N, GRANTHAM H S, WILSON J, et al. Achieving fisheries and conservation objectives within marine protected areas: zoning the Raja Ampat network[J]. The Nature Conservancy, Indonesia Marine Program, Denpasar, Inodnesia, 2012. |
| [96] |
SCHMIING M, DIOGO H, SERRãO SANTOS R, et al. Marine conservation of multispecies and multi-use areas with various conservation objectives and targets[J]. ICES Journal of Marine Science, 2015, 72(3): 851-862. |
| [97] |
FERNANDES L, DAY J, LEWIS A, et al. Establishing representative no‐take areas in the Great Barrier Reef: large‐scale implementation of theory on marine protected areas[J]. Conservation biology, 2005, 19(6): 1733-1744. |
| [98] |
WANG J L. Application of the one-migrant-per-generation rule to conservation and management[J]. Conservation Biology, 2004, 18(2): 332-343. |
 2023, Vol. 47
2023, Vol. 47


