文章信息
- 陈一, 刘二田, 战欣, 顾志峰, 王爱民. 2023.
- CHEN Yi, LIU Er-tian, ZHAN Xin, GU Zhi-feng, WANG Ai-min. 2023.
- 企鹅珍珠贝非致死性DNA提取方法的研究
- Nonlethal DNA extraction methods for Pteria penguin
- 海洋科学, 47(2): 55-62
- Marine Sciences, 47(2): 55-62.
- http://dx.doi.org/10.11759/hykx20220315004
-
文章历史
- 收稿日期:2022-03-15
- 修回日期:2022-09-10
现代分子生物学技术已广泛应用于双壳贝类的选择育种研究中, 许多学者从DNA水平研究了贝类的遗传多样性, 为品质改良、养殖和培育新品种提供理论依据[1-2]。在珍稀贝类的保护遗传学研究中, 通常需要在保持贝类存活的情况下进行。在贝类分子育种实践中, 经常需要在贝类生存或正常生理状态不受影响的前提下, 对大规模育种群体进行基因组DNA提取, 用于后续实验[3]。因而开发一种经济、简便的非致死性DNA提取技术对贝类遗传育种及珍稀贝类保护至关重要。足丝由细胞外蛋白质纤维组成[4], 可以在体外完成组装并发挥功能, 能够在潮湿条件下附着在各种坚硬的底物上[5-6], 可以抵御海浪冲击以及捕食者[7]。企鹅珍珠贝(Pteria penguin)的足丝易于获得且不会损伤贝内部软体组织, 同时其贝壳外缘薄而易碎, 用剪刀即可完成取样, 足丝及贝壳样品易于获得、可再生且对实验贝影响小。
目前海洋双壳贝类的DNA提取通常从软体组织样品获得[8], 而软体组织取样所带来的死亡率问题不可忽略。最常用的酚/氯仿抽提法需通过有机溶剂反复抽提获得高质量DNA, 此方法消耗大量软体组织, 取材量对于实验贝通常是致死性的[9-11]。Wang等[12]在进行牡蛎基因组DNA提取时, 通过将牡蛎(Crassostrea gigas)浸泡在硫酸镁(MgSO4)溶液中麻醉后打开外壳, 取少量的外套膜组织, 也造成了牡蛎的高死亡率(> 16.7%)。故建立一种非侵入性、安全有效的替代取样方式是十分必要的, 如通过软体动物的贝壳来进行DNA的提取。双壳贝类的贝壳是由生物矿化形成的, 用来保护其内部的软体组织免受捕食和干燥环境的侵害[13-14], 生物矿化源于有机和无机分子之间的相互作用以及碳酸钙(CaCO3)晶体壳的沉积[15]。软体动物的壳通过复杂有序的生物过程形成, 这种过程受各种有机分子的调节[16]。一些研究人员从古老的软体动物外壳中提取了DNA[17], Kuehn等[18]和Villanea等[19]的研究表明, DNA遗传信息可以从贝壳样品中成功提取主要依赖于晶体内沉积作用使其在内层壳的方解石中被完好保存, 且壳内DNA在贝壳取样50年后依然可以维持良好的保存状态, 这使得从软体动物的贝壳中提取DNA成为可能。因此, 海洋双壳类软体动物的贝壳作为遗传分析的DNA来源, 与软体组织相比更加具有优越性。足丝是由足腺分泌的蛋白质构成, 其沿着贝类足的长度在足腹沟中合成[7]。当老的足丝腐烂断裂时, 贝类会不断产生新的足丝[20], 此过程中足部细胞可能脱落在足丝中, 为从足丝中提取DNA提供可能。
目前已在多种水产动物中开展贝壳DNA提取方法的研究, 张凤梅等[21]通过对虾夷扇贝(Patinopecten yessoensis)、栉孔扇贝(Chlamys farreri)、牡蛎和侏儒蛤(Mulimia lateralis)4种双壳贝类进行壳DNA提取并评价, 提出了一种适用于双壳贝类的壳DNA提取方法。吕曼等[22]的研究表明, 胍裂解缓冲液法(GLB)在从玻璃质类底栖有孔虫卷转虫属壳中提取DNA的方法与其他方法相比更加有效。Jiang等[23]的研究则在Wang等[24]提出的苯酚-氯仿萃取法的基础上进行改进, 建立了一种改良版有机溶剂萃取法(OSE法), 同时将该方法与胍裂解缓冲液法(GLB)进行效率和质量比较, 发现改良版有机溶剂萃取法(OSE)提取效果优于GLB法。此外, Jiang等[23]的研究还表明从牡蛎腹部外缘取样可获得DNA含量较高的基因组DNA。
本研究在Jiang等[23]开发的有机溶剂萃取法(OSE)提取贝壳DNA的基础上, 结合Tiangen海洋动物组织基因组DNA提取试剂盒(DP324), 并通过脱钙与不脱钙两种处理方式对样品进行处理, 提出了一种新的非致死性DNA提取方法——足丝DNA提取, 以期望降低贝类取样时的死亡率, 提高DNA提取的效率和纯度, 为企鹅珍珠贝的亲缘鉴定和分子标记辅助育种等研究提供前期基础。
1 材料与方法 1.1 样品采集及清理企鹅珍珠贝实验个体为广西北海海区养殖的1龄群体, 暂养于海南海昌养殖基地水泥池中, 暂养期间每天投喂球等鞭金藻(Isochrysis galbana)和角毛藻(Chaetoceros calcitrans)混合藻水, 两天换水一次, 所用海水为经沙滤沉淀的新鲜天然海水, 盐度32~34, 水温23~24 ℃。随机挑选无明显损伤, 表观无病变活力好的个体1 000个, 随机剪取其中50个个体的贝壳和足丝用于后续死亡率的计算, 其中24个个体的贝壳和足丝用于DNA提取。
用眼科剪剪取约100 mg贝壳及100 mg足丝, 样品清洗在黄超敏[25]方法的基础上略有修改。剪取的贝壳和足丝分别置于50 mL离心管中, 超纯水浸泡5 min。加入生理盐水以清洗样品表面污染物2次, 每次3 min, 上下晃动以充分洗涤。超纯水浸泡洗涤10 min, 无尘纸吸干水分后, 用镊子将足丝撕开呈丝状。在50 mL离心管中再次加入生理盐水, 上下颠倒洗涤贝壳和足丝样品3次, 每次2 min, 于超声清洗仪中处理15 min以去除附着物。使用超纯水清洗贝壳和足丝2次, 每次5 min; 无尘纸吸干水分后, 将贝壳和足丝样品放入干净且干燥的1.5 mL离心管中备用。
1.2 企鹅珍珠贝死亡率的计算剪取贝壳和足丝的50个个体及剩余950个个体继续养殖一周, 并统计企鹅珍珠贝的死亡率。死亡率计算公式为: 死亡率=死亡个体数/总个体数×100%。
1.3 贝壳及足丝样品脱钙处理① 清洗后的企鹅珍珠贝边缘新生贝壳及足丝转移到1.5 mL离心管, 灭菌双蒸水清洗后用剪刀剪碎样品, 液氮预冷10 min, 使用多样品组织研磨仪研磨(60 HZ, 60 s)成粉末。
② 各取100 mg左右贝壳及足丝粉末转移至新的1.5 mL离心管中, 加入1 mL的EDTA(pH=8.0)螯合Ca2+进行脱钙处理。
③ 将离心管以1 000 r/min在37 ℃的恒温金属浴上震荡12 h, 以诱导脱钙。
④ 4 000 r/min离心15 min, 弃上清液以去除多余的盐离子, 并用200 μL去离子水清洗沉淀。
1.4 海洋动物组织基因组DNA提取试剂盒法提取贝壳及足丝DNA各取100 mg脱钙及未脱钙的贝壳及足丝样品粉末采用Tiangen海洋动物组织基因组DNA提取试剂盒(DP324)提取DNA, 在溶解产物时添加20 μL TE缓冲液, 其余步骤均按照说明书进行。提取的DNA产物编号分别为: SK(未脱钙贝壳试剂盒法提取的DNA), DSK(脱钙贝壳试剂盒法提取的DNA), BK(未脱钙足丝试剂盒法提取的DNA)和DBK(脱钙足丝试剂盒法提取的DNA)。
1.5 有机溶剂萃取法提取贝壳及足丝DNA(OSE法)各取100 mg脱钙和未脱钙的贝壳及足丝样品粉末采用Jiang等[23]的OSE法提取样品DNA, 最后将提取产物溶于20 μL TE缓冲液中, 于–20 ℃保存备用。提取的DNA产物编号分别为: SO(未脱钙贝壳OSE法提取的DNA), DSO(脱钙贝壳OSE法提取的DNA), BO(未脱钙足丝OSE法提取的DNA)和DBO(脱钙足丝OSE法提取的DNA)。
1.6 DNA提取质量及纯度检测使用超微量紫外可见光分光光度计nanodrop2000测定DNA质量。根据陈雅琦等[26]描述的方法计算DNA提取率。取1 μL DNA提取液, 利用光度计测定仪测定DNA的质量浓度(ng/μL)及纯度(在波长260、280和230 nm处的吸光值), 根据其DNA浓度对DNA得率进行定量, 即DNA含量(μg)=质量浓度×TE缓冲液体积(μL)×10–3, DNA提取率(μg/mg)=DNA含量/新鲜样品重量。
1.7 企鹅珍珠贝DNA提取产物PCR扩增检测目标基因表达将不同处理及提取方法获得的DNA用作模板, 利用企鹅珍珠贝COI基因序列及18S rRNA基因序列, 用Primer 5设计引物, COI及18S rRNA的引物(表 1)由铂尚生物技术(上海)有限公司合成, PCR产物测序由海南楠山生物技术有限公司完成。
| 引物名称 | 引物序列(5′-3′) | 扩增片段长度/bp |
| COI | F: TTAGGATAGTCAGGTGGAG | 400~500 |
| R: CTTACAACGACAACGAAA | ||
| 18S rRNA | F: CGTTCTTAGTTGGTGGAGCG | 100~200 |
| R: AACGCCACTTGTCCCTCTAA |
PCR反应在50 μL体系中进行, 包含5 μL基因组DNA、0.25 μL TaKaRa Taq DNA聚合酶(5 U/μL)、5 μL 10×PCR Buffer(Mg2+ plus)、2.5 μL正向引物(10 μmol/L)和2.5 μL反向引物(10 μmol/L)、灭菌双蒸水补足体积至50 μL。PCR反应条件为: 94 ℃预变性5 min, 进行35个循环反应: 94 ℃ 30 s, 58.5 ℃ 30 s, 72 ℃ 30 s, 最后在72 ℃延伸7 min。
1.8 琼脂糖凝胶电泳提取的贝壳和足丝DNA用1.0%琼脂糖凝胶电泳检测(160 V, 15 min), PCR扩增产物用2.0%琼脂糖凝胶电泳检测(160 V, 15 min), 并采用凝胶成像系统进行拍照观察。
1.9 数据分析数据用平均值±标准差(Mean±SD)表示。利用Excel 2019和SPSS 19.0软件对实验数据进行处理。单因素方差分析采用One-way ANOVA进行, 差异显著水平为P < 0.05。
2 结果 2.1 企鹅珍珠贝死亡率比较剪取贝壳及足丝进行DNA提取的企鹅珍珠贝(n=50)在取样后, 继续养殖一周, 未出现死亡个体, 死亡率为0%。未进行贝壳及足丝取样的企鹅珍珠贝(n=950)养殖一周后也无死亡个体, 死亡率为0%, 因此, 剪取贝壳及足丝并未造成企鹅珍珠贝的死亡。
2.2 贝壳和足丝样品DNA提取方法的比较贝壳及足丝DNA提取方法的结果见图 1和表 2, 不同提取方法获得的DNA产物浓度与DNA提取效率趋势一致。贝壳DNA提取方法中, 未脱钙处理贝壳的试剂盒法其DNA提取效率为(0.006 6±0.000 2) μg/mg, 显著高于其他三种贝壳DNA提取方法。足丝DNA提取方法中, 有机溶剂法(OSE法)提取效率及获得的DNA浓度显著高于试剂盒法, 脱钙处理足丝与未脱钙处理足丝的DNA提取效率则无显著性差异, 分别为(0.016 7±0.002 9) μg/mg和(0.016 1±0.003 1) μg/mg。同时, 足丝DNA的OSE法提取效率显著高于足丝试剂盒法及贝壳DNA的所有提取方法。未脱钙处理足丝DNA提取的OSE法获得的DNA产物纯度最优, A260/280值为1.400 0±0.040 0, A260/230值显著高于贝壳DNA及足丝DNA的其他处理提取方法, 为0.910 0± 0.080 0。
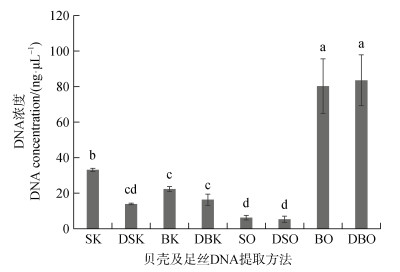 |
| 图 1 企鹅珍珠贝贝壳和足丝的DNA浓度 Fig. 1 Pteria penguin shell and byssus DNA concentrations 注: SK为试剂盒法提取的未脱钙贝壳DNA; DSK为试剂盒法提取的脱钙贝壳DNA; BK为试剂盒法提取的未脱钙足丝DNA; DBK为试剂盒法提取的脱钙足丝DNA; SO为有机溶剂法提取的未脱钙贝壳DNA; DSO为有机溶剂法提取的脱钙贝壳的DNA; BO为有机溶剂法提取的未脱钙足丝DNA; DBO为有机溶剂法提取的脱钙足丝DNA |
| 方法 | 样品 | A260/280 | A260/230 | 提取效率(μg/mg) |
| 试剂盒法 | 未脱钙贝壳 | 1.550 0±0.070 0b | 0.450 0±0.070 0c | 0.006 6±0.000 2b |
| 脱钙贝壳 | 1.810 0±0.160 0b | 0.220 0±0.060 0d | 0.002 8±0.000 1de | |
| 未脱钙足丝 | 1.790 0±0.030 0b | 0.520 0±0.050 0bc | 0.004 5±0.000 3c | |
| 脱钙足丝 | 1.840 0±0.060 0b | 0.210 0±0.050 0d | 0.003 3±0.000 6cd | |
| 有机溶剂萃取法 | 未脱钙贝壳 | 1.200 0±0.110 0b | 0.560 0±0.080 0b | 0.001 2±0.000 3ef |
| 脱钙贝壳 | 2.950 0±2.080 0a | 0.190 0±0.060 0d | 0.001 1±0.000 3f | |
| 未脱钙足丝 | 1.400 0±0.040 0b | 0.910 0±0.080 0a | 0.016 1±0.003 1a | |
| 脱钙足丝 | 1.480 0±0.040 0b | 0.570 0±0.090 0bc | 0.016 7±0.002 9a | |
| 注: 同一列上标字母相同表示差异不显著(P > 0.05), 不同字母表示存在显著性差异(P < 0.05) | ||||
以提取的企鹅珍珠贝贝壳和足丝基因组DNA作为模板, 进行COI和18S rRNA的PCR扩增, 结果发现, 贝壳有机溶剂法提取的DNA扩增条带不够清晰, 且脱钙贝壳OSE法未能扩增出COI基因片段, 但足丝试剂盒法、OSE法及贝壳试剂盒法提取的DNA扩增条带清晰明亮, 扩增效果较好(图 2), 这与图 1及表 2中的DNA提取结果一致。
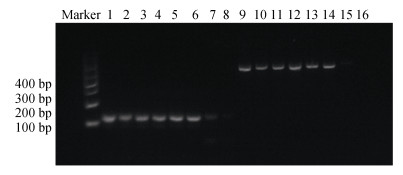 |
| 图 2 不同方法提取贝壳DNA和足丝DNA的PCR扩增产物的琼脂糖凝胶电泳图谱 Fig. 2 Agarose gel electrophoresis patterns of the PCR amplification products extracted from the shell and byssus using different methods 注: 1-8为18S rRNA的PCR扩增条带; 9-16位COI的PCR扩增条带。模板DNA分别为: BK, DBK, BO, DBO, SK, DSK, SO, DSO |
为确定贝壳DNA及足丝DNA是否来源于企鹅珍珠贝, 以足丝和贝壳DNA做模板, 对18S rRNA和COI基因进行PCR扩增和测序, 试剂盒法及有机溶剂法扩增出的足丝和贝壳18S rRNA和COI基因片段与NCBI的核酸数据库中企鹅珍珠贝相应基因的序列相似度分别为98.75%和99.39%(图 3)。
 |
| 图 3 贝壳和足丝18S rRNA(A)和COI(B)基因的序列比对结果 Fig. 3 Sequence alignment results of the shell and byssus 18S rRNA and COI genes 注: SK, DSK, BK, DBK, SO, DSO, BO, DBO为PCR产物测序结果, Pp为NCBI数据库企鹅珍珠贝18S rRNA和COI序列 |
在本项研究中, 我们首次从企鹅珍珠贝足丝中提取出DNA, 其浓度和纯度可满足后续PCR实验需求。足丝是一种外源性黏附结构[27], 由一个柔软的胶原内核组成, 周围环绕着硬化、固化的多酚蛋白。构成足丝的黏附蛋白是由贝类的足部产生的, 这些蛋白储存在足部, 然后分泌或释放到足腹沟中, 从而在水下形成了牢固黏附的足丝[28-29]。企鹅珍珠贝足丝粗壮, 丝部的足丝线黏连在一起, 末端成离散状[30], 而DNA是一种细胞成分[24], 因此, 我们推测足丝中可能存在着足部脱落的细胞, 从而使得从足丝中提取DNA成为可能。
随着现代生物学技术的发展, 分子标记技术成为辅助贝类育种、品质改良和珍稀贝类保护遗传学研究中的重要技术手段, 这类研究开展的前提是需要在保持贝类正常生理状态下大规模获取样品的基因组DNA。然而, 采用酚/氯仿抽提法、CTAB法、SDS法、高盐法以及商业化试剂盒[31-32]等方法提取外套膜[33]、鳃丝[34]等软体组织的DNA, 组织取样量可能会造成贝类感染、畸形甚至死亡等问题, 因此许多学者对贝类非致死性DNA提取方法进行了探索。Kurita等[35]通过注射器吸取太平洋牡蛎体腔液来获得基因组DNA, Holman等[36]通过过滤牡蛎养殖环境中的海水来获取基因组DNA, 虽达到了非致死性取材的目的, 但这类取样方法操作繁琐, 大批量取样效率低下。窦海龙[37]提出了从动物粪便中提取DNA的方法, 然而由于粪便中所含杂质过多, 如细菌、病毒、TaqDNA聚合酶抑制剂等, 提取出的DNA纯度不高, 不适用于后续实验。本研究采用企鹅珍珠贝贝壳及足丝进行DNA提取, 能够在不导致实验贝死亡的前提下快速完成样品的取样, 且未脱钙足丝OSE法的DNA提取效率、DNA产物纯度均高于足丝试剂盒法及贝壳DNA的所有提取方法, 经序列比对发现, 足丝DNA及贝壳DNA均来自于企鹅珍珠贝基因组, 研究结果为贝类大规模开展分子标记辅助育种、亲缘关系分析等研究提供前期基础。
Jiang等[23]采用了OSE法及胍裂解法(GLB法)对100 mg脱钙的太平洋牡蛎腹部外缘贝壳进行了DNA提取, DNA提取效率分别为(0.152 5±0.001 7) μg/mg和(0.111 8±0.000 7) μg/mg, 高于本研究中的所有提取方法。A260/280值分别为1.320 0±0.020 0和0.970 0± 0.010 0, 其DNA纯度低于企鹅珍珠贝的贝壳DNA和足丝DNA。足丝OSE法提取的DNA产物纯度最优, 但A260/A280和A260/A230的比值仍未达到2, 可能与DNA提取过程中蛋白质和盐分杂质较多有关, 这与Jiang等[23]对牡蛎壳DNA提取的结果一致。试剂盒法获得的DNA杂质较少, 其A260/A280值高于OSE法, 可能是试剂盒中使用的离心吸附柱中吸附膜的作用, 使蛋白质类和酚类杂质减少。张天琦等[3]通过棉签和滤纸擦拭虾夷扇贝、栉孔扇贝及海湾扇贝(Argopecten irradians)软体组织的方法进行DNA提取, 棉签擦拭的DNA产物浓度高于滤纸擦拭, 分别为97.66、97.22及82.18 ng/μL。本研究采用的足丝OSE法获得的DNA产物浓度在(80.32±15.37) ng/μL~(83.58±14.35) ng/μL间, 显著高于贝壳试剂盒法及OSE法, 且DNA产物浓度及纯度与上述擦拭法接近。
本研究同样比较了脱钙或未脱钙对贝壳DNA、足丝DNA的提取效果, 结果显示, 脱钙贝壳OSE法其A260/A280值高于2, 表明提取的DNA中可能存在RNA污染, 同时脱钙与未脱钙处理下贝壳OSE法提取的DNA浓度及提取效率均为最低, 这可能是其PCR扩增条带不清晰的原因。230 nm处波长是碳水化合物的最高吸收峰的吸收波长, 故A260/A230的大小反映了DNA样品中存在的糖类、盐类或有机溶剂等的污染情况[32]。足丝OSE法的A260/A230比值高于贝壳的所有处理和提取方法, 而未脱钙足丝OSE法的A260/A230值显著高于脱钙足丝OSE法, 这可能是因为在脱钙过程中EDTA脱钙液中含有的盐离子未能被完全清洗除去。未脱钙贝壳试剂盒法的提取效率显著高于脱钙贝壳试剂盒法, 这可能与贝壳样品是否脱钙有关。软体动物的壳是由生物矿化形成的[13], 生物矿化源于有机和无机分子之间的相互作用以及碳酸钙(CaCO3)晶体壳的沉积[15]。不同的软体动物其有机质和无机质的占比有所区别, 但都以无机基质为主, 甚至牡蛎的贝壳中无机质部分(以CaCO3为主)占到了牡蛎贝壳质量的90%以上[38]。由于EDTA能够螯合Mg2+、Ca2+等金属离子, 抑制脱氧核糖核酸酶对DNA的降解作用(DNase作用需要一定的金属离子做辅基), 故是否脱钙也会影响贝壳DNA的提取效率及纯度[10]。Fremy[39]检验了不同种类贝的足丝在脱钙后的残余物, 并将其命名为“贝壳硬蛋白(conchiolin)”。Schlossberger和Krukenberg[39]对贻贝足丝进行了初步分析, 并将其与贝壳硬蛋白进行了比较, 他们得出的结论是二者是相同的, 即脱钙后的足丝与未脱钙足丝成分相同, 这可能是本研究中不同处理下的足丝样品在相同提取方法中提取效率无显著性差异的原因。
本研究提出的非致死性DNA提取方法中实验贝的死亡率为0%, 有效降低了双壳贝类取样过程中的死亡率。在贝壳DNA和足丝DNA的提取方法中, 未脱钙足丝有机溶剂萃取法(OSE法)的提取效率、获得的DNA浓度及DNA纯度均高于足丝试剂盒法和贝壳DNA所有提取方法。综合考虑, 对于具足丝的珍稀贝类, 采用OSE法提取足丝DNA具有取样速度快、对贝的刺激小的优点, 既可以保证实验样本的存活率, 又可以获得纯度更高的基因组DNA, 为珍稀贝类的遗传结构分析、经济贝类的遗传育种研究提供前期基础。
| [1] |
王小玉. 海水珍珠贝分子标记分析及遗传图谱构建[D]. 武汉: 华中农业大学, 2006. WANG Xiaoyu. Study on molecular markers of some pearl oysters in Pinctada and genetic mapping of P. fucata (Gould)[D]. Wuhan: Huazhong Agricultural University, 2006. |
| [2] |
王永丽. 企鹅珍珠贝野生与养殖群体的微卫星标记及形态学对比分析[D]. 湛江: 广东海洋大学, 2010. WANG Yongli. Study on microsatellite marker and morphological contrastive analysis of wild and cultivated Pteria penguin (Röding)[D]. Zhanjiang: Guangdong Ocean University, 2010. |
| [3] |
张天琦, 刘平平, 吕佳, 等. 高效非损伤性扇贝DNA提取方法的建立及效果评价[J]. 中国海洋大学学报: 自然科学版, 2021, 51(5): 24-31. ZHANG Tianqi, LIU Pingping, LV Jia, et al. Development and evaluation of a rapid, cost-effective and non-destructive DNA extraction method in scallops[J]. Periodical of Ocean University of China, 2021, 51(5): 24-31. |
| [4] |
O'DONNELL M J, GEORGE M N, CARRINGTON E. Mussel byssus attachment weakened by ocean acidification[J]. Nature Climate Change, 2013, 3(6): 587-590. DOI:10.1038/nclimate1846 |
| [5] |
TAMARIN A, LEWIS P, ASKEY J. The structure and formation of the byssus attachment plaque in Mytilus[J]. Journal of Morphology, 1976, 149(2): 199-221. DOI:10.1002/jmor.1051490205 |
| [6] |
WAITE J H. Mussel adhesion-essential footwork[J]. Journal Experiment Biology, 2017, 220(4): 517-530. DOI:10.1242/jeb.134056 |
| [7] |
PRIEMEL T, DEGTYAR E, DEAN M N, et al. Rapid self-assembly of complex biomolecular architectures during mussel byssus biofabrication[J]. Nature Communications, 2017, 8: 14539. DOI:10.1038/ncomms14539 |
| [8] |
SUQUET M, KERMOYSAN G D, ARAYA R G, et al. Anesthesia in Pacific oyster, Crassostrea gigas[J]. Aquatic Living Resources, 2009, 22(1): 29-34. DOI:10.1051/alr/2009006 |
| [9] |
WINNEPENNINCKX B, BACKELJAU T, WACHTE R D. Extraction of high molecular weight DNA from molluscs[J]. Trends in Genetics, 1993, 9(12): 407. DOI:10.1016/0168-9525(93)90102-N |
| [10] |
鲍毅新, 孙波, 张龙龙, 等. 对动物组织DNA提取方法的改进及PCR检测[J]. 浙江师范大学学报(自然科学版), 2009, 32(3): 317-321. BAO Yixin, SUN Bo, ZHANG Longlong, et al. The improvement for method DNA extraction from animal tissue and PCR detection[J]. Journal of Zhejiang Normal University, 2009, 32(3): 317-321. |
| [11] |
徐伟丽, 杜明, 李启明, 等. 动物肌肉组织基因组DNA两种提取方法的比较[J]. 食品工业科技, 2011, 32(12): 81-84. XU Weili, DU Ming, LI Qiming, et al. Comparison of the effects of two methods for four animal muscular tissue DNA extraction[J]. Science and Technology of Food Industry, 2011, 32(12): 81-84. |
| [12] |
WANG X, LI L, XU F, et al. Genomic DNA extraction from in vivo sampled tissue of pacific oyster, Crassostrea gigas[J]. Israeli Journal of Aquaculture-Bamidgeh, 2011, 63(6): 235-243. |
| [13] |
OBERLANDER H. The invertebrate integument: biology of the integument[J]. Science, 1984, 226(4671): 162-162. |
| [14] |
FREDERIC M. The formation and mineralization of mollusk shell[J]. Frontiers in Bioscience, 2012, 4(3): 1099-1125. |
| [15] |
MANN S, ARCHIBALD D D, DIDYMUS J M, et al. Crystallization at inorganic-organic interfaces: biominerals and biomimetic synthesis[J]. Science, 1993, 261(5126): 1286-1292. DOI:10.1126/science.261.5126.1286 |
| [16] |
SAMATA T. Structure and function of the organic matrix in the nacreous layer of Pinctada fucata [C]//SUGA S, NAKAHARA H. Mechanisms and phylogeny of mineralization in biological systems. Tokyo: Springer Japan, 1991: 23-24.
|
| [17] |
DER SARKISSIAN C, PICHEREAU V, DUPONT C, et al. Ancient DNA analysis identifies marine mollusc shells as new metagenomic archives of the past[J]. Molecular Ecology Resources, 2017, 17(5): 835-853. DOI:10.1111/1755-0998.12679 |
| [18] |
KUEHN R, GEIST J, WUNDERLICH H. Use of mollusc shells for DNA-based molecular analyses[J]. Jour nal of Molluscan Studies, 2008, 74(4): 337-343. DOI:10.1093/mollus/eyn025 |
| [19] |
VILLANEA F A, PARENT C E, KEMP B M. Reviving Galápagos snails: ancient DNA extraction and amplification from shells of probably extinct endemic land snails[J]. Journal of Molluscan Studies, 2016, 82(3): 449-456. DOI:10.1093/mollus/eyw011 |
| [20] |
MOESER G M, CARRIGTON E. Seasonal variation in mussel byssal thread mechanics[J]. Journal Experiment Biology, 2006, 209(10): 1996-2003. DOI:10.1242/jeb.02234 |
| [21] |
张凤梅, 连姗姗, 刘思诺, 等. 双壳类软体动物贝壳DNA高效提取及方法学评价[J]. 中国海洋大学学报(自然科学版), 2020, 50(S01): 41-47. ZHANG Fengmei, LIAN Shanshan, LIU Sinuo, et al. Efficient extraction of bivalve mollusks shell DNA and its methodological evaluation[J]. Periodical of Ocean University of China, 2020, 50(S01): 41-47. |
| [22] |
吕曼, 类彦立, 李铁刚. 玻璃质壳类底栖有孔虫卷转虫属Ammonia spp. 的DNA保存方式和提取效能比较研究[J]. 海洋与湖沼, 2016, 47(2): 346-353. LV Man, LEI Yanli, LI Tiegang. Efficiency of DNA preservation and extraction from benthic hyaline of Ammonia spp. : a methodological comparison[J]. Ocea no logia et Limnologia Sinica, 2016, 47(2): 346-353. |
| [23] |
JIANG Q Y, WEI L, GAI C W, et al. An improved extraction method reveals varied DNA content in different parts of the shells of pacific oysters[J]. Aquatic Living Resources, 2019, 32(5). |
| [24] |
WANG X, SONG X, LI L, et al. An improved method of DNA extraction from the shell of the pacific oyster, Crassostrea gigas[J]. Israeli Journal of Aquaculture- Bamidgeh, 2012, 64(404): 557-560. |
| [25] |
黄超敏. 四种海水双壳贝类腹足和足丝的比较解剖研究[D]. 海口: 海南大学, 2011. HUANG Chaomin. Study on the comparative anatomy of four marine bivalve molluscs gastropods and byssus[D]. Haikou: Hainan University, 2011. |
| [26] |
陈雅琦, 苏楷淇, 李春杰. 醉马草DNA提取方法的对比与优化[J]. 草学, 2020, 5: 32-38. CHEN Yaqi, SU Kaiqi, LI Chunjie. The comparison and optimization of DNA extraction Methods from Achnatherum inebrians (Drunken horse grass)[J]. Journal of Grassland and Forage Science, 2020, 5: 32-38. |
| [27] |
SILVERMAN H G, ROBERTO F F. Understanding marine mussel adhesion[J]. Marine Biotechnology, 2007, 9(6): 661-681. DOI:10.1007/s10126-007-9053-x |
| [28] |
YU M, DEMING T J. Synthetic polypeptide mimics of marine adhesives[J]. Macromolecules, 1998, 31(15): 4739-4745. DOI:10.1021/ma980268z |
| [29] |
YU M, HWANG J, DEMING T J. Role of L-3, 4-dihydroxyphenylalanine in mussel adhesive proteins[J]. Journal of the American Chemical Society, 1999, 121(24): 5825-5826. DOI:10.1021/ja990469y |
| [30] |
VASQUEZ H E, ZHENG X, GU Z, et al. Relationships between shell morphological traits and the byssus dimensions in the winged pearl oyster Pteria penguin (Röding, 1798) cultivated in Sanya, Hainan Island, China[J]. Journal of Shellfish Research, 2017, 36(3): 669-676. DOI:10.2983/035.036.0316 |
| [31] |
李联泰, 安贤惠. 几种海水经济贝类DNA提取方法探讨[J]. 淮海工学院学报(自然科学版), 2005, 14(4): 66-69. LI Liantai, AN Xianhui. Methods for DNA extraction from shellfishes[J]. Journal of Huaihai Institute of Tech no logy (Natural Science Edition), 2005, 14(4): 66-69. |
| [32] |
程浩, 查勇, 高丹丹, 等. 不同动物肌肉组织中DNA提取方法研究[J]. 西北民族大学学报(自然科学版), 2020, 41(2): 73-79. CHENG Hao, ZHA Yong, GAO Dandan, et al. Study on methods of DNA extraction from different muscle tissues in animal[J]. Journal of Northwest University for Nationalities (Natural Science), 2020, 41(2): 73-79. |
| [33] |
BERG D J, HAAG W R, GUTTMAN S I, et al. Mantle biopsy: a technique for nondestructive tissue-sampling of freshwater mussels[J]. Journal of the North American Benthological Society, 1995, 14(4): 577-581. DOI:10.2307/1467542 |
| [34] |
MAO J X, LV J, MIAO Y, et al. Development of a rapid and efficient method for non-lethal DNA sampling and genotyping in scallops[J]. PLoS One, 2013, 8(7): 68096. |
| [35] |
KURITA Y, KIJIMA A. A noninvasive method for extracting bivalve DNA from the water-filled mantle cavity[J]. Hydrobiologia, 2019, 829(1): 237-243. |
| [36] |
HOLMAN L E, HOLLENBACK C M, ASHTON T J, et al. Demonstration of the use of environment DNA for the noninvasive genotyping of a bivalve mollusk, the European flat oyster (Ostrea edulis)[J]. Frontiers in Genetics, 2019, 10: 1159. |
| [37] |
窦海龙. 非损伤性取样提取动物DNA方法探讨[J]. 中国草食动物科学, 2012, 32(3): 54-56. DOU Hailong. Preliminary study on the noninvasive sampling of animal DNA[J]. China Herbivore Science, 2012, 32(3): 54-56. |
| [38] |
苗建银, 赵海培, 李超柱, 等. 牡蛎壳的开发利用[J]. 新农业, 2012, 6: 62-63. MIAO Jianyin, ZHAO Haipei, LI Chaozhu, et al. The exploitation of oyster shells[J]. Modern Agriculture, 2012, 6: 62-63. |
| [39] |
BROWN C H. Some structural proteins of Mytilus edulis[J]. Journal of Cell Science, 1952, 93(4): 487-503. |
 2023, Vol. 47
2023, Vol. 47

