文章信息
- 包群靖, 毕芳玲, 蔡春尔, 李可俊, 何培民. 2023.
- BAO Qun-jing, BI Fang-ling, CAI Chun-er, LI Ke-jun, HE Pei-min. 2023.
- 抗生素对浒苔形态及共附生细菌多样性的影响
- Effects of antibiotics on the morphology and associated bacterial diversity of Ulva prolifera
- 海洋科学, 47(4): 108-116
- Marine Sciences, 47(4): 108-116.
- http://dx.doi.org/10.11759/hykx20220519002
-
文章历史
- 收稿日期:2022-05-19
- 修回日期:2022-06-07
2. 水域环境生态上海高校工程研究中心, 上海 201306
2. Water Environment and Ecology Engineering Center of Higher Education, Shanghai 201306, China
大型海藻是近海生态系统中非常重要的初级生产者。大型海藻表面有大量共附生细菌, 这是细菌和藻类相互选择的结果[1-3]。海藻的表面为微生物附着提供了适合的基质, 同时海藻分泌出各种有机物质, 为细菌繁殖和生长提供了营养物质[4-5]。同样藻类也依赖细菌来获取外源钴胺素、硫胺素和生物素等才能生长[6]。研究进一步发现细菌对藻类生长、孢子萌发、病原体抗性具有重要作用, 特别是共附生细菌对于藻类诱导形态建成具有促进作用[7], 实验已证明无菌培养的大型绿藻和褐藻生长缓慢或形态发生异常[8-13]。Spoerner等[14]与Alsufyani等[15]通过无菌培养Ulva mutabilis配子, 发现再生藻体的形态不正常, 发育为愈伤组织状, 由细胞壁畸形的未分化细胞组成。王瑜[16]利用5种抗生素联合处理浒苔孢子, 获得的无菌浒苔愈伤组织也缺乏典型浒苔形态。目前大部分的无菌藻类都是利用抗生素联合培养的方式获得, 进一步研究发现抗生素联合培养很难去除与石莼属共生的所有微生物[17], 大型海藻共附生细菌有外生菌和内生菌之分, 且同一种藻类不同植株之间的共赴生细菌种类是存在差异的[18-19]。为了更好研究内生菌和外生菌对藻类的作用, 最近研究发现采用石英砂共涡旋振荡方式可以去除外生菌且不影响内生菌, 这将有助于分析藻类外生菌和内生菌的群落组成和宿主特异性[20-21]。
大型藻类和细菌之间的化学物质交换和物理接触是两者共生的基础[22], 越来越多的证据表明, 细菌释放一些化学物质对藻类形态发育有影响。U. mutabilis藻体发育需要藻类生长和形态发育促进因子(AGMPF)[23]。由Maribacter spp. 释放的细菌半萜烯类化合物——thallusin是目前唯一已知的AGMPF[12, 24]。当无菌U. mutabilis藻体同时接种2种海洋细菌Roseovarius sp. MS2和Maribacter sp. MS6时, 藻体会从愈伤组织样表型发育成由双层细胞和组织组成的表型[14]。其中Roseovarius sp. MS2促进细胞分裂和藻叶片细胞生长, 作用类似于陆地植物中的细胞分裂素功能[25], 而Maribacter sp. MS6诱导藻类细胞壁和初级根状细胞的形成, 作用类似于陆地植物中的生长素。细菌除了对大型绿藻形态有诱导作用, 还发现丝状单胞菌Hyphomonas(属于α-变形菌门)可以诱导红藻Neoporphyra yezoensis原生质体的正常形态发生[26]。可见藻类与其相关细菌之间的相互作用对藻类的形态和生长具有不可忽视的影响。
本研究采用抗生素联合处理的方式[27], 使用3种抗生素(青霉素G、硫酸新霉素和多黏菌素B)联合处理观察浒苔形态的变化, 利用thallusin探究其对浒苔生长的影响, 并采用16S rDNA高通量测序分析浒苔共附生细菌群落变化, 为浒苔共附生细菌功能的深入研究提供重要参考。
1 材料与方法 1.1 藻种来源与培养黄海绿潮漂浮浒苔(Ulva prolifera)藻体于2008年7月在山东省青岛市海边采集, 在实验室纯化, 利用浒苔配子具有趋光性, 判断藻体为配子体浒苔, 并在10℃低温保种。浒苔藻体养条件为: 温度20±1℃, 光照条件120 μmol·m−2·s−1, L∶D = 12 h∶12 h。采用高浓度海水(金山区, 上海)稀释成25~30盐度的海水, 并进行高压灭菌后, 用无菌注射器和0.22 μm针头式滤器加入营养盐配制VSE培养基[28]备用。
1.2 抗生素培养液配制用无菌注射器和0.22 μm针头式滤器将青霉素G、硫酸新霉素和多黏菌素B液体抗生素加入到灭菌的VSE培养基中, 摇匀。混匀后的液体抗生素终浓度为: 青霉素G 0.1 g/L; 硫酸新霉素0.1 g/L; 多黏菌素B 0.03 g/L[29], 配制全程均采用无菌操作。
1.3 浒苔配子体培养实验抗生素处理组(K组): 将浒苔藻体放散的配子加入到含有3种抗生素的VSE培养基中进行培养, 每5 d更换1次含有抗生素的VSE培养基, 直到幼苗发育至长度为3 mm时转移到圆形培养瓶, 继续用抗生素的培养基培养。
对照组(KH组): 即用不添加抗生素的VSE培养基培养浒苔配子。
每个组均设立3个重复平行组, 采用无菌冲气培养, 共培养25 d, 每5 d测量一次浒苔藻体长度以及更换1次培养液, 培养过程均采用无菌操作。
1.4 浒苔共生细菌的分离分别取培养25 d后的KH组和K组浒苔藻体, 利用无菌海水和无菌钢珠在冷冻研磨仪(上海沪析)以70 Hz频率研磨浒苔植株120 s, 使浒苔藻株完全破碎。离心管直接以3 000 rpm的速度离心30 s, 随后在超净工作台中将离心管内的上清液转移到新的灭菌离心管当中, 继续3 000 rpm离心2 min, 浒苔共附生菌则沉积在离心管壁上[16]。
1.5 浒苔共附生菌DNA的提取与片段扩增采用E.Z.N.A.® soil DNA kit试剂盒提取共附生菌的DNA。用1%的琼脂糖凝胶电泳检测细菌DNA, 在条带清晰、没有拖尾或杂带的情况下, 用NanoDrop 2000测定细菌DNA的浓度和纯度, DNA稀释到10 ng/μL。经鉴定质量合格的细菌基因组DNA进行细菌16S rRNA基因的V3—V4可变区巢式PCR扩增。引物序列如下表 1, 用于后续多样性分析。第一轮PCR扩增体系(20 μL)如下: 5×TransStart FastPfu缓冲液4 μL, 2.5 mmol/L dNTPs 2 μL, 正反引物(5 μmol/L)0.8 μL, 样本DNA 1 μL, ddH2O 11.4 μL。第二轮PCR扩增体系与第一次相同, 样本DNA更换为第一轮PCR产物。第一轮PCR反应条件为: 95℃预变性3 min; 95℃变性30 s, 46℃退火30 s, 72℃延伸30 s, 30个循环; 72℃延伸10 min。第二轮PCR反应条件为: 95℃预变性3 min; 95℃变性30 s, 55℃退火30 s, 72℃延伸30 s, 27个循环; 72℃延伸10 min。
| 引物 | 序列 |
| 799F | 5′-AACMGGATTAGATACCCKG-3′ |
| 1392R | 5′-ACGGGCGGTGTGTRC-3′ |
| 799F | 5′-AACMGGATTAGATACCCKG-3′ |
| 1193R | 5′-ACGTCATCCCCACCTTCC-3′ |
将同一样品的PCR产物混合后回收, 回收的产物使用AxyPrep DNA凝胶提取试剂盒(Axygen Biosciences, Union City, CA, USA)进行纯化, 并用2% 琼脂糖凝胶电泳检测。使用NEXTFLEX Rapid DNA-SEQ Kit进行文库构建, 最后采用Illumina Miseq PE300平台进行测序(上海美吉生物医学科技有限公司)。
下机的原始序列采用Trimmomatic软件和FLASH软件分别进行质控和拼接。使用UPARSE软件(7.1版)(http://drive5.com/uparse/), 按照97% 的相似度对OTU序列进行聚类并剔除嵌合体。使用RDP分类器(http://rdp.cme.msu.edu/)对每个序列进行注释进行物种分类, 并将Silva数据库(SSU128)与70% 的阈值进行比较。Alpha多样性指数(Shannon、Simpson、Chao1、ACE、coverage)分析采用Mothur软件对扩增子基因序列基于OTU进行。基于bray_curtis距离算法对所有样本进行聚类分析, 使用R软件中的ggplot2包来降维进行主成分分析(PCoA), 使用函数anosim进行相似性分析(Anosim), 运用Student’s t双尾检验两组细菌群落分度差异的物种。论文中的数据以平均值±标准差的形式展示。
1.7 thallusin与浒苔共培养实验thallusin获自日本德岛文理大学Matsuo[10], 浓度为1 000 fmol/mL。通过以往研究可知, thallusin对Gayralia oxyspermum的有效浓度为1 fg/mL~ 1 ag/mL[10, 17], 设置5个不同浓度梯度的thallusin与抗生素联合处理之后形态发生异常的3 mm浒苔培养。浓度从10 pmol/L开始, 以10倍稀释, 共分为5组。经过20 d培养, 筛选出促进浒苔藻体长度的最佳浓度。添加该浓度thallusin的培养基作为T组, 不添加抗生素和thallusin的培养基作为B组, 继续添加抗生素的培养基作为K组。抗生素联合处理之后形态发生异常的3 mm浒苔放置在3组培养基中培养25 d, 每组重复3个样本, 观察浒苔藻体长度变化情况。
2 结果与分析 2.1 抗生素对浒苔生长抑制作用KH组和K组浒苔配子经过培养均能形成幼苗, 图 1a和图 1b分别为KH组和K组浒苔配子培养50 d后的幼苗生长状态, 可以看出, 抗生素处理显著抑制浒苔配子体幼苗生长, KH组小苗比K组分枝更长、更多。并且抗生素处理显著改变藻体细胞大小, KH组和K组藻体细胞直径分别约为12 μm和8 μm(图 1c、1d), 可见KH组细胞明显大于K组, K组浒苔分枝瘦弱, 且大部分分枝只由1列细胞组成(图 1d)。
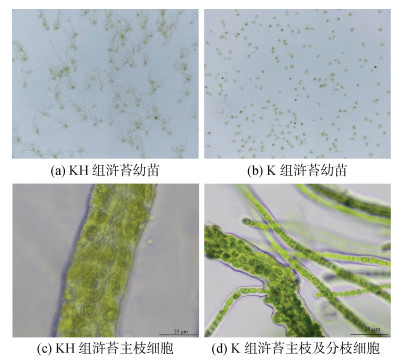 |
| 图 1 KH组和K组浒苔配子体幼苗 Fig. 1 U. prolifera gametophyte seedlings in the KH and K groups |
图 2a为KH组和K组浒苔培养25 d藻体长度变化, 可见KH组藻体在10 d后迅速生长, 而K组浒苔藻体长度几乎不发生变化, 随着培养时间的增加, 两组浒苔藻体长度差异越来越大。两组培养了25 d后, 其藻体形态如图 2b所示, 显示抗生素显著抑制浒苔生长, 浒苔生长减缓, 没有明显分枝, 整个藻体发育为愈伤组织样的表型。
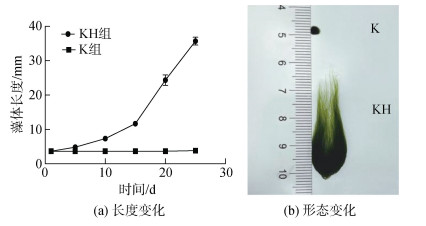 |
| 图 2 浒苔培养25 d藻体的长度及形态变化 Fig. 2 Changes in the algal body length and morphology of U. prolifera cultured for 25 days |
为了降低测序数据量的不同导致的差异, 首先对本次数据进行抽平。测序得到的reads平均长度为375 bp, KH组OTU平均数为149, K组OTU平均数为79。对获得的原始数据进行Alpha多样性分析(表 2), K组的Alpha多样性指数均低于KH组。Shannon指数和Simpson指数表明KH组细菌群落多样性略大于K组, ACE指数和Chao指数表明KH组细菌群落丰富度大于K组(P < 0.05)。Coverage指数指出样本数据覆盖率为99%, 测序结果准确率高。所有结果表明, 在抗生素联合处理浒苔共附生细菌的多样性比对照组降低。
| 样品名称 | 香农指数 | 辛普森指数 | ACE指数 | Chao指数 | Coverage指数 |
| K | 1.75±0.04 | 0.22±0.01 | 61.69±8.64 | 59.94±5.15 | 0.99±0 |
| KH | 1.97±0.34 | 0.25±0.07 | 111.77±20.75 | 109.72±25.73 | 0.99±0 |
为了解不同组别样本之间的相似性或差异性, 通过样本群落层级聚类(图 3a)和主坐标分析(图 3b)进行探索。根据本次实验样本层级聚类分析结果, 可以将K组和KH组浒苔共附生细菌分成两大类, 结果表明添加抗生素之后浒苔的共附生细菌群落组成与对照组相比有明显不同, 主成分分析(PCoA)也证明了这一结论。为了进一步探究分组是否有意义, 基于Bray-Curtis相异度进行ANOSIM相似性分析(图 3c)。结果表明组间差异大于组内差异, P < 0.05, 说明分组有意义, 也说明在KH组中和K组中存在不同的细菌群落。
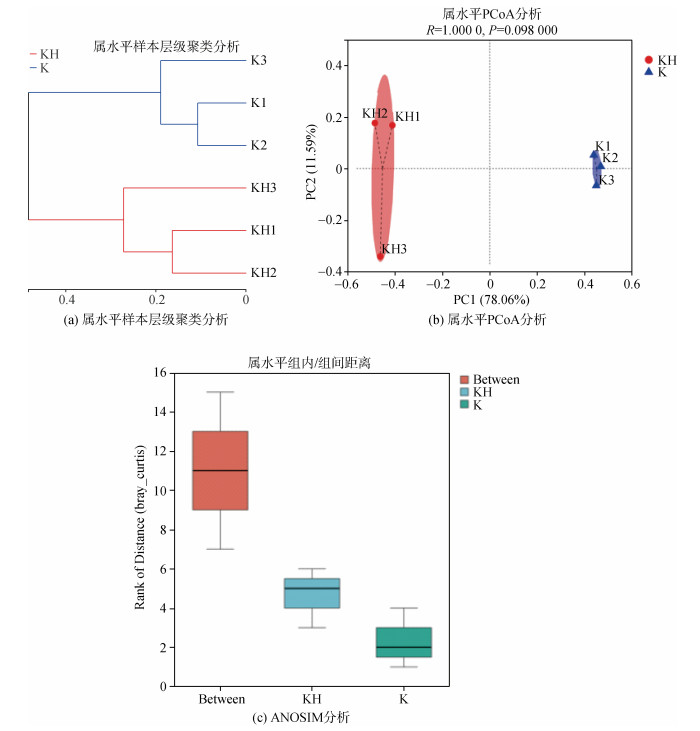 |
| 图 3 Beta多样性分析 Fig. 3 Beta diversity analysis |
在门分类水平上(图 4a), KH组细菌占比依次为变形菌门(Proteobacteria)47.2%、拟杆菌门(Bacteroidota) 49.8%、放线菌门(Actinobacteriota)2.7%和一些其他细菌。K组细菌占比依次为变形菌门90.6%、蓝细菌门(Cyanobacteria)2.5% 和一些其他细菌。KH组变形菌门和拟杆菌门占比含量高, K组变形菌门占比高, 表明在抗生素的作用下对拟杆菌门的杀伤效果明显。
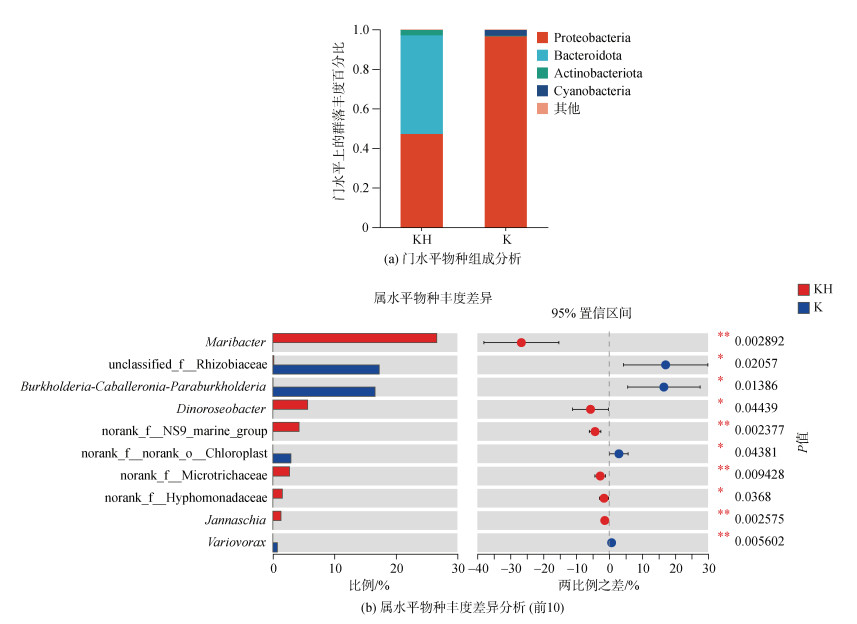 |
| 图 4 门水平物种组成和属水平物种丰度差异分析 Fig. 4 Phylum-level community composition analysis and genus-level differences in species abundance |
图 4b显示了属水平K组和KH组细菌群落中表现出的丰度差异前十的物种。Maribacter在两组中丰度差异最大, 大量存在于KH组中。其余有显著差异(P < 0.05)的物种分别是在K组有高丰度的根瘤菌属Burkholderia-Caballeronia-Paraburkholderia以及在KH组有高丰度的Dinoroseobacter和NS9_marine_group。
2.5 thallusin促进浒苔藻体长度增加为了探究thallusin对浒苔促进作用的最佳浓度, 进行浓度梯度实验(图 5)。通过20 d的培养发现, 在1 pmol/L的浓度下, 浒苔分枝数量最多, 平均藻体长度最长。
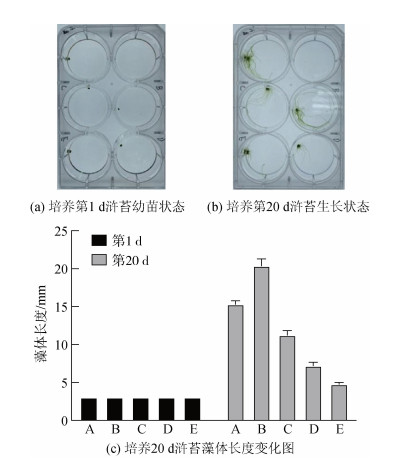 |
| 图 5 添加thallusin浒苔藻体长度变化情况 Fig. 5 Algal body length of U. prolifera with thallusin 注: A−E浓度分别为10 pmol/L、1 pmol/L、1×10−1 pmol/L、1×10−2 pmol/L、1×10−3 pmol/L |
使用含1 pmol/L thallusin的VSE培养基培养愈伤组织样表型的浒苔作为T组, 与B组、K组浒苔共同培养25 d后的生长变化如图 6a所示。第10 d开始, B组和T组藻体长度大于K组, 第15 d开始T组藻体长度大于B组, 随着培养时间的增加, 三组浒苔藻体长度差异更大。25 d后形态如图 6b所示, K组浒苔藻体形态基本不变, B组、T组浒苔发育为典型形态, T组浒苔藻体长度大于B组。
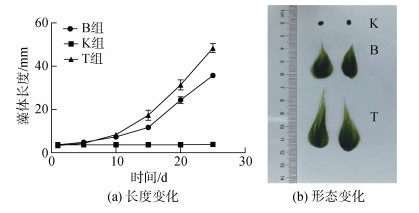 |
| 图 6 培养25 d浒苔藻体长度及形态变化 Fig. 6 Changes in the body length and morphology of algae cultured for 25 days |
微生物在大型藻类的生长发育中发挥着重要作用。据报道, 细菌可以直接调节Ulva sp.物种的生长和形态发生[14, 30]。本研究主要是为了探究浒苔(U. prolifera)共附生细菌对浒苔形态发育的影响, 利用青霉素G、硫酸新霉素和多黏菌素B三种抗生素联合处理浒苔配子, 发现浒苔幼苗无法形成典型形态, 浒苔藻体共附生细菌多样性发生改变, 为探索影响浒苔生长发育的关键细菌提供了新的证据。
从实验结果可知, 浒苔配子暴露在抗生素环境中, 可以形成幼苗但无法形成典型藻体形态(图 1), 而K组浒苔与典型的浒苔形态不同, 藻体呈现为类似于愈伤组织状的表型, 没有明显的假根细胞, 浒苔分枝短而卷曲, 整体缩为一小团, 且颜色很深, 与王瑜[16]和Marshall等[31]构建的无菌浒苔和缘管浒苔形态基本一致。抗生素除了会抑制浒苔的生长, Lestonde等[32]用不同抗生素对U. lactuca做了实验, 发现磺胺噻唑对U. lactuca生长也有抑制作用。同样的, 本次实验结果发现, 利用抗生素培养的浒苔藻体生长速度发生显著改变(图 2), 抗生素可以显著抑制浒苔生长, 推测抗生素抑制某些细菌生长, 导致浒苔无法形成典型的形态, 一旦停止添加抗生素, 细菌生长不受抗生素的抑制, 细菌丰度得到恢复, 故浒苔可恢复正常形态。但当抗生素浓度过高, 也会对浒苔细胞本身造成伤害, 过高浓度的抗生素会对浒苔游孢子和小苗产生致死作用[33], 本实验浒苔配子仍然存活, 并可以发育出幼苗, 但是无法形成典型形态, 所以推测抗生素是通过抑制细菌从而导致形态发生变化。
Burke等[34]对与U. australis共附生细菌群落进行宏基因组测序, 发现其细菌多样性组成为变形菌门(64.0 %)、拟杆菌门(27.6 %)和浮霉菌门(3.4 %), 与本次实验结果趋于一致, 也进一步说明了大型藻类共附生细菌群落在较高的分类等级上(如门、纲)几乎没有区别[35]。为了筛选影响浒苔形态发育的关键细菌, 从属水平比较分析不同组别的细菌丰度差异。丰度差异最大的细菌属为Maribacter, 在KH组含量显著高于K组。已知形态异常的无菌U. mutabilis与Roseovarius sp. (MS2)和Maribacte sp. (MS6)共培养, 两种细菌会产生各种刺激性化学介质完全恢复U. mutabilis形态[24], 本实验K组浒苔同样缺乏Maribacter, 这或许是浒苔藻体形态异常的主要原因。根据金柘的研究, 将几种细菌单独添加到缺乏典型浒苔形态的藻体中, 有4种细菌对浒苔形态恢复有促进作用, 这4种细菌在属水平上分类属于Roseobacter、Marivita、Bacillus sp.、Maribacter[36], 该研究也表明Maribacter对于浒苔形态发育有影响。本实验未检测到Marivita sp. 和Bacillus sp., 可能是因为所用的浒苔经过多年实验室环境下纯化培养, 部分细菌已经消失。黄杆菌属和噬甲基菌属是本次实验中除了Maribacter外丰度差异较大的两个细菌属, 一方面说明三种广谱抗生素可能对黄杆菌属和噬甲基菌属的细菌杀伤性强, 另一方面黄杆菌属和噬甲基菌属可能对浒苔的生长和形态发育有着很强的促进作用, 需要进一步从浒苔共附生菌中分离并进行藻菌共培养来研究对浒苔的影响, 所以去除藻体表面外生菌来研究细菌对藻类的影响就显得尤为重要[20]。
thallusin是由Maribacter spp. 释放的细菌半萜烯类化合物, 在不同的Maribacter培养基中均检测到thallusin的存在[24], 是目前唯一已知的藻类形态发育促进因子[23], 经过测序Maribacter在抗生素作用下丰度显著降低, 所以推测抗生素可能抑制Maribacter的丰度, 导致thallusin含量过低, 进而影响浒苔形态的发育。通过thallusin与浒苔的共培养实验, 发现thallusin对于浒苔的生长具有显著促进作用, 佐证了Maribacter对浒苔形态发育的促进作用。这也表明细菌对藻类形态的影响不仅可以通过直接接触, 还可以通过分泌化合物来介导。
4 结论利用3种抗生素联合处理浒苔配子, 发现浒苔藻体形态在发育过程中发生明显的变化。在抗生素的作用下, 浒苔细胞直径变小, 没有明显的假根细胞和主枝, 藻体生长速度减慢, 由未分化的细胞堆积形成类似于愈伤组织样的表型。为了研究抗生素作用导致浒苔形态发生变化的关键细菌, 利用16S rDNA高通量测序分析3种抗生素联合对浒苔共附生细菌的影响。通过16S rDNA测序结果表明, Maribacter是抗生素组中显著减少的一种细菌属, 共培养实验证明Maribacter spp.分泌的化合物thallusin可以使形态异常的浒苔恢复典型形态, 促进浒苔藻体生长更快, 推测抗生素诱导浒苔形态变化可能是因为Maribacter含量低, 无法对浒苔的生长起到促进作用。综上所述, 浒苔共附生菌对浒苔的生长和形态发育具有促进作用, 而在三种抗生素联合处理胁迫条件下会导致相应的共附生菌丰度减少, 从而影响浒苔的生长和形态发育。
| [1] |
CHRISTIE-OLEZA J A, SOUSONI D, LLOYD M, et al. Nutrient recycling facilitates long-term stability of marine microbial phototroph-heterotroph interactions[J]. Nature Microbiology, 2017, 2: 17100. DOI:10.1038/nmicrobiol.2017.100 |
| [2] |
MURRAY R E, COOKSEY K E, PRISCU J C. Stimulation of bacterial DNA synthesis by algal exudates in attached algal-bacterial consortia[J]. Applied & Environmental Microbiology, 1986, 52(5): 1177-1182. |
| [3] |
JIANG L J, YANG L Y, XIAO L, et al. Quantitative studies on phosphorus transference occuring between Microcystis aeruginosa and its attached bacterium (Pseudomonas sp.)[J]. Hydrobiologia, 2007, 581: 161-165. DOI:10.1007/s10750-006-0518-0 |
| [4] |
STEINBERG P D, DE NYS R, KJELLEBERG S. Chemical cues for surface colonization[J]. Journal of Chemical Ecology, 2002, 28(10): 1935-1951. DOI:10.1023/A:1020789625989 |
| [5] |
STAUFENBERGER T, THIEL V, WIESE J, et al. Phylogenetic analysis of bacteria associated with Laminaria saccharina[J]. FEMS Microbiology Ecology, 2010, 64(1): 65-77. |
| [6] |
CROFT M T, WARREN M J, SMITH A G. Algae need their vitamins[J]. Eukaryotic Cell, 2006, 5(8): 1175-1183. DOI:10.1128/EC.00097-06 |
| [7] |
FUENTES J L, GARBAYO I, CUARESMA M, et al. Impact of microalgae-bacteria interactions on the production of algal biomass and associated compounds[J]. Marine Drugs, 2016, 14(5): 100. DOI:10.3390/md14050100 |
| [8] |
PEDERSEN M. The demand for iodine and bromine of three marine brown algae grown in bacteria‐free cultures[J]. Physiologia Plantarum, 1969, 22(4): 680-685. DOI:10.1111/j.1399-3054.1969.tb07423.x |
| [9] |
KINGMAN A R, MOORE J. Isolation, purification and quantitation of several growth regulating substances in Ascophyllum nodosum (Phaeophyta)[J]. Botanica Marina, 1982, 25(4): 149-153. |
| [10] |
MATSUO Y, IMAGAWA H, NISHIZAWA M, et al. Isolation of an algal morphogenesis inducer from a marine bacterium[J]. Science, 2005, 307(5715): 1598. DOI:10.1126/science.1105486 |
| [11] |
GHADERIARDAKANI F, COATES J C, WICHARD T. Bacteria-induced morphogenesis of Ulva intestinalis and Ulva mutabilis (Chlorophyta): a contribution to the lottery theory[J]. FEMS Microbiology Ecology, 2017, 93(8): fix094. |
| [12] |
WEISS A, COSTA R, WICHARD T. Morphogenesis of Ulva mutabilis (Chlorophyta) induced by Maribacter species(Bacteroidetes, Flavobacteriaceae)[J]. Botanica Marina, 2017, 60(2): 197-206. |
| [13] |
PROVASOLI L, PINTNER I J. Bacteria induced polymorphism in an axenic laboratory strain of Ulva lactuca (Chlorophyceae)[J]. Journal of Phycology, 2010, 16(2): 196-201. |
| [14] |
SPOERNER M, WICHARD T, BACHHUBER T, et al. Growth and thallus morphogenesis of Ulva mutabilis (Chlorophyta)depends on a combination of two bacterial species excreting regulatory factors[J]. Journal of Phycology, 2012, 48(6): 1433-1447. DOI:10.1111/j.1529-8817.2012.01231.x |
| [15] |
ALSUFYANI T, WEISS A, WICHARD T. Time course exo-metabolomic profiling in the green marine macroalga Ulva (Chlorophyta) for identification of growth phase-dependent biomarkers[J]. Marine Drugs, 2017, 15(1): 14. DOI:10.3390/md15010014 |
| [16] |
王瑜. 黄海绿潮浒苔生长发育的分子机理研究[D]. 青岛: 山东大学(青岛), 2020. WANG Yu. Molecular mechanism of growth and development of the green tides forming alga Ulva prolifera in the Yellow Sea[D]. Qingdao: Shandong University (Qingdao), 2020. |
| [17] |
WICHARD T. Exploring bacteria-induced growth and morphogenesis in the green macroalga order Ulvales (Chlorophyta)[J]. Frontiers in Plant Science, 2015, 6: 00086. |
| [18] |
DELBRIDGE L, COULBURN J, FAGERBERG W, et al. Community profiles of bacterial endosymbionts in four species of Caulerpa[J]. Symbiosis, 2004, 37(1/3): 335-344. |
| [19] |
FU H H, JIANG P, ZHAO J, et al. Comparative genomics of Pseudomonas sp. strain SI-3 associated with macroalga Ulva prolifera, the causative species for green tide in the Yellow Sea[J]. Frontiers in Microbiology, 2018, 9: 01458. DOI:10.3389/fmicb.2018.01458 |
| [20] |
LIU X J, ZHAO J, JIANG P. Easy removal of epiphytic bacteria on Ulva (Ulvophyceae, Chlorophyta) by vortex with silica sands[J]. Microorganisms, 2022, 10(2): 476. DOI:10.3390/microorganisms10020476 |
| [21] |
MEI X Y, WU C H, ZHAO J, et al. Community structure of bacteria associated with drifting Sargassum horneri, the causative species of golden tide in the Yellow Sea[J]. Frontiers in Microbiology, 2019, 10: 01192. DOI:10.3389/fmicb.2019.01192 |
| [22] |
CROFT M T, LAWRENCE A D, RAUX-DEERY E, et al. Algae acquire vitamin B-12 through a symbiotic relationship with bacteria[J]. Nature, 2005, 438(7064): 90-93. DOI:10.1038/nature04056 |
| [23] |
WICHARD T, CHARRIER B, MINEUR F, et al. The green seaweed Ulva: a model system to study morphogenesis[J]. Frontiers in Plant Science, 2015, 6(72): 00072. |
| [24] |
TAGHREED A, GIANMARIA C, MICHAEL D, et al. Macroalgal-bacterial interactions: Identification and role of thallusin in morphogenesis of the seaweed Ulva (Chlorophyta)[J]. Journal of Experimental Botany, 2020, 71(11): 3340-3349. DOI:10.1093/jxb/eraa066 |
| [25] |
KESSLER R W, WEISS A, KUEGLER S, et al. Macroalgal–bacterial interactions: Role of dimethylsulfoniopropionate in microbial gardening by Ulva (Chlorophyta)[J]. Molecular Ecology, 2017, 27(8): 1808-1819. |
| [26] |
FUKUI Y, ABE M, KOBAYASHI M, et al. Isolation of Hyphomonas strains that induce normal morphogenesis in protoplasts of the marine red alga Pyropia yezoensis[J]. Microbial Ecology, 2014, 68(3): 556-566. DOI:10.1007/s00248-014-0423-4 |
| [27] |
周玲洁, 何培民, 黄希文, 等. 一种浒苔的无菌化培养方法, CN201310666901. X[P/OL]. [2013-20131210]. ZHOU Lingjie, HE Peimin, HUANG Xiwen, et al. A sterile culture method of Ulva prolifera, CN201310666901. X [P/OL]. [2013-20131210]. |
| [28] |
OTT F D. Synthetic media and techniques for the xenic cultivation of marine algae and flagellate[J]. Virginia J Sci, 1965, 16: 205-218. |
| [29] |
CHEN R, CAI C, JIANG T, et al. Growth and metagenomics analysis of Ulva prolifera after antibiotic treatment[J]. International Journal of Agriculture and Biology, 2019, 21(5): 1031-1035. |
| [30] |
BRADLEY P M. Plant hormones do have a role in controlling growth and development of algae[J]. Journal of Phycology, 2004, 27(3): 317-321. |
| [31] |
MARSHALL K, JOINT I, CALLOW M E, et al. Effect of marine bacterial isolates on the growth and morphology of axenic plantlets of the green alga Ulva linza[J]. Microbial Ecology, 2006, 52(2): 302-310. DOI:10.1007/s00248-006-9060-x |
| [32] |
LESTON S, NUNES M, VIEGAS I, et al. The influence of sulfathiazole on the macroalgae Ulva lactuca[J]. Chemosphere, 2014, 100: 105-110. DOI:10.1016/j.chemosphere.2013.12.038 |
| [33] |
叶静, 张喆, 李富超, 等. 大型绿藻浒苔转化表达系统选择标记的筛选[J]. 生物技术通报, 2006(3): 63-67. YE Jing, ZHANG Zhe, LI Fuchao, et al. Screen of selectable marker for transformation and expression system of green seaweed Enteromorpha Clathrata[J]. Biotechnology Bulletin, 2006(3): 63-67. |
| [34] |
BURKE C, THOMAS T, LEWIS M, et al. Composition, uniqueness and variability of the epiphytic bacterial community of the green alga Ulva australis[J]. ISME Journal, 2011, 5(4): 590-600. DOI:10.1038/ismej.2010.164 |
| [35] |
HOLLANTS J, LELIAERT F, CLERCK O D, et al. What we can learn from sushi: a review on seaweed-bacterial associations[J]. FEMS Microbiology Ecology, 2012, 83(1): 1-16. |
| [36] |
金柘. 绿潮浒苔共附生菌的多样性以及重要共附生菌影响其形态发育的研究[D]. 济南: 山东师范大学, 2018. JIN Zhe. Study on the diversity of the Ulva prolifera epiphytic bacteria in green tide and the effect of important Co-bacteria on the morphological development[D]. Jinan: Shandong Normal University, 2018. |
 2023, Vol. 47
2023, Vol. 47


