
文章信息
- 宁子君, 刘玉萍, 高天翔, 蒋日进, 杨天燕. 2023.
- NING Zi-jun, LIU Yu-ping, GAO Tian-xiang, JIANG Ri-jin, YANG Tian-yan. 2023.
- 东海几种鳗鲡目鱼类DNA条形码研究
- DNA barcoding analysis of some Anguilliformes fishes in the East China Sea
- 海洋科学, 47(4): 126-136
- Marine Sciences, 47(4): 126-136.
- http://dx.doi.org/10.11759/hykx20220607002
-
文章历史
- 收稿日期:2022-06-07
- 修回日期:2022-10-26
2. 浙江省海洋水产研究所, 浙江 舟山 316021
2. Zhejiang Marine Fisheries Research Institute, Zhoushan 316021, China
鳗鲡目(Anguilliformes)在分类学上隶属于硬骨鱼纲(Osteichthyes)、辐鳍亚纲(Actinopterygii)、鳗鲡总目(Anguillomorpha), 该类群的鱼类繁多, 除地球两极和近两极寒冷海域, 在大西洋、印度洋、太平洋等均有分布, 有一些种类可以进入淡水[1]。全球共有鳗鲡目鱼类15科141属约791种, 中国有12科55属135种, 主要分布在东海以南的亚热带和热带海域, 少数种类可分布至黄渤海[1, 2]。由于该类群种类众多且近缘种形态相似度较高, 目前国内外在鳗鲡目大部分类群的系统分类与命名上一直存在许多争议。据《东海鱼类志》记载, 中国东海有鳗鲡目鱼类9科17属18种[3], 近年来该海域陆续有新种被发现[4-6], 在丰富该类群物种多样性的同时, 也为东海鳗鲡目鱼类分类学研究和鉴定带来了新的困难和挑战。
生物的鉴定通常基于传统形态学方法, 但该方法对于损坏的样品外部形态特征会发生变化从而难以识别, 过分依赖鉴定者的个人经验和观察手段, 而且不同研究者的鉴定结论时常不一致, 甚至出现误鉴[7-9]。DNA条形码技术(DNA barcoding)指的是通过对一个或多个标准目的基因的DNA序列进行分析从而进行物种鉴定的技术[10]。作为一种新兴的技术, DNA条形码在物种分类识别上有着快捷简单的优点, 结合已公布的序列数据资料, 可对形态上难以辨认或处于未成熟阶段的物种进行鉴定, 从而增加物种鉴定的加准确性和可信度[11]。近年来, 随着生物分类和分子生物学研究的深入, 越来越多的学者意识到DNA条码(DNA barcoding)可作为传统分类学的有效补充[12-15]。线粒体细胞色素C氧化酶亚基I(Cytochrome C oxidase subumit I, COI)基因是动物DNA条形码研究中使用最为广泛的标准基因之一, 与线粒体上其他蛋白质编码基因相比, 该基因具有进化速度较快、序列长度适中且易被通用引物扩增等特点, 已成为物种鉴定和系统发育研究最有利的工具[16]。
本研究以分布于舟山海域的前肛鳗(Dysomma anguillaris)、短尾蛇鳗(Ophichthus brevicaudatus)、艾氏蛇鳗(Ophichthus evermanni)、海鳗(Muraenesox cinereus)、黑尾吻鳗(Rhynchoconger ectenurus)、微鳍新鳗(Neenchelys parvipectoralis)、大头蚓鳗(Moringua macrocephalus)、梅氏美体鳗(Ariosoma meeki)和星康吉鳗(Conger myriaster)共6科8属9种鳗鲡目鱼类作为研究对象, 通过PCR扩增和测序的方法获得线粒体COI基因片段序列, 计算Kimura双参数模型遗传距离并采用最大似然法构建系统发生树, 探讨COI基因作为DNA条码标准基因对于鳗鲡目不同物种间进行鉴定的有效性, 以期对该类群鱼类分类研究和渔业管理提供一定的理论依据。
1 材料与方法 1.1 实验材料实验所用样品于2021年8—2021年11月间采自浙江舟山和福建厦门近海, 样品冷冻带回实验室进行初步形态鉴定并拍照后浸泡于95%酒精中固定(图 1)。所有鱼类样品均保存在浙江海洋大学渔业生态与生物多样性实验室。
 |
| 图 1 本研究中采集的9种鳗鲡目鱼类照片 Fig. 1 Photographs of the nine species of Anguilliformes collected and assessed in this study |
剪取样品背部肌肉, 采用苯酚–氯仿法提取DNA[17]。扩增线粒体DNA COI基因片段所用引物序列如表 1所示[18]。PCR反应总体积25 μL, 其中10× DNA buffer 2 μL、正反向引物各1 μL、DNTPs 2 μL、ExTaq酶0.15 μL、灭菌水17.5 μL、ExTaq酶0.15 μL以及DNA模板1 μL。反应条件为: 94℃预变性5 min; 94℃变性45 s, 51℃退火30 s, 72℃延伸45 s, 共35个循环; 最后72℃延伸10 min。扩增产物经电泳检测后, 送生工生物工程(上海)股份有限公司进行双向测序。
| 引物名称 | 5′→3′ | 适用物种 |
| FishF1 | TCAACCAACCACAAAGACATTGGCAC | 前肛鳗、短尾蛇鳗、艾氏蛇鳗、大头蚓鳗、黑尾吻鳗、梅氏美体鳗、星康吉鳗、微鳍新鳗 |
| FishR1 | TAGACTTCTGGGTGGCCAAAGAATCA | |
| FishF2 | TCGACTAATCATAAAGATATCGGCAC | 海鳗 |
| FishR2 | ACTTCAGGGTGACCGAAGAATCAGAA |
将测序得到的mtDNA COI基因片段用DNAStar软件包中的Seqman软件进行序列拼接和比对, 并辅以人工校正[19]; 采用NCBI在线BLAST(basic local alignment search tool)工具选取匹配率超过98%的序列用于物种鉴定[20]; 使用MEGA-X软件计算碱基组成和Kimura 2-parameter遗传距离[21]。下载GenBank数据库中2科5属5种鳗鲡目鱼类16条同源基因序列(均为不同的单倍型), 与本研究获得的6科8属9种鱼类56条COI基因序列进行比较分析, 并以光海鳝Muraena argus(GenBank登录号: HQ010100)和细点海鳝M. augusti(GenBank登录号: EU921740)作为外群[2, 22-25], 运用MEGA-X软件筛选模型, 并基于最大似然法(maximum likelihood method, ML)构建系统树分析不同鱼类间的亲缘关系[26], 对所得系统树进行1 000次重复自展法检验各分支节点的支持率[27]。分析所用物种的相关信息如表 2所示。
| 科名 | 属名 | 种名 | 样本数 | 单倍型编号 | GenBank序列号 |
| 前肛鳗科 (Dysommidae) |
前肛鳗属(Dysomma) | 前肛鳗 (Dysomma anguillaris) |
15 | Q1-Q6 | OP703573- OP703578* |
| 蛇鳗科 (Ophichthyidae) |
蛇鳗属(Ophichthus) | 短尾蛇鳗 (Ophichthus brevicaudatus) |
11 | D1-D5 | OP703564- OP703568* |
| 艾氏蛇鳗 (Ophichthus evermanni) |
1 | X1 | OP703578* | ||
| 尖吻蛇鳗 (Ophichthus apicalis) |
4 | O1-O4 | KY472814、KX215176、KX215177、KY472815 | ||
| 须鳗属(Cirrhimuraena) | 中华须鳗 (Cirrhimuraena chinensis) |
1 | CC1 | KX215194 | |
| 豆齿鳗属(Pisodonophis) | 杂食豆齿鳗 (Pisodonophis boro) |
2 | P1、P2 | MK572488、MK572491 | |
| 海鳗科 (Muraenesocidae) |
海鳗属(Muraenesox) | 海鳗 (Muraenesox cinereus) |
6 | H1、H2 | OP703569、OP703570* |
| 新鳗科 (Neenchelyidae) |
新鳗属(Neenchelys) | 微鳍新鳗 (Neenchelys parvipectoralis) |
2 | C1、C2 | OP720914、OP720915* |
| 蚓鳗科 (Moringuidae) |
蚓鳗属(Moringua) | 大头蚓鳗 (Moringua macrocephalus) |
2 | XM1、XM2 | OP704124、OP704125* |
| 康吉鳗科 (Congridae) |
康吉鳗属(Conger) | 星康吉鳗(Conger myriaster) | 5 | XK1-XK3 | OP703580- OP703582* |
| 日本康吉鳗(Conger aponicus) | 2 | CJ1、CJ2 | EF607456、MK843774 | ||
| 美体鳗属(Ariosoma) | 梅氏美体鳗(Ariosoma meeki) | 7 | M1、M2 | OP703571、OP703572* | |
| 尾鳗属(Uroconger) | 尖尾鳗(Uroconger lepturus) | 7 | U1-U7 | EU595337、HQ564291、HQ564520、KX215174、KY026491、MK348181、MK348182 | |
| 吻鳗属(Rhynchoconger) | 黑尾吻鳗 (Rhynchoconger ectenurus) |
7 | B1-B4 | OP703560- OP703563* | |
| 海鳝科(Muraenidae) | 海鳝属(Muraena) | 光海鳝(Muraena argus) | 1 | HQ010100 | |
| 细点海鳝(Muraena augusti) | 1 | EU921740 | |||
| 注: *. 本研究 | |||||
前肛鳗: 体褐色, 腹部色较浅, 背鳍、臀鳍白色。吻尖锐, 上颌长于下颌。口大, 左右鳃孔分离。无前颌骨齿, 犁骨齿显著大于上颌齿。眼小, 退化, 埋于皮下, 位于口裂中央上方, 肛门显著前位。
短尾蛇鳗: 头部和体侧呈灰棕色, 腹部浅白色。吻短而钝。前鼻孔短管形, 位于吻侧面近吻尖处, 后鼻孔裂缝状, 位于眼前缘下方上唇边缘外侧。臀鳍起点紧接肛门后方, 胸鳍发达, 后缘圆形。无尾鳍, 尾端尖突。
艾氏蛇鳗: 体延长呈淡褐色, 背侧色较深且无明显条带, 体侧有20条左右通达背鳍的不规则的褐色云状斑, 颈部也可见褐色斑纹。背鳍始于胸鳍末端上方, 与臀鳍在尾端呈菱形。
海鳗: 体灰褐色, 腹部灰白色, 背、尾鳍边缘黑色, 胸鳍无色。侧线孔明显。躯干近圆柱状, 尾部较侧扁。头细长、吻较尖。前鼻孔短管状, 位于吻前端。
黑尾吻鳗: 体黑绿色, 尾部渐转深黑色。吻突出, 吻端圆钝。口中等大, 口裂超过眼前缘下方。下颌较上颌短。两颌齿小, 呈圆锥状。尾鞭状延长。
微鳍新鳗: 体淡褐色, 腹侧色稍浅。躯干部圆柱状, 较宽大。头短小, 尖锥状。吻短, 尖突。胸鳍甚小。背鳍起始于肛门前上方, 达胸鳍尖端后上方。尾鳍细小, 边缘黑色, 上下叶等长。
大头蚓鳗: 体色较浅, 腹部淡黄色, 躯干细长, 尾端侧扁。吻短钝, 头部较尖, 下颔稍长。背鳍起点在身体的远后方, 臀鳍起点位于肛门稍后方; 两者均不发达, 向后与尾端相连续。胸鳍极短小, 位于鳃孔之后。侧线孔不明显。
梅氏美体鳗: 体浅棕色, 腹侧白色。躯干部粗壮, 尾侧扁。头中等大, 吻圆钝。眼大, 吻长约等于眼径。口宽大, 口裂伸达眼中部后下方。齿小, 密列, 毛刷状。背鳍起始于胸鳍基上方。背鳍、臀鳍边缘黑色。眼后缘上、下侧各有一褐色长斑。
星康吉鳗: 体前部圆筒形, 尾部侧扁。头中等大, 锥状。吻部近平扁。舌宽阔, 前端游离, 两颌牙细。背鳍起始于胸鳍末端的正上方。侧线发达, 侧线孔有白色斑点, 侧线孔上方还有1列小白点。
2.2 COI基因片段序列分析PCR扩增产物经测序后, 通过拼接、人工校正和比对得到56条长度为633 bp的线粒体DNA COI基因序列片段, 符合条形码的长度条件。56条序列共定义了27个单倍型, 分布情况如下: 前肛鳗6个, 短尾蛇鳗5个, 黑尾吻鳗4个, 星康吉鳗3个, 海鳗、微鳍新鳗、大头蚓鳗和梅氏美体鳗各2个, 艾氏蛇鳗1个。
从GenBank数据库下载了16条同源单倍型序列如表 2所示。43条单倍型序列的4种碱基平均含量为: T为27.4 %, C为28.2%, A为25.8 %, G为18.6%, 平均A+T含量(53.2%)高于G+C含量(46.8%)。密码子3个位点的碱基组成如表 3所示, 从中可以看出: 密码子第一位点G含量显著偏低, 而A碱基含量最高, 为36.7%; 密码子第二位点A、C碱基含量大致相同, T碱基含量相对较低, 为17.0%; 第三位点的各碱基含量差别相对较大, T碱基含量最高, 为41%。第三密码子位点A+T平均含量(56.2%)高于第一密码子位点(53.2%)和第二密码子位点(42.5%)。
| 碱基 | 总频率(%) | 密码子第1位点(%) | 密码子第2位点(%) | 密码子第3位点(%) |
| T | 27.4 | 24.0 | 17.0 | 41.0 |
| C | 28.2 | 30.0 | 25.6 | 28.9 |
| A | 25.8 | 36.7 | 25.5 | 15.2 |
| G | 18.6 | 9.4 | 31.8 | 14.7 |
利用MEGA-X软件基于Kimura 2-parameter模型计算不同分类阶元间的遗传距离。不同种间遗传距离范围在0.043 7~0.287 0, 最大值出现在黑尾吻鳗和梅氏美体鳗之间, 最小值出现在尖吻蛇鳗和中华须鳗之间, 平均种间遗传距离为0.218 8(表 4); 不同属间遗传距离最小值出现在蛇鳗属和须鳗属之间, 其遗传距离为0.117 3, 最大值出现在吻鳗属和美体鳗属之间, 其遗传距离为0.287 0, 平均为0.225 0(表 5); 不同科间遗传距离最小值存在于新鳗科和蛇鳗科之间, 为0.193 3, 最大遗传距离在蚓鳗科和海鳗科之间, 为0.270 7, 平均为0.232 7(表 6)。
| 尖尾鳗 | 海鳗 | 中华须鳗 | 尖吻蛇鳗 | 梅氏美体鳗 | 星康吉鳗 | 黑尾吻鳗 | 艾氏蛇鳗 | 日本康吉鳗 | 杂食豆齿鳗 | 前肛鳗 | 短尾蛇鳗 | 微鳍新鳗 | 大头蚓鳗 | |
| 尖尾鳗 | ||||||||||||||
| 海鳗 | 0.238 5 | |||||||||||||
| 中华须鳗 | 0.192 9 | 0.232 1 | ||||||||||||
| 尖吻蛇鳗 | 0.185 2 | 0.217 1 | 0.043 7 | |||||||||||
| 梅氏美体鳗 | 0.241 4 | 0.274 9 | 0.223 8 | 0.216 2 | ||||||||||
| 星康吉鳗 | 0.217 5 | 0.229 4 | 0.219 2 | 0.215 5 | 0.247 5 | |||||||||
| 黑尾吻鳗 | 0.227 8 | 0.237 1 | 0.210 7 | 0.203 6 | 0.287 0 | 0.227 9 | ||||||||
| 艾氏蛇鳗 | 0.189 3 | 0.236 1 | 0.170 3 | 0.177 2 | 0.248 0 | 0.227 3 | 0.220 9 | |||||||
| 日本康吉鳗 | 0.228 1 | 0.120 4 | 0.216 9 | 0.209 3 | 0.260 0 | 0.212 7 | 0.216 6 | 0.253 8 | ||||||
| 杂食豆齿鳗 | 0.139 9 | 0.233 8 | 0.176 3 | 0.167 1 | 0.258 3 | 0.199 8 | 0.234 2 | 0.171 5 | 0.230 3 | |||||
| 前肛鳗 | 0.232 8 | 0.260 8 | 0.217 3 | 0.212 5 | 0.238 2 | 0.239 3 | 0.196 7 | 0.212 1 | 0.250 3 | 0.231 9 | ||||
| 短尾蛇鳗 | 0.183 3 | 0.229 5 | 0.165 6 | 0.171 3 | 0.246 2 | 0.240 3 | 0.227 7 | 0.123 8 | 0.238 4 | 0.149 5 | 0.206 8 | |||
| 微鳍新鳗 | 0.215 9 | 0.248 7 | 0.180 1 | 0.176 1 | 0.286 9 | 0.237 9 | 0.219 0 | 0.219 1 | 0.255 0 | 0.184 2 | 0.209 1 | 0.208 1 | ||
| 大头蚓鳗 | 0.263 0 | 0.270 7 | 0.218 5 | 0.216 4 | 0.240 0 | 0.251 6 | 0.248 9 | 0.250 9 | 0.279 2 | 0.252 7 | 0.220 1 | 0.240 9 | 0.260 3 |
| 尾鳗属 | 海鳗属 | 须鳗属 | 蛇鳗属 | 美体鳗属 | 康吉鳗属 | 吻鳗属 | 豆齿鳗属 | 前肛鳗属 | 新鳗属 | 蚓鳗属 | |
| 尾鳗属 | |||||||||||
| 海鳗属 | 0.238 5 | ||||||||||
| 须鳗属 | 0.192 9 | 0.232 1 | |||||||||
| 蛇鳗属 | 0.184 6 | 0.225 2 | 0.117 3 | ||||||||
| 美体鳗属 | 0.241 4 | 0.274 9 | 0.223 8 | 0.234 4 | |||||||
| 康吉鳗属 | 0.221 8 | 0.185 8 | 0.218 3 | 0.228 7 | 0.252 5 | ||||||
| 吻鳗属 | 0.227 8 | 0.237 1 | 0.210 7 | 0.217 4 | 0.287 0 | 0.223 4 | |||||
| 豆齿鳗属 | 0.139 9 | 0.233 8 | 0.176 3 | 0.158 7 | 0.258 3 | 0.212 0 | 0.234 2 | ||||
| 前肛鳗属 | 0.232 8 | 0.260 8 | 0.217 3 | 0.209 6 | 0.238 2 | 0.243 7 | 0.196 7 | 0.231 9 | |||
| 新鳗属 | 0.215 9 | 0.248 7 | 0.180 1 | 0.196 4 | 0.286 9 | 0.244 7 | 0.219 0 | 0.184 2 | 0.209 1 | ||
| 蚓鳗属 | 0.263 0 | 0.270 7 | 0.218 5 | 0.232 1 | 0.240 0 | 0.262 6 | 0.248 9 | 0.252 7 | 0.220 1 | 0.260 3 |
| 康吉鳗科 | 海鳗科 | 蛇鳗科 | 前肛鳗科 | 新鳗科 | 蚓鳗科 | |
| 康吉鳗科 | ||||||
| 海鳗科 | 0.227 6 | |||||
| 蛇鳗科 | 0.207 1 | 0.227 0 | ||||
| 前肛鳗科 | 0.228 4 | 0.260 8 | 0.213 6 | |||
| 新鳗科 | 0.232 5 | 0.248 7 | 0.193 3 | 0.209 1 | ||
| 蚓鳗科 | 0.257 2 | 0.270 7 | 0.234 2 | 0.220 1 | 0.260 3 |
根据1978年由SCHWARZ[28]提出的贝叶斯信息准则(Bayesian information criterion, BIC), 评分值最低的模型通常可作为最优替代模型。使用MEGA-X软件筛选最优核苷酸替代模型为HKY+G+I, 以单倍型序列基于最大似然法构建的系统关系树如图 2所示, 隶属于蛇鳗科的5种鱼类首先聚类, 其中混杂了来自于GenBank的4条尖尾鳗序列(MK348181、MK348182、KY026491、EU595337); 梅氏美体鳗和大头蚓鳗相聚之后再与前肛鳗聚为一支; 星康吉鳗、日本康吉鳗、黑尾吻鳗和海鳗聚到了一起; 而GenBank下载的另外3条尖尾鳗(KX215174、HQ564291、HQ564520)则聚在了系统进化树的最外层。聚类结果显示除蛇鳗科外, 同科不同属间的聚类效果不够分明, 有不同程度的混杂现象。
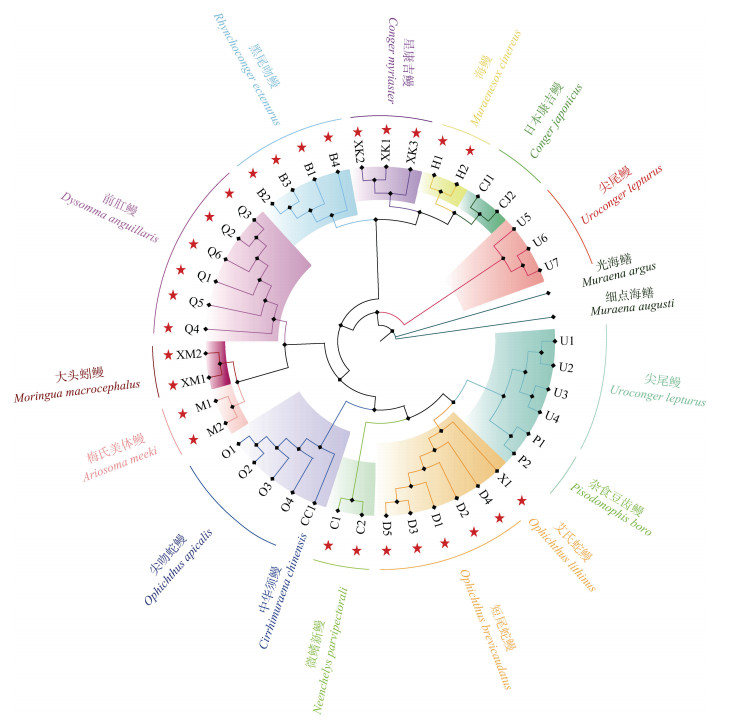 |
图 2 基于单倍型序列构建的鳗鲡目鱼类的ML系统树 Fig. 2 ML tree based on the haplotypes of Anguilliformes fishes  红色星号代表本研究采集的样本 红色星号代表本研究采集的样本 |
DNA条形码是可辅以物种鉴定的, 在不同种间差异明显的一段或几段DNA序列, 不同生物类群适用的DNA条形码存在一定差别[29-32]。其中, 线粒体COI基因已成为鱼类鉴定首选的DNA条形码, 目前被广泛应用于海洋鱼类的分类鉴定和遗传多样性分析[33-37]。在鳗鲡目鱼类的分类鉴定和系统发育研究中, STEIN等[38]首次利用线粒体COI基因, 对国际贸易中未能提供物种鉴定报告的欧洲鳗的非法交易行为进行了判定; 龚晓玲等[39]对鳗鲡属6种鱼类线粒体COI基因序列组成特点、分类有效性进行比较分析, 表明该基因可以对鳗鲡属鱼类进行有效的分子鉴定; 张稚兰等[40]以蛇鳗科鱼类为研究对象, 证实COI基因序列能够满足该类群分类研究的需要; 陈文炳等[41]则进一步采用多基因DNA条形码(COI、16S rRNA和Cyt b)的手段, 成功对6个鳗鱼物种进行了分子鉴定。上述研究表明, 线粒体COI基因可作为辅助性物种鉴定标记应用于鳗鲡目的某些科内种间分类, 但不同科间的适用性研究尚未见到相关报道。
本研究采用COI基因序列标记, 在外部形态学鉴定的基础上, 对采自我国东海的几种鳗鲡目不同科间的鱼类样本分类结果开展进一步分析探讨。通过统计得出6科11属14种鳗鲡目鱼类72条COI基因序列(43种单倍型)的平均GC含量为46.8%, 表现出一定的碱基组成偏向性, 这与脊椎动物COI基因碱基组成中普遍存在的AT含量高于GC含量的特征一致[42]。国内外学者在其他类群鱼类的研究中也发现这一现象, 例如姜郦轩等[37]分析了97条南极鱼类DNA条形码的碱基组成特征, 发现AT平均含量(52.7%)显著高GC平均含量(47.3%); 周建设等[43]对采集自西藏水系的260尾裂腹鱼类线粒体COI基因进行分析发现AT碱基偏倚明显; FADLI等[44]基于COI序列对144尾购买自印度尼西亚9个农贸市场的石斑鱼进行了分子鉴定, 统计得出平均碱基含量为A(24.15 %)、T(29.56 %)、C(28.14 %)、G(18.14 %), AT含量高于GC含量。WARD等[18]推测硬骨鱼类COI基因较高的GC含量可能与其密码子第3位点的GC偏倚有关。本研究中密码子第1位点的GC含量最低(39.4%), 而密码子第3位点的GC含量最高(57.4%), 符合这一普遍性规律。
种间K2P遗传距离的大小是进行物种鉴别的重要标准之一。WARD等[18]对澳大利亚207种鱼类的COI基因序列进行了统计分析, 得出平均种间遗传变异为9.93%, HUBERT等[45]比较了193种加拿大淡水鱼类COI基因序列, 发现种间平均遗传距离为8.30%。本研究基于Kimura双参数法计算鳗鲡目鱼类COI基因序列的平均种间遗传距离为0.218 8, 满足HEBERT等[16]推荐的物种鉴定最小种间遗传距离2%的阈值。不同物种的平均遗传距离间是否存在条形码间隙(barcoding gap)是衡量DNA条形码的另一个重要指标, 即序列的种内变异足够小且种间变异足够大(后者约为前者的10倍标准)[16, 46]。本研究所涉及的鳗鲡目鱼类种间遗传距离(0.218 8)约为种内遗传距离(0.003 2)的68倍, 能够形成一个明显的间隔区, 表明不同属种间的物种能够通过DNA条形码得到较好的区分。
本研究中, 鳗鲡目鱼类遗传距离整体上随着分类阶元的提高呈现出逐渐增大的趋势, 但增长速度减缓。ZHANG等[47]曾对日本海域分布的158种海洋鱼类(隶属于21目93科144属)的COI序列进行分析, 也发现了类似的规律。研究人员对229条序列统计得出种内个体间、属内种间、科内属间、目内科间、纲内目间平均遗传距离分别为0.3%、17.6%、18.5%、23.2%、24.6%, 除种内和种间遗传距离差距最大外, 其他阶元间遗传距离的增幅不大。有学者认为COI条形码序列仅适用于低分类阶元系统进化的分析, 而对科以上分类阶元的物种鉴定存在一定局限性[48, 49], 推测与高阶元间的遗传距离无法形成明显的条码间隔, 种内和种间存在交叉重叠有关。本研究中系统进化关系拓扑结构显示, 来自于GenBank数据库中的4尾尖尾鳗与蛇鳗科鱼类聚到了一起, 可能由于错误的物种鉴定导致。系统树中仅蛇鳗科种类能够得到较好的聚类, 证实了张稚兰等[40]对于COI基因在蛇鳗科鱼类鉴定具有适用性的研究结论, 而其他物种都没能形成相对独立的支系, 属间存在混合分布的现象。因此, 在鳗鲡目鱼类的分类研究中, 仅靠一个长度约为600 bp的COI基因片段进行系统发育分析可靠度不高。有学者采用其他DNA条形码如线粒体12S rRNA和16S rRNA序列对海鲢总目(Elopomorpha)鱼类的系统进化关系进行探讨, 初步证实了鳗鲡目为单系起源, 蛇鳗科和海鳗科互为姊妹群且处于较进化的位置, 而海鳝科和鳗鲡科则是较原始的类群, 但科下分类阶元的亲缘关系仍比较混乱[22-23]。
综上所述, 由于鳗鲡目鱼类种类和数量众多且分布范围广泛, 加之DNA条形码数据库尚不完整且存在错误序列等因素均会导致物种鉴定不够准确, 在一定程度上限制DNA条形码技术的应用。因此, 有必要采用来自不同分布地域、不同种群的样品单倍型序列信息以全面反应物种的种内差异[50], 同时借助高通量测序技术手段[51], 弥补传统形态学鉴定方法的局限和不足并丰富DNA条形码数据库, 以期为鳗鲡目的分类研究提供更加坚实的理论依据, 也为该类群渔业资源保护和可持续开发利用提供参考。
| [1] |
Nelson J S. Fishes of the world (Fourth Edition)[M]. New Jersey: Wiley, 2006.
|
| [2] |
张春光, 唐文乔, 张世义, 等. 中国动物志 硬骨鱼纲 鳗鲡目 背棘鱼目[M]. 北京: 科学出版社, 2010. ZHANG Chunguang, TANG Wenqiao, ZHANG Shiyi, et al. Fauna sinica, Osteichthyes, Anguilliformes, Notacanthiformes[M]. Beijing: Science Press, 2010. |
| [3] |
朱元鼎, 张春霖, 成庆泰. 东海鱼类志[M]. 北京: 科学出版社, 1963. ZHU Yuanding, ZHANG Chunlin, CHENG Qingtai. Fish fauna of the East China Sea[M]. Beijing: Science Press, 1963. |
| [4] |
朱元鼎, 伍汉霖, 金鑫波. 中国蛇鳗科和新鳗科的4新种[J]. 水产学报, 1981, 5(1): 21-27. ZHU Yuangding, WU Hanlin, JIN Xinbo. Four new species of the families Ophichthyidae and Neenchelidae[J]. Journal of Fisheries of China, 1981, 5(1): 21-27. |
| [5] |
唐文乔, 张春光. 中国蛇鳗科鱼类一新种(鱼纲: 鳗鲡目: 蛇鳗科)[J]. 动物分类学报, 2002, 27(4): 854-856. TANG Wenqiao, ZHANG Chunguang. One newspecies of the family Ophichthidae from East China Sea(Pisces: Anguilliformes)[J]. Acta Zootaxonomica Sinica, 2002, 27(4): 854-856. DOI:10.3969/j.issn.1000-0739.2002.04.029 |
| [6] |
TANG W Q, ZHANG C G. A new species of the genus Cirrhimuraena from the East China Sea(Pisces, Anguilliformes, Ophichthidae)[J]. Acta Zootaxonomica Sinica, 2003, 28(3): 551-553. |
| [7] |
KO H L, WANG Y T, CHIU T S, et al. Evaluating the accuracy of morphological identification of larval fishes by applying DNA barcoding[J]. PLOS ONE, 2013, 8(1): e53451. DOI:10.1371/journal.pone.0053451 |
| [8] |
PUNCHER G N, ALEMANY F, ARRIZABALAGA H, et al. Misidentification of bluefin tuna larvae: a call for caution and taxonomic reform[J]. Reviews in Fish Biolology and Fisheries, 2015, 25(3): 485-502. DOI:10.1007/s11160-015-9390-1 |
| [9] |
BECKER R A, SALES N G, SANTOS G M, et al. DNA barcoding and morphological identification of neotropical ichthyoplankton from the upper Paraná and São Francisco[J]. Journal of Fish Biology, 2015, 87(1): 159-168. DOI:10.1111/jfb.12707 |
| [10] |
HEBERT P D N, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270: 313-322. DOI:10.1098/rspb.2002.2218 |
| [11] |
彭居俐, 王绪桢, 何舜平. DNA条形码技术的研究进展及其应用[J]. 水生生物学报, 2008, 32(6): 916-919. PENG Juli, WANG Xuzhen, HE Shunping. The progress and application of DNA barcoding[J]. Acta Hydrobiologica Sinica, 2008, 32(6): 916-919. |
| [12] |
HAJIBABAEI M, SINGER G A C, HEBERT P D N, et al. DNA barcoding: how it complements taxonomy, molecular phylogenetics and population genetics[J]. Trends in Genetics, 2007, 23(4): 167-172. DOI:10.1016/j.tig.2007.02.001 |
| [13] |
VALENTINI A, POMPANON F, TABERLET P. DNA barcoding for ecologists[J]. Trends in Ecology and Evolution, 2009, 24(2): 110-117. DOI:10.1016/j.tree.2008.09.011 |
| [14] |
WAUGH J. DNA barcoding in animal species: progress, potential and pitfalls[J]. Bioessays, 2010, 29(2): 188-197. |
| [15] |
莫帮辉, 屈莉, 韩松, 等. DNA条形码识别I. DNA条形码研究进展及应用前景[J]. 四川动物, 2008, 27(2): 303-306. MO Banghui, QU Li, HAN Song, et al. DNA barcoding identificationI. research progress and applied perspective of DNA barcoding[J]. Sichuan Journal of Zoology, 2008, 27(2): 303-306. |
| [16] |
HEBERT P D N, Ratnasingham S, deWaard J R. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proceedings of the Royal Society B: Biological Sciences, 2003, 270(Suppl 1): 96-99. |
| [17] |
MANIATIS T, FRITSCH E F, SAMBROOK J. Molecular cloning: a laboratory manual[M]. New York: Cold Spring Harbor Laboratory Press, 1982.
|
| [18] |
WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia's fish species[J]. Philosophical transactions of the royal society B: biological sciences, 2005, 360: 1847-1857. DOI:10.1098/rstb.2005.1716 |
| [19] |
BURLAND T G. DNASTAR's Lasergene sequence analysis software[J]. Methods in Molecular Biology, 2000, 132: 71-91. |
| [20] |
STOVER N A, Cavalcanti A. Using NCBI bLAST[J]. Current Protocols Essential Laboratory Techniques, 2014, 8: 1-36. |
| [21] |
SUDHIR K, GLEN S, LI M, et al. MEGA X: Molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549. DOI:10.1093/molbev/msy096 |
| [22] |
OBERMILLERA L E, PFEILER E. Phylogenetic relationships of elopomorph fishes inferred from mitochondrial ribosomal DNA sequences[J]. Molecular Phylogenetics and Evolution, 2003, 26(2): 202-214. DOI:10.1016/S1055-7903(02)00327-5 |
| [23] |
WANG C H, KUO C H, MOK H K, et al. Molecular phylogeny of elopomorph fishes inferred from mitochondrial 12S ribosomal RNA sequences[J]. Zoologica Scripta, 2003, 32: 231-241. DOI:10.1046/j.1463-6409.2003.00114.x |
| [24] |
INOUE1 J H, MIYA M, MILLER M J, et al. Deep- ocean origin of the freshwater eels[J]. Biology Letters, 2010, 6: 363-366. DOI:10.1098/rsbl.2009.0989 |
| [25] |
SANTINI F, KONG X H, SORENSON L, et al. A multi- locus molecular timescale for the origin and diversification of eels(Order: Anguilliformes)[J]. Molecular Phyloge netics and Evolution, 2013, 69(3): 884-894. DOI:10.1016/j.ympev.2013.06.016 |
| [26] |
SAITOU N. Maximum likelihood method[J]. Methods in Enzymology, 1990, 183: 584-598. |
| [27] |
FELSENSTEIN J. Confidence limits on phylogenies: An approach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.2307/2408678 |
| [28] |
SCHWARZ G E. Estimating the dimension of a model[J]. Annals of Statistics, 1978, 6(2): 461-464. |
| [29] |
宁淑萍, 颜海飞, 郝刚, 等. 植物DNA条形码研究进展[J]. 生物多样性, 2008, 16(5): 417-425. NING Shuping, YAN Haifei, HAO Gang, et al. Current advances of DNA barcoding study in plants[J]. Biodiversity Science, 2008, 16(5): 417-425. DOI:10.3321/j.issn:1005-0094.2008.05.001 |
| [30] |
VIRGILIO M, BACKELJAU T, NEVADO B, et al. Comparative performances of DNA barcoding across insect orders[J]. BMC Bioinformatics, 2010, 11(1): 1-10. DOI:10.1186/1471-2105-11-1 |
| [31] |
张宇, 郭良栋. 真菌DNA条形码研究进展[J]. 菌物学报, 2012, 31(6): 809-820. ZHANG Yu, GUO Liangdong. Progress of fungal DNA barcode[J]. Mycosystema, 2012, 31(6): 809-820. DOI:10.13346/j.mycosystema.2012.06.017 |
| [32] |
RAMESH M, SEN A, VACHHER M, et al. Delineating bacteria using DNA barcoding[J]. Molecular Genetics, Microbiology and Virology, 2021, 36(1): 65-73. |
| [33] |
WEIGHT L A, BALDWIN C C, DRISKELL A, et al. Using DNA barcoding to assess Caribbean reef fish biodiversity: expanding taxonomic and geographic coverage[J]. PLoS ONE, 2012, 7(7): e41059. DOI:10.1371/journal.pone.0041059 |
| [34] |
CHANG C H, LIN H Y, REN Q, et al. DNA barcode identification of fish products in Taiwan: government- commissioned authentication cases[J]. Food Control, 2016, 66: 38-43. DOI:10.1016/j.foodcont.2016.01.034 |
| [35] |
李渊, 张然, 宋普庆, 等. 基于DNA条形码对南极Yelcho站周边海域鱼类的种类鉴定[J]. 极地研究, 2018, 30(2): 192-198. LI Yuan, ZHANG Ran, SONG Puqing, et al. Identification of antarctic fish from the sea adjacent to Yelcho station based on DNA barcoding[J]. Chinese journal of polar research, 2018, 30(2): 192-198. DOI:10.13679/j.jdyj.20170010 |
| [36] |
罗纯, 章群, 黄志基, 等. 中日沿海部分鱼类DNA条形码研究[J]. 海洋渔业, 2021, 43(1): 1-11. LUO Chun, ZHANG Qun, HUANG Zhiji, et al. DNA barcoding of some common coastal fishes from Japan and China[J]. Marine Fisheries, 2021, 43(1): 1-11. |
| [37] |
姜郦轩, 张吉昌, 樊钢洲, 等. 南极鱼亚目鱼类DNA条形码及分子系统学[J]. 中国水产科学, 2021, 28(11): 1396-1405. JIANG Lixuan, ZHANG Jichang, FAN Gangzhou, et al. DNA barcoding and molecular systematics of Notothenioidei[J]. Journal of Fishery Sciences of China, 2021, 28(11): 1396-1405. DOI:10.12264/JFSC2021-0149 |
| [38] |
STEIN M, WONG J C Y, SHENG V, et al. First genetic evidence of illegal trade in endangered European eel (Anguilla anguilla)from Europe to Asia[J]. Conservation Genetics Resources, 2016, 8(4): 1-5. |
| [39] |
龚小玲, 岳丽佳, 崔忠凯, 等. 鳗鲡属6种鱼类线粒体COⅠ和COⅡ基因序列分析和分类的有效性[J]. 上海海洋大学学报, 2013, 22(4): 524-530. GONG Xiaoling, YUE Lijia, CUI Zhongkai, et al. The sequencing analysis and taxonomic effectiveness of Anguilla six eels based on mtDNA COⅠ and COⅡ gene sequence[J]. Journal of Shanghai Ocean Uuniversity, 2013, 22(4): 524-530. |
| [40] |
张稚兰, 林汝榕, 邢炳鹏. COⅠ基因序列在蛇鳗科鱼类种类鉴定中的适用性研究[J]. 应用海洋学学报, 2017, 36(3): 411-416. ZHANG Zhilan, LIN Rurong, XING Bingpeng. Suitability analysis of mitochondrial COⅠ gene used as DNA barcode for Ophichthyidae[J]. Journal of Applied Oceanography, 2017, 36(3): 411-416. DOI:10.3969/J.ISSN.2095-4972.2017.03.015 |
| [41] |
陈文炳, 邵碧英, 缪婷玉, 等. 多基因DNA条形码鉴定6个鳗鱼物种[J]. 食品科学, 2018, 39(2): 163-169. CHEN Wenbing, SHAO Biying, MIAO Tingyu, et al. Identification of six eel species using polygenic DNA barcoding[J]. Food Science, 2018, 39(2): 163-169. |
| [42] |
桂建芳. 脊椎动物线粒体DNA的进化遗传学[J]. 动物学杂志, 1990, 25(1): 50-55. GUI Jianfang. Evolutionary genetics of vertebrate mitochondrial DNA[J]. Chinese Journal of Zoology, 1990, 25(1): 50-55. |
| [43] |
周建设, 张驰, 刘海平, 等. DNA条形码在西藏水系裂腹鱼亚科鱼类鉴定中的研究[J]. 淡水渔业, 2019, 49(6): 33-41. ZHOU Jianshe, ZHANG Chi, LIU Haiping, et al. Studies on identification of Schizothoracinae population in Tibet water system by DNA barcode[J]. Freshwater Fisheries, 2019, 49(6): 33-41. |
| [44] |
FADLI N, MUCHLISIN Z A, Siti-Azizah M N. DNA barcoding of commercially important groupers(Epinephelidae)in Aceh, Indonesia[J]. Fisheries Research, 2021, 234: 1-10. |
| [45] |
HUBERT N, HANNER R, HOLM E, et al. Identifying Canadian freshwater fishes through DNA barcodes[J]. PLOS ONE, 2008, 3(6): e2490. |
| [46] |
MEYER C P, PAULAY G. DNA barcoding: error rates based on comprehensive sampling[J]. PLOS Biology, 2005, 3(12): e422. |
| [47] |
ZHANG J B, HANNER R. DNA barcoding is a useful tool for the identification of marine fishes from Japan[J]. Biochemical Systematics and Ecology, 2011, 39(1): 31-42. |
| [48] |
王鹤, 林琳, 柳淑芳, 等. 中国近海习见头足类DNA条形码及其分子系统进化[J]. 中国水产科学, 2011, 18(2): 245-255. WANG He, LIN Lin, LIU Shufang, et al. DNA barcoding for Cephalopoda in classification and phylogeny[J]. Journal of Fishery Sciences of China, 2011, 18(2): 245-255. |
| [49] |
杨凡. 中国鲽形目鱼类的DNA分子条形码及褐牙鲆的遗传多样性研究[D]. 广州: 暨南大学, 2010. YANG Fan. DNA barcoding of the Pleuronectiformes and genetic diversity of Paralichthys olivaceus in China[D]. Guangzhou: Jinan University, 2010. |
| [50] |
关申民, 高邦权. COⅠ序列: 影响动物分类学与生态学的DNA barcode[J]. 2008, 生态学杂志, 27(8): 1406-1412. GUAN Shenmin, GAO Bangquan. COⅠ sequence, the DNA barcode affecting animal taxonomy and ecology[J]. Chinese Journal of Ecology, 27(8): 1406-1412. |
| [51] |
LIU S L, YANG C T, ZHOU C R, et al. Filling reference gaps via assembling DNA barcodes using high- throughput sequencing-moving toward barcoding the world[J]. Gigascience, 2017, 6(12): 1-8. |
 2023, Vol. 47
2023, Vol. 47


