
文章信息
- 贾真, 彭业韶, 孔令鑫, 秦越, 黎林杰, 梁柳, 朱鹏, 佘智彩. 2023.
- JIA Zhen, PENG Ye-shao, KONG Ling-xin, QIN Yue, LI Lin-jie, LIANG Liu, ZHU Peng, SHE Zhi-cai. 2023.
- 香港牡蛎糖原合酶与糖原磷酸化酶基因多态性研究及糖原含量性状相关分子标记挖掘
- Polymorphisms of glycogen synthase and glycogen phosphorylase genes and identification of glycogen content-associated molecular markers in Crassostrea hongkongensis
- 海洋科学, 47(7): 35-43
- Marine Sciences, 47(7): 35-43.
- http://dx.doi.org/10.11759/hykx20220627003
-
文章历史
- 收稿日期:2022-06-27
- 修回日期:2022-09-03
香港牡蛎(Crassostrea hongkongensis), 俗称大蚝, 主要分布于中国的广东、广西等地, 是华南沿海地区的主要经济贝类之一[1]。香港牡蛎曾以个大肥满, 肉质脆嫩, 肉味鲜美闻名全国。糖原是牡蛎体内的一种重要的营养物质, 与牡蛎的肉味鲜美[2]、繁殖、生长、能量代谢和抗逆性等密切相关[3-4]。其含量可达牡蛎干重的20%~40%[5], 是牡蛎品质性状的重要组成部分, 对牡蛎的口感和鲜味具有重要影响[6]。糖原是牡蛎进行正常生命活动的基础和保障, 尤其是繁殖季节, 香港牡蛎体内糖原快速消耗[7], 引起品质急剧下降。因此, 提高香港牡蛎糖原含量, 培育高糖原含量新品系对提高香港牡蛎的品质及其经济价值具有重要意义[8]。
使用传统的育种方式进行选育, 不仅周期长, 而且精度低。随着分子生物学的发展和分子标记辅助选择(Marker-assisted Selection, MAS)的出现, 人们可以在生物学性状和遗传物质之间直接建立联系, 从而显著提高选育的精准度, 缩短育种进程, 提高选育的效率[9]。而性状关联关键分子标记的筛选和鉴定是分子标记辅助选择工作的前提和基础[10]。候选基因是对目标性状具有明确的关键作用, 且功能已得到鉴定的基因。候选基因关联分析成本较低, 多态位点选择简单, 操作简便, 是鉴定性状关联分子标记的经济且有效的手段[11-12]。
因此, 本研究在转录组测序基础上, 利用FLDAS- PCR技术对香港牡蛎糖原代谢关键基因糖原合酶和糖原磷酸化酶进行基因多态性研究, 开发单核苷酸多态性(SNP)标记, 并使用三墩群体(SD)和大风江群体(DFJ)对SNP标记与糖原含量进行关联分析, 以挖掘糖原含量性状相关的SNP分子标记, 为选育高糖原含量香港牡蛎新品系奠定基础。
1 实验材料和方法 1.1 实验材料本实验共用到2个香港牡蛎群体, 一个是用于SNP标记开发及关键SNP筛选的群体, 来源于广西钦州市三墩海域(N21°62′98″, E108°68′46″), 记为SD群体, 共60只个体, 群体内的个体均在同一时间附苗, 同一自然环境下养殖。另一个是用于对筛选出的SNP进行验证的群体, 来源于广西钦州市大风江入海口(E108°86′16″, N21°67′77″), 记为DFJ群体, 共90只个体, 群体内的个体也是在同一时间附苗, 且在同一自然环境下养殖。上述两个群体均为繁殖前的2龄个体, 在2020年6月24日将其运至实验室, 分离闭壳肌、鳃组织, 投入液氮中速冻后于−80℃冷冻保存, 用于后续的糖原含量检测与总RNA提取。
1.2 糖原含量检测样品糖原含量委托上海酶联生物科技有限公司采用双抗体夹心法酶联免疫吸附ELISA试剂盒进行测定。使用预冷后的PBS冲洗牡蛎闭壳肌, 称重剪碎后与一定量的PBS混匀, 使用匀浆机进行破碎匀浆, 收集上清液。设置空白孔、标准孔和样品孔, 标准孔各加不同浓度的标准品, 样品孔预先加样品稀释液, 后加入样品。向各孔加入酶标试剂(空白孔除外), 用封板膜封板后置37 ℃温育60 min, 温育后弃去液体, 用洗涤液重复洗涤5次, 加入显色剂, 轻轻振荡混匀, 37 ℃避光显色15 min后加终止液终止反应。以空白孔调零, 450 nm波长依序测量各孔的吸光度(OD值), 依据标准样品的吸光度计算待测样品的糖原含量。
1.3 SNP多态标记开发 1.3.1 引物设计选取糖原代谢关键基因糖原合酶和糖原磷酸化酶基因作为研究对象, 以香港牡蛎转录组测序预测的SNP位点作为候选SNPs。利用比对软件STAR对每个样本的Reads与Unigene序列进行比对, 然后通过GATK软件进行SNP识别。根据FLDAS-PCR原理, 使用Oligo7软件进行引物设计。针对候选SNP位点设计2条3′末端分别与突变类型的2个等位基因配对的上游(若上游不适合设计则改换设计下游)引物, 同时为增加2条引物的特异性在3′端的第3或第4位碱基引入人为错配, 在其中1条或2条引物的5′末端添加不同长度的核苷酸序列, 使2条引物长度有4~8 bp的差异。下游引物则按照普通引物设计的方法进行设计, 扩增的DNA片段的长度在100~200 bp。引物委托生工生物工程(广州)股份有限公司合成。
1.3.2 候选SNPs的验证使用FLDAS-PCR及聚丙烯酰胺凝胶电泳相结合的方法进行候选SNPs的验证。在SD群体中随机抽取4只个体的鳃组织, 用Transzol法提取总RNA并反转录成cDNA, 作为FLDAS-PCR的模板。使用PCR试剂盒(CWBIO) 25 μL反应体系进行DNA扩增: 2×ES Taq Master Mix 12.5 μL, ddH2O 9.5 μL, 3种引物各0.7 μL, DNA模板1 μL。PCR循环参数设置如下: 94 ℃预变性3 min, 94 ℃ 30 s, 最适Tm 30 s, 72 ℃ 30 s, 35个循环, 72 ℃延伸5 min。PCR产物经12%非变性聚丙烯酰胺凝胶在300 V电压下电泳35 min进行检测验证。使用GeneGreen核酸染料和凝胶成像系统对扩增结果进行观察, 其中扩增结果较好, 目标条带清晰且单一的候选SNP位点即为验证的SNPs。
1.4 SNP标记分型检测取SD群体及DFJ群体所有个体的鳃组织, 用Transzol法提取总RNA并反转录成cDNA。以反转录的cDNA为模板, 使用上述FLDAS-PCR的反应体系和循环参数进行PCR扩增, 扩增后的产物使用12%非变性聚丙烯酰胺凝胶电泳进行检测, 根据电泳结果读取基因型。
1.5 FLDAS-PCR分型结果准确性检验从分型成功的SNP位点中, 随机挑选几只个体的PCR产物, 然后进行PCR产物回收, 连接T载体, 转化入大肠杆菌, 培养后将菌液送至生工生物工程(广州)股份有限公司进行测序。
1.6 关联分析对于在SD群体中分型成功的SNP位点, 使用IBM SPSS Statistics 24软件, 对糖原含量数据和基因型数据利用单因素方差分析或T检验的方法进行关联分析, 同一位点不同基因型之间, 糖原含量存在显著差异的被认为是与糖原含量关联的SNP标记。针对筛选出的SNP标记, 使用IBM SPSS Statistics 24软件在DFJ群体中再次进行关联分析。
2 实验结果 2.1 实验群体糖原含量SD群体糖原含量分布如图 1a所示, 最高值Max=1.85 mg/g, 最低值Min=1.51 mg/g, 糖原含量平均值Mean= 1.68±0.10 mg/g, 变异系数CV=5.95%。正态分布检验显示实验群体糖原含量符合正态分布(P > 0.05)。DFJ群体糖原含量分布如图 1b所示, 最高值Max=1.82 mg/g, 最低值Min=1.48 mg/g, 糖原含量平均值Mean=1.66± 0.10 mg/g, 变异系数CV =6.02%。正态分布检验显示实验群体糖原含量符合正态分布(P > 0.05)。
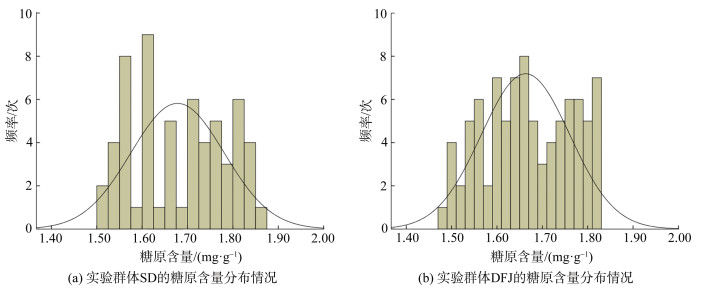 |
| 图 1 实验群体糖原含量分布图 Fig. 1 Distribution of glycogen content in the experimental population |
根据香港牡蛎转录组测序结果, 糖原合酶基因共预测出候选SNP位点9个, 共设计FLDAS-PCR引物9组, 最终9个SNP位点全部得到验证, 成功开发出SNP标记9个。糖原合酶基因SNP标记及其引物信息如表 1所示。糖原磷酸化酶基因共预测出候选SNP位点38个, 共设计FLDAS-PCR引物30组, 最终16个SNP位点得到验证, 成功开发出SNP标记16个。糖原磷酸化酶基因SNP标记及其引物信息如表 2所示。
| SNP名称 | 突变类型 | 引物名称 | 引物序列(5′—3′) |
| Ch_GS25 | A/G | GS25_A | AACAGCATACTAGAATGCACGA |
| GS25_G | ATATAACAGCATACTAGAATGCGCAG | ||
| GS25_R | AGTGCTATTTACAAATGTTAAACCT | ||
| Ch_GS97 | A/C | GS97_A | CCCAGTTATTCATGAAAGTCGCAA |
| GS97_C | GCTATCCCAGTTATTCATGAAAGTCTTAC | ||
| GS97_R | GTTGGACATGAATGATCCTGATTAGA | ||
| Ch_GS233 | G/A | GS233_C | CCTGATTAGATGATTATAGTGTGTC |
| GS233_T | ATATCCTGATTAGATGATTATAGTGTGTT | ||
| GS233_R | ACCCAGTTATTCATGAAAGTCTC | ||
| Ch_GS1166 | C/T | GS1166_A | TCCTAGGTGTATGAATGGTTAGAGACA |
| GS1166_G | AGGTGTATGAATGGTTAGACTCG | ||
| GS1166_R | ATGGGCAAATGGGATATGATAA | ||
| Ch_GS1206 | G/A | GS1206_C | GCTCTAGGATTAAAGAAGTGTTACC |
| GS1206_T | TTAAGCTCTAGGATTAAAGAAGTGTATCT | ||
| GS1206_R | GGAGATTTCAGGCAGTACATTG | ||
| Ch_GS1211 | C/T | GS1211_G | TCTGCTCTAGGATTAAAGATGTG |
| GS1211_A | GGGATCTGCTCTAGGATTAAAGAACTA | ||
| GS1211_R | GGAGATTTCAGGCAGTACATTG | ||
| Ch_GS1248 | G/A | GS1248_G | CCTTCAGAATGCTACTTTTCGAG |
| GS1248_A | ATTAACCTTCAGAATGCTACTTTTCCTA | ||
| GS1248_R | AGAATCATTATCTGCTACATACAAGG | ||
| Ch_GS688 | G/T | GS688_G | GCCCATCCAAGTTAATAAATCCTG |
| GS688_T | TAGCGCCCATCCAAGTTAATAAATGCCT | ||
| GS688_R | TATACAACAAAAGGTCAGCTAGAAGG | ||
| Ch_GS680 | C/T | GS680_T | CGGGCCAAGTTAATAAATGCTGATTGCTAT |
| GS680_C | CAAGTTAATAAATGCTGATTGTGAC | ||
| GS680_R | TATACAACAAAAGGTCAGCTAGAAGG |
| SNP名称 | 突变类型 | 引物名称 | 引物序列(5′—3′) |
| Ch_GP25 | A/G | GP25_A | AACAGCATACTAGAATGCACGA |
| GP25_G | ATATAACAGCATACTAGAATGCGCAG | ||
| GP25_R | AGTGCTATTTACAAATGTTAAACCT | ||
| Ch_GP53 | A/G | GP53_A | GGGCCACATTTAGTTGATGGTTACAATGCA |
| GP53_G | ACATTTAGTTGATGGTTACAATGGG | ||
| GP53_R | GAAACGTACACCAAACTAGGA | ||
| Ch_GP233 | C/T | GP233_C | CCTGATTAGATGATTATAGTGTGTC |
| GP233_T | ATATCCTGATTAGATGATTATAGTGTGTT | ||
| GP233_R | ACCCAGTTATTCATGAAAGTCTC | ||
| Ch_GP461 | A/G | GP461_A | ATATATATCTTTGGCATGAGGCTGA |
| GP461_G | ATCTTTGGCATGAGGGAGG | ||
| GP461_R | CAGCTCCGGAAGATTCTCA | ||
| Ch_GP579 | A/G | GP579_A | TATAGGGCGAATTTCATGTTTTCA |
| GP579_G | GGGGGAATTTCATGTTTGCG | ||
| GP579_R | TCCTGTGATTGGACGTAAACT | ||
| Ch_GP623 | G/T | GP623_G | CTTCTTTCACTCATTATACAGTAAG |
| GP623_T | GGGGCCTTCTTTCACTCATTATACAGCAGT | ||
| GP623_R | CAAGCCGTAAAGAACAATCCG | ||
| Ch_GP944 | G/T | GP944_G | TCAATATTCAAAAAGGCGTTTAATCGCTAG |
| GP944_T | TTCAAAAAGGCGTTTAATCGGCAT | ||
| GP944_R | CTCTCTTTGGGTCCTTCTCATAGT | ||
| Ch_GP992 | G/A | GP992_G | TGGGCAAGAGAGAGGTAGAAG |
| GP992_A | TATCCGTGGGCAAGAGAGAGGTAATAA | ||
| GP992_R | TCAGTATTCGCGGTATTGCTC | ||
| Ch_GP1013 | A/C | GP1013_A | ATATACTACTACCTCTCTCTTGCCGAA |
| GP1013_C | CTACTACCTCTCTCTTGCTCAC | ||
| GP1013_R | GTCCTGCCCATGTAAAACTCC | ||
| Ch_GP1253 | G/T | GP1253_G | AATAATCTGGGTAATGGAGGACTTGGG |
| GP1253_T | TCTGGGTAATGGAGGACTTGGT | ||
| GP1253_R | TTTCTGGGCAAAGATTCCATAGT | ||
| Ch_GP1598 | G/A | GP1598_G | ATTCGGGTTGAAGAAGTGCAGGCTG |
| GP1598_A | GCGTTGAAGAAGTGCAGGATA | ||
| GP1598_R | CAGGGGTCAAGTGGGTGGA | ||
| Ch_GP1604 | A/G | GP1604_A | CGGTATCCCAACAGCTTCAACTTA |
| GP1604_G | TCCCAACAGCTTCAACCCG | ||
| GP1604_R | AGAAAAGAAGTTATCGTTGGGGT | ||
| Ch_GP1773 | C/G | GP1773_C | AACTTAGCAGAAAACATTCCC |
| GP1773_G | AGGCAACTTAGCAGAAAACATTACG | ||
| GP1773_R | GCTGCCACCACGAAATATTCC | ||
| Ch_GP1962 | A/G | GP1962_A | TTATTAGAAGAGGAGGGAAAGGAACCA |
| GP1962_G | GAAGAGGAGGGAAAGGAGCCG | ||
| GP1962_R | GTCCAAAACTGCCTGAATGTAA | ||
| Ch_GP2286 | C/T | GP2286_C | ATCAATCTGGGTCTGCATAAC |
| GP2286_T | CGTAATCAATCTGGGTCTGCAAAAT | ||
| GP2286_R | CCTAATCCAGCATCTTCTTCCA | ||
| Ch_GP2291 | G/A | GP2291_G | ACCAATGAGCGCTTCCAAAACAAATCG |
| GP2291_A | GTGAGCGCTTCCAAAACAAAATA | ||
| GP2291_R | CCCATTCCTCCCCGATTTTC |
经SNP标记分型检测, 糖原合酶最终有2个SNP位点在SD群体中分型成功, 分别为SNP Ch_GS680和SNP Ch_GS1248。其中SNP Ch_GS680在DFJ群体中也分型成功。糖原磷酸化酶最终也有2个SNP位点在SD群体中分型成功, 分别为SNP Ch_GP992和SNP Ch_GP1962。上述4个SNP位点各基因型在实验群体中的分布统计结果见表 3和图 2。
| SNP位点 | 实验群体 | 基因型 | 个体数 |
| Ch_GS680 | SD | TT | 53 |
| TC | 6 | ||
| CC | 1 | ||
| Ch_GS1248 | SD | AA | 2 |
| AG | 7 | ||
| GG | 51 | ||
| Ch_GP992 | SD | AG | 6 |
| AA | 54 | ||
| Ch_GP1962 | SD | AG | 1 |
| GG | 59 | ||
| Ch_GS680 | DFJ | TT | 85 |
| TC | 5 |
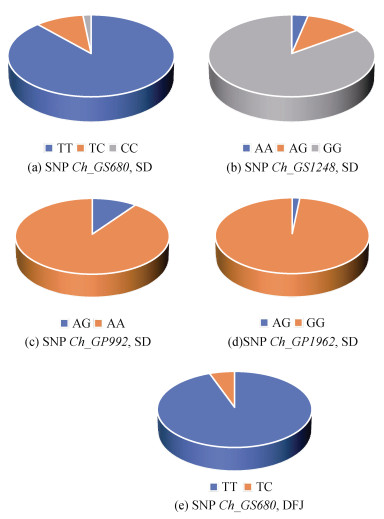 |
| 图 2 SNP位点基因型在实验群体中的分布图 Fig. 2 Distribution of genotypes of SNPs in the experimental population |
测序结果显示, 使用FLDAS-PCR方法的分型结果与测序的完全一致, 表明FLDAS-PCR分型结果是准确可靠的(图 3)。
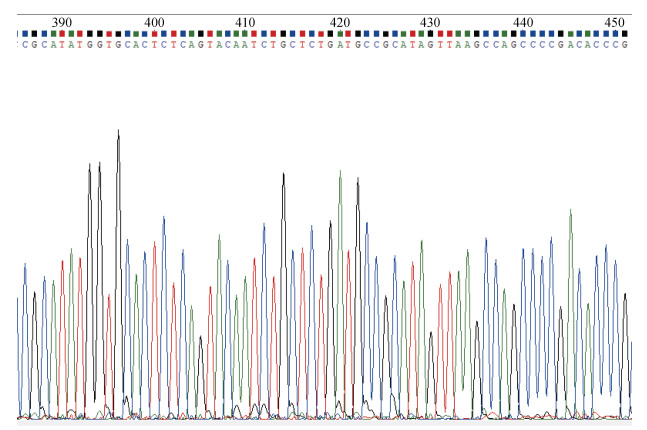 |
| 图 3 GS1248_G引物PCR扩增产物的测序结果 Fig. 3 Sequencing results of PCR amplification products of primer GS1248_G |
经单因素方差分析, 发现SNP Ch_GS680在SD群体中不同基因型之间的糖原含量存在显著差异(P < 0.05), 说明SNP Ch_GS680是与糖原含量关联的SNP位点。对该SNP位点在DFJ群体中再次进行关联分析以对该SNP进行验证, 分型结果显示SNP Ch_GS680在DFJ群体中只有TT和TC两种基因型(图 2), 所以使用独立样本T-检验进行关联分析, 但结果显示TT和TC两种基因型之间的糖原含量并无显著差异(P > 0.05)。SNP Ch_GS680在SD群体中不同基因型之间的两两比较结果见表 4。
| 基因型Ⅰ | 基因型Ⅱ | 平均值差值(Ⅰ-Ⅱ) | P值 |
| CC | TC | −0.25* | 0.03 |
| TT | −0.16 | 0.11 | |
| TC | CC | 0.25* | 0.03 |
| TT | 0.08 | 0.05 | |
| TT | CC | 0.16 | 0.11 |
| TC | −0.08 | 0.05 | |
| 注: *, 平均值差值的显著性水平为0.05。 | |||
SNP Ch_GS1248在SD群体不同基因型之间的糖原含量不存在显著差异(P > 0.05), 说明SNP Ch_ GS1248与糖原含量之间无关联关系。经独立样本T-检验, 发现SNP Ch_GP992和SNP Ch_GP1962在SD群体中不同基因型之间的糖原含量均不存在显著差异(P > 0.05), 说明SNP Ch_GP992和SNP Ch_GP1962与糖原含量之间也无关联关系。各SNP位点在SD群体中与糖原含量的关联分析结果见表 5。
| SNP位点 | P值 | 是否和性状关联 |
| Ch_GS680 | 0.039 | 是 |
| Ch_GS1248 | 0.273 | 否 |
| Ch_GP992 | 0.17 | 否 |
| Ch_GP1962 | 0.76 | 否 |
本研究所用香港牡蛎糖原含量为1.48~1.85 mg/g, 平均值(1.68±0.10)~(1.66±0.10) mg/g, 变异系数达到6.02%。这与长牡蛎相比, 糖原含量及变异系数均较低。She等[13]检测长牡蛎(Crassostrea gigas)糖原含量为2.1~10.4 mg/g, 平均为(5.3±1.7) mg/g, 这可能是由物种间的差异造成的。王卫军等[14]在我国多个长牡蛎产地选取不同发育阶段、不同养殖方式和不同年龄的77份长牡蛎样品, 测得长牡蛎糖原含量为0.13~ 10.57 mg/g, 平均为(4.14±1.99) mg/g, 变异系数高达48.07%, 这可能与长牡蛎的糖原含量测量方法、所用组织及样品来源不同有关。本实验所用糖原检测方法为ELISA试剂盒(上海酶联, 中国), 而王卫军等[14]所用方法为EnzyChromTM糖原试剂盒(BioAssay Systems, USA), 不同方法检测结果可能不同; 本实验所用组织为闭壳肌, 而王卫军等[14]所用组织为整个软体部, 不同组织糖原含量不同, 会造成测量结果的差异; 本实验所用香港牡蛎均来自广西茅尾海, 而王卫军等[14]所用长牡蛎来自我国多个不同产区, 可能牡蛎间差异较大, 因此糖原含量变异系数较大。刘思玮等依据蒽酮比色法, 将长牡蛎性腺组织经过冷冻干燥后研磨成粉末, 测得糖原含量为(30.59%±2.10%)~(35.24%±2.22%), 这种方法所测糖原含量为样品干重的糖原含量, 因此, 远远高于本实验所测样品湿重的糖原含量[15-16]。香港牡蛎与其他贝类如菲律宾蛤仔(Ruditapes philippinarum)、缢蛏(Sinonovacula constricta)、褶牡蛎(Ostrea plicatula)相比, 糖原含量也处于较低水平, 而和皱纹盘鲍(Haliotis discus hannai)的糖原含量水平相当[17]。
3.2 SNP标记开发及分型效率本研究通过转录组测序预测出香港牡蛎糖原合酶基因编码区2 210 bp, 包含9个候选SNP位点, 针对这9个候选SNP位点, 我们共设计了9组特异性引物, 利用FLDAS-PCR和聚丙烯酰胺凝胶技术成功开发出SNP标记9个, SNP开发成功率为100%(9/9), 远远大于使用高分辨率溶解曲线(high resolution melting curve, HRM)技术对长牡蛎糖原合酶进行SNP开发的成功率40.9%(9/22)[13]。通过转录组测序预测出糖原磷酸化酶基因编码区7 488 bp, 包含38个候选SNP位点, 针对这38个候选SNP位点, 我们共设计了30组特异性引物, 成功开发出SNP标记16个, SNP开发成功率为53.3%(16/30)。使用HRM技术对长牡蛎糖原磷酸化酶进行SNP开发的成功率33.3%(14/42)[13], 使用HRM技术对长牡蛎转录组进行SNP开发的成功率43.9%(1 301/2 962)[18], 说明FLDAS-PCR技术进行SNP开发的成功率比较高。但FLDAS-PCR技术通量较低, 不适合进行大规模SNP的开发检测。
本研究所得香港牡蛎糖原合酶基因编码区SNPs的平均密度为1/246(9/2 210), 香港牡蛎糖原磷酸化酶基因编码区SNPs的平均密度为1/468(16/7 488), 这远远低于通过测序验证的长牡蛎糖原合酶基因编码区的SNPs平均密度为1/35 bp与长牡蛎糖原磷酸化酶基因编码区SNPs平均密度为1/25 bp[15-16], 这可能是因为本研究转录组预测的SNP位点密度较低所致。在群体分型中, 香港牡蛎糖原合酶与糖原磷酸化酶分别有2个SNPs分型成功, 分型效率分别为22.2%(2/9)和12.5%(2/16), 使用HRM技术对长牡蛎糖原合酶与糖原磷酸化酶SNP的分型效率分别为55.6%(5/9)和7.1%(1/14)[13], 说明FLDAS-PCR技术的分型效率较低且不稳定。
3.3 关键SNP标记挖掘本研究在香港牡蛎糖原合酶基因编码区开发出9个SNP位点, 通过关联分析在SD群体检测到1个SNP可能与香港牡蛎糖原含量显著相关(P < 0.05), 但在DFJ群体未检测到该相关性, 这可能与本研究分型成功的用于关联分析的SNPs较少有关, 也可能是因为本研究所鉴定的SNP位点具有较强的群体特异性。在香港牡蛎糖原磷酸化酶基因编码区开发出16个SNP位点, 但未检测到与糖原含量显著相关的SNP位点, 这也可能是因为分型成功的SNP较少所致。
目前, 已有研究在长牡蛎糖原合酶基因外显子区检测到41个SNP位点, 其中6个SNPs与糖原含量显著相关(P < 0.01), 且具有单倍型H2(GAGGAT)的个体在糖原含量上达到极显著差异(P < 0.000 1)[15]。在长牡蛎糖原磷酸化酶基因中共检测到82个SNPs位点, 但未检测到与糖原含量相关的SNPs[16]。在长牡蛎糖原磷酸化酶基因5′UTR上游2 773 bp处检测到1个SNP与糖原含量显著相关, 说明糖原磷酸化酶基因中可能不存在与糖原含量显著关联的SNP位点[13]。
另有研究发现位于长牡蛎基因组scaffold1597的36 675碱基处的SNP标记, GG基因型个体相比较于AA基因型个体, 糖原含量显著提高4.5%[19]。蛋白磷酸酶1调节亚基3B(糖原靶向亚基)启动子区有3个与糖原含量显著关联的SNP, 同时拥有3个优势基因型个体糖原含量比劣势基因型组合个体显著提高10%以上[20]。在缢蛏(S. constricta)糖原合酶基因中共筛选出2个与糖原含量相关的SNP位点, 其优势基因组合个体的糖原含量比群体均值高11.8%[21]。这些结果说明利用关联分析的方法鉴定糖原含量相关的关键SNP标记是比较准确的, 这些标记可以为标记辅助选育提供基础。
4 结论本研究利用FLDAS-PCR技术共成功开发香港牡蛎糖原合酶基因SNP位点9个, 香港牡蛎糖原磷酸化酶基因SNP位点16个。利用ELISA方法测得香港牡蛎糖原含量为1.48~1.85 mg/g, 平均值(1.68±0.10)~ (1.66±0.10) mg/g, 变异系数达到6.02%。关联分析结果显示位于香港牡蛎糖原合酶基因的SNP位点Ch_GS680与糖原含量显著关联(P < 0.05), 可作为香港牡蛎分子标记辅助选育的潜在分子标记。
| [1] |
潘英, 李坚明, 黄伟德. 广西贝类养殖现状及产业发展策略建议[J]. 海洋科学, 2015, 39(11): 132-137. PAN Ying, LI Jianming, HUANG Weide. The perspectives of molluscan mariculture and its developmental suggestions in Guangxi[J]. Marine Sciences, 2015, 39(11): 132-137. |
| [2] |
刘文广, 李琪, 高凤祥, 等. 长牡蛎繁殖周期、生化成分的季节变化与环境因子的关系[J]. 热带海洋学报, 2011, 30(3): 88-93. LIU Wenguang, LI Qi, GAO Fengxiang, et al. Seasonal variation in reproductive cycle and biochemical composition in relation to environmental factors in the Pacific oyster Crassostrea gigas[J]. Journal of Tropical Oceanography, 2011, 30(3): 88-93. |
| [3] |
ROACH P J, DEPAOLI-ROACH A A, HURLEY T D, et al. Glycogen and its metabolism: Some new developments and old themes[J]. Biochemical Journal, 2012, 44(3): 763-787. |
| [4] |
LI B S, SONG K, MENG J, et al. Integrated application of transcriptomics and metabolomics provides insights into glycogen content regulation in the Pacific oyster Crassostrea gigas[J]. BMC Genomics, 2017, 18: 713. DOI:10.1186/s12864-017-4069-8 |
| [5] |
陈骞, 杨瑞金, 顾聆琳. 牡蛎糖原的研究(II)——牡蛎糖原的分子结构[J]. 食品科学, 2005, 26(7): 43-46. CHEN Qian, YANG Ruijin, GU Linglin. Studies on oyster glycogen (II)——the fine structure of oyster glycogen[J]. Food Science, 2005, 26(7): 43-46. |
| [6] |
陈骞, 杨瑞金, 顾聆琳. 牡蛎糖原的研究(I)——牡蛎糖原的分离提取和化学组成[J]. 食品科学, 2005, 26(6): 99-101. CHEN Qian, YANG Ruijin, Gu Linling. Studies on oyster glycogen part I: Extraction and purification and chemical composition of oyster glycogen[J]. Food Science, 2005, 26(6): 99-101. |
| [7] |
SAMAIN J F. Review and perspectives of physiological mechanisms underlying genetically based resistance of the Pacific oyster Crassostrea gigas to summer mortality[J]. Aquatic Living Resources, 2011, 24(3): 227-236. DOI:10.1051/alr/2011144 |
| [8] |
张国范, 李莉, 阙华勇. 中国牡蛎产业的嬗变——新认知、新品种和新产品[J]. 海洋与湖沼, 2020, 51(4): 740-749. ZHANG Guofan, LI Li, QUE Huayong. An evolution of oyster mariculture industry in China: New knowledge, variety and product[J]. Oceanologia et Limnologia Sinica, 2020, 51(4): 740-749. |
| [9] |
李春燕. 牡蛎营养品质等重要经济性状的遗传定位与基因解析[D]. 青岛: 中国科学院海洋研究所. 2017: 3-11. LI Chunyan. Genetic mapping and gene dissection of nutritional and other important economic traits of the oyster[D]. Qingdao: Institute of Oceanology, Chinese Academy of Sciences. 2017: 3-11. |
| [10] |
陈秀荔, 李咏梅, 陈晓汉. 分子标记技术在牡蛎遗传育种中应用研究进展[J]. 广西农业科学, 2008, 39(5): 691-695. CHEN Xiuli, LI Yongmei, CHEN Xiaohan. Advance in applications of molecular marker technologies in genetics and breeding of oyster[J]. Journal of Southern Agriculture, 2008, 39(5): 691-695. |
| [11] |
LI B S, LI L, MENG J, et al. Association and functional analyses revealed that PPP1R3B plays an important role in the regulation of glycogen content in the Pacific oyster Crassostrea gigas[J]. Frontiers in Genetics, 2019, 10: 106. |
| [12] |
LIU S W, LI Q, YU H, et al. Relationship between single nucleotide polymorphism of glycogen synthase gene of Pacific oyster Crassostrea gigas and its glycogen content[J]. Journal of Ocean University of China, 2017, 16(1): 168-174. |
| [13] |
SHE Z C, LI L, QI H G, et al. Candidate gene polymorphisms and their association with glycogen content in the Pacific oyster Crassostrea gigas[J]. PloS One, 2015, 10(5): e0124401. |
| [14] |
王卫军, 杨建敏, 董迎辉, 等. 长牡蛎(Crassostrea gigas)鲜样组织八种成分含量近红外(NIR)模型的建立[J]. 海洋与湖沼, 2015, 46(4): 845-852. WANG Weijun, YANG Jianmin, DONG Yinghui, et al. Establishment of near infrared models of eight components on fresh tissue of Pacific oyster Crassostrea gigas[J]. Oceanologia et Limnologia Sinica, 2015, 46(4): 845-852. |
| [15] |
刘思玮. 长牡蛎糖原代谢酶基因多态性与生长及糖原含量相关性研究[D]. 青岛: 中国海洋大学, 2013, 24-31. LIU Siwei. Polymorphisms of glycogen metabolism enzyme gene and their association with growth performance and glycogen content in the Pacific oyster Crassostrea gigas[D]. Qingdao: Ocean University of China, 2013, 24-31. |
| [16] |
刘思玮, 李琪, 于红, 等. 长牡蛎糖原磷酸化酶基因SNPs与生长性状和糖原含量的相关性分析[J]. 中国水产科学, 2013, 20(3): 481-489. LIU Siwei, LI Qi, YU Hong, et al. Single nucleotide polymorphisms in glycogen phosphorylase gene and their association with growth performance and glycogen content in Pacific oyster Crassostrea gigas[J]. Journal of Fishery Sciences of China, 2013, 20(3): 481-489. |
| [17] |
孙莎, 钟婵, 章骞, 等. 4种海产贝类中主要呈味物质含量与季节变化的关系[J]. 集美大学学报(自然科学版), 2016, 21(6): 420-427. SUN Sha, ZHONG Chan, ZHANG Qian, et al. Seasonal variation of taste-active component contents in four species of marine shellfish[J]. Journal of Jimei University(Natural Science), 2016, 21(6): 420-427. |
| [18] |
WANG Jiafeng, QI Haigang, LI Li, et al. Discovery and validation of genic single nucleotide polymorphisms in the Pacific oyster Crassostrea gigas[J]. Molecular Ecology Resources, 2014, 11: 123-135. |
| [19] |
张国范, 李莉, 孟杰, 等. 用于筛选长牡蛎高糖原含量亲贝的方法及其相关引物对[P]. 山东省: CN107354234B, 2020-12-25. ZHANG Guofan, LI Li, MENG Jie, et al. Methods for screening high glycogen content mollusk and related primer pairs of Crassostrea gigas[P]. Shandong: CN107354234B, 2020-12-25. |
| [20] |
刘圣, 李莉, 孟杰, 等. 长牡蛎糖原含量相关基因与SNP标记鉴定及高糖原个体筛选方法[P]. 山东省: CN109355400A, 2019-02-19. LIU Sheng, LI Li, MENG Jie, et al. Identification of genes and SNP markers related to glycogen content in Crassostrea gigas and screening of individuals with high glycogen[P]. Shandong: CN109355400A, 2019-02-19. |
| [21] |
陈燕园, 刘圣, 何京, 等. 浙江缢蛏群体糖原含量变化与GYS基因多态性的关联分析[J]. 水产学报, 2021, 45(3): 415-423. CHEN Yanyuan, LIU Sheng, HE Jing, et al. Glycogen content fluctuation and association analysis with polymorphism of gys gene of Sinonovacula constricta in the Zhejiang population[J]. Journal of Fisheries of China, 2021, 45(3): 415-423. |
 2023, Vol. 47
2023, Vol. 47


