文章信息
- 韩宜晓, 任艺飞, 庄英瑞, 陈尧, 钟秋连, 邱其樱, 陈德福, 王高歌. 2023.
- HAN Yi-xiao, REN Yi-fei, ZHUANG Ying-rui, CHEN Yao, ZHONG Qiu-lian, QIU Qi-ying, CHEN De-fu, WANG Gao-ge. 2023.
- 一株可诱导海带配子体产生白化病的细菌的分离与鉴定
- Isolation and identification of a pathogenic bacterium causing bleaching disease in the gametophytes of Saccharina japonica
- 海洋科学, 47(7): 53-64
- Marine Sciences, 47(7): 53-64.
- http://dx.doi.org/10.11759/hykx20210411001
-
文章历史
- 收稿日期:2021-04-11
- 修回日期:2021-05-12
2. 中国海洋大学 海洋生物多样性与进化研究所, 山东 青岛 266003;
3. 福建省连江官坞海产开发有限公司, 福建 官坞 350511
2. Institute of Evolution & Marine Biodiversity, Ocean University of China, Qingdao 266003, China;
3. Fujian Lianjiang Guanwu Seafood Developing Product Co., Ltd., Guanwu 350511, China
大型藻类表面附生细菌的种类繁多, 它们既可能对宿主有利, 也可使宿主致病。例如, 海藻表面的一些附生细菌有利于海藻的生长和发育[1]。而在当环境条件改变时, 比如温度升高、光照强度变化情况下, 大型藻类的表面附生细菌可能变成致病菌, 导致病害的暴发。这些细菌被称为条件致病菌[2-3]。通常情况下, 可以运用传统微生物分离、纯化培养以及复染实验来鉴定致病菌。
国际上, 运用传统微生物分离培养的方法鉴定出经济养殖海带(Saccharina japonica)病害的致病菌[4-5]。Ezura等人于1988年证实Pseudoalteromonas bacteriolytica是海带幼苗红斑病(red-spotted)的致病菌[6]。P. bacteriolytica生长温度在15~35 ℃之间, 是一种革兰氏阴性菌。Yumoto等人1989年从红斑病海带上分离出一株交替单胞菌(Alteromonas)致病菌[7]。1992年Sawabe等人从腐烂海带藻体上分离一株具有降解海藻酸钠的致病菌—Alteromonas sp.[8]。1998年Swabe从海带的病烂组织中分离出了另一种致病菌—假交替单胞菌(Pseudoalteromonas), 它可导致海带红斑病[9]。假交替单胞菌—Pseudoalteromonas elyakovii是Sawabe等人从烂孔病海带上分离出的一种褐藻酸降解菌, 能分泌具有分解褐藻酸和纤维素的褐藻酸酶, 降解海带细胞壁的主要成分褐藻酸, 可以使细胞原生质体分离, 从而导致使海带病烂[10]。随后, Vairappan等人发现Alteromonas sp.可诱导皱海带(Laminaria religiosa)产生白化病[11]。尽管目前已经分离、鉴定出一些经济养殖海带病害的致病菌, 但对致病菌的致病机制知之甚少。主要原因是与大多数海藻致病菌一样, 海带的致病菌多为条件致病菌[2], 致病菌在实验室条件下培养几代后往往会失去致病性。因此, 目前还没有一个稳定的致病菌与大型经济养殖海藻稳定的实验模型来研究致病菌的致病机理。
海带是世界性经济海藻, 具有较高的食用和商业价值。中国海带与褐藻胶的产量分别占世界总产量的60%和90%[12]。自1958年海带在中国成功实现规模化人工养殖以来, 在苗期及养殖期常常暴发病害, 严重时可造成20%~30%的减产[13]。中国学者在海带致病菌的分离、鉴定方面做了大量的研究工作。首先, 运用传统的微生物培养纯化方法以及复染实验证实褐藻酸降解菌是海带苗期最具破坏性的病害—绿烂/脱苗病的致病菌[14-16]。这些褐藻酸降解菌产生的复染病症与苗期观察到的病害病症相类似[17]。Lin等人2004年报道绿烂幼孢子体上的褐藻酸降解菌的数量(4.88×107 cfu/g)是健康幼孢子体的100~500倍[(0.8~4.3)×105 cfu/g][18]。这里褐藻酸降解菌指的不是分类学意义上的种, 而是指所有具有降解海带细胞壁的细菌[14]。目前, 褐藻酸降解菌被普遍认为是经济养殖海带病害的条件致病菌[3]。除了褐藻酸降解菌以外, 假单胞菌属(Pseudomonas)的细菌由于能够分泌褐藻酸酶, 可降解海带细胞壁, 从而引起海带藻体腐烂, 是导致海带腐烂病的一种主要致病菌[19-20]。畸形病是配子体及幼孢子体时期的一种常见病害。现已证实, 硫酸盐还原菌、可产生硫化氢的腐生菌以及微球菌是导致畸形病的致病菌[19-21]。另外, 麦氏交替单胞菌(Alteromonas macleodii)是在实验室条件下导致健康海带病烂的一种条件致病菌[22-23]。2006年, 埃氏交替单胞菌(Alteromonas espejiana)被鉴定为海带白化病的致病菌[24]。Wang 等人报道了假交替单胞菌(Pseudoalteromonas)、弧菌(Vibrio)、盐单胞菌(Halomonas)以及芽孢杆菌(Bacillus)是孔烂病海带孢子体的优势菌群[25]。然而, 这些优势菌是否具有致病性, 目前还需复染实验证实。
2019年在福建省连江县海带育苗厂发生了脱苗病。脱苗病表现为海带幼苗从苗帘上脱落, 苗帘上出现斑秃的区域。显微镜下观察脱苗病海带幼苗的藻体呈现白化并伴有绿烂症状, 且藻体上覆盖大量的硅藻。本研究旨在从苗期脱苗病海带幼苗藻体中分离、鉴定致病菌。运用16S rDNA基因比对及生理生化指标进行分子鉴定。本研究结果将有助于建立一个稳定的致病与海带之间的实验模型, 为深入研究致病菌的致病机理奠定工作基础。同时, 也可为预防及减轻海带苗期脱苗病的暴发提供理论依据。
1 材料与方法 1.1 脱苗病海带幼苗样品的采集脱苗病海带幼苗样品于2019年11月20日采集自福建省连江县官坞海洋海产开发有限公司(26°19′27″N, 119°48′33″E)。取10 cm脱苗病海带苗帘, 装入50 mL无菌离心管中, 并立即放入装有冰袋的保温箱中, 在12 h内运送到实验室。采集样品设置三个重复。
1.2 细菌分离将脱苗病海带幼苗带回实验室后, 使用无菌海水冲洗去除表面杂质, 将三段苗帘分别放入装有无菌玻璃珠与30 mL无菌海水的50 mL无菌离心管中。震荡5 min后收集悬液, 悬液分别稀释10–1, 10–2, 10–3, 10–4及10–5五个梯度。各取100 μL涂布至1/2 2216E培养基及2216E培养基[26], 放入25 ℃培养箱培养24 h。每个重复挑取细菌8~9株, 采用平板三区划线的方式进行分离纯化, 每株细菌至少纯化3次。将纯化后的菌株储存在30%(v/v)的甘油溶液中, −80 ℃保存。
1.3 复染实验及显微观察复染实验的海带雌、雄配子体A24来自于中国海洋大学海洋生命学院藻类分子病理学实验室保存的品系。首先活化、扩增保存的雌、雄配子体。将雌、雄配子体放入灭菌的三角瓶中利用超声破碎机破碎配子体(300 W, 4 min), 而后加入无菌海水及营养盐(营养盐的终浓度为: 硝酸钾200 μmol/L, 磷酸二氢钾20 μmol/L)。将装有配子体的三角瓶放入摇床中(10℃, 150 r/min), 光照30 µE·m2·s−1(24 h持续光照)的条件下连续培养3周。期间每周更换一次无菌海水。
将纯化后的菌株进行活化, 取60 μL菌液涂布至平板培养(Zobell 2216E培养基与 1/2 Zobell 2216E培养基), 培养48 h后长出菌落。取活化的菌落至Zobell 2216E液体培养基, 振荡培养箱培养(25 ℃, 120 r/min), 各菌株分别摇至对数期(0.7<OD<1)。取活化好的海带雌、雄配子体悬浮液(1 000 μL)分别放入无菌24孔板(生工, 上海), 添加菌液(侵染体系菌悬液浓度为1.0×107 CFU/mL), 以无菌海水作为对照, 每组设置三个重复。然后在10℃、90 µE·m2·s−1(光照∶黑暗=12∶12 h)下培养35 h进行初筛。筛选出致病菌株再次进行复染, 然后每3 h、9 h、24 h在显微镜下观察病症。
1.4 致病菌扫描电镜、透射电镜的形态学观察将致病菌接种在Zobell 2216E液体培养基中活化至稳定期(25 ℃, 120 r/min), 取10 mL活化好的菌液4 000 r/min离心20 min, 使用无菌枪头吸除上清液, 对沉淀细菌进行后续操作。
扫描电镜样品制备。1)固定: 使用2.5%戊二醛(天津博迪化工股份有限公司)固定4 h, 0.1 mol/L磷酸缓冲液漂洗。1%锇酸[Johnson matthey chemicals ltd osmic acid(99.6%)crystals batch 081101]固定90 min, 0.1 mol/L磷酸缓冲液漂洗。2)脱水: 酒精梯度脱水, 在30%、50%、70%、80%、100%依次脱水, 其中100%酒精2次, 每次10 min。3)置换: 乙酸异戊脂(天津巴斯夫化工有限公司)置换, 50%一次, 100%两次。4)喷金: Eiko公司XD-1型二氧化碳临界点干燥器干燥, Eiko公司IB-3型离子镀金仪喷金镀膜。5)TESCAN公司VEGA3扫描电镜观察。
透射电镜样品制备。1)固定: 使用2.5%戊二醛4 ℃固定4 h, PBS缓冲液冲洗3次, 每次10 min。1%锇酸4℃固定2 h。PBS缓冲液冲洗3次, 每次10 min。2)脱水: 酒精梯度脱水, 在30%、50%、70%、90%、100%依次脱水, 其中100%酒精2次, 每次10 min。3)包埋: Epon812环氧树脂包埋, 37 ℃、45 ℃、65 ℃温箱固化, 每级温度24 h。4)半薄切片定位: UltracutE超薄切片机超薄切片。5)染色: 醋酸双氧铀硝酸铅染色。6)JEOL公司JEM—1200EX透射电镜观察。
1.5 菌株的分子鉴定致病菌株的分子鉴定参照Fisher等人的方法[27]。DNA提取采用Plant DNA kit试剂盒(TIANGEN, China), 按照厂家说明书操作。16S rDNA的PCR扩增使用通用引物(7F: AGAGTTTGATCCTGGCTCA; 1429R: GGTTACCTTGTTACGACTT)[28]。扩增片段由北京擎科生物科技有限公司测序。将致病菌的16S rDNA全长序列与NCBI数据库(https://blast.ncbi.nlm. nih.gov)的细菌的16S rDNA序列进行比对, 进行分子鉴定。从NCBI数据库(www. ncbi.nlm.nih. Gov)中检索相关菌株的16S rDNA序列, 并使用Clustal X 1.81程序进行比对[29]。基于邻接法建系统发育树[30]。采用MEGA X软件进行系统发育分析[31-32]。
1.6 采用Biolog Gen III孔板进行表型分析使用Biolog微生物鉴定系统(Biolog, Hayward, USA)对致病菌的生理指纹图谱进行分类。将致病菌菌株在Zobell 2216E培养基上划线培养25 ℃培养24 h。按照用户指南制备菌悬液, 刮取菌落使其在接种液中悬浮, 运用浊度仪测定细菌悬液的透光率为95%。在每个Gen III孔板接种100 µL悬浮液, 25℃孵育24 h。利用标准化的Micro station TM系统(Biolog Inc., USA), 设置为600 nm, Biolog结果自动生成。然后在Biolog GEN III数据库中进行结果分析。
2 结果 2.1 脱苗病海带幼苗形态的显微观察脱苗病的症状为: 由于海带幼苗从苗帘上脱落, 导致苗帘呈现秃斑空白的区域(图1)。显微观察显示: 脱苗病海带幼苗有明显的白化、病烂的症状, 藻体上覆盖大量的硅藻(图2), 组织已经溃烂(图3)。
 |
| 图 1 脱苗病海带苗帘(比例尺为2 cm) Fig. 1 Falling-off diseased S. japonica sporeling curtains (Scale bar: 2 cm) |
 |
| 图 2 脱苗病海带幼苗(比例尺为125 μm) Fig. 2 Falling-off diseased S. japonica sporelings (Scale bar: 125 μm) |
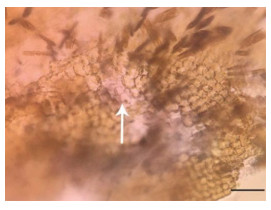 |
| 图 3 显微镜下脱苗病海带幼苗的组织形态(比例尺为25 μm) Fig. 3 Microscopic histological morphology of falling-off diseased S. japonica sporelings (Scale bar: 25 μm) |
从脱苗病海带幼苗共分离出49株附生细菌, 其中Zobell 2216E培养基25株, 1/2 Zobelle 2216E培养基24株。将这49株菌分别侵染海带雌、雄配子体, 经过初筛后, 选出3株可导致海带雌雄配子体产生绿烂病的致病菌。重复复染实验后, 挑选出一株致病力强的菌株LJ2-4。
复染的显微观察结果如图4-图15所示。与健康雌、雄配子体对照相比(图4, 图6), 复染3 h时, 雌、雄配子体无明显变化(图5, 图7); 复染9 h时, 与对照相比(图8, 图10), 受染雌、雄配子体均出现白化现象(图9, 图11); 复染24 h后, 雌、雄配子体白化现象加剧, 并伴有轻微变绿的现象(图13, 图15)。对照组中(图12, 图14), 雌、雄配子体24 h后未观察到白化和细胞变绿的现象。
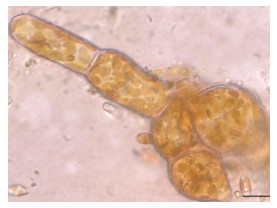 |
| 图 4 3 h雌配子体对照(比例尺为10 μm) Fig. 4 Control groups of female gametophytes at 3 h (Scale bar: 10 μm) |
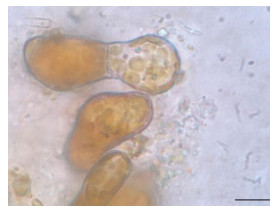 |
| 图 5 复染3 h后的雌配子体(比例尺为10 μm) Fig. 5 Reinfected healthy female gametophyte at 3 h (Scale bar: 10 μm) |
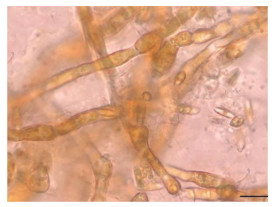 |
| 图 6 3 h雄配子体对照(比例尺为10 μm) Fig. 6 Control groups of male gametophytes at 3 h (Scale bar: 10 μm) |
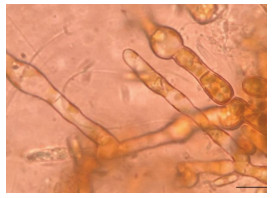 |
| 图 7 复染3 h后的雄配子体(比例尺为10 μm) Fig. 7 Reinfected healthy male gametophyte at 3 h (Scale bar: 10 μm) |
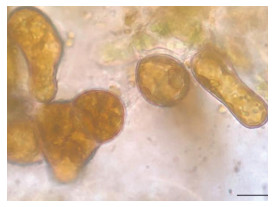 |
| 图 8 9 h雌配子体对照(比例尺为10 μm) Fig. 8 Control groups of female gametophytes at 9 h (Scale bar: 10 μm) |
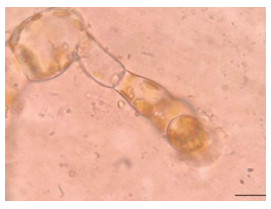 |
| 图 9 复染9 h后的雌配子体(比例尺为10 μm) Fig. 9 Reinfected healthy female gametophyte at 9 h (Scale bar: 10 μm) |
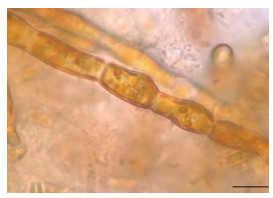 |
| 图 10 9 h雄配子体对照(比例尺为10 μm) Fig. 10 Control groups of male gametophytes at 9 h (Scale bar: 10 μm) |
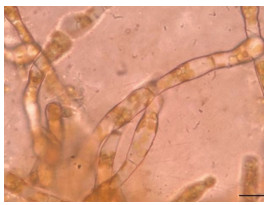 |
| 图 11 复染9 h后的雄配子体(比例尺为10 μm) Fig. 11 Reinfected healthy male gametophyte at 9 h (Scale bar: 10 μm) |
 |
| 图 12 24 h雌配子体对照(比例尺为10 μm) Fig. 12 Control groups of female gametophytes at 24 h (Scale bar: 10 μm) |
 |
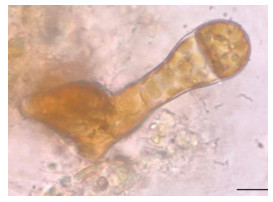
| 图 13 复染24 h后的雌配子体(比例尺为10 μm) Fig. 13 Reinfected healthy female gametophyte at 24 h (Scale bar: 10 μm) |
 |
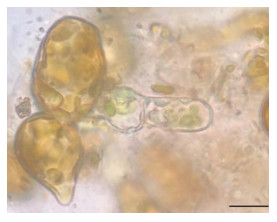
| 图 14 24 h雄配子体对照(比例尺为10 μm) Fig. 14 Control groups of male gametophytes at 24 h (Scale bar: 10 μm) |
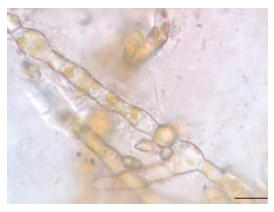 |
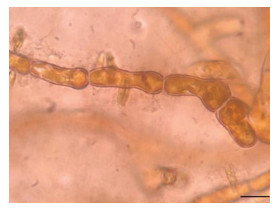
| 图 15 复染24 h后的雄配子体(比例尺为10 μm) Fig. 15 Reinfected healthy male gametophyte at 24 h (Scale bar: 10 μm) |
LJ2-4菌落呈浅橙色, 直径1~2 mm, 呈边缘完整的圆形菌落(图16), 革兰氏阳性菌。此外, 扫描电镜(图17)和透射电镜(图18)的结果表明, LJ2-4菌株为球形/棒状菌株, 长0.5~1.2 µm, 宽0.4~0.7 µm, 无鞭毛。
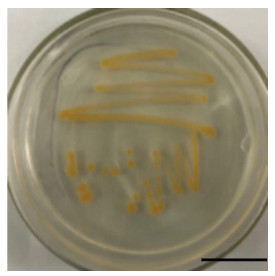 |
| 图 16 Zobell 2216E培养基上的LJ2-4菌落形态(比例尺为2 cm) Fig. 16 LJ2-4 colonies on Zobell 2216E culture media (Scale bar: 2 cm) |
 |
| 图 17 扫描电镜观察LJ2-4的形态(比例尺为1 µm) Fig. 17 Morphology of LJ2-4 observed by SEM (Scale bar: 1 μm) |
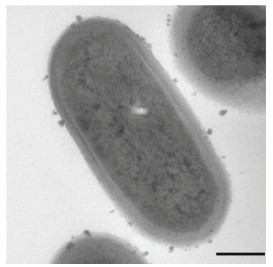 |
| 图 18 透射电镜观察LJ2-4的形态(比例尺为200 nm) Fig. 18 Morphology of LJ2-4 observed by TEM (Scale bar: 200 nm) |
LJ 2-4的16S rDNA序列全长1 467 bp。在NCBI数据库中对其16S rDNA序列进行比对后, 其与Planococcus okeanokoites IFO 12536T的相似性为99.45%。选择亲缘关系相近的21株细菌, 基于它们的16S rDNA序列构建了系统进化树(图19)。在系统发育树中, LJ 2-4明显与标准菌株Planococcusokeanokoites IFO 12536T聚类。LJ2-4的16S rDNA序列已提交NCBI数据库, 登录号为MW699592。
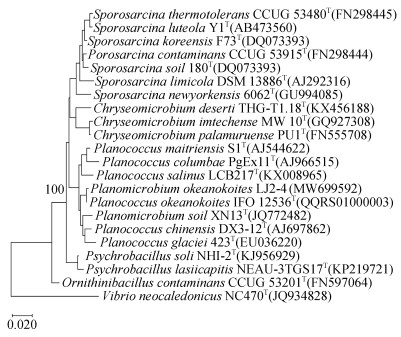 |
| 图 19 LJ2-4的系统发育树 Fig. 19 Phylogenetic tree of LJ2-4 注: 基于16S rDNA序列的系统发育树展示LJ2-4与其他近缘种相邻连接的位置, 以Vibrio neocaledonicus为外源菌群。 |
从LJ2-4复染后绿烂的海带雌、雄配子体分离出来的菌株, 经16S rDNA测序、比对, 证明与LJ2-4是同一菌株。符合科赫法则, 说明LJ2-4是在实验室条件下可引起健康海带雌、雄配子体发生白化病的条件致病菌。
2.5 Biolog GEN III孔板测试的生理生化特征使用Biolog Gen III型微孔板检测LJ2-4的生化生理特性。结果展示了LJ2-4与碳水化合物、氨基酸和其他化学衍生物的作用。其中代谢的碳水化合物有D-Cellobiose、D-Sorbitol、α-D-Lactose、Gentiobiose、N-Acetyl-D-Gal actosamine、D-Arabitol、D-Trehalose和D-Fucose; 代谢的氨基酸、核苷酸包括N-Acetyl Neuraminic Acid、Inosine以及Acetic Acid; 该菌株对Sodium Butyrate、Potassium Tellurite、Nalidixic Acid、Lincomycin、Aztreonam以及Lithium Chloride敏感, 能耐受1%的NaCl。基于碳水化合物、氨基酸和其他化学衍生物的LJ2-4代谢图谱如表1所示。LJ2-4菌株的生理生化特征与Planococcus okeanokoites IFO 12536T相似。基于LJ2-4的16S rDNA序列以及生理生化特征, 将其命名为Planomicrobium okeanokoites LJ2-4。
| 与以下底物/实验反应呈阳性 | ||
| D-纤维二糖 | D-山梨醇 | α-D-乳糖 |
| 龙胆二糖 | N-乙酰-D-半乳糖胺 | D-阿拉伯糖醇 |
| D-海藻糖 | N-乙酰神经氨糖酸 | D-岩藻糖 |
| 丁酸钠 | 乙酰乙酸 | 亚碲酸钾 |
| 肌苷 | 乙酸 | 萘啶酸 |
| 林可霉素 | 氨曲南 | 氯化锂 |
| 1% 氯化钠 | ||
| 与以下底物/实验反应呈弱阳性 | ||
| L-半乳糖酸酯 | 8% 氯化钠 | 3-甲基葡萄糖 |
| D-葡萄糖二酸 | L-精氨酸 | 蔗糖 |
| 吐温40 | L-苹果酸 | 夫西地酸 |
| D-果糖 | D-甘露醇 | 葡罗酰胺 |
| 四唑蓝 | D-棉子糖 | D-葡糖酸 |
| 丙酸 | D-水杨苷 | L-岩藻糖 |
| 4% 氯化钠 | D-蜜二糖 | β-甲基-D-葡糖苷 |
| D-甘露糖 | 肌醇 | N-乙酰-β-D-甘露糖胺 |
| 四唑紫 | ||
| 与以下底物/实验反应呈阴性 | ||
| 明胶 | 甲酸 | 对羟基苯乙酸 |
| γ-氨基丁酸 | 丙酮酸甲酯 | α-羟基丁酸 |
| 甘氨酸-L-脯氨酸 | D-麦芽糖 | D-半乳糖 |
| L-丙氨酸 | α-酮丁酸 | 柠檬酸 |
| 粘酸 | L-焦谷氨酸 | D-丝氨酸 |
| 溴代丁二酸 | 果胶 | D-乳酸甲酯 |
| α-酮戊二酸 | L-乳酸 | β-羟基-D L-丁酸 |
| 水苏糖 | L-组氨酸 | L-谷氨酸 |
| 奎尼酸 | L-天冬氨酸 | D-葡萄糖-6-磷酸 |
| 糊精 | 溴酸钠 | L-丝氨酸 |
| D-丝氨酸 | N-乙酰-D-葡萄糖胺 | D-松二糖 |
| D-半乳糖醛酸 | D-苹果酸 | 盐酸胍 |
| pH 5 | α-D-葡萄糖 | 醋竹桃霉素 |
| D-葡糖醛酸 | D-果糖-6-磷酸 | 甘油 |
| 1% 乳酸钠 | 万古霉素 | L-鼠李糖 |
| pH 6 | 利福霉素 | D-天冬氨酸 |
| 硫酸四癸钠 | 米诺环素 | |
我们从脱苗病海带幼苗表面分离、鉴定出一株致病菌LJ2-4。在实验室条件下, 运用复染实验和科赫法则证实LJ2-4可在实验室条件下引起健康海带雌、雄配子体白化病。16S rDNA序列比对结果显示: 比LJ2-4与Planococcus okeanokoites IFO 12536T的相似度为99.45%, 且LJ2-4的特征符合Planomicrobium的生理生化特性, 我们将LJ2-4命名为Planomicrobium okeanokoites LJ2-4。
Planococcus okeanokoites IFO 12536T在历史上多次变换名称。1944年Zobell 和Upham[33]从海洋沉积物中分离出来一株细菌, 首次命名为Flavobacterium okeanokoites。Nakagawa等人[34]于1996年根据其细胞形态的特性以及DNA-rRNA杂交结果将Flavobacterium okeanokoites转至Planococcus属, 模式菌株为Planococcus okeanokoites IFO 12536T。Yoon等人[35]根据Planococcus okeanokoites的细胞会随着培养时间的延长, 由球状或短棒状细胞变成棒状细胞, 这是Planococcus物种所没有的形态学特征, 结合P. okeanokoites其他生理生化特征, Yoon等人建议将Planococcus okeanokoites归至Planomicrobium新属, 命名为Planomicrobium okeanokoites。Planomicrobium属的细胞呈棒状或球形/短棒状, 革兰氏阳性菌, 不形成孢子, 依靠一根极性鞭毛或周生鞭毛运动, 菌落的颜色为黄色、橙色或淡橙色, 为好氧菌。
目前, Planomicrobium属有11个种。2001年Yoon等人[35]从韩国传统发酵海洋食物中分离出Planomicrobium koreense sp., 并将Planococcus okeanokoites[34]和Planococcus mcmeekinii[36]转移到新属Planomicrobium。Dai等[37]从中国福建省东部沿海沉积物中分离出Planomicrobium chinense sp., 并将从南极洲麦克默多站的湖泊中分离出的Planococcus psychrophilus sp.[38]和在潮间带海滩沉积物中分离出的Planococcus alkanoclasticus sp.转移至Planomicrobium属[39]。2009年, Jung等人[40]从海洋日晒盐田中分离得到Planomicrobium flavidum sp., 并将从印度喜马拉雅山脉的冻原上分离出的Planococcus stackebrandtii sp.[41]移至Planomicrobium属。Zhang等人[42]从中国一号冰川中分离出Planomicrobium glaciei sp.。2014年, Luo等人[43]从内蒙古阿拉善国家地质公园土壤样品中分离到Planomicrobium soli sp.。Ramezani等人[44]在伊朗中部梅甘高盐湿地分离出一种新的微嗜盐细菌Planomicrobium iranicum sp.。
关于Planomicrobium 的功能, 知之甚少。1998年Junge等人发现Planomicrobium mcmeekinii具有将硝酸盐还原为亚硝酸盐的功能, 能生活在低盐的环境中[36]。P. chinense[37]以及P. glaciei[42]也具有还原硝酸盐的能力。Planomicrobium alkanoclasticus分离自原油污染的潮间滩沉积物, 它能够降解原油中C11 至 C33支链烷烃和正构烷烃, 但不能降解芳香烃[39]。迄今为止, 未见在海藻中分离获得Planomicrobium属物种以及该属细菌作为海藻致病菌的报道, P. okeanokoites的功能也不清楚。本文首次发现P. okeanokoites是诱导海藻配子体产生白化病的致病菌, 丰富了海带致病菌的信息。
通过传统的微生物分离纯化培养方法, 从各种病烂海带藻体上分离鉴定出多种致病菌, 包括Pseudoalteromonas sp.[9-10, 25]、Alteromonas sp.[22-23]、Vibrio sp.[25]。而16S rDNA扩增子高通量测序研究结果显示: 与健康幼苗比较, 在绿烂病幼苗附生菌群中占优势的有Granulosicoccus, Litorimonas, Tenacibaculum, 以及Blastopirellula[45], 这些属可能与海带幼苗绿烂病相关。Sulfitobacter、Loktanella以及Roseobacter在养殖海带绿烂组织中丰度较高, 表明这些类群可能是养殖期海带绿烂病的条件致病菌[46]。不同的结果可能是由于研究方法的不同以及不同的病害由不同的致病菌所诱导。此外, 目前由微生物分离纯化培养法和16S rDNA扩增子测序的获得的结果鉴海带致病菌无法进行比较, 因为这些结果针对的是不同的海带病害。因此, 为了对养殖海带的致病菌有更深、更全面的认识, 建议结合传统微生物分离纯化培养法和16S rDNA高通量测序两种实验方法来研究同一种海带病害。此外, 通过16S rDNA高通量测序鉴定出的潜在致病菌是否为真实致病菌还需要通过复染实验进行验证。
硅藻与海带苗是共生共存的, 在海带苗期, 始终把硅藻作为头号敌害, 对于硅藻的防控贯穿于从种海带培育到采苗以及育苗管理的每一个环节。通常在生产上只能通过洗苗来减少硅藻的数量, 以减少硅藻对海带苗生长最大限度的危害。本次脱苗病幼苗采自福建官坞海带育苗场。海带育苗场使用白色的维尼纶苗帘, 在育苗的早期更有利于硅藻的生长。从脱苗病幼苗藻体覆盖大量的硅藻推测, 硅藻是本次脱苗病暴发的原因之一。在实际育苗生产中, 硅藻的去除问题一直难以控制。主要有以下原因: 1)不同年份由于气候原因而造成自然海域中的硅藻品种和数量的不同与多寡, 直接影响海带育苗生产中硅藻品种和数量不同; 2)在苗期生产管理上, 如蓄水池的清洗, 水质过滤器的铺设, 洗苗人员的责任心, 育苗进回水的日常管理等都会直接影响硅藻的繁殖; 3)天气的影响, 育苗房由于使用年限久了, 下雨时苗房就会漏水, 如果遇到多台风和常常下雨的年份, 洗苗次数就会受到影响, 从而导致硅藻的数量大幅增加; 4)苗帘上幼苗密度的影响, 采苗密度和发育转化率直接决定了密度, 一旦出现密度偏低时, 决策上洗苗的程度也会减少以增加除苗量。也会导致硅藻的数量增加。
目前, 对养殖海带病害致病菌的研究还处于起步阶段, 养殖海带细菌性病害的主要问题是病害发生的复杂性。在不同的年份, 育苗期和海上养殖阶段病害的暴发变化多变, 表现出不同的病害症状。分离、鉴定海带致病菌的难点在于: 1)海带的致病菌多为条件致病菌, 其存在于健康的海带藻体上, 只有在养殖环境恶化的条件下, 如温度升高、光照强度变化等才会转变为致病菌; 2)只有大约1%的海带表面附生细菌是可以培养的, 运用传统的分离、纯化培养方法可能无法培养出真正的致病菌; 3)当疾病暴发时, 可能是多种致病菌共同作用导致的, 很难确定哪些细菌是第一个引起海带病害的致病菌。
4 结论由于海藻致病菌多为条件致病菌, 且运用传统微生物分离培养方法分离获得的致病菌在实验室培育几代后, 容易失去致病性, 导致无法深入地解析致病菌的致病机理。因此, 海藻致病菌的分离、鉴定仍是世界性难题。结合传统微生物分离培养方法及侵染实验, 本文从脱苗病海带幼苗藻体分离、鉴定出一株致病菌——Planococcus okeanokoites LJ2-4 (LJ2-4)。虽然LJ2-4在实验室条件下可使健康海带雌、雄配子体产生白化的病症, 但其是否是苗期脱苗病的致病菌, 需进一步实验验证。
| [1] |
SINGH R P, REDDY C R K. Seaweed-microbial interactions: key functions of seaweed-associated bacteria[J]. FEMS Microbiology Ecology, 2014, 88(2): 213-230. DOI:10.1111/1574-6941.12297 |
| [2] |
EGAN S, HARDERr T, BUEKE C, et al. The seaweed holobiont: understanding seaweed-bacteria interactions[J]. FEMS Microbiology Reviews, 2013, 37(3): 462-476. DOI:10.1111/1574-6976.12011 |
| [3] |
WANG G, LU B, LI S, et al. Microbial diseases of nursery and field-cultivated Saccharina japonica (Phaeophyta) in China[J]. Biocontrol Science and Technology, 2014, 145(1): 39-51. |
| [4] |
GACHON C M M, TÉLESPHORE S, STRITTMATTER M, et al. Algal diseases: spotlight on a black box[J]. Trends in Plant Science, 2010, 15(11): 633-640. DOI:10.1016/j.tplants.2010.08.005 |
| [5] |
EGAN S, FERNANDES N D, KUMAR V, et al. Bacterial pathogens, virulence mechanism and host defence in marine macroalgae[J]. Environmental Microbiology, 2014, 16(4): 925-938. DOI:10.1111/1462-2920.12288 |
| [6] |
EZURA Y, YAMAMOTO H, KIMURA T. Isolation of a marine bacterium that produces red-spots on the culture bed of Makonbu Laminaria japonica cultivation[J]. Nippon Suisan Gakkaishi, 1988, 54(4): 665-672. DOI:10.2331/suisan.54.665 |
| [7] |
YUMOTO I, YAMAGUCHI K, YAMADA K, et al. Relationship between bacterial flora and occurrence of the Alteromonas sp. the causative agent of red-spots on the culture bed of makonbu Laminaria japonica, in the coastal area of funka bay[J]. Bulletin of the Japanese Society of Scientific Fisheries, 1989, 55(11): 1907-1914. DOI:10.2331/suisan.55.1907 |
| [8] |
SAWABE T, EZURA Y, KIMURA T. Characterization of an alginolytic marine bacterium from decaying Rishiri kombu Laminaria japonica var. ochotensis[J]. Nippon Suisan Gakkaishi, 1992, 58(1): 141-145. DOI:10.2331/suisan.58.141 |
| [9] |
SAWABE T, MAKINO H, TATSUMI M, et al. Pseudoalteromonas bacteriolytica sp. nov. a marine bacterium that is the causative agent of red spot disease of Laminaria japonica[J]. International Journal of Systematic Bacteriology, 1998, 48(3): 769-774. DOI:10.1099/00207713-48-3-769 |
| [10] |
SAWABE T, NARITA M, TANAKA R, et al. Isolation of Pseudoalteromonas elyakovii strains from spot-wounded fronds of Laminaria japonica[J]. Nippon Suisan Gakkaishi, 2000, 66(2): 249-254. DOI:10.2331/suisan.66.249 |
| [11] |
VAIRAPPAN C S, SUZUKI M, MOTOMURA T, et al. Pathogenic bacteria associated with lesions and thallus bleaching symptoms in the Japanese kelp Laminaria religiosa Miyabe (Laminariales, Phaeophyceae)[J]. Hydrobiologia, 2001, 445(1): 183-191. |
| [12] |
Food and Agriculture Organization of the United Nations. The state of world fisheries and aquaculture 2016: Contributing to food security and nutrition for all[M]. Rome: FAO, 2016: 1-200.
|
| [13] |
GACHON C M M, TELESPHORE S, STRITTMATTER M, et al. Algal diseases: spotlight on a black box[J]. Trends in Plant Science, 2010, 15(11): 633-640. DOI:10.1016/j.tplants.2010.08.005 |
| [14] |
陈騳, 林光恒, 沈世泽. 褐藻酸降解菌的研究Ⅰ. 褐藻酸降解菌与褐藻酸酶对海带藻体的作用[J]. 海洋与湖沼, 1979, 10(4): 329-333,402. CHEN Du, LIN Guangheng, SHEN Shize. Studies on alginic acid-decomposing bacteria, I. Action of alginic acid-decomposing bacteria and alginase on Laminaria japonica[J]. Oceanologia et Limnologia Sinica, 1979, 10(4): 329-333,402. |
| [15] |
陈騳, 林光恒, 沈世泽. 褐藻酸降解菌的研究Ⅱ. 海带夏苗培育中褐藻酸降解菌与烂苗的关系[J]. 海洋与湖沼, 1981, 12(2): 133-137. CHEN Du, LIN Guangheng, SHEN Shize. Studies on alginic acid-decomposing bacteria, II. Rot disease of Laminaria summer sporeling caused by alginic acid- decomposing bacteria[J]. Oceanologia et Limnologia Sinica, 1981, 12(2): 133-137. |
| [16] |
王艳玲, 唐学玺, 杨震. 褐藻酸降解菌的筛选及其生长条件[J]. 中国水产科学, 2003, 10(1): 51-54. WANG Yanling, TANG Xuexi, YANG Zhen. The selection and growth conditions of alginic acid decomposing bacteria[J]. Journal of Fishery Sciences of China, 2003, 10(1): 51-54. |
| [17] |
刘成圣, 王丽丽, 王蒙, 等. 褐藻酸降解菌对海带感染能力差异性分析[J]. 海洋科学, 2002, 26(6): 44-47. LIU Chengsheng, WANG Lili, WANG Meng, et al. Difference analysis of infection activity of alginic acid decomposing bacteria infecting Laminaria japonica[J]. Scientia Marina, 2002, 26(6): 44-47. |
| [18] |
林伟, 张伟伟, 严小军, 等. 褐藻酸降解菌在海带(Laminaria japonica)幼苗藻体表面数量分布特点及其对海带回染的初步研究[J]. 海洋与湖沼, 2004, 35(6): 562-567. LIN Wei, ZHANG Weiwei, YAN Xiaojun, et al. Distribution and reinfection of alginic acid decomposing bacteria on juvenile Laminaria japonica[J]. Oceanologia et Limnologia Sinica, 2004, 35(6): 562-567. |
| [19] |
陈騳, 刘秀云, 刘秀珍, 等. 褐藻酸降解菌的研究Ⅳ. 褐藻酸降解菌在海带栽培区中的生态分布及其重要性[J]. 海洋与湖沼, 1986, 17(2): 137-143. CHEN Du, LIU Xiuyun, LIU Xiuzhen, et al. Studies on alginic acid decomposing bacteria IV. Distribution of alginic acid decomposing bacteria in Laminaria farm and its ecological significance[J]. Oceanologia et Limnologia Sinica, 1986, 17(2): 137-143. |
| [20] |
丁美丽. 环境因子对褐藻酸降解菌引起海带病烂影响研究[J]. 海洋学报(中文版), 1990, 12(2): 224-230. DING Meili, The influence of environmental factors on rot disease of Laminaria caused by alginic acid decomposing bacteria[J]. Acta Oceanologica Sinica, 1990, 12(2): 224-230. |
| [21] |
WU C. Cultivation of temperate seaweeds in the Asia Pacific region: Regional Workshop on the Culture and Utilization of Seaweeds[C]. Cebu City (Philippines): FAO, 1990.
|
| [22] |
WANG G, LIN W, ZHANG L, et al. Programmed cell death in Laminaria japonica (Phaeophyta) tissues infected with alginic acid decomposing bacterium[J]. Progress in Natural Science, 2004, 14(12): 1064-1068. DOI:10.1080/10020070412331344811 |
| [23] |
WANG G, WEI L, YAN X, et al. Study on the enzymatic activity of Caspase-3 in response to alginic acid decomposing bacteria in Laminaria japonica Aresch. (Phaeophyta)[J]. High Technol Lett (English Edition), 2005, 11(1): 80-84. |
| [24] |
WANG Y, TANG X X, YANG Z, et al. Effect of alginic acid decomposing bacterium on the growth of Laminaria japonica (Phaeophyceae)[J]. Journal of Environmental Sciences, 2006, 18(3): 543-551. |
| [25] |
WANG G, LI S, LI Y, et al. Phylogenetic analysis of epiphytic marine bacteria on Hole-Rotten diseased sporophytes of Laminaria japonica[J]. Journal of Applied Phycology, 2008, 20(4): 403-409. DOI:10.1007/s10811-007-9274-4 |
| [26] |
YING J J, ZHANG S L, HUANG C Y, et al. Algicoccus marinus gen. nov. sp. nov. , a marine bacterium isolated from the surface of brown seaweed Laminaria japonica. Archives of Microbiology, 2019, 201(7): 943-950.
|
| [27] |
FISHER M M, WILCOX L W, GRAM L E. Molecular characterization of epiphytic bacterial communities on charophycean green algae[J]. Applied and Environmental Microbiology, 1998, 64(11): 4384-4389. DOI:10.1128/AEM.64.11.4384-4389.1998 |
| [28] |
MORENO C, ROMERO J, ESPEJO R T. Polymorphism in repeated 16S rRNA genes is a common property of type strains and environmental isolates of the genus Vibrio[J]. Microbiology, 2002, 148(4): 1233-1239. DOI:10.1099/00221287-148-4-1233 |
| [29] |
THOMPSON J D, GIBSON T J, PLEWNIAK F, et al. The CLUSTAL_X Windows Interface: Flexible Strategies for Multiple Sequence Alignment Aided by Quality Analysis Tools[J]. Nucleic Acids Research, 1997, 25(24): 4876-4882. DOI:10.1093/nar/25.24.4876 |
| [30] |
TAMURA K, NEI M. Estimation of the number of nucleotide substitutions in the control region of mitochondrial DNA in humans and chimpanzees.[J]. Molecular Biology & Evolution, 1993, 10(3): 512-526. |
| [31] |
FELSENSTEIN J. Confidence limits on phylogenies: An approach using the bootstrap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.2307/2408678 |
| [32] |
KUMAR S, STECHER G, LI M, et al. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms[J]. Molecular Biology & Evolution, 2018, 35(6): 1547-1549. |
| [33] |
ZOBELL C E, UPHAM H C. A list of marine bacteria including descriptions of sixty new species[M]. Berkeley, Los Angeles: University of California Press, 1994, 5: 239-292.
|
| [34] |
NAKAGAWA Y, SAKANE T, YOKOTA A. Emendation of the Genus Planococcus and Transfer of Flavobacterium okeanokoites Zobell and Upham 1944 to the Genus Planococcus as Planococcus okeanokoites comb. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 1996, 46(4): 866-870. |
| [35] |
YOON J H, KANG S S, LEE K C, et al. Planomicrobium koreense gen. nov. sp. nov. a bacterium isolated from the Korean traditional fermented seafood jeotgal, and transfer of Planococcus okeanokoites (Nakagawa et al. 1996) and Planococcus mcmeekinii (Junge et al. 1998) to the genus Planomicrobium[J]. International Journal of Systematic & Evolutionary Microbiology, 2001, 51(4): 1511-1520. |
| [36] |
JUNGE K, GOSINK J, HOPPE H, et al. Arthrobacter, Brachybacterium and Planococcus isolates identified from Antarctic Sea Ice Brine. description of Planococcus mcmeekinii, sp. nov.[J]. Systematic & Applied Microbiology, 1998, 21(2): 306-314. |
| [37] |
DAI X. Planomicrobium chinense sp. nov. isolated from coastal sediment, and transfer of Planococcus psychrophilus and Planococcus alkanoclasticus to Planomicrobium as Planomicrobium psychrophilum comb. nov. and Planomicrobium alkanoclasticum comb. nov.[J]. International Journal of Systematic & Evolutionary Microbiology, 2005, 55(2): 699. |
| [38] |
REDDY G, PRAKASH J, VAIRAMANI M, et al. Planococcus antarcticus and Planococcus psychrophilus spp. nov. isolated from cyanobacterial mat samples collected from ponds in Antarctica[J]. Extremophiles, 2002, 6(3): 253-261. DOI:10.1007/s00792-001-0250-7 |
| [39] |
ENGELHARDT M A, DALY K, SWANNELL R P J, et al. Isolation and characterization of a novel hydrocarbon- degrading, Gram-positive bacterium, isolated from intertidal beach sediment, and description of Planococcus alkanoclasticus sp. nov.[J]. Journal of Applied Microbiology, 2001, 90(2): 238-238. |
| [40] |
JUNG Y T, KANG S J, OH T K, et al. Planomicrobium flavidum sp. nov. isolated from a marine solar saltern, and transfer of Planococcus stackebrandtii Mayilraj et al. 2005 to the genus Planomicrobium as Planomicrobium stackebrandtii comb. nov.[J]. International Journal of Systematic & Evolutionary Microbiology, 2009, 59(12): 2929. |
| [41] |
MAYILRAJ S, PRASAD G S, SURESH K, et al. Planococcus stackebrandtii sp. nov. isolated from a cold desert of the Himalayas, India[J]. International Journal of Systematic & Evolutionary Microbiology, 2005, 55(1): 91-94. |
| [42] |
ZHANG D C, LIU H C, XIN Y H, et al. Planomicrobium glaciei sp. nov. a psychrotolerant bacterium isolated from a glacier[J]. International Journal of Systematic & Evolutionary Microbiology, 2009, 59(6): 1387-1390. |
| [43] |
LUO X, ZHANG J, LI D, et al. Planomicrobium soli sp. nov. isolated from soil[J]. International Journal of Systematic & Evolutionary Microbiology, 2014, 64(8): 2700-2705. |
| [44] |
RAMEZANI M, MAHDI M, MOHAMMAD P, et al. Planomicrobium iranicum sp. nov., a novel slightly halophilic bacterium isolated from a hypersaline wetland[J]. International Journal of Systematic and Evolutionary Microbiology, 2019, 69(5): 1433-1437. DOI:10.1099/ijsem.0.003332 |
| [45] |
LI J, PANG S, SHAN T, et al. Changes of microbial community structures associated with seedlings of Saccharina japonica at early stage of outbreak of green rotten disease[J]. Journal of Applied Phycology, 2020, 32(2): 1323-1327. DOI:10.1007/s10811-019-01975-7 |
| [46] |
ZHANG R, CHANG L R, XIAO L Y, et al. Diversity of the epiphytic bacterial communities associated with commercially cultivated healthy and diseased Saccharina japonica during the harvest season[J]. Journal of Applied Phycology, 2019, 32(3): 2071-2080. |
 2023, Vol. 47
2023, Vol. 47


