文章信息
- 李向乾, 李晓东, 林永豪, 张伟, 徐晗, 杨桂琴, 路玮静, 俞道进, 陈新华. 2023.
- LI Xiang-qian, LI Xiao-dong, LIN Yong-hao, ZHANG Wei, XU Han, YANG Gui-qin, LU Wei-jing, YU Dao-jin, CHEN Xin-hua. 2023.
- 三沙湾大肠杆菌和肠球菌抗生素耐药性分析
- Antibiotic resistance of Escherichia coli and Enterococcus spp. in Sansha Bay, China
- 海洋科学, 47(7): 138-148
- Marine Sciences, 47(7): 138-148.
- http://dx.doi.org/10.11759/hykx20220524003
-
文章历史
- 收稿日期:2022-05-24
- 修回日期:2022-10-26
2. 福建农林大学 动物科学学院 蜂学学院 福建省中西兽医结合与动物保健重点实验室, 福建 福州 350002
2. Fujian Key Laboratory of Traditional Chinese Veterinary Medicine and Animal Health, College of Animal Sciences (Bee Sciences), Fujian Agriculture and Forestry University, Fuzhou 350002, China
近年来抗生素耐药已成为一种全球性的公共卫生威胁[1]。抗生素具有抑菌杀菌、促生长等多种功能, 被大量应用于医疗保健与畜牧养殖[2]。但抗生素被使用后约85%以上通过原药或代谢产物的形式进入环境[3], 其中大部分进入水体导致耐药菌与耐药基因的出现与扩散, 给人类健康与畜牧养殖带来不利影响[4-5]。例如我国东部湖泊就存在着不同程度抗生素污染, 并且与人类活动存在相关性[6]。抗生素或耐药菌/基因还会随着陆地径流进入海洋[7-9], 使近海可能成为抗生素耐药的重灾区。目前已有研究表明我国海域分离的细菌对多种抗生素具有耐药性并且多重耐药情况严重[10]。作为世界上最大的海水养殖国家, 我国近海密集养殖中抗生素的大量使用也会导致当地细菌耐药显著增加[11-12]。华南和华东部分沿海养殖水体均检测出耐药菌[13-14]。水体耐药菌可能通过鱼类携带[15], 在食物[16]、人体以及肠道菌群中传递[17], 对人类健康造成威胁。
大肠杆菌和肠球菌广泛分布于各种水体, 因其能够表征人类活动对环境的影响, 常被作为环境指示菌监测水域耐药情况。在大连海水浴场中大肠杆菌表现出对8种抗生素不同程度耐药[18]; 泰安滦河中ESBLs大肠杆菌同样表现出多重耐药现象[19]; 闽江流域中肠球菌则表现出对土霉素和红霉素的高耐药性[20]。宁德三沙湾水文条件稳定, 水体与外界交换较少[21], 是中国最大的大黄鱼养殖区。密集养殖过程中用药及人类活动可能导致严重的耐药菌污染, 但目前对该地水体中细菌耐药性探究不多。本实验通过分离当地水源性环境指示菌(大肠杆菌和肠球菌), 检测其对常用抗生素的耐药性及相关耐药基因, 从而了解该海域环境中细菌耐药情况。实验结果对该海域水质管理以及渔业健康养殖有着重要意义。
1 材料和方法 1.1 样品采集与处理在三沙湾设立13个采样站位(图 1, 表 1), 于2021年6月20日采集各站位表层水样(0.5 m)250 mL密封保存, 24 h内转移至实验室内处理。使用0.22 μm孔径滤膜过滤(负压≈0.8 MPa)收集细菌并转移至BHI肉汤中增菌培养8 h(37 ℃、200 r/min, 下同)。
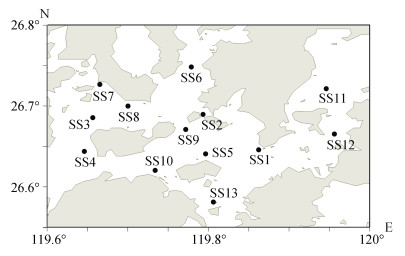 |
| 图 1 三沙湾采样点站位图 Fig. 1 Sampling locations map of Sansha Bay |
| 采样站位 | SS1 | SS2 | SS3 | SS4 | SS5 | SS6 | SS7 | SS8 | SS9 | SS10 | SS11 | SS12 | SS13 |
| 经度 | 119.8623 | 119.7932 | 119.6564 | 119.6458 | 119.7963 | 119.7786 | 119.6651 | 119.7 | 119.7717 | 119.7336 | 119.9461 | 119.9561 | 119.8059 |
| 纬度 | 26.6457 | 26.6898 | 26.6855 | 26.6439 | 26.6408 | 26.7486 | 26.7267 | 26.7 | 26.6710 | 26.6203 | 26.7215 | 26.6654 | 26.5812 |
| 大肠杆菌 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | — | — | 检出 |
| 肠球菌 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | 检出 | — | 检出 | 检出 |
细菌划线接种于SBM(肠球菌选择培养基CM0377)培养基和MAC培养基, 37 ℃恒温培养18~24 h后挑取典型菌落。纯化后的肠球菌和大肠杆菌菌株分别接种到BHI与LB肉汤中增菌培养, 取1 mL菌液水煮法提取DNA, 进行PCR菌种鉴定。
据PremixTaq说明书配制反应体系, 反应条件: 98 ℃预变性2 min, 98 ℃变性10 s, 退火10 s, 72 ℃延伸10 s, 33个循环, 最后72 ℃延伸2 min。引物参考GenBank公布大肠杆菌和肠球菌16S rDNA序列设计(大肠杆菌F: CGATTCCGTTTCAGGGTT, R: TTTCT GATAGGACCGAGCAT; 肠球菌F: GTGTCGCTGAT GGATGG, R: GCAAGCCGAACTGAGAGA)。PCR产物在135 V、120 mA条件下进行琼脂糖凝胶电泳后观察拍照并保存。将已鉴定菌株和无菌LB(BHI)甘油按照1︰1, –20 ℃保存。
1.3 药敏试验使用美国临床和实验室标准协会(CLSI)微量肉汤稀释法测定分离菌株对几种常用抗生素(大肠杆菌: 庆大霉素、氨苄西林、头孢唑啉、头孢噻肟、环丙沙星、四环素、呋喃妥因、氯霉素; 肠球菌: 氨苄西林、万古霉素、红霉素、四环素、环丙沙星、氯霉素、利福平、呋喃妥因; 均购自中国药品生物制品检定所)的最小抑菌浓度(MIC)。质控菌株为粪肠球菌ATCC29212与大肠埃希氏菌ATCC25922, 均购自中国药品生物制品检验所。分离菌株与质控菌分别接种到LB肉汤(大肠杆菌)或BHI肉汤(肠球菌)中增菌培养4 h后稀释成OD值0.08~0.1之间, 即0.5麦氏比浊(1×108~2×108 CFU/mL)备用。96孔平板每孔预先加入100 μL MH肉汤。第一列分别加入100 μL 512 μg/mL抗生素, 倍比稀释至0.5 μg/mL抗生素, 第12列不加药物做阳性对照。接种时菌液再稀释10倍, 每孔加10 μL, 第11列不加菌液做阴性对照。培养箱37 ℃培养24 h后, 在光源下第一个澄清孔即为抗生素对细菌的MIC。
1.4 耐药基因检测检测基因及其引物(表 2)。根据1.1×CataAmp Taq Plus Mix说明书配置20 μL反应体系。反应条件(参照北京开拓赛思生物技术有限公司提供说明书)如下: 初始变性温度98 ℃, 3 min。变性温度98 ℃, 10 s, 退火温度60 ℃, 10 s, 延伸温度72 ℃, 20 s/kb, 循环35次。PCR产物进行琼脂糖凝胶电泳成像观察。
| 菌种 | 抗生素种类 | 基因 | 基因功能 | 引物序列(5′ -3′) | 片段长度/bp | 参考文献 |
| 大肠杆菌 | 硝基呋喃类 | nfsA | 编码氧不敏感的硝基还原酶nfsA | F: ATTTTCTCGGCCAGAAGTGC R: AGAATTTCAACCAGGTGACC |
1036 | [22] |
| nfsB | 编码氧不敏感的硝基还原酶nfsB | F: CCCGCTAAATCTTCAACCTG R: AAAAGAGTGCGTCCAGGCTA |
913 | |||
| 喹诺酮类 | oqxA | 与oqxB基因共同编码外排泵OqxAB | F: CTCGGCGCGATGATGCT R: CCACTCTTCACGGGAGACGA |
596 | ||
| oqxB | 与oqxA基因共同编码外排泵OqxAB | F: TTCTCCCCCGGCGGGAAGTAC R: CTCGGCCATTTTGGCGCGTA |
513 | [23] | ||
| qnrS | 保护DNA促旋酶和拓扑异构酶Ⅳ | F: ATGGAAACCTACAATCATAC R: AAAAACACCTCGACTTAAGT |
492 | [24] | ||
| 氯霉素类 | cat1 | 产生氯霉素乙酰转移酶 | F: AGTTGCTCAATGTACCTATAACC R: TTGTAATTCATTAAGCATTCTGCC |
547 | [25] | |
| cmlA | 编码外排泵蛋白 | F: AAGTCTTTCTGGGGCTTCGG R: TCACACGCCCCATAAAACGA |
510 | |||
| β-内酰胺类 | blaTEM | 编码TEM型β-内酰胺酶 | F: ATGAGTATTCAACATTTCCG R: CAATGCTTAATCAGTGAGG |
859 | ||
| 四环素类 | tetA | 编码外排泵蛋白 | F: GTAATTCTGAGCACTGTCGC R: CTGCCTGGACAACATTGCTT |
957 | [22] | |
| tetB | 编码外排泵蛋白 | F: TTGGTTAGGGGCAAGTTTTG R: GTAATGGGCCAATAACACCG |
659 | |||
| 肠球菌 | tetM | 编码核糖体保护蛋白 | F: ACAGAAAGCTTATTATATAAC R: TGGCGTGTCTATGATGTTCAC |
171 | [5] | |
| tetL | 编码外排泵蛋白 | F: TCGTTAGCGTGCTGTCATTC R: GTATCCCACCAATGTAGCCG |
267 | |||
| tetS | 编码核糖体保护蛋白 | F: CATAGACAAGCCGTTGACC R: ATGTTTTTGGAACGCCAGAG |
667 | |||
| TetG | 编码外排泵蛋白 | F: GCTCGGTGGTATCTCTGCTC R: AGCAACAGAATCGGGAACAC |
468 | [10] | ||
| 大环内酯类 | mefA | 编码外排泵蛋白 | F: AGTATCATTAATCACTAGTGC R: TTCTTCTGGTACTAAAAGTGG |
400 | ||
| ermA | 编码RNA甲基化酶 | F: TCTAAAAAGCATGTAAAAGAA R: CTTCGATAGTTTATTAATATTAGT |
645 | [26] | ||
| ermB | 编码RNA甲基化酶 | F: GAAAAGAGTACTCAACCAAATA R: AGTAACGGTACTTAAATTGTTTAC |
639 | [5] | ||
| 氟喹诺酮类 | gyaA | 降低药物亲和度 | F: CGGGATGAACGAATTGGGTGTGA R: AATTTTACTCATACGTGCTTCGG |
241 | ||
| 万古霉素 | vanB | 产生D-丙氨酰-D-乳酸二肽替代D-丙氨酰-D-丙氨酸 | F: ATGGGAAGCCGATAGTC R: GATTTCGTTCCTCGACC |
635 |
使用Surfer软件对耐药分布进行空间作图, 分析耐药菌在空间的分布情况(表 3, 表 4)。多重耐药性: 细菌对3种或3种以上抗生素耐药[27]。耐药率: 耐药菌株数量与分离菌株数量之比。多重耐药率: 多重耐药菌株数量与分离菌株数量之比。
| 耐药程度 | 敏感 | 中介 | 耐药 |
| 庆大霉素 | ≤4 | 8 | ≥16 |
| 氨苄西林 | ≤8 | 16 | ≥32 |
| 头孢唑啉 | ≤2 | 4 | ≥8 |
| 头孢噻肟 | ≤1 | 2 | ≥4 |
| 环丙沙星 | ≤1 | 2 | ≥4 |
| 四环素 | ≤4 | 8 | ≥16 |
| 呋喃妥因 | ≤32 | 64 | ≥128 |
| 氯霉素 | ≤8 | 16 | ≥32 |
| 耐药程度 | 敏感 | 中介 | 耐药 |
| 氨苄西林 | ≤8 | ≥16 | |
| 万古霉素 | ≤4 | 8~16 | ≥32 |
| 红霉素 | ≤0.5 | 1~4 | ≥8 |
| 四环素 | ≤4 | 8 | ≥16 |
| 环丙沙星 | ≤1 | 2 | ≥4 |
| 氯霉素 | ≤8 | 16 | ≥32 |
| 利福平 | ≤1 | 2 | ≥4 |
| 呋喃妥因 | ≤32 | 64 | ≥128 |
由表 5可知, 实验分离到11株大肠杆菌和12株肠球菌。大肠杆菌对环丙沙星最敏感, 其次是庆大霉素。大肠杆菌对其余抗生素均有一定的耐药性, 从高到低对四环素、氨苄西林、氯霉素、头孢唑啉、头孢噻肟、呋喃妥因耐药率分别为36.36%、36.36%、27.27%、27.27%、18.18%、18.18%, 其中对氨苄西林、四环素、呋喃妥因、氯霉素的耐药水平较高。肠球菌中对利福平耐药率最高, 为83.3%, 其次是呋喃妥因和红霉素, 耐药率分别为66.67%和50%。肠球菌对氨苄西林、万古霉素和氯霉素最敏感, 耐药率为0%。肠球菌对呋喃妥因、四环素、利福平的耐药水平高, MIC≥64 μg/mL比例高于50%, 四环素MIC≥128 μg/mL为33.33%。肠球菌对其余5种抗生素的耐药水平均较低, 均在16 μg/mL以下。
| 菌种 | 耐药程度 | 四环素 | 氨苄西林 | 氯霉素 | 头孢唑啉 | 头孢噻肟 | 呋喃妥因 | 庆大霉素 | 环丙沙星 |
| 大肠杆菌 | 耐药 | 36.36% | 36.36% | 27.27% | 27.27% | 18.18% | 18.18% | 9.09% | 0 |
| 中介 | 0 | 9.09% | 0 | 18.18% | 0 | 0 | 0 | 9.09% | |
| 敏感 | 63.64% | 54.55% | 72.73% | 54.55% | 81.82% | 81.82% | 90.91% | 90.91% | |
| 肠球菌 | 耐药程度 | 利福平 | 呋喃妥因 | 红霉素 | 环丙沙星 | 四环素 | 万古霉素 | 氯霉素 | 氨苄西林 |
| 耐药 | 83.33% | 66.67% | 50.00% | 33.33% | 33.33% | 0 | 0 | 0 | |
| 中介 | 8.34% | 25% | 33.33% | 16.67% | 0 | 16.67% | 0 | 0 | |
| 敏感 | 8.33% | 8.33% | 16.67% | 50.00% | 66.67% | 83.33% | 100% | 100% |
由耐药谱型(表 6)知, 11株大肠杆菌有1株对6种抗生素耐药, 1株对4种抗生素耐药, 2株对3种抗生素耐药, 多重耐药率(≥3重)为36.36%。大多数多重耐药菌分布于水流交换缓慢的水产养殖区, 耐药谱有一定的重合。这表明该海域大肠杆菌多重耐药情况较为严重。12株肠球菌中仅3株肠球菌无多重耐药性, 多重耐药率(≥3重)为75%。多重耐药菌中5株肠球菌耐药谱为红霉素*利福平*呋喃妥因, 其余都对环丙沙星和利福平耐药, 肠球菌耐药谱重合严重。
| 大肠杆菌 | 肠球菌 | |||
| 耐药谱型 | 菌株数量(站位) | 耐药谱型 | 菌株数量(站位) | |
| GM-CZ-CTX-F-AMP-C | 1(2号) | TET-CIP-RIF-F | 1(4号) | |
| CZ-F-AMP-TET | 1(6号) | E-RIF-F | 5(1、8、9、12、13号) | |
| CZ-AMP-C-TET | 1(13号) | CIP-RIF-F | 1(2号) | |
| CTX-C-TET | 1(9号) | E-CIP-RIF | 1(3号) | |
| TET-CIP-RIF | 1(7号) | |||
| 注: 庆大霉素(GM); 头孢唑啉(CZ); 头孢噻肟(CTX); 呋喃妥因(F); 氨苄西林(AMP); 氯霉素(C); 四环素(TET); 环丙沙星(CIP); 利福平(RIF); 呋喃妥因(F); 红霉素(E)。 | ||||
综上所述, 大肠杆菌和肠球菌对5种相同抗生素耐药性差异较大, 肠球菌对部分抗生素耐药率(≥50%)较高。大肠杆菌多重耐药现象较小, 多重耐药菌主要分布于2号站位。肠球菌耐药现象严重, 多数站位肠球菌都对三种以上抗生素耐药。
2.2 大肠杆菌对抗生素耐药性空间分布分析由图 2可知, 除四环素外, 其余抗生素高值区都主要分布于水产养殖区的2号站位, 其次6、13号站位。大肠杆菌对庆大霉素、头孢噻肟、环丙沙星的耐药水平高值区集中于2号站位。大肠杆菌对头孢唑啉、呋喃妥因、氨苄西林的耐药高值区主要位于2、6、13号站位, 但是氨苄西林高值区还存在于3号站位。四环素和氯霉素高值区主要位于9、13号站位, 但是氯霉素高值区分布广泛而四环素高值区分布更加集中。
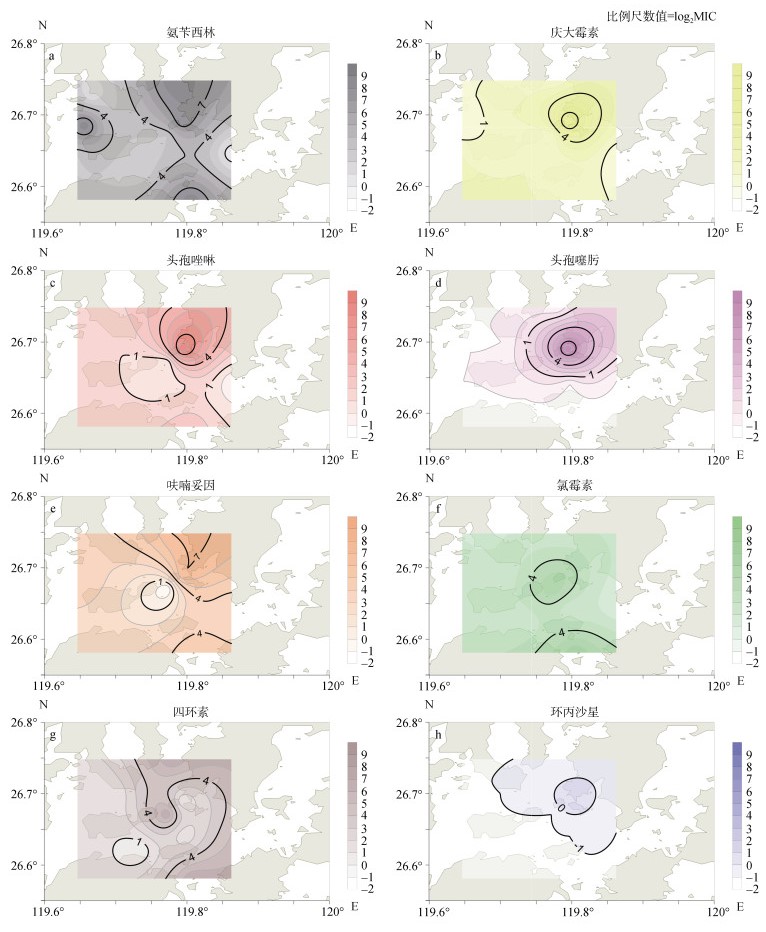 |
| 图 2 大肠杆菌对抗生素耐药空间分布图 Fig. 2 Spatial distribution of Escherichia coli resistance to antibiotics |
由图 3可知, 在调查水域中, 各站位的肠球菌对氨苄西林、氯霉素、呋喃妥因的耐药水平差异小。肠球菌对氯霉素、呋喃妥因的耐药水平高值区分布于1、2、5、9、12、13号站位, 其中氯霉素高值区在6、11号站位也有分布。红霉素、万古霉素、利福平高值区都分布于1、3、4、7、8、9、12、13号站位。同时红霉素在2、11号站位, 万古霉素在5、6、10、11号站位, 利福平在2、10号站位也都存在细菌耐药水平高值区。环丙沙星耐药水平高值区主要分布于2、3、4、6、7号站位。四环素耐药水平高值区主要分布于4、5、7、10号站位。
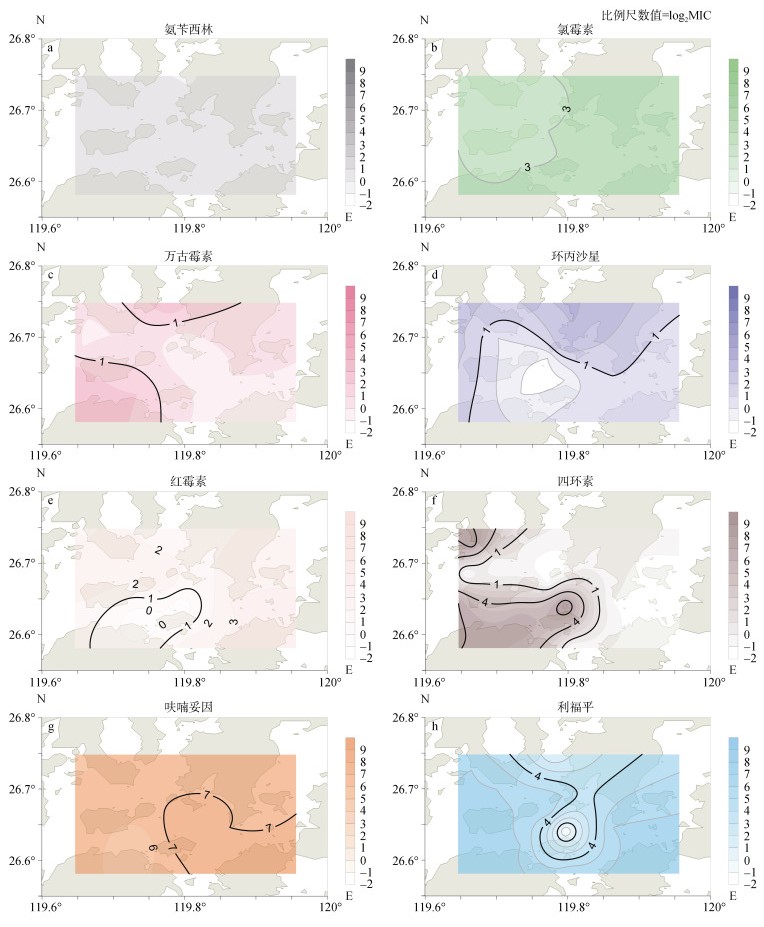 |
| 图 3 肠球菌对抗生素耐药空间分布图 Fig. 3 Spatial distribution of Enterococcus resistance to antibiotics |
大肠杆菌相关耐药基因检出情况如图 4a所示: TEM基因、cat1基因、nfsB基因检出率最高为100%, 无地域差异; 其次是nfsA基因、oqxA基因, 除2号(nfsA基因)、9号(oqxA基因)站位未检出, 检出率为90.91%; 其余耐药基因检出率较低, 分布狭隘。喹诺酮类oqxB基因和qnrs基因均在9号站位检出, 检出率分别为9.1%、36.36 %, qnrs基因还分布于2、6、9、13号站位。氯霉素类仅13号站位检出cmlA基因, 为9.1%。四环素耐药基因检出率均低, tetA基因分布于1、9、13号站位, tetB基因存在于6号站位, 检出率分别为27.27%、9.1%。13号站位耐药基因检出最多, 耐药基因污染较为严重。
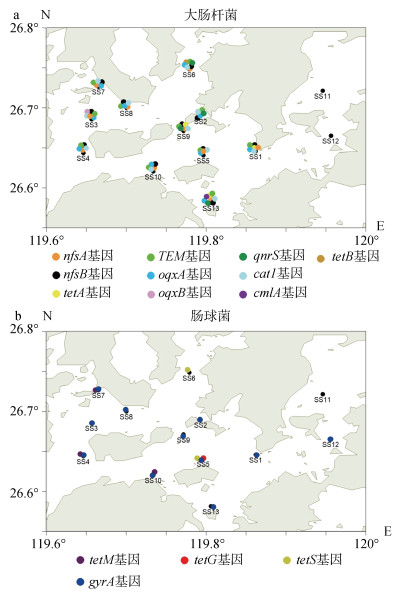 |
| 图 4 细菌耐药基因分布图 Fig. 4 Distribution of bacterial antimicrobial-resistant genes |
肠球菌相关耐药基因检出情况如图 4b所示, 氟喹诺酮类(gyrA基因)检出率最高, 仅6号站位未检出, 为91.67%。四环素类耐药基因(tetM、tetS、tetG、tetL)从高到低依次为25%、16.67%、8.33%、0%。tetM基因分布于4、7、10号站位, tetS基因在5、6号站位被检出, tetG基因仅存在于5号站位。大环内酯类耐药基因(ermA、ermB、mefA)以及万古霉素(vanB)耐药基因均未检出。
3 讨论实验结果中大肠杆菌和肠球菌对同种抗生素的耐药性有一定差异。两种细菌对同种抗生素耐药差异可能与耐药机制有关。即使是同样对青霉素耐药, 大肠杆菌主要通过β-内酰胺酶降解氨苄西林, 而肠球菌主要依靠改变药物与青霉素结合蛋白(PBPs)的结合位点[28]。同种抗生素耐药基因类型不同也是导致两种细菌耐药差异的原因, 如gyrA基因突变可能使细菌对氟喹诺酮类抗生素产生低水平耐药, 在此基础上若发生parC基因突变则可产生高水平耐药[29]。但细菌仅有qnr基因只能降低对喹诺酮类药物敏感性产生低水平耐药[30]。这与实验耐药基因检出和耐药性相符合。
细菌耐药性表达受多种条件影响, 如重金属、温度、抗生素浓度。重金属与抗生素会发生协同作用, 使细菌产生耐药性, 如Cu2+与红霉素共存, 使细菌从敏感变为耐药[31]。有研究表明, 对Cu2+耐受的细菌对万古霉素、四环素、氯霉素耐药性显著增加[32]。温度改变影响细菌对抗生素耐药性, 如温度改变影响肠球菌对多种抗生素的耐药性[33], 适应高温的大肠杆菌对利福平具有耐药性[34]。抗生素浓度也是影响细菌耐药性的重要因素, 由于抗生素高频使用, 水体残药浓度高诱导细菌产生耐药性[11, 35]。有研究表明, 鲍曼不动杆菌耐药性与抗生素使用频率存在相关性[36]。
大肠杆菌耐药菌空间分布主要分布于2号站位, 其次是6、13站位。有研究发现2、13号站位附近海域由于水产养殖导致水体多种抗生素的浓度和耐药基因丰度较高, 这可能会诱导细菌产生耐药性[35, 37-38]。这与本实验在2、13号站位大肠杆菌对多种抗生素耐药结果相符合, 因此水产养殖可能是影响细菌耐药性因素之一。也有研究发现6号站位存在重金属(Cu2+、Zn2+)高值区, 而Cu2+、Zn2+可以通过协同作用诱导耐药性增加, 因此重金属可能与6号站位大肠杆菌具有耐药性有一定关系[40-41]。对四环素、环丙沙星耐药的肠球菌主要分布在入河口和水产养殖区附近, 这与在九龙江、莱州湾基本相同[42-43]。而在三沙湾表层沉积物中发现Cu、Zn、Pb在3、4、7、13号站位附近存在高值区, 这与肠球菌对红霉素的耐药高值区相重合。Zn、Pb、Cu同样对红霉素耐药基因有选择作用[44-45]。这进一步证明环境细菌对抗生素的耐药性可能与环境中的重金属含量存在一定的关系。
已有研究表明三沙湾存在一定程度的抗生素污染, 但其环境浓度并不高, 远低于其对常见细菌的MIC值[38], 但是细菌可能会在亚抑菌浓度下产生耐药性或耐药基因[38, 46]。如大肠杆菌在低浓度四环素中持续暴露, 可以提高突变率和耐药性。低浓度红霉素可促进猪链球菌大量表达ABC转运蛋白[47-48]。三沙湾海域海水中四环素类抗生素分布最为普遍[38], 这可能与该海域四环素高耐药相关, 但是耐药菌分布与三沙湾抗生素浓度分布也具有一定差异, 因此还须对该海域细菌耐药性的成因做进一步的探索。
本研究发现大肠杆菌和肠球菌对多种抗生素存在耐药性。大肠杆菌只对庆大霉素和环丙沙星无耐药性。肠球菌对氨苄西林、万古霉素、氯霉素敏感。肠球菌对利福平、呋喃妥因、红霉素耐药率在50%以上。水域中耐药基因以nfsA基因、oqxA基因、cat1基因、gyrA基因为主要耐药基因, 其余耐药基因检出率低。实验发现, 抗生素耐药菌(大肠杆菌)主要分布于水产养殖区, 其中2、6、13号站位大肠杆菌表现出对多种抗生素耐药。耐药肠球菌空间分布均匀, 其中环丙沙星和四环素主要分布于入河口附近。
| [1] |
徐彤武. 当代全球卫生安全与中国的对策[J]. 国际政治研究, 2017, 38(3): 9-37. XU Tongwu. Contemporary global health security and China's strategy[J]. International Political Studies, 2017, 38(3): 9-37. |
| [2] |
葛林科, 任红蕾, 鲁建江, 等. 我国环境中新兴污染物抗生素及其抗性基因的分布特征[J]. 环境化学, 2015, 34(5): 875-883. GE Linke, REN Hongli, LU Jianjiang, et al. Occurrence of antibiotics and corresponding resistance genes in the environment of China[J]. Environmental Chemistry, 2015, 34(5): 875-883. |
| [3] |
易秀丽. 水源粪肠球菌耐药情况调查及耐药影响因素研究[D]. 福州: 福建农林大学, 2008. YI Xiuli. Study on antibiotics resistance and influence factors of water source Enterococcus faecalis[D]. Fuzhou: Fujian Agriculture and Forestry University, 2008. |
| [4] |
KIM T W, JOUNG Y, HAN J H, et al. Antibiotic resistance among aquatic bacteria in natural freshwater environments of Korea[J]. J Water Health, 2015, 13(4): 1085-1097. DOI:10.2166/wh.2015.032 |
| [5] |
吴丽云, 洪娟, 孙彤, 等. 水源性肠球菌对苯扎溴铵及部分抗菌药的耐药性研究[J]. 中国兽医科学, 2018, 48(11): 1406-1414. WU Liyun, HONG Juan, SUN Tong, et al. Study on resistance of water-borne Enterococcus to benzalkonium bromide and antibiotics[J]. Chinese Veterinary Science, 2018, 48(11): 1406-1414. |
| [6] |
张晶晶, 陈娟, 王沛芳, 等. 中国典型湖泊四大类抗生素污染特征[J]. 中国环境科学, 2021, 41(9): 4271-4283. ZHANG Jingjing, CHEN Juan, WANG Peifang, et al. Pollution characteristics of four-type antibiotics in typical lakes in China.[J]. China Environmental Science, 2021, 41(9): 4271-4283. |
| [7] |
刘畅, 林绅辉, 焦学尧, 等. 粤港澳大湾区水环境状况分析及治理对策初探[J]. 北京大学学报(自然科学版), 2019, 55(6): 1085-1096. LIU Chang, LIN Shenhui, JIAO Xueyao, et al. Problems and treatment countermeasures of water environment in Guangdong-Hong Kong-Macao Greater Bay area[J]. Acta Scientiarum Naturalium Universitatis Pekinensis, 2019, 55(6): 1085-1096. |
| [8] |
ZOU S, XU W, ZHANG R, et al. Occurrence and distribution of antibiotics in coastal water of the Bohai Bay, China: impacts of river discharge and aquaculture activities[J]. Environ Pollut, 2011, 159(10): 2913-2920. DOI:10.1016/j.envpol.2011.04.037 |
| [9] |
何春明, 那广水, 陆紫皓, 等. 莱州湾近岸海域中典型抗生素与抗性细菌分布特征及其内在相关性[J]. 生态毒理学报, 2015, 10(5): 115-123. HE Chunming, NA Guangshui, LU Zihao, et al. Distribution and correlation of typical antibiotics and antibiotic-resistant bacteria in the coastal marine environment of Laizhou Bay, China[J]. Asian Journal of Ecotoxicology, 2015, 10(5): 115-123. |
| [10] |
金晶磊, 周前进, 邵鑫斌, 等. 东海温州水域4类常见耐药基因的流行情况研究[J]. 海洋与湖沼, 2021, 52(4): 947-959. JIN Jinglei, ZHOU Qianjin, SHAO Xinbin, et al. Investigation on resistance genes against four types of common antibiotics in Wenzhou water of the east China sea[J]. Oceanologia et Limnologia Sinica, 2021, 52(4): 947-959. |
| [11] |
赵小慧, 苏洁, 樊景凤, 等. 海洋环境中细菌耐药性研究进展[J]. 中国抗生素杂志, 2019, 44(4): 406-412. ZHAO Xiaohui, SU Jie, FAN Jingfeng, et al. Research progress on bacterial resistance in marine environment[J]. Chinese Journal of Antibiotics, 2019, 44(4): 406-412. |
| [12] |
朱远芳. 福建省渔药使用状况调查与研究[D]. 厦门: 集美大学, 2013. ZHU Yuanfang. The investigation of fishery drugs usage in Fujian[D]. Xiamen: Jimei University, 2013. |
| [13] |
刘开放. 华南沿海哈维氏弧菌耐药性、质粒多性样及其相关性分析[D]. 海口: 海南大学, 2017. LIU Kaifang. Resistance to drugs、plasmid diversity and its correlation analysis of Vibrio harveyi strains isolated from coastal areas of South China[D]. Haikou: Hainan Univeristy, 2017. |
| [14] |
李健. 海水养殖源弧菌耐药性调查与外排泵抑制剂对其耐药性的影响[D]. 上海: 上海海洋大学, 2017. LI Jian, Investigation of drug resistance on phenicols and quinolone by efflux pump inhibitors[D]. Shanghai: Shanghai Ocean University, 2017. |
| [15] |
吴红岩. 鳜肌肉渔药残留、重金属含量与营养成分的质量安全分析[D]. 上海: 上海海洋大学, 2020. WU Hongyan. Quality and safety analysis on fishery drugResidues, heavy metals and nutritional components in muscle of mandarin Fish (Siniperca chuatsi)[D]. Shanghai: Shanghai Ocean University, 2020. |
| [16] |
张健, 朱秋华, 聂长丽, 等. 辽宁部分地区海产品多药耐药菌污染情况调查[C]//中国陕西西安: 2020. ZHANG Jian, ZHU Qiuhua, NIE Changli, et al. Investigation on the contamination of multi -drug resistant bacteria in seafood products in Liaoning Province[C]// Abstracts of the 17th Annual Meeting of CIFST. Xi'an, Shaanxi, China: China Food Science and Technology Society, 2020. |
| [17] |
BACANLI M, BAŞARAN N. Importance of antibiotic residues in animal food[J]. Food Chem Toxicol, 2019, 125: 462-466. DOI:10.1016/j.fct.2019.01.033 |
| [18] |
赵莎. 大连付家庄海水浴场中大肠杆菌耐药性及质粒介导的耐药性传递研究[D]. 大连: 大连海洋大学, 2017. ZHAO Sha. Study on drug-resistance and plasmid-mediated transfer resistance of Escherichia coli in Dalian Fujiazhuang bathing beach[D]. Dalian: Dalian Ocean University, 2017. |
| [19] |
衣首静. 泰安渿河中产ESBLs大肠杆菌耐药性及耐药基因的传递规律初步探究[D]. 泰安: 山东农业大学, 2020. YI Shoujing. Antibiotic resistance and antibiotic resistance gene transfer of ESBLs-Producing Escherichia coli in Naihe, Taian[D]. Taian: Shandong Agricultural University, 2020. |
| [20] |
陈玉红. 闽江流域水源性肠球菌抗生素耐药与水体氮磷营养盐的相关性研究[D]. 福州: 福建农林大学, 2015. CHEN Yuhong. Correlative study of antibiotics resistance of water source enterococcus and nitrogen and phosphorus nutrients in MinJiang river[D]. Fuzhou: Fujian Agriculture and Forestry University, 2015. |
| [21] |
叶海桃, 王义刚, 曹兵. 三沙湾纳潮量及湾内外的水交换[J]. 河海大学学报(自然科学版), 2007, 1: 96-98. YE Haitao, WANG Yigang, CAO Bing. Tidal prism of Sansha Bay and its water exchange with the open sea[J]. Journal of Hohai University(Natural Sciences), 2007, 1: 96-98. |
| [22] |
王宇翔. 福建省内动物园圈养野生动物肺炎克雷伯菌的耐药表型及基因型研究[D]. 福州: 福建农林大学, 2020. WANG Yuxiang. Study on drug resistant phenotype and resistance genes of Klebsiella pneumoniae from wild animals in zoos in Fujian Province[D]. Fuzhou: Fujian Agriculture and Forestry University, 2020. |
| [23] |
赵静雯. 苏北地区奶牛乳腺炎病原菌流行病学调查及大肠杆菌耐药性与毒力基因相关性分析[D]. 扬州: 扬州大学, 2022. ZHAO Jingwen. Epidemiological investigation of bovine mastitis in Northern Jiangsu and correlation analysis between drug resistance and virulence genes of Escherichia coli[D]. Yangzhou: Yangzhou University, 2022. |
| [24] |
刘聪. 山东地区猪牛养殖废水中多重耐药大肠杆菌与重金属残留的分布特征和风险评估[D]. 泰安: 山东农业大学, 2022. LIU Cong. Distribution Characteristics and Risk Assessment of Multi-drug Resistant Escherichia coli and Heavy Metal Residues in Swine and Dairy Farm Wastewater in Shandong Province[D]. Taian: Shandong Agricultural University, 2022. |
| [25] |
梁晶晶. 九龙江流域耐药细菌及其特定耐药基因和整合子的研究[D]. 厦门: 集美大学, 2015. LIANG Jingjing. Studies on Antibiotic-resistant Bacteria Isolated from Jiulong River and Their Carrying Resistant Genes and Integrons[D]. Xiamen: Jimei University, 2015. |
| [26] |
李晓云, 赵志慧, 闫明, 等. 东北地区猪链球菌对大环内酯类药物耐药机制的研究[J]. 中国预防兽医学报, 2008, 8: 612-617. LI Xiaoyun, ZHAO Zhuihui, YAN Ming, et al. Mechanism of Streptococcus suis resistance to Macrolides in Northeast China[J]. Chinese Journal of Preventive Veterinary Medicine, 2008, 8: 612-617. |
| [27] |
李琴, 孙成波, 陈美领, 等. 湛江东海岛对虾育苗场水体细菌的抗生素耐药性[J]. 热带生物学报, 2014, 5(1): 8-14. LI Qin, SUN Chengbo, CHEN Meiling, et al. Antibiotic Resistance of Bacteria Isolated from Shrimp Hatcheries at Donghai Island, Zhanjiang[J]. Journal of Tropical Biology, 2014, 5(1): 8-14. |
| [28] |
MUNITA J. M., ARIAS C. A. Mechanisms of Antibiotic Resistance[J]. Microbiol Spectr, 2016, 4(2): 136-142. |
| [29] |
郭丽娜. 耐喹诺酮类肠球菌耐药机制研究[J]. 系统医学, 2021, 6(13): 192-194. GUO Lina. Study on the Drug Resistance Mechanism of Quinolone -resistant Enterococci[J]. Systems Medicine, 2021, 6(13): 192-194. |
| [30] |
谭爱萍, 邓玉婷, 姜兰, 等. 水产动物源气单胞菌喹诺酮类耐药与PMQR基因、QRDR突变相关性分析[J]. 中国农业大学学报, 2019, 24(4): 102-111. TAN Aiping, DWNG Yuting, JIANG Lan, et al. Correlation between PMQR genes/QRDR mutations and quinolones resistance in Aeromonas from aquatic animals[J]. Journal of China Agricultural University, 2019, 24(4): 102-111. |
| [31] |
陈颖欣, 陈佳华, 张志瑾, 等. 畜禽养殖场抗生素耐药基因残留及传播研究进展[J]. 家畜生态学报, 2021, 42(3): 1-7. CHEN Yingxin, CHEN Jiahua, ZHANG Zhijin, et al. Research progress of antibiotic resistance gene residues and dissemination in livestock farms[J]. Journal of Domestic Animal Ecology, 2021, 42(3): 1-7. |
| [32] |
BERG J, THORSEN M. K., HOLM P. E., et al. Cu exposure under field conditions coselects for antibiotic resistance as determined by a novel cultivation-independent bacterial community tolerance assay[J]. Environ Sci Technol, 2010, 44(22): 8724-8728. |
| [33] |
JACKSON C R, FEDORKA-CRAY P J, JACKSON-HALL M C, et al. Effect of media, temperature and culture conditions on the species population and antibiotic resistance of enterococci from broiler chickens[J]. Lett Appl Microbiol, 2005, 41(3): 262-268. |
| [34] |
RODRÍGUEZ-VERDUGO A, GAUT B S, TENAILLON O. Evolution of Escherichia coli rifampicin resistance in an antibiotic-free environment during thermal stress[J]. BMC Evol Biol, 2013, 13: 50. |
| [35] |
张彤, 李炳. 水环境中抗生素耐药性的科学研究前沿、环境健康风险评估和控制阻断策略[J]. 科学通报, 2020, 65(24): 2543-2554. ZHANG Tong, LI Bing. Antibiotic resistance in water environment: Frontiers of fundamental research, risk assessment and control strategies[J]. Chinese Science Bulletin, 2020, 65(24): 2543-2554. |
| [36] |
付远清, 莫逢伟, 花南霞. 鲍曼不动杆菌对8种抗生素耐药性与用药频度相关性分析[J]. 现代医药卫生, 2013, 29(17): 2655-2656. FU Yuanqing, MO Fengwei, HUA Nanxia. Correlation analysis between antibiotic resistance of Acinetobacter baumannii to 8 antibiotics and drug frequency[J]. Journal of Modern Medicine & Health, 2013, 29(17): 2655-2656. |
| [37] |
曹佳雯. 三沙湾海水养殖环境中抗生素抗性基因的时空分布特征研究[D]. 厦门: 厦门大学, 2019. CAO Jiawen. Study on the spatial and temporal distribution characteristics of antibiotic resistance genes in mariculture environment in Sansha Bay[D]. Xiamen: Xiamen University, 2019. |
| [38] |
周柯举. 集约化海水养殖区抗生素的污染特征[D]. 厦门: 厦门大学, 2020. ZHOU Keju. Contamination of antibiotics in intensive mariculture areas: A case study of Sansha Bay in Ningde, Fujian province[D]. Xiamen: Xiamen University, 2020. |
| [39] |
霍云龙, 陈金民, 林彩, 等. 三沙湾表层沉积物重金属含量分布及生态风险评估[J]. 应用海洋学学报, 2015, 34(3): 356-364. HUO Yunlong, CHEN Jinmin, LIN Cai, et al. Distribution of heavy metal contents in surface sediments of Sansha Bay and ecological risk assessment[J]. Journal of Applied Oceanography, 2015, 34(3): 356-364. |
| [40] |
史亚楠. 重金属铜锌对病原菌及环境细菌耐药性的影响[D]. 广州: 广东药科大学, 2017. SHI Yanan. Effects of heavy metal copper and zinc on drug resistance of pathogenic bacteria and environmental bacteria[D]. Guangzhou: Guangdong Pharmaceutical University, 2017. |
| [41] |
张佳奇, 徐艳, 罗义, 等. 重金属协同选择环境细菌抗生素抗性及其机制研究进展[J]. 农业环境科学学报, 2016, 35(3): 409-418. ZHANG Jiaqi, XU Yan, LUO Yi, et al. Co-selection mechanisms of bacterial resistance to heavy metals and antibiotics[J]. Journal of Agro-Environment Science, 2016, 35(3): 409-418. |
| [42] |
欧丹云, 陈彬, 陈灿祥, 等. 九龙江下游河口水域抗生素及抗性细菌的分布[J]. 中国环境科学, c2013, 33(12): 2243-2250. OU Danyun, CHEN Bin, CHEN Canxiang, et al. Distribution of antibiotics residue and resistant bacteria in the downstream and estuarine area in Jiulong River[J]. China Environmental Science, c2013, 33(12): 2243-2250. |
| [43] |
张瑞杰, 张干, 郑芊, 等. 喹诺酮类抗生素在莱州湾及主要入海河流中的含量和分布特征[J]. 海洋环境科学, 2012, 31(1): 53-57. ZHANG Ruijie, ZHANG Gan, ZHENG Qian, et al. Concentrations and spatial distributions of selected quinolones antibiotics in Laizhou Bay and main rivers flowing into the bay[J]. Marine Environmental Science, 2012, 31(1): 53-57. |
| [44] |
GAO P, HE S, HUANG S, et al. Impacts of coexisting antibiotics, antibacterial residues, and heavy metals on the occurrence of erythromycin resistance genes in urban wastewater[J]. Appl Microbiol Biotechnol, 2015, 99(9): 3971-3980. |
| [45] |
李侃竹, 高品, 王凯, 等. 污水中抗生素与重金属对红霉素抗药性基因的选择性效应[J]. 中国环境科学, 2015, 35(3): 889-896. LI Kanzhu, GAO Pin, WANG Kai, et al. Selective pressure of antibiotics and heavy metals on erythromycin resistance genes in wastewater[J]. China Environmental Science, 2015, 35(3): 889-896. |
| [46] |
谷宇锋, 王淑歌, 郝海红, 等. 亚抑菌浓度抗生素对细菌耐药性和毒力影响的研究进展[J]. 中国抗生素杂志, 2018, 43(11): 1321-1331. GU Yufeng, WANG Shuge, HAO Haihong, et al. Effects of sub-inhibitory concentration of antibiotics on bacterial resistance and virulence: A review[J]. Chinese Journal of Antibiotics, 2018, 43(11): 1321-1331. |
| [47] |
杨艳北, 许晶, 沈城辉, 等. 低浓度红霉素对猪链球菌蛋白表达、交叉耐药性与荚膜多糖的影响[J]. 微生物学报, 2022, 62(5): 1843-1850. YANG Yanbei, XU Jing, SHEN Chenghui, et al. Effects of low-concentration erythromycin on protein expression, cross resistance and capsular polysaccharides of Streptococcus suis[J]. Acta Microbiologica Sinica, 2022, 62(5): 1843-1850. |
| [48] |
裴梦珂. 亚抑菌浓度四环素暴露对大肠杆菌及生物生态耦合处理系统中菌群抗性基因变化的影响[D]. 上海: 上海交通大学, 2019. PEI Mengke. Effects of subinhibitory concentration tetracycline on changes of antibiotic resistance genes in E. coli and microbe in bio-ecological wastewater treatment system[D]. Shanghai: Shanghai Jiaotong University, 2019. |
 2023, Vol. 47
2023, Vol. 47


