文章信息
- 刘丽娴, 郭栗, 杨官品. 2023.
- LIU Li-xian, GUO Li, YANG Guan-pin. 2023.
- CRISPR/Cas9基因编辑系统及其在微藻中的应用
- Application of the CRISPR/Cas9 gene editing system in microalgae
- 海洋科学, 47(8): 101-111
- Marine Sciences, 47(8): 101-111.
- http://dx.doi.org/10.11759/hykx20221014001
-
文章历史
- 收稿日期:2022-10-14
- 修回日期:2022-11-08
成簇规则间隔短回文重复(clustered regularly interspaced short palindromic repeats, CRISPR)于1987年在大肠杆菌中首次被发现[1-4]。之后陆续在许多细菌中也发现了CRISPR序列, 并逐渐阐明了其生物学意义。CRISPR/Cas系统广泛用于动植物遗传育种, 微生物工程, 合成生物学等研究, 并在不断改进, 以满足多样化的研究和应用需求。近年来, CRISPR/Cas9系统在微藻中的应用越来越多。本文简要介绍了CRISPR/Cas系统, 重点综述了CRISPR/ Cas9系统在微藻中的应用, 以期为微藻的相关研究提供参考。
1 CRISPR/Cas系统成簇规则间隔短回文重复序列由前导、重复和间隔序列构成。前导序列长20~534 bp, 腺嘌呤和胸腺嘧啶含量高, 是CRISPR的启动子[5-6]。重复序列长21~48 bp, 其转录本可形成发夹结构[7-8]。间隔序列各异, 长26~72 bp, 是免疫外源DNA的效应序列。病毒或外源DNA的部分序列可插入到CRISPR引导序列端, 形成新的间隔序列。当相应的病毒或外源DNA再次入侵时, CRISPR便会发挥免疫作用[5, 7, 9-11]。
CRISPR合作蛋白Cas的编码基因位于CRISPR基因附近[5]。CRISPR和Cas协同发挥作用, 使细菌免疫有特定间隔序列的病毒或外源DNA的效应序列, 防止入侵。CRISPR/Cas系统介导的免疫过程分为适应、表达和干扰3步, 参与的蛋白各异。表 1列出适应过程Cas1和Cas2将入侵的DNA片段整合到CRISPR基因座中, 进入表达步骤, CRISPR被转录成前体crRNA (pre-crRNA), 前体crRNA结合到Cas蛋白上并经RNase切割, 最终形成成熟的crRNA (CRISPR RNA)。当外源DNA再次侵入时, 干扰步骤发挥作用, crRNA与外源DNA互补, Cas蛋白行使核酸酶功能切割外源DNA[1, 12-14], 实现免疫。CRISPR/ Cas系统存在于约一半已测序细菌基因组和几乎所有的古细菌基因组中。但在真核生物和病毒中却不存在[5, 8, 11, 15-16]。
Sapranauskas等[19]证明嗜热链球菌CRISPR/Cas系统可转移到大肠杆菌中发挥免疫作用, 保护转化的质粒和转染的噬菌体。2012年, 双RNA分子被发现可引导Cas9切割细菌基因组任意位置[20], Cas9的可操纵性和编辑功能在许多生物中通用, 仅需改变与靶DNA互补结合的引导RNA分子。这种高效、通用的系统迅速获得许多研究者的青睐, 他们也因此获得2020年诺贝尔化学奖。这一划时代的发现推动了各种生物的基因编辑进程。
常用的CRISPR/Cas9基因编辑技术属于Ⅱ型CRISPR/Cas系统。参与CRISPR/Cas作用的各组分如下: (1) crRNA。crRNA5′端的20个核苷酸各不相同, 结合不同的靶DNA。该部分由外源的DNA序列插入CRISPR序列的间隔区, 先转录形成pre-crRNA, 再切割成crRNA[12, 21]。(2) tracrRNA(trans-activating crRNA)。tracrRNA通过RNase Ⅲ触发pre-crRNA加工, 随后通过Cas9激活crRNA引导的靶DNA切割[20-21](图 1a)。(3) sgRNA(single guide RNA)。crRNA和tracrRNA连接, 形成长约100 bp的单分子引导RNA[22], GC含量约40%~60%[23](图 1b)。(4) Cas9蛋白。Cas9蛋白是2个RNA引导的DNA核酸内切酶。Cas9有2个结构域。HNH结构域切割与crRNA互补的DNA链, RuvC结构域切割非互补DNA链[20]。Cas9识别crRNA互补的20 bp DNA序列和三核苷酸[12]。(5) PAM(protospacer-adjacent motif)序列。PAM序列与目的基因常为NGG, 是Cas9核酸酶特异性识别位点。PAM序列对间隔区的选择有CRISPR类型特异性[24]。质粒DNA会在PAM序列上游3个碱基对的位置切割并产生平末端。而短线型双链DNA, 与crRNA序列互补的目的基因DNA链在PAM序列上游3个碱基对的位置被切割, 非互补DNA链在PAM上游的3~8个碱基对内的1个或多个位点被切割。非互补DNA链首先被核酸内切酶切割, 随后被3′-5′端的外切核酸酶切割[20]。PAM序列在平均每16个碱基中出现1次, 限制了可以建立的目标数量[25]。
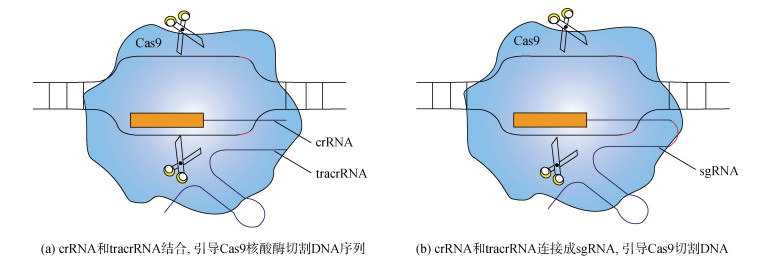 |
| 图 1 CRISPR/Cas9系统切割原理 Fig. 1 Functional mechanism of CRISPR/Cas9 |
2014年, CRISPR/Cas9技术首次在微藻中成功应用。Jiang等[26]将sgRNA和Cas9基因转入莱茵衣藻, 证明Cas9和sgRNA在莱茵衣藻中能正常发挥作用, 介导靶向基因修饰。表达CRISPR/Cas9系统的构建物转化进藻细胞后, sgRNA与目标DNA结合, 引导Cas9切割DNA, 产生双链DNA断裂(double strand break, DSB)。DSB经过非同源末端连接(non-homologous end joining, NHEJ)或同源重组修复(homologous recombination, HR), 引入插入或缺失, 敲除基因(基因失活)。若外源DNA和表达CRISPR/Cas构建物同时转化, 则可插入编辑的基因[27]。可根据不同目的设计特定的sgRNA, 获得编辑藻株或转化藻株。Cas9和sgRNA也可以纯化后融合成复合物, 转化进细胞, 实现编辑。
实现CRISPR/Cas9基因编辑的微藻越来越多。表 2根据Cas9的表达位置, 可以将微藻CRISPR/Cas9基因编辑的方式可以分为3类: (1) Cas9基因整合到微藻基因组中发挥功能, 常用细菌质粒为构建物进行转化。(2) Cas9以游离蛋白方式在微藻基因组中发挥功能, 通常是先形成核糖核蛋白复合体(Cas9-gRNA ribonucleoprotein)再导入微藻细胞。(3) 构建游离的附加体(episome)转入微藻细胞。附加体上Cas9和gRNA表达后发挥功能, 但附加体不整合进基因组, 去除抗生素选择压后, 附加体因生物稀释而消失, 如图 2所示。
| CRISPR/Cas9合成方式 | 构建物 | 藻名 | 编辑的基因 | 表现型 | 参考文献 |
| 整合进基因组, 组成型表达 | 细菌质粒构建物 | 莱茵衣藻(Chlamydomonas reinhardtii) | FKB12 | 雷帕霉素抗性 | [21, 28] |
| 团藻(Volvox carteri) | glsA/regA/invA | 无性腺(Gls A)表型 | [30] | ||
| 小球藻(Chlorella vulgaris) | Fad3; NR/APT | 脂质升高; 不能利用硝酸盐/2-氟腺嘌呤抗性 | [29, 45] | ||
| 微拟球藻(Nannochloropsis) | BGS/TGS; 光捕获复合基因 | 脂质积累增多; 光捕获天线尺寸减小 | [32, 35] | ||
| 三角褐指藻(Phaeodactylum tricornutum) | CpSRP54; 脲酶基因; | 不能在含尿素培养基上生长 | [31, 33] | ||
| 假微型海链藻(Thalassiosira pseudonana) | 脲酶基因 | 不能在含尿素培养基上生长 | [34] | ||
| 不整合, 瞬时表达 | 真核附加体构建物 | 三角褐指藻(Phaeodactylum tricornutum) | NR | 不能在硝酸盐培养基上生长 | [39] |
| 微拟球藻(Nannochloropsis) | Chr30染色体; NR | 精确和连续切除100kb大片段; NR-KO突变株 | [37-38] | ||
| RNP核糖核蛋白复合物 | 莱茵衣藻(Chlamydomonas reinhardtii) | Cpftsy; MAA7/CpRP43/ChlM; ZEP; PLA2 | 淡绿色; 浅绿色; 玉米黄质升高; 脂质生产力提高; 突变体生长劣势 | [27, 41, 45-47] | |
| 杜氏盐藻(Dunaliella salina) | Dschyb | β-胡萝卜素升高, 玉米黄质降低 | [40] | ||
| 不整合, 瞬时表达 | RNP核糖核蛋白复合物 | 扁藻(Tetraselmis sp.) | AGP | 脂质生产力提高 | [48] |
| 胶球藻(Coccomyxa sp.) | FTSY | 淡绿色 | [42] | ||
| 微绿藻(Picochlorum celeri) | CrtISO | 多等位基因功能丧失 | [43] | ||
| 小球藻(Chlorella vulgaris) | NR/APT; | 不能利用硝酸盐/ 2-氟腺嘌呤抗性 | [45] | ||
| 三角褐指藻(Phaeodactylum tricornutum) | PtAureo1a | 叶绿素荧光的非光化学猝灭降低 | [44] | ||
| 裸藻(Euglena gracilis) | EgGSL2 | 少而大的副淀粉颗粒 | [49] |
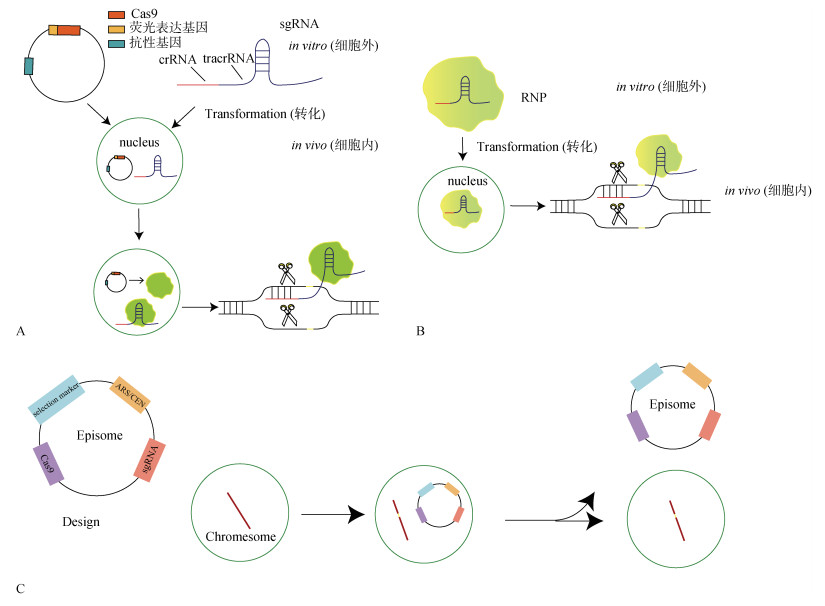 |
| 图 2 CRISPR/Cas9技术的不同应用方式 Fig. 2 Different functional mechanisms of CRISPR/Cas9 system 注: A, 以细菌质粒为扩增方法构建Cas9基因和gRNA表达构建物, 整合进微藻基因组, 组成型表达; B, 合成和纯化Cas9和gRNA, 形成核糖核蛋白复合物, 瞬时发挥功能; C, 以episome为载体构建Cas9和gRNA表达构建物转入微藻, 但不整合进基因组, 基因表达后瞬时发挥作用, 去除抗生素选择压, 附加体因生物稀释而消失。 |
细菌质粒是常用的表达Cas9-gRNA基因的构建物。质粒是细菌体系, 构建方便, 分子量小, 在植物、动物等的基因工程研究中使用广泛。在微藻中应用CRISPR/Cas系统, 要用细菌质粒为框架, 构建Cas9-gRNA基因表达构建物, 把Cas9基因和sgRNA单元表达元件集中在同一质粒或不同质粒上, 用合适的转化方法, 将细菌质粒构建物(重组质粒)转入微藻细胞并整合在微藻基因组DNA中, 实现Cas9蛋白基因和sgRNA的组成型表达(图 2A)。
2014年, Jiang等[26]构建CRISPR/Cas9细菌质粒构建物, 实现莱茵衣藻内源FKB12基因靶向敲除。这是CRISPR/Cas9技术在微藻中的首次应用。虽然获得了突变体, 但是莱茵衣藻的编辑效率并不很高。为了提高莱茵衣藻的编辑效率, 2017年, 该研究团队又将sgRNA表达组件插入Cas9基因的内含子中, 实现两个成员共享一套表达控制元件, 使莱茵衣藻的编辑效率提高50倍[28]。
在微藻中, CRISPR/Cas9技术是新兴的技术, 需要通过对已知基因的编辑, 证明其可行性。Lin等[29]首次在小球藻中实现CRISPR/Cas9技术应用, 他们把表达Cas9和sgRNA的细菌质粒构建物转化进小球藻(Chlorella vulgaris), 敲除其fad3(omega-3 fatty acid desaturase, ω-3脂肪酸去饱和酶)基因, 突变株与野生型相比, 染色后脂质积累荧光增加了67%。Ortega-Escalante等[30]将表达sgRNA和Cas9基因的细菌质粒构建物首次转入团藻(Volvox carteri)细胞, 获得了发育基因glsA(gonidialess A, 无性腺A)基因缺陷突变株, 验证了CRISPR/Cas9技术在团藻基因编辑中的可行性。三角褐指藻(Phaeodactylum tricornutum)是硅藻的研究模式藻。Nymark等[31]构建含CRISPR/Cas9系统的细菌质粒构建物并转化进三角褐指藻, 是CRISPR/ Cas9在硅藻中的首次尝试, 获得CpSRP54(chloroplast signal recognition particle 54, 叶绿体信号识别因子54)基因缺陷的突变藻株, 突变体对强光敏感。
CRISPR/Cas9技术编辑微藻已知基因成功后, 就可以用来研究微藻未知基因功能功能, 获得有益突变株。Vogler等[32]将编码Cas9-GFP融合蛋白基因的细菌质粒构建物转入微拟球藻(Nannochloropsis gaditana)细胞, 获得了β-葡萄糖合成酶和糖基化酶基因缺陷的突变株, 突变株中不溶于甲醇的水溶性碳水化合物积累减少但无生长缺陷, β-葡聚糖合酶基因的敲除可导致细胞自发荧光显著变化。Slattery等[33]和Hopes等[34]分别利用CRISPR/Cas技术靶向敲除三角褐指藻和假微型海链藻的urease(脲酶)基因, 突变株无法利用尿素作为生长的氮源, 藻细胞生长量降低, 藻细胞尺寸减小。
另外, 因Cas9组成型表达对某些藻类, 如莱茵衣藻, 有毒性。因此, 诱导型启动子的应用可解决Cas9的毒性问题[26, 35]。
3.2 Cas9蛋白基因和sgRNA瞬时表达附加体(episome)类似质粒, 通常比细菌质粒大, 在微藻中有一定程度的自我复制能力, 是真核生物染色体外遗传结构。与细菌质粒构建物的最大区别是, episome不整合到基因组中。在微藻中转化含CRISPR/Cas9系统的episome构建物, 可发挥编辑作用但不整合到宿主基因组中, 在去除抗生素选择压后, 因生物稀释作用, 附加体随微藻繁殖而逐渐消失[36](图 2C)。目前, 在微拟球藻和三角褐指藻中, 微藻附加体构建物均有应用。
Wang等[37]通过构建含有CRISPR/Cas9系统的附加体, 实现微拟球藻30号染色体中精确连续切除约10 kb的大片段。缺失大片段的突变株生长与野生型相比没有显著变化。因此, 用此方法可得到最小的微拟球藻功能基因组(底盘基因组)。Poliner等[38]构建有抗生素选择标记和有CRISPR系统的附加体构建物, CRISPR/Cas9系统可精确地在微拟球藻目标位置产生双链DNA断裂, 引入插入突变, 去除抗生素选择压力后, 消除附加体, 从而产生无标记的非转基因藻株。Moosburner等[39]将人工构建的episome(Cas9-ShBle: sgRNA)转化到含有Pta-MOB质粒的大肠杆菌中, 再通过细菌转染将episome转入三角褐指藻, 实现单基因和多基因敲除, 效率分别是48%和25%。
利用构建附加体方式进行CRISPR/Cas9基因编辑, 不整合到基因组中, 能产生定点突变并可以避免Cas9基因对藻细胞的长期毒害。但是, 构建附加体方式在微藻中应用不多, 大多都是在模式微藻中进行的。
3.3 核糖核蛋白复合体(Ribonucleoproteins, RNP)直接导入RNP直接导入是将Cas9蛋白体外表达纯化并与sgRNA融合形成RNP复合物后, 将RNP共同导入细胞(图 2B)。复合物的直接导入需充分考虑半衰期、转化效率等问题[40]。对于低等植物和单细胞生物, RNP直接导入成功率高。微藻是单细胞低等生物, RNP直接导入非常适合在微藻中使用。有研究表明, 莱茵衣藻转化Cas9基因表达靶向敲除低效率可能是载体驱动的Cas9基因长期在细胞内表达诱导的毒性造成的[41]。RNP直接转入瞬时表达, 可以避免Cas9基因表达相关的细胞毒性效应和脱靶效应。
2016年, Shin等[41]将Cas9蛋白和sgRNA复合体导入莱茵衣藻, 证明这种方法显著提高了CRISPR/ Cas9系统在衣藻中的诱变效率和特异性, 与转化含Cas9基因的质粒相比, 靶向突变效率提高了100倍, 能最大限度减少脱靶, 同时降低Cas9蛋白毒性。
同步敲除莱茵衣藻已知功能的MAA7(色氨酸合酶β亚基基因)、CpSRP43(叶绿体SRP43触角组装基因)和ChlM(叶绿素生物合成基因), 可建立基于5-氟吲哚和利用菌落颜色的筛选方法[41]。将RNP导入胶球藻(Coccomyxa sp.), 敲除了FTSY(叶绿体信号识别因子基因), 藻落呈淡绿色[42]。靶向敲除微绿藻(Picochlorum celeri)NR(硝酸还原酶基因)和CrtISO (类胡萝卜素异构酶基因), 获得基因“功能丧失”敲除藻株[43], 突变株不能利用硝酸盐作为氮源。Serif等[44]鉴定并用RNP敲除了三角褐指藻2个内源性标记基因和光感受器转录因子, 实现了多基因同步敲除, 证明了RNP直接导入可实现硅藻的基因编辑; Kim等[45]选择同时使用质粒表达Cas9的方法和RNP方法转化小球藻, 分别敲除其NR和APT(腺嘌呤磷酸核糖转移酶基因)。
进行科学研究的最终目的是要能够应用到实际生产中, RNP直接导入可编辑筛选高生产力藻株。Baek等[46]用RNP敲除莱茵衣藻CpFTSY(叶绿体信号识别因子基因), 光合效率大幅提升, 生产力提高。后又敲除ZEP(玉米黄质环氧化酶基因), 实现莱茵衣藻黄斑色素光合自养生产, 并用做蛋鸡饲料[47]。Shin等[48]用RNP敲除莱茵衣藻PLA2(磷脂酶A2)基因, 提高了莱茵衣藻脂质生产力。Chang等[49]用RNP编辑扁藻(Tetraselmis sp.)AGP(ADP葡萄糖焦磷酸化酶)基因, 提高了脂质生产力。Hu等[40]用RNP编辑杜氏盐藻(Dunaliella salina)β-胡萝卜素羟化基因, 使β-胡萝卜素含量比野生株提高2.2倍。Nomura等[50]用RNP编辑细小裸藻(Euglena gracilis)EgGSL2(葡聚糖合酶基因), 实现单链寡核苷酸精准敲入, 使裸藻储存更少但更大的副淀粉颗粒。
RNP直接导入已成功应用于许多微藻的基因标记, 瞬时存在的Cas9蛋白, 不会对目标微藻产生长期毒害。这种方式也有局限性, RNP复合物的形成需要Cas9纯度较高, 但其获得较为困难。因此, 选择用细菌质粒为框架, 构建Cas9基因和gRNA单元表达构建物会更加简便。
4 应用于微藻的其他CRISPR/Cas系统与Cas9核酸酶有相同作用的Cas12a(Cpf1)也是被单RNA引导的酶。与Cas9识别PAM序列不同, Cpf1内切酶识别富含胸腺嘧啶的DNA序列, 如5′-TTTN-3′, 产生黏性双链断裂[51]。Cas12a(Cpf1)核酸内切酶在距PAM 18~23 bp的位置切开DNA, 引入DSB。因此, 非同源末端修复可能不影响Cas9需要的PAM和识别序列, Cas12a可再次切割同一位点[52]。用CRISPR/Cpf1系统的RNP复合物和单链寡核苷酸高效编辑莱茵衣藻CpFTSY(叶绿体信号识别因子基因)、CpSRP43(参与叶绿素捕光复合物的组装)和PHT7(磷酸盐转运蛋白基因) 3个基因, 获得淡绿色、小型化、生长慢的藻株[53]。Naduthodi等[52]纯化Francisella novicida的Cas12a (FnCas12a)蛋白(Cpf1), 将crRNA与Cas12a组合成RNP复合物, 实现了海洋微拟球藻硝酸还原酶基因敲除。
除了Cpf1和Cas9外, 还有其他Cas9变体, 如Cas9n(Cas9 nickase)可以造成单链DNA的断裂, 类似限制性核酸酶功能, 产生一个切口, 减少脱靶效应的发生。
5 CRISPR/Cas9系统的验证CRISPR/Cas9系统每一个元件都是基因编辑不可或缺的。表达Cas9的重组质粒常有整合失败的情况, Cas9基因成功表达需要验证。与Cas9基因共表达一个报告基因, 如Nlux、tdTomato基因等, 通过荧光可初步确定Cas9的成功表达[36, 38]。Jiang等[26]设计的Cas9基因表达构建物上有一个20 bp位点, 可被ApaL Ⅰ酶切消化。如果Cas9基因成功整合, PCR扩增产物不含这段序列, 因而抗ApaL Ⅰ消化。Wang等[54]对转化后的微藻基因组DNA进行巢式PCR扩增片段, 用限制酶Pvu Ⅱ处理。Pvu Ⅱ可将野生型PCR片段消化成2条带, 而突变型无限制酶位点, 抗限制酶消化。
编辑微藻肉眼或显微镜可辨表型的对应基因就是验证CRISPR/Cas9系统的直接方法。莱茵衣藻CpSRP43和ChlM基因分别编码叶绿体SRP43触角组装和Mg-原卟啉IXS-腺苷甲硫氨酸O-甲基转移酶编辑成功后获得肉眼可辨的浅绿色、黄色或棕色藻落[41]。常用的NR和NIR分别编码硝酸还原酶和亚硝酸还原酶, 敲除后藻株无法利用硝酸盐或亚硝酸盐氮源, 肉眼上识别藻落呈黄色, 但能在含铵盐的f/2培养基中正常生长, 藻落呈正常绿色[45, 55]。
因此, 通过验证可辨表型的基因编辑方法, 在很大程度上减少了抗生素的使用, 不仅可以节省实验成本, 而且可以减少对环境的污染。
6 微藻中CRISPR介导的表达干扰(CRISPR-interference, CRISPRi)CRISPRi仍用gRNA引导突变的Cas9(deficient Cas9, dCas9)识别和结合DNA, 干扰基因转录和基因表达[56]。dCas9的RuvC和HNH2个核酸酶结构域沉默突变, D10A和H840A, 失去内切酶活性, 但保留了DNA识别和结合活性, 与sgRNA共表达时可形成DNA识别复合体, 特异性干扰基因转录[20, 56-57]。CRISPRi可以同时干扰多个靶基因转录, 干扰作用可逆[56, 58]。CRISPRi在蓝细菌中较早运用。Gordon等[59]用CRISPRi实现了蓝藻中心碳代谢动态调节和通量操控。Huang等[60]用CRISPRi调控蓝藻基因表达, 增加了琥珀酸合成。成功应用CRISPRi的第一个真核微藻是三角褐指藻。Nymark等[31]构建含dCas9基因的质粒构建物, 转化三角褐指藻, 实现了CpSRP(叶绿体信号识别因子基因)基因沉默, 突变藻株对强光敏感。Kao等[61]在莱茵衣藻中用CRISPRi干扰了PEPC(磷酸烯醇式丙酮酸羧化酶基因)表达, 降低了叶绿素含量, 提高了脂质积累。Ajjawi等[62]用CRISPR/Cas9系统引入反向遗传学插入突变, 调控微拟球藻(Nannochloropsis gaditana)ZnCys基因表达, 在半连续生长条件下, 使脂质含量较野生型提高2倍, 但突变株生长缓慢。Stukenberg等[63]构建编码dCas9基因的细菌质粒构建物, 转化三角褐指藻, 沉默了Vtc2(液泡转运伴侣复合体基因, vacuolar transport chaperone complex gene)和Pho4(假定存在的磷酸盐转运蛋白基因, putative phosphate transporter gene), 突变体在磷限制的条件下, Vtc2基因表达上调10倍, 而Pho4基因表达下调2倍。
dCas9仍具有结合sgRNA的功能, 而不具备剪切功能。sgRNA结合至基因的转录起始位点会沉默基因功能, sgRNA结合至转录抑制物或活化物, 会激活或抑制基因的转录。因此, 可以根据需要设计不同的sgRNA实现研究目的。CRISPRi和CRISPRa(转录激活, CRISPR activation)均是可逆的, 不会对基因组DNA产生永久性的改变。
7 讨论和展望在微藻中, 研究目标基因的功能, 在不影响其生长的情况下, 最优的方法是实现基因的完全敲除, 再进行表型或生化指标的后续验证。利用同源重组方法可以实现这种目的, 但是真核生物同源重组受非同源性末端修复抑制, 仅存在于细胞周期的特定阶段。我们可以通过干扰非同源末端修复的特异DNA连接酶, 或在细胞周期同源重组特定阶段进行转化, 来提高同源重组效率。在设计同源重组敲除基因过程中有RNP参与, 定点切割可提高转化效率[27]。有证据表明ZFN在莱茵衣藻中HDR(homology-directed repair)效率高于CRISPR/Cas9系统[64]。ZFN和Cas9定点敲除可实现目标微藻同源敲除验证微藻中绝大多数基因的功能, 将有益基因过表达, 可获得高生产力的藻株。同样, 组合使用Cas9和Cre重组酶, 在微拟球藻中已实现连续敲除内源基因和敲入新基因[35], 敲除生化过程中的酶基因, 可增加上游产物的积累。
在CRISPR/Cas9设计的过程中gRNA侧翼增加有核酶(ribozyme)HH和HDV, 可以更加精确切割[37], 用内源核定位信号(nuclear located signals, NLSs)替代哺乳动物NLSs提高Cas9靶向精度[54], 在设计sgRNA时提高特异性, 降低Cas蛋白与其它类似sgRNA分子结合, 避免非特异性切割[51], 选择GC含量低于70%的靶序列可降低脱靶效应[65]。模式微藻莱茵衣藻, 化脓性链球菌Cas9(SpCas9)对其毒性较强, 而金黄色葡萄球菌Cas9(SaCas9)毒性较弱, 因此更适合莱茵衣藻的基因编辑[30]。但是, SaCas9的精确靶向度不如SpCas9。最新鉴定出的SaCas9-HF是SaCas9的变体, 提高了SaCas9的精确度, 应用于人体细胞, 将脱靶活性降低了大约90%[66], 而SpCas9的变体MiCas9也显著提高单链寡核苷酸介导的精确编辑, 提高长片段基因插入效率, 减少缺失突变的产生[67]。在微藻中我们也可以利用这些高精确靶向的Cas9蛋白, 获得更高的编辑效率。
Cas9n(Cas9 nickase)与修饰单碱基酶连用构成CRISPR碱基编辑系统, 可以避免转化过程中引入缺失突变, 精确地对单链DNA中的碱基进行编辑[68]。此系统可检验微藻单碱基改变对微藻性状的影响。Cas9n(Cas9 nickase)和反转录酶连用构成引导编辑(CRISPR prime edition)系统, 可打断特定DNA位点的一条链, 引入一段序列, 高效造成插入突变。Cas9n是单链内切酶, 不产生双链DNA断裂, 最高只能精确插入约40 bp, 删除最高约80 bp[68-69]。
微藻生长快, 易培养, 基因组相对简单。莱茵衣藻、三角褐指藻、微拟球藻、小球藻等许多微藻都是基因工程研究常用微藻藻种。优秀藻种是高效生产多种多样物质的基础。基因编辑技术就是微藻藻种遗传改良的技术之一。CRISPR/Cas9技术成功率高、易操作、成本低, 比TALEN、ZFN等应用更广泛。目前, 以CRISPR/Cas9系统为基础而衍生出的多种基因调控, 例如CRISPR激活、CRISPR prime编辑、CRISPR碱基编辑、编辑组蛋白标记、甲基化编辑等, 都可以成为日后进行微藻基因编辑的有力工具。因此, 如何整合现有编辑技术、修饰现有编辑技术、阐明微藻经济性状遗传基础、培育优质微藻藻种是重要的研究领域之一。
| [1] |
HORVATH P, BARRANGOU R. CRISPR/Cas, the immune system of bacteria and archaea[J]. Science, 2010, 327(5962): 167-170. DOI:10.1126/science.1179555 |
| [2] |
JANSEN R, VAN EMBDEN J, GAASTRA W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43: 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [3] |
ISHINO Y, SHINAGAWA H, MAKINO K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product[J]. Journal of Bacteriology, 1987, 169(12): 5429-5433. DOI:10.1128/jb.169.12.5429-5433.1987 |
| [4] |
MOJICA F J, DÍEZ-VILLASEÑOR C, SORIA E, et al. Biological significance of a family of regularly spaced repeats in the genomes of Archaea, Bacteria and mitochondria[J]. Molecular Microbiology, 2000, 36(1): 244-246. DOI:10.1046/j.1365-2958.2000.01838.x |
| [5] |
JANSEN R, EMBDEN J D, GAASTRA W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Molecular Microbiology, 2002, 43(6): 1565-1575. DOI:10.1046/j.1365-2958.2002.02839.x |
| [6] |
HORVATH P, ROMERO D A, COÛTÉ-MONVOISIN A C, et al. Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus[J]. Journal of Bacteriology, 2008, 190(4): 1401-1412. DOI:10.1128/JB.01415-07 |
| [7] |
DEVEAU H, GARNEAU J E, MOINEAU S. CRISPR/ Cas system and its role in phage-bacteria interactions[J]. Annual Review of Microbiology, 2010, 64: 475-493. DOI:10.1146/annurev.micro.112408.134123 |
| [8] |
AL-ATTAR S, WESTRA E R, VAN DER OOST J, et al. Clustered regularly interspaced short palindromic repeats (CRISPRs): the hallmark of an ingenious antiviral defense mechanism in prokaryotes[J]. Biological Chemistry, 2011, 392(4): 277-289. |
| [9] |
BARRANGOU R, FREMAUX C, DEVEAU H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712. DOI:10.1126/science.1138140 |
| [10] |
MOJICA F J, DÍEZ-VILLASEÑOR C, GARCÍA-MARTÍNEZ J, et al. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. Journal of Molecular Evolution, 2005, 60(2): 174-182. DOI:10.1007/s00239-004-0046-3 |
| [11] |
GRISSA I, VERGNAUD G, POURCEL C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8: 172. DOI:10.1186/1471-2105-8-172 |
| [12] |
HOWARD J A, DELMAS S, IVANČIĆ-BAĆE I, et al. Helicase dissociation and annealing of RNA-DNA hybrids by Escherichia coli Cas3 protein[J]. Biochemical Journal, 2011, 439(1): 85-95. DOI:10.1042/BJ20110901 |
| [13] |
BROUNS S J, JORE M M, LUNDGREN M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960-964. DOI:10.1126/science.1159689 |
| [14] |
HALE C R, ZHAO P, OLSON S, et al. RNA-guided RNA cleavage by a CRISPR RNA-Cas protein complex[J]. Cell, 2009, 139(5): 945-956. DOI:10.1016/j.cell.2009.07.040 |
| [15] |
SOREK R, KUNIN V, HUGENHOLTZ P. CRISPR-a widespread system that provides acquired resistance against phages in bacteria and archaea[J]. Nature Reviews Microbiology, 2008, 6(3): 181-186. DOI:10.1038/nrmicro1793 |
| [16] |
KARGINOV F V, HANNON G J. The CRISPR system: small RNA-guided defense in bacteria and archaea[J]. Molecular Cell, 2010, 37(1): 7-19. DOI:10.1016/j.molcel.2009.12.033 |
| [17] |
MAKAROVA K S, HAFT D H, BARRANGOU R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577 |
| [18] |
MAKAROVA K S, ZHANG F, KOONIN E V. SnapShot: Class 1 CRISPR-Cas Systems[J]. Cell, 2017, 168(5): 946-946, e941. DOI:10.1016/j.cell.2017.02.018 |
| [19] |
SAPRANAUSKAS R, GASIUNAS G, FREMAUX C, et al. The Streptococcus thermophilus CRISPR/Cas system provides immunity in Escherichia coli[J]. Nucleic Acids Research, 2011, 39(21): 9275-9282. DOI:10.1093/nar/gkr606 |
| [20] |
JINEK M, CHYLINSKI K, FONFARA I, et al. A programmable dual-RNA─guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816. DOI:10.1126/science.1225829 |
| [21] |
DELTCHEVA E, CHYLINSKI K, SHARMA C M, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase Ⅲ[J]. Nature, 2011, 471(7340): 602-607. DOI:10.1038/nature09886 |
| [22] |
SHOJAEI S, GARDANOVA Z R, ZEKIY A O, et al. Optimizing sgRNA to improve CRISPR/Cas9 knockout efficiency: Special focus on human and animal cell[J]. Frontiers in Bioengineering & Biotechnology, 2021, 9: 775309. |
| [23] |
LIU X X, HOMMA A, SAYADI J, et al. Sequence features associated with the cleavage efficiency of CRISPR/Cas9 system[J]. Scientific Reports, 2016, 6: 19675. DOI:10.1038/srep19675 |
| [24] |
MOJICA F J M, DÍEZ-VILLASEÑOR C, GARCÍA-MARTÍNEZ J, et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system[J]. Microbiology, 2009, 155(Pt 3): 733-740. |
| [25] |
SCOLLAN M E. Chapter 2 - CRISPR-Cas orthologs and variants[C]//TSANG STEPHEN H. CRISPR genome surgery in stem cells and disease tissues. United States: Academic Press, 2022: 7-38.
|
| [26] |
JIANG W Z, BRUEGGEMAN A J, HORKEN K M, et al. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii[J]. Eukaryot Cell, 2014, 13(11): 1465-1469. DOI:10.1128/EC.00213-14 |
| [27] |
ANGSTENBERGER M, DE SIGNORI F, VECCHI V, et al. Cell synchronization enhances nuclear transformation and genome editing via Cas9 enabling homologous recombination in Chlamydomonas reinhardtii[J]. ACS Synthetic Biology, 2020, 9(10): 2840-2850. DOI:10.1021/acssynbio.0c00390 |
| [28] |
JIANG W Z, WEEKS D P. A gene-within-a-gene Cas9/ sgRNA hybrid construct enables gene editing and gene replacement strategies in Chlamydomonas reinhardtii[J]. Algal Research, 2017, 26: 474-480. DOI:10.1016/j.algal.2017.04.001 |
| [29] |
LIN W R, NG I S. Development of CRISPR/Cas9 system in Chlorella vulgaris FSP-E to enhance lipid accumulation[J]. Enzyme & Microbial Technology, 2020, 133: 109458. |
| [30] |
ORTEGA-ESCALANTE J A, JASPER R, MILLER S M. CRISPR/Cas9 mutagenesis in Volvox carteri[J]. Plant Journal, 2019, 97(4): 661-672. DOI:10.1111/tpj.14149 |
| [31] |
NYMARK M, SHARMA A K, SPARSTAD T, et al. A CRISPR/Cas9 system adapted for gene editing in marine algae[J]. Scientific Reports, 2016, 6: 24951. DOI:10.1038/srep24951 |
| [32] |
VOGLER B W, ASHFORD A, POSEWITZ M C. CRISPR/Cas9 disruption of glucan synthase in Nannochloropsis gaditana attenuates accumulation of β-1, 3-glucose oligomers[J]. Algal Research, 2021, 58: 102385. DOI:10.1016/j.algal.2021.102385 |
| [33] |
SLATTERY S S, DIAMOND A, WANG H, et al. An expanded plasmid-based genetic toolbox enables Cas9 genome editing and stable maintenance of synthetic pathways in Phaeodactylum tricornutum[J]. ACS Synthetic Biology, 2018, 7(2): 328-338. DOI:10.1021/acssynbio.7b00191 |
| [34] |
HOPES A, NEKRASOV V, KAMOUN S, et al. Editing of the urease gene by CRISPR-Cas in the diatom Thalassiosira pseudonana[J]. Plant Methods, 2016, 12: 49. DOI:10.1186/s13007-016-0148-0 |
| [35] |
VERRUTO J, FRANCIS K, WANG Y, et al. Unrestrained markerless trait stacking in Nannochloropsis gaditana through combined genome editing and marker recycling technologies[J]. Proceedings of the National Academy of Sciences, 2018, 115(30): E7015. |
| [36] |
NADUTHODI M I S, SÜDFELD C, AVITZIGIANNIS E K, et al. Comprehensive genome engineering toolbox for microalgae Nannochloropsis oceanica based on CRISPR-Cas systems[J]. ACS Synthetic Biology, 2021, 10(12): 3369-3378. DOI:10.1021/acssynbio.1c00329 |
| [37] |
WANG Q T, GONG Y H, HE Y H, et al. Genome engineering of Nannochloropsis with hundred-kilobase fragment deletions by Cas9 cleavages[J]. Plant Journal, 2021, 106(4): 1148-1162. DOI:10.1111/tpj.15227 |
| [38] |
POLINER E, TAKEUCHI T, DU Z Y, et al. Nontransgenic marker-free gene disruption by an episomal CRISPR system in the oleaginous microalga, Nannochloropsis oceanica CCMP1779[J]. ACS Synthetic Biology, 2018, 7(4): 962-968. DOI:10.1021/acssynbio.7b00362 |
| [39] |
MOOSBURNER M A, GHOLAMI P, MCCARTHY J K, et al. Multiplexed knockouts in the model diatom Phaeodactylum by episomal delivery of a selectable Cas9[J]. Frontiers in Microbiology, 2020, 11: 5. DOI:10.3389/fmicb.2020.00005 |
| [40] |
HU L N, FENG S Y, LIANG G F, et al. CRISPR/Cas9-induced β-carotene hydroxylase mutation in Dunaliella salina CCAP19/18[J]. AMB Express, 2021, 11(1): 83. DOI:10.1186/s13568-021-01242-4 |
| [41] |
SHIN S E, LIM J M, KOH H G, et al. CRISPR/Cas9-induced knockout and knock-in mutations in Chlamydomonas reinhardtii[J]. Scientific Reports, 2016, 6(1): 27810. DOI:10.1038/srep27810 |
| [42] |
YOSHIMITSU Y, ABE J, HARAYAMA S. Cas9-guide RNA ribonucleoprotein-induced genome editing in the industrial green alga Coccomyxa sp. strain KJ[J]. Biotechnology for Biofuels, 2018, 11(1): 326. DOI:10.1186/s13068-018-1327-1 |
| [43] |
KRISHNAN A, CANO M, BURCH T A, et al. Genome editing using Cas9-RNA ribonucleoprotein complexes in the high-productivity marine alga Picochlorum celeri[J]. Algal Research, 2020, 49: 101944. DOI:10.1016/j.algal.2020.101944 |
| [44] |
SERIF M, DUBOIS G, FINOUX A L, et al. One-step generation of multiple gene knock-outs in the diatom Phaeodactylum tricornutum by DNA-free genome editing[J]. Nature Communication, 2018, 9(1): 3924. DOI:10.1038/s41467-018-06378-9 |
| [45] |
KIM J, CHANG K S, LEE S, et al. Establishment of a genome editing tool using CRISPR-Cas9 in Chlorella vulgaris UTEX395[J]. International Journal of Molecular Sciences, 2021, 22(2): 480. DOI:10.3390/ijms22020480 |
| [46] |
BAEK K, KIM D H, JEONG J, et al. DNA-free two-gene knockout in Chlamydomonas reinhardtii via CRISPR-Cas9 ribonucleoproteins[J]. Scientific Reports, 2016, 6(1): 30620. DOI:10.1038/srep30620 |
| [47] |
BAEK K, YU J, JEONG J, et al. Photoautotrophic production of macular pigment in a Chlamydomonas reinhardtii strain generated by using DNA-free CRISPR-Cas9 RNP-mediated mutagenesis[J]. Biotechnology Bioengineering, 2018, 115(3): 719-728. DOI:10.1002/bit.26499 |
| [48] |
SHIN Y S, JEONG J, NGUYEN T H T, et al. Targeted knockout of phospholipase A(2) to increase lipid productivity in Chlamydomonas reinhardtii for biodiesel production[J]. Bioresource Technology, 2019, 271: 368-374. DOI:10.1016/j.biortech.2018.09.121 |
| [49] |
CHANG K S, KIM J, PARK H, et al. Enhanced lipid productivity in AGP knockout marine microalga Tetraselmis sp. using a DNA-free CRISPR-Cas9 RNP method[J]. Bioresource Technology, 2020, 303: 122932. DOI:10.1016/j.biortech.2020.122932 |
| [50] |
NOMURA T, INOUE K, UEHARA-YAMAGUCHI Y, et al. Highly efficient transgene-free targeted mutagenesis and single-stranded oligodeoxynucleotide-mediated precise knock-in in the industrial microalga Euglena gracilis using Cas9 ribonucleoproteins[J]. Plant Biotechnology Journal, 2019, 17(11): 2032-2034. DOI:10.1111/pbi.13174 |
| [51] |
KIM D, KIM J, HUR J K, et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells[J]. Nature Biotechnology, 2016, 34(8): 863-868. DOI:10.1038/nbt.3609 |
| [52] |
NADUTHODI M I S, MOHANRAJU P, SÜDFELD C, et al. CRISPR-Cas ribonucleoprotein mediated homology-directed repair for efficient targeted genome editing in microalgae Nannochloropsis oceanica IMET1[J]. Biotechnology for Biofuels, 2019, 12: 66. DOI:10.1186/s13068-019-1401-3 |
| [53] |
FERENCZI A, PYOTT D E, XIPNITOU A, et al. Efficient targeted DNA editing and replacement in Chlamydomonas reinhardtii using Cpf1 ribonucleoproteins and single-stranded DNA[J]. Proceedings of the National Academy of Sciences, 2017, 114(51): 13567. DOI:10.1073/pnas.1710597114 |
| [54] |
WANG Q T, LU Y D, XIN Y, et al. Genome editing of model oleaginous microalgae Nannochloropsis spp. by CRISPR/Cas9[J]. Plant Journal, 2016, 88(6): 1071-1081. DOI:10.1111/tpj.13307 |
| [55] |
KILIAN O, BENEMANN C S E, NIYOGI K K, et al. High-efficiency homologous recombination in the oil-producing alga Nannochloropsis sp.[J]. Proceedings of the National Academy of Sciences, 2011, 108(52): 21265. DOI:10.1073/pnas.1105861108 |
| [56] |
QI L S, LARSON M H, GILBERT L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022 |
| [57] |
BIKARD D, JIANG W Y, SAMAI P, et al. Programmable repression and activation of bacterial gene expression using an engineered CRISPR-Cas system[J]. Nucleic Acids Research, 2013, 41(15): 7429-7437. DOI:10.1093/nar/gkt520 |
| [58] |
LV L, REN Y L, CHEN J C, et al. Application of CRISPRi for prokaryotic metabolic engineering involving multiple genes, a case study: Controllable P(3HB-co-4HB) biosynthesis[J]. Metabolic Engineering, 2015, 29: 160-168. DOI:10.1016/j.ymben.2015.03.013 |
| [59] |
GORDON G C, KOROSH T C, Cameron J C, et al. CRISPR interference as a titratable, trans-acting regulatory tool for metabolic engineering in the cyanobacterium Synechococcus sp. strain PCC 7002[J]. Metabolic Engineering, 2016, 38: 170-179. DOI:10.1016/j.ymben.2016.07.007 |
| [60] |
HUANG C H, SHEN C R, Li H, et al. CRISPR interference (CRISPRi) for gene regulation and succinate production in cyanobacterium S. elongatus PCC 7942[J]. Microbial Cell Factories, 2016, 15(1): 196. DOI:10.1186/s12934-016-0595-3 |
| [61] |
KAO P H, NG I S. CRISPRi mediated phosphoenolpyruvate carboxylase regulation to enhance the production of lipid in Chlamydomonas reinhardtii[J]. Bioresource Technology, 2017, 245: 1527-1537. DOI:10.1016/j.biortech.2017.04.111 |
| [62] |
AJJAWI I, VERRUTO J, AQUI M, et al. Lipid production in Nannochloropsis gaditana is doubled by decreasing expression of a single transcriptional regulator[J]. Nature Biotechnology, 2017, 35(7): 647-652. DOI:10.1038/nbt.3865 |
| [63] |
STUKENBERG D, ZAUNER S, DELL'AQUILA G, et al. Optimizing CRISPR/Cas9 for the diatom Phaeodactylum tricornutum[J]. Frontiers in Plant Science, 2018, 9: 740. DOI:10.3389/fpls.2018.00740 |
| [64] |
GREINER A, KELTERBORN S, EVERS H, et al. Targeting of photoreceptor genes in Chlamydomonas reinhardtii via zinc-finger nucleases and CRISPR/Cas9[J]. Plant Cell, 2017, 29(10): 2498-2518. DOI:10.1105/tpc.17.00659 |
| [65] |
RUSSO M T, AIESE CIGLIANO R, SANSEVERINO W, et al. Assessment of genomic changes in a CRISPR/ Cas9 Phaeodactylum tricornutum mutant through whole genome resequencing[J]. PeerJ, 2018, 6: e5507. DOI:10.7717/peerj.5507 |
| [66] |
TAN Y, CHU A H Y, BAO S, et al. Rationally engineered Staphylococcus aureus Cas9 nucleases with high genome-wide specificity[J]. Proceedings of the National Academy of Sciences, 2019, 116(42): 20969-20976. DOI:10.1073/pnas.1906843116 |
| [67] |
MA L Y, RUAN J X, SONG J, et al. MiCas9 increases large size gene knock-in rates and reduces undesirable on-target and off-target indel edits[J]. Nature Communication, 2020, 11(1): 6082. DOI:10.1038/s41467-020-19842-2 |
| [68] |
KIM H K, YU G, PARK J, et al. Predicting the efficiency of prime editing guide RNAs in human cells[J]. Nature Biotechnology, 2021, 39(2): 198-206. DOI:10.1038/s41587-020-0677-y |
| [69] |
ANZALONE A V, RANDOLPH P B, DAVIS J R, et al. Search-and-replace genome editing without double-strand breaks or donor DNA[J]. Nature, 2019, 576(7785): 149-157. DOI:10.1038/s41586-019-1711-4 |
 2023, Vol. 47
2023, Vol. 47


