文章信息
- 万圆圆, 赵峰, 王超锋, 张越, 徐奎栋. 2024.
- WAN Yuanyuan, ZHAO Feng, WANG Chaofeng, ZHANG Yue, XU Kuidong. 2024.
- 基于高通量测序的表层水体纤毛虫多样性评估方法比较与优化
- Comparison and optimization of high-throughput sequence processing strategies: a case study regarding ciliate diversity in surface seawater
- 海洋科学, 48(4): 83-96
- Marine Sciences, 48(4): 83-96.
- http://dx.doi.org/10.11759/hykx20220823001
-
文章历史
- 收稿日期:2022-08-23
- 修回日期:2022-10-08
2. 中国科学院大学, 北京 100049;
3. 中国科学院海洋研究所 海洋生态与环境科学重点实验室, 山东 青岛 266071
2. University of Chinese Academy of Sciences, Beijing 100049, China;
3. CAS Key Laboratory of Marine Ecology and Environmental Sciences, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China
纤毛虫是原生生物的重要类群, 具有多样性高、体积小、生命周期短、代谢率高、对环境变化敏感等特点, 在土壤、海洋、淡水中广泛分布[1]。在海洋生态系统中, 纤毛虫作为浮游和底栖微食物网的重要组成部分, 具有十分多样化的生态策略, 从自由生活到共生生活, 从异养到混合营养等[2], 在生物地球化学循环、能量流动、食物链交互等过程中都有着重要的作用[3-6]。
物种丰富的砂壳纤毛虫是浮游原生动物中形态最多样化的类群之一, 通常存在于全球的海洋表层水[7]。砂壳纤毛虫有壳, 易采集和保存, 可依据壳体形态进行鉴定, 已有大量针对砂壳纤毛虫形态特征的描述性文献[8-9]。基于扩增子的高通量测序技术广泛应用于微型生物多样性的研究, 通过高通量测序技术检获了更高的物种多样性, 提供了更全面和更准确的原生生物多样性和分布格局信息, 极大拓展了对纤毛虫等原生生物的认知[10-16]。环境DNA和环境RNA测序技术均可展现纤毛虫的多样性和群落构成, 但检获结果存在差异[10]。随着海量测序数据的获得, 多样化的数据分析软件不断涌现, 例如USEARCH[17]、VSEARCH[18]、QIIME[19]等。为了更大程度上提高数据分析的速度和精确度, 不断有算法被推出, 例如UPARSE[20]、UNOISE3[21]、DADA2[22]等, 并且算法仍在不断优化和更新迭代[23-26]。
分析基于扩增子的高通量测序数据时, 通常根据序列相似度聚类划分可操作分类单元(operational taxonomic units, OTU), 97%序列相似度应用最广[20, 27-29]。近年, 基于单核苷酸变异分析方法的提出撼动了基于97%序列相似度划分OTU的地位[22]。每个单核苷酸变异体对应一个准确的序列[30], 剔除测序错误的序列, 可更加真实的反应出物种的丰度[31-32]。目前基于单核苷酸变异的分析方法主要包括DADA2分析法与UNOISE算法。通过DADA2获得扩增序列变异(amplicon sequence variants, ASV), 该算法的核心在于测序错误校正[22, 33]。UNOISE也采用测序错误校正的算法, 得到的操作单元称为zOTU (zero-radius OTU), 其也可看成是聚类100%而得到的OTU[34]。二者的不同在于所采用的公式存异, 且UNOISE默认去除序列数小于8的序列, DADA2默认去掉单体(singleton)。有研究系统比较了不同DADA2、UNOISE等算法[35], 结果表明DADA2和UNOISE3所计算的微生物群落组成、相对丰度、β多样性等结果相似, 但UNOISE3的运行速度比DADA2快1 200倍[35]。并且DADA2对计算机配置要求更高, 占用资源更多。
近年来越来越多的研究者在数据分析中采用单核苷酸变异分析方法[33], 然而前期基于纤毛虫单细胞的高通量测序发现, 不同物种之间细胞内核糖体小亚基基因的拷贝差异可达97%[36]。Gong和Wang等人验证了纤毛虫的OTU聚类阈值在95%~99%的可靠性[37-38]。这一系列研究结果, 挑战着基于单核苷酸变异分析方法的精准度。总体来讲, 单核苷酸变异和相似度聚类等不同分析方法如何影响多样性分析结果仍然不明, 需要对分子分析方法进行有效的评估, 判断不同操作分类单元和单核苷酸序列变异体对微型生物多样性的影响。
此外, 数据处理的其他关键步骤也直接影响多样性的评估结果, 例如嵌合体处理、聚类相似度的选择、合并相似分类单元以及去除稀有类群等的影响[11, 20, 34, 39-40]。物种注释分析主要依赖于参考数据库[41], 由于现有参考数据库的不完备, 经常会导致下游出现多个OTUs比对至同一物种的现象[42-44]。因此, 已有研究者对原始OTU表的准确度存疑, 并使用二次处理再聚类的操作以提高物种表的稳定性和准确率[45]。合并这些OTUs是否能改善分子数据的结果有待考究。另外, 高通量测序技术通常检获高比例的稀有类群, 稀有类群的筛滤是否会对分子数据造成影响仍然不明[46-47]。因此, 现亟需对高通量测序数据的分析方法及主要参数进行系统评估和比较, 为分子多样性分析提供参考。
本研究选以易于形态鉴定的砂壳纤毛虫作为研究对象, 通过比较DNA测序、RNA (cDNA)测序两种分子手段以及与形态学手段所检获的砂壳虫多样性, 探究基于单核苷酸变异和相似度聚类等不同分析方法对纤毛虫多样性和物种组成分析的影响; 评估序列分析流程中关键步骤: 嵌合体处理、分析方法选择、合并相似分类单元以及去除稀有类群等对研究结果影响。以期为纤毛虫等真核微生物的分子多样性研究提供科学的数据分析流程, 促进真核微生物多样性研究标准化体系构建。
1 材料与方法 1.1 调查站位与样品采集本研究样品于2019年5月搭乘“科学”号科学考察船于西太平洋采集, 共计9个站位点, 分别为R29 (131.583°E, 20.133°N)、R30(132.65°E, 19.067°N)、R31(133.717°E, 17.95°N)、R32(134.8°E, 16.833°N)、R33(135.8°E, 15.8°N)、R34(136.833°E, 14.7°N)、R35(137.733°E, 13.75°N)、R36(138.817°E, 12.633°N)和R37(139.65°E, 11.567°N)。
每个站位采集20 L表层海水, 海水样品通过200 µm孔径的筛绢过滤预处理, 而后通过0.22 µm孔径的聚碳酸酯滤膜过滤处理, 将20 L海水过滤至一张膜上。过滤时, 为确保细胞的完整性, 需控制蠕动泵的转速及压力。过滤后将滤膜放入2 mL无RNA酶的冻存管中, 快速置于在液氮中冻存, 随后将保存的冻存管带回实验室, 保存于−80 ℃冰箱内。在每次使用后, 对采水和过滤器材进行无菌蒸馏水清洗, 防止样品间交叉污染。
1.2 DNA和RNA提取及PCR扩增滤膜总DNA和总RNA提取选用All Prep DNA/RNA Mini Kit (Qiagen, Germany), 此试剂盒可同时完成滤膜上的DNA和RNA提取。总RNA采用PrimerScript Ⅱ1st strand cDNA Synthesis Kit反转录成cDNA。每个海水样品各一份DNA以及cDNA, 通过巢式PCR对纤毛虫18S rRNA基因V4区进行特异性扩增[48]。首先, 采用纤毛虫特异性引物CilF(碱基序列: 5′-TGGTAGTGTATTGGACWACCA-3′)和CilRI-Ⅲ(碱基序列: 5′-TCTGATTGTCTTTGATCCCCT-3′) 针对纤毛虫的18S rRNA基因进行扩增, 扩增长度约600 bp[49]; 然后, 以第一轮PCR产物为模板, 采用真核特异性引物EukF(碱基序列: 5′-CCAGCASCYGCGGTAATTCC-3′)和EukR(碱基序列: 5′-ACTTTCGTTCTTGATYRA-3′)对V4高变区进行特异性扩增, 扩增长度约400 bp[50]。以上PCR处理每份DNA/cDNA进行三个重复。
1.3 测序以及数据分析琼脂糖凝胶电泳检测PCR产物质量, 若符合要求, 则将来自同一样品的3个重复的DNA/cDNA的PCR产物分别进行合并。样品进行Illumina Hiseq测序, 测序过程如下: 使用NEB Next Ultra DNA Library Prep Kit (New England Biolabs, USA)试剂盒构建文库, 检测文库, 合格后上机测序。
分子分析方法采用软件USEARCH (v. 10.0.240)[17], VSEARCH (v. 2.15.2)[18], 去冗余参数miniuniquesize = 8, 参考数据库选用Protist Ribosomal Reference (PR2, v. 4.14.0)[51], VSEARCH生成特征表id = 0.97[40]。基于Liu等[40]提供的数据分析流程, 多样性分析的主要步骤依次为序列去嵌合体、不同聚类相似度的选择、分类单元的合并、稀有OTU的过滤处理(图 1)。如前所述, 单核苷酸变异分析的两种方法DADA2和UNOISE获得的ASV或zOTU对结果影响不大, 但DADA2运行较慢, UNOISE3的运行时间比DADA2快1 200倍[35], 且DADA2对计算机配置要求更高, 占用资源更多。因此我们的单核苷酸序列变异体是通过UNOISE3算法去噪获得[52], 利用UNOISE3的denoise和-cluster_smallmem命令获得zOTU; 序列相似度的聚类OTU使用UPARSE聚类[20], 获得不同相似度聚类OTUs: OTU99%, OTU98%, OTU97%, OTU96%, OTU95%, OTU90%。
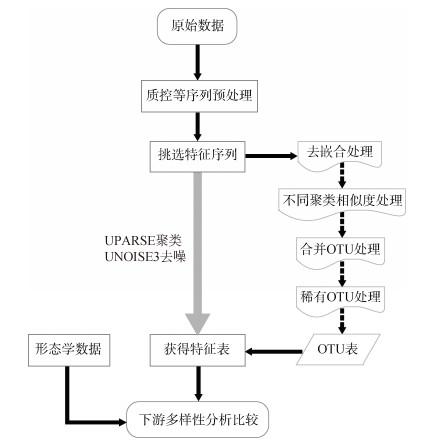 |
| 图 1 数据分析流程图 Fig. 1 Data analysis flowchart |
97%聚类相似度一直作为领域内应用最多的聚类阈值, 因此有关不同方法去嵌合体的结果比较, 序列相似度聚类方法以相似度97%聚类的数据为结果, 单核苷酸变异法以通过UNOISE3获得的zOTU数据为结果。根据USEARCH官网(http://www.drive5.com/usearch/)的建议, zOTU去噪手段是将相似度的标准设为100%进行聚类[34]。
分类单元合并原则是将比对注释至同一物种的OTUs, 并且与PR2数据库(v. 4.14.0)序列比对率为99%以上合并成1个分类单元; 每个未注释的物种将仍视作为1个分类单元, 不作合并。合并所选用数据为zOTU、OTU99%、OTU98%、OTU97%的去嵌合体的数据。
相对丰度(即类群序列数占总序列数的比例)小于0.1%的OTUs设为稀有OTUs。探究稀有类群对群落构成分析影响时, 设置11个过滤梯度, 包括不去除OTU, 去除相对丰度低于0.01%、0.02%、0.03%、0.04%、0.05%、0.06%、0.07%、0.08%、0.09%和0.10%的OTUs。
1.4 砂壳纤毛虫样品采集和形态鉴定在各站位使用船载走航采水系统对表层海水(100 L)用10 μm的筛绢进行过滤, 过滤后网滴管内的海水浓缩样品约100 mL用鲁戈试剂(终浓度1%)固定, 4 ℃黑暗处保存。实验室内, 用量筒测量每个样品的体积(mL); 取16 mL于沉降杯中沉降24 h, 在Olympus IX71倒置显微镜下(100×或400×)计数砂壳纤毛虫(N)。依据壳的形状和尺寸, 参考分类学文献对砂壳纤毛虫进行种类鉴定[53-54]。
1.5 数据统计分析通过对主要步骤的比较, 对输出的分子特征表进行结果优化, 与基于形态学手段所检获得的砂壳纤毛虫数据比较评估。
由于参考数据库中砂壳虫相关序列不完整, 未注释类群中(Unassigned)可能存在砂壳虫, 因此进一步结合了NCBI数据库中代表的砂壳虫序列和分子手段提取的砂壳虫序列建立系统发育树。NCBI(National Center for Biotechnology Information)的砂壳纤毛虫参考序列号如下: JQ408215.1, JQ408214.1, JQ408211.1, JQ408209.1, JQ408206.1, JQ408204.1, JQ408203.1, JQ408202.1, JQ408193.1, JQ408183.1, JQ408188.1, MN185012.1, JQ408179.1, JQ408174.1, MH673409.1, KY290299.1, KY290295.1, KY290293.1, KU715801.1, KT390000.1, KT389951.1, EU024990.1, AY143563.1。系统发育树的结果显示, 未注释的分子序列含有砂壳虫, 例如波膜虫属(Undella), 而该砂壳纤毛虫属通过形态学手段检获。同时也确定了分子手段未检获的囊坎虫属(Ascampbelliella)和叶状虫属(Petalotricha)。囊坎虫作为形态学检获优势种, 可能由于其分子不易提取、PCR/引物偏好性、NCBI参考序列有误等原因, 分子手段未检获到。
本实验制图通过R中的ggplot2包完成, 系统发育树建立命令同样采用数据分析流程中的命令操作, 使用IQ-TREE构建最大似然树。我们通过Kappa检验的方法来评估不同方法检获群落的相似度, Kappa统计检验分析于SPSSAU project平台(2022.Version 22.0. https://www.spssau.com)。
2 实验结果 2.1 嵌合体的处理的比较DNA测序共检获780 594条序列, RNA测序共检获364 337条序列。数据分析过程中, 不同算法检测到的嵌合体数据存在差异: 通过UNOISE3算法, DNA测序检获1 020 zOTUs, 1 374个嵌合体。RNA测序检获727 zOTUs, 657个嵌合体; 通过UPARSE算法, 在97%序列相似度下聚类, DNA测序检获488 OTUs, 1 189个嵌合体。RNA测序检获279 OTUs, 607个嵌合体。
DNA测序中, 未去嵌合体所检获的砂壳虫共14属、151 zOTUs。去嵌合体zOTU检获15属、53 zOTUs (表 1)。97%聚类相似度一直作为领域内应用最多的聚类阈值, 因此本部分去嵌合体OTU以相似度97%聚类, 检获12属、22 OTUs。RNA测序中, 未去嵌合体所检获砂壳虫12属、129 zOTUs, 去嵌合体zOTU检获14属、51 zOTUs, 去嵌合体OTU检获11属、21 OTUs。未去嵌合体数据的未注释率(Unassigned占比)平均为0.82% (DNA测序: 1.10%, RNA测序: 0.53%), zOTU去嵌合体的平均未注释率为0.72% (DNA测序: 0.82%, RNA测序: 0.61%), OTU去嵌合体的平均未注释率为0.50%(DNA测序: 1.55%, RNA测序: 0.46%)。去嵌合体可适当降低分子数据未注释的比例。
| 检获方法 | 砂壳虫属数 | 砂壳虫种数 | |||||
| DNA | RNA | 形态学 | DNA | RNA | 形态学 | ||
| 未去嵌合体 | 14 | 12 | — | 151 | 129 | — | |
| zOTU | 15 | 14 | — | 53 | 51 | — | |
| 97%OTU | 12 | 11 | — | 22 | 21 | — | |
| 形态学 | — | — | 15 | — | — | 32 | |
| 注: “—”表示无此数据 | |||||||
形态学总检获砂壳虫15属、32种。未去嵌合体时, DNA测序检获结果与形态学检获结果的Kappa值为−0.514 (P=0.015), RNA测序Kappa值为−0.522 (P=0.012)。去嵌合体zOTU的DNA测序Kappa值为−0.583 (P=0.005), RNA测序Kappa值为−0.571 (P=0.007)。去嵌合体OTU的DNA测序Kappa值为−0.522 (P=0.012), RNA测序Kappa值为−0.474 (P=0.020)。
虽然去嵌合体会造成假阴性, 但分析结果表明去嵌合体并未对砂壳虫群落组成结构造成太大影响(图 2a-c)。但从总检获量来说, 未去嵌合体检获的的砂壳虫种数比形态学所检获的砂壳虫将近大5倍(表 1)。另外, 将每个站点的砂壳虫数据进一步提取后得到的结论也和前相似(图 2d), 未去嵌合体的数据离形态学数据偏差较大, 而去嵌合体后的两种分子手段与形态学数据更接近。
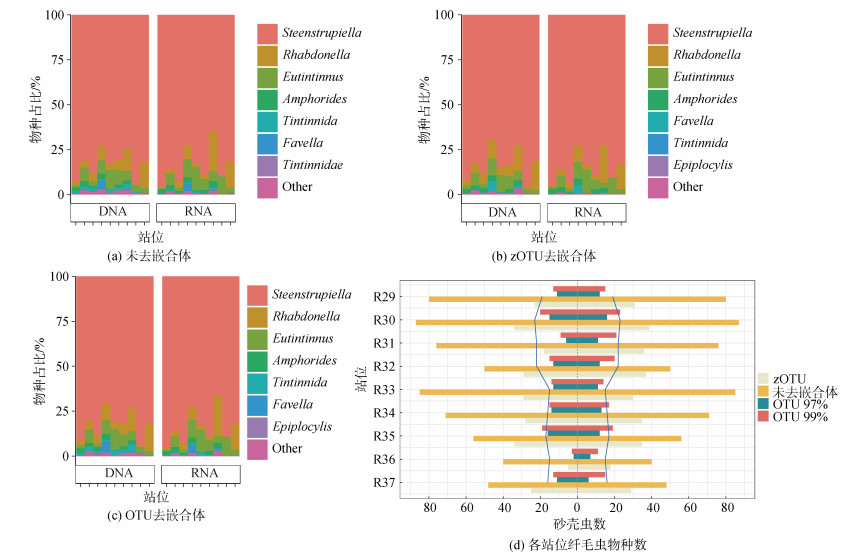 |
| 图 2 嵌合体处理下砂壳虫物种组成及多样性情况 Fig. 2 Taxonomic composition and diversity of tintinnids under chimera processes 注: 中轴线以左为DNA测序所得物种数, 右为RNA测序所得物种数 |
对比聚类梯度下丰富度后发现, 总体趋势呈现相似度越高, 得到的砂壳虫OTUs越多, 丰富度越高(图 3a), 检测到的嵌合体也越多(表 2), 且DNA测序获得的结果都比RNA测序的结果高。相似度97%以上(即97%OTU, 98%OTU, 99%OTU, zOTU)所得的群落结构结果相似(图 3c), 阈值为95%的组和90%的组的结果与97%~100%相似度的结果差异较大。如图 3b, 通过95%及以下相似度检获的砂壳虫占所有检测物种的比例低。在阈值为95%以上的几个组中, 聚类相似度为96%的时候砂壳虫检测比例最高, 其次是97%相似度, OTU聚类相似度99%略高于zOTU相似度组, 相似度为98%时砂壳虫占比最低。在不同聚类相似度中, OTU99%所获物种囊括了zOTU, OTU98%, OTU97%和OTU96%全部所检获的砂壳虫属。
 |
| 图 3 不同聚类相似度检获砂壳虫结果比较 Fig. 3 Comparison of results derived from different cluster similarities of tintinnids |
| 相似度 | 嵌合体 | 比对率 | 平均长度/bp | 序列数 | 砂壳虫属数 | 砂壳虫种数 | ||||||
| 数量 | 比例 | 总 | 砂壳虫 | DNA | RNA | DNA | RNA | |||||
| zOTU | 315 | 25.2% | 93.55% | 372 | 1 146 202 | 278 603 | 15 | 14 | 53 | 51 | ||
| OTU99% | 181 | 25.0% | 97.70% | 372 | 1 123 475 | 268 443 | 16 | 15 | 29 | 28 | ||
| OTU98% | 106 | 22.1% | 84.63% | 373 | 1 036 960 | 230 455 | 13 | 12 | 24 | 23 | ||
| OTU97% | 73 | 19.5% | 67.46% | 373 | 826 550 | 230 401 | 12 | 11 | 22 | 21 | ||
| OTU96% | 56 | 18.2% | 63.17% | 373 | 773 966 | 205 600 | 12 | 12 | 19 | 19 | ||
| OTU95% | 44 | 17.6% | 42.49% | 373 | 520 567 | 11 159 | 10 | 10 | 15 | 15 | ||
| OTU90% | 14 | 10.7% | 12.17% | 374 | 149 087 | 6 043 | 4 | 4 | 6 | 6 | ||
在不同聚类相似度中与形态结果比较, OTU99% (DNA测序检获16属, RNA测序检获15属)与形态学数据(15属)最为接近, 两种方法共同检测到的物种有8个属, 分别为翁状虫属(Amphorides)、网梯虫属(Climacocylis)、类管虫属(Dadayiella)、网膜虫属(Epiplocylis)、号角虫属(Salpingella)、真铃虫属、条纹虫属以及细翁虫属。根据各站位砂壳虫数比较, OTU99%仍是最为接近形态学数据的(图 2d)。OTU99%和zOTU检获的砂壳虫物种数量较其他几个相似度检获的砂壳虫多2~3个属, 但zOTU所检获的砂壳虫数(DNA测序检获53个OTUs, RNA测序检获51个OTUs)与形态学结果(32种)比较存在严重的数据膨胀。因此, 砂壳虫的聚类相似度选择99%。
此外, 通过不同砂壳虫相对丰度的计算(图 3d, e, 表 3), 图 3d, e和表 3中的DNA和RNA方法检获的相对丰度为96%~100%聚类梯度相对丰度的平均值。由图可见, 分子和形态学方法共同检获的砂壳虫的趋势基本一致。96%的聚类相似度所得的共有砂壳虫相对丰度较其他组低, 与之前结论类似, 97%以上的聚类相似度与形态学检获的结果相近。形态方法和分子方法对各砂壳虫类群检获的平均相对丰度见表 3。两种分子手段之间检获的平均相对丰度结果类似, 而两种分子手段与形态学之间除了细翁虫属的相对丰度有较大的差别, 其他的检获结果基本一致, 虽然两种手段对细翁虫属在相对丰度数值上相差较大, 但不论是分子手段还是形态学方法, 细翁虫属均为当前检获相对丰度最高的类群。
| 砂壳虫属 | Mor | DNA | RNA |
| 翁状虫属Amphorides | 2.39% | 2.10% | 1.61% |
| 网梯虫属Climacocylis | 0.11% | 0.02% | 0.01% |
| 网膜虫属Epiplocylis | 0.62% | 0.09% | 0.03% |
| 真铃虫属Eutintinnus | 5.27% | 2.12% | 1.54% |
| 条纹虫属Rhabdonella | 13.09% | 6.81% | 9.45% |
| 号角虫属Salpingella | 0.30% | 0.06% | 0.03% |
| 细翁虫属Steenstrupiella | 34.72% | 84.98% | 85.96% |
zOTU检获的DNA数据与形态学数据Kappa检验结果见表 4。聚类相似度为99%时Kappa值最大, 即接近形态学数据。
| 相似度 | DNA | RNA |
| OTU100% | −0.583 (P = 0.005) | −0.571 (P = 0.007) |
| OTU99% | −0.481 (P = 0.021) | −0.467 (P = 0.029) |
| OTU98% | −0.485 (P = 0.023) | −0.444 (P = 0.035) |
| OTU97% | −0.522 (P = 0.012) | −0.474 (P = 0.020) |
| OTU96% | −0.522 (P = 0.012) | −0.522 (P = 0.012) |
| OTU95% | −0.500 (P = 0.010) | −0.500 (P = 0.010) |
DNA和RNA测序所得序列, 基于单核苷酸变异分析获得的砂壳纤毛虫zOTU数和基于形态学检获的物种数见表 1。DNA测序检获的砂壳虫数普遍相较RNA测序以及形态学方法获得的物种多1~22个(表 5)。DNA测序检获的砂壳虫共15属、53 zOTUs。RNA测序检获的砂壳虫共14属、51 zOTUs。形态学检测到的砂壳虫共15属、32种。三种方法共同检获的有7属、5种, DNA比RNA测序多检获的一个拟网膜虫属(Epiplocyloides)。有8个属仅通过形态学鉴定获得, 而8个属仅通过分子手段检获(图 4a)。在属阶元上, 两种分子方法检测得到的砂壳虫群落结果类似(图 4b)。DNA测序检获砂壳虫物种前三依次为细翁虫属(Steenstrupiella)、条纹虫属(Rhabdonella)以及真铃虫属(Eutintinnus), 占砂壳虫群落比分别为84.26%、7.01%、4.65%。RNA测序检获砂壳虫物种前3依次为细翁虫属(85.83%)、条纹虫属(7.29%)以及真铃虫属(4.68%)。而形态学方法获检砂壳虫较高占比的依次为细翁虫属、囊坎虫属、条纹虫属以及真铃虫属, 占比分别为37.42%、25.97%、14.10%、5.68%。与先前建树结果一致, 囊坎虫属仅为形态学检获, 而两种分子手段所检获不到。
| 站位 | DNA | RNA | Mor |
| R29 | 30 | 22 | 19 |
| R30 | 38 | 33 | 23 |
| R31 | 35 | 18 | 22 |
| R32 | 36 | 28 | 22 |
| R33 | 29 | 28 | 15 |
| R34 | 34 | 27 | 16 |
| R35 | 34 | 33 | 17 |
| R36 | 17 | 5 | 15 |
| R37 | 26 | 25 | 16 |
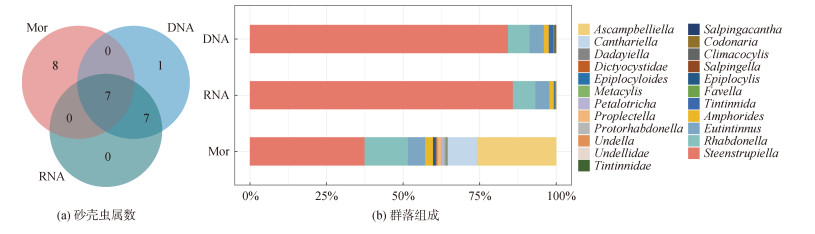 |
| 图 4 形态学方法和分子方法检获结果比较 Fig. 4 Comparison of results obtained by morphological and DNA/RNA sequencing techniques |
经过合并相似分类操作单元, DNA和RNA方法所减少的OTUs数目基本相似, 合并后的OTUs还是保持了一定的数目(表 6)。通过对比合并前后数据, OTU99%得到的结果还是比其他相似度组的砂壳虫检获结果更接近形态学检获结果。此外, 合并OTU/zOTU对去噪获得的zOTU结果具有较好的作用, 可以通过合并zOTU来减少去噪检获的数据噪音。但对于其他相似度, 特别是OTU99%来说, 因检获的砂壳虫数本身没有去噪所得的zOTUs多, 经过合并OTU以后会致其检获砂壳虫数更少。例如99%相似度在合并前DNA检获砂壳虫29 OTUs, 合并后减少为27 OTUs。而形态学检获砂壳虫为32属, 因此合并会使得其结果与形态学数据更加偏差, 导致误差加大。
| 分子方法 | 合并前 | 合并后 | |||
| DNA | RNA | DNA | RNA | ||
| zOTU | 53 | 51 | 41 | 40 | |
| OTU99% | 29 | 28 | 27 | 26 | |
| OTU98% | 24 | 23 | 22 | 21 | |
| OTU97% | 22 | 21 | 21 | 20 | |
如图 5, 随着稀有OTU过滤梯度的增大, 分子学方法与形态学方法所共同检获的砂壳虫属数下降, 仅分子手段检获的砂壳虫属数也同步下降。
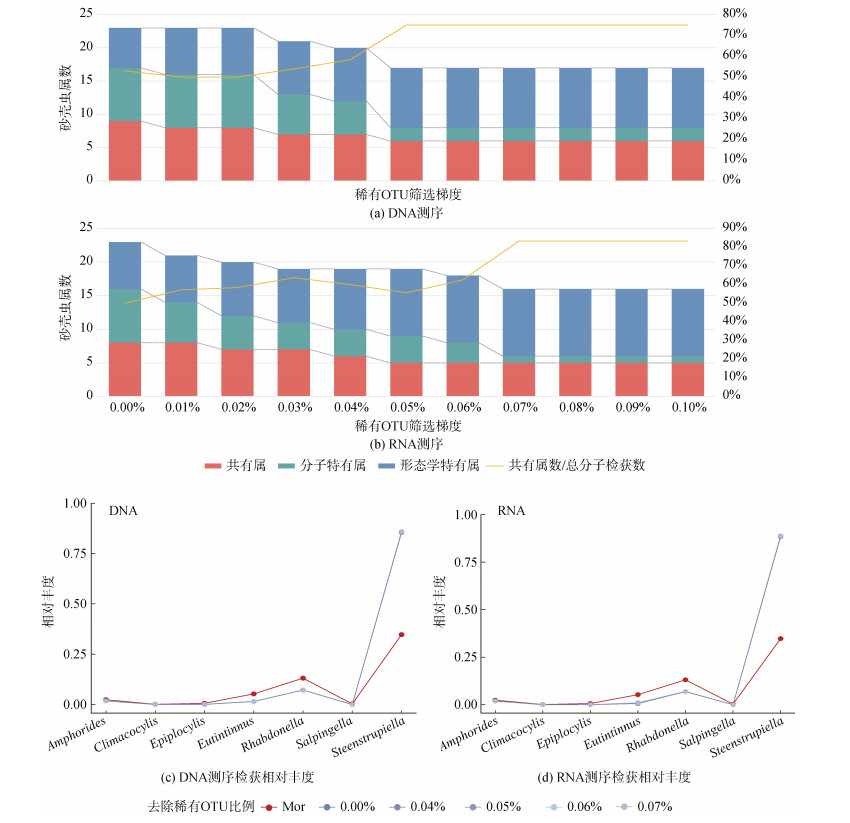 |
| 图 5 稀有OTU筛选结果比较 Fig. 5 Comparison of rare OTU screening results |
DNA测序结果中, 形态学方法和分子方法共同检获的属比分子总检获属数在0.04%~0.05%大幅上涨, 即在此过滤区间可以筛掉大量的分子特有属, 有效改善与形态学检获结果偏差。另外, 随着梯度的增大Kappa值也随之增大。在过滤梯度为0.04%时的Kappa值为−0.444 (P=0.035), 此时检获量为12属, 与形态学共有属为7属, 仅分子检获5属。梯度为0.05%时的Kappa值达到最大并持平(Kappa=−0.238, P=0.11), 此时检获量为8属, 共有属6属, 仅分子检获2属。
RNA测序结果中, 共同检获的属比分子总检获属数在0.06%~0.07%大幅上涨。在过滤梯度为0.06%时的Kappa值为−0.345 (P=0.034), 此时检获量为8属, 与形态学共有属为5属, 仅分子检获3属。梯度为0.07%时的Kappa值达到最大并持平(Kappa=−0.128, P=0.182), 此时检获量为6属, 共有属5属, 仅分子检获1属。即对于RNA测序检获结果, 稀有OTU过滤区间为0.06%~ 0.07%时, 更接近形态学检获的结果。
3 讨论环境DNA (eDNA)技术能够识别特定物种和群落特征[55], 并有效评估其多样性变化[56], 是一种高效的生物监测方法[57]。环境RNA (eRNA)技术同样作为一种生物多样性的检测技术[58]。与DNA不同, RNA在细胞死亡后会迅速分解, 因此在水体中保存的时间短[59-60]。Wood等[61]首次系统的比较了环境DNA和环境RNA在水体中的衰变情况, 发现eRNA在水体中的保存时间远不及eDNA。
前人比较了DNA和RNA测序手段检获的土壤中的真菌群落, 发现在丰富度、某些类群的丰度和整体群落组成方面存在差异[62-64]。尤其是沉积物中, eRNA检测的多样性和群落结构与eDNA差异很大[65]。然而, 我们的数据显示针对海水样品, eDNA和eRNA两种不同的分子手段检获OTUs数和物种组成基本一致, 没有明显差异。这与地下水层真菌的研究结果一致, DNA手段和RNA手段获检获的丰富度没有明显差异[66]。在土壤、沉积物中, DNA和RNA两种检测方法所导致的结果差异, 而水体中差异不显著, 是由于环境异质性导致。与水体不同, 在土壤及沉积物中, 死去的生物仍会保留存在于介质中, RNA方法主要检测活性生物, 而DNA方法检测的既有死亡生物也有活性生物。此前也有学者发现浮游类的纤毛虫(包括砂壳虫)具有包囊[67-68], 其沉降速率较快[69], 因此DNA测序所检获的纤毛虫沉积物既有活动虫体也有以包囊形式存在的纤毛虫, 故DNA和RNA检测结果有别。综上, 无论是海水还是淡水, DNA手段和RNA手段在水体中基本可以检获相似的生物多样性与群落结构。进一步比较发现, 分子手段与形态学手段检获的主要类群基本一致, 但两种方法检获的各物种相对丰度比例存在差异。这主要由于分子手段和形态学手段均存在局限性, 尤其是分子手段检获的群落组成及相对丰度受各个物种rDNA拷贝数及PCR扩增的影响, 不能完全反映出各个类群的相对丰度, 因此造成与形态学手段检获相对丰度存在差异。但通过比较分子手段与形态学手段共同检获的物种的相对丰度, 二者检获的趋势是一致的。这表明基于扩增子的高通量测序技术检获的砂壳纤毛虫相对丰度具有一定参考价值。因此, 鉴于RNA测序方法较为繁琐, 对于浮游水体中的砂壳虫类群, 用易操作的DNA测序方法, 就可以真实反映不同类群的变化。
针对高通量测序数据分析方法, 目前主流方法包括基于单核苷酸变异和序列相似度聚类的分析方法。近年来, 在基于单核苷酸变异的ASV与zOTU分析方法的提出后[22, 34, 70], 有学者针对单核苷酸变异和相似度聚类这两种分析方法进行了比较, 发现针对土壤生物多样性, 二者所得的α-多样性结果相异, 并且其检获的物种相对丰度也有所不同[35, 71]。部分学者认为单核苷酸变异方法在微型生物多样性分析中具有优势[31-33], 基于单核苷酸变异的方法所得到的结果较OTU方法精确度更高[72]。然而, 我们对比分子数据与形态数据, 发现相较于zOTU分析, 99%相似度下聚类的结果更加接近形态学数据。尽管两种不同的砂壳虫可能也会有超过99%相似的序列[11-12], 低于99%阈值可能会导致不同物种, 甚至不同属的混合[73]。但是基于单核苷酸变异分析砂壳纤毛虫时会导致物种数虚高。在95%序列相似度以下划分纤毛虫OTUs, 物种数量均呈现大幅下降的趋势[74]。因此, 对于砂壳纤毛虫, 建议序列相似度在99%聚类更为合理。
除上述选择合适的分类阶元划分标准, 去嵌合体的处理, 也可以提高物种注释率, 且不会对群落组成结构造成太大的影响, 去嵌合手段可以有效降低砂壳虫分子检获数与形态学存在的较大偏差, 更接近形态学数据, 这与先前的研究结论一致[75-76], 嵌合体的存在会使得分子多样性数据变得繁琐, 导致数据冗余化。此外, 纤毛虫稀有OTU比例较高, 达80%[46-47], 本实验砂壳虫稀有OTU比例约为48%。高比例的稀有物种会影响多样性与群落构成的分析, 有学者去掉所有稀有类群, 会丢失一部分真实存在的稀有物种。因此, 本研究设置一系列过滤梯度, 经筛选, 当过滤0.04%~0.05%的稀有OTU后, DNA测序检获的砂壳虫结果是最接近形态学结果的, RNA测序手段的最佳过滤区间为0.06%~0.07%, 在这两段区间内能很好的过滤分子手段检获的特有属。
基于序列相似度聚类获得OTUs, 常出现多个OTUs比对至同一物种的现象[42-44]。因此, 仍需进一步处理, 才可以更好地反映群落构成, 但目前仍无国际通用的方式, 我们结合前期研究经验和本文数据特点, 确定合并原则为“比对至同一物种且相似度在99%以上的分类单元合并”, 用以探讨聚类后处理方式对分子多样性研究结果的影响。本研究表明合并比对到同一物种的OTU对zOTU方法的数据有较好的作用, 可以减少大量噪音, 减缓其数据膨胀现象。然而, 在本实验中, 合并采取的只是简单原则, 这种粗糙的合并可能会导致OTU的不稳定, 进而影响下游多样性计算等进一步分析[77]。合并OTU是否可以改善分子手段检获的结果有待考究, 建议选择较完善的参考数据库, 更精确的合并原则或许能进一步优化分子手段检获的数据。
4 总结本文通过将分子检获数据与形态学观察数据比较, 表明DNA测序和RNA测序两种分子手段与形态学方法检获结果趋势基本一致。分子手段能够检获到形态学手段所检获的主要物种, 并且能在一定程度上可以反映出自然界中不同类群的相对丰度。研究阐明了原生生物多样性的评估结果因分析方法而异, 通过优化下游数据分析流程, 例如数据去嵌合体、选择99%聚类相似度阈值、过滤合适的稀有OTU等, 能够使得分子手段检获结果趋近于传统方法学检获结果。针对砂壳纤毛虫下游的数据分析, 我们的研究提供了可供参考的数据分析参数和阈值信息, 对微生物监测的分子方法选择提供了参考, 以改善数据产出与成本的平衡。对未来微生物多样性研究选择最佳流程具有重要的指导意义, 并为后续多样性研究分析提供理论依据。
致谢: 中国科学院海洋研究所“科学”号科学考察船及船员协助样品采集, 谨致谢忱。
| [1] |
SINGER D, SEPPEY C V W, LENTENDU G, et al. Protist taxonomic and functional diversity in soil, freshwater and marine ecosystems[J]. Environment International, 2021, 146: 106262. DOI:10.1016/j.envint.2020.106262 |
| [2] |
LYNN D H. The ciliated protozoa: Characterization, classification, and guide to the literature: Third edition[M]. New York: Springer, 2008: 1-605.
|
| [3] |
FALKOWSKI P G, BARBER R T, SMETACEK V. Biogeochemical controls and feedbacks on ocean primary production[J]. Science, 1998, 281(5374): 200-206. DOI:10.1126/science.281.5374.200 |
| [4] |
PERNTHALER J. Predation on prokaryotes in the water column and its ecological implications[J]. Nature Reviews: Microbiology, 2005, 3(7): 537-546. DOI:10.1038/nrmicro1180 |
| [5] |
FENCHEL T. The microbial loop – 25 years later[J]. Journal of Experimental Marine Biology and Ecology, 2008, 366(1): 99-103. |
| [6] |
WORDEN A Z, FOLLOWS M J, GIOVANNONI S J, et al. Rethinking the marine carbon cycle: factoring in the multifarious lifestyles of microbes[J]. Science, 2015, 347(6223): 1257594. DOI:10.1126/science.1257594 |
| [7] |
PIERCE R, TURNER J. Global biogeography of marine tintinnids[J]. Marine Ecology Progress Series, 1993, 94(1): 11-26. |
| [8] |
MODIGH M, ZOOLOGICA S, DOHRN A, et al. Seasonal variations of photosynthetic ciliates at a Mediterranean coastal site[J]. Aquatic Microbial Ecology, 2001, 23: 163-175. DOI:10.3354/ame023163 |
| [9] |
FILEMAN E S, LEAKEY R J G. Microzooplankton dynamics during the development of the spring bloom in the north-east Atlantic[J]. Journal of the Marine Biological Association of the United Kingdom, 2005, 85(4): 741-753. DOI:10.1017/S0025315405011653 |
| [10] |
GINER C R, FORN I, ROMAC S, et al. Environmental sequencing provides reasonable estimates of the relative abundance of specific picoeukaryotes[J]. Applied and Environmental Microbiology, 2016, 82(15): 4757-4766. DOI:10.1128/AEM.00560-16 |
| [11] |
BACHY C, DOLAN J R, LóPEZ-GARCíA P, et al. Accuracy of protist diversity assessments: morphology compared with cloning and direct pyrosequencing of 18S rRNA genes and ITS regions using the conspicuous tintinnid ciliates as a case study[J]. The ISME Journal, 2013, 7(2): 244-255. DOI:10.1038/ismej.2012.106 |
| [12] |
SANTOFERRARA L F, MCMANUS G B, ALDER V A. Utility of genetic markers and morphology for species discrimination within the order Tintinnida (Ciliophora, Spirotrichea)[J]. Protist, 2013, 164(1): 24-36. DOI:10.1016/j.protis.2011.12.002 |
| [13] |
DOHERTY M, TAMURA M, VRIEZEN J A C, et al. Diversity of oligotrichia and choreotrichia ciliates in coastal marine sediments and in overlying plankton[J]. Applied and Environmental Microbiology, 2010, 76(12): 3924-3935. DOI:10.1128/AEM.01604-09 |
| [14] |
MASSANA R, GOBET A, AUDIC S, et al. Marine protist diversity in European coastal waters and sediments as revealed by high-throughput sequencing[J]. Environmental Microbiology, 2015, 17(10): 4035-4049. DOI:10.1111/1462-2920.12955 |
| [15] |
LI G, SU L, ZHANG Q, et al. Molecular diversity and biogeography of benthic ciliates in the Bohai Sea and Yellow Sea[J]. Acta Oceanologica Sinica, 2019, 38(2): 78-86. DOI:10.1007/s13131-018-1236-y |
| [16] |
ZHAO F, FILKER S, WANG C, et al. Bathymetric gradient shapes the community composition rather than the species richness of deep-sea benthic ciliates[J]. The Science of the Total Environment, 2021, 755(Pt 2): 142623. |
| [17] |
EDGAR R C. Search and clustering orders of magnitude faster than BLAST[J]. Bioinformatics, 2010, 26(19): 2460-2461. DOI:10.1093/bioinformatics/btq461 |
| [18] |
ROGNES T, FLOURI T, NICHOLS B, et al. VSEARCH: a versatile open source tool for metagenomics[J]. PeerJ, 2016, 4: e2584. DOI:10.7717/peerj.2584 |
| [19] |
CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J]. Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303 |
| [20] |
EDGAR R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10): 996-998. DOI:10.1038/nmeth.2604 |
| [21] |
EDGAR R C, BATZOGLOU S. Multiple sequence alignment[J]. Current Opinion in Structural Biology, 2006, 16(3): 368-373. DOI:10.1016/j.sbi.2006.04.004 |
| [22] |
CALLAHAN B J, MCMURDIE P J, ROSEN M J, et al. DADA2: High-resolution sample inference from Illumina amplicon data[J]. Nature Methods, 2016, 13(7): 581-583. DOI:10.1038/nmeth.3869 |
| [23] |
FLYNN J M, BROWN E A, CHAIN F J J, et al. Toward accurate molecular identification of species in complex environmental samples: testing the performance of sequence filtering and clustering methods[J]. Ecology and Evolution, 2015, 5(11): 2252-2266. DOI:10.1002/ece3.1497 |
| [24] |
MAJANEVA M, HYYTIäINEN K, VARVIO S L, et al. Bioinformatic amplicon read processing strategies strongly affect eukaryotic diversity and the taxonomic composition of communities[J]. PLoS One, 2015, 10(6): e0130035. DOI:10.1371/journal.pone.0130035 |
| [25] |
HARDGE K, NEUHAUS S, KILIAS E S, et al. Impact of sequence processing and taxonomic classification approaches on eukaryotic community structure from environmental samples with emphasis on diatoms[J]. Molecular Ecology Resources, 2018, 18(2): 204-216. DOI:10.1111/1755-0998.12726 |
| [26] |
XU J, HAN J, SU H, et al. Diversity patterns of protists are highly affected by methods disentangling biological variants: a case study in Oligotrich (s. l.) Ciliates[J]. Microorganisms, 2022, 10(5): 913. DOI:10.3390/microorganisms10050913 |
| [27] |
RIDEOUT J R, HE Y, NAVAS-MOLINA J A, et al. Subsampled open-reference clustering creates consistent, compreheoutve OTU definitions and scales to billions of sequences[J]. PeerJ, 2014, 2: e545. DOI:10.7717/peerj.545 |
| [28] |
SCHLOSS P D, HANDELSMAN J. Introducing DOTUR, a computer program for defining operational taxonomic units and estimating species richness[J]. Applied and Environmental Microbiology, 2005, 71(3): 1501-1506. DOI:10.1128/AEM.71.3.1501-1506.2005 |
| [29] |
SCHLOSS P D, WESTCOTT S L, RYABIN T, et al. Introducing mothur: open-source, platform-independent, community-supported software for describing and comparing microbial communities[J]. Applied and Environmental Microbiology, 2009, 75(23): 7537-7541. DOI:10.1128/AEM.01541-09 |
| [30] |
CALLAHAN B J, MCMURDIE P J, HOLMES S P. Exact sequence variants should replace operational taxonomic units in marker-gene data analysis[J]. The ISME Journal, 2017, 11(12): 2639-2643. DOI:10.1038/ismej.2017.119 |
| [31] |
BROWN E A, CHAIN F J J, CREASE T J, et al. Divergence thresholds and divergent biodiversity estimates: can metabarcoding reliably describe zooplankton communities?[J]. Ecology and Evolution, 2015, 5(11): 2234-2251. DOI:10.1002/ece3.1485 |
| [32] |
BEHNKE A, ENGEL M, CHRISTEN R, et al. Depicting more accurate pictures of protistan community complexity using pyrosequencing of hypervariable SSU rRNA gene regions[J]. Environmental Microbiology, 2011, 13(2): 340-349. DOI:10.1111/j.1462-2920.2010.02332.x |
| [33] |
FORSTER D, LENTENDU G, FILKER S, et al. Improving eDNA-based protist diversity assessments using networks of amplicon sequence variants[J]. Environmental Microbiology, 2019, 21(11): 4109-4124. DOI:10.1111/1462-2920.14764 |
| [34] |
EDGAR R C. Updating the 97% identity threshold for 16S ribosomal RNA OTUs[J]. Bioinformatics, 2018, 34(14): 2371-2375. DOI:10.1093/bioinformatics/bty113 |
| [35] |
NEARING J T, DOUGLAS G M, COMEAU A M, et al. Denoising the denoisers: an independent evaluation of microbiome sequence error-correction approaches[J]. PeerJ, 2018, 6: e5364. DOI:10.7717/peerj.5364 |
| [36] |
ZHAO F, FILKER S, XU K, et al. Effects of intragenomic polymorphism in the SSU rRNA gene on estimating marine microeukaryotic diversity: A test for ciliates using single-cell high-throughput DNA sequencing[J]. Limnology and Oceanography: Methods, 2019, 17(10): 533-543. DOI:10.1002/lom3.10330 |
| [37] |
GONG J, DONG J, LIU X, et al. Extremely high copy numbers and polymorphisms of the rDNA operon estimated from single cell analysis of Oligotrich and Peritrich Ciliates[J]. Protist, 2013, 164(3): 369-379. DOI:10.1016/j.protis.2012.11.006 |
| [38] |
WANG C, ZHANG T, WANG Y, et al. Disentangling sources of variation in SSU rDNA sequences from single cell analyses of ciliates: impact of copy number variation and experimental error[J]. Proceedings of the Royal Society B: Biological Sciences, 2017, 284(1859): 20170425. DOI:10.1098/rspb.2017.0425 |
| [39] |
EGGE E, BITTNER L, ANDERSEN T, et al. 454 pyrosequencing to describe microbial eukaryotic community composition, diversity and relative abundance: a test for marine haptophytes[J]. PLoS One, 2013, 8(9): e74371. DOI:10.1371/journal.pone.0074371 |
| [40] |
LIU Y X, QIN Y, CHEN T, et al. A practical guide to amplicon and metagenomic analysis of microbiome data[J]. Protein & Cell, 2021, 12(5): 315-330. |
| [41] |
GASOL J M, KIRCHMAN D L. Microbial ecology of the oceans[M]. Hoboken, NJ, USA: John Wiley & Sons, 2018.
|
| [42] |
SCHLOSS P D. Amplicon sequence variants artificially split bacterial genomes into separate clusters[J]. MSphere, 2021, 6(4): e0019121. DOI:10.1128/mSphere.00191-21 |
| [43] |
OBIOL A, GINER C R, SáNCHEZ P, et al. A metagenomic assessment of microbial eukaryotic diversity in the global ocean[J]. Molecular Ecology Resources, 2020, 20(3): 718-731. DOI:10.1111/1755-0998.13147 |
| [44] |
SALAZAR G, PAOLI L, ALBERTI A, et al. Gene expression changes and community turnover differentially shape the global ocean metatranscriptome[J]. Cell, 2019, 179(5): 1068-1083. DOI:10.1016/j.cell.2019.10.014 |
| [45] |
SéBASTIEN T, CHRISTOPHE D, CORENTIN J, et al. ReClustOR: a re-clustering tool using an open-reference method that improves OTU definition[J]. Methods in Ecology and Evolution, 2020, 11: 168-180. DOI:10.1111/2041-210X.13316 |
| [46] |
ZHAO F, XU K. Molecular diversity and distribution pattern of ciliates in sediments from deep-sea hydrothermal vents in the Okinawa Trough and adjacent sea areas[J]. Deep Sea Research Part I: Oceanographic Research Papers, 2016, 116: 22-32. DOI:10.1016/j.dsr.2016.07.007 |
| [47] |
ZHAO F, FILKER S, STOECK T, et al. Ciliate diversity and distribution patterns in the sediments of a seamount and adjacent abyssal plains in the tropical Western Pacific Ocean[J]. BMC Microbiology, 2017, 17(1): 192. DOI:10.1186/s12866-017-1103-6 |
| [48] |
STOCK A, EDGCOMB V, ORSI W, et al. Evidence for isolated evolution of deep-sea ciliate communities through geological separation and environmental selection[J]. BMC Microbiology, 2013, 13: 150. DOI:10.1186/1471-2180-13-150 |
| [49] |
LARA E, BERNEY C, HARMS H, et al. Cultivation-independent analysis reveals a shift in ciliate 18S rRNA gene diversity in a polycyclic aromatic hydrocarbon-polluted soil[J]. FEMS Microbiology Ecology, 2007, 62(3): 365-373. DOI:10.1111/j.1574-6941.2007.00387.x |
| [50] |
STOECK T, BASS D, NEBEL M, et al. Multiple marker parallel tag environmental DNA sequencing reveals a highly complex eukaryotic community in marine anoxic water[J]. Molecular Ecology, 2010, 19(Suppl 1): 21-31. |
| [51] |
GUILLOU L, BACHAR D, AUDIC S, et al. The Protist Ribosomal Reference database (PR2): a catalog of unicellular eukaryote Small Sub-Unit rRNA sequences with curated taxonomy[J]. Nucleic Acids Research, 2012, 41(D1): D597-D604. DOI:10.1093/nar/gks1160 |
| [52] |
EDGAR R C, FLYVBJERG H. Error filtering, pair assembly and error correction for next-generation sequencing reads[J]. Bioinformatics (Oxford, England), 2015, 31(21): 3476-3482. |
| [53] |
张武昌, 丰美萍, 于莹, 等. 砂壳纤毛虫图谱[M]. 北京: 科学出版社, 2012: 1-499. ZHANG Wuchang, FENG Meiping, YU Ying, et al. An illustrated guide to contemporary tintinnids in the world[M]. Beijing: Science Press, 2012: 1-499. |
| [54] |
WANG C, LI H, DONG Y, et al. Planktonic ciliate trait structure variation over Yap, Mariana, and Caroline seamounts in the tropical western Pacific Ocean[J]. Journal of Oceanology and Limnology, 2021, 39(5): 1705-1717. DOI:10.1007/s00343-021-0476-4 |
| [55] |
FUKAYA K, MURAKAMI H, YOON S, et al. Estimating fish population abundance by integrating quantitative data on environmental DNA and hydrodynamic modelling[J]. Molecular Ecology, 2021, 30(13): 3057-3067. DOI:10.1111/mec.15530 |
| [56] |
CARRARO L, HARTIKAINEN H, JOKELA J, et al. Estimating species distribution and abundance in river networks using environmental DNA[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(46): 11724-11729. |
| [57] |
MIYA M. Environmental DNA Metabarcoding: A novel method for biodiversity monitoring of marine fish communities[J]. Annual Review of Marine Science, 2022, 14: 161-185. DOI:10.1146/annurev-marine-041421-082251 |
| [58] |
VEILLEUX H D, MISUTKA M D, GLOVER C N. Environmental DNA and environmental RNA: Current and prospective applications for biological monitoring[J]. The Science of the Total Environment, 2021, 782: 146891. DOI:10.1016/j.scitotenv.2021.146891 |
| [59] |
EIGNER J, BOEDTKER H, MICHAELS G. The thermal degradation of nucleic acids[J]. Biochimica et Biophysica Acta, 1961, 51(1): 165-168. DOI:10.1016/0006-3002(61)91028-9 |
| [60] |
MENGONI A, TATTI E, DECOROSI F, et al. Comparison of 16S rRNA and 16S rDNA T-RFLP approaches to study bacterial communities in soil microcosms treated with chromate as perturbing agent[J]. Microbial Ecology, 2005, 50(3): 375-384. DOI:10.1007/s00248-004-0222-4 |
| [61] |
WOOD S A, BIESSY L, LATCHFORD J L, et al. Release and degradation of environmental DNA and RNA in a marine system[J]. The Science of the Total Environment, 2020, 704: 135314. DOI:10.1016/j.scitotenv.2019.135314 |
| [62] |
BALDRIAN P, KOLAŘíK M, STURSOVá M, et al. Active and total microbial communities in forest soil are largely different and highly stratified during decomposition[J]. The ISME Journal, 2012, 6(2): 248-258. DOI:10.1038/ismej.2011.95 |
| [63] |
COX F, NEWSHAM K K, ROBINSON C H. Endemic and cosmopolitan fungal taxa exhibit differential abundances in total and active communities of Antarctic soils[J]. Environmental Microbiology, 2019, 21(5): 1586-1596. DOI:10.1111/1462-2920.14533 |
| [64] |
ROMANOWICZ K J, FREEDMAN Z B, UPCHURCH R A, et al. Active microorganisms in forest soils differ from the total community yet are shaped by the same environmental factors: the influence of pH and soil moisture[J]. FEMS Microbiology Ecology, 2016, 92(10): fiw149. DOI:10.1093/femsec/fiw149 |
| [65] |
KITAHASHI T, SUGIME S, INOMATA K, et al. Meiofaunal diversity at a seamount in the pacific ocean: A comprehensive study using environmental DNA and RNA[J]. Deep Sea Research Part Ⅰ: Oceanographic Research Papers, 2020, 160: 103253. DOI:10.1016/j.dsr.2020.103253 |
| [66] |
NAWAZ A, PURAHONG W, HERRMANN M, et al. DNA- and RNA- derived fungal communities in subsurface aquifers only partly overlap but react similarly to environmental factors[J]. Microorganisms, 2019, 7(9): 341. DOI:10.3390/microorganisms7090341 |
| [67] |
DOHERTY M, TAMURA M, VRIEZEN JAN A C, et al. Diversity of Oligotrichia and Choreotrichia ciliates in coastal marine sediments and in overlying plankton[J]. Applied and Environmental Microbiology, 2010, 76(12): 3924-3935. DOI:10.1128/AEM.01604-09 |
| [68] |
TORTI A, LEVER M A, JøRGENSEN B B. Origin, dynamics, and implications of extracellular DNA pools in marine sediments[J]. Marine Genomics, 2015, 24: 185-196. DOI:10.1016/j.margen.2015.08.007 |
| [69] |
KIM Y-O, HA S, TANIGUCHI A. Morphology and in situ sedimentation of the cysts of a planktonic oligotrich ciliate, Strombidium capitatum[J]. Aquatic Microbial Ecology, 2008, 53: 173-179. DOI:10.3354/ame01241 |
| [70] |
AMIR A, MCDONALD D, NAVAS-MOLINA J A, et al. Deblur rapidly resolves single-nucleotide community sequence patterns[J]. mSystems, 2017, 2(2): e00191-16. |
| [71] |
NOGUERALES V, MERAMVELIOTAKIS E, CASTRO-INSUA A, et al. Community metabarcoding reveals the relative role of environmental filtering and spatial processes in metacommunity dynamics of soil microarthropods across a mosaic of montane forests[J]. Molecular Ecology, 2023, 32: 6110-6128. DOI:10.1111/mec.16275 |
| [72] |
CARUSO V, SONG X, ASQUITH M, et al. Performance of microbiome sequence inference methods in environments with varying biomass[J]. mSystems, 2019, 4(1): e00163-18. |
| [73] |
BACHY C, GÓMEZ F, LÓPEZ-GARCÍA P, et al. Molecular phylogeny of tintinnid ciliates (Tintinnida, Ciliophora)[J]. Protist, 2012, 163(6): 873-887. DOI:10.1016/j.protis.2012.01.001 |
| [74] |
DOHERTY M, COSTAS B, MCMANUS G, et al. Culture-independent assessment of planktonic ciliates in coastal northwest Atlantic waters[J]. Aquatic Microbial Ecology, 2007, 48: 141-154. DOI:10.3354/ame048141 |
| [75] |
THORNHILL D J, LAJEUNESSE T C, SANTOS S R. Measuring rDNA diversity in eukaryotic microbial systems: how intragenomic variation, pseudogenes, and PCR artifacts confound biodiversity estimates[J]. Molecular Ecology, 2007, 16(24): 5326-5340. DOI:10.1111/j.1365-294X.2007.03576.x |
| [76] |
FONSECA V G, NICHOLS B, LALLIAS D, et al. Sample richness and genetic diversity as drivers of chimera formation in nSSU metagenetic analyses[J]. Nucleic Acids Research, 2012, 40(9): e66. DOI:10.1093/nar/gks002 |
| [77] |
HE Y, CAPORASO J G, JIANG X T, et al. Stability of operational taxonomic units: an important but neglected property for analyzing microbial diversity[J]. Microbiome, 2015, 3: 20. DOI:10.1186/s40168-015-0081-x |
 2024, Vol. 48
2024, Vol. 48


