
文章信息
- 王馥容, 张雨, 冯诗语, 王宇航, 郑伊宁, 张家豪, 王连顺, 卢亚楠, 王丽, 谷晶, 丛玉婷, 杨国军, 王华. 2024.
- WANG Furong, ZHANG Yu, FENG Shiyu, WANG Yuhang, ZHENG Yining, ZHANG Jiahao, WANG Lianshun, LU Yanan, WANG Li, GU Jing, CONG Yuting, YANG Guojun, WANG Hua. 2024.
- 新型生物脱氮技术及其相关酶系的研究进展
- Research progress on novel biological nitrogen removal techniques and related enzyme systems
- 海洋科学, 48(6): 106-117
- Marine Sciences, 48(6): 106-117.
- http://dx.doi.org/10.11759/hykx20230812001
-
文章历史
- 收稿日期:2023-08-12
- 修回日期:2023-10-12
2. 大连海洋大学 设施渔业教育部重点实验室, 辽宁 大连 116023;
3. 辽宁省贝类良种繁育工程技术研究中心, 辽宁 大连 116023
2. Key Laboratory of Environment Controlled Aquaculture, Dalian Ocean University, Ministry of Education, Dalian 116023, China;
3. Engineering Research Center of Shellfish Culture and Breeding in Liaoning Province, Dalian 116023, China
氮素在水体中主要以氨态氮、亚硝态氮和硝态氮等形式存在, 垃圾渗滤液和工业都会产生大量的氨氮废水。随着全球人口增长、人民生活水平的提高和工农业的快速发展, 城市污水和工业废水等水体中氮素浓度急剧升高, 大大增加了水中氮素的营养负荷, 导致水体富营养化, 严重危害水域生态环境, 含氮污水的基础理论研究及氮污染废水处理已经成为全球广泛关注的焦点和重点研究对象[1]。
水体中过高的氮素浓度极易造成水体的富营养化, 进而导致水体生态系统中食物链的结构发生变化, 使食物网从以底栖微藻和大型植物生产为主转为以浮游植物为主的系统[2], 同时降低水体的透光度, 显著抑制底栖浮游生物和深海水生植物的生长和繁殖, 造成水体污染和破坏水域生态环境。同时, 氮素营养负荷会产生许多种科学问题, 包括产生缺氧“死亡区”, 严重影响鱼类和贝类的产量, 例如, 密西西比河排放的营养盐和有机物在墨西哥湾产生超过15 000 km2的缺氧死区[3]。缓慢流动河流中的富营养化还可能导致有毒藻类大量暴发性增殖, 例如在澳大利亚的默里-达令河系会发生部分河段的富营养化导致藻类增殖, 造成牲畜的大量死亡, 并会对人类健康造成了严重影响[4]。例如, 当NO3–高于10 mg/L时可导致婴儿高铁血红蛋白血症[5], 同时其也是人体内分泌干扰物[6], 危害人体的健康, 引起人体味觉和嗅觉等问题; 影响饮用水和水生食品供应的安全, 刺激温室气体的排放, 以及使周边水域文化和社会价值发生生态退化[7]。基于氮素对环境和人类健康造成的严重危害, 以及水体中氮素污染的急剧增加, 污水处理的成本也大大增加。因此, 如何经济和高效地控制水体中氮元素污染已成为全球关注的重点研究方向。
目前, 去除水体中氮素的方法主要是生物脱氮法, 生物脱氮法分为传统生物脱氮和新型生物脱氮两部分。传统生物脱氮法主要包括氨化作用、硝化作用和反硝化作用3个部分, 发生的反应方程式如下[8]:
1) 氨化作用:
2) 硝化作用:
3) 反硝化作用:
虽然传统生物脱氮的工艺更加成熟, 脱氮效果更好, 但存在工艺流程长、占地大、常需外加碳源、能耗大和成本高等缺点。因此激发了新型生物脱氮技术的发展。
综上, 本文从新型生物脱氮技术的生物脱氮机制以及相关催化酶的酶学性质表征等方面进行系统的介绍, 并对相关领域的研究进展进行讨论, 对新型生物脱氮技术和相关催化酶的研究进行展望, 旨在为提高新型生物脱氮技术在水体氮素处理中的效率及相关酶的研究提供理论支撑。
1 新型生物脱氮技术由于化学物理法和传统生物脱氮技术存在着许多问题和缺点, 因此研究者一直在探索研究脱氮效果更好的新型生物脱氮技术。近年来, 随着生物技术的发展, 人们提高了对传统生物脱氮技术的理解和认识, 对生物脱氮的理论基础得到进一步的认识和提升, 提出比传统生物脱氮技术优点更多的新型生物脱氮技术。如短程硝化反硝化、厌氧氨氧化和异养硝化好氧反硝化等新型生物脱氮技术。
1.1 短程硝化反硝化去除水体特别是污水处理厂(WWTPs)中含游离氨和NH4+的氨氮废水主要通过生物硝化和反硝化来实现, 因为相关生物的生长速度较慢, 所以生物硝化是氮去除过程中的主要限制步骤。一个完整的生物硝化过程包括氨氧化成亚硝酸盐到氧化成硝酸盐, 分别由氨氧化细菌(AOB)和亚硝酸盐氧化细菌(NOB)进行。而反硝化过程则是在缺氧条件下, 将硝酸盐还原成亚硝酸盐再生成氮气的过程[9]。从硝化和反硝化的路径可以看出, 硝化过程中将亚硝酸盐氧化成硝酸盐和反硝化过程中硝酸盐还原成亚硝酸盐的过程正好相反, 如果将其从反应路径中去除, 则为短程硝化反硝化的过程, 即将氨氧化成亚硝酸盐再生成氮气的过程(图 1)[10]。
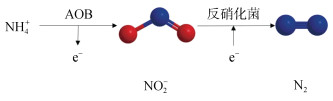 |
| 图 1 短程硝化反硝化电子传递示意图 Fig. 1 Schematic of electron transport during short-range nitrification and denitrification |
其中反应所需的AOB可以在甲胺上生长[11], 其主要依赖氨的有氧氧化, 所以它们必须在其生境中竞争氨和O2。在低O2环境中AOB可能与厌氧氨氧化细菌(anaerobic ammonia oxidizing bacteria, AnAOB)竞争, 其是一种不使用氧气就能氧化氨的细菌[12]。尽管好氧AOB和AnAOB都消耗氨, 但它们的分解代谢产物完全不同: AOB通过硝化作用消耗氧气并产生亚硝酸盐, 并与NOB竞争利用亚硝酸盐, 而AnAOB不依赖于O2, 消耗NOx并生成氮气作为完全反硝化[13]。
在分类学上, AOB来源于变形菌纲两个不同属的单系类群。多数人工培养的AOB属于β-变形菌的亚硝化单胞菌科(Nitrosomonadaceae), 而亚硝基球菌(Nitrosococcus)的海洋AOB是γ-变形菌的着色菌科(Chromatiaceae)。只包括AOB的亚硝化单胞菌科(Nitrosomonadaceae)分为亚硝化单胞菌(Nitrosomonas sp.)、亚硝化螺菌(Nitrosospira sp.)和亚硝化弧菌属(Nitrosovibrio sp.)3个属[14]。
相比生物硝化和反硝化工艺而言, 短程硝化反硝化工艺因为略去了亚硝酸盐向硝酸盐的转化步骤, 短程硝化反硝化节省了25%的曝气能耗、减少40%的碳源投加量以及降低污泥产量等优点[15]; 因为省去了硝酸盐还原为亚硝酸盐步骤, 根据反应计量学: 1 mol硝酸盐和1 mol亚硝酸盐还原为氮气, 分别需要2.86 mol和1.71 mol的化学需氧量[16], 可节约反硝化碳源40%左右[17]。综上, 因为反应路径的缩短, 反应成本也会相应降低。
然而想要将硝化过程完全控制在亚硝酸盐阶段是十分困难的, 常见的影响亚硝酸盐积累的条件为: 温度、pH、溶解氧等。因此应在短程硝化过程中对上述条件进行控制。常见的短程硝化主要有3种工艺[18](表 1)。
| 工艺类型 | 优点 | 缺点 |
| 基于序批式活性污泥法反应器(sequencing batch reactor activated sludge process, SBR) | 操作灵活方便、可由软件自动化控制 | 不利于污水处理厂的直接改造, 重新建设设备花费较大, 进水不连续, 处理周期长 |
| 基于完全混合反应器(continuous stirred tank reactor, CSTR) | 结构简单、操作方便、改造容易、投资费用少、有利于工业实际应用 | 易产生污泥膨胀[19], 不利于亚硝酸盐氮积累[20] |
| 基于膜生物反应器(membrane bio-reactor, MBR) | 容积负荷高、占地面积小、剩余污泥量低、截留高效、泥龄可以和水力停留时间分开、亚硝酸盐氮累计率比CSTR工艺高 | 单纯的MBR短程硝化对氮素的脱除效果不够好, 常需要和其他工艺进行组合 |
厌氧氨氧化(anammox)是一种低成本高效的废水处理微生物工艺其具有广阔的应用前景, 受到了广泛关注。与传统的硝化相比, 厌氧氨氧化是一种更先进的技术, 因为它减少了溶解氧(DO)和外源碳源的需求[21]。厌氧氨氧化过程以铵(NH4+)为电子供体, 将亚硝酸盐(NO2−)还原为氮气(N2)(图 2)[22]。
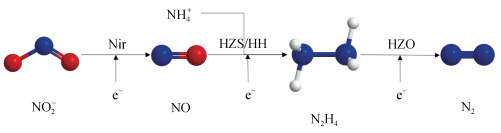 |
| 图 2 厌氧氨氧化电子传递示意图 Fig. 2 Schematic of electron transport in anammox |
与传统的硝化-反硝化工艺相比, 可以节省曝气能量(0.9 kW·h/kg N, 与3.5 kW·h/kg N相比)和碳供应(0 kg CH3OH/kg N, 与3 kg CH3OH/kg N相比)在内的运行成本[23-24]。可减少90%的过剩污泥的产生, 大幅度减少CO2和N2O在内的温室气体的排放。此外, 与较低的常规脱氮能力(小于0.5 kg/(m3·d)相比, 厌氧氨氧化表现出极高的脱氮潜力和能力[24]。Tang等人[25]报道在实验室规模的上升流式厌氧污泥床反应器中, 氮去除率可以达到74.6~76.7 kg/(m3·d)。此外, 厌氧氨氧化工艺不仅应用于垃圾渗滤液、畜禽废水和抗生素废水等各种富氨废水的处理, 而且还应用于城市废水的规模化处理, 并取得了良好的效果和经济效益[26]。
本文列举了一些厌氧氨氧化工程应用以供参考, 见表 2。
| 应用方向 | 工艺类型 | 总氮去除率/% | 总氮去除负荷/kg·(m 3 ·d)−1 |
| 垃圾渗滤液 | 短程硝化反硝化-厌氧氨氧化-硫自养反硝化工艺[27] | 99.5 | 0.85 |
| 前置反硝化与短程硝化-厌氧氨氧化串联工艺[28] | 98.6 | — | |
| 短程硝化-厌氧氨氧化-土壤渗滤串联工艺[29] | 89 | — | |
| 基于移动床生物膜反应器(MBBR)对亚硝化-厌氧氨氧化-反硝化工艺[30] | 88 | 0.9 | |
| 污泥消化液 | 接种短程硝化污泥和厌氧氨氧化生物膜填料[31] | 94.4 | 0.27 |
| 生活污水 | 缺氧MBBR反应器[32] | — | — |
| 一体化ABR-短程硝化-厌氧氨氧化工艺[33] | — | — | |
| 其他废水 | CANON工艺[34] | 98 | — |
| 向厌氧氨氧化工艺中投加其他菌种[35] | — | 9.4 | |
| 短程硝化-厌氧氨氧化-全程硝化工艺[36] | — | — |
综上所述, 厌氧氨氧化技术能够有效去除高氨氮废水中的含氮污染物及其他污染物。
1.2.1 亚硝酸盐还原酶(nitrite reductase, Nir)亚硝酸盐还原酶(nitrite reductase, Nir)可以使NO2–得到一个电子生成不能被其他大部分生物利用的气态产物NO, 是生物脱氮途径中的中心酶[37]。Nir分为两种主要形式: 一种是含有细胞色素c和d1的cd1-NiR、一种是在每个活性位点上含有两个铜中心的Cu-NiR。其中cd1-NiR是一种双功能酶, 由nirS基因编码, 能将亚硝酸盐还原为NO和N2O的混合物, 能使O2得到4个电子生成H2O, 并催化亚硝酸得到1个电子转变成NO[38]。Cu-NiR即可溶性含铜亚硝酸盐还原酶, 是一种同源三聚体, 由nirK基因编码, 位于外周胞质, 催化产物也是NO和N2O的混合物。尽管这两种亚硝酸盐还原酶的结构不同, 但这两种形式的亚硝酸盐还原酶在类型、功能和生理上是等效的[39]。其中nirS基因分布更广, 而nirK基因虽然比较少, 但可以在更广泛的生理基团中被发现[40]。
为确定在实验室培养的Nir的类型, 可以使用以下4种方法: 一是用二乙基二硫代氨基甲酸酯鉴定含nirK的反硝化菌[41]; 二是用Nir抗血清(dNirS和dNirK), 这种方法可以实现在菌株水平上非常具体的检测[42]; 三是使用nirK或nirS基因探针, 主要针对所研究的菌株[43]; 四是用一对引物靶向nirS亚硝酸盐还原酶基因的PCR方法, 这种方法比杂交实验具有更高的特异性[44]。
Nir酶活可以在10 mmol/L磷酸盐缓冲溶液(pH=7.4)和0.200 mmol/L NADH的条件下根据反应前后亚硝酸钠的减少量进行测定[45], 其蛋白质浓度可采用考马斯亮蓝法测定[46]。刘玮等人的研究表明植物乳杆菌(Lactobacillus plantarum)中的Nir具有较强降解亚硝酸盐能力, 最适温度为50 ℃、最适pH值为6.5、Vmax 为5.84 mg/ (L·min)、Km值为7.79 mmol/L, 当金属离子的添加浓度在1 mmol/L~10 mmol/L时, Fe2+等金属离子对Nir酶活力有显著作用(P < 0.05), 但乙二胺四乙酸(ethylenediaminetetraacetic acid, EDTA)对酶活无显著影响[47]。嗜盐古菌(Halophiles)获得的亚硝酸盐还原酶NirKSrb为蓝色铜型亚硝酸盐还原酶, 在广阔的盐度温度范围内具有较高的活性, 同时对一些有机试剂具有良好的耐受性, 其在富含亚硝酸盐的食品加工生产中具有广泛的应用潜力[48]。
1.2.2 肼合成酶(hydrazine synthetase, HZS)厌氧氨氧化(anammox)是一种在1977年被Broda通过热力学反应自由能预测的将NO氧化成肼的生物化学反应过程[49], 在全球氮循环特别是N-N键的形成中起着非常重要的作用, 主要用于富氨废水的处理。其中肼合成酶(hydrazine synthetase, HZS)等多种含血红素的酶共同参与厌氧氨氧化过程[50], 血红素蛋白种类繁多, 广泛用作生物催化剂、传感器和分子转运体[51]。
HZS由hzsABC基因簇编码, 是催化一氧化氮(NO)和氨(NH3)生成肼(N2H4)过程中唯一且不可替代的生物催化剂, 该基因被广泛用作功能基因生物标志物, 用于检索不同生态位厌氧氨氧化细菌的多样性、丰度和活性[51]。其功能不可被其他生物催化剂所替代, 使HZS基因成为厌氧氨氧化细菌的可靠的功能性生物标志物和PCR检测技术手段[52]。虽然HZS基因在厌氧氨氧化菌的基因组中被频繁地发现, 但其基因拷贝数在任何基因组中都是未知的。例如, 在斯图加蒂斯库尼尼亚念珠菌中首次发现了两个可能编码HZS的候选基因簇[53], 但该结果并未得到后续分子代谢机制研究的实验验证。
1.2.3 肼氧化酶(hydrazine oxidase, HZO)肼氧化酶(hydrazine oxidase, HZO)在低底物浓度下可以将肼氧化成氮气[54]。HZO是一种三聚体蛋白, 其光谱被连二亚硫酸钠还原后在419 nm、552 nm和523 nm处有最大吸光度, 且在472 nm处有一个独特的峰, 这与厌氧氨氧化富集培养物的HAO[55]和硝化细菌欧洲亚硝化单胞菌(Nitrosomonas europaea)的HAO是不同的[56], 这表明与厌氧氨氧化细菌的HAO相比, HZO对肼具有较高的亲和力。
邢崇阳等[57]通过实时荧光定量聚合酶链反应(RT-qPCR)技术和16S rRNA高通量测序方法进行实验, 表明了HZO酶的转录水平变化可以更精确地测定厌氧氨氧化颗粒对羟胺的耐受范围。为检测环境样品中hzo基因的分布和多样性的方法是通过简并PCR引物和PCR反应[58]。HIRSCH等[59]比较了hzoA/hzoB基因检测的不同简并引物, 通过对16S rRNA和hzoA/hzoB基因序列的比较分析, 揭示了各种水生生态系统中厌氧氨氧化细菌的多样性和分布。
1.3 异养硝化好氧反硝化废水中氨氮的去除主要包括异养硝化好氧反硝化过程, 其中异养硝化过程的第一步是通过氨单加氧酶(AMO)将NH4+氧化为羟胺。第二步是通过羟胺氧化还原酶(HAO)将羟胺氧化成亚硝酸盐, 再由亚硝酸盐氧化还原酶(NOR)通过将亚硝酸盐氧化为硝酸盐(图 3)[60]。这一硝化过程可以通过全程氨氧化微生物, 第一篇关于全程氨氧化微生物的报道与硝化螺旋菌属(Nitrospira)有关, 其基因组包含一整套AMO、HAO和NOR基因[61]。
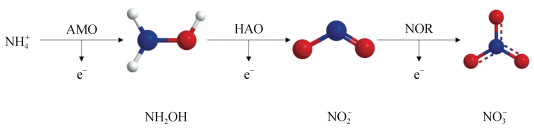 |
| 图 3 异养硝化电子传递示意图 Fig. 3 Schematic of electron transport during heterotrophic nitrification |
在反硝化过程中硝酸盐还原菌是关键类群。反硝化的第一步是通过硝酸盐还原酶催化硝酸盐生成亚硝酸盐。第二步是使亚硝酸盐通过亚硝酸盐还原酶(NIR)转化为NO, 然后通过一氧化氮还原酶(NOR)转化为一氧化二氮, 最后通过一氧化二氮还原酶(NOS)转化为氮气(图 4)[62]。
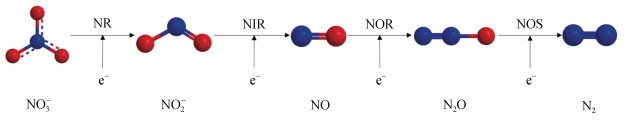 |
| 图 4 好氧反硝化电子传递示意图 Fig. 4 Schematic of electron transport during aerobic denitrification |
然而硝化和反硝化对营养和溶氧需求具有不一致的局限性, 因此在不断地深入研究下, 荷兰科学家Robertson和Kuenen在1984年首次分离纯化得到一株好氧反硝化菌脱氮副球菌(Thiosphaera pantotropha), 该菌株同时具备了异养硝化和好氧反硝化功能[63]。研究人员由此提出了异养硝化好氧反硝化菌(heterotrophic nitrification-aerobic denitrification, HN-AD), 这种菌株可以在好氧条件下同时完成上述硝化和反硝化反应过程[44]。当碳源充足时, HN-AD菌株可以快速生长形成高效的硝化反硝化生物脱氮能力, 显著降低生物脱氮成本[64-65]。
HN-AD菌来源较广泛, 对环境的适应能力相对较强, 可应用养殖废水、城镇生活废水、制药废水等多种环境中进行生物脱氮, 具有突出的脱氮能力(表 3)。
| 应用方向 | 菌株名称 | 除氮种类 | 脱氮效率/% |
| 养殖废水 | Acinetobacter sp. T1不动杆菌[66] | 化学需氧量(COD) | 64 |
| 氨氮(NH4+-N) | 42 | ||
| 总氮(TN) | 28 | ||
| Marinomonas aquimarina SD167SA1海草海单胞菌[67] | NH4+-N | 100 | |
| Halomonas titanicae SD136WN1泰坦尼克号盐单胞菌[67] | NH4+-N | 100 | |
| 生活废水 | Acinetobacter sp. ND7不动杆菌[68] | NH4+-N(51. 0 mg/L) | 99.8 |
| NO3–-N(52.1 mg/L) | 97.18 | ||
| NO2–-N(51.8 mg/L) | 96.2 | ||
| Enterobacter cloacae CF-S27阴沟肠杆菌[69] | NH4+-N | 81 | |
| NO3–-N | 99.9 | ||
| NO2–-N | 92.8 | ||
| 制药废水 | Acinetobacter sp. JR1不动杆菌[70] | NH4+-N | 98.5 |
| NO3–-N | 91.1 |
氨单加氧酶(ammonia monooxygenase, AMO)是一种依赖于铜的膜结合酶, 可以将NH4+催化成NH2OH, 是硝化反应的第一步[71]。AMO有助于甲烷和一氧化碳的全球循环以及碳氢化合物和卤化化合物的降解[71]。编码AMO基因普遍存在于HN-AD中, 有时还会存在amoD和amoE基因, 这两个基因具有高度序列保守性, 并在AMO操纵子上有相似的定位[72], 表明这两个基因在氨氧化过程中起重要的催化作用[14]。
amoA、amoB和amoC基因组成了AMO操纵子[73], 其存在于迄今已检测的变形菌门β-亚门的所有氨氧化剂菌株中[73]。相比之下在γ-亚门海洋亚硝化球菌(Nitrosococcus oceani)和亚硝基球菌属(Nitrosococcus sp. C-113)中只发现了单一的AMO操作子[55]。基于GenBank中可用的AMO序列, amoA的内部部分已从环境样品中扩增和克隆, 证明了amoA作为氨氧化细菌的功能标记物的有用性[74]。
1.3.2 羟胺氧化还原酶(hydroxylamine oxidase, HAO)羟胺氧化还原酶(hydroxylamine oxidase, HAO)位于细胞膜周质中, 可以将羟胺转化为亚硝酸盐。Schalk等人在厌氧条件下从厌氧氨氧化富集培养基中纯化并鉴定了羟胺氧化还原酶(HAO)[54]。Yamanaka等人通过研究表明羟胺在好氧和厌氧条件下都能被HAO氧化[75]。Otte等人通过实验揭示了N2O是羟胺氧化反应的副产物之一[76]。因此, HAO在N2O的生成中也起着重要的作用。
为研究HAO的酶学性质, 可以在使用渗透压休克法提取HAO的粗酶液后采用DEAE Sefinose TM FF离子交换层析和Sefinose TM CL-6B凝胶过滤层析, 并通过Brandford法测定蛋白浓度[77]。然后通过在Tris-HCl (pH=7.4)和EDTA-2Na等反应体系内对反应体系中羟胺的减少量进行HAO的酶活测定, 由此得出HAO反应的最适pH为8.0, 最适温度为30 ℃[78]。
1.3.3 硝酸盐还原酶(nitrate reductase, NR)硝酸盐还原酶(nitrate reductase, NR)可以将硝酸盐还原为亚硝酸盐, 是催化反硝化过程中的第一步反应。根据硝酸盐还原酶的细胞位置、操纵子和活性位点结构分为周质硝酸盐还原酶(Nap)、呼吸性硝酸盐还原酶(Nar)和同化性硝酸盐还原酶(Nas)[79], 其中Nar镶嵌于细胞膜内, Nap位于细胞质膜和细胞壁之间。在有氧条件下Nap优先催化硝酸盐还原反应, 且在厌氧条件下也可以正常表达, 而Nar只能在厌氧条件下进行表达[80]。
Nar酶活测定方法与NIR酶活测定方式相似, 根据反应前后硝酸钠的减少量进行酶活测定[45]。最早是在真养产碱杆菌(Alcaligenes eutrophus)的结构基因中鉴定出编码Nap的基因, 其NAP系统在好氧条件下的稳定期有最大的表达量[81]。
1.3.4 一氧化氮还原酶(nitric oxide reductase, NOR)一氧化氮还原酶(nitric oxide reductase, NOR)是一种含铁的金属酶, 催化NO生成温室气体一氧化二氮(N2O)。此外NOR可以解决囊性纤维化肺厌氧呼吸产生的NO的细胞毒性, 因此NOR在像绿脓杆菌(Pseudomonas aeruginosa)等病原体引起的急性和慢性感染的致病菌临床和制药上也很重要[82]。在生物脱氮方面, 细菌中的NOR还原NO的过程中, Glu配体可能会与FeB短暂分离, 在双核中心创造足够的空间, 使两个NO分子容纳在血红素b3和FeB双核中心上[83]。
为测定NOR的酶活, 可以由抗坏血酸钠提供电子, 由吩嗪硫酸甲酯传递电子, 由NO接受电子, 通过气相色谱法以反应体系内氧化亚氮的增加量来计算酶活性[84]。由考马斯亮蓝法测定酶液中总蛋白质的浓度测定[85]; 由二苯胺法测定酶液中总核酸(DNA)的浓度[86]。
2 新型生物脱氮技术的其他应用A/O步进式活性污泥工艺因其具有良好的脱氮潜力而受到越来越多的关注, 不仅在技术上具有先进性, 而且在经济上也有很显著的优越性。A/O将前段缺氧段和后段好氧段串联在一起, 可以在同一槽中反应缺氧/好氧(anoxic/oxic), 系统简称为A/O。在缺氧阶段污水中的有机氮可以被反硝化菌所利用, 减轻好氧阶段的有机负荷, 同时反硝化反应产生的碱度可以满足后续硝化反应对碱度的需求。该工艺可以降低工程造价, 所以这种形式有利于对人为高效生物脱氮过程的改造。在A/O的基础上, 人们提出A2/O污水处理系统, 即使污水经过厌氧、缺氧及好氧3个生物处理过程, 达到同时去除生化耗氧量、氮和磷的目的[87]。
此外微藻和蓝藻细胞也被应用于废水的处理。存在于淡水和海洋系统的微藻和蓝藻细胞可以单独存在, 也可以链状或成群存在, 其特点是相对较快的生长和对恶劣条件的适应性。在废水处理中, 它们进行光合作产生O2, 将有机废物分解为简单的无机营养物。此外, 微藻和蓝藻也可以去除废水水体中的无机营养物质[88]。微藻和蓝藻的快速生长需要大量的氮和磷, 这可以通过从废水中吸收氮和磷来有效地提供。它们的氮回收潜力是这些微生物的特殊优势。微藻和蓝藻脱氮能力依赖于亚硝酸盐的氨化和同化还原作用形成氨, 其吸收的硝酸盐和氨被转化为生物量, 而不是通过异化硝酸盐还原作用生成氮气释放到大气中[89]。
微藻光合作用所需的CO2溶解度受pH值和温度的负面影响。pH和温度的升高也会导致NH4+挥发, 对微藻对NH4+的去除过程产生不利影响[90]。同时微藻的最佳生长和脱氮效率也受光的质量(波长)和数量以及温度的影响。不同种类微藻生长的最佳温度不同, 从低于10 ℃到中等温度(10~20 ℃), 有些甚至超过30 ℃。但当微藻在较低温度下生长减少时, 温度升高通常会增加代谢活性[91]。
微藻或微藻与细菌的混合培养使其具有广泛的代谢活动, 使它们能够适应各种条件。这些有价值的群落可以在不同的环境和不同的营养负荷中生长。菌群中微生物的协同代谢相互作用可以实现废水中营养物质的去除[92]。在这样一个有益的组合中, 微藻光合作用产生的氧气被异养好氧菌利用来生物降解废水中的有机污染物。异养细菌可以释放二氧化碳, 在有光的情况下, 微藻可以利用这些二氧化碳。
3 结论与展望面对愈加严重的水体氮素污染状况, 寻求经济高效的处理方法刻不容缓。氨氮废水的处理方法多种多样, 优缺点各有不同, 在这些方法中寻找到成本和效率之间的平衡重中之重。相比于其他脱氮技术, 生物脱氮技术更加经济高效, 其中新型生物脱氮技术与传统生物脱氮技术相比具有更多的优点和更新更准确的理论基础。更加深刻地了解与研究新型生物脱氮技术, 对脱氮优良菌株进行筛选, 集中在C/N、温度、pH值和DO等环境因素对菌株及相关酶系脱氮性能的影响方面进行研究。由于不同菌株表达的酶系有所不同, 系统地研究异养硝化好氧反硝化过程中酶的种类、活性和代谢产物的种类尤为重要。并且针对不同的氨氮浓度使用多种技术的耦合手段, 通过分子生物学等方式对基因进行编码等手段提高HN-AD菌胞内电子传递效率, 通过分子技术和蛋白质工程方法将生物脱氮酶进行基因克隆和异源表达直接应用于高氮素废水的生物脱氮, 同时, 提高异养硝化好氧反硝化效能是今后发展的方向, 采用拥耐高温和耐盐度等优点的菌群在未来的生物脱氮技术领域也具有较大的潜力。
| [1] |
ALDAYA M M, RODRIGUEZ C I, FERNANDEZ-POULUSSEN A, et al. Grey water footprint as an indicator for diffuse nitrogen pollution: The case of Navarra, Spain[J]. Science of the Total Environment, 2020, 698: 134338. DOI:10.1016/j.scitotenv.2019.134338 |
| [2] |
MOSS B, JEPPESEN E, SØNDERGAARD M, et al. Nitrogen, macrophytes, shallow lakes and nutrient limitation: resolution of a current controversy?[J]. Hydrobiologia, 2012, 710(1): 3-21. |
| [3] |
SCAVIA D, BERTANI I, OBENOUR D R, et al. Ensemble modeling informs hypoxia management in the northern Gulf of Mexico[J]. Proceedings of the National Academy of Sciences, 2017, 114(33): 8823-8828. DOI:10.1073/pnas.1705293114 |
| [4] |
PAERL H W, OTTEN T G. Environmental science. Blooms bite the hand that feeds them[J]. Science, 2013, 342(6157): 433-434. DOI:10.1126/science.1245276 |
| [5] |
FEWTRELL L. Drinking-water nitrate, methemoglobinemia, and global burden of disease: a discussion[J]. Environmental Health Perspectives, 2004, 112(14): 1371-1374. DOI:10.1289/ehp.7216 |
| [6] |
POULSEN R, CEDERGREEN N, HAYES T, et al. Nitrate: An environmental endocrine disruptor? A review of evidence and research needs[J]. Environmental Science & Technology, 2018, 52(7): 3869-3887. |
| [7] |
WURTSBAUGH W A, PAERL H W, DODDS W K. Nutrients, eutrophication and harmful algal blooms along the freshwater to marine continuum[J]. Wiley Interdisciplinary Reviews: Water, 2019, 6(5): e1373. DOI:10.1002/wat2.1373 |
| [8] |
王紫琪, 杨基先, 何伟华, 等. 一株异养硝化-好氧反硝化菌Bacillus flexus X1-L的分离鉴定及效能研究[J]. 环境科学学报, 2020, 40(10): 3742-3748. WANG Ziqi, YANG Jixian, HE Weihua, et al. Isolation and characterization of a heterotrophic nitrification-aerobic denitrification strain Bacillus flexus X1-L[J]. Acta Scientiae Circumstantiae, 2020, 40(10): 3742-3748. |
| [9] |
WANG F, LIU Y, WANG J, et al. Influence of growth manner on nitrifying bacterial communities and nitrification kinetics in three lab-scale bioreactors[J]. Journal of Industrial Microbiology and Biotechnology, 2012, 39(4): 595-604. DOI:10.1007/s10295-011-1065-x |
| [10] |
张周, 赵明星, 阮文权, 等. 短程硝化反硝化工艺处理低C/N餐厨废水[J]. 环境工程学报, 2015, 9(9): 4165-4170. ZHANG Zhou, ZHAO Mingxing, RUAN Wenquan, et al. Short-cut nitrification and denitrification process for treating low C/N food wastewater[J]. Chinese Journal of Environmental Engineering, 2015, 9(9): 4165-4170. |
| [11] |
REGAN J M, HARRINGTON G W, NOGUERA D R. Ammonia- and nitrite-oxidizing bacterial communities in a pilot-scale chloraminated drinking water distribution system[J]. Applied and Environmental Microbiology, 2002, 68(1): 73-81. DOI:10.1128/AEM.68.1.73-81.2002 |
| [12] |
SCHMIDT I, HERMELINK C, VAN DE PAS-SCHOONEN K, et al. Anaerobic ammonia oxidation in the presence of nitrogen oxides (NO(x)) by two different lithotrophs[J]. Applied and Environmental Microbiology, 2002, 68(11): 5351-5357. DOI:10.1128/AEM.68.11.5351-5357.2002 |
| [13] |
REIS M P, AVILA M P, KEIJZER R M, et al. The effect of human settlement on the abundance and community structure of ammonia oxidizers in tropical stream sediments[J]. Frontiers in Microbiology, 2015, 6: 898. |
| [14] |
ARP D J, CHAIN P S, KLOTZ M G. The impact of genome analyses on our understanding of ammonia-oxidizing bacteria[J]. Annual Review of Microbiology, 2007, 61: 503-528. DOI:10.1146/annurev.micro.61.080706.093449 |
| [15] |
高春娣, 赵楠, 安冉, 等. FNA对短程硝化污泥菌群结构的影响[J]. 中国环境科学, 2019, 39(5): 1977-1984. GAO Chundi, ZHAO Nan, AN Ran, et al. Effect of FNA on microorganism community structures of partial nitrification sludge[J]. China Environmental Science, 2019, 39(5): 1977-1984. |
| [16] |
JENNI S, VLAEMINCK S E, MORGENROTH E, et al. Successful application of nitritation/anammox to wastewater with elevated organic carbon to ammonia ratios[J]. Water Research, 2014, 49: 316-326. DOI:10.1016/j.watres.2013.10.073 |
| [17] |
TOBINO T, CHEN J, SAWAI O, et al. Inline thickener-MBR as a compact, energy efficient organic carbon removal and sludge production devise for municipal wastewater treatment[J]. Chemical Engineering and Processing-Process Intensification, 2016, 107: 177-184. DOI:10.1016/j.cep.2015.11.010 |
| [18] |
张晟伟, 王洪涛. 生物脱氮技术研究进展[C]//北京力学会. 北京力学会第二十九届学术年会论文集(续二). 中国矿业大学(北京)地球科学与测绘工程学院; 秦皇岛市自来水有限公司山海关水厂, 2023: 4.
|
| [19] |
张婷, 吴鹏, 沈耀良, 等. CSTR和MBR反应器的短程硝化快速启动[J]. 环境科学, 2017, 38(8): 3399-3405. ZHANG Ting, WU Peng, SHEN Yaoliang, et al. Fast start-up of shortcut nitrification in a CSTR and an MBR[J]. Environmental Science, 2017, 38(8): 3399-3405. |
| [20] |
张杰, 张艳辉, 李冬, 等. 不同厌氧方式对连续流亚硝化恢复的影响[J]. 哈尔滨工业大学学报, 2017, 49(2): 1-5. ZHANG Jie, ZHANG Yanhui, LI Dong, et al. Effects of different anaerobic modes on recovery of partial nitrification in continuous flow process[J]. Journal of Harbin Institute of Technology, 2017, 49(2): 1-5. |
| [21] |
MA B, WANG S, CAO S, et al. Biological nitrogen removal from sewage via anammox: Recent advances[J]. Bioresource Technology, 2016, 200: 981-990. DOI:10.1016/j.biortech.2015.10.074 |
| [22] |
WANG C, QIAO S, ZHOU J. Strategy of nitrate removal in anaerobic ammonia oxidation-dependent processes[J]. Chemosphere, 2022, 137586. |
| [23] |
WANG B, GUO Y, ZHAO M, et al. Achieving energy-efficient nitrogen removal and excess sludge reutilization by partial nitritation and simultaneous anammox denitrification and sludge fermentation process[J]. Chemosphere, 2019, 218: 705-714. DOI:10.1016/j.chemosphere.2018.11.168 |
| [24] |
TRINH H P, LEE S H, JEONG G, et al. Recent developments of the mainstream anammox processes: challenges and opportunities[J]. Journal of Environmental Chemical Engineering, 2021, 9(4): 105583. DOI:10.1016/j.jece.2021.105583 |
| [25] |
TANG C J, ZHENG P, WANG C H, et al. Performance of high-loaded ANAMMOX UASB reactors containing granular sludge[J]. Water Research, 2011, 45(1): 135-144. DOI:10.1016/j.watres.2010.08.018 |
| [26] |
PHAN T N, VAN TRUONG T T, HA N B, et al. High rate nitrogen removal by ANAMMOX internal circulation reactor (IC) for old landfill leachate treatment[J]. Bioresour Technology, 2017, 234: 281-288. DOI:10.1016/j.biortech.2017.02.117 |
| [27] |
彭荷衢, 刁兴兴, 张伟军, 等. 两级自养反硝化实现垃圾渗滤液的深度脱氮[J]. 环境工程学报, 2019, 13(9): 2113-2120. PENG Hequ, DIAO Xingxing, ZHANG Weijun, et al. Deep denitrification of landfill leachate by twostage autotrophic denitrification process[J]. Chinese Journal of Environmental Engineering, 2019, 13(9): 2113-2120. |
| [28] |
王凡, 陆明羽, 殷记强, 等. 反硝化-短程硝化-厌氧氨氧化工艺处理晚期垃圾渗滤液的脱氮除碳性能[J]. 环境科学, 2018, 39(8): 3782-3788. WANG Fan, LU Mingyu, YIN Jiqiang, et al. Treatment of old landfill leachate via a denitrification-partial nitritation-ANAMMOX process[J]. Environmental Science, 2018, 39(8): 3782-3788. |
| [29] |
LIANG Z, LIU J X, LI J. Decomposition and mineralization of aquatic humic substances (AHS) in treating landfill leachate using the Anammox process[J]. Chemosphere, 2009, 74(10): 1315-1320. DOI:10.1016/j.chemosphere.2008.11.073 |
| [30] |
徐晓晨, 周亮, 王超, 等. SNAD-MBBR处理垃圾渗滤液厌氧出水的脱氮研究[J]. 中国给水排水, 2018, 34(23): 21-25. XU Xiaochen, ZHOU Liang, WANG Chao, et al. Nitrogen removal from Anaerobic Effluent of Landfill Leachate by SNAD-MBBR Process[J]. China Water & Wastewater, 2018, 34(23): 21-25. |
| [31] |
杨延栋, 黄京, 韩晓宇, 等. 一体式厌氧氨氧化工艺处理高氨氮污泥消化液的启动[J]. 中国环境科学, 2015, 35(4): 1082-1087. YANG Yandong, HUANG Jing, HAN Xiaoyu, et al. Start-up of one-stage partial nitrification/anammox process treating ammonium-rich reject water[J]. China Environmental Science, 2015, 35(4): 1082-1087. |
| [32] |
杨岚, 彭永臻, 李健伟, 等. 缺氧MBBR耦合部分厌氧氨氧化强化城市生活污水深度脱氮[J]. 环境科学, 2019, 40(8): 3668-3674. YANG Lan, PENG Yongzhen, LI Jianwei, et al. Advanced denitrification of municipal wastewater achieved via partial ANAMMOX in anoxic MBBR[J]. Environmental Science, 2019, 40(8): 3668-3674. |
| [33] |
吴鹏, 张诗颖, 宋吟玲, 等. ABR工艺ANAMMOX耦合短程硝化协同脱氮处理城市污水[J]. 环境科学, 2016, 37(8): 3108-3113. WU Peng, ZHANG Shiying, SONG Yinling, et al. Nitrogen removal of municipal wastewater by ANAMMOX coupled shortcut nitrification in anaerobic baffled reactor[J]. Environmental Science, 2016, 37(8): 3108-3113. |
| [34] |
DAVEREY A, SU S H, HUANG Y T, et al. Partial nitrification and anammox process: A method for high strength optoelectronic industrial wastewater treatment[J]. Water Research, 2013, 47(9): 2929-2937. DOI:10.1016/j.watres.2013.01.028 |
| [35] |
TANG C J, ZHENG P, CHEN T T, et al. Enhanced nitrogen removal from pharmaceutical wastewater using SBA-ANAMMOX process[J]. Water Research, 2011, 45(1): 201-210. DOI:10.1016/j.watres.2010.08.036 |
| [36] |
薛占强, 李玉平, 李海波, 等. 短程硝化/厌氧氨氧化/全程硝化工艺处理焦化废水[J]. 中国给水排水, 2011, 27(1): 15-19. XUE Zhanqiang, LI Yuping, LI Haibo, et al. Shortcut nitrification / anaerobic ammonium oxidation / complete nitrification process for treatment of coking wastewater[J]. China Water & Wastewater, 2011, 27(1): 15-19. |
| [37] |
YE R W, AVERILL B A, TIEDJE J M. Denitrification: production and consumption of nitric oxide[J]. Applied and Environmental Microbiology, 1994, 60(4): 1053-1058. DOI:10.1128/aem.60.4.1053-1058.1994 |
| [38] |
FARVER O, KRONECK P M, ZUMFT W G, et al. Allosteric control of internal electron transfer in cytochrome cd 1 nitrite reductase[J]. Proceedings of the National Academy of Scie nces, 2003, 100(13): 7622-7625. DOI:10.1073/pnas.0932693100 |
| [39] |
CASCIOTTI K L, WARD B B. Dissimilatory nitrite reductase genes from autotrophic ammonia-oxidizing bacteria[J]. Applied and Environmental Microbiology, 2001, 67(5): 2213-2221. DOI:10.1128/AEM.67.5.2213-2221.2001 |
| [40] |
COYNE M, ARUNAKUMARI A, AVERILL B, et al. Immunological identification and distribution of dissimilatory heme cd1 and nonheme copper nitrite reductases in denitrifying bacteria[J]. Applied and Environmental Microbiology, 1989, 55(11): 2924-2931. DOI:10.1128/aem.55.11.2924-2931.1989 |
| [41] |
SHAPLEIGH J, PAYNE W. Differentiation of c, d 1 cytochrome and copper nitrite reductase production in denitrifiers[J]. FEMS microbiology letters, 1985, 26(3): 275-279. |
| [42] |
KLOOS K, FESEFELDT A, GLIESCHE C G, et al. DNA-probing indicates the occurrence of denitrification and nitrogen fixation genes in Hyphomicrobium. Distribution of denitrifying and nitrogen fixing isolates of Hyphomicrobium in a sewage treatment plant[J]. FEMS microbiology ecology, 1995, 18(3): 205-213. DOI:10.1111/j.1574-6941.1995.tb00177.x |
| [43] |
BRAKER G, FESEFELDT A, WITZEL K P. Development of PCR primer systems for amplification of nitrite reductase genes (nirK and nirS) to detect denitrifying bacteria in environmental samples[J]. Applied and Environmental Microbiology, 1998, 64(10): 3769-3775. DOI:10.1128/AEM.64.10.3769-3775.1998 |
| [44] |
MULDER A, VAN DE GRAAF A A, ROBERTSON L, et al. Anaerobic ammonium oxidation discovered in a denitrifying fluidized bed reactor[J]. FEMS Microbiology Ecology, 1995, 16(3): 177-183. DOI:10.1111/j.1574-6941.1995.tb00281.x |
| [45] |
ZHAO B, HE Y L, HUANG J, et al. Heterotrophic nitrogen removal by Providencia rettgeri strain YL[J]. Journal of Industrial Microbiology and Biotechnology, 2010, 37(6): 609-616. DOI:10.1007/s10295-010-0708-7 |
| [46] |
郭金玲, 陈程鹏, 周一郎, 等. 黑曲霉β-葡萄糖苷酶的分离纯化及酶学性质研究[J]. 中国酿造, 2021, 40(2): 83-87. GUO Jinling, CHEN Chengpeng, ZHOU Yilang, et al. Isolation, purification and enzymatic properties of β-glucosidase from Aspergillus niger[J]. China Brewing, 2021, 40(2): 83-87. |
| [47] |
刘玮, 邱崇顺, 何宇星, 等. 降解亚硝酸盐乳杆菌的筛选鉴定及其NiRs酶学性质[J]. 食品研究与开发, 2022, 43(13): 164-171. LIU Wei, QIU Chongshun, HE Yuxing, et al. Screeningand Identification of Nitrite-Degrading Lactobacillus and Characterization of Its Nitrite Reductase[J]. Food Research and Development, 2022, 43(13): 164-171. |
| [48] |
鲍晨曦. 近海滩涂与沉积物中嗜盐古菌多样性及其亚硝酸盐还原酶研究[D]. 镇江: 江苏大学, 2022. BAO Chenxi. Biodiversity and nitrite reductases of halophilic archaea from offshore beaches and sediment[D]. Zhen Jiang: Jiangsu University, 2022. |
| [49] |
KUYPERS M M, MARCHANT H K, KARTAL B. The microbial nitrogen-cycling network[J]. Nature Reviews Microbiology, 2018, 16(5): 263-276. DOI:10.1038/nrmicro.2018.9 |
| [50] |
KARTAL B, KUENEN J G, VAN LOOSDRECHT M C M. Sewage treatment with anammox[J]. Science, 2010, 328(5979): 702-703. DOI:10.1126/science.1185941 |
| [51] |
YANG Y, LI M, LI X Y, et al. Two identical copies of the hydrazine synthase gene clusters found in the genomes of anammox bacteria[J]. International Biodeterioration & Biodegradation, 2018, 132: 236-240. |
| [52] |
HARHANGI H R, LE ROY M, VAN ALEN T, et al. Hydrazine synthase, a unique phylomarker with which to study the presence and biodiversity of anammox bacteria[J]. Applied and Environmental Microbiology, 2012, 78(3): 752-758. DOI:10.1128/AEM.07113-11 |
| [53] |
STROUS M, PELLETIER E, MANGENOT S, et al. Deciphering the evolution and metabolism of an anammox bacterium from a community genome[J]. Nature, 2006, 440(7085): 790-794. DOI:10.1038/nature04647 |
| [54] |
SCHALK J, DE VRIES S, KUENEN J G, et al. Involvement of a novel hydroxylamine oxidoreductase in anaerobic ammonium oxidation[J]. Biochemistry, 2000, 39(18): 5405-5412. DOI:10.1021/bi992721k |
| [55] |
NORTON J M, ALZERRECA J J, SUWA Y, et al. Diversity of ammonia monooxygenase operon in autotrophic ammonia-oxidizing bacteria[J]. Archives of Microbiology, 2001, 177(2): 139-149. |
| [56] |
ARCIERO D M, BALNY C, HOOPER A B. Spectroscopic and rapid kinetic studies of reduction of cytochrome c554 by hydroxylamine oxidoreductase from Nitrosomonas europaea[J]. Biochemistry, 1991, 30(48): 11466-11472. DOI:10.1021/bi00112a014 |
| [57] |
邢崇阳, 范禹辰, 陈璇, 等. 羟胺对厌氧氨氧化污泥群落的影响[J]. 环境科学, 2020, 41(7): 3365-3372. XING Chongyang, FAN Yuchen, CHEN Xuan, et al. Effect of Hydroxylamine on Community of ANAMMOX Sludge[J]. Environmental Science, 2020, 41(7): 3365-3372. |
| [58] |
SCHMID M C, HOOPER A B, KLOTZ M G, et al. Environmental detection of octahaem cytochrome c hydroxylamine/hydrazine oxidoreductase genes of aerobic and anaerobic ammonium-oxidizing bacteria[J]. Environmental Microbiology, 2008, 10(11): 3140-3149. DOI:10.1111/j.1462-2920.2008.01732.x |
| [59] |
HIRSCH M D, LONG Z T, SONG B. Anammox bacterial diversity in various aquatic ecosystems based on the detection of hydrazine oxidase genes (hzoA/hzoB)[J]. Microbial Ecology, 2011, 61(2): 264-276. DOI:10.1007/s00248-010-9743-1 |
| [60] |
CAPONE D G. Discovery of new nitrite-oxidizing bacteria increases phylogenetic and metabolic diversity within this niche[J]. MBio, 2018, 9(5): e01619-1618. |
| [61] |
VAN KESSEL M A, SPETH D R, ALBERTSEN M, et al. Complete nitrification by a single microorganism[J]. Nature, 2015, 528(7583): 555-559. DOI:10.1038/nature16459 |
| [62] |
BERGAUST L, VAN SPANNING R J M, FROSTEGARD A, et al. Expression of nitrous oxide reductase in Paracoccus denitrificans is regulated by oxygen and nitric oxide through FnrP and NNR[J]. Microbiology (Reading), 2012, 158(Pt 3): 826-834. |
| [63] |
ROBERTSON L, KUENEN J. Aerobic denitrification—old wine in new bottles?[J]. Antonie van Leeuwenhoek, 1984, 50: 525-544. DOI:10.1007/BF02386224 |
| [64] |
CHEN Q, NI J. Ammonium removal by Agrobacterium sp. LAD9 capable of heterotrophic nitrification-aerobic denitrification[J]. Journal of bioscience and bioengineering, 2012, 113(5): 619-623. DOI:10.1016/j.jbiosc.2011.12.012 |
| [65] |
黄诗玮, 田云, 马述, 等. 异养硝化-好氧反硝化菌氮代谢特性研究进展[J]. 生物学杂志, 2023, 40(1): 91-97. HUANG Shiwei, TIAN Yun, MA Shu, et al. Research progress on nitrogen metabolism characteristics of heterotrophic nitrification-aerobic denitrification bacteria[J]. Journal of Biology, 2023, 40(1): 91-97. |
| [66] |
CHEN S, HE S, WU C, et al. Characteristics of heterotrophic nitrification and aerobic denitrification bacterium Acinetobacter sp. T1 and its application for pig farm wastewater treatment[J]. Journal of Bioscience and Bioengineering, 2019, 127(2): 201-205. DOI:10.1016/j.jbiosc.2018.07.025 |
| [67] |
CHEN Q, NI J. Heterotrophic nitrification–aerobic denitrification by novel isolated bacteria[J]. Journal of Industrial Microbiology & Biotechnology, 2010, 38(9): 1305-1310. |
| [68] |
XIA L, LI X, FAN W, et al. Heterotrophic nitrification and aerobic denitrification by a novel Acinetobacter sp. ND7 isolated from municipal activated sludge[J]. Bioresource Technology, 2020, 301. |
| [69] |
PADHI S K, TRIPATHY S, MOHANTY S, et al. Aerobic and heterotrophic nitrogen removal by Enterobacter cloacae CF-S27 with efficient utilization of hydroxylamine[J]. Bioresource Technology, 2017, 232: 285-296. DOI:10.1016/j.biortech.2017.02.049 |
| [70] |
YANG J R, WANG Y, CHEN H, et al. Ammonium removal characteristics of an acid-resistant bacterium Acinetobacter sp. JR1 from pharmaceutical wastewater capable of heterotrophic nitrification-aerobic denitrification[J]. Bioresource Technology, 2019, 274: 56-64. DOI:10.1016/j.biortech.2018.10.052 |
| [71] |
HYMAN M R, MURTON I B, ARP D J. Interaction of ammonia monooxygenase from Nitrosomonas europaea with alkanes, alkenes, and alkynes[J]. Applied and Environmental Microbiology, 1988, 54(12): 3187-3190. DOI:10.1128/aem.54.12.3187-3190.1988 |
| [72] |
SAYAVEDRA-SOTO L, HOMMES N, ALZERRECA J, et al. Transcription of the amoC, amoA and amoB genes in Nitrosomonas europaea and Nitrosospira sp. NpAV[J]. FEMS Microbiology Letters, 1998, 167(1): 81-88. DOI:10.1111/j.1574-6968.1998.tb13211.x |
| [73] |
NORTON J M, LOW J M, KLOTZ M G. The gene encoding ammonia monooxygenase subunit A exists in three nearly identical copies in Nitrosospira sp. NpAV[J]. FEMS Microbiology Letters, 1996, 139(2/3): 181-188. |
| [74] |
PURKHOLD U, POMMERENING-RöSER A, JURETSCHKO S, et al. Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA and amoA sequence analysis: implications for molecular diversity surveys[J]. Applied and Environmental Microbiology, 2000, 66(12): 5368-5382. DOI:10.1128/AEM.66.12.5368-5382.2000 |
| [75] |
YAMANAKA T, SAKANO Y. Oxidation of hydroxylamine to nitrite catalyzed by hydroxylamine oxidoreductase purified from Nitrosomonas europaea[J]. Current Microbiology, 1980, 4: 239-244. DOI:10.1007/BF02605864 |
| [76] |
OTTE S, SCHALK J, KUENEN J, et al. Hydroxylamine oxidation and subsequent nitrous oxide production by the heterotrophic ammonia oxidizer Alcaligenes faecalis[J]. Applied Microbiology and Biotechnology, 1999, 51: 255-261. DOI:10.1007/s002530051390 |
| [77] |
刘泽英. Acinetobacter sp. Y1的氨氮去除性能及其关键酶的研究[D]. 太原: 太原理工大学, 2016. LIU Zeying. Study on ammonia nitrogen removal characteristic and its key enzymes of Acinetobacter sp. Y1[D]. Tai Yuan: Taiyuan University of Technology, 2016. |
| [78] |
王瑶, 刘玉香, 安华, 等. 金属离子对粪产碱杆菌C16的脱氮和亚硝酸盐积累的影响[J]. 微生物学通报, 2014, 41(11): 2254-2263. WANG Yao, LIU Yuxiang, AN Hua, et al. Influence of metal ions on nitrogen removal and NO2–-N accumulation by Alcaligenes faecalis C16[J]. Microbiology China, 2014, 41(11): 2254-2263. |
| [79] |
SPARACINO-WATKINS C, STOLZ J F, BASU P. Nitrate and periplasmic nitrate reductases[J]. Chemical Society Reviews, 2014, 43(2): 676-706. DOI:10.1039/C3CS60249D |
| [80] |
BELL L C, RICHARDSON D J, FERGUSON S J. Periplasmic and membrane-bound respiratory nitrate reductases in Thiosphaera pantotropha. The periplasmic enzyme catalyzes the first step in aerobic denitrification[J]. FEBS letters, 1990, 265(1/2): 85-87. |
| [81] |
SIDDIQUI R, WARNECKE-EBERZ U, HENGSBERGER A, et al. Structure and function of a periplasmic nitrate reductase in Alcaligenes eutrophus H16[J]. Journal of Bacteriology, 1993, 175(18): 5867-5876. DOI:10.1128/jb.175.18.5867-5876.1993 |
| [82] |
HINO T, MATSUMOTO Y, NAGANO S, et al. Structural basis of biological N2O generation by bacterial nitric oxide reductase[J]. Science, 2010, 330(6011): 1666-1670. DOI:10.1126/science.1195591 |
| [83] |
MOëNNE-LOCCOZ P. Spectroscopic characterization of heme iron–nitrosyl species and their role in NO reductase mechanisms in diiron proteins[J]. Natural Product Reports, 2007, 24(3): 610-620. DOI:10.1039/B604194A |
| [84] |
HE Z X, YUAN L J, WEI Y N, et al. N2O emission and hydroxylamine oxidase (HAO) activity in a nitrogen removal process based on activated sludge with three COD/NH4+ ratios[J]. Water Environment Research, 2017, 89(5): 387-395. DOI:10.2175/106143017X14839994523668 |
| [85] |
BRADFORD M M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976, 72(1/2): 248-254. |
| [86] |
BURTON K. A study of the conditions and mechanism of the diphenylamine reaction for the colorimetric estimation of deoxyribonucleic acid[J]. Biochemical Journal, 1956, 62(2): 315. DOI:10.1042/bj0620315 |
| [87] |
兰淑澄, 司亚安, 朱燕民. 污水处理与回用新工艺——A/O系统在污水处理与回用中的应用[J]. 环境保护, 1988(1): 20-22, 9. |
| [88] |
POULIOT Y, BUELNA G, RACINE C, et al. Culture of cyanobacteria for tertiary wastewater treatment and biomass production[J]. Biological Wastes, 1989, 29(2): 81-91. DOI:10.1016/0269-7483(89)90089-X |
| [89] |
TAZIKI M, AHMADZADEH H, MURRY M A, et al. Nitrate and nitrite removal from wastewater using algae[J]. Current Biotechnology, 2015, 4(4): 426-440. |
| [90] |
GONÇALVES A L, PIRES J C, SIMõES M. A review on the use of microalgal consortia for wastewater treatment[J]. Algal Research, 2017, 24: 403-415. DOI:10.1016/j.algal.2016.11.008 |
| [91] |
LI X, HU H Y, ZHANG Y P. Growth and lipid accumulation properties of a freshwater microalga Scenedesmus sp. under different cultivation temperature[J]. Bioresource Technology, 2011, 102(3): 3098-3102. DOI:10.1016/j.biortech.2010.10.055 |
| [92] |
RENUKA N, SOOD A, RATHA S K, et al. Evaluation of microalgal consortia for treatment of primary treated sewage effluent and biomass production[J]. Journal of Applied Phycology, 2013, 25: 1529-1537. DOI:10.1007/s10811-013-9982-x |
 2024, Vol. 48
2024, Vol. 48


