文章信息
- 魏星, 张海静, 潘宝平. 2015.
- WEI Xing, ZHANG Hai-Jing, PAN Bao-Ping. 2015.
- 青蛤(Cyclina sinensis)IκB基因的克隆及其在鳗弧菌(Vibrio anguillarum)刺激下的表达分析
- CLONING OF IκB GENE FROM CYCLINA SINENSIS AND THE EXPRESSION ANALYSIS UNDER VIBRIO ANGUILLARUM STIMULATION
- 海洋与湖沼, 46(4): 793-799
- Oceanologia et Limnologia Sinica, 46(4): 793-799.
- http://dx.doi.org/10.11693/hyhz20150500147
-
文章历史
- 收稿日期:2015-05-24
- 改回日期:2015-06-08
青蛤(Cyclina sinensis)是我国常见的海产养殖贝类,因其肉质鲜美,营养丰富,目前已成为重要的海水养殖对象之一(高晶等,2015)。但是随着近年来国内青蛤养殖业的扩大,加之水质污染严重,青蛤由病原微生物引起的疾病时有发生(曹华,2004)。其中,鳗弧菌(Vibrio anguillarum)是引起海洋生物死亡的一种主要的革兰氏阴性菌,并且对青蛤有明显毒害作用(葛端阳等,2012)。由于青蛤缺乏特异性免疫系统,仅仅依赖其先天性免疫系统防御入侵的病原微生物。因此,对免疫信号通路相关分子的研究有助于更深入地了解软体动物的先天性免疫防御机制,为贝类养殖疾病防控提供新思路。
无脊椎动物为先天性免疫防御系统,也称为非特异性免疫(Iwanaga et al,2005)。非特异性免疫系统可以通过Toll样受体(Toll-like receptors,TLRs)对病原相关分子模式(PAMPs)的识别,从而激活下游信号通路,最终导致入侵病原体的清除(Janeway et al,2004)。所有的Toll样受体家族成员均依赖于TIR(Toll/IL-1R homologous region)结构域向细胞内转导其识别的信号。一类包含TIR结构域的接头分子如MyD88、TIRAP、TRIF、TRAM可募集到不同的Toll样受体(张炜等,2006),TLRs信号通路依据接头分子的不同可以分为MyD88依赖型信号通路和MyD88非依赖型信号通路(Akir et al,2004)。在TLRs 信号通路中,IκB(inhibitor of NF-κB)是一类重要的信号分子,是核转录因子κB(Nuclear factor-κB,NF-κB)的抑制蛋白(Vallabhapurapu et al,2009)。在没有受到刺激的细胞中,NF-κB二聚体通过与IκB结合而以失活状态存在于细胞质中。当受到外界刺激后,IκB通过磷酸化和泛素化而被蛋白酶降解,活化的NF-κB随即转位到核内与其相关的DNA基序结合以诱导靶基因的转录(Baldwin,1996)。
IκB家族成员存在三个特征结构域: 锚蛋白重复序列(ankyrin repeat,ANK),ANK是哺乳动物中与NF-κB蛋白相互作用的重要结构域; N端调控区,包含两个保守的丝氨酸残基及降解序列(degradation motif); 以及PEST结构域(PEST motif),富含脯氨酸、甘氨酸、丝氨酸、以及苏氨酸残基(Baldwin,1996)。在哺乳动物中IKBs有7种结构类型,其中 IκBа、IκBβ、IκBε这3种IκB含有N端调控区(郭伟等,2006)。近年来,在一些海洋双壳类软体动物中也发现了IκB基因,例如合浦珠母贝、海湾扇贝、菲律宾蛤仔、太平洋牡蛎、文蛤(Zhang et al,2009,2011; Mu et al,2010; Yang et al,2011; Lee et al,2013),它们对病原微生物刺激的研究结果表明,IκB基因参与这些生物的先天免疫信号转导。
1 材料与方法 1.1 实验青蛤青蛤采自于天津市大港滩涂,选取平均壳长(29.12±1.49)mm,平均壳高(29.86±1.57)mm,平均壳宽(19.56±0.47)mm的个体,暂养于通气海水中,水温20—23°C,海水密度1.020—1.040 g/cm3,海水pH 7.0,投喂5‰的小球藻,一周后进行试验。
1.2 菌种实验室保存的鳗弧菌用2216E培养基于28°C下培养24h。
1.3 侵染实验将培养好的鳗弧菌用无菌海水洗脱,浓度调至OD600=0.4(1OD = 1×108 bacteria/mL)。实验随机分组,每次设10个平行组。在注射前称取青蛤闭壳肌、鳃、肝脏、外套膜和性腺各50mg,并迅速放入装有液氮的预冷研钵中按1︰9的质量体积比加入预冷生理盐水并研磨,研磨后于4°C,4500 r/min离心15min,取上清液备用。实验组的每只青蛤注射50μL鳗弧菌菌液,空白组注射等量灭菌的生理盐水,对照组注射等量的灭菌海水。分别于注射后0h、3h、6h、9h、12h、24h、48h、96h提取血淋巴,并于4°C条件下,8000 r/min离心10min,收集血细胞,加入1mL Trizol,置于-80°C超低温冰箱以备使用。
1.4 转录组文库的构建及筛选Trizol法提取健康青蛤各个组织的总RNA,使用QIAGEN公司的Oligotex mRNA Kits法分离纯化总RNA。纯化后的mRNA用随机引物逆转录法合成cDNA。纯化的双链cDNA再经末端修复、加尾等步骤完成整个文库制备。采用Illumina MiSeq二代测序仪完成转录组测序。去冗余后的数据采用Unigene编码蛋白框ORF预测分析,并与NR数据库等BLAST比对分析,通过Blast2GO软件完成Unigene的GO注释。根据KEGG数据库注释并进一步对Unigene进行Pathway通路的注释和预测(Pan et al,2015),从中筛选得到青蛤IκB(CsIκB)基因类似序列。
1.5 生物信息学分析根据从转录组文库中筛选出的CsIκB基因类似序列设计特异性引物CsIκB-S、CsIκB-A(表 1)进行基因克隆并测序后与GenBank核酸数据库进行BLASTX(http: //www.ncbi.nlm.nih.gov)分析; ORF Finder(http: //www.ncbi.nlm.nih.gov/projects/gorf/)预测开放阅读框(ORF); ProtParam工具在线预测序列的分子式、分子量和等电点; ExPASy(http: //web.expasy. org/compute_pi/)分析蛋白质性质; SMART(http: //smart.embl-heidelberg.de/)查找蛋白质结构域; PEST find 用于预测PEST结构域(http: //www.at.embnet. org/toolbox/pestfind/),Proscan(http: //pbil.ibcp.fr/)用于推断酪蛋白激酶Ⅱ磷酸化位点。使用ClustalX1.83对氨基酸序列进行比对,根据邻接法(NJ),使用MEGA4.1构建基因系统进化树,采用bootstrap1000个循环检验拓扑结构的置信度。
1.6 Cs IκB基因在各组织内的表达分析
参照1.3方法,将青蛤肝脏、外套膜、闭壳肌、鳃和性腺的上清液与青蛤注射后0h的血细胞分别置于1mL TRIZOL中提取总RNA,并根据TaKaRa公司的PrimerScriptTM1st Str and cDNA Synthesis Kit使用说明反转录成cDNA,以cDNA为模板,β-actin基因为内参基因进行实时荧光定量PCR。根据β-actin基因和克隆得到的CsIκB基因序列分别设计引物为: β-actin F、β-actin R、CsIκB-F、CsIκB-R(表 1)。反应体系为20μL,在Rotor-Gene 6000实时定量PCR仪上进行,反应程序为: 95°C 30s; 94°C 5s,60°C 30s,72°C 30s,40个循环。采用2−ΔΔCT法计算Cs IκB基因表达量(Livak et al,2001),并使用SPSS软件进行数据分析。
| 引物名称 | 引物序列(5′—3′) |
| Cs IκB-S | GAGCAAGTGGACTCGGGTAT |
| Cs IκB-A | GTCAGTGGCAAGTTCAATCAA |
| β-actin F | CACCACAACTGCCGAGAG |
| β-actin R | CCGATAGTGATGA CCTGACC |
| Cs IκB-F | GAACAGCCCGAAGGTTACTC |
| Cs IκB-R | TCTAAACTTCCGCTTCTCAA |
分别利用1.2.1方法中鳗弧菌刺激后0h、3h、6h、12h、24h、48h、96h的血细胞提取总RNA,并反转录成cDNA进行实时荧光定量PCR,方法及引物同1.6。反应程序为: 95°C 30s; 95°C 5s,58°C 30s,72°C 30s,40个循环。采用2−ΔΔCT法处理数据,使用SPSS软件对同一时间点的实验组和对照组以及实验组与空白组的表达量进行单因素方差分析。
2 结果 2.1 Cs IκB 基因的序列分析从青蛤转录组文库筛选得到CsIκB基因类似序列,设计引物克隆并测序,BLASTX进行序列比对及ORF分析后结果显示CsIκB基因cDNA序列ORF为1134bp,预测编码377个氨基酸(图 1),分子量为41.79 kDa,等电点为4.98,氨基酸组成中亮氨酸(Leu)含量最高,占10.6%。C端存在6个ANK。N端存在DS64GILS68序列,与保守的DSGXXS降解序列一致。利用PEST序列查找软件分析表明,CsIκB不存在PEST结构域,但是C端存在保守的酪蛋白激酶Ⅱ磷酸化位点(S373DDES377),通常位于PEST结构域。Cs IκB基因在GenBank中的注册号为KP067202。
 |
| 图 1 CsIκB基因的cDNA序列的开放阅读框及推导的氨基酸序列 Fig. 1 The cDNA sequence of CsIκB ORF and its deduced amino acid sequence方框且加粗部分代表起始密码子,*代表终止密码子,下划线代表降解域,圆圈部分代表保守的丝氨酸残基,灰色底纹部分代表 6个ANK,方框代表酪蛋白激酶Ⅱ磷酸化位点 |
经GenBank BLASTX研究表明,Cs IκB基因氨基酸序列与菲律宾蛤仔IκB(Ruditapes philippinarum,ACF93446)基因的氨基酸序列同源性最高,达到59%,与海蜗牛(Aplysia californica,XP_005111546)、长牡蛎(Crassostrea gigas,ADX06856)、布氏新亮丽鲷(Neolamprologus brichardi,XP_006792655)、合浦珠母贝(Pinctada fucata,ACF93446)和红鲍(Haliotis rufescens,AGZ03662)IκB基因的氨基酸序列同源性为40%、40%、41%、42%和44%。利用无脊椎动物和脊椎动物代表性的IκB氨基酸序列构建的系统进化树显示(图 2),无脊椎动物与脊椎动物的IκB氨基酸序列出现一个明显的进化分歧。CsIκB基因与菲律宾蛤仔IκB基因聚为一支,与其它无脊椎动物IκB基因聚类。
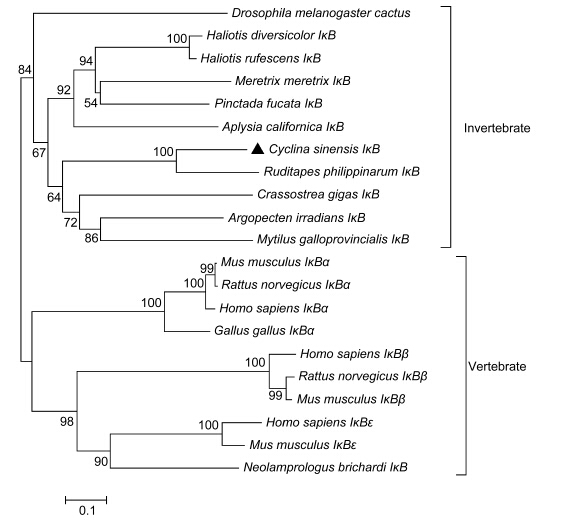 |
| 图 2 CsIκB和其它物种的IκB氨基酸序列构建的系统树 Fig. 2 The phylogenetic tree constructed by the amino acid sequence of CsIκB and IκBs from other species 建立系统树所用的物种以及序列号为: Drosophila melanogaster(AAA85908); Haliotis diversicolor(AHM27300); Haliotis rufescens(AGZ03662); Meretrix meretrix(ADK74377); Pinctada fucata(ACF93446); Aplysia californica(XP_005111546); Ruditapes philippinarum(AEB92230); Crassostrea gigas(ADX06856); Argopecten irradians(ACZ34178); Mytilus galloprovincialis(AHI17300); Homo sapiens IκBа,IκBβ,IκBε(NP_065390,AAH15528,AAC51216); Mus musculus IκBа,IκBβ,IκBε(AAH46754,AAH21938,AAH30923); Rattus norvegicus IκBа,IκBβ(Q63746,AAH85729); Neolamprologus brichardi(XP_006792655); Cyclina sinensis(KP067202) |
通过实时荧光定量PCR技术检测CsIκB基因在青蛤不同组织中的表达情况,结果显示,CsIκB基因在所检测的血淋巴、肝脏、外套膜、闭壳肌、鳃和性腺6种组织中均有表达,并且在血淋巴中表达量最高,其次为闭壳肌和性腺,而在肝脏中表达量最低(图 3)。
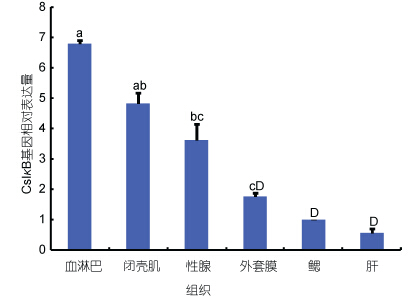 |
| 图 3 CsIκB基因在青蛤不同组织中的表达分布Fig. 3 The distribution of CsIκB expression in different tissues of C. sinensis 不同小写字母代表差异显著(P<0.05),不同大写字母代表差异极显著(P<0.01) |
青蛤在鳗弧菌侵染刺激后,在青蛤血淋巴中的CsIκB基因的时序性表达情况如图 4所示,实验组的表达量在刺激后3h迅速升高,与对照组及空白组相比均有极显著性差异(P<0.01); 在6h时表达量达到最大值,约为对照组的2.3倍左右,并且与对照组及空白组均差异极显著(P<0.01)。表达量于6h后开始下降,并逐渐接近正常水平。对照组没有显著性变化。
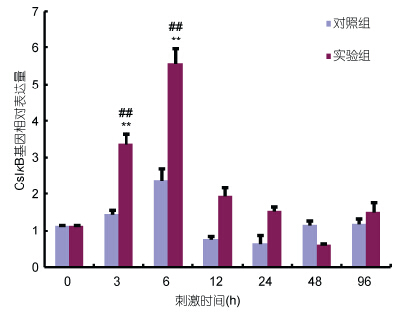 |
| 图 4 鳗弧菌刺激下在青蛤血淋巴中CsIκB基因的时序性表达情况Fig. 4 The temporal expression of CsIκB in hemocytes after V. anguillarum stimulation ##代表该时间点实验组基因的表达量同组注射前(0h)相比差异极显著(P<0.01),**代表相同时间点实验组与对照组基因的表达量差异极显著(P<0.01) |
本研究从青蛤转录组文库中筛选并进一步克隆得到了TLRs通路信号分子CsIκB的cDNA序列。分子系统学分析表明,CsIκB在从无脊椎动物到脊椎动物不同物种的进化上极为保守,青蛤、菲律宾蛤仔海湾扇贝等软体动物动物的IκB聚为一类。由此推测,这些软体动物IκB蛋白有相似的生物学功能,在其先天性免疫防御中起着重要作用。CsIκB基因包含6个ANK,降解域以及保守的酪蛋白激酶Ⅱ磷酸化位点。研究发现合浦珠母贝、海湾扇贝、菲律宾蛤仔、太平洋牡蛎(Zhang et al,2009,2011; Mu et al,2010; Lee et al,2013)等的IκB基因也存在6个ANK,然而文蛤的IκB基因存在5个ANK(Yang et al,2011)。ANK数量的差异可能与其相互作用的NF-κB/Rel蛋白的不同有关。
根据前人对哺乳动物IκB基因的研究表明,降解域中丝氨酸残基的磷酸化是由多种细胞外刺激介导的(Luque et al,2000)。特别是IKK激活IκB的降解域,导致泛素化和蛋白酶体降解(Schmid et al,2008)。CsIκB基因N端存在保守的降解域(DS64GILS68),表明CsIκB可能同样是由IKK和NF-κB调节的。IκB基因的C端通常存在PEST结构域,会导致含有它们的细胞内蛋白的快速降解( Rogers et al,1986)。然而CsIκB基因与一些软体动物的IκB基因一样缺少PEST结构域,如合浦珠母贝、海湾扇贝、太平洋牡蛎(Zhang et al,2009,2011; Mu et al,2010)。但是CsIκB的C端存在酪蛋白激酶Ⅱ磷酸化位点(S373DDES377),说明也许一些IκB基因的降解不需要PEST结构域,而是酪蛋白激酶Ⅱ磷酸化位点起到关键作用。
哺乳动物IκB蛋白在不同组织的表达差异是由于它们对NF-κB二聚体的亲和力不同所致(Hoffmann et al,2006)。也是由于NF-κB参与多种生理过程,如免疫反应、炎症反应、细胞增殖和凋亡、细胞应激反应以及组织重建(Li et al,2002)。本研究通过实时定量PCR技术分析了CsIκB基因在青蛤不同组织中的表达情况,结果显示CsIκB基因在青蛤血淋巴、肝脏、外套膜、闭壳肌、鳃和性腺6种组织中均有表达但表达量不同,表明NF-κB基因作为一种多效性的转录因子在各个组织中被屏蔽的程度不同。CsIκB基因在血淋巴中的表达量最高,该结果与合浦珠母贝poI-κB、菲律宾蛤仔Rp-IκB、太平洋牡蛎IκB1(Zhang et al,2009,2011; Lee et al,2013)在血淋巴中的表达情况相似。血淋巴是软体动物免疫系统的重要组成成分,介导了软体动物内部防御中对于入侵病原微生物的吞噬和包埋作用(Humphries et al,2003)。
为了揭示CsIκB基因在青蛤免疫体系中的作用机制,本研究进一步检测了青蛤血淋巴中CsIκB基因在鳗弧菌刺激不同时间后的表达量,结果表明; CsIκB基因在刺激后3h上调,6h后其表达量开始下降,并逐渐恢复至正常水平。与合浦珠母贝、菲律宾蛤仔、太平洋牡蛎、文蛤(Zhang et al,2009,2011; Yang et al,2011; Lee et al,2013)的研究结果类似,说明CsIκB基因参与了青蛤的先天性免疫信号转导。CsIκB的诱导机制可能是通过TLRs信号通路识别病原微生物,从而引发下游信号级联反应(Kratsovnik et al,2005)。已有研究显示,在栉孔扇贝中存在一条原始的MyD88依赖型TLRs信号通路(Wang et al,2011)。目前我们已克隆得到青蛤TLR2基因,以及MyD88依赖型TLRs信号通路中最关键的接头分子MyD88,并证明它们参与了青蛤的先天性免疫反应(任毅鹏等,2014; 高晶等,2015),本研究克隆得到TLRs信号通路下游重要的信号分子CsIκB基因的cDNA序列,这些研究表明青蛤可能存在一条TLRs信号通路并且该通路参与了青蛤对于鳗弧菌刺激的免疫应答。
| 任毅鹏, 高 晶, 潘宝平等, 2014. 青蛤(Cyclina sinensis)TLR2基因的克隆与表达分析. 海洋与湖沼, 45(5): 1037—1043 |
| 张 炜, 袁金铎, 安利国, 2006. 由不同接头分子介导的Toll样受体信号通路. 细胞与分子免疫学杂志, 22(4): 544—546 |
| 高 晶, 任毅鹏, 潘宝平等, 2015. 鳗弧菌(Vibrio anguillarum)侵染对青蛤(Cyclina sinensis)髓样分化因子88基因表达的影响. 海洋与湖沼, 46(2): 440—445 |
| 郭 伟, 王 奇, 陈云波, 2006. 细胞核因子κB信号转导途径内的活化与调节机制研究进展. 实用医学杂志年, 22(11): 1332—1334 |
| 曹 华, 2004. 沿海滩涂青蛤死亡原因及对策. 中国水产, (4): 57—58 |
| 葛端阳, 赵 婷, 潘宝平, 2012. 鳗弧菌(Vibrio anguillarum)对青蛤(Cyclina sinensis)的毒性及半致死浓度研究. 海洋与湖沼, 43(6): 1192—1195 |
| Akir S, Takeda K, 2004. Toll-like receptor signalling. Nat Rev Immunol, 4(7): 499—511 |
| Baldwin Jr A S, 1996. The NF-κB and IκB proteins: new discoveries and insights. Annu Rev Immunol, 14: 649—681 |
| Hoffmann A, Natoli G, Ghosh G, 2006. Transcriptional regulation via the NF-kappaB signaling module. Oncogene, 25(51): 6706—6716 |
| Humphries J E, Yoshino T P, 2003. Cellular receptors and signal transduction in molluscan hemocytes: connections with the innate immune system of vertebrates. Integr Comp Biol 43(2): 305–312 |
| Iwanaga S, Lee B L, 2005. Recent advances in the innate immunity of invertebrate animals. J Biochem Mol Biol, 38(2): 128—150 |
| Janeway Jr C A, Medzhitov R, 2004. Innate immune recognition. Annu Rev Immunol, 20: 197–216 |
| Kratsovnik E, Bromberg Y, Sperling O et al, 2005. Oxidative stress activates transcription factor NF-κB-mediated protective signaling in primary rat neuronal cultures. J Mol Neurosci, 26(1): 27—32 |
| Lee Y, Wickamarachchi W D, Whang I et al, 2013. Immune response-related gene expression profile of a novel molluscan IkB protein member from Manila clam (Ruditapes philippinarum). Mol Biol Rep, 40(2): 1519—1527 |
| Li X X, Stark G R, 2002. NFκB-dependent signaling pathways. Exp Hematol, 30(4): 285—296 |
| Livak K J, Schmittgen T D, 2001. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCTmethods. Methods, 25(4): 402—408 |
| Luque I, Zong W X, Chen C L et al, 2000. N-terminal determinants of IκBα necessary for the cytoplasmic regulation of c-Rel. Oncogene, 19(9): 1239—1244 |
| Mu C, Yu Y, Zhao J et al, 2010. An inhibitor kappaB homologue from bay scallop Argopecten irradians. Fish Shellfish Immunol, 28(4): 687—694 |
| Pan B P, Ren Y P, Gao J et al, 2015. De novo RNA-Seq analysis of the Venus clam, Cyclina sinensis, and the identification of immune-related genes. PLoS One, 10(4): e123296 |
| Rogers S, Wells R, Rechsteiner M, 1986. Amino acid sequences common to rapidly degraded proteins: the PEST hypothesis. Science, 234(4774): 364—368 |
| Schmid J A, Birbach A, 2008. IκB kinase β (IKKβ/IKK2/IKBKB)-A key molecule in signaling to the transcription factor NF-κB. Cytokine Growth Factor Rev, 19(2): 157—165 |
| Vallabhapurapu S, Karin M, 2009. Regulation and function of NF-kappaB transcription factors in the immune system. Annu Rev Immunol, 27: 693—733 |
| Wang M Q, Yang J L, Zhou Z et al, 2011. A primitive Toll-like receptor signaling pathway in mollusk Zhikong scallop Chlamys farreri. Dev Comp Immunol, 35(4): 511—520 |
| Yang Q, Yang Z, Li H, 2011. Molecular characterization and expression analysis of an inhibitor of NF-kB (IkB) from Asiatic hard clam Meretrix meretrix. Fish Shellfish Immunol, 31(3): 485—490 |
| Zhang D, Jiang S, Qiu L et al, 2009. Molecular characterization and expression analysis of the IkB gene from pearl oyster Pinctada fucata. Fish Shellfish Immunol, 26(1): 84—90 |
| Zhang Y, He X, Yu Z, 2011. Two homologues of inhibitor of NF-kappa B (IkB) are involved in the immune defense of the Pacific oyster, Crassostrea gigas. Fish Shellfish Immunol, 30(6): 1354—1361 |
 2015, Vol. 46
2015, Vol. 46


