
中国海洋湖沼学会主办。
文章信息
- 黄媛, 方序, 褚文珂, 陈敏. 2015.
- HUANG Yuan, FANG Xu, CHU Wen-Ke, CHEN Min. 2015.
- 杭州西溪湿地沉积物细菌的群落结构和多样性
- BACTERIAL DIVERSITY AND COMMUNITY STRUCTURE IN SEDIMENTS OF XIXI WETLAND, HANGZHOU
- 海洋与湖沼, 46(5): 1202-1209
- Oceanologia et Limnologia Sinica, 46(5): 1202-1209.
- http://dx.doi.org/10.11693/hyhz20150700192
-
文章历史
- 收稿日期: 2015-07-21
- 收修改稿日期: 2015-07-30
2. 浙江省微生物研究所 杭州 310012
2. Zhejiang Institute of Microbiology, Hangzhou 310012, China
湿地是位于水陆交界处形成的独特的生态系统。湿地生态系统中,沉积物是一个由各种微生物参与的、物质发生频繁交换的、具有高度生物活性的特殊生境(徐长君等,2009)。沉积物中微生物的多样性对整个水体系统有重要影响(何建瑜等,2013)。与水体中的悬浮微生物比较,沉积物微生物往往在单位数量和功能多样性上更为丰富(Klammer et al,2002),因此对水体生态系统平衡的重要性也更大。目前,沉积物微生物正受到越来越多研究者的关注(任丽娟等,2013)。Li等(2011)利用荧光原位杂交方法(fluorescence in situ hybridization,FISH)和变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术,研究了海底表层沉积物中细菌的群落结构,发现属于β-变形菌纲(β-Proteobacteria)的氨氧化菌(AOB)的丰度达到了(1.87—3.53)×105 cells/g,其群落结构组成与沉积物的盐度、温度、呼吸作用和总有机碳(TOC)等因子密切相关,可以作为海域沉积物硝化作用的间接指示因子。时玉等(2014)利用定量PCR(qPCR)和变性梯度凝胶电泳(DGGE)技术,对青藏高原淡水湖普莫雍错和盐水湖阿翁错湖底沉积物进行细菌多样性的比较研究,结果发现青藏高原淡水与盐水湖泊沉积物细菌丰度与群落结构具有明显的差异; 同时,沉积物细菌群落结构在不同深度也表现出差异。
杭州西溪国家湿地公园位于杭州市区西部,是在自然湿地基础上由一千多年农渔耕作用下形成的罕见的城中次生湿地,曾经的稻、桑(柿)、鱼、蚕的农业模式是西溪湿地的一大特色。近20多年来,随着经济和社会的发展,西溪湿地的面积由原来的60km2 萎缩到现在的不足12km2,并且因为人类活动的日趋频繁,湿地的生态系统也遭到了不同程度的破坏(陈久和,2003)。在此背景下,2003年正式启动了西溪湿地综合保护工程,到2008年保护工程一、二、三期基本建成并投入使用。西溪湿地植物园是西溪国家湿地公园二期建设的一部分,其目标是通过恢复一定比例的湿地植物群落,将原来大面积的鱼塘-基-渚湿地演变成典型的湿地景观,形成自然的湿生生态系统,这对于西溪湿地的保护和修复有着重要的实践意义。本文以西溪湿地植物园区域内不同水生植物生境下的表层沉积物为研究对象,采用MiSeq高通量测序的方法,研究沉积物中细菌的群落特征和多样性,以期为丰富西溪湿地生态系统内涵,开发西溪湿地的微生物资源,进而为西溪湿地的保护和修复提供理论和实践依据。
1 材料与方法 1.1 沉积物样品沉积物样品于2014年6月采自杭州西溪国家湿地公园保护区湿地植物园野外观测样地(120°4′18″E,130°16′26″N)。湿地植物园的群落构建遵循了优势种培育模式,即在大面积宽阔的水域大量种植一种或数种水生植物,发展大面积的优势群落。选择的水生植物主要包括:(1)挺水植物群落(蒲苇,白茅等);(2)浮水植物群落(睡莲,凤眼莲等);(3)沉水植物群落(金鱼藻,狐尾藻等);(4)湿生植物群落(美人蕉,黄菖蒲等)。以上述4类不同水生植物生境下的表层沉积物(0—10cm)为研究对象,每种类型的样品进行3个重复采样,即每个样点距离2m以上随机取点,混匀后样品编号为1—4号。采集后的样品迅速放入无菌保鲜袋中,-20°C保存。
1.2 土壤理化性质测定沉积物pH值采用测定其间隙水的方法,称取数克沉积物样品,离心,取上清液,用pH计测定其pH值。总有机碳含量采用Analytik multi N/C 3100分析仪测定; 总氮含量采用EURO EA元素分析仪测定; 总磷含量采用H2SO4-HClO4双酸消煮-钼锑抗比色法测定(赵亚杰等,2015)。
1.3 高通量测序及数据分析 1.3.1 MiSeq高通量测序用EZNA Soil DNA Kit(OMEGA公司)提取基因组DNA,对16S rRNA V3、V4区进行扩增,引物: 338F(5′-ACTCCTACGGGAG GCAGCA-3′); 806R(5′-GGACTACHVGGGTWTCTA AT-3′)。PCR扩增反应体系(20μL): 5×FastPfu Buffer 4μL,2.5mmol/L dNTPs 2μL,Forward Primer(5μmol/L)0.4μL,Reverse Primer(5μmol/L)0.4μL,FastPfu Polymerase 0.4μL,Template DNA 10ng。反应条件: 94°C预变性120s,94°C变性30s,55°C退火30s,72°C延伸45s; 28个循环。每个样品3个重复,将同一样品的PCR产物混合后用2%琼脂糖凝胶电泳检测,AxyPrep DNA凝胶回收试剂盒切胶回收PCR产物,Tris-HCl洗脱,2%琼脂糖电泳检测。
参照电泳初步定量结果,将PCR产物用QuantiFluor™-ST蓝色荧光定量系统(Promega公司)进行检测定量,之后按照每个样品的测序量要求,进行相应比例的混合,委托上海美吉生物医药科技有限公司进行Illumina MiSeq高通量测序(李靖宇等,2014)。
1.3.2 测序数据优化处理为了保证后续生物信息学分析的准确性,对测序数据进行质量控制。通过过滤read尾部质量值20以下的碱基,利用Trimmomatic、FLASH等软件筛选拼接序列的overlap区错配比率低于0.2,barcode错配数为0,最大引物错配数为2的优化序列进行后续分析。
1.3.3 OTU聚类分析及分类学分析利用Usearch(vsesion 7.1 http: //qiime.org/)软件平台,在97%的相似水平对所有序列进行可操作分类单元(OUT)划分。为了得到每个OTU对应的物种分类信息,采用RDP法(Wang et al,2007)(置信度阈值为0.7)对97%相似水平的OTU代表序列进行分类学分析。比对数据库为16S细菌数据库: Silva(Release115 http: //www.arb- silva.de)。
1.3.4 群落a多样性分析基于优化处理的OTUs及相关分析软件Mothur(version v.1.30.1 http: //www. mothur.org/wiki/Schloss_SOP#Alpha diversity)绘制了各样品的稀释曲线,并在重取样的基础上计算了种群丰富度指数Chao值(http: //www.mothur.org/wiki/ Chao)和物种多样性指数Shannon-Wiener指数(http: //www.mothur.org/wiki/Shannon)。
2 结果与分析 2.1 沉积物理化性质沉积物中的总有机碳、总氮和总磷等含量是反映营养状况和污染程度的双重指标。表 1结果显示,4个沉积物样品的理化特性有较大的差异,其中总有机碳和总氮最高的是1号挺水植物生境样品,其次是4号湿生植物生境样品; 而总磷最高的是4号湿生植物生境样品,其次是1号挺水植物生境样品。2号浮水植物和3号沉水植物生境样品无论是总有机碳、总氮还是总磷明显低于1号挺水、4号湿生植物生境样品。样品的pH均偏酸性,其中2号浮水植物生境样品的pH为5.17,酸性最强。
| 样品编号 | 采样地 | pH | 总有机碳(g/kg) | 总氮(g/kg) | 总磷(g/kg) |
| 1 | 挺水植物区 | 6.60±0.09 | 49.313±1.003 | 9.423±0.057 | 0.665±0.015 |
| 2 | 浮水植物区 | 5.17±0.11 | 20.073±0.656 | 4.900±0.065 | 0.245±0.004 |
| 3 | 沉水植物区 | 6.43±0.19 | 18.157±0.363 | 4.726±0.069 | 0.555±0.021 |
| 4 | 湿生植物区 | 6.44±0.15 | 31.250±0.565 | 6.593±0.064 | 0.910±0.009 |
采用RDP法对97%相似水平的OTU代表序列进行分类学分析,在数据库中没有相应分类单元的序列,以norank标记。结果表明,沉积物样品具有很高的细菌多样性。在门的水平有变形菌门(Proteobacteria)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、硝化螺旋菌门(Nitrospirae)、酸杆菌门(Acidobacteria)、绿菌门(Chlorobi)、螺旋菌门(Spirochaetae)、梭杆菌门(Fusobacteria)、放线菌门(Actinobacteria)、疣微菌门(Verrucomicrobia)和芽单胞菌门(Gemmatimonadetes)等30个门(数据未显示)。在属的水平上共鉴定有252个属(图 1所示,已将丰度极低的部分合并为other在图中显示),其中丰度较高且所有样品均有分布的主要有铁杆菌属(Ferribacterium)、乳球菌属(Lactococcus)、厌氧粘细菌属(Anaeromyxobacter)、硫杆菌属(Thiobacillus)、假单胞菌属(Pseudomonas)、螺旋体属(Spirochaeta)、硫碱螺旋菌属(Thioalkalispira)、Sulfuritalea属、泉发菌属(Crenothrix)、互养菌属(Syntrophus)和Pseudorhodoferax属。1号和4号的优势类群为铁杆菌属,含量分别为8.29%和12.30%; 2号和3号的优势类群是乳球菌属,含量分别为13.52%和8.87%。有些细菌类群仅在特定的样品中出现,如1号样品特有的菌群包括醋菌属(Actibacter)、贝日阿托氏菌属(Beggiatoa)和Ferruginibacter属等6个属; 2号样品特有的菌群有铁氧化细菌(Ferritrophicum)、纤维堆囊菌(Sorangium)和新衣原体属(Neochlamydia)等10个属; 3号样品特有的菌群只有蛭弧菌属(Bdellovibrio); 4号样品特有的菌群有甲基暖菌属(Methylocaldum)和Propionivibrio属。这些特有的菌群通常丰度较低。此外,尚有未培养(Uncultured)和未分类(Unclassified)细菌类群,前者占比在30%—50%,后者占比在10%—20%。说明西溪湿地沉积物中蕴藏有较多的潜在新物种。
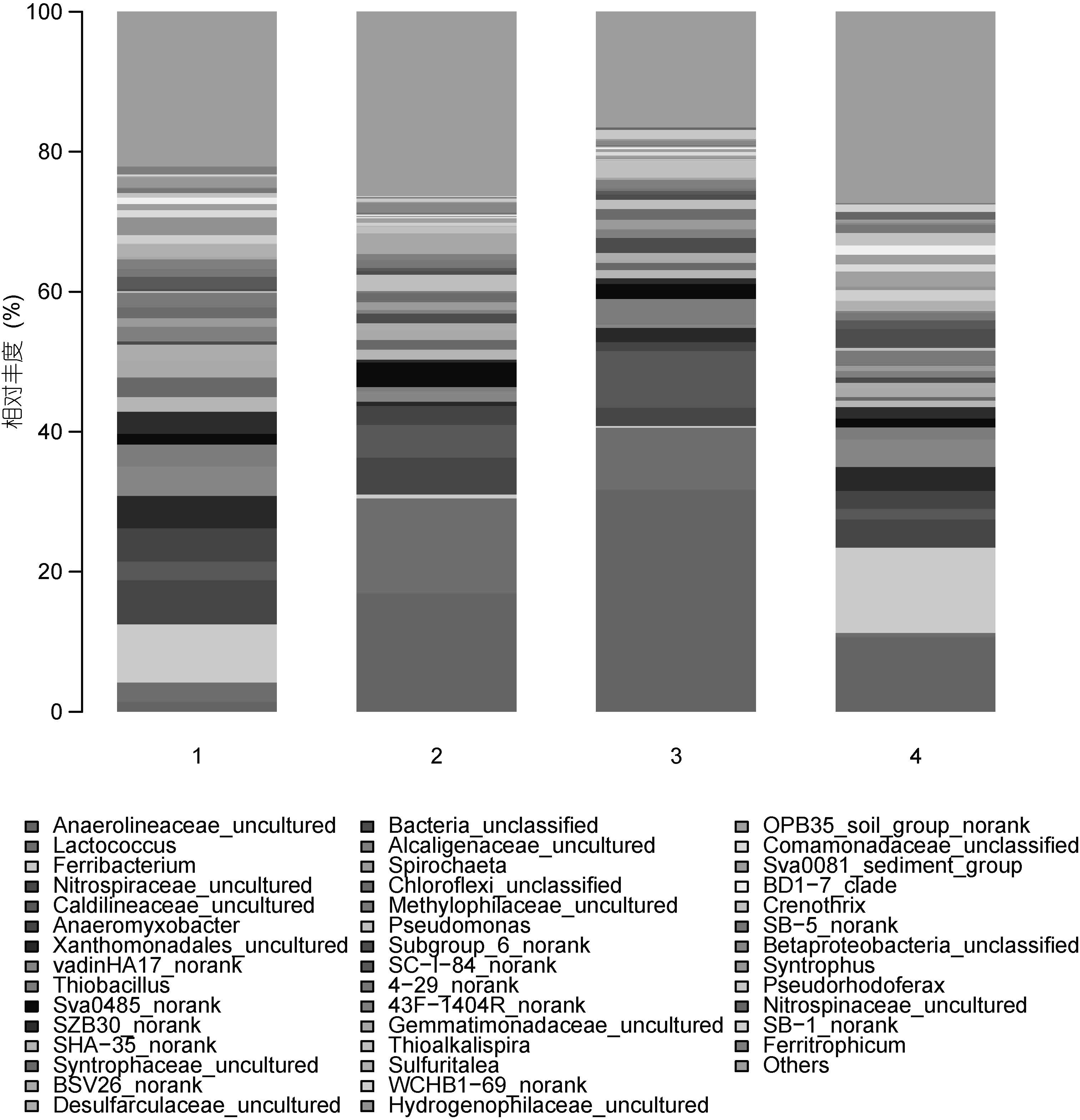 |
| 图 1 各样品优势细菌群落结构Fig. 1 Dominant bacterial community of each sample at the genus level |
利用高通量测序技术,过滤掉低质量的序列后,4个样品共获得有效序列67734条,根据barcode标签进行样品序列拆分,并对初始序列进行去冗余处理以获得16S rDNA Unique Reads,在97%相似度下将其聚类为用于物种分类的OTU,统计各样品在不同OTU 中的丰度信息,4个样品共产生2181个OTU,经优化处理后,得到平均长度为441 bp的序列,其中片段长度>400 bp的序列数占到了总序列数的99.74%。各样品Reads和OTU数量如表 2所示。
| 样品编号 | 重取样前 | 重取样后 | 多样性指数 | |||
| 读数 | OTU | 读数 | OTU | Chao | Shannon | |
| 1 | 7544 | 496 | 4823 | 454 | 542 | 5.04 |
| 2 | 12917 | 680 | 4823 | 567 | 636 | 5.32 |
| 3 | 4823 | 472 | 4823 | 472 | 568 | 5.06 |
| 4 | 7122 | 533 | 4823 | 483 | 569 | 5.20 |
采用对测序序列进行随机抽样的方法,以抽到的序列数与它们所能代表的数目构建稀释性曲线(图 2)。从图 2中可以看出,4个样品的稀释曲线在0.97相似性水平下趋于平坦,但仍未达到饱和。因此,为了判断这些数据量是否合理,绘制了各样品的Shannon- Wiener曲线(图 3),结果显示,开始时曲线直线上升,是由于测序条数远不足覆盖样品; 当测序数值增至2000以上时,曲线趋向平坦并达到了平台期,说明4个样品的测序量足以覆盖样品中的绝大部分微生物。
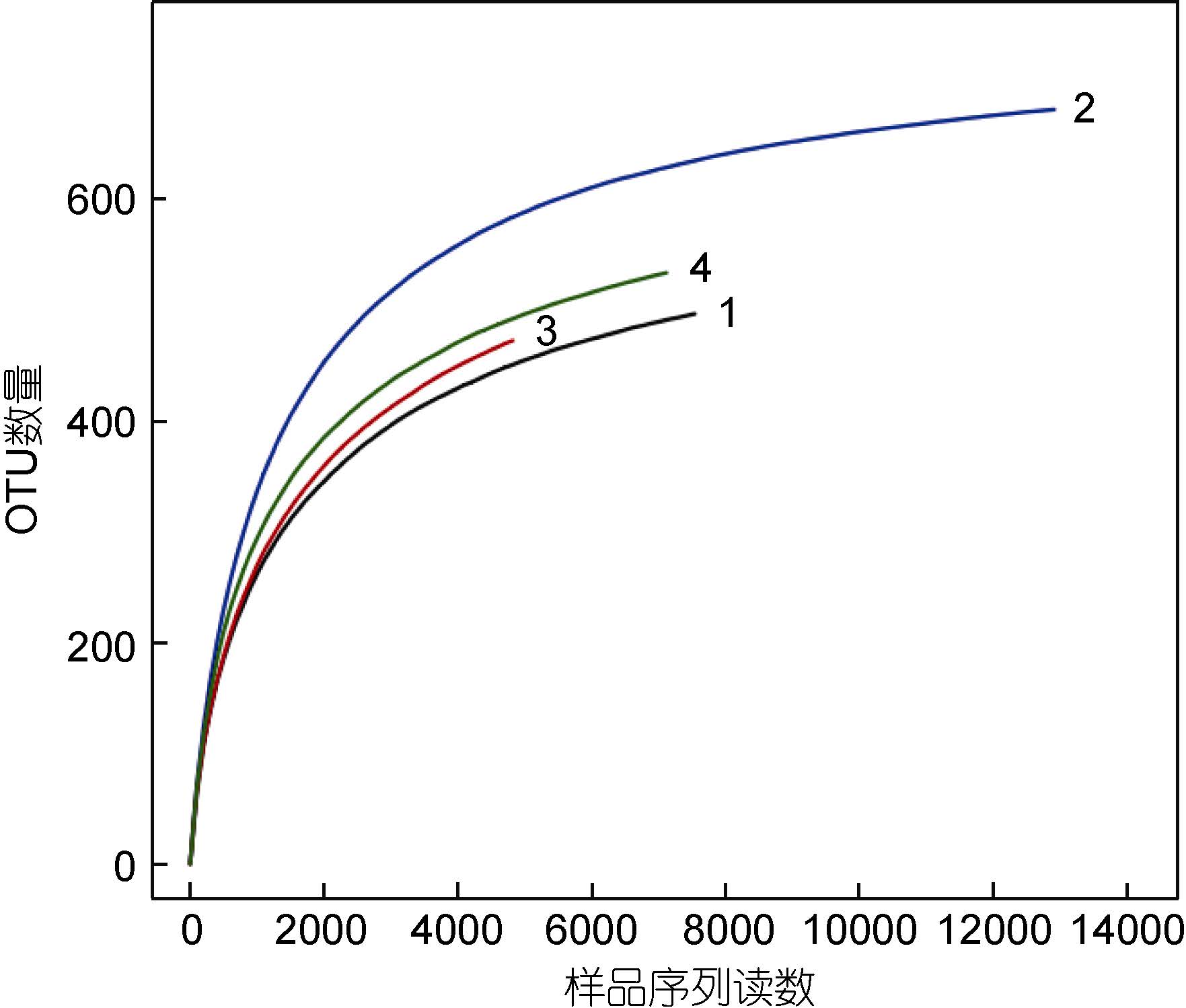 |
| 图 2 各样品的稀释性曲线Fig. 2 Rarefaction curves of each sample |
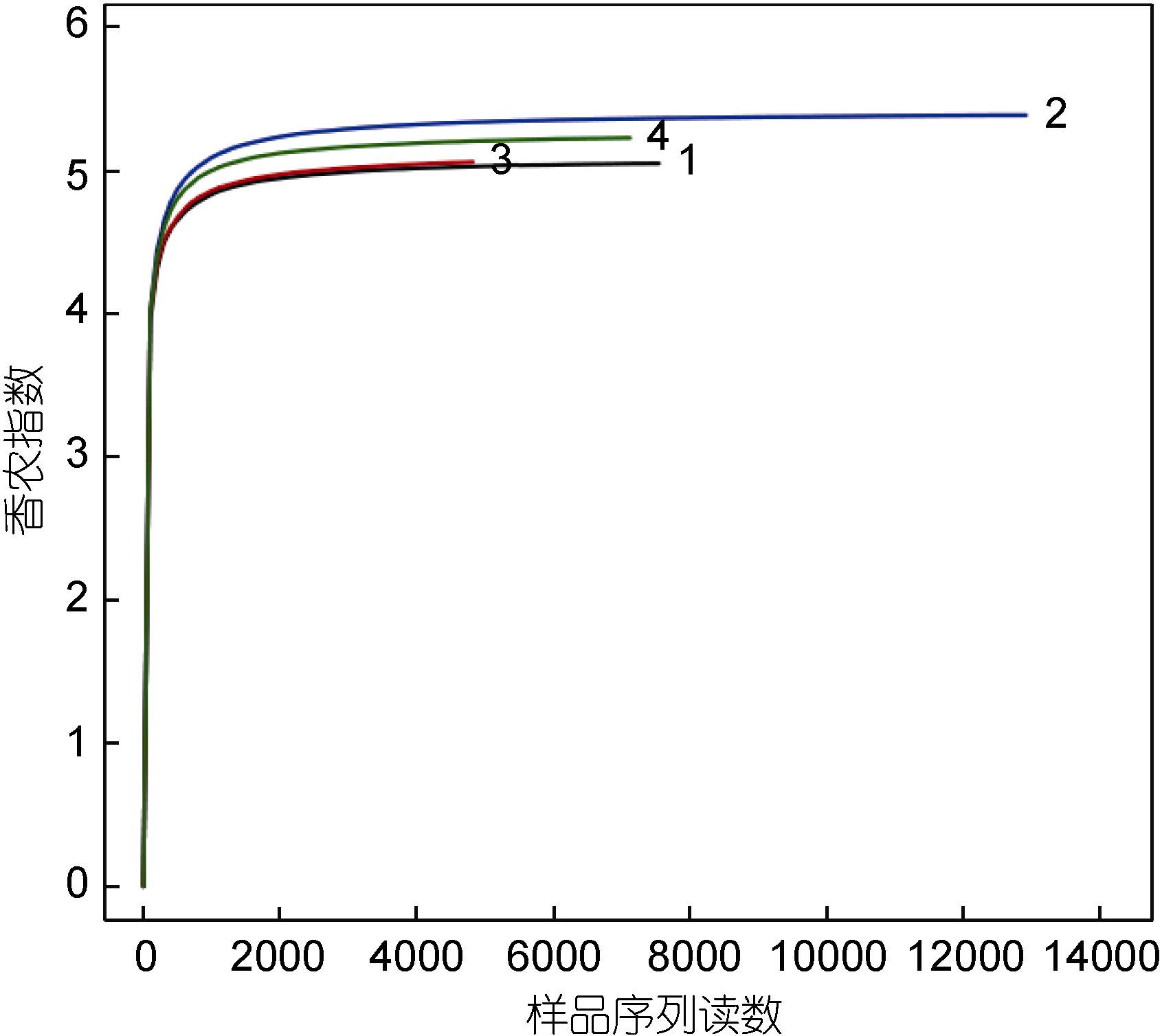 |
| 图 3 各样品微生物Shannon-Wiener曲线Fig. 3 Shannon-Wiener curves of each sample |
由于测序系统本身的缺陷,虽然在上样测序前,已对各样品的3个重复PCR产物进行了混合,但每个样品所测出的序列数目仍不一致(表 2)。而OTU数和Chao指数会受到序列数的较大影响,因此有必要对每个样品进行重取样。根据图 3的Shannon指数研究结果,确定每个样品随机重取样序列数为4823条。通过Mothur软件计算各样品的α-多样性指数,指数值越大,表明细菌群落多样性越高。结果显示,4个样品的Chao 指数和Shannon指数均较大,其中2号样品的丰度最高多样性最高,4号样品其次,1号和3号样品相对较低。
2.4 沉积物样品细菌群落结构相似性比较对4个样品的OTU进行统计,其中1号样品454个,2号样品567个,3号样品472,4号样品483个。维恩图结果显示(图 4),4个样品共有的OTU为224个,占总数的11.34%。1号和4号样品间共有的OTU为264个,2号和3号样品间共有的OTU为293个,比其他样品间共有OTU数目都要多,表明1号与4号样品,2号与3号样品之间的细菌群落结构较为相似。
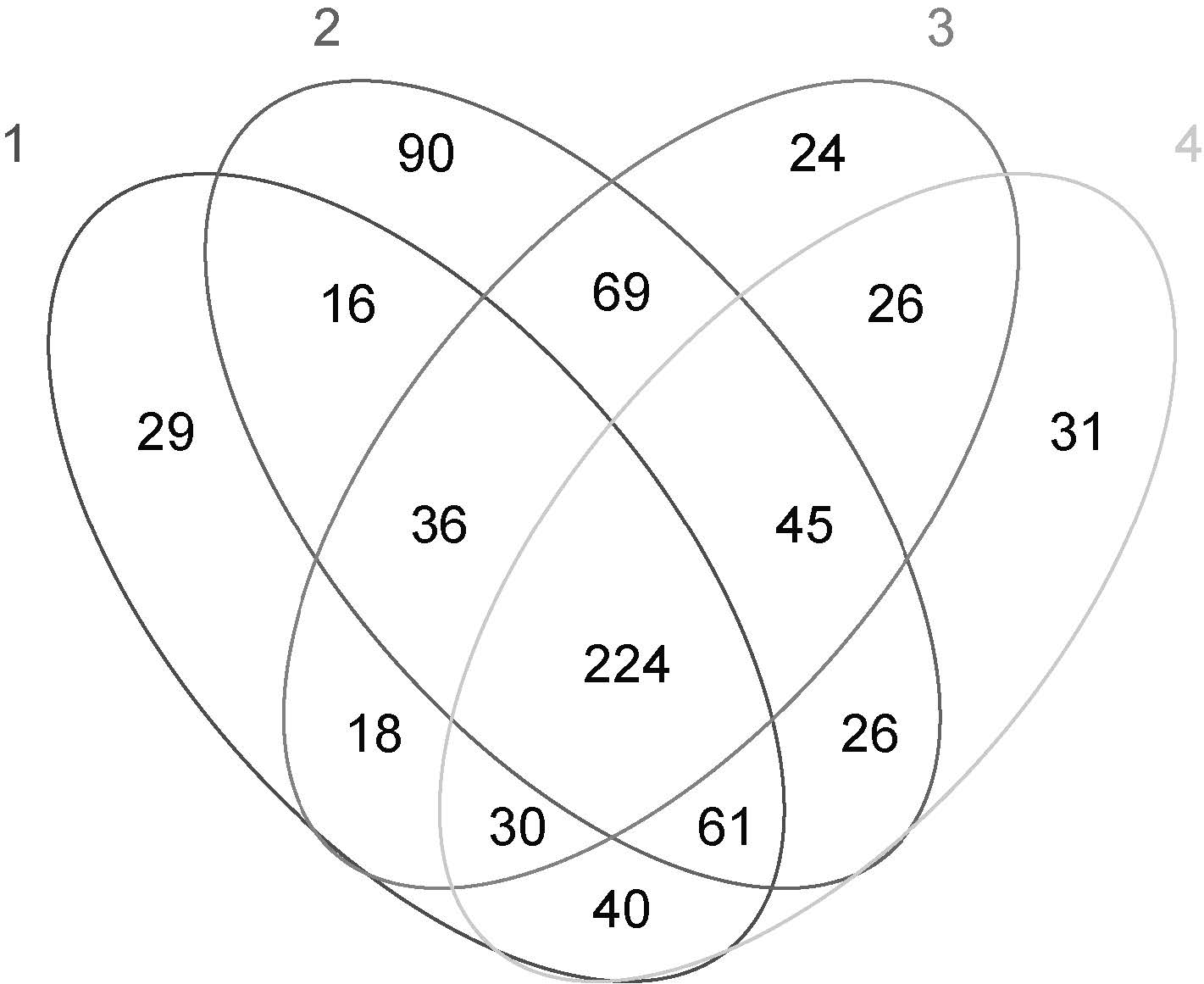 |
| 图 4 OTU分布Venn图Fig. 4 Venn diagram showing the unique and shared OTUs (3% distance level) |
同样,利用树枝结构描述和比较4个样品间的相似性和差异关系(图 5),结果表明,1号和4号样品的细菌群落结构组成及丰度相对接近而聚在一个分枝,同样2号和3号样品相似性较高聚在另一分枝上,末端竖线表示样品聚在一起相似度较高。此分析结果与维恩图结果一致。
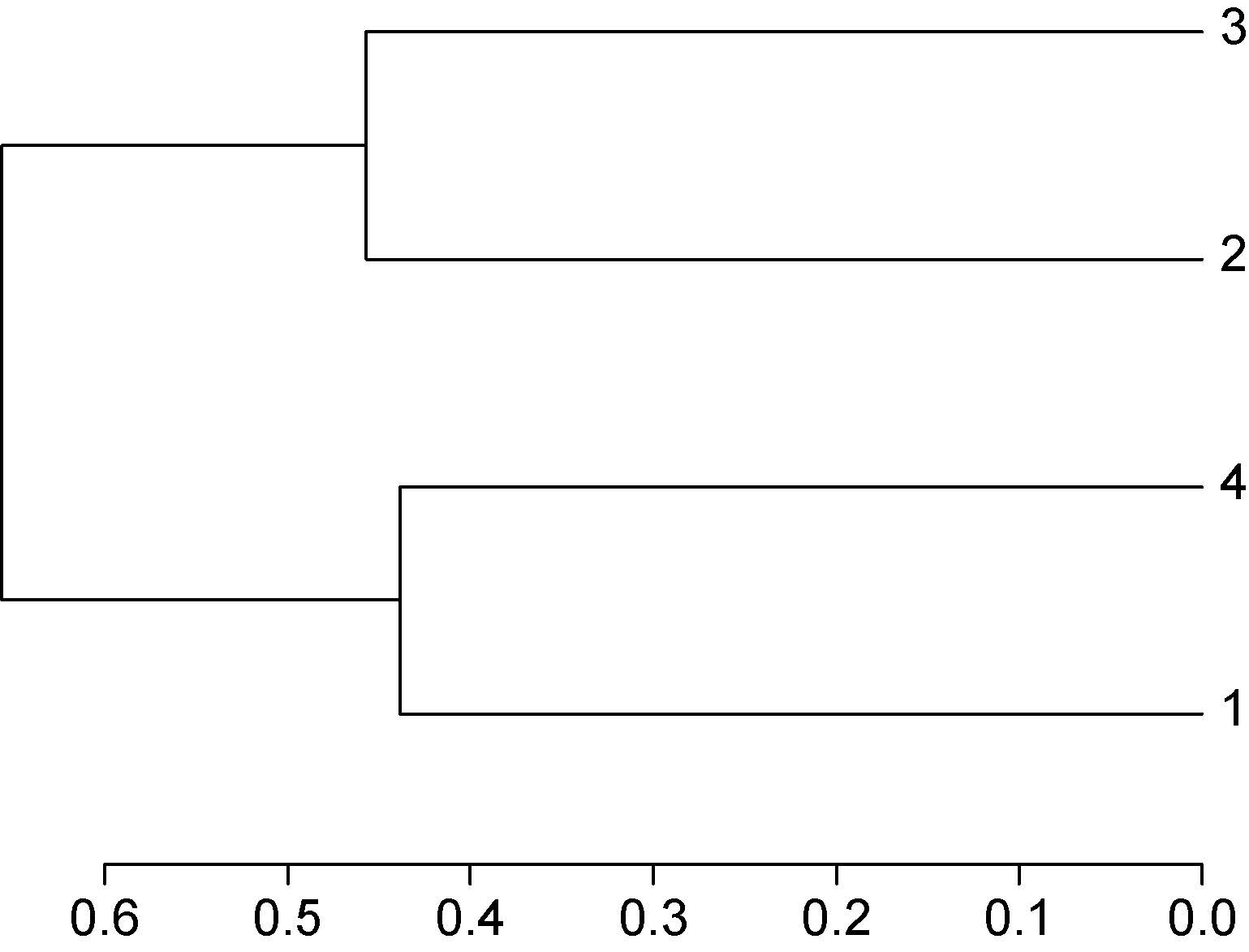 |
| 图 5 多样品相似度树状图Fig. 5 Multiple samples similarity tree |
微生物是湿地生态系统的主要分解者,推动系统的物质循环和能量流动,对维持生态平衡、涵养水源、调节气候、降解污染物等方面起着十分重要的作用。研究湿地生态系统微生物的变化规律及群落结构,有助于更深层次了解湿地微生物多样性及湿地生态系统结构和功能。目前国内关于湿地微生物的研究已有较多的报道,大多采用基于PCR的构建克隆文库方法、变性梯度凝胶电泳(DGGE)及荧光原位杂交(FISH)等技术。本研究采用高通量测序技术,首次对西溪湿地4个不同植被下沉积物样品的细菌群落组成进行了比较分析,结果表明,沉积物中具有很高的细菌多样性,鉴定到的共有30门252属。其中变形菌门(Proteobaeteria)是最优势的菌群,在1号至4号样品中的所占比例分别为64.7%、31.0%、31.5%和53.3%。据报道,变形菌门在各类环境包括水体沉积物中普遍存在,是细菌中最大的类群(Cifuentes et al,2000; Madrid et al,2001)。Ravenschlag等(2001)分析北极群岛海洋沉积物微生物群落结构时发现,γ-变形菌纲(γ-Proteobacteria)和δ-变形菌纲(δ-Proteobacteria)在变形菌门类群中占主导地位; Li等(2009)用16S rRNA基因文库的方法,分析了南海地区的表层沉积物细菌的多样性,研究结果也同样支持这一观点。本研究也得到了相似的结果,变形菌门中以β-变形菌纲、γ-变形菌纲及δ-变形菌纲为优势类群,这些菌群具有极为丰富的代谢多样性,在西溪湿地沉积物的C、N、S、Fe等元素循环中发挥着重要功能。
根据研究,与氮素循环密切相关的硝化细菌分散在变形菌门中的α、β、γ、δ类,当环境中大量氨存在时,这类细菌的数量将会较多。本研究中4个沉积物样品硝化细菌的类群主要分布在亚硝化球菌属(Nitrosococcus)、硝化螺菌属(Nitrospira)、亚硝化单胞菌科(Nitrosomonadaceae_uncultured)、Nitrospinaceae_ uncultured和硝化螺菌科(Nitrospiraceae_uncultured),其中硝化螺菌科是硝化细菌的优势种群,尚属未培养类群。
硫氧化细菌和硫(或硫酸盐)还原细菌都是硫素循环的重要菌群,主要分布于变形菌门中的β、γ类,通过鉴定,4个样品都有且为优势种属的硫氧化细菌是硫杆菌属(Thiobacillus); 硫碱螺旋菌属(Thioalkalispira)只在样品2、3中发现,贝日阿托菌属(Beggiatoa)只在样品1中存在。而硫(或硫酸盐)还原细菌的优势种群是脱硫盒菌科(Desulfarculaceae_ uncultured),其次是脱硫叶菌属(Desulfobulbus)、杆状脱硫菌属(Desulforhabdus)、Desulfocapsa属、Desulfobacca属和脱硫单胞菌目(Desulfuromonadales_ unclassified)。硫酸盐还原细菌通常生活在含有丰富有机质和高含量硫酸盐的水体或沉积物中(Higashioka et al,2013),据报道,被还原的S-2能与沉积物中的多种重金属离子如Zn2+、Cu2+、Hg2+及Pb2+等形成溶度很小的金属硫化物,重金属硫化物的沉淀是沉积物固定重金属的主要机制(王文卿等,1999)。
甲烷营养菌大部分属于γ变形细菌,种类多样,在碳循环中有重要作用,能够将甲烷分解后转化为细胞物质。4个样品中鉴定到的甲烷营养菌类群主要有甲基杆菌属(Methylobacter)、甲基孢囊菌属(Methylocystis)、甲基微球菌属(Methylomicrobium)、甲基暖菌属(Methylocaldum)、Methylogaea属、Methylotenera属、甲基球菌科(Methylococcaceae_ unclassified)和嗜甲基菌科(Methylophilaceae_uncultured),其中甲基杆菌属(Methylobacter)是所有甲烷营养菌的优势种属。
绿弯菌门是本研究中的第二大类群,在1号至4号样品中的占比分别是7.0%、24.9%、43.0%和13.5%。这类细菌通过光合作用产生能量,兼性厌氧,有些类群为专性厌氧菌(Grégoire et al,2011)。绿弯菌主要分布于深海和湖泊有机物丰富的沉积物中,在许多相关的研究中频繁被发现(魏曼曼等,2012),但有关绿弯菌功能特性的报道较少,Björnsson等(2002)实验证明,绿弯菌确实是活性污泥的组成成分之一; 杨小丽等(2013)研究发现,绿弯菌与COD去除具有较好的相关性; Sorokin等(2012)也发现,绿弯菌可能与亚硝酸盐氧化作用有关; 曹新垲等(2012)对工业废水中的萘进行高效生物处理时发现,属于绿弯菌门的Levilinea对于染料中的萘有一定的去除作用。因此推测,作为西溪湿地沉积物的优势菌群之一,绿弯菌在湿地环境污染物的降解方面可能发挥有重要作用。
此外,本研究的4个样品中约有10%—15%的序列属于无法确定分类位置(Unclassified)的类群,这些细菌很可能是新的细菌分类单元,另有30%—50%的序列与未培养细菌具有高度相似性,表明湿地中存在大量未知的功能菌群。以上结果反映了西溪湿地沉积物中独特的细菌组成,同时也揭示了沉积物中有许多菌种资源尚待发掘和认知。
3.2 细菌多样性与环境因素的关系物种多样性是评价群落质量的重要指标,本研究采用Chao 指数和Shannon指数对细菌群落结构进行综合分析。从分析结果来看,4个样品的Chao 指数和Shannon指数均较大,表明西溪湿地水生植物园的沉积物中细菌群落的丰度和多样性较高,这一结果可能与沉积物富含有机物等营养物质有关。有实验表明,在土壤营养物质丰富时,微生物群落的数量、生物量等与土壤有机碳、总氮等呈明显正相关(Córdova- Kreylos et al,2006)。本研究中,4个沉积物样品中有机碳丰富,其含量在18.16—49.31mg/g,总氮、总磷含量也较高,前者4.73—9.42mg/g,后者0.25—0.91mg/g。说明细菌对富营养生境有一定的耐受性,一定程度的富营养有利于细菌的生长和繁殖,导致群落结构复杂,多样性更丰富; 但如果富营养过度,也可能对一些敏感类群产生抑制作用,或是丰富的营养促进了特定几类细菌的大量生长,从而限制了其他许多细菌的生长(郑艳玲等,2012),如1号样品的有机碳、总氮和总磷含量显著高于2号和3号样品(表 1),但前者的丰度及多样性指数反而较低。
从相似性分析来看,1号与4号样品,2号与3号样品之间的细菌群落结构及丰度较为相似(图 4和图 5)。1号和4号样品的优势种属是铁杆菌属(Ferribacterium),相对丰度分别是8.29%和12.30%。据报道,在缺氧的湖泊沉积物中,Fe+3的还原作用通常会普遍发生,Ferribacterium能将Fe+3还原成Fe+2,导致铁从水生生境中的转移(Cumming et al., 1999)。据此推测,样品1和4的生境中可能都富含铁,从而使铁杆菌成为优势种群。2号和3号样品的优势种属是乳球菌属(Lactococcus),相对丰度分别是13.52%和8.87%。乳球菌是一类耐氧菌,其适宜生长的生境一般是植物体,据此推测,2号和3号沉积物中乳球菌数量较多可能与采样地的水生植物相关,至于其在沉积物中的生态学意义少见报道,需进一步的研究确定。因此,细菌的群落结构和多样性,还可能与采样地植物种类和生长状况有关。在有水生植物的环境中,水生植物的残体及根系代谢物等也可能对沉积物中细菌的种属多样性产生影响。总之,湿地沉积物是一个非常复杂的生态系统,各种环境因子、生物因子相互作用和影响,很多情况下并不能用简单的单因子之间的相关性来描述其和微生物之间的关系,沉积物综合环境条件的差异,才是导致其多样性和优势微生物种属发生变化的根本原因。对这方面的研究,必须进行多年的试验观察,才能比较全面地分析微生物和环境因子之间的相互作用。
3.3 微生物多样性的研究方法微生物群落多样性的研究主要涉及对生态系统中微生物的种类、丰度、分布均匀性和结构变化等的解析,但传统的纯培养技术因其方法具有较大的局限性,很难体现自然生境中的微生物群落特征(王保军等,2013)。因此,在过去的40多年里,微生物多样性的研究方法已经从传统的培养分离发展到了无需依赖纯培养的现代分子生物学技术。这些技术主要包括核酸杂交法、DNA指纹图谱技术和宏基因组学等。核酸杂交技术(如FISH和DNA芯片技术)对环境中具特殊功能微生物的鉴定非常有效,但是由于目前尚无法得到所有环境微生物的探针序列,因此有一定的局限性。基因指纹图谱技术包括末端限制性片断长度多态(terminal restriction fragment length polymorphism,T-RFLP)、变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、DNA单链构象多态性(single str and conformation polymorphism,SSCP)等,由于具有操作简便以及可同时分析多个样品的优点,已经被广泛地用于环境微生物群落结构和多样性评价以及动态监测(Singh et al,2011; Ding et al,2012),但同时每种方法几乎都有缺点,例如SSCP的重现性较差; 对于超过500bp的DNA片段,DGGE的分离灵敏度会降低等等。
1998年H and elsman等首次提出宏基因组学的概念(H and elsman et al,1998)。高通量测序技术作为宏基因组学最成熟的关键技术,其全面性、准确性以及信息的深入程度都令其他技术无法企及(孙欣等,2013)。本研究利用Illumina Miseq高通量测序技术,对西溪湿地4个采样点沉积物的细菌多样性进行了分析,鉴定出细菌OTU达到2181个,这是其他方法很难实现的。但如何从这些海量数据中挖掘有效信息成为一大难题,即生物信息学的发展将成为微生物研究的瓶颈。最近,Deng等(2012)基于宏基因组学技术的高通量数据成功构建了分子生态网络(Molecular Ecological Networks,MENs),该网络可根据数据固有特性自动选择阈值,可较好地反映环境中微生物之间的相关性,并且对高通量技术普遍存在的高噪音问题有很好的耐受性。总之,每种技术都有各自的局限性,因此将两种不同的方法结合起来进行综合研究,将是今后发展的方向(孙欣等,2013)。
| 王文卿,林鹏, 1999.红树林生态系统重金属污染的研究.海洋科学,(3):45-47 |
| 王保军,刘双江, 2013.环境微生物培养新技术的研究进展.微生物学通报, 40(1):6-17 |
| 任丽娟,何聃,邢鹏等, 2013.湖泊水体细菌多样性及其生态功能研究进展.生物多样性, 21(4):421-432 |
| 孙欣,高莹,杨云锋, 2013.环境微生物的宏基因组学研究新进展.生物多样性, 21(4):393-400 |
| 李靖宇,杜瑞芳,赵吉, 2015.乌梁素海富营养化湖泊湖滨湿地过渡带细菌群落结构的高通量分析.微生物学报, 55(5):598-606 |
| 杨小丽,周娜,陈明等, 2013. FISH技术解析不同氨氮浓度MBR中的微生物群落结构.东南大学学报(自然科学版), 43(2):380-385 |
| 时玉,孙怀博,刘勇勤等, 2014.青藏高原淡水湖普莫雍错和盐水湖阿翁错湖底沉积物中细菌群落的垂直分布.微生物学通报, 41(11):2379-2387 |
| 何建瑜,刘雪珠,赵荣涛等, 2013.东海表层沉积物纯培养与非培养细菌多样性.生物多样性, 21(1):28-37 |
| 陈久和, 2003.城市边缘湿地生态环境脆弱性研究--以杭州西溪湿地为例.科技通报, 19(5):395-398, 402 |
| 郑艳玲,侯立军,陆敏等, 2012.崇明东滩夏冬季表层沉积物细菌多样性研究.中国环境科学, 32(2):300-310 |
| 赵亚杰,赵牧秋,鲁彩艳等, 2015.施肥对设施菜地土壤磷累积及淋失潜能的影响.应用生态学报, 26(2):466-472 |
| 徐长君,张国发, 2009.大庆湿地的生物多样性及其保护策略.中国农学通报, 25(11):215-219 |
| 曹新垲,杨琦,郝春博, 2012.厌氧污泥降解萘动力学与生物多样性研究.环境科学, 33(10):3535-3541 |
| 魏曼曼,陈新华,周洪波, 2012.深海热液喷口微生物群落研究进展.海洋科学, 36(6):113-121 |
| Björnsson L, Hugenholtz P, Tyson G W et al, 2002. Filamentous Chloroflexi(green non-sulfur bacteria) are abundant in wastewater treatment processes with biological nutrient removal. Microbiology, 148(8):2309-2318 |
| Cifuentes A, Antón J, Benlloch S et al, 2000. Prokaryotic diversity in Zostera noltii-colonized marine sediments. Appl Environ Microbiol, 66(4):1715-1719 |
| Córdova-Kreylos A L, Cao Y P, Green P G et al, 2006. Diversity, composition, and geographical distribution of microbial communities in California salt marsh sediments. Appl Environ Microbiol, 72(5):3357-3366 |
| Cumming D E, Caccavo F Jr, Sping S et al, 1999. Ferribacterium limneticum, gen. nov., sp. nov., an Fe(III)-reducing microorganism isolated from mining-impacted freshwater lake sediments. Arch Microbiol, 171(3):183-188 |
| Deng Y, Jiang Y H, Yang Y et al, 2012. Molecular ecological network analyses. BMC Bioinformatics, 13:113 |
| Ding X, Peng X J, Peng X T et al, 2012. Diversity of bacteria and archaea in the deep-sea low-temperature hydrothermal sulfide chimney of the Northeastern Pacific Ocean. Afr J Biotech, 11(2):337-345 |
| Grégoire P, Fardeau M-L, Joseph M et al, 2011. Isolation and characterization of Thermanaerothrix daxensis gen. nov., sp. nov., a thermophilic anaerobic bacterium pertaining to the phylum "Chloroflexi", isolated from a deep hot aquifer in the Aquitaine Basin. Syst Appl Microbiol, 34(7):494-497 |
| Handelsman J, Rondon M R, Brady S F et al, 1998. Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products. Chem Biol, 5(10):R245-R249 |
| Higashioka Y, Kojima H, Watanabe M et al, 2013. Desulfatitalea tepidiphila gen. nov., sp. nov., a sulfate-reducing bacterium isolated from tidal flat sediment. Int J Syst Evol Microbiol, 63:761-765 |
| Klammer S, Posch T, Sonntag B et al, 2002. Dynamics of bacterial abundance, biomass, activity, and community composition in the oligotrophic Traunsee and the Traun river(Austria). Water Air Soil Pollut:Focus, 2(4):137-163 |
| Li H R, Yu Y, Luo W et al, 2009. Bacterial diversity in surface sediments from the Pacific Arctic Ocean. Extremophiles, 13(2):233-246 |
| Li J L, Bai J, Gao H W et al, 2011. Distribution of ammonia-oxidizing Betaproteobacteria community in surface sediment off the Changjiang River Estuary in summer. Acta Oceanol Sinica, 30(3):92-99 |
| Madrid V M, Aller J Y, Aller R C et al, 2001. High prokaryote diversity and analysis of community structure in mobile mud deposits off French Guiana:identification of two new bacterial candidate divisions. FEMS Microbiol Ecol, 37(3):197-209 |
| Ravenschlag K, Sahm K, Amann R, 2001. Quantitative molecular analysis of the microbial community in marine Arctic sediments(Svaibard). Appl Environ Microbiol, 67(1):387-395 |
| Singh R, Sheoran S, Sharma P et al, 2011. Analysis of simple sequence repeats(SSRs) dynamics in fungus Fusarium graminearum. Bioinformation, 5(10):402-404 |
| Sorokin D Y, Lücker S, Vejmelkova D et al, 2012. Nitrification expanded:discovery, physiology and genomics of a nitrite-oxidizing bacterium from the phylum Chloroflexi. ISME J, 6(12):2245-2256 |
| Wang Q, Garrity G M, Tiedje J M et al, 2007. Naïve Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy. Appl Environ Microbiol, 73(16):5261-5267 |
 2015, Vol. 46
2015, Vol. 46


