中国海洋湖沼学会主办。
文章信息
- 孙向楠, 胡晓珂, 王慧. 2015.
- SUN Xiang-Nan, HU Xiao-Ke, WANG Hui. 2015.
- 海洋沉积物细菌群落结构对芘和苯甲酸钠胁迫的响应
- RESPONSE OF BACTERIAL COMMUNITIES TO PYRENE AND BENZOATE SODIUM IN MARINE SEDIMENTS
- 海洋与湖沼, 46(6): 1304-1311
- Oceanologia et Limnologia Sinica, 46(6): 1304-1311.
- http://dx.doi.org/10.11693/hyhz20141200345
-
文章历史
- 收稿日期:2014-12-10
- 改回日期:2015-04-7
2. 中国科学院大学 北京 100049
2. University of Chinese Academy of Sciences, Beijing 100049, China
随着工业的发展和海上石油开采运输等活动的增多,大量多环芳烃(polycyclic aromatic hydrocarbons,PAHs)类物质进入海洋环境,这些多环芳烃极可能通过食物链富集作用危害人类健康(崔志松等,2006)。 在海洋环境中,多环芳烃降解中很重要的一部分是由海洋特有的微生物来完成(Rehmann et al,1998)。高分子量的多环芳烃(含4 个和4 个以上芳香环)通常是由一群微生物协同代谢完成(Juhasz et al,2000)。多环芳烃在微生物的一系列氧化酶和脱氢酶作用下,经过开环反应,逐渐减少苯环数目,最终变为邻苯二甲酸、邻苯二酚、龙胆酸等单苯环芳香族化合物,单苯环化合物再经过开环反应而进入三羧酸循环。在多环芳烃中,芘是具有四个苯环连接的高分子量芳香烃代表,是致癌、致畸、致突变有害污染物。已报告能够降解芘的菌属包括,分支杆菌属(Mycobacterium)和假单胞菌属(Pseudomonas)等(Rehmann et al,1998)。其中分支杆菌属(Mycobacterium)一些菌株能够在添加营养盐的矿物培养基中矿化芘(Heitkamp et al,1988)。
苯甲酸是多种芳香族化合物生物代谢的中间产物,是一种常见的环境污染物,常存在于化工、食品和医药工业废水中并广泛应用于塑料、染料和医药工业等方面,对人类健康有害(Parales et al,2002; 张晓云等,2012)。在自然界中,微生物能够通过好氧和厌氧途径降解苯甲酸类化合物。迄今为止,已经发现很多细菌能够降解苯甲酸钠,例如,盐单胞菌属(Halomonas)(Oie et al,2007)、不动杆菌属(Acinetobacter)(Collier et al,1997; Kim et al,2003)、红假单胞菌属(Rhodopseudomonas)(Geissler et al,1988)、假单胞菌属(Pseudomonas)(Parsek et al,1992)都有能够降解苯甲酸细菌的报道。
芘是多环芳烃上游代谢途径的代表化合物,因其分子亲脂性、低水溶性、低生物可利用性、高稳定性等,造成的环境污染较难消除。在一系列的脱氢酶、加氧酶的作用下,芘分子逐步加氢、氧化将苯环打开,形成一系列低分子量的苯环类化合物,进而开环进入中心代谢途径。苯甲酸钠是只有一个苯环的化合物,相比于芘更容易被微生物降解进入下游相关代谢途径,随后进入三羧酸循环。通过比较芘和苯甲酸钠对环境中菌群的影响,可以初步分析得出在多环芳烃降解上游代谢途径和下游代谢途径中微生物群落组成的差异。
本研究在实验室条件下,向海洋沉积物样品中添加芘和苯甲酸钠,研究芘和苯甲酸钠胁迫下沉积物样品细菌群落结构的变化特征,初步分析海洋沉积物中微生物群落对不同芳香族类化合物的响应。
1 材料与方法 1.1 样品采集海底表层沉积物样品于2013 年9 月采集于龙口港(37°39′N,120°18′E),采集后的样品4°C 冰盒内带回实验室,4°C 保存。
1.2 样品处理实验组1(P1,P2): 称取5g 沉积物样品加入编号为P1,P2 的250mL 三角瓶,添加0.1g 芘及100mL 灭菌海水;
实验组2(B1,B2): 称取5g 沉积物样品加入编号为B1,B2 的250mL 三角瓶,添加0.2g 的苯甲酸钠及 100mL 灭菌海水;
生物对照组(Y1,Y2): 在编号为Y1,Y2 的250mL 三角瓶中加入5g 沉积物样品及100mL 灭菌海水;
化学对照组1(Pa,Pb): 在编号为Pa,Pb 的250mL 三角瓶中加入0.1g 芘及100mL 灭菌海水;
化学对照组2(Ba,Bb): 在编号为Pa,Pb 的250mL 三角瓶中加入0.2g 的苯甲酸钠及100mL 灭菌海水;
将实验组1、实验组2、生物对照组、化学对照组1、化学对照组2 放置于30°C 水平摇床,180r/min,连续培养15 天。生物对照组用于与实验组1、实验组2 比较细菌群落的变化; 化学对照组用于与实验组 1、实验组2 比较实验结束时芘、苯甲酸钠的残留。
1.3 基因组DNA 的提取实验结束时,分别从P1,P2,B1,B2,Y1,Y2 样品瓶中取少量沉积物,6000g 离心收集沉积物及菌体。应用 PowerSoil® DNA 提取试剂盒(MoBio Laboratories,Solana Beach,USA)提取环境基因组DNA。Nanodrop(NanoDrop 2000/2000C,Thermo Scientific,USA)对所提取的DNA 样品进行定量分析,所有样品浓度均大于10 ng/μL,OD260/OD280 均大于1.8。其中Y1,Y2 样品为无底物胁迫的样品; P1,P2 为添加芘胁迫的样品; B1,B2 为添加苯甲酸钠胁迫的样品。
1.4 Illumina Miseq 测序分析委托上海翰宇生物科技有限公司,在上海人类基因组研究中心基因组测序部使用Illumina Miseq 仪器(美国)进行测序。以检测合格DNA 样本为模板,以设计好含有标签接头的引物V3-F: TACGGRAGGCA GCAG; V4-R: AGGGTATCTAATCCT 进行PCR 反应,扩增目的条带。切胶回收纯化后定量,上机测序。
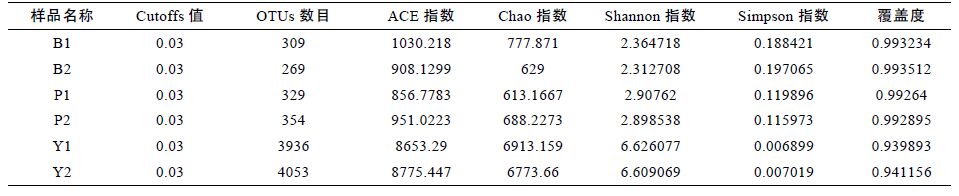
1.5 测序结果分类鉴定经Miseq 平台测序后下机,允许的最低读取序列(read)平均测序质量为Q20,即1%的错误率,去掉N 序列,保留最短长度为100bp,去除较长的均聚物,设置最大聚合值为8。然后根据条码技术的值对处理好的序列进行样本归类,统计样本数目及片段长度分布。使用PyNAST和数据库比对,然后再用 UCHIME(Edgar et al,2011)方法检测并去除嵌合序列; 非嵌合体序列使用GreenGene等核糖体数据库中 aligned 核糖体序列数据比对,去除非目的物种序列污染。采用mothur(Schloss et al,2009)默认的OTU 分析方法获得OTU 代表序列,通过计算序列间未修正的两两距离并使用cluster和97%的相似性对序列进行聚类(Edgar,2010)。然后在每个OTU 聚类里面选择最长的序列作为该OTU 代表序列。以0.03 为cutoff 值划分可操作分类单元(operational taxonomic unit,OTU)。计算cutoff 为0.03 时ACE和Chao 值,绘制稀释曲线。
在cutoff=0.03 情况下分析结果,从门水平(变形菌门从纲水平)对每个实验组两个平行各菌门所占百分比求平均值,将各实验组中微生物各菌门平均组成绘制柱状图,将少于序列总数1%的菌门归为“少数菌群”。此外,分别从对照组、添加苯甲酸钠的实验组、添加芘的实验组中选出属水平数量上占前十位的优势菌OUT 的百分含量绘制heatmap 图,分析群落结构变化(Deng et al,2014)。
1.6 降解率的测定 1.6.1 芘降解率的测定在15 天实验结束时,采用GC-MS 分别测定实验组1(P1,P2)和化学对照组 1(Pa,Pb)中芘的含量。
芘相对降解率=(Pn-P)/Pn×100%,(Pn: 化学对照组1 中芘的残留量; P: 实验组1 中芘的残留)(陈志丹等,2012)。
1.6.2 苯甲酸钠降解率配制2.000mg/L 、 4.000mg/L、6.000mg/L、8.000mg/L、10.000mg/L 的苯甲酸钠系列标准液,测定其吸光光度值并绘制标准曲线。取2mL 实验组或对照组样品,加水定容至 20mL,恒温摇床充分震荡30min,6000r/min 离心 20min,取10mL 上清定容至100 mL。将待测液于石英比色皿在222nm 下测定吸光度。根据吸光度和标准曲线,计算苯甲酸钠的降解率测定(毛宁等,2010)。
苯甲酸钠相对降解率=(Bn-B)/Bn×100%,(Bn: 化学对照组2 中苯甲酸钠的残留量; B: 实验组2 中苯甲酸钠的残留量)。
1.7 序列信息所有的序列都已经上传至NCBI 的SRA 数据库原始数据SRA ID 分别为Y1: SRR1947065; Y2: SRR1948026; P1: SRR1948031; P2: SRR1948037; B1: SRR1948178; B2: SRR1948179。
2 结果 2.1 Illumina MiSeq测序的有效序列数据统计、稀释曲线和多样性分析
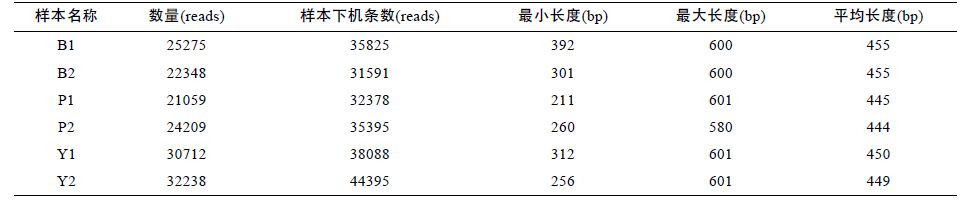
2.1.1 Illumina MiSeq测序的有效序列数据统计 Miseq 测序结果共得到有效序列127174 条,平均每个样品约25434 条序列,有效序列信息见表 1。
 |
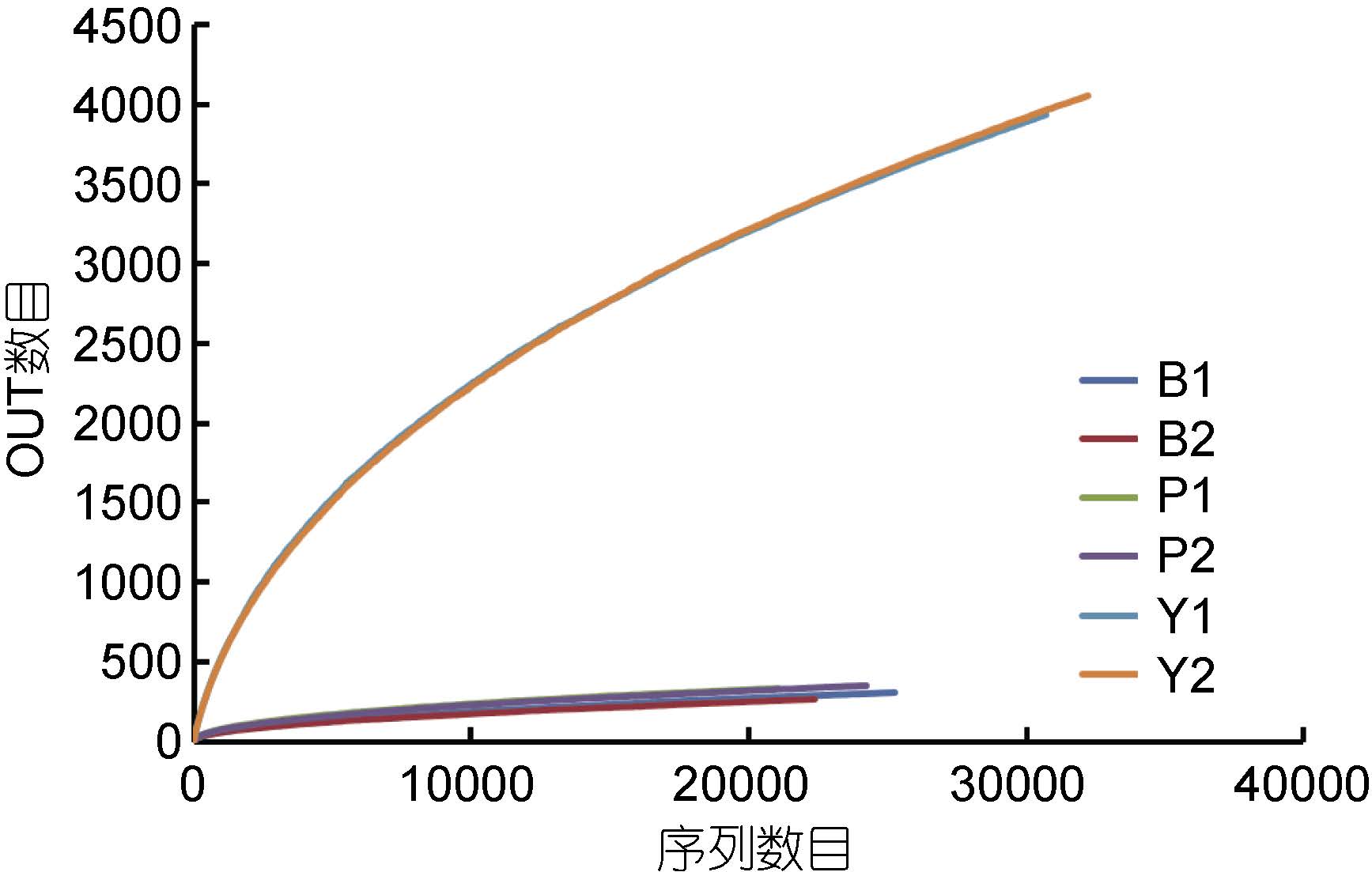
测序的稀释曲线和多样性分析对测序序列进行随机抽样,以抽到的序列数与它们所能代表OTU 的数目构建稀释曲线(rarefaction curve)。分析基于cutoff=0.03 水平,评估对照组Y1,Y2和实验组 B1,B2,P1,P2 样本之间的物种多样性(图 1)。其中样品文库的覆盖率,实验组B1 为0.993234,B2 为0.993512和P1 为0.99264,P2 为0.992895,曲线趋于平坦; 当 cutoff=0.03,Y1、Y2、样品文库的覆盖率分别达到了0.939893和0.941156(见表 2)。本次测序结果基本能够代表样本的真实情况。
|
| 图 1 样品在cutoff=0.03 情况下得到的稀释曲线 Fig. 1 Rarefaction curve obtained in the case of cutoff=0.03 |
Chao 指数和ACE 指数在对照实验组的值远大于添加苯甲酸钠和芘的实验组的值,说明对照组的菌群丰度(community richness)远大于实验组。对照实验组的(Y1,Y2)的Shannon 指数>添加芘的样品的值>添加苯甲酸钠的样品的值,同时对照实验组的(Y1,Y2)的Simpson 指数<添加芘的样品的值<添加苯甲酸钠的样品的值,说明对照实验组的菌群的多样性大于实验组,添加芘的实验组的菌群的多样性大于添加苯甲酸钠的实验组。
2.2 芘或苯甲酸钠降解率的测定经过15 天的摇床培养,相较于化学对照组1(Pa,Pb)实验组1(P1,P2)中的芘的降解率为40.67%; 化学对照组2(Ba,Bb)相比实验组2(B1,B2)中苯甲酸钠的降解率为89.92%。说明在实验过程中,芘和苯甲酸钠在沉积物菌群作用下发生了不同程度的降解。
2.3 苯甲酸钠胁迫下微生物群落结构的变化与响应通过Illumina MiSeq 测序技术,分析在不同污染物的选择压力下沉积物样品中细菌群落演替规律以及预测有降解功能的微生物群落。在添加芘和苯甲酸钠连续培养15 天后菌群多样性极大降低。 相比于对照组微生物菌群,添加苯甲酸钠的实验组和添加芘的实验组,在门的水平上呈现不同的变化特征。
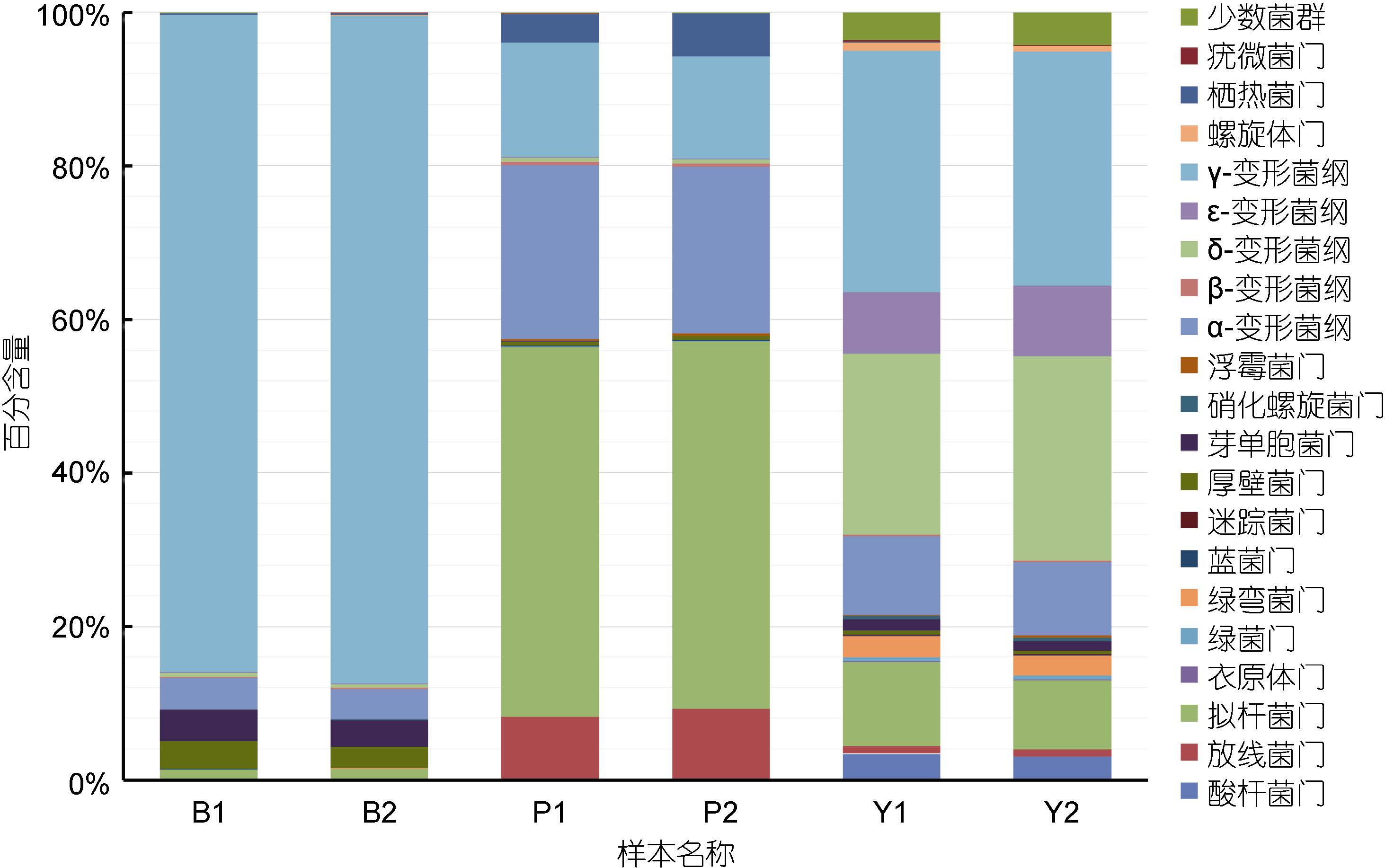
在对照组样品中,占主导地位的是变形菌门。 变形菌门中的γ 变形菌纲、δ 变形菌纲,α 变形菌纲分别占序列总数的31.007%,25.168%,9.857%(百分含量为两个平行试验组的平均值,下同)。此外拟杆菌门占10.024%。添加苯甲酸钠或芘连续培养15 天后的微生物群落与对照组的微生物群落组成有显著的差别。
在添加芘实验组中,菌群主要包括拟杆菌门(48.102%),α 变形菌纲(22.214%),γ 变形菌纲(14.019%),放线菌门(8.735%),热脱硫杆菌门(4.785%)。其中拟杆菌门,α 变形菌纲相对于对照组有所增加。拟杆菌门序列数从对照样品的10.024%增加到48.102%; α 变形菌纲所占比例由对照组的9.857%增加至22.214%。 放线菌门由原位的1.014%增加至8.735%,热脱硫杆菌门所占比例由0.003%增加至4.785%。而变形菌门的γ 变形菌纲,δ 变形菌纲,ξ 变形菌纲相比于原位微生物菌群所占百分比均显著降低,分别从31.007%,25.168%,8.581% 降低至14.019%,0.623%,和0.011%。
Illumina MiSeq 测序结果表明,在添加苯甲酸钠实验组的微生物群落中,变形菌门占了菌群的绝大多数(91.122%)。其中γ 变形菌纲占了整个微生物菌群的86.362%,而对照组菌群中γ 变形菌纲只占了整个群落的31.007%,α 变形菌纲相对于原位菌群的 9.857%有所降低,占4.025%。δ 变形菌纲则相比于对照组的25.168%降低至为0.533%,β 变形菌纲为 0.139%,ε 变形菌纲占0.044%。除变形菌门以外,芽单孢菌门相比于对照组的菌群1.320%增加至3.906%,厚壁菌门的菌群相对于对照组菌群0.493%增加至 3.104% 。拟杆菌门相比于对照组,菌群含量从 10.024%降低至1.480%,变形菌门中的δ 变形菌纲则由对照组的25.168%降低至0.533%。
通过比较可以得出,相比对照组,在添加芘的实验组,拟杆菌门和变形菌门的γ 变形菌纲其富集最为显著,在添加苯甲酸钠的实验组中变形菌门微生物群落极大地富集,其中γ 变形菌纲尤为显著,从对照组的31.007%增加至占菌群的86.362%。
从属的水平上对菌群组成进行分析,发现在对照组中数量居前十位的属大多在1.000%—10.000% 之间,各菌群分布均匀,说明没有芳香族化合物胁迫时没有占主导地位的菌群。
添加苯甲酸钠的实验组,主要菌属包括假海源菌属(Pseudidiomarina)(39.421%)、海杆菌属(Marinobacter)(37.924%)、噬甲基菌属(Methylophaga)(6.254%)、芽孢杆菌属(Bacillus)(2.921%)。而在对照组,假海源菌属(Pseudidiomarina)只占菌群的0.001%,在添加芘的实验组,假海源菌属(Pseudidiomarina)占0.011%; 对照组噬甲基菌属(Methylophaga)只占菌群的0.095%,在添加芘的实验组噬甲基菌属(Methylophaga)数目占菌群总量的0.349%,对照组芽孢杆菌属(Bacillus)占菌群百分数远小于0.001%,添加芘的实验组芽孢杆菌属(Bacillus)数目占菌群总量的0.077%。比较得出,相比于原位菌群的数目,添加苯甲酸钠的实验组假海源菌属(Pseudidiomarina)、海杆菌属(Marinobacter)、 噬甲基菌属(Methylophaga)、芽孢杆菌属(Bacillus)所占百分比数目明显增加。
在添加芘的实验组,Balneola 属(28.892%)、碳酸噬胞菌属(Aequorivita)(13.254%)、黄单胞杆菌科下的 unclassified(7.344%)、叶杆菌科下的unclassified(6.604%)及分支杆菌属(Mycobacterium)(6.541%)占优势地位。而在对照组和添加苯甲酸钠的实验组中 Balneola 属基本检测不到; 对照组中的碳酸噬胞菌属(Aequorivita)和添加苯甲酸钠的实验组中的碳酸噬胞菌属(Aequorivita)OTU 数目基本检测不到。在属的水平,在添加芘实验组中,Balneola 属、碳酸噬胞菌属(Aequorivita)以及在添加苯甲酸钠实验组中海杆菌属(Marinobacter),假海源菌属(Pseudidiomarina)极大地富集,成为优势菌群。
3 讨论Illumina Miseq 技术可为解析微生物群落结构、 阐明海洋沉积物中微生物群落的整体概貌及其在不同污染物胁迫下的演替特征提供技术支持。本文在实验室建立驯化系统,采用Illumina Miseq 技术研究芳香族化合物芘和苯甲酸钠对海洋沉积物中细菌群落结构的影响,初步阐释不同苯环数的芳香族化合物对海洋环境微生物群落的影响。监测海洋沉积物中微生物群落在芘或苯甲酸钠胁迫下的演替特征,有助于揭示芳香烃类污染环境中的优势种群,发现在海洋沉积物中与芘、苯甲酸钠降解相关的微生物。
通常,原位生态环境的破坏会伴随物种丰度和多样性的降低(Peng et al,2013)。Alpha 评估指数和稀释曲线显示,在添加芘和苯甲酸钠的实验组中,海洋沉积物中细菌群落的丰度和多样性都极大降低。添加苯甲酸钠的实验组OTUs 数目分别为309和269,添加苯甲酸钠实验组的OTUs 数目为329和354,添加芘和苯甲酸钠的实验组稀释曲线已经达到饱和。在对照组中(Y1,Y2),OTUs 数目分别为3963和4053,并且随着测序数目的增加尚有增加的趋势。进一步分析 Chao 指数、AC 指数、Shannon 指数和Simplon 指数可知,添加苯甲酸钠和芘的实验组中菌群的丰度和多样性都远远低于对照组。原因可能是添加芳香族化合物导致一些原位微生物失去生存条件,此外添加芳香族化合物可能导致敏感种群的生长受到抑制,同时使某些能够利用或耐受芳香族化合物的菌群得到额外的碳源而富集。
在未添加苯甲酸钠和芘的对照组中,海洋沉积物样品中多样性分布结果如图 2 所示,样品中微生物多样性较高,主要有变形菌门、拟杆菌门、酸杆菌门、 放线菌门等,其中变形菌门为主要的优势菌群,占总序列数的74.801%。在添加芘的实验组中主要包括拟杆菌门、变形菌门、放线菌门、热脱硫杆菌门,其中拟杆菌门占到序列总数的48.102%,变形菌门降低至 47.852%。添加苯甲酸钠的实验组中主要包括变形菌门,占到了序列总数的91.122%,其中γ 变形菌纲占到了总序列数的86.362%。研究结果表明,添加芘和苯甲酸钠的海洋沉积物中微生物群落在门水平呈现不同的变化趋势。变形菌门的γ 变形菌纲在对照组、 添加苯甲酸钠、和添加芘的菌群中都占主导地位。在芘和苯甲酸钠的影响下,一部分细菌失去优势,同时一些细菌能够耐受或者是能够利用芘和苯基酸钠从而实现污染物的降解和自身数量的富集。
|
| 图 2 样品在cutoff=0.03 情况下菌群在门的水平上(变形菌门在纲的水平)的分类情况 Fig. 2 Bacterial classification in phylum level in cutoff=0.03 (Proteobacteria in class level) |
在属水平,对照组中数量占细菌群落前十位的属百分含量介于1.000%—10.000%之间,大多数属于未被明确分类的细菌,说明在对照样品中没有占主导地位的菌属,另一方面也能够说明人们对海洋环境中的细菌群落的认识还不够充分。添加苯甲酸钠的实验组中占前四位的菌属为假海源菌属(Pseudidiomarina)、海杆菌属(Marinobacter)、噬甲基菌属(Methylophaga)、 芽孢杆菌属(Bacillus),分别占39.421%、37.924%、 6.254%、2.921%,而在添加芘的实验组中假海源菌属(Pseudidiomarina)、海杆菌属(Marinobacter)、噬甲基菌属(Methylophaga)、芽孢杆菌属(Bacillus)所占的比例相比于对照组也相应地有所增加,分别占0.011%,0.349%,1.418%,0.077%。在添加芘实验组,占前两位的菌属分别为Balneola 属和碳酸噬胞菌属(Aequorivita)细菌,分别占菌群含量的28.88%,13.25%; 而在对照组和添加苯甲酸钠的实验组中并未检测到以上两者。我们推测Balneola 属和碳酸噬胞菌属(Aequorivita)这两个菌属在耐受或者降解芘的过程中起重要作用,在添加芘后,菌群得到富集成为主导菌群。假海源菌属(Pseudidiomarina)、海杆菌属(Marinobacter)、噬甲基菌属(Methylophaga)、芽孢杆菌属(Bacillus)细菌在添加苯甲酸钠后菌群富集成为优势菌群; 在添加芘的实验组,可能由于环境中芘的降解,在代谢过程中产生的单苯环类化合物导致这四种菌属相比于对照组菌群数量稍微有富集。
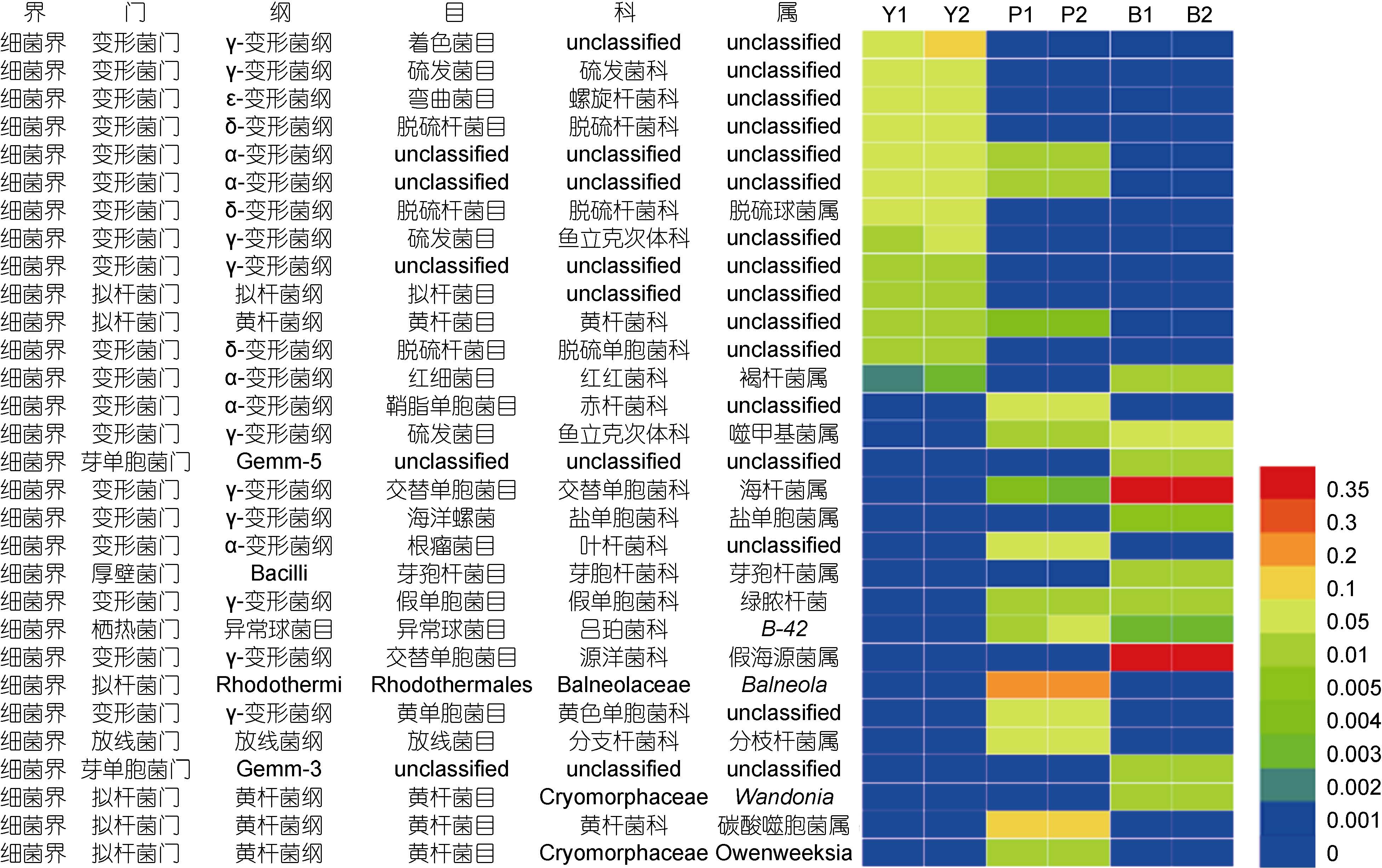
|
| 图 3 Heatmap 图显示有无胁迫样品细菌群落结构的组成与变化 Fig. 3 Heatmap showing the relative abundance of different bacterial genera 柱状图中的不同颜色表征不同种群的测序丰度 |
假海源菌属(Pseudidiomarina)、海杆菌属(Marinobacter)细菌在添加苯甲酸钠实验组中极大地富集成为优势菌群。研究表明海杆菌属(Marinobacter)、 假海源菌属(Pseudidiomarina)能够广泛利用多种碳源作为底物,许多成员已经被证明具有多环芳烃的降解能力。据报道,Marinobacter sp. NCE312 能够降解萘、2 甲基-萘、1-甲基萘( Handley et al,2009)。 Moghadam等(2014)分离到海杆菌属(Marinobacter)和假海源菌属(Pseudidiomarina)菌株能够以芴和菲为唯一碳源和能量来源Kostka等分离的海杆菌属(Marinobacter)和假海源菌属(Pseudidiomarina)细菌在降解石油的过程中发挥了一定作用(Kostka et al,2011; Gao et al,2013)。Li等(2012)发现海杆菌属(Marinobacter)能够在广泛的盐度范围内降解单苯环类化合物,成为主导菌群。Balneola 属、碳酸噬胞菌属(Aequorivita)细菌在芘胁迫下成为添加芘的实验组主要的菌群,有研究表明Balneola 属细菌可以在较广泛的盐浓度范围内有效降解BTEX(张倩,2012),Balneola 属细菌在添加芘的环境中富集还是首次报道。推测在海洋原位环境中Balneola 属、碳酸噬胞菌属(Aequorivita)等细菌协同完成芘的降解和自身数量的富集。
4 结论芘和苯甲酸钠的胁迫对海洋沉积物中微生物群落结构演替产生巨大影响。相对于对照组,添加芘的实验组中Balneola 属、碳酸噬胞菌属(Aequorivita)极大地富集成为优势菌群; 添加苯甲酸钠的实验组中海杆菌属(Marinobacter)和假海源菌属(Pseudidiomarina)极大地富集成为优势菌群。这些菌已经被证明能够参与芳香族化合物的降解。本实验表明,海洋中不同的芳香族化合物胁迫下细菌群落结构发生明显演替。本实验结果能够为芳香族化合物乃至石油等有机污染物的修复提供理论指导。
| 毛宁, 薛泉宏, 唐明. 2010. 2 株放线菌对土壤中苯甲酸和对羟基苯甲酸的降解作用. 西北农林科技大学学报(自然科学版), 38(5): 143-148 |
| 张晓云, 盖忠辉, 台萃等. 2012. 微生物降解苯甲酸的研究进展. 微生物学通报, 39(12): 1808-1816 |
| 张倩. 2012. 高盐条件BTEX 降解菌群多样性、降解基因型及相容性溶质分析. 上海: 华东理工大学硕士论文 |
| 陈志丹, 晁群芳, 杨滨银等. 2012. 一株芘降解菌B2 的降解条件优化及降解基因. 环境工程学报, 6(10): 3795-3800 |
| 崔志松, 邵宗泽. 2006. 一株海洋新鞘氨醇杆菌phe-8(Novosphingobium sp.)的PAHs 降解基因和降解特性. 厦门大学学报(自然科学版), 45(S): 257-261 |
| Collier L S, Nichols N, Neidle E L. 1997. benK encodes ahydrophobic permease-like protein involved in benzoatedegradation by Acinetobacter sp. strain ADP1. Journal ofBacteriology, 179(18): 5943-5946 |
| Deng W K, Wang Y B, Liu Z X et al. 2014. HemI: a toolkit forillustrating heatmaps. PLoS One 9(11): e111988 |
| Edgar R C. 2010. Search and clustering orders of magnitudefaster than BLAST. Bioinformatics, 26(19): 2460-2461 |
| Edgar R C, Haas B J, Clemente J C et al. 2011. UCHIMEimproves sensitivity and speed of chimera detection.Bioinformatics, 27(16): 2194-2200 |
| Gao W, Cui Z S, Li Q et al. 2013. Marinobacter nanhaiticus sp.nov., polycyclic aromatic hydrocarbon-degrading bacteriumisolated from the sediment of the South China Sea. Antonievan Leeuwenhoek, 103(3): 485-491 |
| Geissler J F, Harwood C S, Gibson J. 1988. Purification and propertiesof benzoate-coenzyme A ligase, a Rhodopseudomonas palustrisenzyme involved in the anaerobic degradation of benzoate.Journal of Bacteriology, 170(4): 1709-1714 |
| Handley K M, Héry M, Lloyd J R. 2009. Marinobactersantoriniensis sp. nov., an arsenate-respiring andarsenite-oxidizing bacterium isolated from hydrothermalsediment. International Journal of Systematic andEvolutionary Microbiology, 59(4): 886-892 |
| Heitkamp M A, Franklin W, Cerniglia C E. 1988. Microbialmetabolism of polycyclic aromatic hydrocarbons: isolationand characterization of a pyrene-degrading bacterium.Applied and Environmental Microbiology, 54(10):2549-2555 |
| Juhasz A L, Naidu R. 2000. Bioremediation of high molecularweight polycyclic aromatic hydrocarbons: a review of themicrobial degradation of benzo[a]pyrene. InternationalBiodeterioration & Biodegradation, 45(1-2): 57-88 |
| Kim S II, Song S Y, Kim K W et al. 2003. Proteomic analysis ofthe benzoate degradation pathway in Acinetobacter sp. KS-1.Research in Microbiology, 154(10): 697-703 |
| Kostka J E, Prakash O, Overholt W A et al. 2011. Hydrocarbondegradingbacteria and the bacterial community response ingulf of Mexico beach sands impacted by the deepwaterhorizon oil spill. Applied and Environmental Microbiology,77(22): 7962-7974 |
| Li H, Zhang Q, Wang X L et al. 2012. Biodegradation of benzenehomologues in contaminated sediment of the East China Sea.Bioresource Technology, 124: 129-136 |
| Moghadam M S, Ebrahimipour G, Abtahi B et al. 2014.Biodegradation of polycyclic aromatic hydrocarbons by abacterial consortium enriched from mangrove sediments.Journal of Environmental Health Science and Engineering,12(1): 114 |
| Oie C S I, Albaugh C E, Peyton B M. 2007. Benzoate andsalicylate degradation by Halomonas campisalis, analkaliphilic and moderately halophilic microorganism. WaterResearch, 41(6): 1235-1242 |
| Parales R E, Bruce N C, Schmid A et al. 2002. Biodegradation,biotransformation, and biocatalysis (B3). Applied andEnvironmental Microbiology, 68(10): 4699-4709 |
| Parsek M R, Shinabarger D L, Rothmel R K et al. 1992. Roles ofCatR and cis, cis-muconate in activation of the catBCoperon, which is involved in benzoate degradation inPseudomonas putida. Journal of Bacteriology, 174(23):7798-7806 |
| Peng J J, Li H, Su J Q et al. 2013. Response of bacterialcommunities to short-term pyrene exposure in red soil.Frontiers of Environmental Science & Engineering, 7(4):559-567 |
| Rehmann K, Noll H P, Steinberg C E W et al. 1998. Pyrenedegradation by Mycobacterium sp. strain KR2. Chemosphere,36(14): 2977-2992 |
| Schloss P D, Westcott S L, Ryabin T et al. 2009. Introducing mothur:open-source, platform-independent, community-supported softwarefor describing and comparing microbial communities.Applied and Environmental Microbiology, 75(23): 7537-7541 |
 2015, Vol. 46
2015, Vol. 46