
中国海洋湖沼学会主办。
文章信息
- 郭宝英, 张川, 祁鹏志, 张建设, 管奥, 陈宇, 毋玉婷, 吴常文. 2015.
- GUO Bao-Ying, ZHANG Chuan, QI Peng-Zhi, ZHANG Jian-She, GUAN Ao, CHEN Yu, WU Yu-Ting, WU Chang-Wen. 2015.
- 大黄鱼(Larimichthys crocea)补体调节因子CFH 和 CFHR2 基因的分子特征及表达分析
- CHARACTERIZATION AND EXPRESSION ANALYSIS OF COMPLEMENT REGULATOR FACTOR CFH AND CFHR2 IN LARGE YELLOW CROAKER LARIMICHTHYS CROCEA
- 海洋与湖沼, 46(6): 1405-1412
- Oceanologia et Limnologia Sinica, 46(6): 1405-1412.
- http://dx.doi.org/10.11693/hyhz20150300094
-
文章历史
- 收稿日期:2015-03-28
- 改回日期:2015-10-31
大黄鱼(Larimichthys crocea),隶属鲈形目、石首鱼科(Sciaenidae),俗称黄花鱼,曾广泛分布于中国东海、南海以及黄海南部。大黄鱼肉质细嫩、味道鲜美,是我国沿海传统“四大海产”之一。由于野生资源衰竭,大黄鱼人工养殖业发展迅速,养殖主要集中于浙江、广东和福建等沿海地区。大黄鱼现已发展成为我国海水网箱养殖量最大的鱼种,经济效益巨大(张彩兰等,2002; Zheng et al,2006)。近年来,由弧菌属细菌引起弧菌病呈爆发趋势。该病发病迅速、传播速度快、流行时间长、死亡率高(金珊等,2005),对大黄鱼养殖业造成严重冲击。实际生产中,常用抗生素及化学药物来预防治疗该病,虽然防治效果明显,但会导致耐药性增加,药物残留等一系列不良效果。近年来研究人员试图通过提高大黄鱼自身免疫力来防御病害侵袭,而这需要在深入阐释大黄鱼免疫调控机制的基础上进行。目前,已有相当数量的大黄鱼免疫相关基因被发现,其参与疾病发生的响应模式也被陆续鉴定(Wang et al,2009; Tian et al,2010; Qian et al,2013; Li et al,2014; Zhou et al,2014),大黄鱼基因组测序也已完成(Wu et al,2014; Ao et al,2015)。这些研究为最终阐释大黄鱼响应疾病发生的免疫调节机制提供了理论基础。
补体是鱼类免疫系统重要的组分,在免疫组织中,约有二分之一的补体成分是作为调节因子存在的,而其中大部分都存在于血清和细胞表面中。当受到病原体刺激后,补体各成分依赖于一系列丝氨酸蛋白酶的酶解级联反应被活化,产生具有生物活性的多肽片段,聚合形成具有酶活性的复合体,发挥识别和消除外来病原体,辅助吞噬、裂解细胞,免疫调节等作用。同哺乳动物类似,鱼类补体系统可通过经典途径、旁路途径、凝集素途径被激活,3 条途径既相对独立又相互联系(Boshra et al,2006)。哺乳动物中补体系统的活化是由许多与膜结合的可溶性补体调节蛋白来调节的,这些补体调节蛋白可以避免自身组织由于过度激活而造成损伤(Molina et al,2004)。补体调节因子CFH 属于RCA 家族,是单链血清糖蛋白,在替代途径中可与细胞膜结合膜蛋白发挥作用,也可以以可溶性状态发挥作用。CFH 可以通过和C3b的结合加速C3 转化酶的分解,同时作为CFI 的辅因子,提高了CFI 与C3b 的亲和力,促进丝氨酸蛋白酶切割C3b 分子。研究表明各种补体效应分子能够与多种调控蛋白质相结合,形成复合物,进而识别补体受体,形成补体质膜穿孔复合物,杀伤靶细胞(Buresova et al,2011)。除了CFH 以外,已经有5 种CFH 家族补体调节因子CFHR1-5 在人类中被鉴定克隆出来。它们彼此之间具有高度的同源性,由数量不等的短的重复SCRs 序列复制而成,每一个SCR 结构域包含约60 个进化上高度保守的氨基酸(Hourcade et al,1989)。CFH和CFHR 基因已经在老鼠(Demberg et al,2002)、猪(Hegasy et al,2003)、斑马鱼(sun et al,2010)等生物中被鉴定。虽然补体系统在鱼类机体免疫中发挥至关重要作用,但有关大黄鱼补体研究的报道较少,仅本团队前期对大黄鱼补体分子C3/C4 进行了分子鉴定及表达分析,但对于补体调节因子的研究依然空白。本文鉴定了大黄鱼补体调节因子CFH和CFHR2,并分析了其组织表达特异性及响应溶藻弧菌感染的表达特征。本文研究成果可望为大黄鱼响应溶藻弧菌感染的免疫调节机制的最终阐释提供资料支撑。
1 材料与方法1.1 材料实验用200 条健康大黄鱼均取自浙江海洋学院福建苍南养殖基地,均约500g/尾。实验前将大黄鱼养殖在恒温养殖池内,温度控制在24°C,投喂商业饲料。
两周后随机选取健康大黄鱼6 尾,用致死剂量的MS-222 处死。迅速解剖,取脑、肌肉、心脏、鳃、肠、肝脏、脾脏和肾脏等8 种组织,置于RNA 保存液中,-80°C 保存备用。
将实验室保存的溶藻弧菌(Vibrio alginolyticus)菌种活化,扩大培养,然后稀释菌液到108 CFU/mL 备用。随机选取100 条健康大黄鱼,等分为实验组和对照组,每组50 尾。实验组采用腹腔注射法每尾注射200μL 溶藻弧菌悬液,对照组每尾注射200μL PBS。分别于注射后0h,6h,12h,24h,36h和48h 采样。每次取样4 尾,取肝、脾、肾3 种组织,-80°C 保存备用。用Trizol(Invitrogen)提取总RNA,试剂盒(Fermentas)逆转录成cDNA,用于大黄鱼CFH和CFHR2 基因的克隆扩增及表达检测。
1.2 Lc-cfh和Lc-cfhr2 基因克隆及全长cDNA 的获得分析本实验室测序的大黄鱼全基因组序列,获取大黄鱼CFH和CFHR2 的基因全长。在起始密码子区和终止密码子区设计特异性引物P1和P2,PCR 扩增获得大黄鱼CFH和CFHR2 基因(Lc-cfh和Lc-cfhr2)ORF 区全长。
1.3 序列分析采用NCBI 数据库中ORF finder 程序预测Lc-cfh和Lc-cfhr2 氨基酸序列,并进行同源序列比对。采用SMART(http: //smart.embl-heidelberg.de/)和SignalP 4.0 程序(http: //www.cbs. dtu.dk/services/ SignaIP/)分别进行结构域分析和信号肽预测。利用CLUSTALX软件进行氨基酸序列的多重比对。采用邻接法(Neighbour-joining)构建系统发育树,通过自举分析(bootstrap)进行置信度检测,重复次数为1000 次。
1.4 Lc-cfh和Lc-cfhr2 特异性表达分析根据Lc-cfh和Lc-cfhr2 基因的cDNA 序列跨内含子区设计一对特异性引物P3和P4,用于荧光定量分析(qPCR),选取β-actin 作为内参基因(黄左安等,2011)。
荧光定量检测试剂采用SYBR 定量检测试剂盒(TaKaRa),qPCR 采用10μL 反应体系,含SYBR Premix ExTaq(2×)缓冲液5μL,正向和反向引物(10μmol/L)各0.6μL,cDNA 模板0.5μL,灭菌水补足至10μL。扩增反应在Applied Biosystems 7500P 荧光定量 PCR 仪上进行,95 °C 变性120 s 后,按以下程序进行40 个循环: 95°C 15s,59°C 45s,72°C 30s。实验结束时采用熔解曲线分析,流程为95°C 30s,72°C 60s,95°C 30s,每样品重复3 次。采用2-ΔΔCt(Livak et al,2001)分析相对定量结果,显著性水平采用SPSS18.0 软件中的单因素方差分析(One-way ANOVA)进行统计,P<0.05 为差异显著。
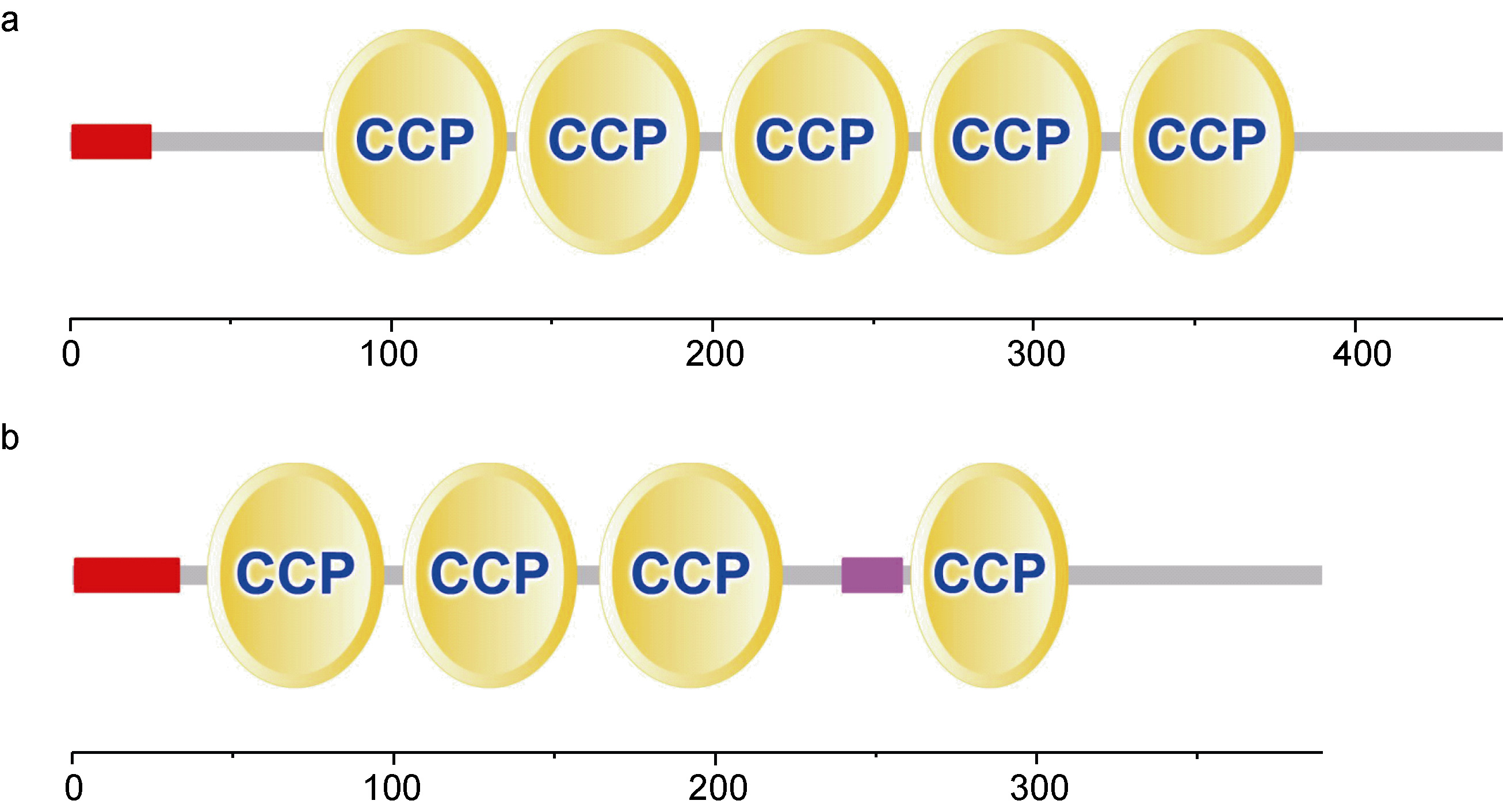
Lc-cfh 基因cDNA 序列全长1332bp 个核苷酸(GenBank 登录号: KP710858),编码一个由443 个氨基酸组成的前体蛋白,其预测的相对分子质量为49.96 kDa,等电点为6.66。前体蛋白N 端24 个氨基酸为信号肽序列。利用SMART 数据库对结构域检测分析显示,Lc-cfh 蛋白包含5 个CCP 结构域(图 1a)。序列存在1 个潜在的N-糖基化位点(Asn237)。Lc-cfhr2基因cDNA 序列全长1170bp 个核苷酸(GenBank 登录号: KP710859),编码一个由389 个氨基酸组成的前体蛋白,相对分子质量43.53 kDa等电点为5.61,前体蛋白N 端32 个氨基酸为信号肽序列,包含4 个CCP结构域(图 1b)。序列存在3 个潜在的N-糖基化位点(Asn90,Asn112,Asn148)。虽然在两个基因中,糖基化位点数不同,但是它们都处于CCP 结构中。同样人的CFH 基因发现与肝素的三个结合位点分别位于SCRs7,SCRs13,SCRs20 上(Krushal et al,1998)。
|
|
图 1 大黄鱼Lc-cfh和Lc-cfhr2 结构域
Fig. 1 Structural domains of Lc-cfh and Lc-cfhr2 putative amino acids sequences a. Lc-cfh 结构域; b. Lc-cfhr2 结构域 |
比对Lc-cfh、Lc-cfhr2、Hs-cfh 及Hs-cfhr2 CCP结构序列,发现半胱氨酸(Cys)残基具有高保守性,所有CCPs 均包含4 个Cys 残基,且排列位置大致相同。同时,脯氨酸、天冬氨酸以及甘氨酸残基也相对保守(图 2)。

|
|
图 2 Lc-cfh、Lc-cfhr2、Hs-cfh 及Hs-cfhr2 CCP 结构序列比对图
Fig. 2 CCP alignment of Lc-cfh,Lc-cfhr2,Hs-cfh and Hs-cfhr2 左边为CCPs 代号,其中CCP1-20 为Hs-cfh CCPs,CCP21-24 为Hs-cfhr2 CCPs,CFH1-5 为Lc-cfh CCPs,CFHR21-24 为Lc-cfhr2 CCPs; 右边数字为CCPs 长度; 空缺氨基酸残基用圆点表示,彩色表示同源性>50%的氨基酸残基,完全相同的氨基酸残基用深蓝色表示 |
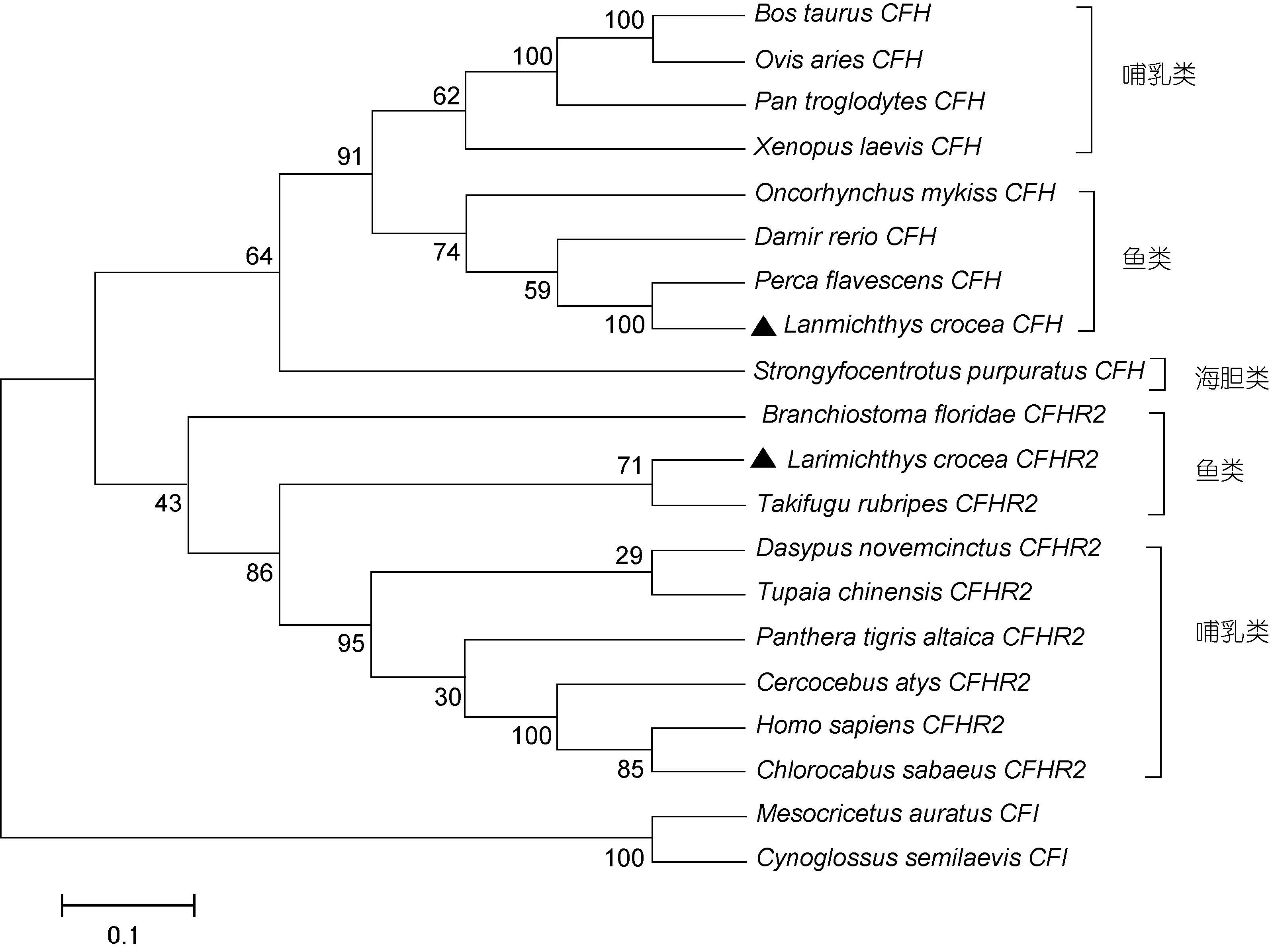
为了研究Lc-cfh、Lc-cfhr2 在进化过程中所处的位置,构建以金仓鼠(Mesocricetus auratus)和半滑舌鳎(Cynoglossus semilaevis)CFI 基因为外群的系统发育树。如图 3 所示,Lc-cfh、Lc-cfhr2 分别和其它鱼类CFHs 及CFHR2s 聚合成单独分支,然后再与哺乳类相关同源物聚成一大类,聚类结果符合传统的分类方法。总体来看,CFH和CFHR2 基因在进化树中聚合成独立的大支,独立于外群CFI 基因,表明两个补体调节因子较近的亲缘关系。
|
|
图 3 基于邻近法的CFH和CFHR2 系统发育树
Fig. 3 The phylogenetic tree of CFH and CFHR2 based on neighbor-joining method 各物种GenBank 登录号如下: Larimichthys crocea CFH(大黄鱼KP710858.1); Oncorhynchus mykiss CFH虹鳟NP 001117899.1); Oreochromis niloticus CFH罗非鱼XP 013131964.1); Pan troglodytes CFH黑猩猩XP 003308708.1); Danio rerio CFH斑马鱼NP 001186119.1; Perca flavescens CFH金鲈ADX97184.1); Homo sapiens CFH人ABB02180.1); Bos taurus CFH牛NP 001029108.1); Oreochromis niloticus CFH罗非鱼XP 005476028.1); Xenopus laevis CFH非洲爪蟾NP 001079684.1); Larimichthys crocea CFHR2大黄鱼KP710859.1); Strongylocentrotus purpuratus CFH紫海胆 XP 003724666.1); Takifugu rubripes CFHR2红鳍东方鲀 XP 011614930.1); Dasypus novemcinctus CFHR2九带犰狳 XP 004476415.1); Panthera tigris altaica CFHR2东北虎 XP 007096167.1); Tupaia chinensis CFHR2树鼩 XP 006155519.1); Mesocricetus auratus CFHR2金仓鼠XP 012971310.1); Cercocebus atys CFHR2白眉猴XP 011891503.1); Chlorocebus sabaeus CFHR2绿猴XP 007987272.1); Mesocricetus auratus CFI 金仓鼠XP 012978061.1); Cynoglossus semilaevis CFI半滑舌鳎AKN79751.1) |
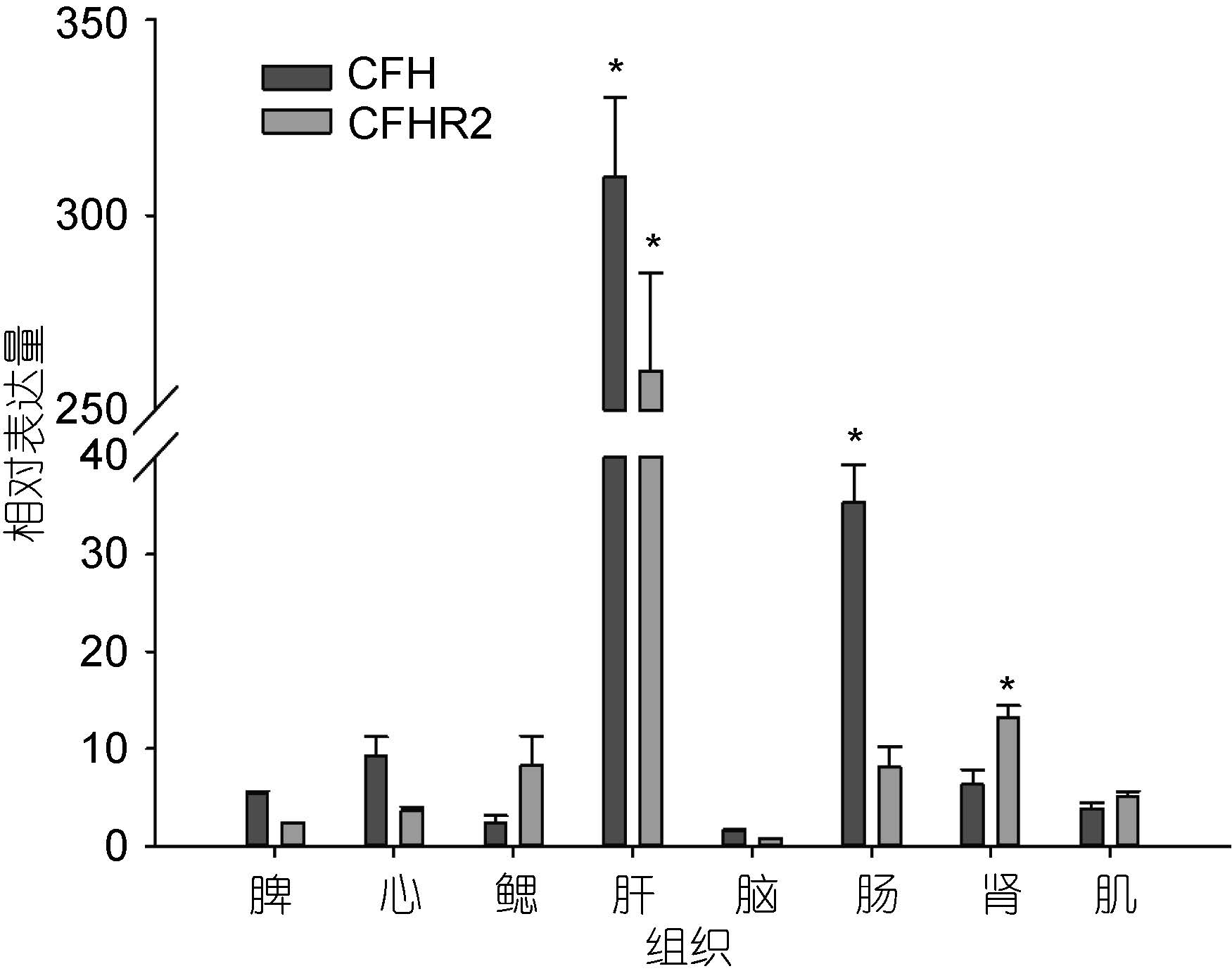
如图 4 所示,在所检测的大黄鱼脾脏、肾、肠、脑、心脏、肌肉、鳃、肝脏等8 种组织中,Lc-cfh和Lc-cfhr2 均有表达,二者均发现在肝脏中表达量最高,显著高于其它组织,而在脑中的二者表达量均最低。除肝脏外,Lc-cfh 在肠道中也检测到较高表达量,显著高于其它组织,而Lc-cfhr2 依次在肾脏、鳃和肠中呈现显著表达。
|
|
图 4 Lc-cfh和Lc-cfhr2 基因组织表达特征分析
Fig. 4 Tissue distributions of Lc-cfh and Lc-cfhr2 *表示显著性差异P<0.05 |
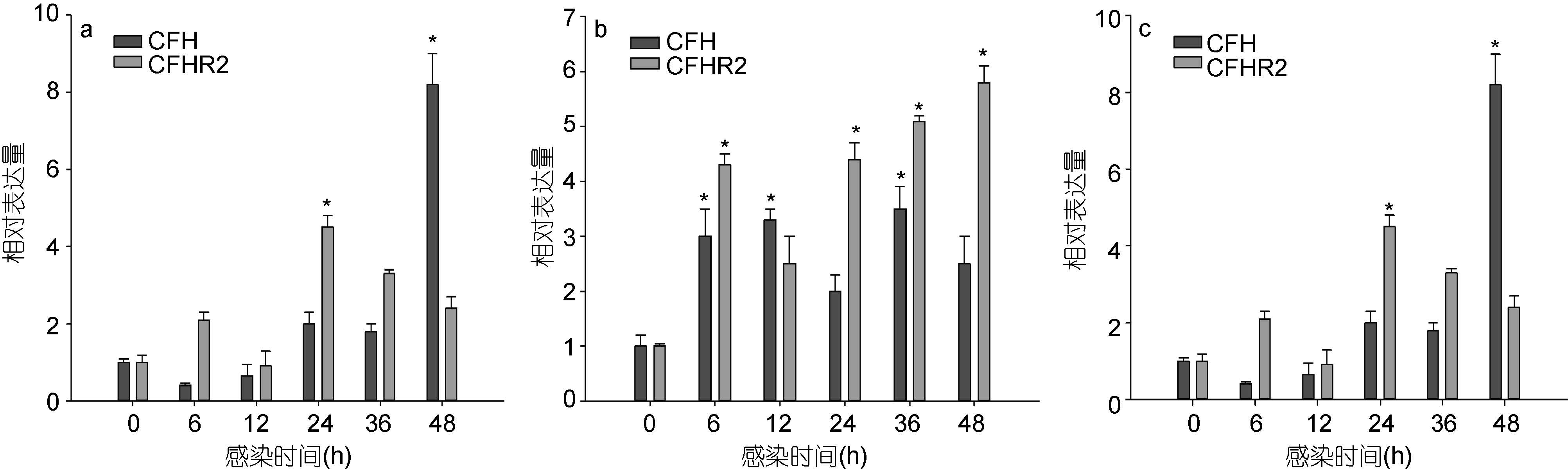
检测了溶藻弧菌感染后,Lc-cfh和Lc-cfhr2 在肝脏、脾脏、肾脏三种组织中的表达情况。如图 5 所示,溶藻弧菌感染后,Lc-cfh和Lc-cfhr2 在肝、脾、肾中的表达量都呈现不同程度上升。在肝脏中,溶藻弧菌感染后6h,Lc-cfh 表达量出现显著升高,感染后24h达到峰值,为正常表达量的11.4 倍,此后Lc-cfh 表达量呈逐渐下降趋势。而Lc-cfhr2 表达量在弧菌感染后呈现温和上升,与Lc-cfh 相似,Lc-cfhr2 也在弧菌感染后24h 达到峰值,但仅为正常表达量的4.3 倍(图 5a)。在肾脏中,溶藻弧菌感染后,Lc-cfh和Lc-cfhr2表达量均呈现明显变化。感染后6h,两基因表达量开始显著升高,并呈现温和上升趋势,Lc-cfh 表达量在36h 时达到峰值,为正常表达量3.5 倍,而Lc-cfhr2 在48h 时达到峰值,为正常表达量的6.1 倍。除感染后12h 外,在其它各时间点,Lc-cfhr2 表达量明显高于Lc-cfh,表明在肾脏组织中,受弧菌刺激后,Lc-cfhr2相比Lc-cfh 具有更高的响应效率(图 5b)。在脾脏中,弧菌刺激后,Lc-cfh 表达量温和上升,感染后24h 表达量显著上升,感染后48h 达到峰值,为正常量的8.4倍,而Lc-cfhr2 在感染后24h 即达到峰值,为正常值4.6 倍,后逐渐降低。暗示受到溶藻弧菌感染,脾脏组织中感染前期Lc-cfhr2 发挥主导作用,而感染24h 后,Lc-cfh 开始发挥主要作用(图 5c)。
|
|
图 5 溶藻弧菌感染后,Lc-cfh和Lc-cfhr2 在肝(a)、肾(b)、脾(c)中表达量变化
Fig. 5 Expression characteristic analysis of Lc-cfh and Lc-cfhr2 in croaker liver(a),kidney(b) and spleen(c)induced by V. alginolyticus *表示显著性差异P<0.05 |
本文利用大黄鱼基因组数据库,获得了大黄鱼补体调节蛋白CFH和CFHR2 的全长cDNA 序列,在ORF 区设计特异引物,获得了Lc-cfh和Lc-cfhr2 ORF全序列(GenBank: KP710858.1,KP710859.1)。序列分析表明,Lc-cfh和Lc-cfhr2 包含多个保守的CCPs 序列(补体控制蛋白),含有CCP 结构域的蛋白质一般分为两类,即补体激活调节蛋白质(regulators of complement activation,RCA)和非补体激活调节蛋白质(non-RCA)(Zipfel et al,2009)。CCP 结构域大约包含60 个氨基酸残基,结构分析表明,CCP 结构域具有“Cys-Cys-Cys-Trp-Cys”基序(LHR),这一序列与其功能直接相关(Soares et al,2005)。Lc-cfh和Lc-cfhr2CCP 结构域均具有4 个高度保守的Cys 残基,具有RCA 蛋白家族的典型特征。CFH 是一种补体激活途径的抑制因子,它在C3b 的表面有多个结合位点,能够有效地抑制FB 与C3b 结合形成C3 转化酶,也可以作为CFI 的辅因子加速C3 转化酶的衰变(Yates et al,2007)。C3b 与CFH 的1—4 个CCP 结构域形成的复合物会改变其结构,使得C3b 的第一个键暴露在外,易于被CFI 切割,而其它的键是随着构象的改变而被切割(Janssen et al,2006; Wu et al,2009)。高度保守的半胱氨酸残基之间可以形成二硫键,使得SCR 成为一个空间环状结构(Hourcade et al,1989)。并且在CCPs 序列中分布若干数量不等的糖基化位点,而糖基化普遍被认为和免疫作用相关(Van Kooyk et al,2008)。尽管所有物种SCRs 结构相对保守,但不同物种CFH 及CFHRs 包含的CCP 数量却有较大差异,CFH 差异更为明显。大黄鱼CFH 含有5 个CCP 结构域,而哺乳动物CFH 一般含有20 个CCP 结构域,非洲爪蟾(Xenopus laevis)含有7 个CCP 机构,而硬骨鱼中的斑马鱼(Danio rerio)含有15 个CCPs,黄金鲈(Perca flavescens)则含有4 个CCP 结构,文昌鱼含有2 个SCRs 结构域(Cai et al,2014),这可能是补体调节因子相关功能区在进化过程中对调控需求不同造成的。系统发育图显示鱼类和哺乳类的CFH、CFHR2分别形成个不同的支系,然后再聚成一大类,基本符合传统分类法。整体上看,CFH和CFHR2 具有较近的亲缘关系。CFH和其相关蛋白CFHRs(CFHR1-5)共同组成CFH 基因家族,它们彼此之间具有高度的同源性,由数量不等的短的重复SCRs 序列复制而成(Skerka et al,2013)。通过比对Lc-cfh和Lc-cfhr2 的CCP 结构,发现所有CCPs 在N 端和C 端都具有高度保守性,CFH1和CFHR21 具有55.1%的相似度,CFH1和CFHR23 具有47.6%的相似度,CFH2和CFHR21 也有49%的相似度。这些序列同源性也表明,补体调节蛋白功能的正常发挥与这些短的同源序列紧密相关。
3.2 大黄鱼CFH和CFHR2 基因表达特性分析组织表达特异性显示,健康大黄鱼CFH 基因和CFHR2 在肝中表达量最高。在斑马鱼(sun et al,2010)和鼠(Hellwage et al,2006)的肝脏组织中,CFH 基因mRNA 表达量也显著高于其它组织。肝脏作为主要的解毒器官,在机体受到外界病原体侵染时对机体免疫起重要作用。同时,肝脏也是许多免疫因子尤其是各补体成分的主要发生器官(Zipfel et al,2002; Wang et al,2008b)。肝脏作为补体调节因子的主要合成器官,也已经在斑马鱼(Sun et al,2010)和虹鳟(Anastasiou et al,2011)上得到了证明。但同时应注意到在不同物种中,肝脏并不作为补体因子的唯一发生器官,这可能与蛋白在每个物种中的特异性功能有关。
溶藻弧菌感染后,Lc-cfh和Lc-cfhr2 mRNA 在肝、脾、肾中的表达量都呈现一个上升趋势。肝脏中CFH 的mRNA 表达量在6h和24h 达到峰值,表达量明显高于对照组。肾脏中CFH 的mRNA 表达量水平在受到感染后6h 增加到对照组的3 倍,并且一直维持这个水平直到48h。在肝、肾中CFHR2 mRNA表达量水平增长同样明显,在24h和48h 达到峰值,分别是对照组的4.6 倍和6.2 倍。说明受到病原体侵袭后,鱼类免疫器官能迅速对此作出免疫应答,大量合成补体调节因子避免补体分子过度激活,从而有效的保护机体不受伤害。此外,CFH 作为一种粘附蛋白,通过细胞表面与CR3 受体结合,清除凋亡细胞、保护细胞对抗慢性炎症(Boshra et al,2006)。在脾脏中CFH mRNA 表达量在6h 下降到低于对照组的水平,直到24h 又高于对照组,而CFHR2 mRNA在6h 的表达量显著高于对照组水平。这说明补体调节因子在免疫途径中的调节有可能是多方面的,CFHR2 的表达量水平可能会对CFH 造成影响,作为同源基因共同在替代途径中发挥调节作用。在溶藻弧菌侵染后6h,CFHR2 的mRNA 表达量在三种免疫组织中都显著增加,说明大黄鱼CFHR2 分子具有重要的免疫调节作用,但其具体的调节机制还需要更加深入的研究。
综上所述,本文报道了大黄鱼补体调节因子CFH和CFHR2 的分子特征,并对其组织表达特异性以及感染溶藻弧菌的大黄鱼肝、脾、肾中的表达量变化进行了相关研究,为大黄鱼抗弧菌免疫的深入研究奠定了基础。
| 张彩兰, 刘家富, 李雅璀等. 2002. 福建省大黄鱼养殖现状分析与对策. 上海水产大学学报, 11(1): 77-83 |
| 金珊, 王国良, 赵青松等. 2005. 海水网箱养殖大黄鱼弧菌病的流行病学研究. 水产科学, 24(1): 17-19 |
| 黄左安, 陈炯, 陆新江等. 2011. 香鱼凝血因子X 基因表达与鳗利斯顿菌感染的相关性. 动物学研究, 32(5): 492-498 |
| Anastasiou V, Mikrou A, Papanastasiou A D et al. 2011. Themolecular identification of factor H and factor I molecules inrainbow trout provides insights into complement C3regulation. Fish Shellfish Immunol, 3: 491-499 |
| Ao J, Mu Y, Xiang L X et al. 2015. Genome sequencing of theperciform fish Larimichthys crocea provides insights intomolecular and genetic mechanisms of stress adaptation.PLoS Genetics, 11(4): 100-118 |
| Boshra H, Li J, Sunyer J O. 2006. Recent advances on thecomplement system of teleost fish. Fish Shellfish Immunol,20: 239-262 |
| Buresova V, Hajdusek O, Franta Z et al. 2011. Functionalgenomics of tick thioester-containing proteins reveal theancient origin of the complement system. J Innate Immun,3(6): 623-630 |
| Demberg T, Pollok-Kopp B, Gerke D et al. 2002. Ratcomplement factor H: molecular cloning, sequencing andquantification with a newly established ELISA. Scand JImmuno, l56: 149-160 |
| Hegasy G A, Willhoeft U, Majno S A et al. 2003. Pigcomplement regulator factor H: molecular cloning andfunctional characterization. Immunogenetics, 55: 462-471 |
| Hellwage J, Eberle F, Babuke T et al. 2006. Two factor H-relatedproteins from the mouse: expression analysis and functionalcharacterization. Immunogenetics, 58: 883-893 |
| Hourcade D, Holers V M, Atkinson J P. 1989. The regulators ofcomplement activation (RCA) gene cluster. Adv Immunol,45: 381-416 |
| Janssen B J, Christodoulidou A, McCarthy A et al. 2006.Structure of C3b reveals conformational changes thatunderlie complement activity. Nature, 444: 213-216 |
| Krushal J, Kemper C, Gigli I. 1998. Ancient origin of humancomplement factor H. J Mol Evol, 47: 625-630 |
| Li C H, Chen J. 2013. Molecular cloning, characterization andexpression analysis of a novel wap65-1 gene fromPlecoglossus altivelis. Comparative Biochemistry andPhysiology Part B: Biochemistry and Molecular Biology,165(2): 144-152 |
| Livak K J, Schmittgen T D. 2001. Analysis of relative geneexpression data using real-time quantitative PCR and the2-ΔΔCT method. Methods, 25(4): 402-408 |
| Lu Cai, Jiu Zhu, Denghua Yin et al. 2014. Identification andcharacterization of complement factor H in Branchiostomabelcheri. Gene, 553: 42-48 |
| Molina H. 2004. Complement and immunity. Rheum Dis ClinNorth Am, 30: 175-191 |
| Qian T, Wang K, Mu Y et al. 2013. Molecular characterizationand expression analysis of TLR 7 and TLR 8 homologs inlarge yellow croaker (Pseudosciaena crocea). Fish andShellfish Immunology, 35(3): 671-679 |
| Rodriguez de Cordoba S, Esparza-Gordillo J, Goicoechea deJorge et al. 2004. The human complement factor H:functional roles, genetic variations and disease associations.Mol Immunol, 41: 355-367 |
| Skerka C, Chen Q, Fremeaux-Bacchi V et al. 2013. Complementfactor H related proteins (CFHRs). Molecular Immunology,56: 170-180 |
| Soares D C, Gerloff D L, Syme N R et al. 2005. Largescalemodeling as a route to multiple surface comparisons of theCCP module family. Protein Eng Des Sel, 18: 379-388 |
| Sun G, Li H, Wang Y et al. 2010. Zebrafish complement factor Hand its related genes: identification, evolution, andexpression. Funct Integr Genomics, 10: 577-587 |
| Tian C, Chen Y, Ao J et al. 2010. Molecular characterization andbioactivity of a CXCL13 chemokine in large yellow croakerPseudosciaena crocea. Fish and Shellfish Immunology,28(3): 445-452 |
| Van Kooyk, Y, Rabinovich G A. 2008. Protein-glycaninteractions in the control of innate and adaptive immuneresponses. Nat Immunol, 9: 593-601 |
| Wang K J, Cai J J, Cai L et al. 2009. Cloning and expression of ahepcidin gene from a marine fish (Pseudosciaena crocea)and the antimicrobial activity of its synthetic peptide.Peptides, 30(4): 638-646 |
| Wang Z, Zhang S, Wang G. 2008. Response of complementexpression to challenge with lipopolysaccharide in embryoslarvae of zebrafish Danio rerio: acquisition of immunologycompetent complement. Fish Shellfish Immunology, 25:264-270 |
| Wu J, Wu Y Q, Ricklin D et al. 2009. Structure of complementfragment C3b-factor H and implications for host protectionby complement regulators. Nat Immunol, 10: 728-733 |
| Zheng W B, Liu G Z, Ao J Q et al. 2006. Expression analysis ofimmune-relevant genes in the spleen of large yellow croaker(Pseudosciaena crocea) stimulated with poly (I: C). FishShellfish Immunol, 21: 414-430 |
| Zhou Q J, Su Y Q, Niu S F et al. 2014. Discovery and molecular cloning of piscidin-5-like gene from the large yellow croaker(Larimichthys crocea). Fish and Shellfish Immunology,41(2): 417-420 |
| Zipfel P F, Skerka C. 2009. Complement regulators andinhibitory proteins. Nat Rev Immunol, 9: 729-740 |
| Zipfel P F, Skerka C, Hellwage J et al. 2002. Factor H familyproteins: on complement, microbes and human diseases.Biochem Soc Trans 30: 971-978 |
 2015, Vol. 46
2015, Vol. 46

