
中国海洋湖沼学会主办。
文章信息
- 蒋然, 王健鑫, 黄备, 张潘, 郑俊威, 俞凯成, 刘明华. 2015.
- JIANG Ran, WANG Jian-Xin, HUANG Bei, ZHANG Pan, ZHENG Jun-Wei, YU Kai-Cheng, LIU Ming-Hua. 2015.
- 椒江口化工园区邻近海域沉积物中细菌多样性初步研究
- BACTERIAL DIVERSITY IN SEDIMENTS IN JIAOJIANG CHEMICAL INDUSTRIAL PARK ADJACENT AREA
- 海洋与湖沼, 46(6): 1420-1437
- Oceanologia et Limnologia Sinica, 46(6): 1420-1437.
- http://dx.doi.org/10.11693/hyhz20150900246
-
文章历史
- 收稿日期:2015-09-19
- 改回日期:2015-10-21
2. 浙江省舟山海洋生态环境监测站 舟山 316021
2. Zhoushan Marine Ecological and Environmental Monitoring Station in Zhejiang Province, Zhoushan 316021, China
椒江口化工园区位于台州湾循环经济聚集区椒江分区的椒南滨海工业区块,是十二五规划的核心建设区域,国家计委、国家经贸委审批的浙江省化学原料药基地正坐落在此。基地建设初期,企业产生的污染物未经处理直接排放到椒江中,对附近环境造成严重污染,2004 年以来,相关部门单位对该区内所有企业的排污口进行封堵,使得该海域生态环境有一定好转(赵永强等,2009)。但除化工园区以外,椒江口上游还有火力、发电等企业,椒江口附近水域中已发现多六六六、溴联苯醚、滴滴涕、重金属等污染物(江锦花等,2006; 余鹏等,2011; 杨华云等,2014)。作为各种污染载体的沉积物,其相关生物承受巨大的环境压力。
微生物多样性及群落结构由于对环境变化十分敏感,常作为环境变化的指示因子(王金成等,2012;Zhu et al,2013)。但关于椒江口及邻近海域的生物与生态的研究主要集中在大型底栖动物、虾蟹、浮游动物、鱼类养殖环境调查等方面(江锦花等,2007; 赵永强等,2009; 杜萍等,2011a; 齐海明等,2013),然而在微生物多样性方面,还比较欠缺: 杜萍等(2011b)首先分析了春季椒江口水体中氮、磷细菌及异氧细菌的分布特征,次年,通过Biolog 及PCR-DGGE 方法研究了椒江口沉积物中的微生物多样性(杜萍等,2012),但主要集中在石油烃及站点多样性的关系,并没有讨论具体的细菌类群; Hu等(2012)分析了椒江口工业园区附近沉积泥中厌氧氨氧化细菌与硝基苯等化学污染物之间的关系; Shen等(2014)使用分子生态学方法,研究了椒江口沉积物中氮依赖型厌氧甲烷氧化细菌。以上两则研究侧重于特殊细菌类别,未曾涉及群落中其它种群细菌及它们多样性的相关信息。
本文采用生理生化及分子鉴定的方法,对椒江化工园区邻近海域沉积物中可培养细菌多样性及群落结构进行研究,并构建环境细菌总DNA 克隆文库,对非培养细菌的多样性及系统发育进行分析,以期为该海域沉积环境生态的相关研究提供科学参考。
1 材料与方法1.1 样品采集2013 年5 月23 日,选择椒江口化工园区附近海域4个站点: C3(28°37'16"N,121°34'19"E)、C4(28°39'54"N,121°37'19"E)、C5(28°37'16"N,121°39'47"E)、C6(28°34'44"N,121°36'43")(图 1),每个站点使用采泥器,采集表层沉积物样品一份,无菌条件下分装于灭菌的样品瓶内,调查船上-20°C 冰柜保存,回实验室后置于-80°C 超低温冰箱长期保存。
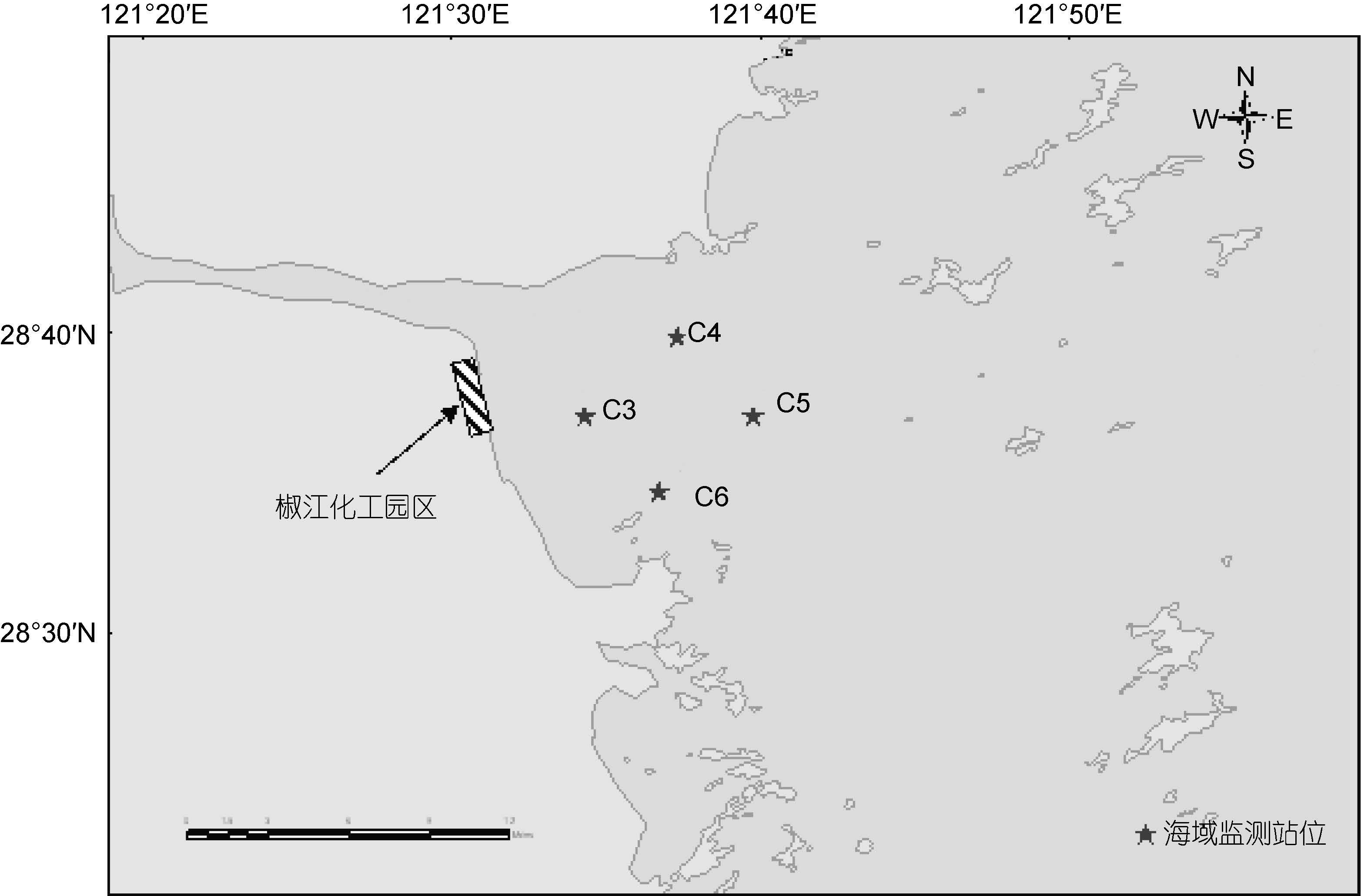
|
| 图 1 椒江口化工园区邻近海域采样站点 Fig. 1 Sampling station in the Jiaojiang chemical industrial park adjacent area |
参照海底沉积物化学分析方法测量常规水化学指标(Heijs et al,2008); 以总有机碳分析仪(TOC-V CSH,日本岛津)确定总有机碳含量(TOC); 以钼酸铵分光光度法(国标GB/T20260-200)确定总磷含量(TP);以蛋白质自动分析仪(SKALAR Primacs SN,荷兰)确定总氮含量(TN)。
1.3 可培养细菌的分离和鉴定 1.3.1 细菌分离纯化向锥形瓶中加入90 mL 已灭菌海水的(含玻璃珠)及10.0 g 沉积物样本,180 r/min 转速震荡30 min。静置后取上清液稀释1000 倍,涂布于Zobell 2216 培养基,25°C 恒温培养,待长出明显菌落后,挑取单个菌落并反复划线纯化菌株(何建瑜等,2013)。
1.3.2 细菌形态学鉴定菌株分离纯化后,首先观察并记录其菌落形态,并通过染色检测其革兰氏阴阳性,在确定是否生成芽孢后,进行以下实验(李振高等,2008):(1)革兰氏阳性无芽孢杆菌: 镜检菌体形态及其随培养时间的变化;(2)革兰氏阳性芽孢杆菌: 测定菌株与氧的关系;(3)革兰氏阴性无芽孢杆菌: 进行氧化酶实验;(4)菌体形态是球形者,要观察其幼龄及老龄菌体形态,确定其没有杆状菌。
1.3.3 细菌生理生化鉴定因为海洋中革兰氏阴性细菌数量远多于兰氏阳性细菌,所以使用法国梅里埃公司的API-20E(肠杆菌和其它革兰阴性杆菌科鉴定系统)及API-20NE(非肠道革兰阴性杆菌科鉴定系统)对所得革兰氏阴性菌进行鉴定。API-20E和API-20NE 能以其自身代谢产物的颜色变化或加入试剂后的颜色变化,对未知菌株加以鉴定,进行数据库检索后可得到相对应种名(黄备等,2009)。
1.3.4 细菌16S rDNA 基因扩增及鉴定挑取典型菌株,利用细菌基因组DNA提取试剂盒(TaKaRa 公司)提取所选菌株DNA,采用细菌16S rDNA 通用引物27F(5'-AGAGTTTGATCCTGGCTCAG-3')和1492R(5'-TACGGTTACCTTGTTACGACTT-3')进行50 μL 反应体系的PCR 扩增。具体为: dNTPs 0.5 μL,Taq 酶(TaKaRa)0.5 μL,Primer 1492R 1μL,Primer 27F 1μL,模板DNA 1 μL,10×buffer 5 μL,H2O41 μL。扩增条件为: 95°C,5 min; 94°C,1 min; 56°C,30 s; 72°C,2 min,循环35 次; 72°C,10 min。PCR 产物由上海美吉生物技术有限公司进行测序。将测序长度超过1300 bp 的序列于BLASTn(www.ncbi.nlm.nih.gov/BLAST/)中进行比对,记录相似度高于97%的序列信息。
1.4 非培养细菌多样性研究 1.4.1 环境总DNA 提取和16S rDNA 扩增对于沉积物环境总DNA,使用FastPrep 快速核酸提取仪(MP Biomedicals 公司)及其配套试剂盒(FastDNA spin kit for soil)进行提取。提取后的DNA 采用核酸蛋白检测仪(Bio-Rad 公司)鉴定其浓度及纯度,纯化后用于PCR 扩增,引物为341F(5'-CCTACGGGAGGCA GCAG-3')和907R(5'-CCGTCAATTCCTTTGAGTTT-3')。扩增体系同1.3.4。
1.4.2 非培养细菌克隆文库构建及序列分析将PCR 回收产物加入pMD-18T vector(TaKaRa 公司)中,16°C 连接过夜。在冰上将获得连接产物,转化到大肠杆菌(E. coli )DH5α 感受态细胞(TaKaRa 公司)中,经蓝白斑筛选,挑选阳性克隆,重新扩增后检测片段是否插入,选择片段长度合适的克隆送至上海美吉生物技术有限公司测序。
获得序列后,采用Usearch 软件筛选并去除嵌合体,后应用Clustal X 软件进行比对分析,再利用BLASTn 程序寻找相似序列,进行系统发育分析。使用PHYLIP 软件包中的DNASIS 程序计算距离矩阵,再应用DOTUR 将相似度大于97%的序列归为一个OTU,计算丰富度及多样性指数并绘制稀疏曲线(Schloss et al,2005)。采用Neighbor-Joining 建树方法,在MEGA 5 软件中建立系统发育树(Kumar et al,2004),建树结果经Bootstrap1000 次检验; 可培养菌株16S rDNA 的序列登录号为: KP016596-KP016699。非培养菌株16S rDNA 的序列登录号为: C3:KP016298-KP016387; C4: KP016388-KP016430; C5:KP016431-KP016488; C6: KP016489-KP016595。
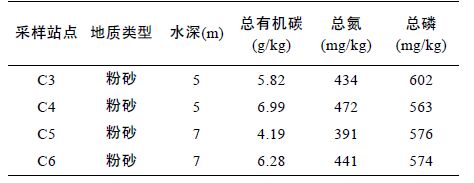
2 结果与分析2.1 样品环境化学指标椒江口化工园区邻近海域四个站点沉积物环境化学指标如表 1 所示。从表 1 可知,C3 为TP 含量最高的站点(602 mg/kg),TP 含量在四个站点排序为:C3>C5>C6>C4; C4 为四个站点中TOC 含量(6.99 g/kg)及TN 含量(472 mg/kg)最高的站点,各站点间TOC和TN 含量由高到低依次为为: C4>C6>C3>C5。
 |
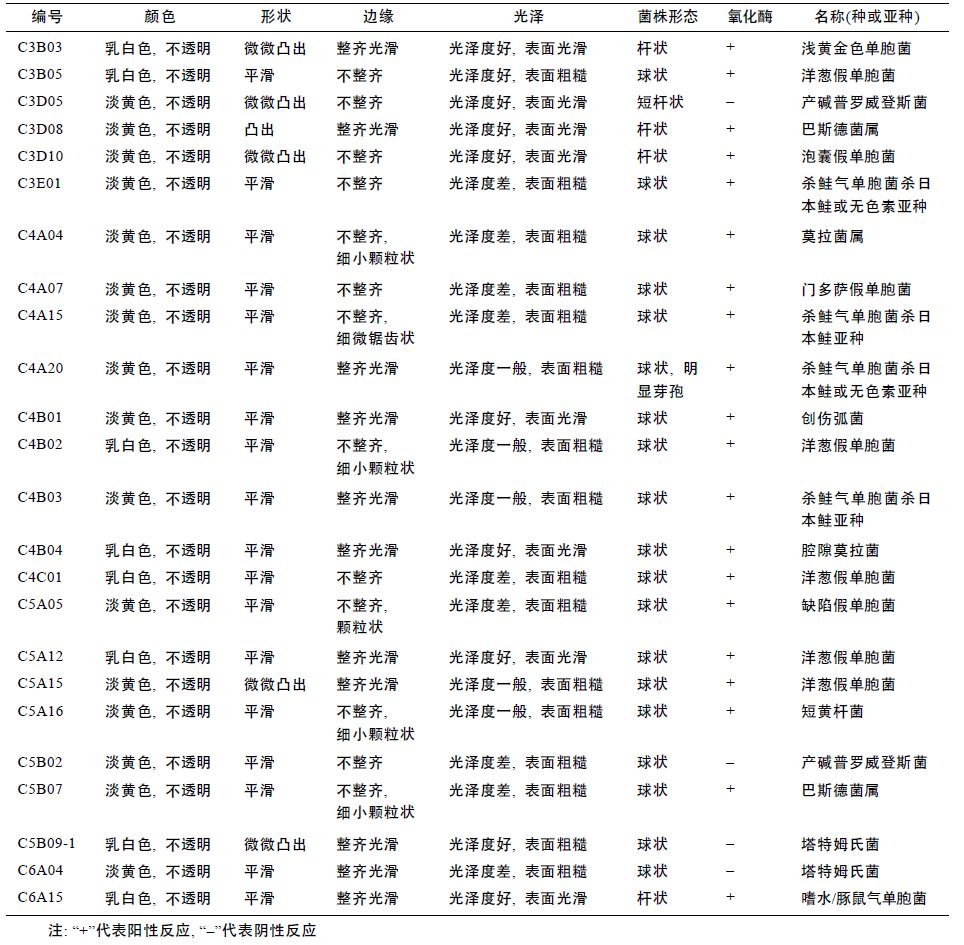
在获得的91 株可培养细菌中,革兰氏阴性菌占65.9%,革兰氏阳性菌占34.1%。显微镜下,球状为细菌的主要形态(64.8%); 且多数细菌氧化酶反应呈阴性(65.9%)。菌落仅存在乳白色和淡黄色(不透明),且比例相当; 菌落边缘的形态中整齐光滑菌落略多于不整齐菌落(53.8%和46.2%)。根据菌落形态及菌体特征,挑取24 株典型革兰氏阴性菌进行API-20NE和API-20E 鉴定,结果如表 2 所示。其中,变形菌门占95.83%,剩余一株为拟杆菌门的短黄杆菌,变形菌门中21.74%为洋葱假单胞菌(Pseudomonas cepacia),杀鲑气单胞菌杀鲑亚种(Aeromonas salmonicida ssp.salmonicida)也占变形菌门中的17.40%; 其余菌株所占比例较小,塔特姆氏菌(Tatumella ptyseos)、产碱普罗威登斯菌(Providencia alcalifaciens)、巴斯德菌属某些种(Pasteurella spp.)各占8.70%,浅黄金色单胞菌(Chryseomonas luteola)、嗜水气单胞菌(Aeromonas hydrophila)、腔隙莫拉菌(Moraxella lacunata)、莫拉菌属某些种(Moraxella spp.)、门多萨假单胞菌(Pseudomonas mendocina)、创伤弧菌(Vibrio vulnificus)、缺陷假单胞菌(Pseudomonas diminuta)、泡囊假单胞菌(Pseulormanas vesicularis)各占5.3%。
 |
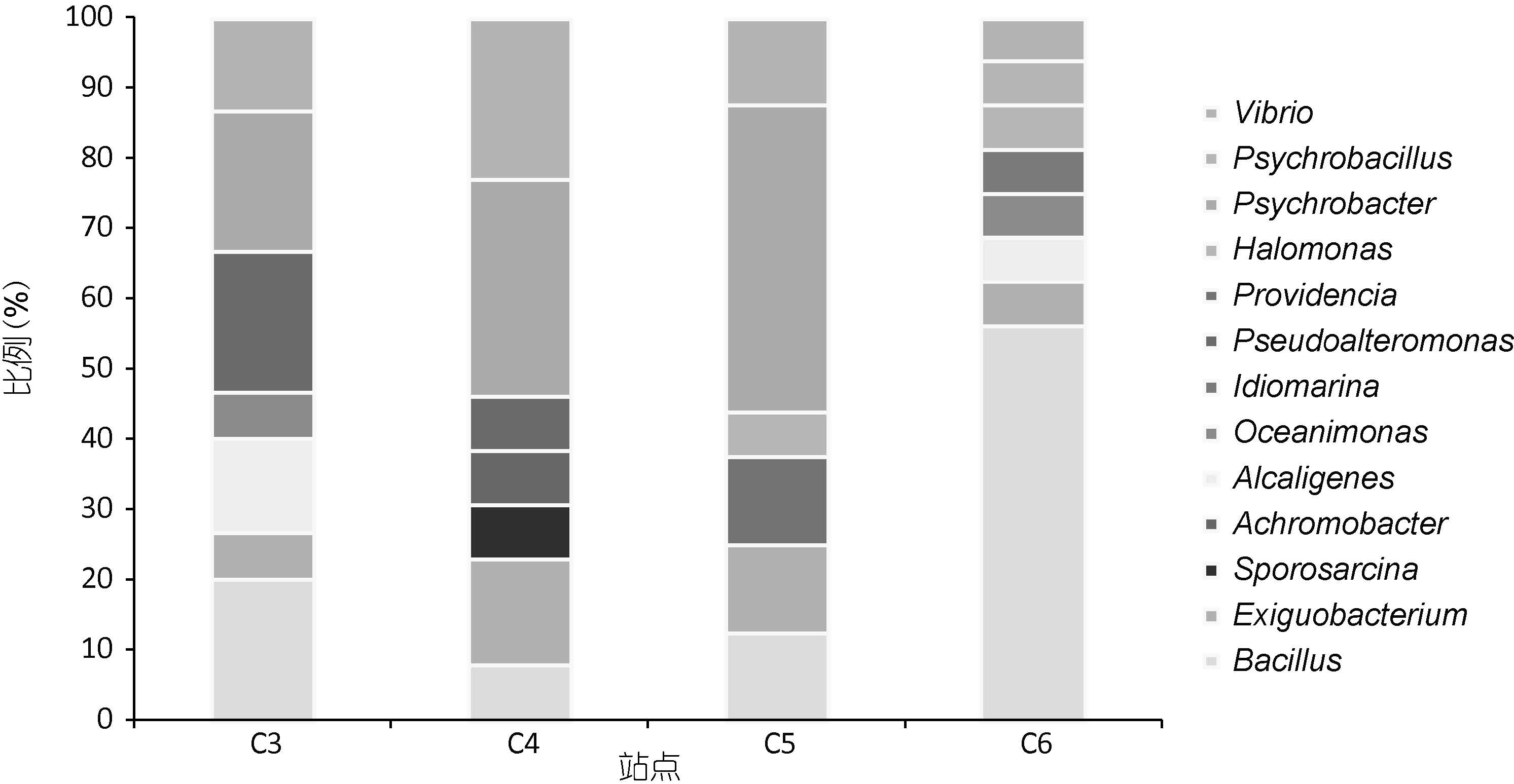
挑选62 株菌株进行测序获得60 个片段长约为1400bp 的有效序列。BLASTn 结果表示: 60 株菌株主要属于厚壁菌门(Firmicutes)、γ-变形菌纲(Gammaproteobacteria)和β-变形菌纲(Betaproteobacteria)。不同类群细菌所占比例如图 2 所示。
|
| 图 2 四个站点菌株的类群比例 Fig. 2 Ratio of groups from clone library of four stations |
微小杆菌(Exiguobacterium)、芽孢杆菌(Bacillus)和弧菌(Vibrio)在四个站点均有分布,但各站点所占比例不同,芽孢杆菌在C6 站点丰度最高(占56.25%),但在C4 站点只占6.67%。相对于C3和C5 而言,C4、C6 站点的多样性更丰富。另外芽胞八叠球菌(Sporosarcina)、无色杆菌(Achromobacter)只在C4 站点分布,海源菌(Idiomarina)、嗜冷芽胞杆菌(Psychrobacillus)只在C6 站点分布,而普罗威登斯菌(Providencia)只出现在C5 站点。
选取的60 个菌株中共含有34 个OTU,构建其系统发育树(如图 3 所示)。由图 3 可知,γ-变形菌纲多样性最为丰富,主要由嗜冷杆菌(Psychrobacter)(占所有菌株的23%)、弧菌(Vibrio)(占所有菌株的13%)、假交替单胞菌(Pseudoalteromonas)、盐单胞菌(Halomonas)及海单胞菌(Oceanimonas)构成,还有少数的普罗威登斯菌(Providencia)和海源菌(Idiomarina)。厚壁菌门多样性仅次于其γ-变形菌纲,芽孢杆菌(Bacillus)为厚壁菌门中的主要类群(占所有菌株的25%),还有部分微小杆菌(Exiguobacterium)(占所有菌株的10%)、嗜冷芽胞杆菌(Psychrobacillus)、和芽胞八叠球菌(Sporosarcina)。β-变形菌门仅有3 个OTU,分别属于氧化木糖无色杆菌(Achromobacter xylosoxidans)及粪产碱菌(Alcaligenes faecalis)。
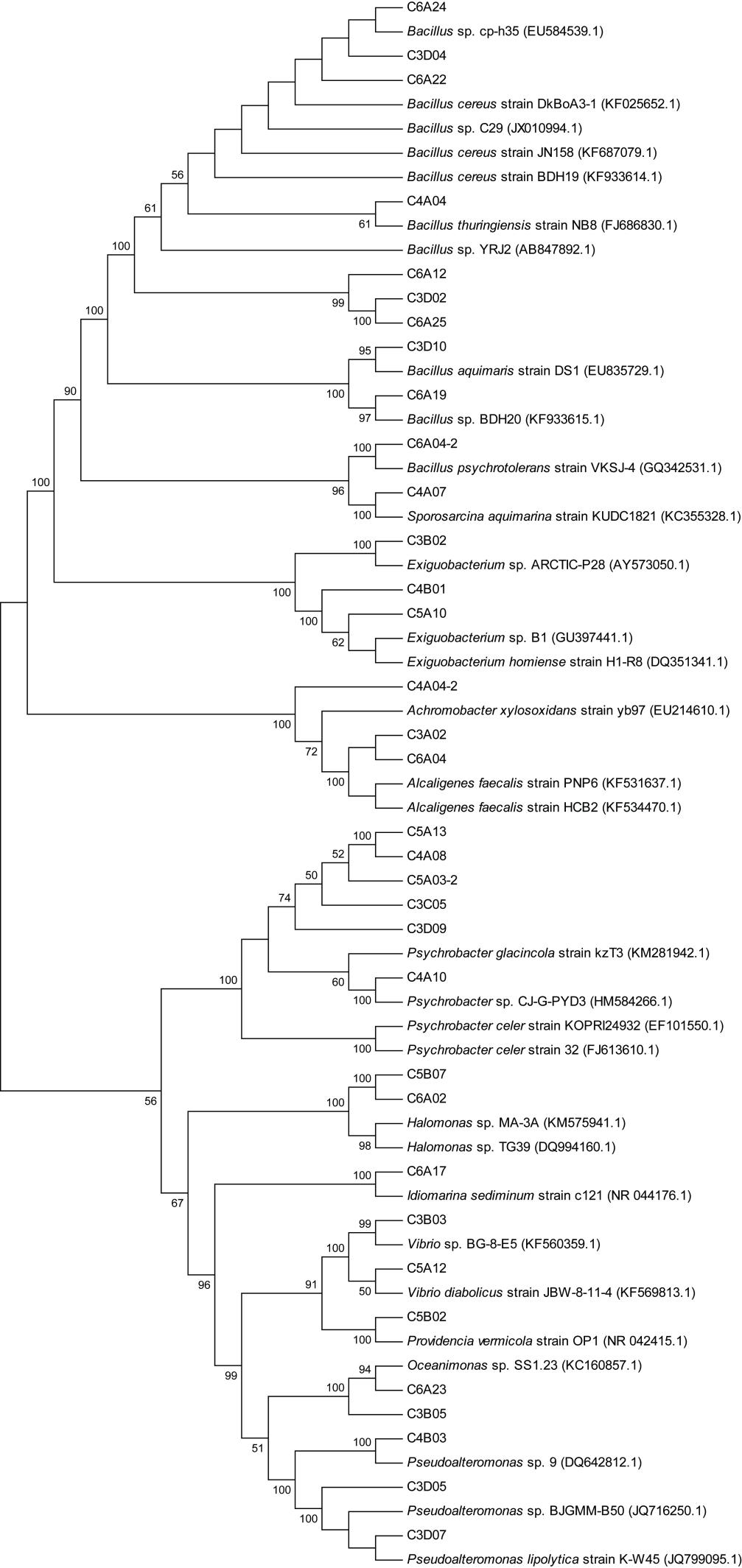
|
| 图 3 根据可培养细菌16S rDNA 基因序列构建的系统发育树 Fig. 3 Phylogenetic tree of culturable bacteria base on 16S rDNA gene sequences |
每个站点均选取约100 个细菌测序,4 个站点最终分别获得115、97、100、104 个有效克隆子,片段长度均为550—700bp; 相似度大于97%的克隆子归为一个OTU,C3、C4、C5和C6 四个站点的OTU 分别为35、60、38和72 个,并计算出四个站点克隆文库的多样性指数(如表 3 所示)。
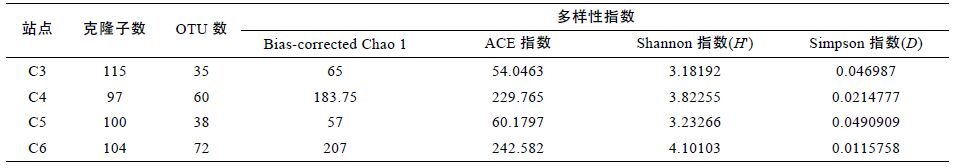 |
由表 3 可知,丰富度指数Chao1 与ACE 指数同时显示C6 站点拥有最高丰富度,C4 站点次之,但C3、C5 站点两个丰富度指数并不一致,Chao1 指数显示C3 站点拥有更高丰富度,但ACE 指数显示C5 站点拥有更高的丰富度。另一种丰富度估计——稀释性曲线(图 4)与ACE 指数所显示的结果一致,丰富度从高到低排列顺序为C6>C4>C5>C3。于多样性指数而言,Shannon 与Simpson 指数同时指示C6 站点多样性最高,C4 站点次之,但C3、C5 两站点的两种多样性指数的指示结果并不相符,Shannon 指数显示C5 站点拥有更高的多样性,而Simpson 指数显示C3 站点拥有更丰富的多样性。
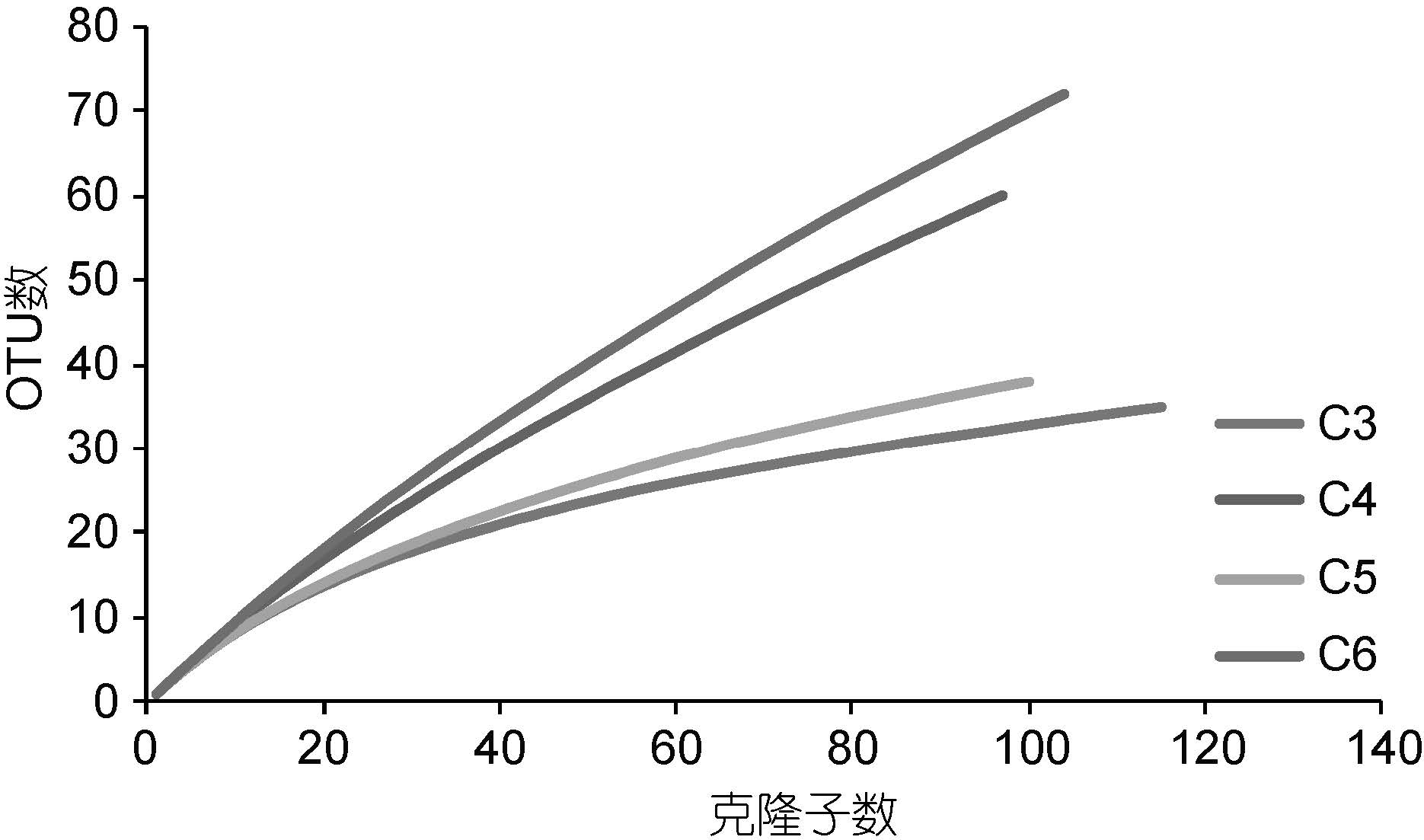
|
| 图 4 四个文库的稀释性曲线 Fig. 4 Rarefaction curve of four clone library |
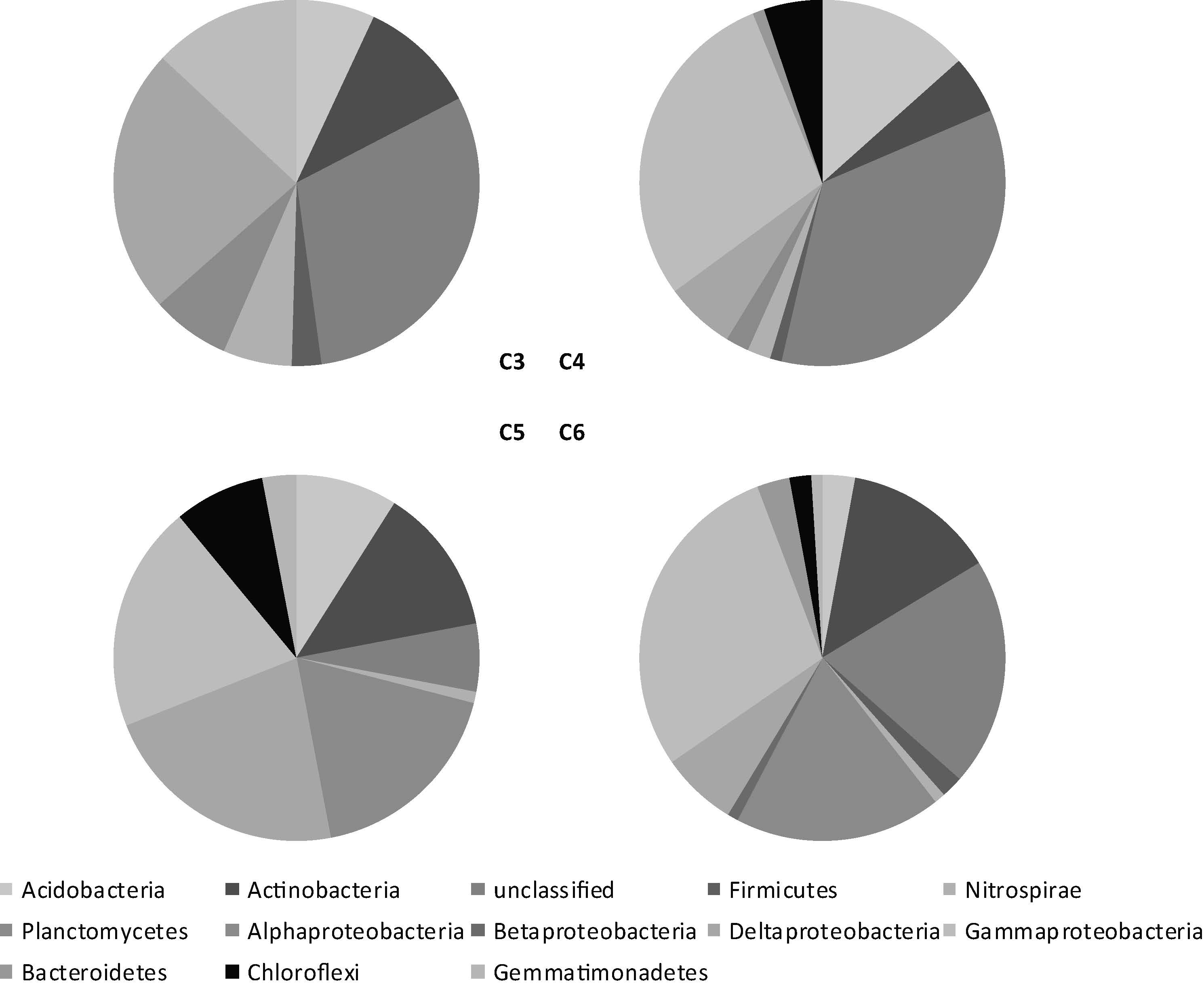
4 个站点共416 个克隆子经BLASTn 检索可分为9 个门(群),分别为变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、硝化螺旋菌门(Nitrospirae)、绿弯菌门(Chloroflexi)、厚壁菌门(Firmicutes)、芽单胞菌门(Gemmatimonadetes)、CFB群(Cytophaga-Flavobacterium-Bacterioides)、浮霉菌门(Planctomycetes); 另有23.1%的细菌分类地位不确定;这些相似序列主要来自于海洋或河流沉积物、海水或河水,少数来自土壤。
由图 5 可看出: 综合4 个站点文库,最大优势类群为变形菌门,占总文库的46.63%,其中,γ-变形菌占22.36%,δ-变形菌占14.90%,α-变形菌占9.13%,β-变形菌占0.24%; 仅次于变形菌门的是放线菌门(占10.58%); 第三大优势菌群为酸杆菌门(占7.93%)。
|
| 图 5 四个站点克隆文库的类群比例 Fig. 5 Ratio of groups from clone libraries of four stations |
于各站点而言: C3 站点不仅OTU 含量最低,其文库所含类群也最少,主要由变形菌门(38.26%)和放线菌门组成(10.43%); 变形菌门主要由δ-、γ-变形菌组成,分别占C3 文库的23.48%和13.04%,另有少量α-变形菌,δ-变形菌所占比例为4 个站点中最高。C4站点文库主要由变形菌门及酸杆菌门细菌构成,分别占37.11%及13.40%,变形菌门所占比例为4个站点最低;变形菌门以γ-变形菌为主要类群,占文库的28.87%,这高于其它3 个站点的γ-变形菌含量,变形菌门中还有少量δ-、α-变形菌,δ-变形菌所占比例为4 个站点中最低(6.19%)。C5 站点文库的主要类群也为放线菌门和变形菌门,分别占13%及57%,变形菌门含量高于剩余3 个站点; 变形菌门也由α-、δ-、γ-变形菌构成,但其分布明显较其它站点均匀,分别为15%、22%、20%。C6 站点文库同样以放线菌门和变形菌门为主要类群(13.46%和54.81%),其放线菌门含量为4 个站点中最高; 变形菌门中α-、γ-变形菌所占比例较高,分别为1 8 . 2 7 % 及2 8 . 8 5 %,其中α-变形菌含量高于其余3 个站点,此站点变形菌门也拥有其它站点没有的β-变形菌,但其含量很低仅占0.96%。
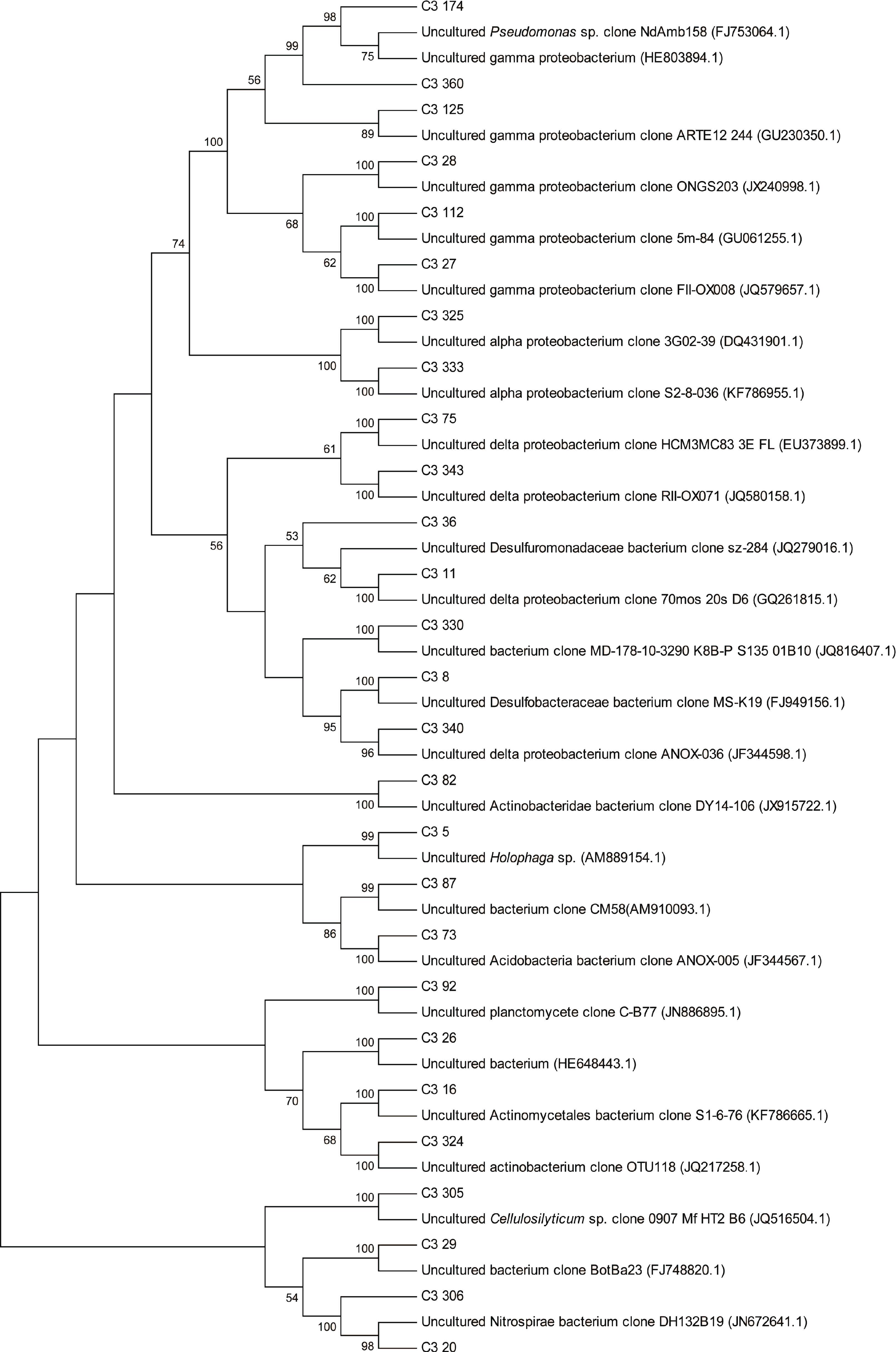
2.5 非培养细菌系统发育分析 2.5.1 C3 站点非培养细菌系统发育分析将C3站点OTU 及其BLASTn 结果建立系统发育树,结果如图 6。
|
| 图 6 C3 站点根据细菌16S rDNA 基因序列构建的系统发育树 Fig. 6 Phylogenetic tree of bacteria according to 16S rDNA gene sequences of station C3 |
由图 6 可知,C3 站点所含δ-变形菌纲最为丰富,基因型C3_8(6 个克隆子)与西班牙马略卡岛沉积物中对芳香烃污染有响应的脱硫杆菌科(Desulfobacteraceae)克隆子MS-K19 有高同源性(98%); 基因型C3_36(10 个克隆子)的相似序列为华北平原地下水中氯代脂肪烃迁移转化有关除硫单胞菌科(Desulfuromonadaceae)克隆子sz-284; 基因型C3_11(3 个克隆子)与蒙特利海底峡谷沉积物中的未培养δ-变形菌克隆子70mos_20s_D6 相似; 基因型C3_75(4 个克隆子)与希腊克里特岛边缘沉积物中未培养δ-变形菌克隆子HCM3MC83_3E_FL 相似度最高(97%); 基因型C3_340 与基因型C3_343(都只有1个克隆子)的相似序列分别为石油污染西班牙Cies 群岛潮下带沉积物中未培养δ- 变形菌克隆子ANOX-036和RII-OX071。γ-变形菌纲在C3 站点也有较多分布,克隆子C3_125(3 个克隆子)与波多黎各拉普拉塔河到南大西洋中未培养γ-变形菌克隆子ARTE12_244 相似; 基因型C3_28(3 个克隆子)与印度临近化工城市巴罗达的坎贝湾的海滨土壤中未培养γ-变形菌克隆子ONGS203 有较高同源性(99%); 基因型C3_112(5 个克隆子)与韩国黄海沿岸海水中未培养γ-变形菌克隆子5m-84 相似度为96%,同时此基因型还与北极Svalbard 海湾的未培养γ-变形菌克隆子SS08_052 相似,这可能表明此基因型分布广泛; 基因型C3_174和基因型C3_360(1 个克隆子)的相似序列都为污染的里沃纳港海床沉积物中未培养γ 变形菌克隆子147; 基因型C3_27(1 个克隆子)与被原油污染的西班牙Cies 群岛潮下带沉积物中未培养γ-变形菌克隆子FII-OX008 相似度为96%。α-变形菌仅有的2 个基因型C3_325、C3_333(都只有1 个克隆子),相似序列分别为墨西哥湾深水地平线区域的未培养α-变形菌克隆子S1-8-10和S2-8-036。
放线菌门在C3 站点文库中分布也比较丰富。基因型C3_16(2 个克隆子)与美国墨西哥湾深水地平线事故区域未培养放线菌克隆子S1-6-76 有较高同源性(99%); 基因型C3_82(4 个克隆子)与黄河三角洲沿岸湿地中未培养放线菌克隆子DY14-106 相似度为99%;基因型C3_324(4 个克隆子)的相似序列为网箱养殖影响的宁波南沙湾沉积物中未培养放线菌克隆子OTU118。
C3 站点还有一定数量的酸杆菌,基因型C3_5(5个克隆子)与突尼斯 Bizerte地区潟湖沉积物中未培养酸杆菌克隆子1'C70 相似; 基因型C3_73(2 个克隆子)与Cies 群岛潮下带沉积物中未培养酸杆菌克隆子ANOX-005 相似度最高(97%)。
在C3 站点的硝化螺旋菌仅属于两个基因型: C3_306(1 个克隆子)和C3_20(6 个克隆子),它们的相似序列都为表层东海沉积物中未培养硝化螺旋菌克隆子DH132B19。 厚壁菌门及放线菌门都只含一个OTU分别为基因型C3_305(3 个克隆子)和C3_92(6 个克隆子),前者与加勒比海Montastraea faveolata 珊瑚中未培养Cellulosilyticum 克隆子0907_Mf_HT2_B6 有较高同源性(97%),后者与西南印度洋脊沉积物中未培养浮霉菌目(Planctomycetales)克隆子C-B77 相似度为97%。
在分类地位不确定的细菌中克隆子中,基因型C3_330(2 个克隆子)与台湾可能含有天然气水合物区域未培养细菌克隆子MD-178-10-3290_K8B-P_S135_01B10 有高同源性(99%); 基因型C3_87(6 个克隆子)与水稻稻田中未培养细菌克隆子CM58 相似; 基因型C3_29(15 个克隆子)的相似序列为珠江口硫酸盐还原菌克隆子BotBa23; 与基因型C3_26(10 个克隆子)有较高同源性(98%)的序列为网箱养殖影响的象山港沉积物中未培养细菌克隆子SdBa81。
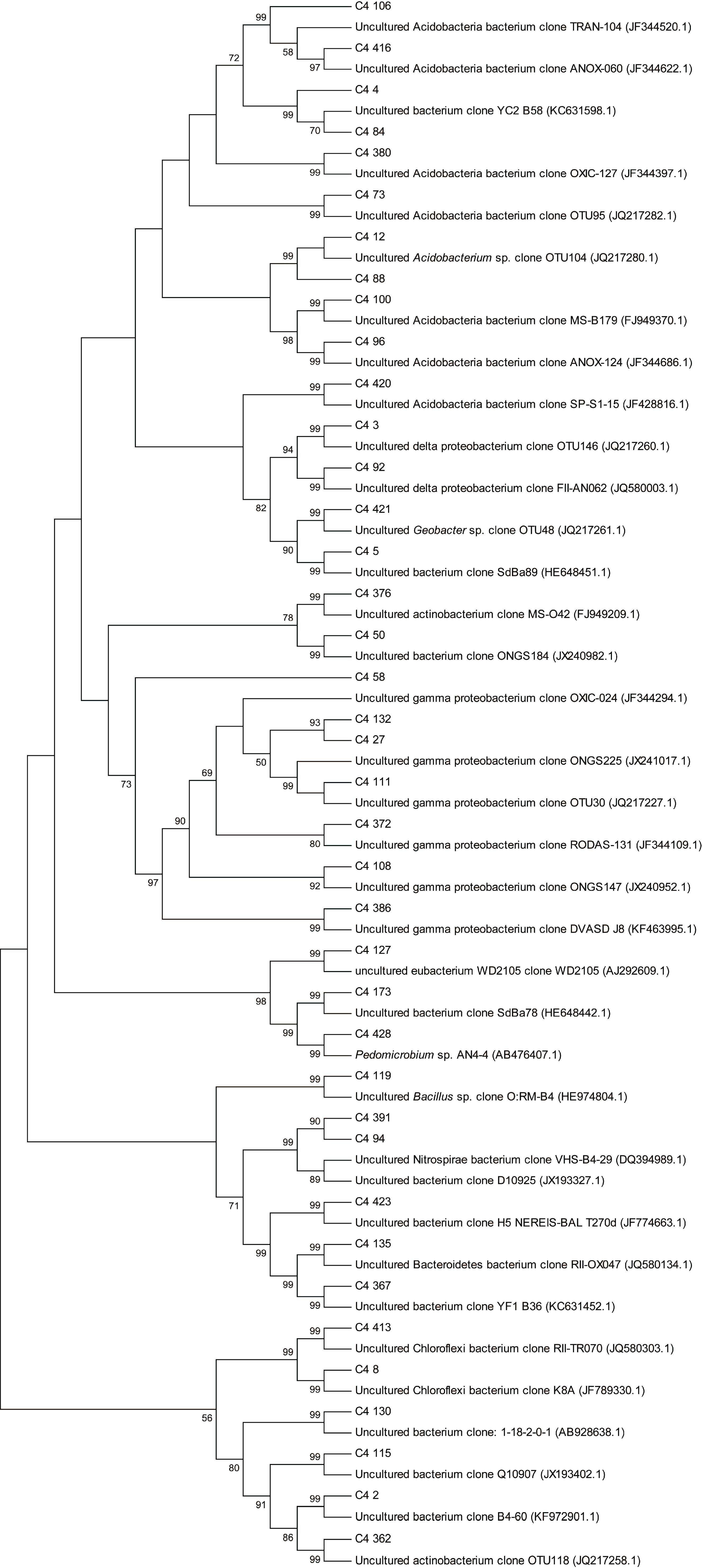
2.5.2 C4 站点非培养细菌系统发育分析将C4站点OTU 及其BLASTn 结果建立系统发育树,结果如图 7。
|
| 图 7 C4 站点根据细菌16S rDNA 基因序列构建的系统发育树 Fig. 7 Phylogenetic tree of bacteria according to 16S rDNA gene sequences of station C4 |
由图 7 可知,γ-变形菌纲为C4 站点最主要的类群,基因型C4_27(10 个克隆子)和基因型C4_372(1 个克隆子)的相似序列分别为Cies 群岛潮下带沉积物中未培养γ-变形菌克隆子OXIC-024和RODAS-131; 基因型C4_132(6 个克隆子)、基因型C4_108(2 个克隆子)、基因型C4_58(3 个克隆子)的相似序列都来自印度坎贝湾的海滨土壤,前者与此地未培养γ-变形菌克隆子ONGS225 有较高同源性(98%),后两者与此地未培养γ- 变形菌克隆子ONGS147 同源性最高; 基因型C4_111(2 个克隆子)的相似序列为网箱养殖影响的宁波南沙湾沉积物中未培养γ 变形菌克隆子OTU30; 基因型C4_386(1 个克隆子)与印度Alang-Sosiya 拆船厂未培养γ-变形菌克隆子DVASD_J8 相似度为96%。δ-变形菌纲中,基因型C4_3(2 个克隆子)和基因型C4_421(1 个克隆子)相似序列分别为来自网箱养殖影响的宁波南沙湾沉积物中未培养δ-变形菌克隆子OTU146和OTU48; 基因型C4_92(1 个克隆子)与Cies群岛潮下带沉积物中未培养δ- 变形菌克隆子FII-AN062 有高同源性(99%)。α-变形菌在C4 站点分布很少,基因型C4_428(1 个克隆子)的相似序列为能降解阿特拉津除草剂微生物群落中的土微菌属(Pedomicrobium)克隆子AN4-4。
C4 站点还分布有较为丰富的酸杆菌,基因型C4_12(2 个克隆子)、基因型C4_88(1 个克隆子)、基因型C4_73(2 个克隆子)的相似序列分别为网箱养殖影响的宁波南沙湾沉积物中未培养酸杆菌克隆子OTU104(前2 个基因型)和OTU95; 基因型C4_420(1个克隆子)与虾塘沉积物中未培养酸杆菌克隆子SP-S1-15 同源性为92%; 基因型C4_380、C4_416、C4_106、C4_96(都仅一个克隆子),它们的相似序列分别为Cies 群岛潮下带沉积物中未培养酸杆菌克隆子OXIC-127、ANOX-060、TRAN-104、ANOX-124;基因型C4_100(1 个克隆子)与人为石油污染西班牙马略卡岛海岸沉积物中未培养酸杆菌克隆子MS-B179有高同源性(99%)。
C4 站点还有一些其它基因型,基因型C4_362(1个克隆子)相似序列与基因型C3_324 一致; 基因型C4_376(1 个克隆子)与芳香烃污染西班牙马略卡岛钙砂质沉积物中未培养放线菌克隆子MS-O42 有较高同源性(97%); 基因型C4_135(1 个克隆子)与基因型C4_413(1 个克隆子)相似序列分别为石油污染西班牙Cies 群岛潮下带沉积物中未培养拟杆菌克隆子RII-OX047 及未培养绿弯菌克隆子RII-TR070; 基因型C4_8(1 个克隆子)与三文鱼养殖海底沉积物中未培养绿弯菌克隆子K8A 相似。基因型C4_391(2 个克隆子)与香港被污染维多利亚港沉积物中未培养硝化螺旋菌克隆子VHS-B4-29 有较高同源性(98%)。基因型C4_119(1 个克隆子)与匈牙利奥伊考被污染赤泥中未培养芽孢杆菌(Bacillus)克隆子O: RM-B4 同源性为99%。
在C4 站点分类地位未明确的细菌中,基因型C4_130(1 个克隆子)相似序列为日本富营养霞浦湖沉积泥中未培养细菌克隆子1-18-2-0-1; 基因型C4_367(1 个克隆子)、基因型C4_84(1 个克隆子)、基因型C4_4(3 个克隆子)相似序列分别为韩国南岸岩鱼、海鲷养殖沉积泥中未培养细菌克隆子YF1_B36、YC2_B58(后两者); 基因型C4_423(1 个克隆子)与法国原油污染Aber-Benoît 潮汐港湾中未培养细菌克隆子H5 NEREIS-BAL T270d 相似; 基因型C4_2(2 个克隆子)与虾养殖系统中未培养细菌克隆子B4-60 有高同源性(99%); 基因型C4_115(1 个克隆子)、C4_94(1个克隆子)相似序列分别为来自秦皇岛和大连受海洋养殖影响的潮间带沉积物中未培养细菌克隆子Q10907和D10925; 基因型C4_50(6 个克隆子)与印度临近化工城市巴罗达的坎贝湾的海滨土壤未培细菌克隆子ONGS184 同源性为96%; 基因型C4_127(1个克隆子)的相似序列为德国威腾伯格市多氯联苯污染土壤中未培养细菌克隆子WD2105; 基因型C4_173(1 个克隆子)、基因型C4_5(4 个克隆子)相似序列分别为象山港网箱养殖影响沉积物中未培养细菌克隆子SdBa78、SdBa89。
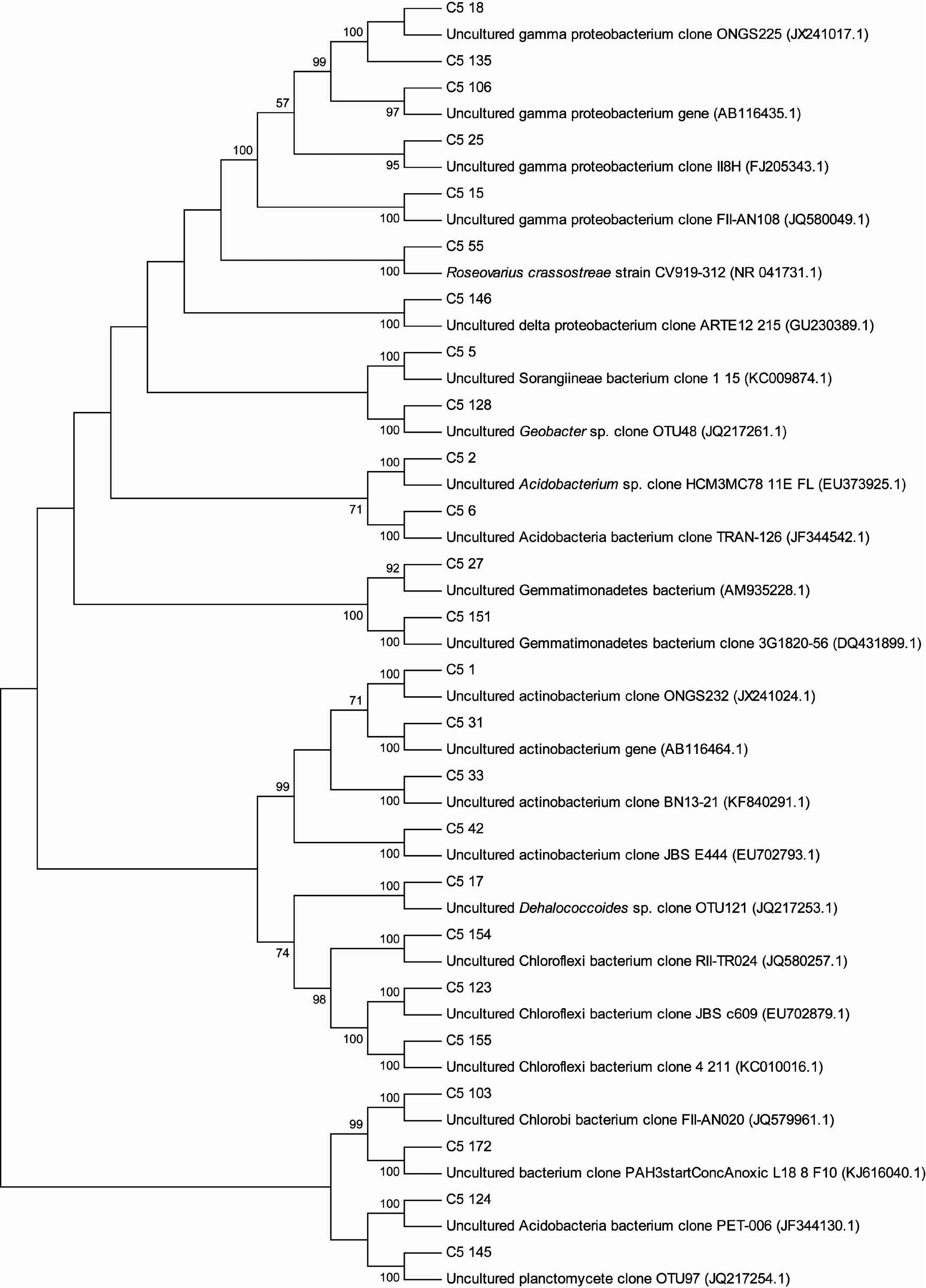
2.5.3 C5 站点非培养细菌系统发育分析将C5站点OTU 及其BLASTn 结果建立系统发育树,结果如图 8。
|
| 图 8 C5 站点根据细菌16S rDNA 基因序列构建的系统发育树 Fig. 8 Phylogenetic tree of bacteria according to 16S rDNA gene sequences of station C5 |
由图 8 可知,变形菌门的α-、δ-、γ-变形菌也是C5 站点的主要优势类群。其中,α-变形菌纲中多数克隆子属于基因型C5_55(14 个克隆子),其相似序列为养殖的东方生蚝中致病玫瑰变色菌属(Roseovarius)克隆子CV919-312。δ-变形菌纲中,基因型C5_128(4 个克隆子)与网箱养殖影响的宁波南沙湾沉积物中未培养地杆菌属(Geobacter)克隆子OTU48 有高同源性(99%); 基因型C5_146(3 个克隆子)与波多黎各拉普拉塔河到南大西洋中未培养δ 变形菌克隆子ARTE12_215 相似; 基因型C5_5(13 个克隆子)相似序列为法国Guiana 海岸与二氧化碳固定有关未培养Sorangiineae 科克隆子1_15。γ-变形菌纲中,基因型C5_106(5 个克隆子)与日本三陆密集养殖贝类海岸沉积泥中未培养γ-变形菌克隆子Y182 有高同源性(99%); 基因型C5_15(2 个克隆子)与Cies 岛潮下带沉积物中未培养γ-变形菌克隆子FII-AN108 相似; 基因型C5_18(1 个克隆子)与基因型C5_135(6 个克隆子)相似序列分别为印度坎贝湾海滨土壤中未培养γ-变形菌克隆子ONGS225、ONGS225; 基因型C5_25(4个克隆子)与东Lau 盆地深海热液中未培养γ-变形菌克隆子II8H 相似。
放线菌门在C5 站点分布仅次于变形菌门,基因型C5_33(2 个克隆子)与北极海洋沉积物中未培养放线菌克隆子BN13-21 相似度为99%; 基因型C5_31(3个克隆子)与日本三陆密集养殖贝类海岸沉积泥中未培养放线菌克隆子Y8 有高同源性(99%); 基因型C5_42(5 个克隆子)相似序列为胶州湾水底未培养放线菌克隆子JBS_E444; 基因型C5_1(2 个克隆子)与印度坎贝湾海滨土壤中未培养放线菌克隆子ONGS232 有较高同源性(98%)。
C5 站点还有一定数量的酸杆菌,基因型C5_2(2个克隆子)与希腊克里特岛边缘沉积物中酸杆菌属(Acidobacterium)未培养克隆子相似; 基因型C5_124(1 个克隆子)与基因型C5_6(5 个克隆子)相似序列分别为西班牙Cies 群岛石油污染潮下带沉积物中未培养酸杆菌克隆子PET-006、TRAN-126。
其它类群在C5 站点也有分布。基因型C5_103(2个克隆子)与基因型C5_154(1 个克隆子)相似序列分别为西班牙Cies 群岛石油污染潮下带沉积物中未培养绿菌克隆子FII-AN020 及RII-TR024。基因型C5_17(1 个克隆子)与宁波南沙湾网箱养殖影响沉积物中未培养Dehalococcoides 克隆子OTU121 有高同源性(99%); 基因型C5_123(2 个克隆子)与胶州湾水底未培养绿弯菌克隆子JBS_c609 相似度为99%; 基因型C5_155(2 个克隆子)相似序列为法国Guiana 海岸与二氧化碳固定有关未培养绿弯菌克隆子4_211。基因型C5_151(2 个克隆子)与佛罗里达圣乔治岛海洋沉积物中未培养芽单胞菌克隆子3G1820-56 有高同源性(99%); 基因型C5_27(1 个克隆子)的相似序列来自修复脂肪族烃污染土壤群落中未培养芽单胞菌克隆子AMFC3。基因型C5_145(2 个克隆子)与网箱养殖影响的宁波南沙湾沉积物中未培养浮霉菌克隆子OTU97 同源性为95%。
在C5 站点分类地位未明确的细菌中,基因型C5_172(1 个克隆子)与波罗的海锰铁结核及沉积物中可能有能力降解原油和PAHs 的未培养细菌克隆子PAH3startConcAnoxic_L18_8_F10 有高同源性(99%)。
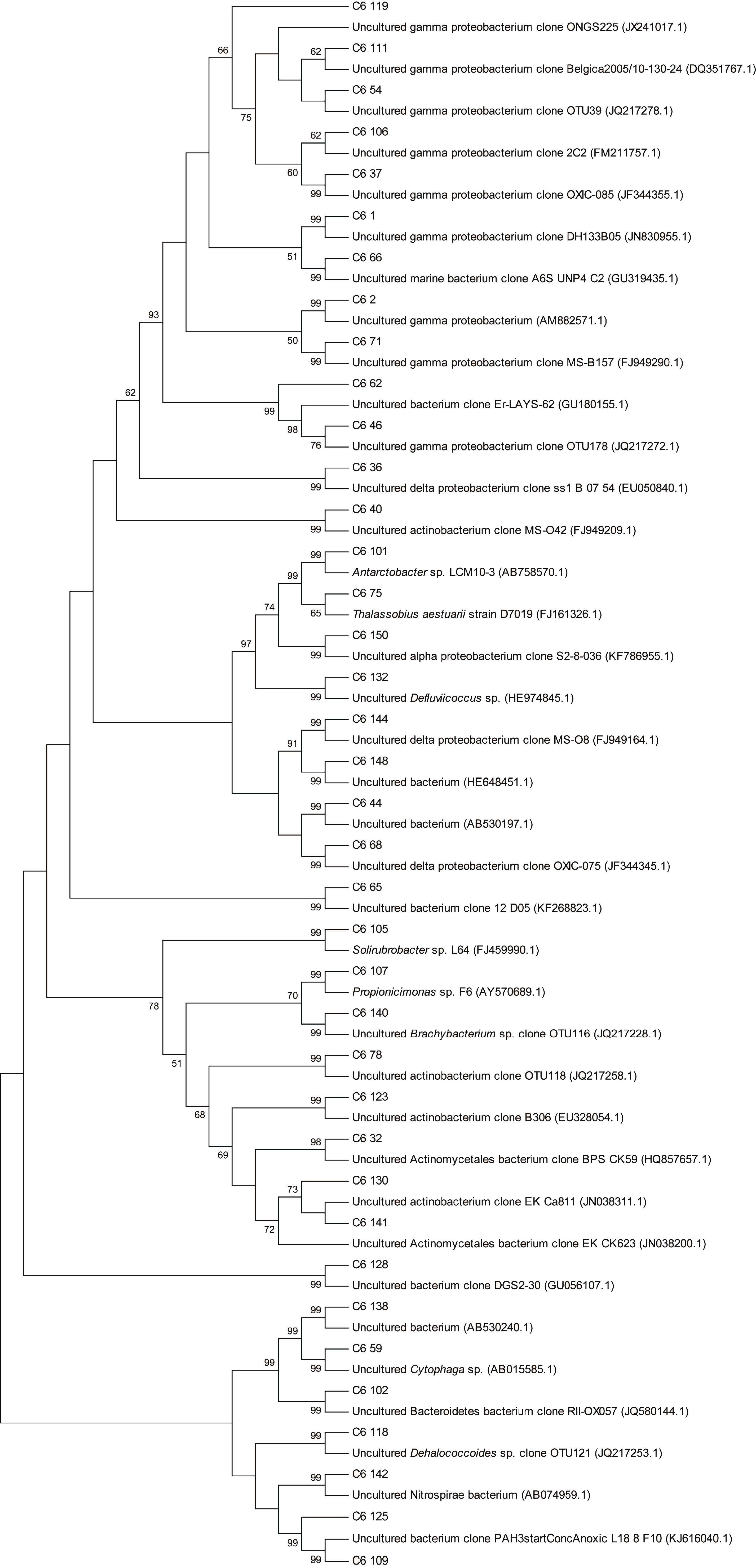
2.5.4 C6 站点非培养细菌系统发育分析将C6站点OTU 及其BLASTn 结果建立系统发育树,结果如图 9。
|
| 图 9 C6 站点根据细菌16S rDNA 基因序列构建的系统发育树 Fig. 9 Phylogenetic tree of bacteria according to 16S rDNA gene sequences of station C6 |
由图 9 可知,C6 站点主要优势种群为变形菌门中γ-变形菌纲及α-变形菌纲。γ-变形菌纲中,基因型C6_1(7 个克隆子)与表层东海沉积物中γ-变形菌克隆子DH133B05 相似度为98%; 基因型C6_2(4 个克隆子)与法国长期石油污染Berre 潟湖沉积物中未培养γ-变形菌克隆子46 EDB3 有高同源性(99%); 基因型C6_71(3 个克隆子)相似序列为存在原油溢出的西班牙马略卡岛海岸沉积物中未培养γ-变形菌克隆子MS-B157; 基因型C6_46(2 个克隆子)与基因型C6_119(1 个克隆子)相似序列分别为宁波南沙湾网箱养殖影响沉积物中未培养γ 变形菌克隆子OTU178、OTU39; 基因型C6_37(2 个克隆子)与Cies 群岛的潮下带沉积物中未培养γ-变形菌克隆子OXIC-085 有较高同源性(98%); 基因型C6_54(5 个克隆子)与印度坎贝湾海滨土壤中未培养γ-变形菌克隆子ONGS225 相似度为99%; 基因型C6_106(2 个克隆子)相似序列为突尼斯 Bizerte 地区潟湖沉积物中未培养 γ-变形菌克隆子2C2; 基因型C6_111(1 个克隆子)与重金属污染比利时大陆板块沉积物中未培养γ-变形菌克隆子Belgica2005/10-130-24 相似度为98%。α-变形菌纲中,基因型C6_150(1 个克隆子)相似序列与基因型C3_333 一致; 基因型C6_101(4 个克隆子)与红藻条斑紫菜原生质体中分离的Antarctobacter 克隆子LCM10-3 有高同源性(99%); 基因型C6_75(2 个克隆子)与山东海岸可培养Thalassobius aestuarii 克隆子D7019 相似度为98%; 基因型C6_132(2 个克隆子)相似序列为匈牙利奥伊考污染赤泥中未培养Defluviicoccus 克隆子O: RM-G8。变形菌门中δ-变形菌在C6 站点也有分布,基因型C6_68(1 个克隆子)与石油污染西班牙Cies 群岛潮下带沉积物中δ-变形菌克隆子OXIC-075 相似度为99%; 基因型C6_36(3 个克隆子)相似序列为北极Svalbard 孔斯峡湾沉积物中未培养δ- 变形菌克隆子ss1_B_07_54; 基因型C6_144(1 个克隆子)与芳香烃污染西班牙马略卡岛钙砂质沉积物中未培养δ-变形菌克隆子MS-O8 相似度为99%。
放线菌门在C6 站点的分布仅次于变形菌门,基因型C6_32(2 个克隆子)与石油烃污染盐碱土壤中未培养放线菌目(Actinomycetales)克隆子BPS_CK59 有较高同源性(98%); 基因型C6_107(1 个克隆子)相似序列为加拿大低温低盐石油储存层水中产丙酸单孢菌属(Propionicimonas)克隆子F6; 基因型C6_141(1个克隆子)与石油污染土壤中放线菌目(Actinomycetales)克隆子EK_CK623 相似度为97%;基因型C6_140(1 个克隆子)和基因型C6_78(3 个克隆子)相似序列分别为网箱养殖影响的宁波南沙湾沉积物中未培养短状杆菌(Brachybacterium)克隆子OTU116 及未培养放线菌克隆子OTU118; 基因型C6_40(1 个克隆子)的相似序列与基因型C4_376 一致;基因型C6_123(1 个克隆子)与原油污染土壤中未培养放线菌克隆子B306 有较高同源性(97%); 基因型C6_130(1 个克隆子)相似序列为石油污染土壤中未培养放线菌克隆子EK_Ca811; 基因型C6_105(1 个克隆子)与TPH 污染土壤中可培养Solirubrobacter 克隆子L64 有高相似度(99%)。
还有一些其它类群也在C6 站点中有一定分布,基因型C6_59(2 个克隆子)相似序列为日本深海沉积物中未培养噬细胞菌(Cytophaga)克隆子BD7-10; 基因型C6_102(1 个克隆子)序列与石油污染西班牙Cies群岛潮下带沉积物中未培养拟杆菌克隆子RII-OX057 相似。基因型C6_118(1 个克隆子)与基因型C5_17 的相似序列一致。基因型C6_142(1 个克隆子)相似序列来自日本久慈贮油腔层水中未培养硝化螺旋菌克隆子KB4。
在C6 站点分类地位未明确的细菌中,基因型C6_62(1 个克隆子)与多氯联苯影响的厌氧河口沉积物中未培养细菌克隆子Er-LAYS-62 有较高同源性(97%); 基因型C6_66(2 个克隆子)序列与以色列埃拉特Acropora eurystoma 珊瑚上未培养细菌克隆子A6S_UNP4_C2 相似; 基因型C6_148(1 个克隆子)与基因型C4_5 的相似序列一致; 基因型C6_128(2 个克隆子)相似序列为中国Ban 876 石油天然气田土壤中未培养细菌克隆子DGS2-30; 基因型C6_65(2 个克隆子)与重金属污染海洋沉积物中未培养细菌克隆子12_D05 相似度为99%; 基因型C6_109(1 个克隆子)、基因型C6_125(1 个克隆子)相似序列都为波罗的海锰铁结核及沉积物中可能有能力降解原油和PAHs 未培养细菌克隆子PAH3startConcAnoxic_L18_8_F10; 基因型C6_44、和C6_138(都仅1 个克隆子),分别与被污染的自苏伊士湾及东京湾表层沉积物中未培养细菌克隆子Suez.16S.Bac.29和Tokyo.16S.Bac.40 同源性为99%。
3 讨论3.1 可培养细菌多样性近年许多研究针对海洋沉积物中可培养细菌展开,Gontang等(2007)就西太平洋群岛潮间带沉积物可培养细菌的多样性研究发现,该区域以放线菌(Actinobacteria)和芽孢杆菌(Bacilli)为主要优势菌群,分别占65.6%和34.4%; 何建瑜等(2013)发现东海表层沉积物中,可培养细菌主要由厚壁菌门(57.5%)和γ-变形菌纲(32.9%)组成; 刘玉娟等(2014)在对南海沉积环境可培养细菌多样性的研究中也发现厚壁菌门在此区域占主导地位(54.5%); 郑莹等(2013)对浙江镇海潮间带沉积物中可培养微生物中研究发现,厚壁菌门占到菌株总量的51.3%。上述研究从一定程度上证明厚壁菌门为广泛海域沉积物中可培养细菌优势菌群,同时还有大量实验结果报道了γ-变形菌纲也是沉积物可培养细菌中的优势类群(霍颖异等,2008;卢婧雯等,2012; 李昭,2013)。本实验对可培养细菌的研究结果(γ-变形菌纲与厚壁菌门占43.33%和50%)与孙风芹等(2008)对南海南沙海域沉积物中可培养微生物多样性的研究结果(γ-变形菌纲与厚壁菌门分别占38.2%和43.6%)类似,也印证了上述结论。
厚壁菌门中的芽孢杆菌是本实验纯培养菌株中最丰富的菌群(占所有菌株的25%),其为好氧异养细菌,一般存在于有机质较为丰富的环境(孙风芹等,2008),而本实验中所测得的较高总碳、总氮、总磷含量也证实了这一观点。γ-变形菌纲中的嗜冷杆菌在纯培养菌株中分布仅次于芽孢杆菌(占所有菌株的23%),其OTU 的相似序列大多分离自韩国裙带菜(Undaria pinnatifida)。此次,实验中还发现某些OTU的相似序列为降解石油烃的细菌,如C4A04和C3C05 两株菌株。
3.2 非培养细菌多样性物种多样性不仅能表征群落结构复杂性,也是评价群落质量的重要指标,具有重要的生态学意义(马克平,1993)。在大多数有关微生物的实验中,作者习惯采用ACE与Chao1(或选取其中一个)作为丰富度指数,且ACE 指数常与Chao1 指数呈正比; Simpson与Shannon(或选取其中一个)作为多样性指数,且Simpson 指数常与Shannon 指数呈反比。但在本实验中,C3、C5 站点的丰富度指数与多样性指数所指示结果并不一致。对于丰富度指示结果的不同,是源于两种指数的算法的差异,在Chao1 指数算法中,一旦某个种有2 个及以上个体,就认为此物种名录是完整的,估计量不会再继续增加(Coddington et al,1996),而ACE指数算法则认为超过10 个才为优势种(Magurran,2011)。在本实验中C3、C5 站点的OTU 中常包含10个以上的克隆子,ACE 指数应比Chao1 指数更加可信,稀释曲线指示结果也与ACE 指数指示结果相同。至于多样性指示结果的不同,可能是因为Simpson 指数对于富集种更加敏感,而Shannon 指数对于稀少种更为敏感的原因(Magurran,1988)。
近年有关河口及附近海域非培养细菌多样性研究方兴未艾,国外研究主要集中在海水中细菌多样性方面(Crump et al,1999; Henriques et al,2006; Kan et al,2006),对沉积物中细菌的研究一般只针对某些特定菌群(Bernhard et al,2005; Beman et al,2006;Mosier et al,2008),相比之下,国内有更多关于河口沉积物细菌多样性的报道: 姜丽晶等(2007)、王新新等(2008)、郭建丽等(2013)分别对珠江口、长江口、双台子河口沉积物细菌多样性进行了研究,结果都发现δ-变形菌纲为这些地区的最大优势菌群。与以上结果不同,本实验中最大优势类群为γ- 变形菌(22.36%),其次才为δ-变形菌(14.90%),这与许多海洋沉积物细菌研究的结果相似(Bowman et al,2003;Inagaki et al,2003; Webster et al,2006)。这可能由于海域来沙影响了椒江口邻近海域沉积物(谢钦春等,1998),而其它河口主要受流域来沙影响(林承坤,1984; 江四义等,2008)。
γ-变形菌与δ-变形菌分别为本次实验最大的优势类群,它们虽然都为变形菌门,但其代谢类型与生态地位不尽相同。γ-变形菌在水体沉积环境中分布广泛,是许多物质代谢的重要组成,如厌氧情况下氨、硫的代谢(Freitag et al,2003; 白洁等,2009)。相比γ-变形菌参与各种物质的循环,δ-变形菌主要通过在厌氧条件下还原硫酸盐的SRB,参与硫酸盐还原代谢,而SRB 是参与有机物矿化与污染物降解的重要物质(Jørgensen,1982)。变形菌门中α-变形菌纲分布略少于γ-、δ-变形菌纲,α-变形菌大多数也参与低价硫化合物的代谢,常以H2S 作为代谢中的电子供体(李涛等,2008)。椒江口化工园区临近海域中丰富的变形菌门分布可能暗示了此处含有丰富的硫化合物。放线菌门含量仅次于变形菌门,Bull等(2005)对历年海洋沉积物放线菌含量数据的整理中发现,在深海及半深海地区放线菌一般为可培养细菌中的优势类群,但由于对海洋放线菌研究较少,其生态地位还不能确定。除以上菌群外,本实验还检测到丰富的酸杆菌门,虽然海洋酸杆菌研究较少,但Barns等(2007)对水下沉积物中酸杆菌含量数据整理中发现,由于其耐酸、重金属污染等特性,导致其在污染、极端环境中广泛分布。
3.3 细菌多样性与环境适应性分析系统发育树中相似序列所处环境,有助于更好地了解微生物与环境间的关系(Clementino et al,2007)。对比椒江口上游沉积物中细菌相似序列(蒋然等,2015),椒江口化工园区临近海域沉积物主要来自石油烃污染环境,如: 石油污染的西班牙Cies 群岛的潮下带沉积物(36 个克隆子),石油污染的西班牙马略卡岛(13 个克隆子),美国墨西哥湾深水地平线事故区域(5 个克隆子)等地区; 而来自重金属污染环境的相似序列较椒江口上游少,仅4 个克隆子。同时椒江口化工园区临近海域中还有大量相似序列来自养殖环境,如: 网箱养殖影响的宁波南沙湾沉积物(30 个克隆子)、网箱养殖影响的象山港沉积物(16 个克隆子)、日本三陆密集养殖贝类海岸沉积物(8 个克隆子)、韩国南岸岩鱼、海鲷养殖沉积物(5 个克隆子)、秦皇岛和大连受海洋养殖影响的潮间带沉积物(2 个克隆子)、虾养殖系统(2 个克隆子)、虾塘沉积物(1 个克隆子)、三文鱼养殖的海底沉积物(1 个克隆子)。对这些相似序列来自养殖环境的克隆子进行统计,我们可以发现,它们主要来自C4 站点,其原因可能是因为C4 站点位于本次采样的最北面,临近三门县渔业养殖基地。三门县地处浙东沿海三门湾畔,可养面积30 万余亩,为全省水产养殖强县,水产养殖品种有蟹、虾、鱼、贝、藻五大类30 多种,是台州市的重要养殖基地。三门县大量的滩涂、围塘养殖可能对其附近海域也造成了一定影响,导致位于其南面海域的C4 站点分布较多养殖环境相关序列。
4 结论本实验采用分离纯化、鉴定和建立克隆文库的方法,分别分析了椒江口化工园区邻近海域四个站点沉积物中细菌的群落结构及多样性,研究发现: 于可培养细菌,分子鉴定较生理生化鉴定方法更加准确,厚壁菌门为四个站点的优势菌群; 于非培养细菌,C6站点多样性最为丰富,γ-变形菌为四个站点的最大优势类群; 将系统发育树上的基因型与相似序列比较,除相当数量的相似序列来自石油烃污染的沉积环境,还有大量相似序列来自养殖污染环境。
| 马克平. 1993. 论述生物多样性的概念. 生物多样性, 1(1):20-22 |
| 王金成, 井明博, 肖朝霞等. 2012. 陇东黄土高原地区石油污染土壤微生物群落及其与环境因子的关系. 水土保持通报, 32(5): 145-151 |
| 王新新. 2008. 长江口、东海沉积环境细菌多样性和邻近西太平洋海域多环芳烃降解菌研究. 青岛: 中国海洋大学硕士学位论文, 21-25 |
| 卢婧雯, 张心齐, 杜丽丽等. 2012. 中国东海及南海近海4 采样点海水可培养细菌的多样性研究. 浙江大学学报(理学版), 39(4): 443-449 |
| 白洁, 李海艳, 赵阳国. 2009. 黄海北部不同站位海洋细菌群落分布特征. 微生物学报, 49(3): 343-350 |
| 刘玉娟, 田新朋, 黄小芳等. 2014. 中国南海沉积环境可培养细菌多样性研究. 微生物学通报, 41(4): 661-673 |
| 齐海明, 孙岳, 徐兆礼等. 2013. 椒江口海域春秋季虾类群落结构及其影响因素. 应用生态学报, 24(12): 3546-3552 |
| 江四义, 郑兆勇. 2008. 从珠江口沉积物粒度参数特征分析泥沙来源及其运移趋势. 中山大学学报( 自然科学版),47(S1): 126-129 |
| 江锦花, 朱利中, 张明. 2006. 椒江口水体和生物体中典型有机污染物的浓度水平及来源初探. 环境化学, 25(5):546-549 |
| 江锦花, 江正玲, 陈希方等. 2007. 椒江口海域重金属含量分布及在沉积物和生物体中的富集. 海洋环境科学, 26(1):58-62 |
| 孙风芹, 汪保江, 李光玉等. 2008. 南海南沙海域沉积物中可培养微生物及其多样性分析. 微生物学报, 48(12):1578-1587 |
| 杜萍, 刘晶晶, 沈李东等. 2012. Biolog 和PCR-DGGE 技术解析椒江口沉积物微生物多样性. 环境科学学报, 32(6):1436-1444 |
| 杜萍, 刘晶晶, 陈全震等. 2011b. 椒江口春季水体异养细菌及氮, 磷细菌的生态分布特征. 海洋环境科学, 30(4):467-472 |
| 杜萍, 徐晓群, 刘晶晶等. 2011a. 椒江口春、秋季浮游动物分布特征及与主要环境因子的关系. 应用与环境生物学报, 17(4): 486-494 |
| 李昭. 2013. 南太平洋环流区深海可培养细菌的多样性研究以及两株海洋新菌的分类鉴定. 青岛: 中国海洋大学博士学位论文, 61-63 |
| 李涛, 王鹏, 汪品先. 2008. 南海南部陆坡表层沉积物细菌和古菌多样性. 微生物学报, 48(3): 323-329 |
| 李振高, 骆永明, 滕应. 2008. 土壤与环境微生物研究法.北京: 科学出版社, 146-147 |
| 杨华云, 吕伟, 季天嘉等. 2014. 椒江中下游沉积物中多溴联苯醚的分布特征. 杭州师范大学学报(自然科学版),13(2): 175-180 |
| 何建瑜, 刘雪珠, 赵荣涛等. 2013. 东海表层沉积物纯培养与非培养细菌多样性. 生物多样性, 21(1): 28-37 |
| 余鹏, 周珊珊, 杨华云. 2011. 椒江口沉积物中有机氯农药的含量及对映体特征. 环境科学与技术, 34(11): 1-5 |
| 林承坤. 1984. 长江口泥沙的来源分析与数量计算. 泥沙研究,(2): 22-32 |
| 郑莹, 霍颖异. 2013. 浙江镇海潮间带沉积物中可培养微生物初步研究. 海洋学研究, 30(4): 65-71 |
| 赵永强, 陈全震, 曾江宁等. 2009. 椒江口化工园区及其邻近区域潮间带大型底栖动物秋冬季分布特征. 生态学报,29(3): 1578-1586 |
| 姜丽晶. 2007. 珠江口沉积物微生物多样性及其与环境相互关系的研究. 厦门: 国家海洋局第三海洋研究所硕士学位论文, 39-68 |
| 郭建丽, 樊景凤, 付慧等. 2013. 双台子河口沉积物中细菌多样性分析. 微生物学通报, 40(9): 1550-1559 |
| 黄备, 闵怀, 唐静亮等. 2009. 浙江沿海海洋微生物群落的研究. 中国环境监测, 25(2): 44-47 |
| 蒋然, 王健鑫, 黄备等. 2015. 椒江口沉积物中细菌多样性初步研究. 海洋与湖沼, 46(4): 887-900 |
| 谢钦春, 李伯根, 夏小明等. 1998. 椒江河口悬沙浓度垂向分布和泥跃层发育. 海洋学报, 20(6): 58-59 |
| 霍颖异, 许学伟, 王春生等. 2008. 浙江苍南近海沉积物细菌物种多样性. 生态学报, 28(10): 5166-5172 |
| Magurran A E. 2011. 生物多样性测度. 张峰译. 北京: 科学出版社, 66-67 |
| Barns S M, Cain E C, Sommerville L et al. 2007. Acidobacteriaphylum sequences in uranium-contaminated subsurfacesediments greatly expand the known diversity within thephylum. Applied and Environmental Microbiology, 73(9):3113-3116 |
| Beman J M, Francis C A. 2006. Diversity of ammonia-oxidizingarchaea and bacteria in the sediments of a hypernutrifiedsubtropical estuary: Bahía del Tóbari, Mexico. Applied andEnvironmental Microbiology, 72(12): 7767-7777 |
| Bernhard A E, Donn T, Giblin A E et al. 2005. Loss of diversityof ammonia-oxidizing bacteria correlates with increasingsalinity in an estuary system. Environmental Microbiology,7(9): 1289-1297 |
| Bowman J P, McCuaig R D. 2003. Biodiversity, communitystructural shifts, and biogeography of prokaryotes withinAntarctic continental shelf sediment. Applied andEnvironmental Microbiology, 69(5): 2463-2483 |
| Bull A T, Stach J E, Ward A C et al. 2005. Marine actinobacteria:perspectives, challenges, future directions. Antonie VanLeeuwenhoek, 87(1): 65-79 |
| Clementino M M, Fernandes C C, Vieira R P et al. 2007.Archaeal diversity in naturally occurring and impactedenvironments from a tropical region. Journal of AppliedMicrobiology, 103(1): 141-151 |
| Coddington J A, Young L H, Coyle F A. 1996. Estimating spiderspecies richness in a southern Appalachian cove hardwoodforest. Journal of Arachnology, 24(2): 111-128 |
| Crump B C, Armbrust E V, Baross J A. 1999. Phylogeneticanalysis of particle-attached and free-living bacterialcommunities in the Columbia River, its estuary, and theadjacent coastal ocean. Applied and EnvironmentalMicrobiology, 65(7): 3192-3204 |
| Freitag T E, Prosser I J. 2003. Community structure ofammonia-oxidizing bacteria within anoxic marine sediments.Applied and Environmental Microbiology, 69(3): 1359-1371 |
| Gontang E A, Fenical W, Jensen P R. 2007. Phylogeneticdiversity of gram-positive bacteria cultured from marinesediments. Applied and Environmental Microbiology,73(10): 3272-3282 |
| Heijs S K, Laverman A M, Forney L J et al. 2008. Comparison ofdeep-sea sediment microbial communities in the EasternMediterranean. FEMS Microbiology Ecology, 64(3): 362-377 |
| Henriques I S, Alves A, Tacão M et al. 2006. Seasonal andspatial variability of free-living bacterial communitycomposition along an estuarine gradient (Ria de Aveiro,Portugal). Estuarine, Coastal and Shelf Science, 68(1-2):139-148 |
| Hu B L, Shen L D, Du P et al. 2012. The influence of intensechemical pollution on the community composition, diversityand abundance of anammox bacteria in the Jiaojiang Estuary(China). PLoS One, 7(3): e33826 |
| Inagaki F, Suzuki M, Takai K et al. 2003. Microbial communitiesassociated with geological horizons in coastal subseafloorsediments from the Sea of Okhotsk. Applied andEnvironmental Microbiology, 69(12): 7224-7235 |
| Jørgensen B B. 1982. Mineralization of organic matter in the seabed-the role of sulphate reduction. Nature, 296(5858): 643-645 |
| Kan J J, Crump B C, Wang K et al. 2006. Bacterioplanktoncommunity in Chesapeake Bay: predictable or randomassemblages. Limnology and Oceanography, 51(5): 2157-2169 |
| Kumar S, Tamura K, Nei M. 2004. MEGA3: integrated softwarefor molecular evolutionary genetics analysis and sequencealignment. Briefings in Bioinformatics, 5(2): 150-163 |
| Magurran A E. 1988. Ecological Diversity and Its Measurement.Princeton: Princeton University PressMosier A C, Francis C A. 2008. Relative abundance and diversityof ammonia-oxidizing archaea and bacteria in the SanFrancisco Bay estuary. Environmental Microbiology, 10(11):3002-3016 |
| Schloss P D, Handelsman J. 2005. Introducing DOTUR, acomputer program for defining operational taxonomic units and estimating species richness. Applied and EnvironmentalMicrobiology, 71(3): 1501-1506 |
| Shen L D, Zhu Q, Liu S et al. 2014. Molecular evidence fornitrite-dependent anaerobic methane-oxidising bacteria inthe Jiaojiang estuary of the East Sea (China). AppliedMicrobiology and Biotechnology, 98(11): 5029-5038 |
| Webster G, Parkes R J, Cragg B A et al. 2006. Prokaryoticcommunity composition and biogeochemical processes indeep subseafloor sediments from the Peru Margin. FEMSMicrobiology Ecology, 58(1): 65-85 |
| Zhu J Y, Zhang J X, Li Q et al. 2013. Phylogenetic analysis ofbacterial community composition in sediment contaminatedwith multiple heavy metals from the Xiangjiang River inChina. Marine Pollution Bulletin, 70(1-2): 134-139 |
 2015, Vol. 46
2015, Vol. 46


