中国海洋湖沼学会主办。
文章信息
- 舒琥, 岳磊, 李强, 蓝昭军, 谷平华, 王杭军, 吴土金. 2015.
- SHU Hu, YUE Lei, LI Qiang, LAN Zhao-Jun, GU Ping-Hua, WANG Hang-Jun, WU Tu-Jin. 2015.
- 不同流域光倒刺鲃(Spinibarbus hollandi)子一代及其亲本的线粒体COI 基因遗传变异分析
- GENETIC VARIABILITY OF mtDNA COI GENES IN SPINIBARBUS HOLLANDI
- 海洋与湖沼, 46(6): 1509-1515
- Oceanologia et Limnologia Sinica, 46(6): 1509-1515.
- http://dx.doi.org/10.11693/hyhz20150800229
-
文章历史
- 收稿日期:2015-08-30
- 改回日期:2015-09-13
2. 韶关市水产研究所 韶关 512006
2. Shaoguan Fisheries Research Institute, Shaoguan 512006, China
光倒刺鲃(Spinibarbus hollandi),隶属鲤形目、鲤科、鲃亚科、倒刺鲃属,主要分布于长江以南各水系(广西壮族自治区水产研究所等,1981),具有体形好、肉质佳、食性杂、适应性强、生长快等优点。目前,由于江河污染的加剧和人类无限制的捕捞,致使自然资源逐渐减少,到了濒危的边缘,因此光倒刺鲃的人工繁殖愈加受到重视,繁育技术取得较大的发展(邓维德等,2013)。线粒体DNA(mtDNA)是真核生物的核外遗传物质,具有基因组结果简单、编码效率高、进化速率快、无组织特异性和母系遗传等特性(董丽娜等,2011),特别是其中的COI 基因具有进化速率适中等特性,可作为系统进化研究的良好标记(付景等,2006)。本实验对光倒刺鲃杂交子一代及其亲本的COI基因进行了测序分析,不仅为光倒刺鲃杂交子代的mtDNA 遗传效应的研究提供理论基础,同时对光倒刺鲃新品种的选育具有指导作用。
1 材料与方法 1.1 材料实验用鱼为长江流域光倒刺鲃雌、雄鱼(平均体质量为 1200g),北江流域光倒刺鲃雌、雄鱼(平均体质量为 1400g),长江流域光倒刺鲃雄鱼与北江流域光倒刺鲃雌鱼杂交子一代(平均体质量为 22.5g),及其长江流域光倒刺鲃雌鱼与北江流域光倒刺鲃雄鱼杂交子一代(平均体质量为14.3g),均取自韶关水产研究所,每组3 个个体,剪鳍条保存于95%的酒精中。
1.2 方法DNA 提取采用上海生工的试剂盒,DNA 放置在–80°C 保存备用。扩增COI 全序列的引物序列为:COI-F: GGGGCTGATAGGAAGAGGAC,COI-R: TTC GATTCCTCCCTTCCTCG。反应总体积为25μL,其中ddH2O7.5μL,Mix 12.5μL,模板DNA 3μL,上下游引物各1μL。反应程序为: 95°C 预变性5min,94°C 变性30s,53°C 退火30s,68°C 延伸2min,共30 个循环;最后68°C 延伸5min。PCR 产物用1%的琼脂糖电泳检测。
对测序结果进行人工拼接与矫正,然后将校准过的序列与已发表的光倒刺鲃线粒体全基因序列进行比对,验证所测得片段就是所需要的目标序列。最后将该校准的序列以FASTA 格式文件保存。运用MegAlign 进行序列间相似度的比对和差异性分析,而其中的碱基含量、变异位点、简约信息位点及序列碱基间替换情况则运用MEGA5.0 统计(Tamura et al,2007),同时采用Kimura 双参数模型(Kimura,1981)计算遗传距离,构建NJ 系统发育树。
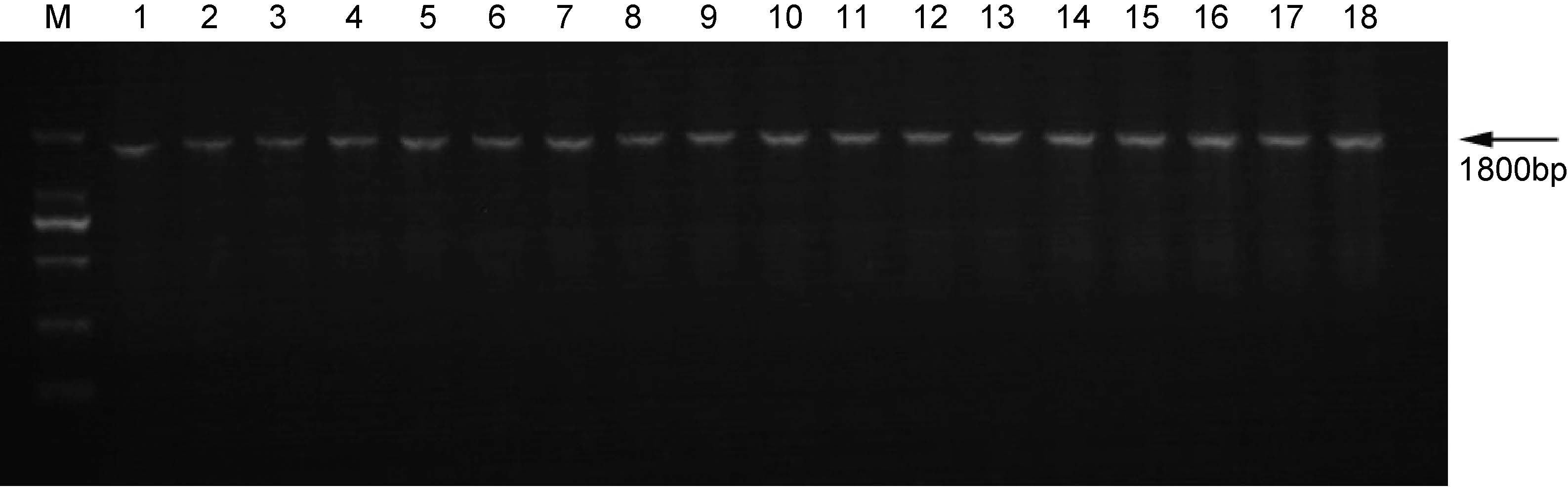
2 结果与分析 2.1 COI 基因的PCR 结果在 6 组共 18 个光倒刺鲃个体中均扩增出约1800bp 的片段如图 1。
|
|
图 1 光倒刺鲃 mtDNA COI 基因片段扩增结果
Fig. 1 The PCR amplification products of mtDNA COI genes of S. hollandi M 为 DNA 2000bp 相对分子质量标准,1—3 为长江流域光倒刺鲃雄鱼,4—6 为长江流域光倒刺鲃雌鱼,7—9 为北江流域光倒刺鲃雄鱼,10—12 为北江流域光倒刺鲃雌鱼,13—18 为杂交子一代 |
对PCR 扩增出来的片段进行序列测定,对比得到 6 组光倒刺鲃 mtDNA COI 1551bp 的碱基序列,由于其中有6 个碱基插入导致对比出来的序列长为1557bp(除去部分tRNA-Tyr 基因序列和部分的tRNA-Ser 基因序列)。在 18 个光倒刺鲃个体的 COI基因序列中,共检测到35 个可变核苷酸位点,其中28 个简约信息位点,另外1522 个为保守核苷酸位点。长江的2个光倒刺鲃母本和北江的3个光倒刺鲃母本共享一个单倍型(C-1),另一个长江的光倒刺鲃母本单倍型为(CC-1),长江的 3 个光倒刺鲃父本和北江的3个光倒刺鲃父本共享一个单倍型(X-1),长江光倒刺鲃母本与北江光倒刺鲃父本杂交子一代 3 个个体共享3个单倍型(Z-1,Z-4,Z-5),长江的光倒刺鲃父本与北江光倒刺鲃母本杂交子一代3个个体共享2个单倍型(Z-2,Z-3)。其中 18 个光倒刺鲃个体的 COI 基因序列中T、C、A、G 碱基的平均含量分别为29.9%、25.4%、27.2%、17.5%。
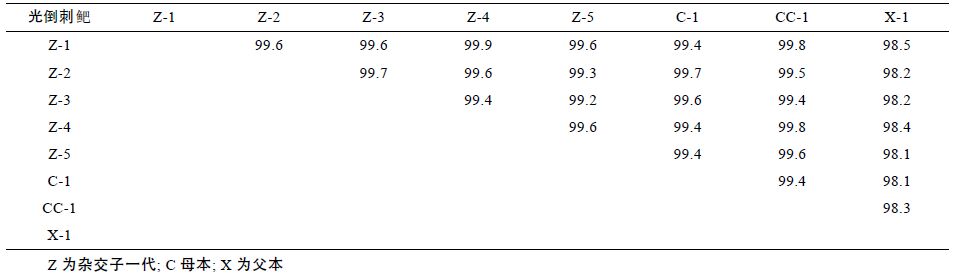
2.3 COI 基因核苷酸序列同源性分析6 组 18 个光倒刺鲃个体的 COI 基因同源性在 98.1%—99.9%之间。子一代单倍型(Z-1)与母本2 种单倍型(C-1,CC-1)的同源性分别为99.4%、99.8%,与父本单倍型(X-1)的同源性为98.5%。子一代单倍型(Z-2)与母本单倍型(C-1)的同源性为99.7%,与父本单倍型(X-1)的同源性为98.2%。子一代单倍型(Z-3)与母本单倍型(C-1)的同源性为99.6%,与父本单倍型(X-1)的同源性为98.2%。子一代单倍型(Z-4)与母本2种单倍型(C-1,CC-1)的同源性分别为99.4%、99.8%,与父本单倍型(X-1)的同源性为98.4%。子一代单倍型(Z-5)与母本2 种单倍型(C-1,CC-1)的同源性分别为99.4%,99.6%,与父本单倍型(X-1)的同源性为98.1%(见表 1)。
对光倒刺鲃杂交子代的单倍型与其亲本的单倍型进行了序列比对(如图 2)。其中存在碱基的插入与缺失,分别为在第612 位点单倍型(Z-2)插入碱基G,在第715 位点单倍型(C-1,Z-5)插入碱基G,在第745位点单倍型(C-1)插入碱基C,在第845 位点单倍型(CC-1)插入碱基C,在第856 位点单倍型(Z-5)插入碱基T,在第879 位点单倍型(Z-4)插入碱基T,在第1556 位点单倍型(C-1,Z-5)存在碱基缺失,在第1557位点处单倍型(Z-1,X-1,Z-3)插入碱基C。除了碱基的缺失与插入,还存在碱基的转换与颠换。在子一代单倍型(Z-1)与其母本2 种单倍型(C-1,CC-1)和父本单倍型(X-1)分析中,发现28 个变异位点,其中子一代单倍型(Z-1)与母本单倍型(C-1)有4 个变异位点,即在第317、350、758、1214 位碱基分别为C、T、A、T,而母本在相应位点分别为T、A、G、C,父本在相应的位点分别为C、T、A、T,子一代单倍型(Z-1)与母本单倍型(CC-1)分析中,发现1 个变异位点,即在第80 位点碱基为A,而母本相应位点分别为G,父本在相应位点为A。在子一代单倍型(Z-4)与母本单倍型(C-1,CC-1)和父本单倍型(X-1)分析中,发现28 个变异位点,其中子一代单倍型(Z-4)与母本单倍型(C-1)有5 个变异位点即在第317、350、758、975、1214位,碱基为C、T、A、T、T,而母本在相应位点为T、A、G、C、C,父本在相应位点为C、T、A、C、T。在子一代单倍型(Z-4)与母本单倍型(CC-1)分析中共1个变异位点,即在第80 位点碱基为A,而母本在相应位点分别为G,父本在相应位点分别为A。在子一代单倍型(Z-5)与母本单倍型(C-1,CC-1)和父本单倍型(X-1)分析中,发现30 个变异位点,其中子一代单倍型(Z-5)与母本单倍型(C-1)分析中共5 个变异位点,即在第317、350、758、784、1214 位点,碱基分别为C、T、G、C、T,而母本在相应位点为T、A、C、T、C,父本在相应位点为C、T、C、T、T。在子一代单倍型(Z-5)与母本单倍型(CC-1)分析中发现3 个变异位点,即在第80、778、784 位点,碱基分别为A、G、C,而母本在相应位点分别为G、C、T,父本在相应位点分别为A、C、T。

|
|
图 2 6 组光倒刺鲃的 8 种单倍型的 mtDNA COI 基因核苷酸比对
Fig. 2 Multiple alignment analysis for mtDNA COI of 8 haplotype in 6 groups of S. hollandi C-1、CC-1 为母本的两种单倍型, X-1 为父本的单倍型, Z 为子代单倍型 |
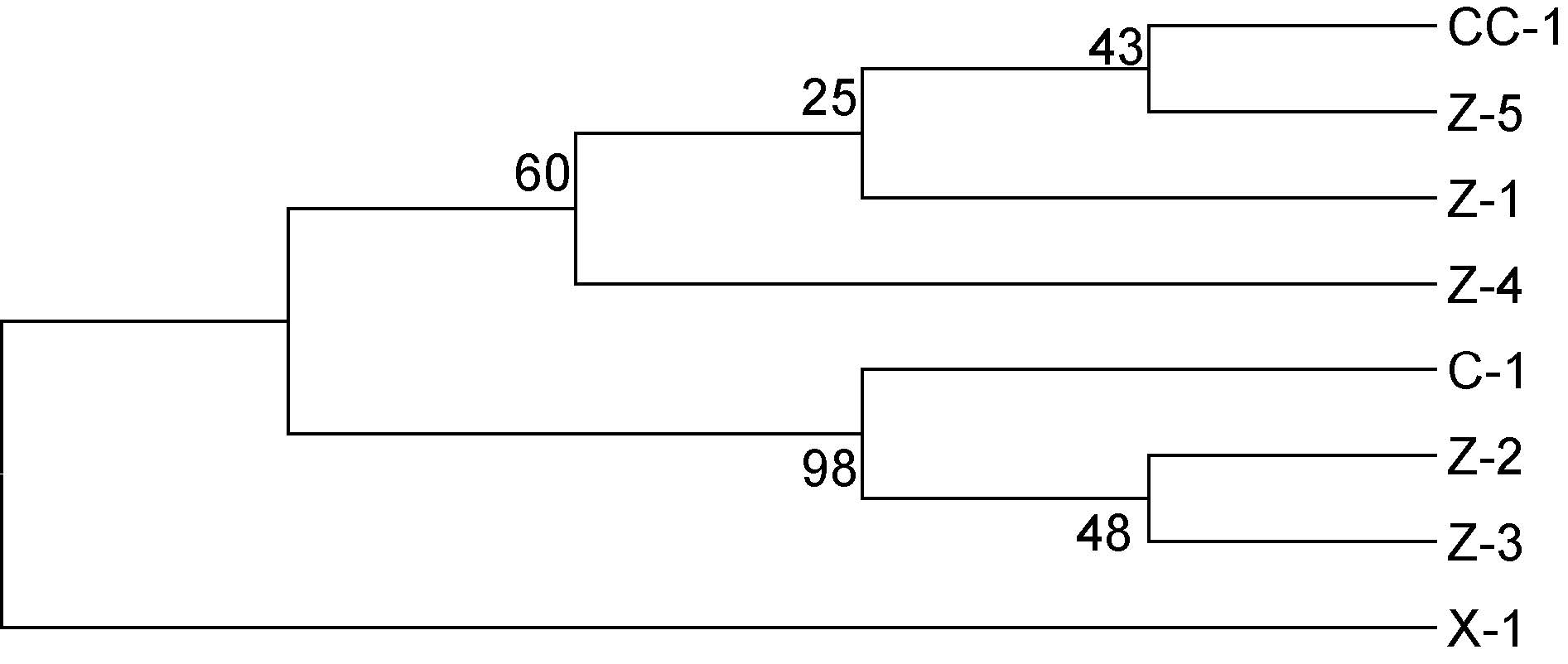
用测得的光倒刺鲃亲本与其杂交子代的 COI 基因单倍型构建系统进化树。根据Kimura's Two-Parameter Method 用邻接法(Neighbor-joining,NJ)构建以上 6 组光倒刺鲃的系统进化树,结果见图 3。
|
|
图 3 邻接法构建 6 组光倒刺鲃 mtDNA COI 基因系统进化树
Fig. 3 The Neighbor-joining phylogenetic tree of the mtDNA COI genes from S. hollandi 图中数字代表 1000 个重复的bootstrape 值,水平线代表遗传距离。Z 为子一代光倒刺鲃; C 为母本; X 为父本 |
线粒体基因已广泛应用于许多生物个体的遗传变异分析和系统进化方面的研究,其中应用细胞色素氧化酶I(COI)进行遗传背景研究的文献报道已经有很多,王太等(2015)分析了DNA 条形码在鱼类物种鉴定中的应用; 黄燕(2014)进行了长江特有鱼类DNA 条形码的研究; 丁春(2014)通过COI 基因进行了河南四种鳊亚科鱼类遗传差异分析; 谢佳燕等(2013)通过COI基因进行鲤科鲌属药用鱼类线粒体的DNA 条形码研究; 有学者对东亚特有鲤科类群的DNA 条形码及其系统发育进行了研究(彭居俐,2007;彭居俐等,2009),利用 DNA 条形码对鲤科鲌属鱼类物种进行了鉴定; 均表明COI 基因具有良好的物种识别能力和保守性。
对于在鱼类中杂交背景的序列变异研究也有很多报道,徐晖等(2 0 0 7)分析了雌性褐牙鲆(Paralichthys olivaceus)和雄性夏牙鲆(P. dentatus)及其杂交子一代的线粒体DNA 16S rRNA 基因部分序列的遗传特性,发现杂交子代的16S rRNA 序列与母本褐牙鲆的序列是一致的,遵循母性遗传的规律。周翰林等(2012)进行了两种杂交石斑鱼子一代及其亲本的线粒体COI 基因遗传变异分析,表明杂交子代遵循母性遗传的特征。郑乐云(2014)对云纹石斑鱼(Epinephelus moara)和赤点石斑鱼(Epinephelus akaara)杂交子一代线粒体相关基因的母性遗传特征进行了分析,表明杂交子代同样是严格遵循母性遗传的。然而郭新红在研究杂交多倍体鲤发现,异源四倍体鲫鲤与其父本鲤和母本红鲫的线粒体DNA 16S rRNA 是遵循母性遗传的。但发现以日本白鲫(Carassium auratus cuvieri)为母本、异源四倍体鲫鲤为父本杂交获得的三倍体湘云鲫的mtDNA 存在父性遗传现象,说明线粒体DNA 在杂交过程中主要表现为母性遗传,同时也存在父性遗传的特殊情况(郭新红,2004)。对于线粒体DNA 的父性遗传现象在其他物种中也有报道,分别在哺乳动物(Shitara et al,1998;Schwartz et al,2002)、鸟类大山雀(Kvist et al,2003)、比目鱼(Platichthys flesus)、昆虫(Lansman et al,1983;Meusel et al,1993)、贝类(Passamonti et al,2001;Dalziel et al,2002; Hoarau et al,2002)等物种中发现了不同程度的父性遗传现象。
本研究使用相同引物扩增上述6组光倒刺鲃COI基因,通过分析表明杂交子一代光倒刺鲃 COI 基因与母本光倒刺鲃的单倍型同源性达到 99.4%—99.8%,而与父本光倒刺鲃的单倍型同源性为 98.1%—98.5%(见表 1)。杂交子一代与母本最多的有9 个[子一代单倍型(Z-4)与其母本单倍型(C-1)]位点不同,但是其呈分散分布,这可能是由于其母本COI 基因的个体差异造成的或者由人工育种造成的点突变。研究表明,虽然父母本为同种鱼类,但与其母本的变异水平明显低于与其父本的变异水平,表明光倒刺鲃杂交线粒体COI 基因严格遵循母性遗传的规律。综上所述,在光倒刺鲃的杂交子代中,线粒体DNA 在COI 基因全序列上严格遵循母性遗传规律,没有发现父性遗传的现象。
| 丁春. 2014. 河南四种鳊亚科鱼类遗传差异研究. 新乡: 河南师范大学硕士学位论文, 23-38 |
| 广西壮族自治区水产研究所, 中国科学院动物研究所. 1981.广西淡水鱼类志. 南宁: 广西人民出版社, 69-70 |
| 王太, 张艳萍, 管丽红等. 2015. 甘肃省鱼类资源现状及DNA 条形码在鱼类物种鉴定中的应用. 生物多样性,23(3): 306-313 |
| 邓维德, 邹佩贞, 吴伟荣等. 2013. 不同温度条件下光倒刺鲃胚的发育. 韶关学院学报, 34(6): 54-57 |
| 付景, 张迎春. 2006. 27 种瓢虫mtDNA-COI 基因序列分析及系统发育研究(鞘翅目: 瓢虫科). 昆虫分类学报, 28(3):179-186 |
| 周翰林, 杨森, 高川等. 2012. 两种杂交石斑鱼子一代及其亲本的线粒体COⅠ基因遗传变异分析. 热带生物学报,3(1): 2-10 |
| 郑乐云. 2014. 云纹石斑鱼和赤点石斑鱼杂交子一代线粒体相关基因的母性遗传特征分析. 上海海洋大学学报, 23(3):351-358 |
| 徐晖, 肖志忠, 孔晓瑜等. 2007. 褐牙鲆(♀)、夏鲆(♂)及其杂交子一代线粒体16S rDNA 序列遗传特性的初步研究. 热带海洋学报, 26(5): 60-63 |
| 郭新红. 2004. 不同倍性鱼类mtDNA 及三倍体湘云鲫Sox 基因研究. 长沙: 湖南师范大学博士学位论文, 81-117 |
| 黄燕. 2014. 长江上游特有鱼类DNA 条形码研究. 重庆: 西南大学硕士学位论文, 19-24 |
| 彭居俐. 2007. 东亚特有鲤科类群的DNA 条形码研究及其系统发育分析. 中国科学院研究生院(水生生物研究所)硕士学位论文, 9-12 |
| 彭居俐, 王绪祯, 王丁等. 2009. 基于线粒体CO1 基因序列的DNA 条形码在鲤科鲌属鱼类物种鉴定中的应用. 水生生物学报, 33(2): 271-276 |
| 董丽娜, 黄梓荣, 艾红等. 2011. 北部湾3 种金线鱼属鱼类COI 基因序列的比较分析. 中国水产科学, 18(3):508-514 |
| 谢佳燕, 李军德, 黄钰淞. 2013. 鲤科鲌属药用鱼类线粒体COI 基因的DNA 条形码研究. 中国中药杂志, 38(7):943-946 |
| Dalziel A C, Stewart D T. 2002. Tissue-specific expression ofmale-transmitted mitochondrial DNA and its implicationsfor rates of molecular evolution in Mytilus mussels (Bivalvia:Mytilidae). Genome, 45(2): 348-355 |
| Hoarau G, Holla S, Lescasse R et al. 2002. Heteroplasmy andevidence for recombination in the mitochondrial controlregion of the flatfish Platichthys flesus. Mol Biol Evol,19(12): 2261-2264 |
| Kimura M. 1981. Estimation of evolutionary distances betweenhomologous nucleotide sequences. Proc Natl Acad Sci USA,78(1): 454-458 |
| Kvist L, Martens J, Nazarenko A A et al. 2003. Paternal leakageof mitochondrial DNA in the great tit (Parus major). MolBiol Evol, 20(2): 243-247 |
| Lansman R A, Avise J C, Huettel M D. 1983. Criticalexperimental test of the possibility of "paternal leakage" ofmitochondrial DNA. Proc Natl Acad Sci USA, 80(7):1969-1971 |
| Meusel M S, Moritz R F A. 1993. Transfer of paternalmitochondrial DNA during fertilization of honeybee (Apismellifera L.) eggs. Curr Genet, 24(6): 539-543 |
| Passamonti M, Scali V. 2001. Gender-associated mitochondrialDNA heteroplasmy in the venerid clam Tapes philippinarum(Mollusca Bivalvia). Curr Genet, 39(2): 117-124 |
| Shitara H, Hayashi J-I, Takahama S et al. 1998. Maternalinheritance of mouse mtDNA in interspecific hybrids:segregation of the leaked paternal mtDNA followed by theprevention of subsequent paternal leakage. Genetics, 148(2):851-857 |
| Schwartz M, Vissing J. 2002. Paternal inheritance of mitochondrialDNA. N Engl J Med, 347(8): 576-580 |
| Tamura K, Dduley J, Nei M et al. 2007. MEGA4: molecularevolutionary genetics analysis (MEGA) software version 4.0.Mol Biol Evol, 24(8): 1596-1599 |
 2015, Vol. 46
2015, Vol. 46