
中国海洋湖沼学会主办。
文章信息
- 纪仁平, 李焕军, 冯艳微, 张荣良, 王卫军, 杨建敏. 2015.
- JI Ren-Ping, LI Huan-Jun, FENG Yan-Wei, ZHANG Rong-Liang, WANG Wei-Jun, YANG Jian-Min. 2015.
- 长牡蛎(Crassostrea gigas)微卫星多重PCR 体系构建及其在家系鉴定中的应用
- DEVELOPMENT OF MULTIPLEX PCR OF MICROSATELLITES AND ITS APPLICATION TO KINSHIP ANALYSIS IN CRASSOSTREA GIGAS
- 海洋与湖沼, 46(6): 1542-1548
- Oceanologia et Limnologia Sinica, 46(6): 1542-1548.
- http://dx.doi.org/10.11693/hyhz20150900231
-
文章历史
- 收稿日期:2015-09-06
- 改回日期:2015-09-24
2. 山东省海洋资源与环境研究院 海洋生态修复重点实验室 烟台 264006
2. Shandong Marine Resource and Environment Research Institute, Shandong Provincial Key Laboratory of Restoration for Marine Ecological Restoration, Yantai 264006, China
长牡蛎(Crassostrea gigas),地方名蚝、蛎黄、海蛎子、蚵等,属于珍珠贝目、牡蛎科、巨蛎属。长牡蛎自然分布于西太平洋海域,是一种广温、广盐性贝类。牡蛎因其肉质鲜美,营养丰富,环境适应性强,生长快速,而成为我国乃至全世界养殖产量最大的一种经济贝类。近几年长牡蛎的养殖规模越来越大,产量也大幅度提高,但其种质却出现明显的退化。因此,良种选育是我国长牡蛎养殖产业健康可持续发展亟待解决的问题,而微卫星分子标记技术可为长牡蛎群体遗传多样性分析和家系亲权鉴定等方面的研究提供技术支持。
在水产动物研究领域,微卫星标记因其具有高多态性,共显性遗传等特点,已在分子标记辅助育种和家系分析等方面得到广泛的应用。20 世纪80 年代,Chamberlain等(1988)首次尝试了多重PCR 反应模式,即在同一PCR 反应体系中加入多对微卫星引物,同时进行多个目标序列的扩增分析。多重PCR 技术具有省时高效、高通量、低成本等优点,且可有效减少靶基因片段的假阳性现象,检验效率得到明显提高(罗伟等,2013)。因此,微卫星多重PCR 为水生生物亲权关系分析(Lerceteau-Köhler et al,2006)、种质鉴定(王婷等,2013)、群体遗传分析(Li et al,2007)、遗传分离模式分析(聂鸿涛等,2013)等研究提供了有力的工具。目前关于长牡蛎微卫星多重PCR 已有报道,但一般都是三重或四重微卫星PCR 反应体系(Li et al,2010; Taris et al,2005)。本研究从已报道的长牡蛎微卫星中开发出了两组微卫星五重PCR 反应体系,并在2012 年构建的0527 组和0612 组的各27 个全同胞家系的亲权鉴定中得以应用。
1 材料与方法1.1 家系样品的构建本实验家系材料的构建于2012 年5 月27 日(记为0527 组)和6 月12 日(记为0612 组)在山东省烟台海益苗业有限公司莱州育苗场(LZ,37°26′1″N,120°02′15″E)进行。实验所用亲贝为经过四代选育的中日韩三个群体。每个群体分别选取3 个父本和3 个母本个体作为亲贝,0527 组和0612 组都采用表 1 所示的部分因子实验设计的方式分别建立27 个全同胞家系; 当各家系发育至D 形幼虫期时,计密度并将各家系幼虫等量混合,然后进行混合培育。取0527 组和0612 组各18 个亲本的闭壳肌保存于70%乙醇中用于DNA 的提取。
当养成至560 日龄时,对两个实验组进行取样。随机选取0527 组653 个子代和0612 组382 个子代个体的闭壳肌保存于70%乙醇中用于提取DNA。各实验组子代和亲本基因组DNA 的提取按照Li等(2002)氯仿/异戊醇法,通过分光光度计和1%琼脂糖凝胶检测DNA 样品的浓度和纯度,并将DNA 浓度调至30ng/μL,–20°C 保存备用。
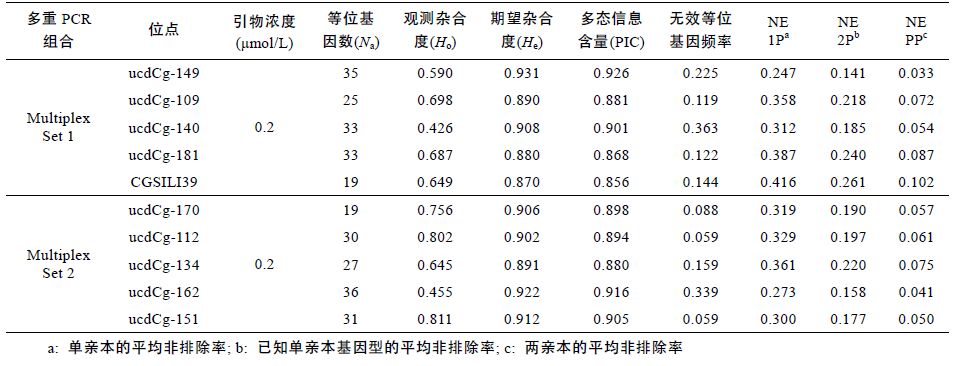
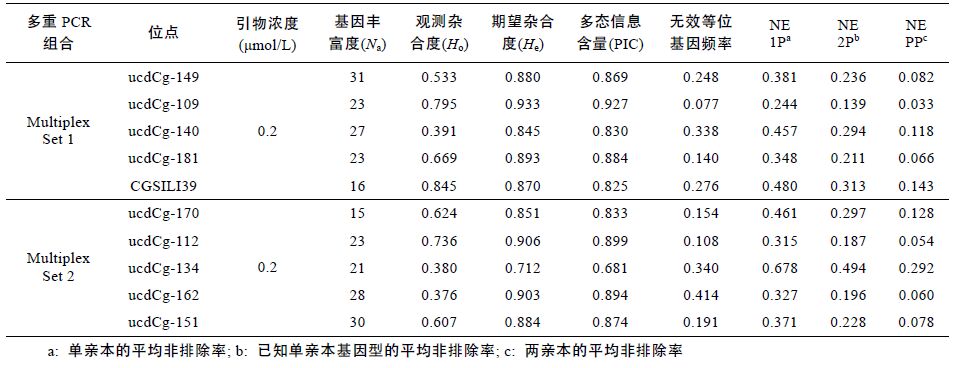
1.3 长牡蛎微卫星位点的筛选从已开发的长牡蛎微卫星中,根据微卫星位点的多态性、序列重复单位、连锁信息,以及扩增效果等筛选出43 个位点(Li et al,2003; Sauvage et al,2009)。微卫星引物由上海生工生物工程技术有限公司合成。每个位点最佳PCR 扩增条件用10 个个体检测。以引物的多态性,条带大小及清晰程度,扩增效率等作为筛选条件,挑选出扩增效果最好的10 个微卫星位点用于多重PCR 的开发,ucdCg149,ucdCg109,ucdCg140,ucdCg181,ucdCg170,ucdCg112,ucdCg129,ucdCg134,ucdCg162,ucdCg151(Li et al,2003)和CGSILI39(Sauvage et al,2009)(表 2)。将10 个位点分为两组,每组中用四种颜色的荧光标记(HEX,FAM,ROX和TAMRA),构成两组微卫星五重PCR反应体系(Multiplex Set 1,MS 1; Multiplex Set 2,MS 2)。
 |
PCR 扩增产物与高度去离子甲酰胺(HIDI)和分子量内标GeneScan ROX500/ LIZ500(美国ABI 公司)以产品说明中要求的比例进行混合,变性5min 后迅速冷却,于3730 XL 测序列分析仪(美国ABI 公司)上进行检测分析。用Genemapper v.3.7 软件(美国Applied Biosystems 公司)分析DNA 片段大小。
1.5 微卫星标记优化组合根据适于同时扩增的位点最大化且同一荧光标记的位点间等位基因不重叠的原则(Li et al,2010)将这些位点进行五重PCR 组合。通过对退火温度、反应体系、循环参数(退火温度,延伸时间)等条件进行优化,确定五重PCR 的最佳反应条件。
1.6 数据统计分析首先运行CERVUS 3.0(Kalinowski et al,2007)软件中的模拟功能估算两组长牡蛎家系鉴定所需要的位点数及其鉴定效率。软件操作具体参数设置如下:模拟子代数目设置为10000(循环重复数),父母本数根据软件要求均为实际父母本数设为9,亲本检测率设置为100%,位点检测率为100%,分型误差率为1%,置信区间设置为95%,亲本性别已知。家系鉴定运用CERVUS 3.0 中基于似然性的计算方法从非排除亲本中选择最可能的亲本。
同时也采用CERVUS 3.0 对各微卫星位点的等位基因数(Number of alleles,Na),多态信息含量(Polymorphism Information Content,PIC)和位点的非排除能力(NE-1P,NE-2P,NE-PP)进行统计分析。亲本对子代的贡献差异由SPSS 14.0(SPSS Inc.,Chicago,IL,USA)进行非参数卡方检验。
2 结果2.1 五重PCR 的优化设计的引物组合经过筛选优化后获得了两组微卫星五重PCR 体系。
两组五重PCR 反应体系25μL 包括: 模板DNA1.0μL(30ng),10×PCR Buffer 2.5μL,25mmol/L MgCl22.0μL,10mmol/L dNTPs 0.5μL,10mmol/L 引物每对各0.2μL,5U/μL Taq DNA 聚合酶0.2μL,纯水17.8μL。
两组五重PCR 反应程序均为: 95°C 预变性3min; 95°C 变性30s,退火温度60°C 30s,72°C 延伸30s,循环10 次; 95°C 变性30s,退火温度55°C 30s,72°C 延伸30s,循环20 次; 最后72°C 延伸6min;4°C 保存。
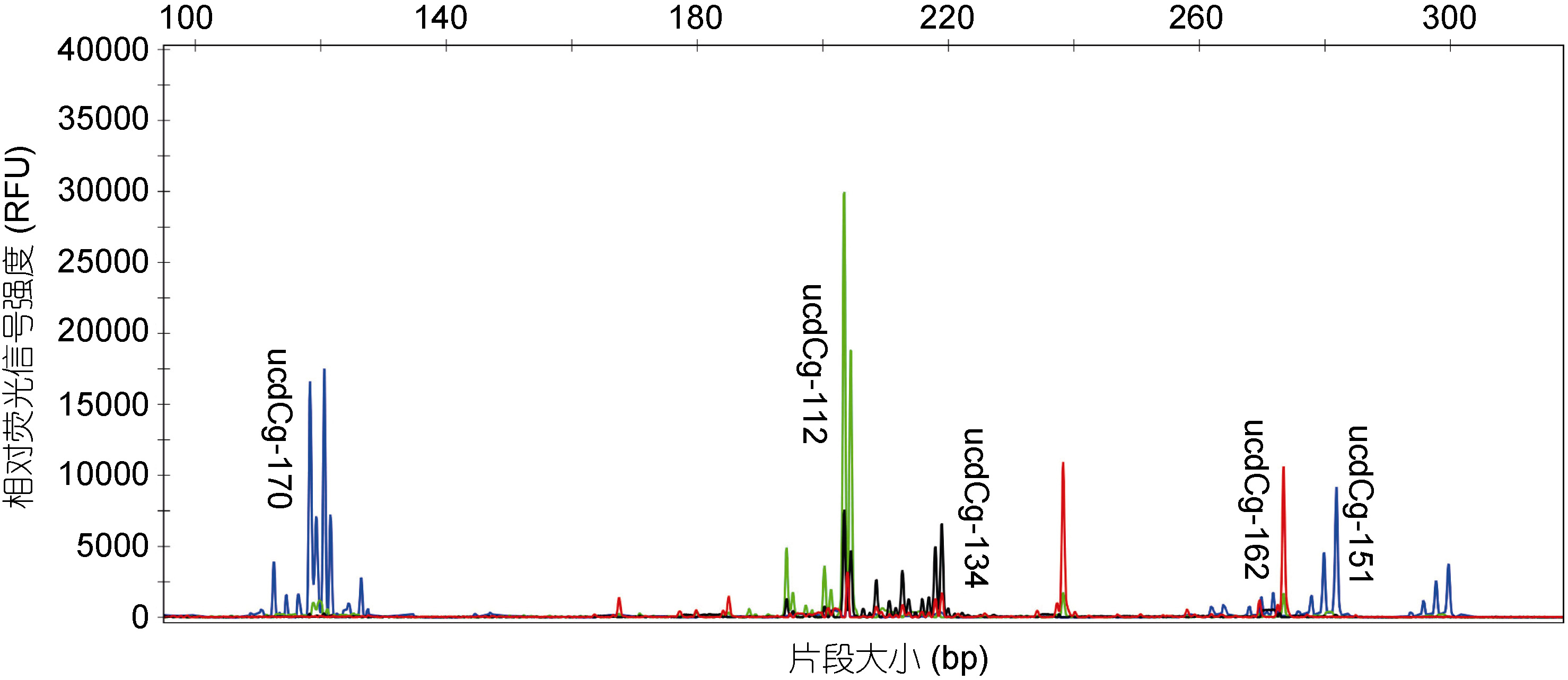
各体系的引物组合、引物浓度详见表 3、表 4。部分微卫星五重PCR 的毛细管电泳结果如图 1。
 |
 |
|
|
图 1 微卫星五重PCR 的毛细管电泳图谱
Fig. 1 The capillary electrophoresis amplified by SSR 5-multiplex PCR 图示为随机选取的0612-R291 号个体用MS 2 扩增后的毛细管电泳图谱 |
10 个微卫星位点的等位基因数(Na)范围从19 到36,平均值为28.8,位点平均多态信息含量为0.8924,反映了这两组五重PCR 在家系鉴定中具有较高的排除能力。两组五重PCR 的观测杂合度(Observed heterozygosity,Ho)范围为0.426—0.811,平均观测杂合度为0.6519,期望杂合度(Expected heterozygosity,He)范围为0.870—0.931,平均期望杂合度为0.9011,无效等位基因频率为5.9%—36.3%(表 3)。
2.2.2 0612 实验组经筛选后得到10 个微卫星位点(表 4)。每个位点的等位基因数(Na)为15—31,平均值为23.7; 观测杂合度(Ho)为0.376—0.845,平均观测杂合度为0.5588; 期望杂合度(He)为0.712—0.933,平均期望杂合度为0.8652; 位点平均多态信息含量为0.8516; 无效等位基因频率为7.7%—41.4%。
2.3 微卫星五重PCR 在家系鉴定中的应用 2.3.1 0527 实验组使用两组微卫星五重PCR 对27 个家系的653 个个体进行了亲子鉴定,经用CERVUS 3.0 软件进行分析,使用这两组微卫星五重PCR 的亲子鉴定成功率为100%; 只用MS 1,可以将96%的子代鉴定到亲本; 只用第二组MS 2,可以将97%的子代鉴定到亲本。
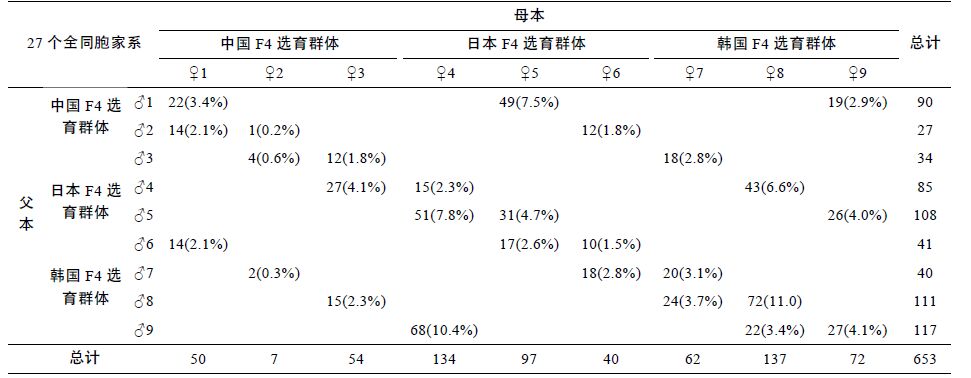
对653 个长牡蛎子代个体进行基因分型,将其全部成功分配至唯一亲本对,子代在27 个全同胞家系中的分布情况见表 5。家系间子代数目差异非常大,家系F23(♀8×♂8)有72 个子代,占子代总数的11.0%;而家系F04(♀2×♂2)仅有1 个子代,占子代总数的0.2%。通过卡方检验可知,父本(χ2=148.02,P=0.00)和母本(χ2=208.55,P=0.00)对子代的贡献率有显著性差异: 母本产生的子代数最多为137 个(♀8),贡献率为21.0%; 最少的为7 个(♀2),贡献率为1.1%。父本产生的子代数最多的为117 个(♂9),贡献率为17.9%;最少为27 个(♂2),贡献率为4.1%(表 5)。
 |
使用两组微卫星五重PCR 对0612 组27 个全同胞家系382 个个体进行亲子鉴定的准确率为100%; 只用MS 1,亲子鉴定准确率为96%;只用MS 2,可以将95%的子代鉴定到亲本。
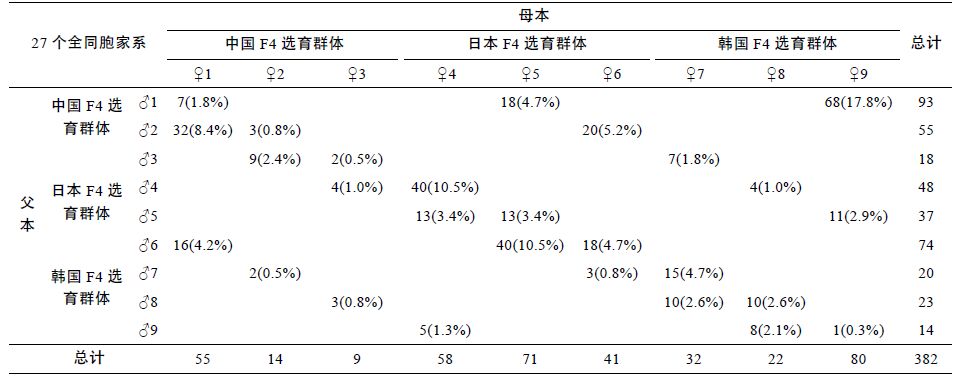
表 6 为0612 组382 个长牡蛎子代个体在27 个全同胞家系中的分布情况。子代在建立的 27 个家系中呈现不均等分布。不同家系间的子代数有较大差异,从1(F27,♀9×♂9)到68(F25,♀9×♂1)不等。卡方检验结果显示,母本(χ2=119.86,P=0.00)和父本(χ2=142.88,P=0.00)对子代的贡献率有显著性差异: 母本产生的子代数最多和最少分别为80(♀9)和9(♀3),贡献率分别为20.9%和2.4%; 父本产生的子代数最多和最少分别为93(♂1)和14(♂9),贡献率分别为24.3%和3.7%。
 |
微卫星中位点的突变、插入或缺失是无效等位基因产生的主要原因(Callen et al,1993; Pemberton et al,1995)。无效等位基因的出现是降低家系分析准确率的主要来源之一(Marshall et al,1998),但并不影响其在家系分析研究中的应用(Li et al,2005)。通过增加微卫星位点数和避免使用无效等位基因频率过高的微卫星位点可提高微卫星位点的家系鉴定能力(Carlsson,2008; Wang et al,2010)。McGoldrick等(2000)曾报道太平洋牡蛎中无效等位基因频率为20%。Li等(2003)发现长牡蛎中51.9%的微卫星位点含有无效等位基因。Hubert等(2004)也报道了在长牡蛎中无效等位基因较高,达51.0%。本实验的10 个微卫星位点在0527 组的27 个全同胞家系鉴定中的无效等位基因频率为5.9%—36.3%,而在0612 组的27 个全同胞家系鉴定中无效等位基因频率为7.7%—41.4%。
3.2 微卫星多重PCR 在家系鉴定中的应用微卫星多重PCR 技术已应用于多种水生动物的家系鉴定分析。聂鸿涛等(2013)用12 个微卫星位点对12 个皱纹盘鲍全同胞家系的372 个子代进行家系鉴定,两组多态信息含量最高的多重PCR 的鉴定成功率分别为90%和86%,在此基础上增加任意一个多重PCR 组合,即可达到100%的鉴定成功率。李佳凯等(2014)建立了大黄鱼微卫星多重PCR 体系,使用3 组微卫星多重PCR 体系进行亲子鉴定准确率为100%。苗贵东等(2011)对大菱鲆7 个人工选育家系进行了亲子鉴定,已知1 个亲本时两组多重PCR的鉴定成功率为96.42%,在双亲未知的情况下亲子鉴定准确率为96.58%。Li等(2010)使用两组多态信息含量最高的微卫星多重PCR 对12 个单对交配长牡蛎家系进行基因分析,达到100%的鉴定成功率。
在本试验中,用两组微卫星五重PCR 鉴别时,在0527 组和0612 组的鉴定成功率均为100%; 只用MS 1,可以将0527 组96%的子代和0612 组96%的子代鉴定到亲本; 只用MS 2,可以将0527 组97%的子代和0612 组95%的子代鉴定到亲本。本研究中较高的家系鉴定成功率可能与所使用的微卫星位点较好及其多态性水平较高有关,这与Gheyas等(2009)所提到的选择更多信息量大、多态性高的位点是达到高鉴定率的必要保障的观点相一致。
作者仅用两组微卫星五重PCR 反应体系就完成了10 个微卫星位点的分型,在短时间内实现长牡蛎群体的遗传多样性分析,充分体现了多重PCR 快速高效、高通量、节约、简便的特点。本研究中筛选出的微卫星多重PCR 体系不仅可以应用于长牡蛎群体的亲子鉴定分析以及分子标记辅助育种,还可为家系群体遗传多样性分析和遗传关系研究等方面的研究提供技术基础。
| 王婷, 徐燕, 陈昌生等. 2013. 基于SCAR 标记的坛紫菜“闽丰1 号”多重PCR 鉴定技术的建立. 水产学报, 37(5):688-695 |
| 李佳凯, 王志勇, 韦信键等. 2014. 大黄鱼微卫星多重PCR 体系的建立及其应用. 水产学报, 38(4): 470-475 |
| 罗伟, 高泽霞, 曾聪等. 2013. 团头鲂微卫星多重PCR 体系的建立及应用. 大连海洋学学报, 28(5): 418-423 |
| 苗贵东, 杜民, 杨景峰等. 2011. 大菱鲆亲子鉴定的微卫星多重PCR 技术建立及应用. 中国海洋大学学报, 41(1-2):97-106 |
| 聂鸿涛, 李琪, 孔令锋. 2013. 皱纹盘鲍微卫星多重PCR 体系构建及其在家系鉴定中的应用. 水产学报, 37(2): 207-215 |
| Callen D F, Thompson A D, Shen Y et al. 1993. Incidence andorigin of “null” alleles in the (AC)n microsatellite markers.Am J Human Genet, 52(5): 922-927 |
| Carlsson J. 2008. Effects of microsatellite null alleles onassignment testing. J Hered, 99(6): 616-623 |
| Chamberlain J S, Gibbs R A, Rainer J E et al. 1988. Deletionscreening of the Duchenne muscular dystrophy locus viamultiplex DNA amplification. Nucl Acids Res, 16(23):11141-11156 |
| Gheyas A A, Woolliams J A, Taggart J B et al. 2009. Heritabilityestimation of silver carp (Hypophthalmichthys molitrix)harvest traits using microsatellite based parentage assignment.Aquaculture, 294(3-4): 187-193 |
| Hubert S, Hedgecock D. 2004. Linkage maps of microsatelliteDNA markers for the Pacific oyster Crassostrea gigas.Genetics, 168(1): 351-362 |
| Kalinowski S T, Taper M L, Marshall T C. 2007. Revising howthe computer program CERVUS accommodates genotypingerror increases success in paternity assignment. Mol Ecol,16(5): 1099-1106 |
| Lerceteau-Köhler E, Weiss S. 2006. Development of a multiplexPCR microsatellite assay in brown trout Salmo trutta, and itspotential application for the genus. Aquaculture, 258(1-4):641-645 |
| Li G, Hubert S, Bucklin K et al. 2003. Characterization of 79microsatellite DNA markers in the Pacific oysterCrassostrea gigas. Mol Ecol Notes, 3(2): 228-232 |
| Li Q, Kijima A. 2005. Segregation of microsatellite alleles ingynogenetic diploid Pacific abalone (Haliotis discus hannai).Mar Biotechnol, 7(6): 669-676 |
| Li Q, Park C, Kijima A. 2002. Isolation and characterization ofmicrosatellite loci in the Pacific abalone, Haliotis discushannai. J Shellfish Res, 21: 811-815 |
| Li Q, Park C, Kobayashi T et al. 2003. Inheritance of microsatellite DNA markers in the Pacific abalone Haliotisdiscus hannai. Mar Biotechnol, 5(4): 331-338 |
| Li R H, Li Q, Cornette F et al. 2010. Development of fourEST-SSR multiplex PCRs in the Pacific oyster (Crassostreagigas) and their validation in parentage assignment.Aquaculture, 310(1-2): 234-239 |
| Li Y T, Wongprasert K, Shekhar M et al. 2007. Development oftwo microsatellite multiplex systems for black tiger shrimpPenaeus monodon and its application in genetic diversitystudy for two populations. Aquaculture, 266(1-4): 279-288 |
| Marshall T C, Slate J, Kruuk L E B et al. 1998. Statisticalconfidence for likelihood-based paternity inference innatural populations. Mol Ecol, 7(5): 639-655 |
| McGoldrick D, Hedgecock D, English L J et al. 2000. Thetransmission of microsatellite alleles in Australian and NorthAmerican stocks of the Pacific oyster (Crassostrea gigas):selection and null alleles. J Shellfish Res, 19: 779-788 |
| Pemberton J M, Slate J, Bancroft D R et al. 1995. Nonamplifyingalleles at microsatellite loci: a caution for parentage andpopulation studies. Mol Ecol, 4(2): 249-252 |
| Sauvage C, Boudry P, Lapègue S. 2009. Identification andcharacterization of 18 novel polymorphic microsatellitemakers derived from expressed sequence tags in the Pacificoyster Crassostrea gigas. Mol Ecol Res, 9(3): 853-855 |
| Taris N, Baron S, Sharbel T F et al. 2005. A combinedmicrosatellite multiplexing and boiling DNA extractionmethod for high-throughput parentage analyses in thePacific Oyster (Crassostrea gigas). Aquac Res, 36(5): 516-518 |
| Wang Y, Wang X X, Wan A M et al. 2010. A 16-microsatellitemultiplex assay for parentage assignment in the easternoyster (Crassostrea virginica Gmelin). Aquaculture, 308(1):S28-S33 |
 2015, Vol. 46
2015, Vol. 46



