中国海洋湖沼学会主办。
文章信息
- 李玉龙, 董婧, 王彬, 孙明, 王爱勇, 王文波. 2016.
- LI Yu-Long, DONG Jing, WANG Bin, SUN Ming, WANG Ai-Yong, WANG Wen-Bo. 2016.
- 基于18S和ITS-5.8S rDNA基因序列的白色霞水母(Cyanea nozakii)的分子鉴定与检测
- MOLECULAR IDENTIFICATION AND DETECTION OF CYANEA NOZAKII BY PARTIAL SEQUENCING OF 18S RDNA AND ITS-5.8S
- 海洋与湖沼, 47(1): 158-165
- Oceanologia et Limnologia Sinica, 47(1): 158-165.
- http://dx.doi.org/10.11693/hyhz20150200051
-
文章历史
- 收稿日期: 2015-02-10
- 收修改稿日期: 2015-08-20
近年来,环境污染、温室效应及过度捕捞等因素使海洋生态系统发生了有利于水母类的变化,全球多处海域发生大型水母类的暴发现象,大型水母暴发在破坏海洋生态系统平衡的同时对海洋渔业造成巨大负面影响,已引起一系列的经济和社会问题(仲霞铭等,2004; 程家骅等,2004; Kawahara et al,2006; Purcell,2007; 孙松,2012a,b)。据已有的文献报道,我国近海也曾多次发生过大型灾害性水母暴发现象,其优势种主要为可食用但低值的沙蜇(Nemopilema nomurai)和无食用价值的海月水母(Aurelia spp.)和霞水母(Cyanea spp.)(董婧等,2013)。
霞水母隶属于刺胞动物门(Cnidaria)、钵水母纲(Scyphomedusae)、旗口水母目(Semaeostomeae)、霞水母科(Cyaneidae),是一种广布性的大型海洋浮游生物,通常以小型浮游动物为饵,在我国沿海均有分布,目前已发现有白色霞水母(Cyanea nozakii)、发形霞水母(Cyanea capillata)、棕色霞水母(Cyanea ferruginea)和紫色霞水母(Cyanea purpurea)四种(董婧等,2013),其中以白色霞水母数量最多、分布范围最广。白色霞水母是在我国常见的曾多次暴发过的水母,自20世纪末起,黄海、东海、南海近海多次发生霞水母暴发现象,因其生长过程中分泌毒素并缠粘网具,造成海洋渔业资源枯竭,海洋捕捞产量严重下降(董婧等,2013),如2004年辽东湾白色霞水母暴发导致的海蜇大面积减产,其主要原因是生物灾难——霞水母旺发,次要原因是海水污染(葛立军等,2004)。
针对白色霞水母等大型灾害水母暴发引起的一系列问题,其早期生活史阶段的有效鉴定与观测对查清在自然海域中白色霞水母的生长发育和暴发性增殖具有重要意义。霞水母具有世代交替生活史(Dong et al,2006),其浮浪幼虫、螅状体、碟状体等个体微小(毫米以下)难于观测,制约了研究者对其早期生长阶段数量变化及其分布扩散等方面的研究。
分子生物技术的快速发展为解决上述难题提供了新契机,目前已有一些学者(Bayha et al,2009; Ki et al,2010; 王建艳等,2013)利用线粒体分子标记如COI、16S等开发了白斑水母(Phyllorhiza spp.)、金黄水母(Chrysaora sp.)和海月水母(Aurelia sp.)螅状体和碟状体的分子鉴定和检测技术; 张姝等(2009)和杨傲傲(2014)分别利用16S rRNA、COI基因片段和核基因β-连环蛋白对我国两种常见大型水母海蜇和沙蜇进行了分子甄别鉴定研究。但上述所研究的水母种类未包括白色霞水母,所用的分子标记主要为线粒体分子标记,利用核基因分子标记如核糖体DNA对大型水母进行的研究较少,目前仅刘敏等(2011)利用18S rDNA对沙蜇和口冠水母(Stomolophus meleagris)的分类关系进行了分析; Bayha等(2010)利用18s和28s rDNA序列分析了钵水母纲的系统和进化关系。
rDNA(核糖体DNA)为多拷贝基因,在真核生物中可以达到1000个重复(Schlötterer,1998),每个重复单元包括编码区18S、5.8S和28S rDNA以及非编码区ITS1和ITS2,其在细胞中以一种协同方式进化,重复序列间基本保持一致。rDNA具有高变异和高保守两种区域,在海洋动物的分类、种质鉴定和系统发育中得到广泛应用(Chu et al,2001; Chen et al,2002; Dawson et al,2005; Wanna et al,2006; Ki et al,2009; Bayha et al,2010)。2004年辽东湾暴发的霞水母经形态鉴定为白色霞水母(董婧等,2005),其暴发使辽东湾海蜇数量锐减并造成生态灾难,当年辽东湾海域高温高盐的外部环境特征是诱使其暴发的重要原因(王彬等,2014)。Dong等(2006,2008)首次揭示了辽东湾白色霞水母的生活史并对各发育阶段的形态进行了描述,但不同水母种类螅状体及碟状体阶段形态相似不易区分且样品易被破坏,这都增加了传统分类的难度。因此,本研究利用rDNA分子标记对白色霞水母各发育阶段的的样品进行分析,为建立白色霞水母早期阶段螅状体和碟状体的物种鉴定和检测方法提供参考,同时为其它大型水母的鉴定和分类以及系统发生研究提供基础资料。
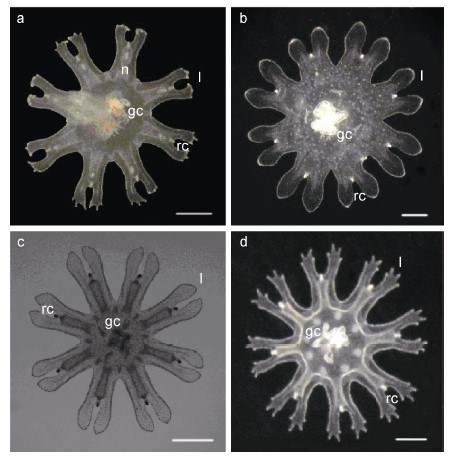
1 材料与方法 1.1 样品采集研究所用白色霞水母包括螅状体、碟状体和水母体。其中螅状体、碟状体为2004年9月在辽东湾捕获的白色霞水母亲体进行人工繁殖所得,水母体为2009年采自辽东湾北部海域。根据董婧等(2013)列出的黄、渤海4种常见大型水母螅状体、碟状体的特征差别(表 1、图 1),对研究所用螅状体、碟状体样品进行了形态分类鉴定,以确保样本采集的准确性。
| 物种 | 触手数量 | 形态 | 触手表面刺胞丛 | 无性生殖 | 横裂生殖 |
| 海蜇R.esculentum | 16 | 高脚杯形柄长、吻长 | 分节,节上具纤毛,刺胞丛凸起明显 | 足囊 | 多碟 |
| 沙蜇N.nomurai | 16 | 圆锥形柄长度中等吻长 | 互生排列,具纤毛,刺胞丛凸起略明显 | 足囊 | 多碟 |
| 海月水母Aureliasp. | 16 | 圆锥形柄短,口盘阔,吻短 | 分节,具纤毛刺胞丛凸起明显 | 螅状体匍匐茎芽 | 多碟(偶见单碟) |
| 白色霞水母C.nozakii | 16 | 圆锥形柄短,口盘阔,吻短 | 分节,具纤毛刺胞丛凸起明显 | 足囊匍匐茎芽 | 单碟 |
采用CTAB法提取基因组DNA后,分别利用引物SSU-U: 5′-AAC CTG GTT GAT CCT GCCAGT- 3′; SSU-L: 5′-TGA TCC TTC TGC AGG TTC ACC TAC- 3′和JF-18F1750 5′-AAAGTCGTAACAAGGTTTCCG- 3′; JF-28R765 5′-TTGGTCCGTGTTTCAAGACG-3′(Ki et al,2009)对白色霞水母1个螅状体样品、1个碟状体样品和3个水母体样品进行扩增,反应体系25μL,包括: 0.2mmol/L每种dNTPs,0.2μmol/L每种引物,1μLDNA模板,1U Taq,2.0mmol/L MgCl2,2.5μL 10×缓冲液,灭菌超纯水补足剩余体系。PCR扩增在Gene Amp9700型PCR仪上进行,反应程序: 95℃预变性3min后,95℃变性30s,50℃退火30s,72℃延伸45s,运行35个循环,最后72℃下延伸5min。扩增后进行双向测序[宝生物工程(大连)有限公司]。
1.3 数据分析测定的18S和ITS-5.8S rDNA序列进行BLAST(http://www.ncbi.nlm.gov/BLAST/)检索,确定序列为目的片段。应用CLUSTAL X 1.8软件(Thompson et al,1997)对序列进行比对及相似性分析。通过MEGA 3.0(Kumar et al,2004)统计碱基含量、变异位点,采用Kimura双参数模型计算种间、属间的遗传距离。用MEGA3.0软件构建NJ(Neighbour-joining)系统树,采用Bootstrap1000检验分子系统树各分支的置信度。
2 结果与分析 2.1 18S和ITS-5.8S rDNA基因序列分析按照CTAB法提取不同生活史阶段(螅状体、碟状体及水母体)的白色霞水母的DNA,所有个体都成功进行PCR扩增,经测序分别得到长度1728bp的18S rDNA基因片段和长度1531bp的18S-ITS-5.8S- 28S rDNA基因序列。本研究中白色霞水母5个个体的18S rDNA基因序列共享一个基因型,与GenBank中采自青岛胶州湾的白色霞水母样品(JX845355)同源序列完全一致。5个白色霞水母样品ITS-5.8S rDNA序列完全一致,ITS1、5.8S rDNA和ITS2长度分别为270bp、158bp、249bp,与GenBank中未知真核生物(表 2)的序列高度相似(≥99%),其序列来源物种的环境信息显示这些序列都是从南海北部表层海水中获得的未指明来源的物种,推测其可能是白色霞水母生活史的早期阶段(卵、浮浪幼虫或碟状体)。
| 种名 | 拉丁名 | 缩写 | GenBank登录信息 | |
| 18S rDNA | ITS1 | |||
| 白色霞水母 | Cyanea nozakii | CNC | KT445894(中国辽东湾), JX845355(胶州湾) | KR338969(中国辽东湾) |
| 未知物种 | uncultured eukaryote ( surface sea water) | CNC | — | GU941271, GU941278, GU941280—82,GU941284, KJ180486, KJ180493(南海) |
| 白色霞水母 | Cyanea nozakii | CNJ | — | AB377551—54(日本) |
| 发形霞水母 | Cyanea capillata | CC | JX995327—28(北海) HM194820(挪威) | AY903053—54(挪威), AY903055—56(美国 卡彻马克湾), U65481 (美国东海岸) |
| 安娜霞水母 | Cyanea annaskala | CA | HM194778(澳大利亚胡昂河口) | AY903061—65(澳大利亚胡昂河口) |
| 玫瑰红霞水母 | Cyanea rosea | CR | — | AY903057—60(澳大利亚梅林布拉湖) |
| 蓝水母 | Cyanea lamarckii | CL | JX995325—26(北海) | — |
| 霞水母 | Cyanea sp. | CS | JX393285—86 (美国), AF358097 | — |
结合GenBank中已有霞水母种类的18S rDNA、ITS1同源序列(表 2),对霞水母属不同种类的18S rDNA和ITS1片段进行了序列分析。5种霞水母属18S rDNA序列经比对后同源序列长度为1709bp,多态位点数33个,约占总序列的1.9%,其中简约信息位点数33个,插入/缺失位点6个。由于GenBank数据库中其它霞水母种类只含核糖体ITS1片段,因此仅对ITS1序列进行了分析。去掉两侧18S rRNA与5.8S rRNA基因序列,ITS1长度为264—379bp,具有明显的种间序列长度多态性,变异幅度较大。比对后保留同源序列,长度为368bp,其中多态位点数为203个,约占总序列的55%,简约信息位点数178个,
单变异位点21个。除种间长度多态性外大部分种类还具有种内序列长度多态性,这种种内序列长度多态性产生的原因基本上是由于微卫星序列插入/缺失所致。
2.2 霞水母属种内和种间遗传距离表 2—表 3列出了基于18S rDNA和ITS1基因序列的霞水母属种内和种间的遗传距离。由表 3可以看出基于18S rDNA计算的种间遗传距离最小值出现在霞水母Cyanea sp.和安娜霞水母、玫瑰红霞水母之间(0.001),最大值出现在白色霞水母和蓝水母之间(0.019),其中白色霞水母与其它几种霞水母的差异较大(0.017—0.019)。平均的种间遗传为0.008,种内遗传距离为0。基于ITS1计算的霞水母种间及种内的K2P遗传距离见表 4,与18S rDNA计算的遗传距离的结果一致,白色霞水母与其它几种霞水母的差异较大(0.325—0.543),其中白色霞水母辽东湾样品与南海未指明来源的物种的序列高度一致,因此将其视为中国白色霞水母样品,其与日本白色霞水母样品间的遗传距离为0.424,这个值甚至大于霞水母不同种间的遗传距离值。白色霞水母日本样品和玫瑰红霞水母之间(0.543)遗传距离最大,安娜霞水母和发形霞水母之间(0.104)的遗传距离最小,平均的种间遗传为0.284,种内的K2P遗传距离范围为0.002—0.056,平均K2P遗传距离为0.019。基于ITS1的种种间遗传距离是种内遗传距离的15倍,适合于进行物种鉴定。
| CNC | CC | CA | CL | D | |
| CNC | 0 | ||||
| CC | 0.018 | 0 | |||
| CA | 0.017 | 0.003 | - | ||
| CL | 0.019 | 0.002 | 0.002 | 0 | |
| CS | 0.018 | 0.002 | 0.001 | 0.001 | 0 |
| D表示种内遗传距离, 各物种全称见表 1 | |||||
| CNC | CNJ | CC | CA | D | |
| CNC | 0.002 | ||||
| CNJ | 0.424 | 0.056 | |||
| CC | 0.325 | 0.499 | 0.027 | ||
| CA | 0.369 | 0.448 | 0.104 | 0.002 | |
| CR | 0.396 | 0.543 | 0.137 | 0.171 | 0.007 |
| D表示种内遗传距离, 各物种全称见表 2 | |||||
基于18S rDNA和ITS1片段构建的霞水母NJ系统树的拓扑结构如图 2所示,结果显示,不同的方法构建的系统树差异不大,同种的不同个体,亲缘关系较近的不同种各自聚支,其聚类结果大致与形态分类吻合,但基于ITS1构建的系统树的聚枝结果比基于18S rDNA构建的系统树得到更高的自举检验的支持,说明ITS1具有丰富的系统发育信息位点,更适于大型水母种、属水平的系统分析研究。
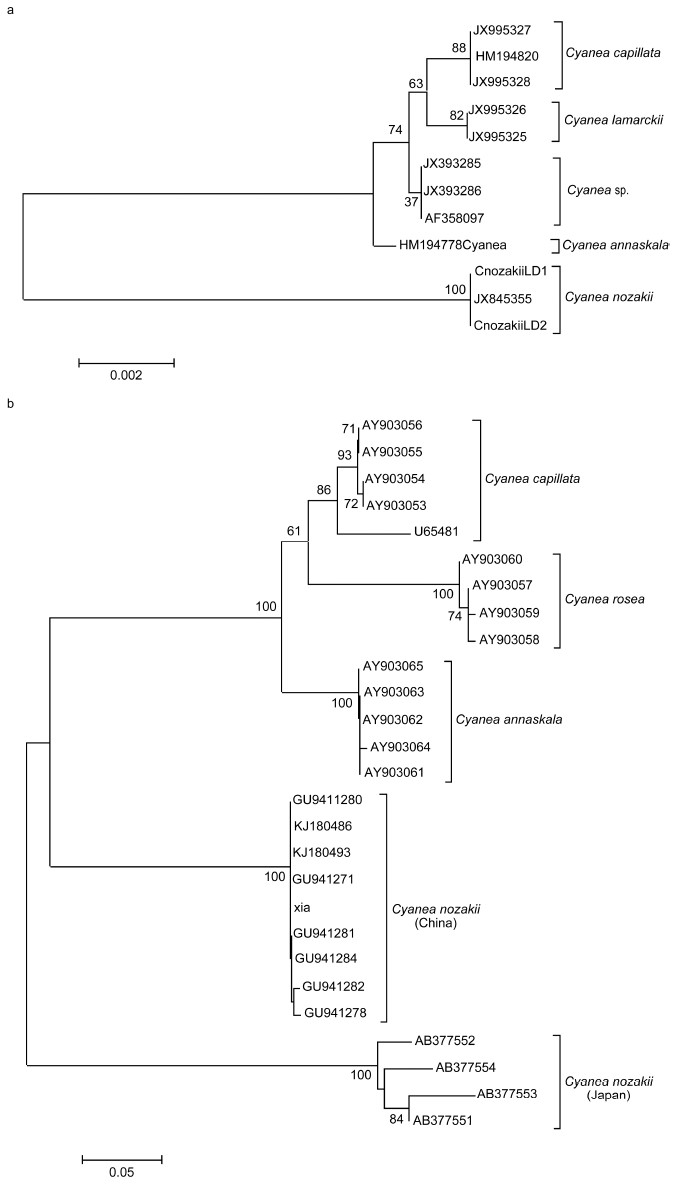 |
| 图 2 基于18S rDNA(a)和ITS1(b)基因片段构建的霞水母属NJ系统树 Fig. 2 Molecular phylogenetic tree of Cyanea jellyfish species based on the fragments of 18S rDNA (a) and ITS1 (b) (NJ method) |
白色霞水母与其它钵水母类相似都具有世代交替的生活史,受精卵经浮浪幼虫、螅状体、碟状体发育为成体水母(Dong et al,2006,2008),除螅状体外,其它都营浮游性生活。通常情况下多种钵水母生活于同一水域,其螅状体、碟状体形态较为相似,难以用肉眼区分不同的种类,给水母初期调查工作种类区分带来很大困难。如海蜇、沙蜇、海月水母和白色霞水母是黄、渤海水域常见的四种大型水母,它们的螅状体形态大致相同,其相同和相异的特征如表 1所示,且螅状体的外观形态与其存活生长状态息息相关,从形态上很难区分(董婧等,2013)。关于四种大型水母的碟状体,一般通过碟状体缘瓣形状、感觉裂缝占缘瓣长度比例和刺细胞团的分布位置加以区分,沙蜇、海蜇碟状体具有爪状的缘瓣,白色霞水母和海月水母碟状体的缘瓣钝圆。白色霞水母碟状体的感觉裂缝较深,占缘瓣长度比例大于1/2,其它种类碟状体感觉裂缝较浅(图 1)。因此,钵水母螅状体、碟状体的形态分类十分繁琐和困难,必须由有经验的专业人员借助显微镜观察才能进行准确区分。
单独挑取1个白色霞水母螅状体、碟状体及水母体进行DNA提取及检测,所有样品都成功扩增出目的片段,结果表明从核酸提取到PCR扩增、测序并进行系统分析的分子生物学技术手段具有很高的灵敏度,只要样品中含1个螅状体或碟状体即可检出。本研究利用18S rDNA和ITS-5.8S基因片段分析了白色霞水母螅状体、碟状体和水母体样品,从分子水平上佐证了采自辽东湾的霞水母样品为白色霞水母(董婧等,2005),这也说明分子生物学方法具有很高的准确性。同时,通过序列比对发现GenBank数据库中从南海北部表层海水中获得的未指明来源的物种的ITS-5.8S
序列与辽东湾白色霞水母样品的同源序列高度相似(≥99%),据此推测该物种可能为自然海域中早期发育阶段(卵、浮浪幼虫或碟状体)的白色霞水母,这与卢振彬等(2003)报道的关于霞水母(主要为白色霞水母)是福建沿海(南海北部)水母类优势种的分布相符。研究结果表明ITS-5.8S基因序列可能是解决白色霞水母不同阶段分类鉴定与检测的有效分子标记。
3.2不同海区霞水母的遗传分化本研究中,基于18S rDNA和ITS-5.8S基因片段计算的霞水母属种间遗传距离(表 3、表 4)表明分布距离越远的霞水母种间遗传差异越明显,如分布于中国及日本附近海域的白色霞水母与其它几种霞水母(主要分布于澳大利亚、美国附近海域)的遗传距离明显高于相距较近的霞水母间的种间遗传距离,漫长的进化历史和长期的隔离分化是导致形成这种现象的主要原因。此外,中国海域白色霞水母样品与日本附近海域白色霞水母样品的遗传距离为0.424,而来自中国的样品间序列差异为0—0.008,日本的样品间序列差异0.03—0.079,两个地方的样品间差异极大甚至大于霞水母属不同种间的遗传距离,说明白色霞水母中国样品与日本样品间有明显的遗传分化; 这种现象在马赛克水母及海月水母中都存在,其原因是长期隔离分化所致(Dawson et al,2005)。另一种可能是样品鉴定错误导致结果出现偏差,这个问题的解决还需要分析更多的样品和更多的DNA片段加以确定。
3.3 核糖体rDNA基因在大型水母类物种鉴定及系统进化分析中的应用很多学者(Bucklin et al,2003)包括国际海洋生物普查组织都认为18S rDNA 序列是适用于进化较慢物种的种类鉴定和系统分析的合适分子标记,但由于其基因序列较为保守,在解决较小分类阶元如属的种间关系尤其是在进行某些近缘种的物种鉴定时具有较大的局限性,其可能更适合进行较高阶元的系统发生关系研究(Hwang et al,1999)。与之相反,核糖体ITS片段处于非编码区,受选择压力较小、进化较快,具有丰富的变异位点信息,适合作为物种鉴定及种、群体水平的分子标记。Chu等(2001)在利用ITS1序列对7种甲壳类动物进行分析时发现不同甲壳动物的ITS1序列差异明显,可作为种间和群体水平的分子标记。本研究利用18S和ITS-5.8S rDNA基因序列对霞水母属不同种类进行的分析表明,序列变异方面所分析的霞水母不同种类间ITS1基因片段明显比18S rDNA片段变异更大,其种间遗传距离平均值是种内遗传距离平均值的15倍,更符合作为物种鉴定的DNA条形码(Hebert et al,2003a,b); 在系统进化方面,两种片段构建的NJ系统发育树都显示同种的不同个体,亲缘关系较近的不同种各自聚枝,但18S rDNA基因进化速率较慢,在解决部分近缘种的进化关系时有较大的局限,可能更适于大型水母科间和科内属间的进化关系研究。因此,在进行大型水母类分子系统进化研究时应联合采用多个进化速率不同的分子标记以得出更为全面准确的结论。
4 结论(1)利用18S rDNA和ITS-5.8S基因片段对白色霞水母螅状体、碟状体和成体水母样品进行的分析表明采自辽东湾的霞水母样品为白色霞水母,白色霞水母样品的ITS-5.8S rDNA序列与GenBank中未知真核生物的序列高度相似(≥99%),推测该物种可能为自然海域中早期发育阶段(卵、浮浪幼虫或碟状体)的白色霞水母。
(2)分布距离越远的霞水母种间遗传差异越明显,中国海域白色霞水母样品与日本附近海域白色霞水母样品的遗传差异极大甚至大于霞水母属不同种间的遗传距离,霞水母漫长的进化历史和长期的隔离分化是导致形成这种遗传分化的主要原因。
(3)霞水母不同种类间ITS1基因片段明显比18S rDNA片段变异更大,具有更丰富的变异位点信息,适合作为大型水母物种鉴定及种、群体水平的分子标记; 18S rDNA基因进化速率较慢,在解决部分近缘种的进化关系时有较大的局限,更适合进行大型水母科间和科内属间的进化关系研究。
| 王彬,李玉龙,沈鸿等,2014. 2005-2013年辽东湾北部近海白色霞水母的数量分布.海洋渔业, 36(2):146-154 |
| 王建艳,甄毓,王国善等,2013.基于mt-16S rDNA和mt-COI基因的海月水母分子生物学鉴定方法和检测技术.应用生态学报, 24(3):847-852 |
| 卢振彬,戴泉水,颜尤明,2003.福建东山岛海域霞水母的渔业生物学研究.应用生态学报, 14(6):973-976 |
| 仲霞铭,汤建华,刘培廷,2004.霞水母(Cyanea nozakii Kisninouye)暴发与海洋生态之关联性探讨.现代渔业信息, 19(3):15-17 |
| 刘敏,马凌波,凌建忠等,2011.东黄海沙海蜇与口冠水母分类关系的辨析——基于核糖体18S rDNA基因序列.海洋渔业, 33(2):131-137 |
| 孙松,2012a.水母暴发研究所面临的挑战.地球科学进展, 27(3):257-261 |
| 孙松,2012b.对黄、东海水母暴发机理的新认知.海洋与湖沼, 43(3):406-410 |
| 杨傲傲,2014.海蜇微卫星标记的开发及应用.上海:上海海洋大学硕士学位论文, 1-79 |
| 张姝,张芳,刘媛等,2009.我国海域两种大型水母的分子鉴定.海洋与湖沼, 41(1):94-101 |
| 葛立军,何德民,2004.生态危机的标志性信号——霞水母旺发,今年辽东湾海蜇大面积减产.中国水产, (9):23-25 |
| 董婧,刘春洋,李文泉等,2005.白色霞水母的形态与结构.水产科学, 24(2):22-23 |
| 董婧,姜连新,孙明等,2013.渤海与黄海北部大型水母生物学研究.北京:海洋出版社 |
| 程家骅,李圣法,丁峰元等,2004.东、黄海大型水母暴发现象及其可能成因浅析.现代渔业信息, 19(5):10-12 |
| Bayha K M, Dawson M N, Collins A G et al,2010. Evolutionary relationships among scyphozoan jellyfish families based on complete taxon sampling and phylogenetic analyses of 18S and 28S ribosomal DNA. Integrative and Comparative Biology, 50(3):436-455 |
| Bayha K M, Graham W M,2009. A new Taqman© PCR-based method for the detection and identification of scyphozoan jellyfish polyps. Hydrobiologia, 616(1):217-228 |
| Bucklin A, Frost B W, Grieve J B et al,2003. Molecular systematic assessment of thirty-four calanoid copepod species of the Calanidae and Clausocalanidae using DNA sequences of mtCOI and nuclear 18S rRNA. Marine Biotechnology, 142:333-343 |
| Chen C A, Chen C-P, Fan T-Y et al,2002. Nucleotide sequences of ribosomal internal transcribed spacers and their utility in distinguishing closely related Perinereis polychaets (Annelida; Polychaeta; Nereididae). Marine Biotechnology, 4(1):l7-29 |
| Chu K H, Li P C, Ho H Y,2001. The first internal transcribed spacer (ITS-1) of ribosomal DNA as a molecular marker for phylogenetic and population analyses in crustacean. Marine Biotechnology, 3(4):355-361 |
| Dawson M N, Gupta A S, England M H,2005. Coupled biophysical global ocean model and molecular genetic analyses identify multiple introductions of cryptogenic species. Proceedings of the National Academy of Sciences of the United States of America, 102(34):11968-11973 |
| Dong J, Liu C Y, Wang Y Q et al,2006. Laboratory observations on the life cycle of Cyanea nozakii (Semeostomida, Scyphozoa). Acta Zoologica Sinica, 52(2):389-395 |
| Dong J, Sun M, Wang B et al,2008. Comparison of life cycles and morphology of Cyanea nozakii and other scyphozoans. Plankton and Benthos Research, 3(Suppl.):118-124 |
| Hebert P D N, Cywinska A, Ball S L et al,2003a. Biological identifications through DNA barcodes. Proceedings of the Royal Society B, 270(1512):313-321 |
| Hebert P D N, Ratnasingham S, de Waard J R,2003b. Barcoding animal life:cytochrome c oxidase subunit 1 divergences among closely related species. Proceedings of the Royal Society B, 270(S1):S96-S99 |
| Hwang U-W, Kim W,1999. General properties and phylogenetic utilities of nuclear ribosomal DNA and mitochondrial DNA commonly used in molecular systematics. The Korean Journal of Parasitology, 37(4):215-228 |
| Kawahara M, Uye S, Ohtsu K et al,2006. Unusual population explosion of the giant jellyfish Nemopilema nomurai (Scyphozoa:Rhizostomeae) in East Asian waters. Marine Ecology Progress Series, 307:161-173 |
| Ki J-S, Hwang D-S, Lee D-S,2010. Simultaneous detection of Aurelia and Chrysaora scyphozoan jellyfish on a DNA microarray. Journal of the Marine Biological Association of the United Kingdom, 90(6):1111-1117 |
| Ki J-S, Kim I-C, Lee D-S,2009. Comparative analysis of nuclear ribosomal DNA from the moon jelly Aurelia sp. 1(Cnidaria:Scyphozoa) with characterizations of the 18S, 28S genes, and the intergenic spacer (IGS). In:Pitrt K A, Purcell J E eds. Jellyfish Blooms:Causes, Consequences, and Recent Advances. Netherlands:Springer, 229-239 |
| Kumar S, Tamura K, Nei M,2004. MEGA 3:Integrated software for molecular evolutionary genetics analysis and sequence alignment. Briefings in Bioinformatics, 5(2):150-163 |
| Purcell J E,2007. Environmental effects on asexual reproduction rates of the scyphozoan Aurelia labiata. Marine Ecology Progress Series, 348:183-196 |
| Schlötterer C,1998. Ribosomal DNA probes and primers. In:Karp A, Isaac P G, Ingram D S eds. Molecular Tools for Screening Biodiversity. Netherlands:Springer, 267-276 |
| Thompson J D, Gibson T J, Plewniak F et al,1997. The Clustal_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25(24):4876-4882 |
| Wanna W, Chotigeat W, Phongdara A et al,2006. Sequence variations of the first ribosomal internal transcribed spacer of Penaeus species in Thailand. Journal of Experimental Marine Biology and Ecology, 331(1):64-73 |
 2016, Vol. 47
2016, Vol. 47