
中国海洋湖沼学会主办。
文章信息
- 韩承慧, 马海涛, 姜海滨, 刘阳, 韩慧宗, 王斐. 2016.
- HAN Cheng-Hui, MA Hai-Tao, JIANG Hai-Bin, LIU Yang, HAN Hui-Zong, WANG Fei. 2016.
- 许氏平鲉(Sebastes schlegeli)微卫星标记开发及野生、养殖群体遗传多样性分析
- ISOLATION OF MICROSATELLITE MARKERS FOR BLACK ROCKFISH SEBASTES SCHLEGELI AND GENETIC DIVERSITY OF WILD AND CULTURED POPULATIONS
- 海洋与湖沼, 47(1): 213-220
- Oceanologia et Limnologia Sinica, 47(1): 213-220.
- http://dx.doi.org/10.11693/hyhz20150900235
-
文章历史
- 收稿日期: 2015-09-09
- 收修改稿日期: 2015-12-26
2. 山东省海洋资源与环境研究院山东省海洋生态修复重点实验室烟台 264006;
3. 中国科学院南海海洋研究所 广州 510301
2. Shandong Provincial Key Laboratory of Restoration for Marine Ecology, Shandong Marine Resource and Environment Research Institute, Yantai 264006, China;
3. South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, China
许氏平鲉(Sebastes schlegeli),又称黑鲪,属鲉形目(Scorpaeniformes)、鲉亚目(Scorpaenoidei)、鲉科(Scorpaenidae)、平鲉亚科(Sebastinae)、平鲉属(Sebastodes)(成庆泰等,1987),广泛分布于东海、黄海和渤海,属于卵胎生鱼类(冯昭信,2003),具有肉质鲜美、营养丰富、生长速度快、抗逆性强等优点,是我国北方海区以及日本、韩国的重要养殖鱼类之一(刘丽娟等,2009)。目前养殖苗种以海捕野生苗为主要来源,养殖成活率较低,长期的过度捕捞也使许氏平鲉资源和遗传多样性遭到破坏,因此,进行其良种选育已成为保护我国许氏平鲉资源、促进其养殖业可持续发展的必要手段(刘立明等,2010)。
微卫星DNA标记,又称简单序列重复(simple sequence repeats,SSR),具有多态性高、稳定性高、信息含量大、共显性等特点(徐莉等,2002),已经被广泛应用于水产动物分子标记与数量性状的相关系分析、遗传结构分析、亲缘关系鉴定、遗传连锁图谱构建和QTL定位中(Zhan et al,2005; 孙国华等,2011; 王文琪等,2012; 初冠囡等,2013)。
目前国内外对于许氏平鲉微卫星标记的研究处于起步阶段,发表的微卫星标记较少。日本学者Yoshida等(2005)首先利用两重PCR扩增的方法获得6个微卫星标记; 韩国学者An等(2009)利用磁珠富集法获得了14个微卫星标记; Yasuike等(2013)利用454测序法筛选了17个微卫星标记。国内方面,Bai等(2011)运用磁珠富集的方法获得了18个微卫星标记; 初冠囡等(2013)发表了17个许氏平鲉微卫星标记; 贾超峰等(2014)报道了15个具有多态性的微卫星标记。这些微卫星标记远远不能满足许氏平鲉群体遗传结构分析、遗传图谱构建和分子标记辅助育种的需要。本研究首次运用高通量测序的方法从许氏平鲉基因组中开发24个多态性较高的微卫星标记,并利用其对野生群体和养殖群体的遗传多样性进行评价。
1 材料与方法 1.1 材料许氏平鲉野生群体采自山东荣成附近海域,养殖群体采自烟台泰华海洋科技有限公司(国家级黑鲪原种场)选育的长岛群体选育子一代,子一代个体为混合群体随机取样。荣成野生群体和烟台养殖群体各取36尾,活体采集部分鱼鳍样品后浸泡于70%乙醇中,带回实验室-20℃保存备用。
1.2 基因组DNA的提取采用常规酚-氯仿抽提法提取基因组DNA(黄培堂,2002)。1%琼脂糖凝胶电泳检测所提取的基因组DNA的完整性,NanoDrop2000分光光度计检测DNA浓度和纯度。使用时稀释到50 ng/μL。
1.3 微卫星引物的设计和筛选选取完整性和纯度高的单个个体DNA送至北京诺禾致源生物信息科技有限公司进行基因组高通量测序,并对含有微卫星核心的序列设计引物,选取200对微卫星引物送由上海生工生物工程有限公司合成。首先选用三个不同个体DNA模板进行初步筛选,选出能稳定扩增的微卫星标记,再通过温度梯度PCR进行退火温度的优化。优化后引物分别用FAM(荧光颜色为蓝色)和HEX(荧光颜色为绿色)进行标记(表 1),将筛选得到的微卫星标记在野生群体中扩增,对微卫星标记进行评价,然后在养殖群体中扩增,对野生群体和养殖群体的遗传多样性进行评价。PCR反应体系为25μL,包括2μL DNA模板(50ng/μL),2.5μL 10×buffer,引物各1μL(50μmol/L),0.5μL dNTP(10 mmol/L),0.2μL Taq DNA聚合酶(U/μL),灭菌水补足。PCR反应程序为; 94℃预变性5min,94℃变性40s,退火反应40s,72℃延伸1min,30个循环,最后72℃延伸10min。PCR产物经由1.5%琼脂糖凝胶电泳检测后,送至上海生工生物工程有限公司进行毛细管电泳。
| 位点 | GenBank号 | 重复基元 | 引物序列(5'—3') | 退火温度Ta(℃) | 产物长度(bp) |
| HJ6-10 | KT260126 | (CA)16 | F: FAM-ATTCTCATTCTACCCCATCCCATT | 60 | 150 |
| R: CTGGCTGAGCTAACACGCATT | |||||
| HJ6-18 | KT260127 | (TA)16 | F:HEX-TCGAGTGATGCGATATATGGTACG | 50 | 130 |
| R: GACATGGAAACTGCAACTTGACTG | |||||
| HJ6-19 | KT260128 | (GT)10 | F:HEX-TTGCAGACAGAATAAAATTCACGG | 60 | 144 |
| R: TGGCTTTACAACCATATCACTCCC | |||||
| HJ6-23 | KT260129 | (TGC)10 | F: FAM-GACGAGCTTCTTCCCATCAGG | 60 | 140 |
| R: TGACCACACCATCACTAGAAGAGC | |||||
| HJ6-30 | KT260130 | (AC)6 | F:HEX-ACTGAGCAACACAAGATGAAGACG | 60 | 159 |
| R: AGTGTGTGTGTGTGTGTGTGTGTG | |||||
| HJ6-31 | KT260131 | (CA)7 | F: HEX-CCGGTCTCTTTTTCCTTCTGTCTT | 60 | 122 |
| R: CTGTTCTGATGTAATGCGCTATGG | |||||
| HJ6-32 | KT260132 | (TG)6 | F: HEX-TGAGGCAAAGCATTACAAAATCTG | 60 | 139 |
| R: ATGTTTTGGTGTCATTTACAGGCA | |||||
| HJ6-56 | KT260133 | (TC)7 | F: FAM-CACAACTGCTGCCAATATTCATGT | 60 | 158 |
| R: CCAAACCCCATTACACACATACAG | |||||
| HJ6-57 | KT260134 | (TG)6 | F: FAM-AGTAATCTGACTGTCTGCGCTGC | 60 | 143 |
| R: AAGGATACCACAAAGCATGGTCAC | |||||
| HJ6-70 | KT260135 | (TAT)4 | F: HEX-TTTTTGTGTGCCTAATCTGAACCA | 60 | 140 |
| R: AAGGTGTTTCATGTGCATTTTGTG | |||||
| HJ6-80 | KT260136 | (TC)15 | F: HEX-CCTCACAACTTTCACCACCTCTCT | 60 | 122 |
| R: TGCAGTAAATCCATTGTCGAAGAA | |||||
| HJ7-2 | KT260137 | (AC)9 | F: FAM-ACACACGTTCTTACAGAAGGACCC | 60 | 144 |
| R: CAATGGCATAATTTGGTGTTGAAA | |||||
| HJ7-20 | KT260138 | (TG)13 | F: HEX-AATCCTCTGATTACCTAGCCAGCA | 62 | 127 |
| R: TACAGCGTTTGGTGTCTCTGAACT | |||||
| HJ7-22 | KT260139 | (TG)14 | F:HEX-AGTTAGCTGGGAAGGAAAAGCACT | 62 | 109 |
| R: ACCTTACTGCACCTGTTCTCTTGG | |||||
| HJ7-38 | KT260140 | (GGC)4 | F: HEX-GCAATCGAATTACCGTCCATTTTA | 60 | 134 |
| R: CAGCTCCACTGAACAGGACTAAGG | |||||
| HJ7-44 | KT260142 | (GA)8 | F: HEX-CTCCTCCTACTCCAGGGTCAAAGT | 60 | 117 |
| R: CGCTGAACTCTAAGCAACCTCATT | |||||
| HJ7-45 | KT260143 | (AAT)4 | F: FAM-GCTGTGTGGCTCCTCTAACCTG | 60 | 160 |
| R: TACTGATGGCAGAACTGCTTCTTG | |||||
| HJ7-46 | KT260144 | (GCA)5 | F: HEX-TAGGCCTATGTGGCTCAAGGTA | 60 | 117 |
| R: TCAATTTTAAGGACATGCATCACA | |||||
| HJ7-59 | KT260145 | (GT)10 | F: FAM-AGCTTACCTGTACCTGAGTGTCGG | 60 | 141 |
| R: CCGGCAGAGTAAAACAGACAAACT | |||||
| HJ7-60 | KT260146 | (TG)15 | F: FAM-ATATGTGTGAAGCTGCTGCATGTC | 60 | 128 |
| R: TCAGGCAAATCAACACTAGTCAGC | |||||
| HJ7-66 | KT260147 | (CA)8 | F: FAM-AATCATGATGTTTCCGATCGTCTT | 62 | 155 |
| R: CACAGTACTCTACGCTCTACGGCA | |||||
| HJ7-67 | KT260148 | (TAA)10 | F:FAM-CCAGAATTGCTGTGGAAGAAAAGT | 62 | 143 |
| R: CCTCTCCTTCCTTTTCCTCATCTT | |||||
| HJ7-68 | KT260149 | (TCTA)4 | F: FAM-ATCTGGTCGCAGATACAGGACAGT | 60 | 158 |
| R: TTTCTAGTGACACATATTTCTGCCCTT | |||||
| HJ7-100 | KT260150 | (AGGT)4 | F: FAM-TTTCCAGGCAGGTAGGTAGTTAGG | 60 | 160 |
| R: GGTGACAAAGTTGAACATGTGAGG |
PopGene32软件统计分析每一个位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He)、香农-威纳指数(I); GenePop软件检验Hardy-Weinberg平衡,并经Bonferroni校正; PIC Calc 6.0计算每个位点的多态信息含量(PIC)。
2 结果与分析 2.1 DNA提取结果所提取的群体基因组DNA溶解于50μL的T.E Buffer(pH=8.0)中,浓度在800—1000 ng/μL,1%琼脂糖凝胶电泳结果显示条带明显,无拖带降解现象,紫外分光光度计测定OD260nm/OD280nm在1.8—1.9之间,说明样品DNA中蛋白质、RNA等残存较少,适合PCR扩增。
2.2 微卫星标记的多态性分析共筛选出24个具有多态性的微卫星标记,峰值多在5000—40000之间,电泳条带清晰,说明这些微卫星标记具有较高的可靠性,能满足群体遗传多样性分析的需要(图 1)。24对微卫星标记在野生群体表现出了较高的多态性(表 2),在野生群体中共检测出204个等位基因,每个微卫星位点的等位基因数(Na)在2—21之间,平均等位基因数为8.5个,其中位点HJ6-30、HJ6-32、HJ6-57和HJ7-46等位基因数最少,各得到2个等位基因,位点HJ7-60的等位基因数最多,得到21个等位基因。有效等位基因数(Ne)为1.3340—15.3373,平均值为4.5484; 观测杂合度(Ho)为0.0417—0.9167,平均值为0.6059; 期望杂合度(He)范围为0.0278—0.9722,平均值为0.6041; 多态信息含量(PIC)在0.1948—0.9496之间,平均值为0.6088,其中高度多态的位点(PIC>0.5)有14个,占总位点数的58.33%,中度多态位点(0.25<PIC<0.5)有6个,占25%,低度多态位点(PIC<0.25)有4个,占16.67%。
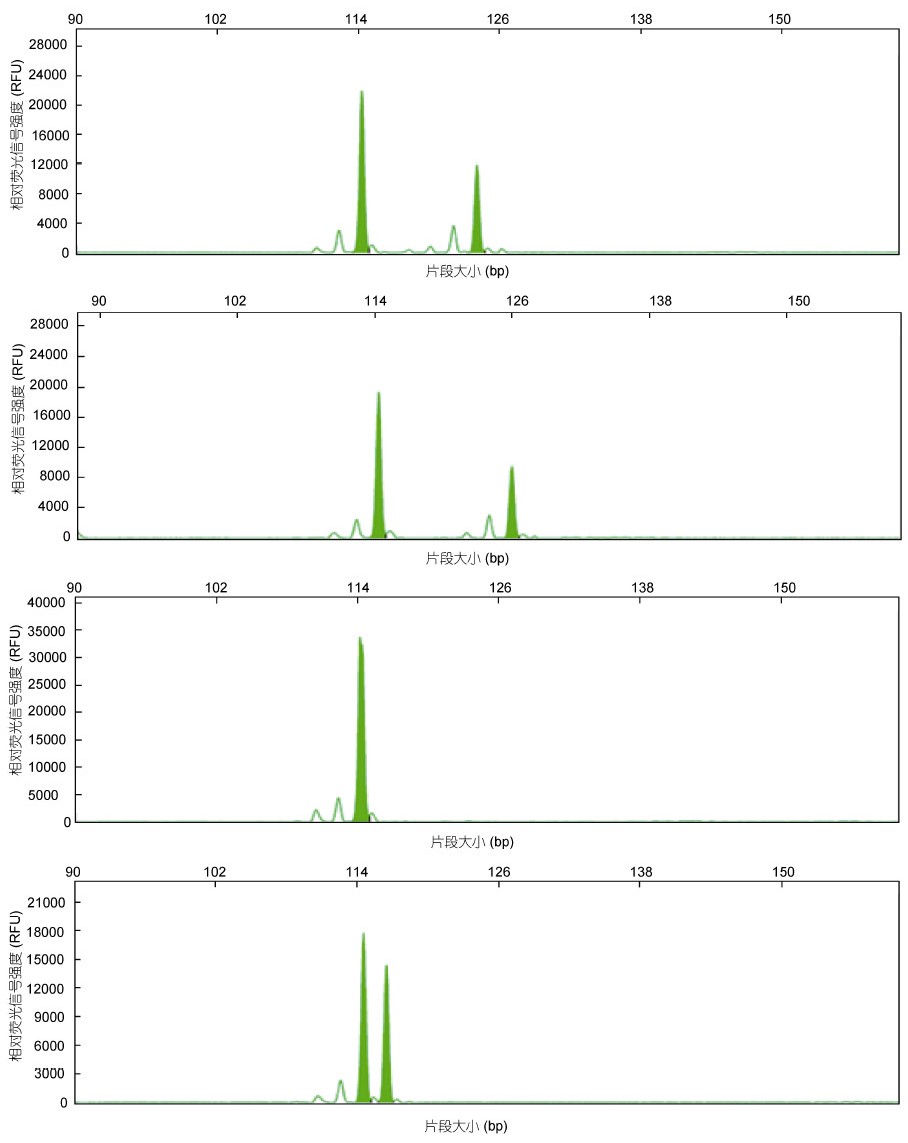 |
| 图 1 位点HJ7-44的部分毛细管电泳结果 Fig. 1 Samples of capillary electrophoresis results for locus HJ7-44 |
| 微卫星 位点 | 群体 | 等位基因数 (Na) | 有效等位基因数 (Ne) | 观测杂合度 (Ho) | 期望杂合度 (He) | 多态信息含量 (PIC) | 香农-威纳指数 (I) | Hardy-Weinberg (P) |
| HJ6-10 | YS | 8 | 4.9560 | 0.6667 | 0.8095 | 0.7674 | 1.7197 | 0.0606 |
| YZ | 6 | 4.2702 | 0.7778 | 0.7766 | 0.7356 | 1.6143 | 0.1503 | |
| HJ6-18 | YS | 13 | 7.4057 | 0.6389 | 0.8772 | 0.8508 | 2.1971 | 0.0005* |
| YZ | 9 | 2.3060 | 0.6111 | 0.5743 | 0.5451 | 1.3082 | 0.4332 | |
| HJ6-19 | YS | 18 | 7.7373 | 0.9167 | 0.8830 | 0.8606 | 2.4290 | 0.9564 |
| YZ | 14 | 5.7093 | 0.9444 | 0.8365 | 0.8053 | 2.0718 | 0.0004* | |
| HJ6-23 | YS | 16 | 9.3237 | 0.8056 | 0.9053 | 0.8835 | 2.4311 | 0.2287 |
| YZ | 13 | 9.2903 | 0.9722 | 0.9049 | 0.8825 | 2.3464 | 0.0010* | |
| HJ6-30 | YS | 2 | 1.9985 | 0.9722 | 0.5067 | 0.3748 | 0.6928 | 0.0000* |
| YZ | 2 | 1.1484 | 0.1389 | 0.1311 | 0.1208 | 0.2522 | 1.0000 | |
| HJ6-31 | YS | 4 | 1.8215 | 0.0278 | 0.4574 | 0.4033 | 0.8312 | 0.0000* |
| YZ | 2 | 1.0571 | 0.0556 | 0.0548 | 0.0526 | 0.1269 | 1.0000 | |
| HJ6-32 | YS | 2 | 1.3846 | 0.3333 | 0.2817 | 0.2392 | 0.4506 | 0.5612 |
| YZ | 2 | 1.2129 | 0.1944 | 0.1780 | 0.1601 | 0.3189 | 1.0000 | |
| HJ6-56 | YS | 6 | 3.5217 | 0.7222 | 0.7261 | 0.6660 | 1.4054 | 0.3286 |
| YZ | 3 | 2.3845 | 0.6389 | 0.5888 | 0.5149 | 0.2522 | 0.3852 | |
| HJ6-57 | YS | 2 | 1.8215 | 0.2500 | 0.2218 | 0.1948 | 0.3768 | 1.0000 |
| YZ | 4 | 1.1520 | 0.1111 | 0.1338 | 0.1282 | 0.3182 | 0.0156 | |
| HJ6-70 | YS | 6 | 2.2618 | 0.4722 | 0.5657 | 0.5169 | 1.1335 | 0.1777 |
| YZ | 9 | 4.0725 | 0.8056 | 0.7653 | 0.7267 | 1.7138 | 0.0342 | |
| HJ6-80 | YS | 19 | 11.5200 | 0.8333 | 0.7422 | 0.9074 | 2.6757 | 0.0000* |
| YZ | 14 | 8.6400 | 0.9722 | 0.8967 | 0.8735 | 2.3333 | 0.0538 | |
| HJ7-2 | YS | 8 | 3.7295 | 0.8333 | 0.7422 | 0.7009 | 1.6088 | 0.3581 |
| YZ | 8 | 2.8452 | 0.6667 | 0.6577 | 0.6208 | 1.4309 | 0.1337 | |
| HJ7-20 | YS | 12 | 8.0000 | 0.8056 | 0.8873 | 0.8634 | 2.2610 | 0.1482 |
| YZ | 11 | 5.3665 | 0.8056 | 0.8251 | 0.7918 | 1.9447 | 0.0028 | |
| HJ7-22 | YS | 9 | 4.5474 | 0.7500 | 0.7911 | 0.9496 | 1.7204 | 0.1473 |
| YZ | 10 | 6.0988 | 0.8333 | 0.8478 | 0.8180 | 2.0174 | 0.0154 | |
| HJ7-38 | YS | 4 | 1.3340 | 0.2778 | 0.2539 | 0.2384 | 0.5340 | 1.0000 |
| YZ | 3 | 1.2186 | 0.1389 | 0.1819 | 0.1702 | 0.3771 | 0.2570 | |
| HJ7-44 | YS | 5 | 2.0506 | 0.5556 | 0.5196 | 0.4592 | 0.9634 | 0.2546 |
| YZ | 4 | 2.7429 | 0.7500 | 0.6444 | 0.5730 | 1.1370 | 0.0338 | |
| HJ7-45 | YS | 5 | 2.0250 | 0.4722 | 0.5133 | 0.4254 | 0.8766 | 0.8105 |
| YZ | 6 | 2.7284 | 0.6667 | 0.6424 | 0.5805 | 1.2092 | 0.1868 | |
| HJ7-46 | YS | 2 | 1.3846 | 0.2222 | 0.2817 | 0.2392 | 0.4506 | 0.2280 |
| YZ | 2 | 1.2800 | 0.1944 | 0.2218 | 0.1948 | 0.3768 | 0.4337 | |
| HJ7-59 | YS | 12 | 5.0625 | 0.7222 | 0.8138 | 0.7839 | 1.9645 | 0.4337 |
| YZ | 9 | 4.5000 | 0.8056 | 0.7887 | 0.7463 | 1.7247 | 0.0106 | |
| HJ7-60 | YS | 21 | 15.3373 | 0.8056 | 0.9480 | 0.9310 | 2.8524 | 0.0306 |
| YZ | 14 | 7.0820 | 0.8333 | 0.8709 | 0.8459 | 2.2601 | 0.0000* | |
| HJ7-66 | YS | 8 | 2.0523 | 0.4167 | 0.5200 | 0.4900 | 1.1574 | 0.0362 |
| YZ | 5 | 2.0653 | 0.6111 | 0.5231 | 0.4797 | 1.0275 | 0.9435 | |
| HJ7-67 | YS | 9 | 3.3402 | 0.5556 | 0.7105 | 0.6743 | 1.5974 | 0.0893 |
| YZ | 7 | 2.2958 | 0.5000 | 0.5724 | 0.5284 | 1.1802 | 0.0114 | |
| HJ7-68 | YS | 10 | 5.3030 | 0.9429 | 0.8232 | 0.7929 | 1.9398 | 0.9049 |
| YZ | 7 | 5.5267 | 0.8889 | 0.8306 | 0.7955 | 1.8070 | 0.0049 | |
| HJ7-100 | YS | 3 | 1.7851 | 0.5000 | 0.4460 | 0.3988 | 0.7834 | 1.0000 |
| YZ | 3 | 2.2797 | 0.8611 | 0.5692 | 0.4847 | 0.9293 | 0.0000* | |
| 平均值 | YS | 8.5000 | 4.5484 | 0.6041 | 0.6421 | 0.6088 | 1.4605 | |
| YZ | 6.9583 | 3.6365 | 0.6157 | 0.5840 | 0.5490 | 1.2834 | ||
| *表示显著偏离Hardy-Weinberg平衡; 野生群体用YS表示,养殖群体用YZ表示 | ||||||||
各个微卫星位点的Hardy-Weinberg平衡检测结果表明(表 2),在野生群体中HJ6-18、HJ6-30、HJ6-31和HJ6-80显著偏离了Hardy-Weinberg平衡,偏离位点数占总位点数的16.67%,在养殖群体中HJ6-19、HJ6-23、HJ7-60和HJ7-100显著偏离平衡,占总位点数的16.67%。其余位点均符合Hardy- Weinberg平衡。
2.4 野生和养殖群体遗传多样性分析将筛选得到的24个微卫星标记在养殖群体中扩增,结果如下; 养殖群体的等位基因数(Na)为2—14个,平均值为6.9583个; 有效等位基因数(Ne)为1.0571—9.2903,平均值为3.6365; 观测杂合度(Ho)范围为0.0556—0.9722,平均值为0.6157; 期望杂合度(He)为0.0548—0.9049,平均值为0.5840; 多态信息含量(PIC)为0.0526—0.8825,平均值为0.5490; 野生群体和养殖群体的香农-威纳多样性指数为0.3768—2.8524、0.1269—2.3464,平均值分别为1.4605和1.2834,比较野生群体和养殖群体这六组数据的均值,除观测杂合度(Ho)外,野生群体都高于养殖群体,但对以上六组数据F检验无显著差异。说明许氏平鲉野生群体的遗传多样性高于养殖群体的遗传多样性,但是两者之间的差异并不显著。
3 讨论微卫星标记是分子遗传研究中的一种重要手段,其获得的方法有多种; 基因组文库法、微卫星富集法、数据库查找法、近缘物种筛选法等(孙波等,2009)。本研究首次使用高通量测序的方法从许氏平鲉基因组中开发微卫星标记,设计微卫星引物200对,结果得到了190对可稳定扩增的引物,占95%,其中24对微卫星引物具有较高多态性,证明这种方法可用于许氏平鲉基因组微卫星标记的大规模开发。Weber(1990)认为只有在2碱基重复序列次数达到12次时微卫星标记才能表现出较高的多态性。低度多态(PIC<0.25)的位点有四个,分别是HJ6-32和HJ6-57为两碱基6重复,HJ7-38为三碱基4重复和HJ7-46为三碱基5重复,中度多态(0.25<PIC<0.5)的位点有4个,HJ6-30、HJ6-31、HJ7-66为两碱基重复,最多重复次数只有8次,HJ7-100为四碱基重复4次,以上8个位点的核心序列重复均少于12次,相反核心序列重复次数多于12次的微卫星标记均检测出较高的杂合度和丰富的多态性,这种现象在诸氏鲻虾虎、三疣梭子蟹、中国明对虾(蔡磊等,2015)中也有报道,因此在设计引物时应选用核心序列重复单元的重复次数在较高水平的序列,从而避免筛选过程中得到较多低多态性的微卫星标记。
24个微卫星位点在野生群体中共检测出204个等位基因,每个位点扩增出的等位基因数目2—21个不等,明显高于贾超峰等(2014)观测到的2—14个和初冠囡等(2013)观测到的2—8个,这可能与所选用的检测方法有关,以上两者选用的为聚丙烯酰胺凝胶电泳,此种方法分辨率较低,微卫星因其多态性较高,核心重复次数相差较少的两条等位基因条带可能相近,导致条带判读产生误差。本实验选用的是毛细管电泳,毛细管电泳技术因其高灵敏度、高分辨率、高速度、低耗样等优势,已经逐步代替了经典电泳技术(毛煜等,2001),得到的结果分辨率和准确性更高。
24个微卫星标记在野生群体和养殖群体中各有四个位点显著偏离了Hardy-Weinberg平衡,主要原因是纯合子过剩。野生许氏平鲉营半定栖生活,活动范围较小,这种生活习性有可能造成近亲交配,从而出现微卫星位点偏离Hardy-Weinberg平衡现象; 而养殖群体所用的亲本数量一般比较少,也可能造成近亲交配并出现微卫星位点偏离Hardy-Weinberg平衡现象。另外,无效等位基因的存在也能导致微卫星位点偏离Hardy-Weinberg平衡现象。研究表明在海洋经济鱼类、贝类、棘皮类的养殖群体中被检测位点偏离Hardy-Weinberg平衡的现象普遍存在(耿慧君等,2009)。
最大限度地维持种内遗传多样性水平,是持续利用种质资源的前提和基础。群体的遗传多样性每丧失10%,就会对其繁育能力、存活率、生长等重要性状产生很大的负面影响(Allendorf et al,1987)。多态信息含量(PIC)是指一个后代所获的某个等位基因标记来自它亲本的同一个等位标记的可能性大小(彭银辉等,2008),野生群体和养殖群体的平均PIC值均大于0.5,表明两个群体均表现出较高的遗传多样性。野生群体的等位基因数、有效等位基因数、Shannon’s指数、期望杂合度和多态信息含量的平均值都高于养殖群体,但对以上参数进行F检验后发现均无显著差异(P>0.05)。一方面,本结果说明许氏平鲉养殖群体仍保持有较高的遗传多样性,原因可能与养殖群体的亲本数量较大和选育代数较少有关,我们在进行许氏平鲉群体选育时,一般会选择较大数量的优质个体作为亲本,从而有效地防止由于亲本数量太少而造成选育子代遗传多样性降低的风险; 另外由于本研究所用养殖群体为群体选育子一代,较少的选育代数减少了近亲交配风险的发生,从而也避免了选育子代遗传多样性降低的发生。另一方面,单从数据上看野生群体的遗传多样性略高于养殖群体,这说明人工定向选育对选育群体的遗传多样性也产生了一定的影响,这提醒我们在下一步的良种选育中,应进一步采取避免选育群体遗传多样性降低的措施,如定期补充野生个体作为亲本等,并利用微卫星标记对其选育后代继续进行遗传监控,从而达到较好的选育效果。
本研究开发的24对许氏平鲉微卫星标记为其遗传连锁图谱构建和分子标记辅助育种提供了更多标记选择,对野生群体和养殖群体的遗传多样性分析结果也为下一步的新品系选育提供了参考。
| 王文琪,张毅,刘梦侠等, 2012.许氏平鲉4个野生群体遗传多样性微卫星分析.海洋科学, 36(1):10-16 |
| 毛煜,徐建明, 2001.毛细管电泳技术和应用新进展.化学研究与应用, 13(1):4-9 |
| 冯昭信, 2003.鱼类学(海洋渔业专业用).北京:中国农业出版社, 191 |
| 成庆泰,郑葆珊, 1987.中国鱼类系统检索(下册).北京:科学出版社, 462-463 |
| 刘立明,杜荣斌,孙颖民等, 2010.黑鲪苗种培育关键技术的研究.海洋科学, 34(3):1-5 |
| 刘丽娟,任利华,姜向阳等, 2009.黑鲪(Sebastes schtegeli)遗传多样性的等位酶研究.海洋与湖沼, 40(4):479-483 |
| 孙波,鲍毅新,赵庆洋等, 2009.微卫星位点获取方法的研究进展.生物学杂志, 28(10):2030-2137 |
| 孙国华,杨建敏,孙孝德等, 2011.刺参微卫星标记与生长性状体重、体长的相关分析.水产学报, 35(4):501-508 |
| 初冠囡,姜黎明,于海洋等, 2013.卵胎生鱼许氏平鲉(Sebastes schlegelii)雌亲家系的微卫星鉴定.海洋与湖沼, 44(3):670-676 |
| 耿慧君,周遵春,董颖等, 2009.中间球海胆野生和养殖群体遗传结构的微卫星分析.水产学报, 33(4):549-556 |
| 贾超峰,马海涛,姜海滨等, 2014.许氏平鲉微卫星标记的开发及评价.上海海洋大学学报, 23(3):345-350 |
| 徐莉,赵桂仿, 2002.微卫星DNA标记技术及其在遗传多样性研究中的应用.西北植物学报, 22(3):714-722 |
| 黄培堂, 2002.分子克隆实验指南(第三版:上册).北京:科学出版社, 483 |
| 彭银辉,刘楚吾,郭昱嵩等, 2008.三种笛鲷的野生群体和养殖群体遗传多样性的微卫星分析.农业生物技术学报, 16(5):810-814 |
| 蔡磊,陈小曲,郑伟强等, 2015.诸氏鲻虾虎鱼多态性微卫星标记的开发及评价.中国实验动物学报, 23(1):57-62 |
| Allendorf F W, Ryman N, 1987. Genetic management of hatchery stocks. In:Ryman N, Utter F eds. Population Genetics and Fishery Management. Seattle:University of Washington Press, 141-159 |
| An H S, Park J Y, Kim M J et al, 2009. Isolation and characterization of microsatellite markers for the heavily exploited rockfish Sebastes schlegeli, and cross-species amplification in four related Sebastes spp. Conservation Genetics, 10(6):1969-1972 |
| Bai C C, Liu S F, Zhuang Z M et al, 2011. Isolation and characterization of microsatellite markers for the Korean rockfish, Sebastes schlegeli. Genetics and Molecular Research, 10(3):2065-2068 |
| Weber J L, 1990. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms. Genomics, 7(4):524-530 |
| Yasuike M, Noda T, Fujinami Y et al, 2013. Tri-, tetra-and pentanucleotide-repeat microsatellite markers for the Schlegel's black rockfish Sebastes schlegelii:the potential for reconstructing parentages. Conservation Genetics Resources, 5(2):577-581 |
| Yoshida K, Nakagawa M, Wada S et al, 2005. Multiplex PCR system applied for analysing microsatellite loci of Schlegel's black rockfish, Sebastes schlegeli. Molecular Ecology Notes, 5(2):416-418 |
| Zhan A B, Bao Z M, Wang X L et al, 2005. Microsatellite markers derived from bay scallop Argopecten irradians expressed sequence tags. Fisheries Science, 71(6):1341-1346 |
 2016, Vol. 47
2016, Vol. 47


