
中国海洋湖沼学会主办。
文章信息
- 周前进, 陈先锋, 蔡怡, 段丽君, 段维军, 苗亮, 陈炯. 2016.
- ZHOU Qian-Jin, CHEN Xian-Feng, CAI Yi, DUAN Li-Jun, DUAN Wei-Jun, MIAO Liang, CHEN Jiong. 2016.
- 孔石莼(Ulva pertusa)LAMP-LFD快速检测方法的建立
- DEVELOPMENT OF A NOVEL LAMP-LFD METHOD FOR RAPID DETECTION OF ULVA PERTUSA
- 海洋与湖沼, 47(2): 380-389
- Oceanologia et Limnologia Sinica, 47(2): 380-389.
- http://dx.doi.org/10.11693/hyhz20141200344
-
文章历史
- 收稿日期:2014-12-10
- 改回日期:2015-10-19
2. 宁波检验检疫科学技术研究院 宁波 315012
2. Academy of Inspection and Quarantine, Ningbo 315012, China
孔石莼(Ulva pertusa)属于绿藻纲,石莼目,石莼科,石莼属。石莼属(Ulva)藻类呈全球性分布,多数种类生活在温带至亚热带海洋中,部分生长在高潮带至低潮带和大干潮线附近岩石上或石沼中。石莼属藻类富含蛋白和多种微量元素,具有很高的食用及药用价值(Torres et al,2014; Montingelli et al,2015; Ghadiryanfar et al,2016)。作为重要的经济藻类,孔石莼(U. pertusa)、浒苔(U. prolifera)等已得到越来越多的开发利用。然而,当石莼属藻类脱离固着基,形成大量的漂浮增殖群体时,将导致绿潮的暴发,破坏底栖生态系统。自20世纪70年代初法国布列塔尼沿海首次报道以来,绿潮发生范围已遍及欧洲沿海、美国东西海岸、东亚和东南亚沿海以及澳大利亚等国,成为世界性的海洋环境问题(Smetacek et al,2013)。
多年以来,石莼属等绿潮藻的分类鉴定主要依赖于显微镜下的形态学观察,通常以其形态、结构特征等作为主要的分类依据。然而,石莼属藻类不同生长阶段的细胞结构和藻体形态有着很好的可塑性,易随季节和环境的变化而变化(Blomster et al,1999,2002),导致形态学分类比较困难。因此,以PCR为代表的分子生物学技术引入到绿潮藻类的检测。其中,rDNA-ITS序列、rDNA-18s序列、二磷酸核酮糖羧化酶大亚基基因(rbcL)等因其种间较大的差异性,通常用作鉴定的靶标,应用于藻类的系统分类和鉴定过程(Blomster et al,2002; Wang et al,2010; Lin et al,2012); 由于该类技术有一定的仪器依赖性,要在特定环境下由专业人员操作,仅限于在实验室开展。
环介导等温扩增(loop-mediated isothermal amplification,LAMP)是2000年发展起来的一种新的核酸扩增方法(Notomi et al,2000)。相较于PCR技术,LAMP方法需针对靶序列的不同区域设计6—8条引物,这些引物在反应过程中需完全与靶序列匹配才能完成扩增反应,使得该方法具有良好的特异性。LAMP反应依赖于一种具有链置换功能的Bst DNA聚合酶,恒温条件下即可完成核酸的大量扩增,无需类似于PCR的反复升降温过程,使得仪器设备的依赖性大幅度降低。因此,自该技术发明以来已广泛应用于细菌、病毒、寄生虫等病原的检测(Niu et al,2012; Nie et al,2013; Notomi et al,2015)。LAMP产物的检测主要以琼脂糖凝胶电泳或浊度检测为主,仍依赖于凝胶成像系统和浊度仪等仪器(Parida et al,2008); 依赖于荧光染料的裸眼观察和实时荧光LAMP法在某些微生物的检测中也取得了应用,但非特异性扩增引起的假阳性成为限制该类检测手段广泛应用的重要因素(Mori et al,2001,2004; Schnetzinger et al,2013)。LAMP-LFD技术是利用生物素标记的引物进行LAMP反应,实现核酸的几何级数扩增,随后将生物素标记的LAMP产物与异硫氰酸荧光素标记的特异性探针进行杂交,杂交产物在经生物素抗体和异硫氰酸荧光素抗体处理的横向流动试纸条(lateral flow dipstick,LFD)上完成检测,阳性的杂交产物会在LFD的检测线位置呈现特征性的条带。相较于其它的LAMP产物检测手段,LFD无需任何仪器设备,易于操作和携带; 而且可通过探针的特异性杂交进一步筛选扩增产物,降低了结果的假阳性。该技术在传染性脾肾坏死病毒(ISKNV)和海豚链球菌等多种水产病原微生物的基层即时检测中展示了良好的适用性(Ding et al,2010; 王瑞娜等,2014); 同时,该方法在绿潮藻扁浒苔的的鉴别诊断中也展示了一定的应用潜力(陈先锋等,2015)。
本研究根据孔石莼核糖体DNA的内转录间隔区序列(rDNA-ITS)的保守区设计3对引物和1条异硫氰酸荧光素标记探针,优化反应条件后,建立LAMP- LFD技术,为水体环境中孔石莼的定期检测和快速检测提供一种快捷可靠的方法。
1 材料与方法 1.1 藻种分离株及培养实验所用的各微藻分离株(赤潮异弯藻、东海原甲藻、塔玛亚历山大藻、无纹环沟藻、锥状斯克里普藻等)由宁波大学海洋学院藻种实验室提供。实验中涉及的石莼属绿藻经母藻细断法分离后(Hiraoka et al,1998),按Duan等(2012)的方法于本实验室培养、保存。具体所用藻类见表 1,孔石莼分离株S66用于LAMP条件优化,灵敏度优化,特异性分析等试验。
| 藻类名称 | 英文名称 | 分离株 | 来源 |
| 孔石莼 | Ulva pertusa | S66 | 宁波检验检疫科学技术研究院培养 |
| 浒苔 | Ulva prolifera | XS5 | 宁波检验检疫科学技术研究院培养(孙东等,2011) |
| 曲浒苔 | Ulva flexuosa | SDF12 | 宁波检验检疫科学技术研究院培养(孙东等,2011) |
| 缘管浒苔 | Ulva linza | HS42 | 宁波检验检疫科学技术研究院培养 |
| - | Ulva ohnoi | FJ4 | 宁波检验检疫科学技术研究院培养 |
| 赤潮异弯藻 | Heterosigma akashiwo | H1 | 周成旭博士惠赠(周成旭等,2006) |
| 东海原甲藻 | Prorocentrum donghaiense | NMBjah045 | 周成旭博士惠赠(周成旭等,2006) |
| 塔玛亚历山大藻 | Alexandrium tamarense | NMBjah048 | 周成旭博士惠赠(王金娜等,2010) |
| 无纹环沟藻 | Gyrodinium instriatum | NMBjah046 | 周成旭博士惠赠(王金娜等,2010) |
按照植物组织基因组DNA提取试剂盒(离心柱型)的步骤,提取各藻类分离株的基因组DNA(Qiagen,Hilden,德国)。获取的基因组DNA溶解于50 μL的无菌去离子水中,经Nanodrop 2000分光光度计(Thermo Fisher Scientific,美国)测定浓度后用作标准品,-30°C贮存备用。
1.3 引物和探针设计根据孔石莼的rDNA-ITS序列(GenBank登录号: HM584747),在保守区域设计3对特异性引物(包括上游外引物UpeITS-F3、下游外引物UpeITS-B3、上游内引物UpeITS-FIP、下游内引物UpeITS-BIP,以及环引物UpeITS-LF和UpeITS-LB),用于LAMP实验(表 2,图 1)。此外,基于该扩增区段设计1条DNA探针UpeITS-HP用于LFD杂交试验(表 2,图 1)。其中,上游内引物UpeITS-FIP的5'端进行生物素(biotin)标记,探针UpeITS-HP的5'端进行异硫氰酸荧光素(FITC)标记。同时,以外引物UpeITS-F3和UpeITS-B3进行常规PCR扩增,扩增片段大小为360 bp。引物的合成和标记均由英潍捷基(上海)贸易有限公司完成。
| 引物 | 长度(碱基数) | 序列(5'—3') | 目的 |
| UpeITS-F3 | 19-mer | TAGCCTCACCTGAACTCAG | LAMP/PCR |
| UpeITS-B3 | 18-mer | ACGGATATCTCGGCTCTC | LAMP/PCR |
| UpeITS-FIP(F1c+F2)a | 39-mer(F1c:21-mer,F2:18-mer) | CGGCTGAAATACAGAGGCTCGTATCCTTCGTGGCTCACA | LAMP |
| UpeITS-BIP(B1c+B2) | 39-mer(B1c:20-mer,B2: 19-mer) | GTTATTCCGACGCTGAGGCAGCAGAATTCCGTGAGTCAT | TLAMP |
| UpeITS-LF | 21-mer | TAGGTAGCTCGCTACTCCTAC | LAMP |
| UpeITS-LB | 20-mer | GTGGTCTCATCCGAAGACTC | LAMP |
| UpeITS-HPb | 20-mer | GCAAGCGCGTGAGGGGTTAT | LFD |
| a上游引物UpeITS-FIP的5'端进行biotin标记; b UpeITS-HP的5'端进行FITC标记 | |||
 |
| 图 1 孔石莼rDNA-ITS序列的LAMP引物设计示意图 Fig. 1 Design of primers and DNA probe targeting ITS1-5.8S-ITS2 of U. pertusa used in LAMP-LFD assay |
LAMP反应体系参照Ding等(2010),具体组成包括(25 μL): UpeITS-F3和UpeITS-B3各0.2μmol/L,UpeITS-FIP和UpeITS-BIP各1.6μmol/L,UpeITS-LF和UpeITS-LB各0.4μmol/L,Tris-HCl(pH 8.80)20mmol/L,KCl 10mmol/L,(NH4)2SO4 10mmol/L,MgSO4 6.5mmol/L,dNTPs 1.4mmol/L,betaine 0.8mol/L,Triton X-100 0.1%,Bst DNA聚合酶(New England BioLabs,美国)8U,藻类基因组DNA标准品1μL,无菌去离子水补足25μL体系。对于反应条件的优化,主要从反应温度和时间两个方面考虑。Bst DNA聚合酶的最佳活性温度范围在60—65°C,根据已经建立的多个LAMP方法,我们得出63°C和65°C是最为常用的温度。因此,本研究中LAMP反应的温度设定为63°C,并在此基础上完成了引物的筛选。为进一步分析DNA模板浓度与LAMP反应效率之间的关系,我们在LAMP反应体系中加入0.1 mmol/L SYTO 9 荧光染料(Invitrogen,美国),在晶芯RT-Cycler实时荧光定量PCR仪(博奥生物有限公司,中国)上进行LAMP反应(即实时荧光LAMP反应),据此可实时观察反应产物的生成情况。操作可大致概括为: 将孔石莼S66的DNA标准品以10倍浓度单位进行梯度稀释,取3.04×102、3.04×101、3.04×100、3.04×10-1、3.04×10-2、3.04×10-3和3.04×10-4pg/L等7个浓度的基因组DNA为模板,在63°C下进行LAMP反应,根据起峰时间和扩增强度分析反应时间与模板浓度之间的关系,并初步确定LAMP反应的时间; 实时荧光LAMP的反应程序如下: 63°C 1min; 63°C15s和63°C 45s,60 个循环,于每个循环的 63°C 45s 末端收集荧光信号。在此基础上,分别选取较高浓度和最低检测浓度的基因组DNA为模板,反应时间依次设置为10、20、30、40、50和60min,进行LAMP反应,扩增产物经1.5%琼脂糖凝胶电泳分析,最终确定其反应时间。
1.5 利用LFD检测LAMP产物经biotin标记的LAMP反应结束后,向反应体系中加入2L 10pmol/L的探针UpeITS-HP,63°C杂交5 min,最后80°C温育5min终止反应,反应产物可利用LFD试纸条(Milenia GenLine HybriDetect,Milenia Biotec,德国)检测,具体操作如下: 取80L Buffer(Milenia GenLine HybriDetect,Milenia Biotec,德国)于1.5mL离心管内,加入5L杂交液混匀,将试纸条检测端竖直浸没预混液中反应5min,肉眼判断结果。当FITC标记的探针与biotin标记的扩增产物特异性杂交后,杂交产物与胶体金标记的FITC抗体结合形成的三元复合物结合在带有biotin抗体的检测线上; 多余的探针则与胶体金标记的FITC抗体结合后越过检测线,最终 结合在质控线上。
1.6 LAMP-LFD的特异性验证选择浒苔(U. prolifera)XS5、曲浒苔(U. flexuosa)SDF12、缘管浒苔(U. linza)HS42、U. ohnoi FJ4等常见石莼属绿藻分离株,以及赤潮异弯藻(Heterosigma akashiwo)H1、东海原甲藻(Prorocentrum donghaiense)、NMBjah045塔玛亚历山大藻(Alexandrium tamarense)NMBjah048、无纹环沟藻(Gyrodinium instriatum)NMBjah046和锥状斯克里普藻(Scrippsiella trochoidea)NMBjah044等国内常见微藻种类,用于LAMP-LFD的特异性实验。用于特异性实验的各藻类分离株的模板浓度统一调整为1.0×102pg/L,产物分别采用1.5%琼脂糖凝胶电泳和LFD进行检测。
1.7 LAMP-LFD的灵敏度分析取稀释后的孔石莼分离株S66的6个不同浓度的基因组DNA(3.04×102—3.04×10-3pg/L)为模板,按优化后的体系和条件进行有生物素标记的LAMP反应,扩增产物分别采用1.5%琼脂糖凝胶电泳和LFD进行检测。
以上述6个浓度的基因组DNA为模板,外引物UpeITS-F3和UpeITS-B3为引物,进行PCR扩增。25L PCR反应体系,包括: 10×PCR Buffer 2.5μL,dNTPs(0.25mmol/L)2μL,0.2μmol/L UpeITS-F3 1μL,0.2μmol/L UpeITS-B3 1μL,5U/μL rTaq DNA聚合酶(TaKaRa,大连,中国)0.25μL,基因组NDA模板1μL,用无菌去离子水补足25μL体系。PCR反应程序如下: 94°C预变性2min; 94°C 30s,52°C 30s,72°C30s,30个循环; 72°C延伸10min。扩增产物经1.5%琼脂糖凝胶电泳检测。
1.8 LAMP-LFD的重复性实验重新培养孔石莼分离株S66,平行制备3个样品,按步骤1.2的方法提取基因组DNA,测定浓度后将浓度稀释至LAMP-LFD的最低检测浓度,并以此为模板,进行有生物素标记的LAMP反应,产物分别采用1.5%琼脂糖凝胶电泳和LFD进行检测。
1.9 海水样品检测从福建和青岛等地区收集海水样品17份。样品经100目的筛绢网过滤后,各取400mL水样培养于500mL的三角烧瓶中若干天,至成熟藻体长成,培养方法参照Duan等(2012)所述。成熟藻体用于形态学鉴定。另取筛绢网滤后水样250mL按步骤1.2的方法提取基因组DNA,利用LAMP-LFD和PCR进行鉴定。
2 结果 2.1 LAMP反应时间的优化在63°C条件下,以不同浓度的基因组DNA为模板进行的实时荧光LAMP实验结果表明,当模板浓度在3.04×102pg/L至3.04×10-2pg/L范围内均可获得明显扩增(图 2A),起峰时间随模板浓度降低而增加(图 2A),范围约在17—33min之间(图 2A,2B),呈典型的线性相关性(图 2B)。各反应均在约50min时,相对荧光强度达到平台期(图 2A)。当以较高浓度(3.04×102pg/L)的基因组DNA为模板时,LAMP扩增30min即可通过琼脂糖凝胶电泳的方式检测到明显的梯形条带,反应至40min及以后产物浓度不再明显增加(图 2C)。当以较低浓度的基因组DNA(3.04×10-2pg/L)为模板时,发现反应时间达到40min才能观察到明显的梯形条带,至50min及以后产物浓度不再显著增加(图 2D),与扩增曲线的结果(图 2A)相一致。因此,确定LAMP的最适反应时间为50min。
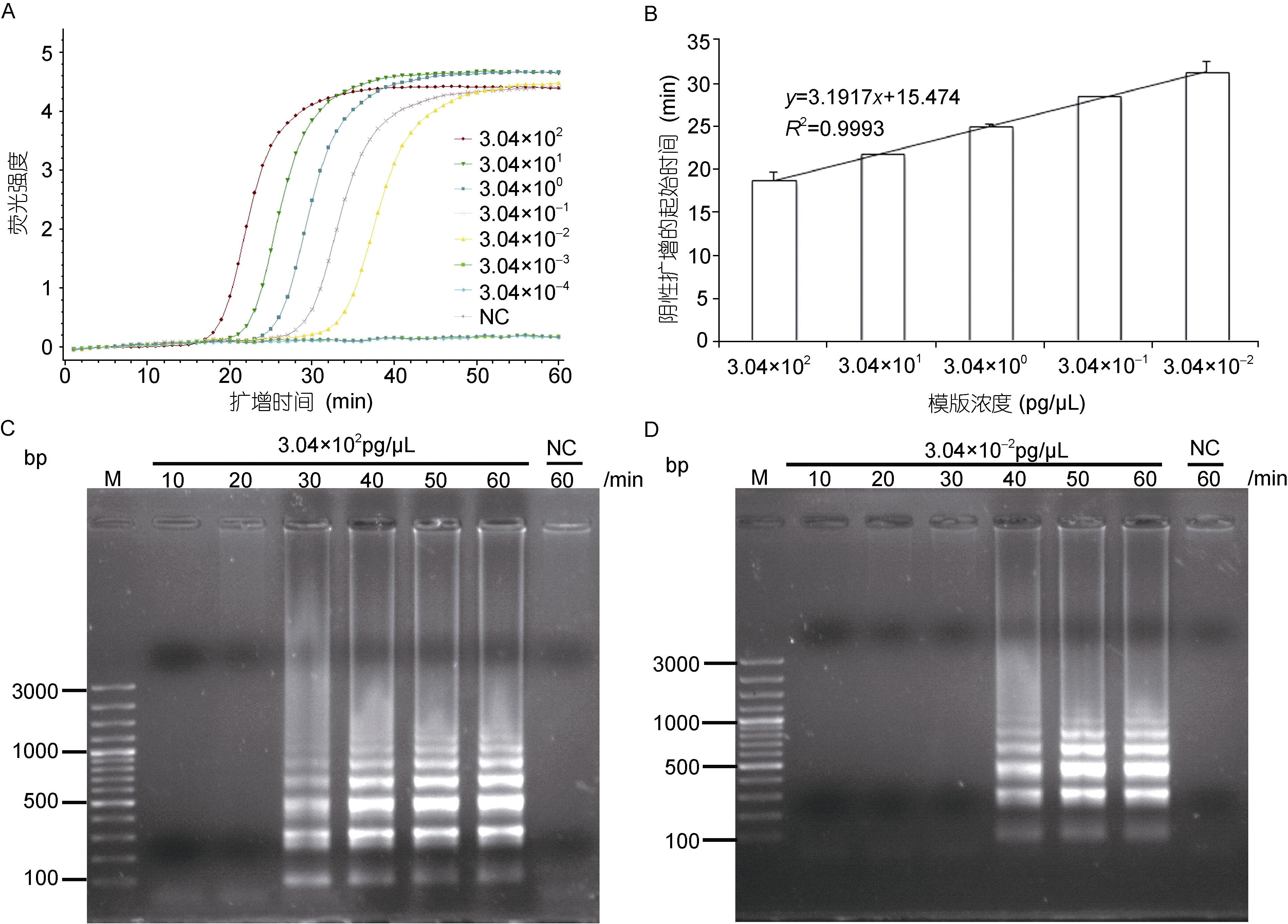 |
| 图 2 LAMP检测孔石莼最适反应时间的确定 Fig. 2 Time optimization of LAMP for detection of U. pertusa |
除孔石莼外,选择其它9株国内常见的石莼属绿藻或微藻的基因组DNA(1.00×102pg/L)为模板,进行有生物素标记的实时荧光LAMP反应,条件为63°C反应50min。结果表明,以孔石莼的基因组DNA为模板的LAMP反应,15min左右即出现明显“S”形荧光曲线(图 3A),利用琼脂糖凝胶电泳检测也出现明显的梯形条带(图 3B),LFD的检测线位置也出现明显的条带(图 3C); 而以其它9种藻类基因组DNA为模板时,实时荧光曲线呈光滑曲线,未见明显扩增(图 3A),琼脂糖凝胶电泳的结果未呈现梯形条带(图 3B),LFD的检测线位置也未能检测到明显条带(图 3C),检测结果为阴性。
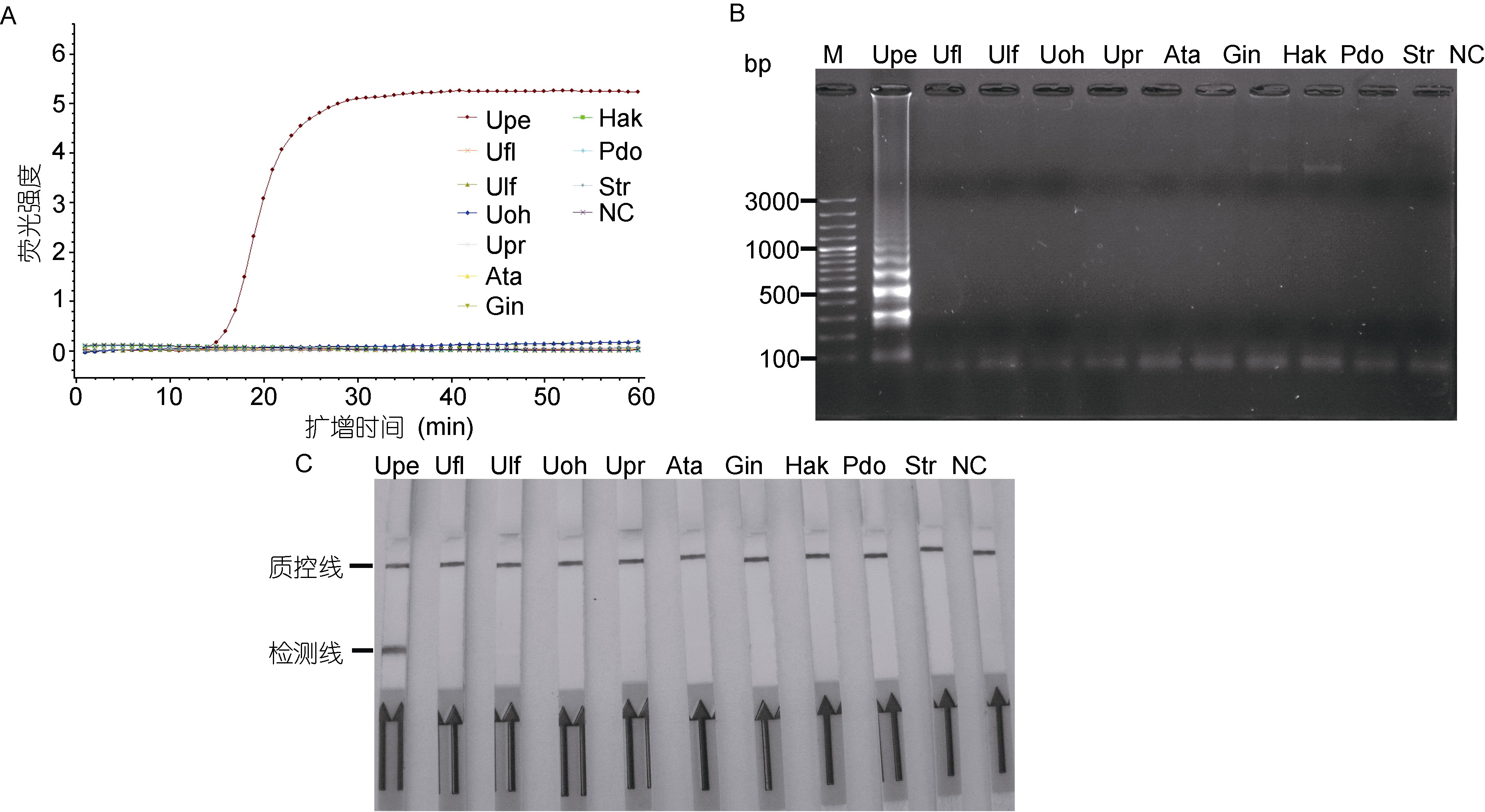 |
| 图 3 LAMP(A、B)和LAMP-LFD(C)的特异性实验结果 Fig. 3 Specificity test of LAMP(A,B)and LAMP-LFD(C)for detection of U. pertusa |
结果表明,当孔石莼的基因组DNA模板浓度稀释至3.04×10-2 pg/L时,利用琼脂糖凝胶电泳仍可清晰地检测到梯形条带(图 4A),LFD检测线位置也呈现明显的条带(图 4B); 当模板浓度进一步稀释至3.04×10-3pg/L时,琼脂糖凝胶电泳未能检测到梯形条带(图 4A),LFD检测线位置也未见条带(图 4B),结果阴性,该结果与实时荧光LAMP的检测结果一致(图 2A); 以外引物UpeITS-F3和UpeITS-B3为特异性引物的PCR反应,检测灵敏度为3.04×101pg/L(图 4C)。上述结果表明LAMP-LFD的灵敏度可达3.04×10-2 pg/L,是常规PCR方法灵敏度的1000倍。
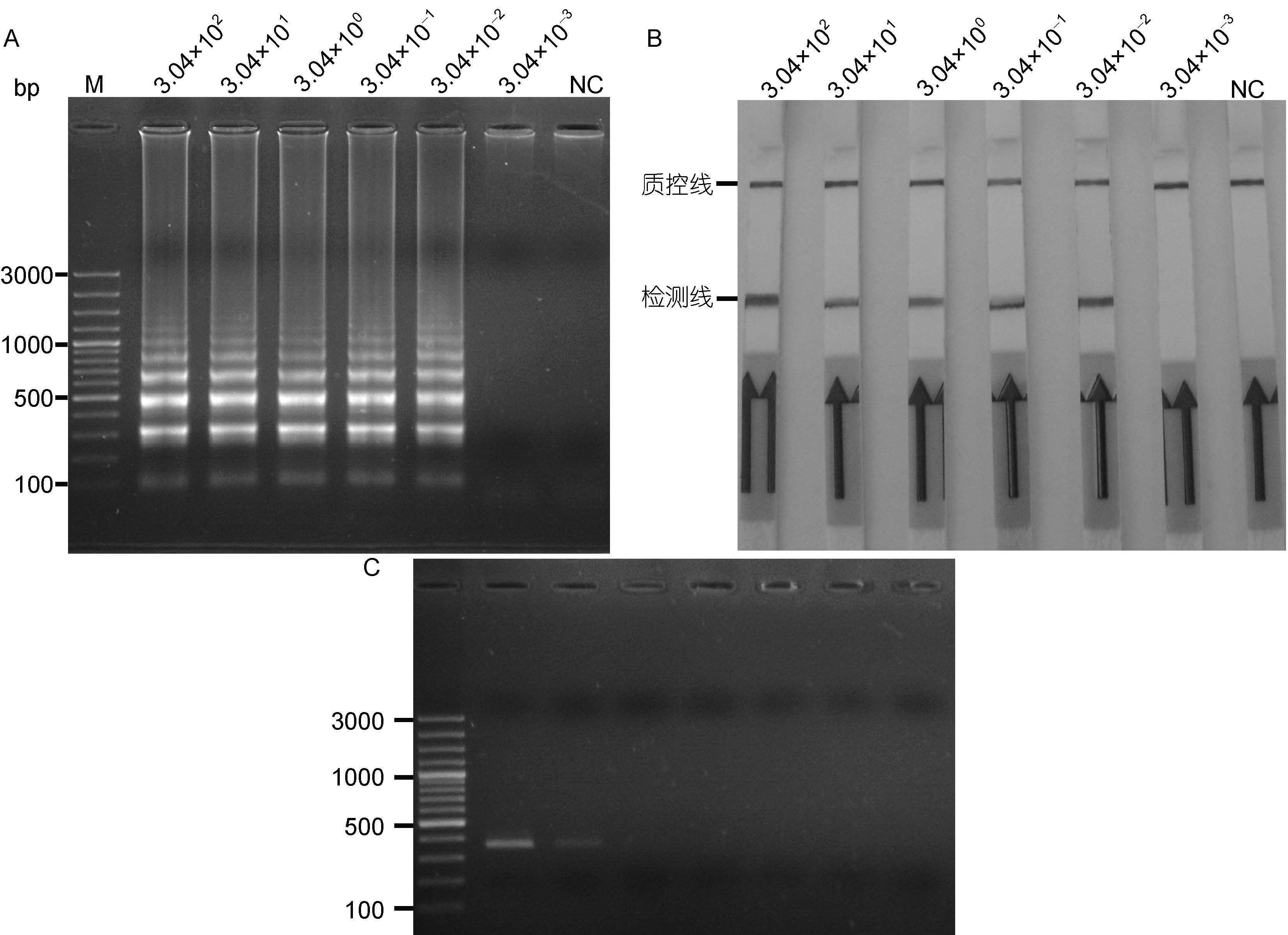 |
| 图 4 LAMP(A)、LAMP-LFD(B)和PCR(C)检测孔石莼的灵敏度比较 Fig. 4 Comparison in detection limit to U. pertusa by LAMP(A),LAMP-LFD(B),and PCR(C) |
结果表明,以灵敏度浓度(约3.04×10-2pg/L)的孔石莼基因组DNA为模板,在63°C反应50min条件下进行的LAMP反应,产物利用LFD试纸条和琼脂糖凝胶电泳两种方式检测,均能稳定地检测到目的产物的有效扩增(图 5); 以无菌去离子水为模板时,结果皆为阴性(图 5); 这说明孔石莼的LAMP-LFD方法具有良好的技术重复性。
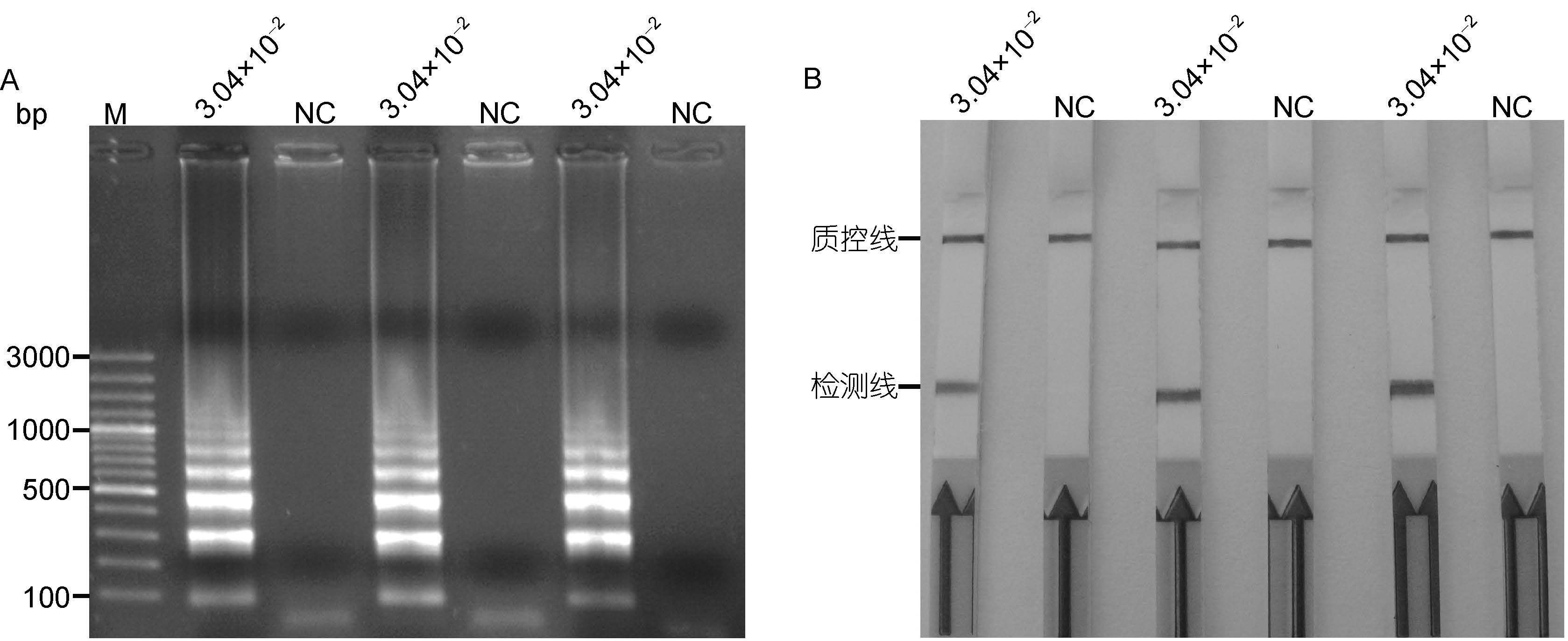 |
| 图 5 LAMP(A)和LAMP-LFD(B)的重复性实验 Fig. 5 Reproducibility of LAMP-LFD(A)and LAMP(B)for detection of U. pertusa |
从青岛和福建等地区采集17份海水样品,对LAMP-LFD方法应用于野外样本检测的适用性进行分析。对收集样本利用传统的显微观察方法鉴定发现来自青岛的ZQ-5、TD-4、S73、S77以及S78等5个样品为孔石莼(表 3); LAMP-LFD的检测结果表明青岛的ZQ-4、ZQ-5、TD-2、TD-4、S73、S77以及S78为孔石莼(表 3); 利用PCR的方法同样获得青岛的ZQ-4、ZQ-5、TD-2、TD-4、S73、S77以及S78为孔石莼(表 3),其余样品呈阴性。
| 样品编号 | 来源地 | 检测方法 | ||
| 显微观察 | LAMP-LFD | PCR | ||
| ZQ-1 | 青岛 | — | — | — |
| ZQ-2 | 青岛 | — | — | — |
| ZQ-3 | 青岛 | — | — | — |
| ZQ-4 | 青岛 | — | + | + |
| ZQ-4 | 青岛 | + | + | + |
| TD1 | — | — | — | |
| TD2 | 青岛 | — | + | + |
| TD3 | — | — | — | |
| TD4 | 青岛 | + | + | + |
| S69 | 青岛 | — | — | — |
| S70 | 青岛 | — | — | — |
| S73 | 青岛 | + | + | + |
| S77 | 青岛 | + | + | + |
| S78 | 青岛 | + | + | + |
| FJ-10 | 福建 | — | — | — |
| FJ-11 | 福建 | — | — | — |
| FJ-12 | 福建 | — | — | — |
富营养化海区暴发的绿潮灾害已逐渐成为我国某些海域的常态化现象,严重影响到当地的海水养殖和旅游业(Hiraoka et al,2004; Sun et al,2008)。研究证实,原本多数具有较好的食用和药用价值的石莼属藻类如浒苔和孔石莼等,如今已成为导致绿潮灾害频发的主要因素(Wang et al,2015; Zhou et al,2015)。传统的基于藻类形态学特征的显微镜观察方法,需经过海水样本的过筛、培养等步骤培养出藻体,除对培养设施和操作人员有较高要求外,整个培养周期过长(30d左右),不能及时有效的反映藻类的分布情况。因此,本研究以孔石莼的作为检测靶标,建立了快速、准确的LAMP-LFD方法,为孔石莼的快速筛选和鉴定提供了一种可靠方法。
耗时短是目前核酸检测类方法追求的重要目标。LAMP的扩增反应通常可在1h内完成,尤其添加环引物,可明显缩短LAMP的反应时间,这也是LAMP方法相较于基于PCR原理的分子检测技术的重要优点。本研究中,在添加环引物的前提下,当DNA模板浓度较高,LAMP反应在20min之内即可出现阳性扩增(图 2A); 通过琼脂糖凝胶电泳检测发现,30min时即可观察到明显的梯形条带(图 2C); 当模板浓度降低至最低检测浓度时,利用琼脂糖凝胶电泳的方法在40min也可以清晰地检测到梯形条带,反应至50min左右时,产物量即可达到最大值(图 2D)。当使用LFD检测时,LAMP产物只需经过与特异性探针杂交5min,再将LFD试纸条浸没入添加有杂交产物的反应液3—5min,即可完成结果判读。因此,考虑到从LAMP反应开始计算,整个检测时程在1h左右。除此之外,LAMP反应的体系组成也是建立LAMP方法过程中重点考虑的因素。本研究使用的体系组成主要参考自Ding等(2010)的方法; 在此基础上,通过添加荧光染料SYTO 9,利用荧光曲线得出LAMP扩增的起峰时间与DNA的浓度成线性关系,模板浓度约低,LAMP反应的起峰时间越延迟(图 2B)。
由于近年来石莼属绿藻引发的绿潮灾害逐渐引起人们的重视,多种基于核酸的分子检测技术在绿潮藻类的检测与鉴定中取得了应用。其中,基于PCR原理的核酸扩增技术占多数。Liu等(2012)借助于核酸测序技术,对2007—2011年间江苏省近海的绿潮藻类情况进行了调查,发现浒苔(U. prolifera)能够抵抗该海域强烈的环境变化,是该海域的主要优势藻类。Duan等(2012)借助于内转录间隔区(ITS)、二磷酸核酮糖羧化酶大亚基基因(rbcL)以及5S rDNA等藻类常用的分子标志,对采集于黄海的部分样本的藻类信息进行了分析,发现该海域中存在有扁浒苔、孔石莼、缘管浒苔,以及浒苔等多种绿潮藻类。Xiao等(2013)基于ITS序列建立了PCR-RFLP技术,应用于黄海海域石莼属和盘苔属藻类的鉴别。Zhao等(2015)利用建立的PCR-ISSR技术(PCR-inter-simple sequence repeat)对黄海海域的漂浮浒苔分析发现,浒苔的一个独特的生态型是引起全球性绿潮的重要原因,固着生长的浒苔种类不是该生态型的物种来源,这两种不同生活方式的浒苔种之间很难发生基因漂流现象。Zhang等(2015)建立了荧光原位杂交技术(FISH),该技术无需依赖于PCR的核酸扩增,借助于针对浒苔5S rDNA序列的特异性探针即可将浒苔与缘管浒苔、曲浒苔、扁浒苔、孔石莼,以及盘苔属藻类区分开来。检测灵敏度是核酸检测类技术的重要指标。段维军等(2012)针对扁浒苔ITS序列建立的PCR方法最低可检测到10 pg的藻类基因组DNA; 本实验室先前也针对扁浒苔的rDNA-ITS序列建立了LAMP-LFD技术,最低可检测到0.1 pg的扁浒苔基因组DNA(陈先锋等,2015)。Chen等(2016)基于多重PCR的原理建立的基因芯片技术能够特异性检测浒苔、扁浒苔等多种藻类,最低检测灵敏度可达0.5 ng的基因组DNA。本研究建立的LAMP-LFD技术,能够将孔石莼与浒苔、曲浒苔和缘管浒苔等加以区分,最低可检测到3.04× 10-2 pg/L 的孔石莼基因组DNA,是以UpeITS-F3和UpeITS-B3为特异性引物的PCR方法的1000倍。17个海水样本的检测结果表明,LAMP-LFD方法检测孔石莼与传统的形态学观察的结果基本一致。因此,该方法作为一种快检技术,可作为孔石莼现场检测的重要工具并加以推广。
4 结论本研究以孔石莼的rDNA-ITS序列为检测靶标建立了LAMP-LFD方法能够特异性地应用于孔石莼的检测。该方法检测时间短,从核酸扩增到LFD结果展示,整个检测过程仅需60min。该方法具有良好的检测灵敏度,最低可检测到3.04×10-2pg/L的孔石莼基因组DNA。由于该技术从核酸扩增开始的整个检测过程能够彻底摆脱对于仪器设备的依赖性,容易操作上手,作为一种新型技术,有望成为我国东部沿海孔石莼快速检测的有效手段之一。
 2016, Vol. 47
2016, Vol. 47


