
中国海洋湖沼学会主办。
文章信息
- 夏骏, 汪岷, 宫政, 姜勇, 马玉, 汪俭, 邵红兵, 王多兵, 许志梦. 2016.
- XIA Jun, WANG Min, GONG Zheng, JIANG Yong, MA Yu, WANG Jian, SHAO Hong-Bing, WANG Duo-Bing, XU Zhi-Meng. 2016.
- 以宏基因组技术探讨渤海秋冬季节病毒多样性
- METAGENOMIC STUDY ON VIRAL DIVERSITY IN AUTUMN AND WINTER IN BOHAI SEA
- 海洋与湖沼, 47(3): 572-580
- Oceanologia et Limnologia Sinica, 47(3): 572-580.
- http://dx.doi.org/10.11693/hyhz20151100293
-
文章历史
- 收稿日期:2015-11-30
- 改回日期:2016-03-25
海洋环境中的病毒数量极为丰富,总共有将近4×1030个,平均丰度约3×109ind./L(Suttle,2005)。海洋病毒以微食物环、侵染微生物等形式影响海洋生物群落组成和碳氮循环(Wilhelm et al,1999),浮游病毒优先杀死占优势宿主的特性能使海域富营养区的赤潮快速消散(Brussaard et al,2005,2008)。渤海的浮游病毒数量相当丰富,2010年从春季至冬季数量变化范围是(6.40×108—3.59×1010)ind./L,并且呈现出中部海域丰度高于近岸海域的情况(王健等,2013)。本文所研究的站位B47(38.66°N,118.97°E)处于渤海远离岸边的海域,受近岸的环境因素影响较小,2011年12月以流式细胞仪测得的病毒丰度为8.97×109ind./L,因此选择这个点的数据来代表渤海浮游病毒群落的大致情况。
宏基因组学(metagenomics)作为研究环境总基因组的有效方法在1998年第一次提出(Handelsman et al,1998),可以直接对环境样品中未经培养的微生物群落复杂的总基因组进行分析,通过遗传物质的提取、测序、拼接、注释、统计分析等步骤(Thomas et al,2012),了解环境微生物群落的物种组成、功能基因结构、进化地位、种间关系以及与环境因子的相关性(Handelsman,2004)。
近十多年来,有多个探索海洋病毒宏基因组的研究成果被发表。有针对海水中病毒组的相关研究(Angly et al,2006; Steward et al,2011; Williamson et al,2012; Hurwitz et al,2013; Winter et al,2014; Brum et al,2015),从病毒物种组成和多样性、各大洋病毒宏基因组之间的关系、病毒种之间的进化关系、病毒群落结构与环境因子之间以及地域、水深、离岸距离的相关性等方面全面阐述了大洋海水中的病毒宏基因组特征; 也有针对海洋沉积物病毒组的相关研究(Breitbart et al,2004; Yoshida et al,2013),对近岸海域以及深海的沉积物病毒组作出具体分析。
目前还没有针对渤海这一海域的病毒宏基因组的相关报道。本文所涉及的综合分析内容可为国内海域真光层病毒群落、微生物、海洋生态系统等各类研究提供相关数据支持。
1 材料与方法 1.1 采样和病毒宏基因组样品的预处理于2010年9月和2011年12月东方红2号国家自然科学基金开放航次中,用潜水泵在渤海B47站(38.66°N,118.97°E)(图 1)采集表层海水样各150L。海水润洗塑料桶三次后泵入海水。先使用直径300mm,孔径3μm的混合纤维素膜进行第一次过滤,再使用直径300mm、孔径0.22μm的混合纤维素膜进行第二次过滤,去除非病毒生物颗粒。滤过液经中型切相流系统(膜包: Pellicon 2 Cassette,材质: 聚醚砜,孔径: 50kDa)浓缩至0.5L,再用小型切相流系统(膜包: Pellicon XL Cassette,材质: 聚醚砜,孔径: 50kDa)浓缩至0.01L。将高度浓缩后的样品置入液氮中速冻,船上-20°C储存,实验室提取DNA前-80°C储存。
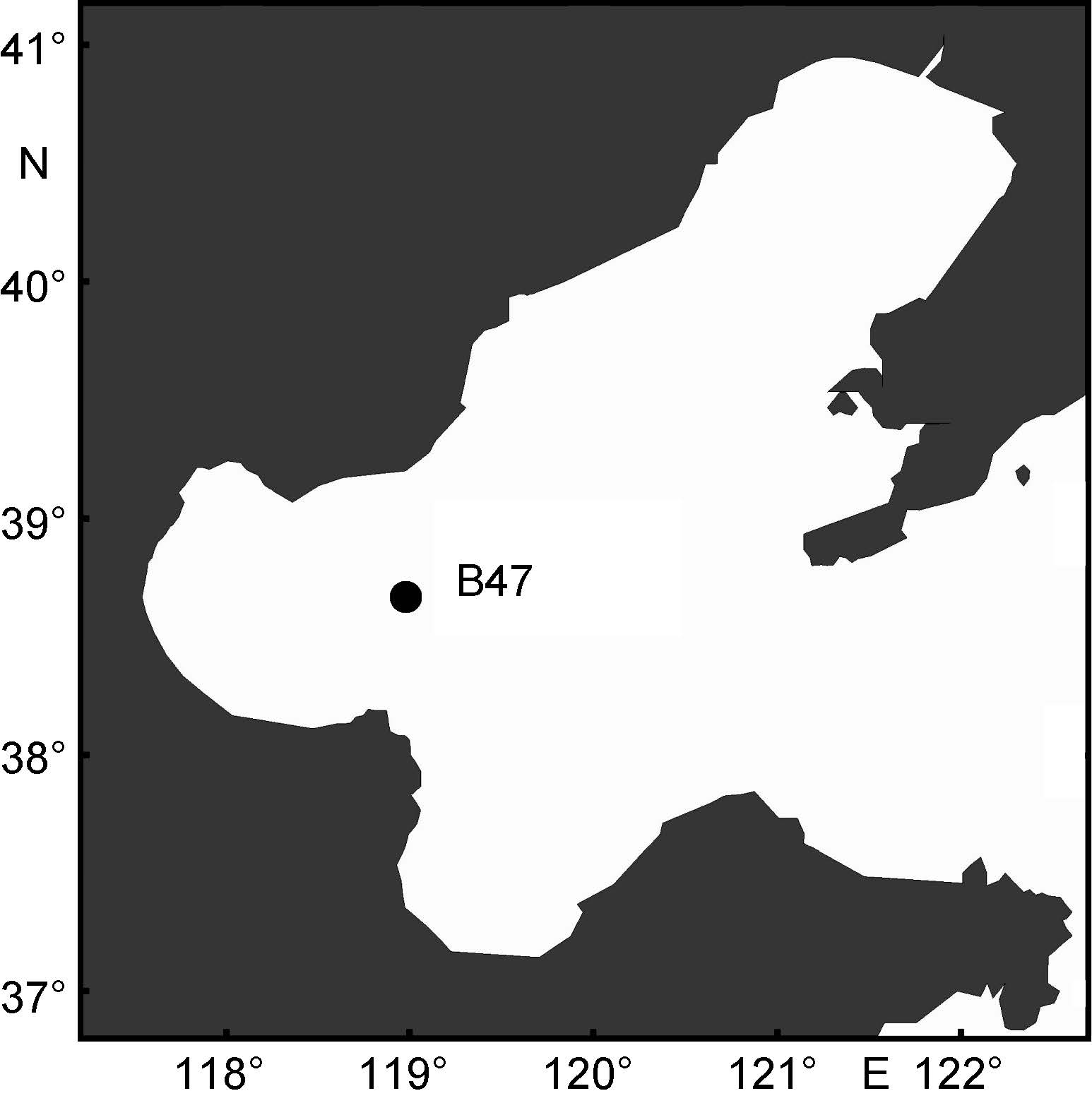 |
| 图 1 渤海病毒宏基因组采样站位图 Fig. 1 Sites map for sampling viral metagenome in Bohai Sea |
样品37°C水浴解冻,加入聚乙二醇(终浓度10%)和NaCl(终浓度0.6%),4°C避光静置24h。在4°C下以13500r/min离心40min,去除上清液,让沉淀悬浮于300μL 钠-镁离子缓冲液。加入100μL KCl,冰浴30min。4°C 12000r/min离心10min,去除沉淀。加入10μL蛋白酶K,混匀后再加入20μL 10%十二烷基硫酸钠溶液,56°C金属浴1h(每10min震荡摇匀一次)。连续两次用215μL的平衡酚和215μL的氯仿-异戊醇萃取,取上清液20°C 12000r/min离心4min,去除有机相。加入43μL醋酸钠,860μL无水乙醇,-20°C静置3h。4°C 12000r/min离心15min,去除上清液,加入0.001L 70%乙醇,使沉淀悬浮,4°C 12000r/min离心5min。去除上清液,加入0.001L无水乙醇,吹打悬浮,4°C 12000r/min离心5min。吹干,使沉淀溶于30μL Tris-EDTA溶液,4°C静置30min,-80°C保存。
1.3 建库、测序和拼接利用Hiseq 2000(Illumina)为样品进行高通量测序,2×100bp双末端测序,插入片段长度为170bp,测序公司为深圳华大基因科技有限公司(BGI)。首先对样品进行检测,然后根据Illumina标准建库测序流程进行测序:(1)电泳回收主要的DNA片段;(2)用T4 DNA Polymerase、Klenow DNA Polymerase和T4多核苷酸激酶将打断形成的黏性末端修复成平末端;(3)通过3端加碱基“A”,使得DNA片段能与3端带有“T”碱基的特殊接头连接;(4)用合格的文库进行cluster制备和测序。对得到的所有reads序列进行进一步处理:(1)去除含N碱基数目总和达到3个或以上的reads;(2)去除与reads序列有15bp的重叠区的接头序列污染;(3)去除质量值连续(默认reads中质量值小于Q20的碱基数目大于36,设置为84、36);(4)去除重复污染;(5)对数据进行去宿主污染的分析,一致性大于等于90%的条件下,与宿主比对上的被认为是宿主污染r eads。最终得到干净数据。用SOAP denovo(1.06版)软件对reads进行拼接,设置多个K值选择质量最高的结果,2010年9月样品选择K值为59,2011年12月样品选择K值为47,参数设置为“SOAP denovo-63mer all-K*-p8-F-M2-d1-R-u-k-o”。拼接完成后过滤掉长度小于500bp的scaffold。
1.4 病毒组物种组成分析上传拼接好的scaffold序列,利用MetaVir网站平台(http://metavir-meb.univ-bpclermont.fr/)在病毒基因组参考序列蛋白库(Refseq complete viral genomes protein sequences database)中进行Blastp比对(e-value<10-3)。用GAAS(Genome relative Abundance and Average Size)软件进行病毒物种分类统计(Roux et al,2014)。
1.5 功能基因分析使用MetaGeneMark(2.10版)软件预测scaffold中的开放阅读框(ORF)(Zhu et al,2010),再用CD-HIT(4.6版)软件去除冗余的ORF。将非冗余的ORF利用Blastp(e-value<10-3)在WebMGA网站上进行COG功能基因数据库比对(Wu et al,2011)。
1.6 系统进化分析两个样品中的病毒超过97%属于双链DNA病毒,所以选择以双链DNA病毒基因的保守结构域(conserved domain)AVS、G20、GP23、MCP、PhoH、Polβ、Polβ2、T7gp17、TerL作为待建树的标记基因,用Blastp(e-value<10-3)在ORF中查找。其中属于Polβ和TerL保守结构域中的各一条基因在样品的ORF和GenBank NR库均有最好的比对结果(e-value<10-10),被 挑选出来进行系统进化分析,分别是DNA聚合酶Polβ中的CyPHP-SSM4(pfam编号: PF00136)和DNA末端酶大亚基TerL中的Q58LJ7_BPPRS(pfam编号: 03237)。在用CLASTALW进行两两比对和多序列比对后,用MEGA(6.06版)软件建立邻接树(Hall,2013)。
2 结果 2.1 病毒物种分类2010年9月和2011年12月的样品中各拼接得到19875条、31319条scaffold,总长度分别为28.29Mbp、33.46Mbp,拼接序列其它数据见表 1。两个样品预测到非冗余ORF 48227个(2010年9月)、63660个(2011年12月)(表 1)。
| 样品 | 序列数 | 总长 (Mbp) | N50 (bp) | N90 (bp) | 最长 (bp) | 最短 (bp) | 序列定位 | 开放阅读框数量 | 开放阅读框平均长度 | ||
| 双末端拼接 | 单末端拼接 | % | |||||||||
| B47_Sep.2010 | 19875 | 28.29 | 1820 | 614 | 73314 | 500 | 4407626 | 1227695 | 37.74 | 48227 | 529 |
| B47_Dec.2011 | 31319 | 33.46 | 1115 | 568 | 38650 | 500 | 1926379 | 756354 | 30.73 | 63660 | 484 |
在NR库(non-redundant protein database,非冗余蛋白库)中比对上(blastx,e-value<10-3)病毒序列的分别有9716条(48.89%)和13599条(43.42%)。两个样品共比对上1240个病毒种。与太平洋深海沉积物中大多数序列属于单链DNA病毒不同(Mitsuhiro et al,2013),在两个样品比对上病毒基因组的序列中,双链DNA病毒占据主要地位,2010年9月的样品中占97.65%,2011年12月的样品中占97.51%。以病毒目来划分,有尾噬菌体目占据主导地位(81.19%、80.63%)。以病毒科来划分,除了有尾噬菌体目下的肌尾病毒科(2010年9月为27.26%,2011年12月为21.08%1)、长尾病毒科(27.83%和25.34%)和短尾病毒科(23.64%和30.90%)外,藻类DNA病毒科(3.04%和3.28%)和米米病毒科(1.38%和0.83%)也占据一定的比例,这五个科的病毒序列数量占到总病毒序列数的83.15%(2010年9月)、81.43%(2011年12月)(图 2)。
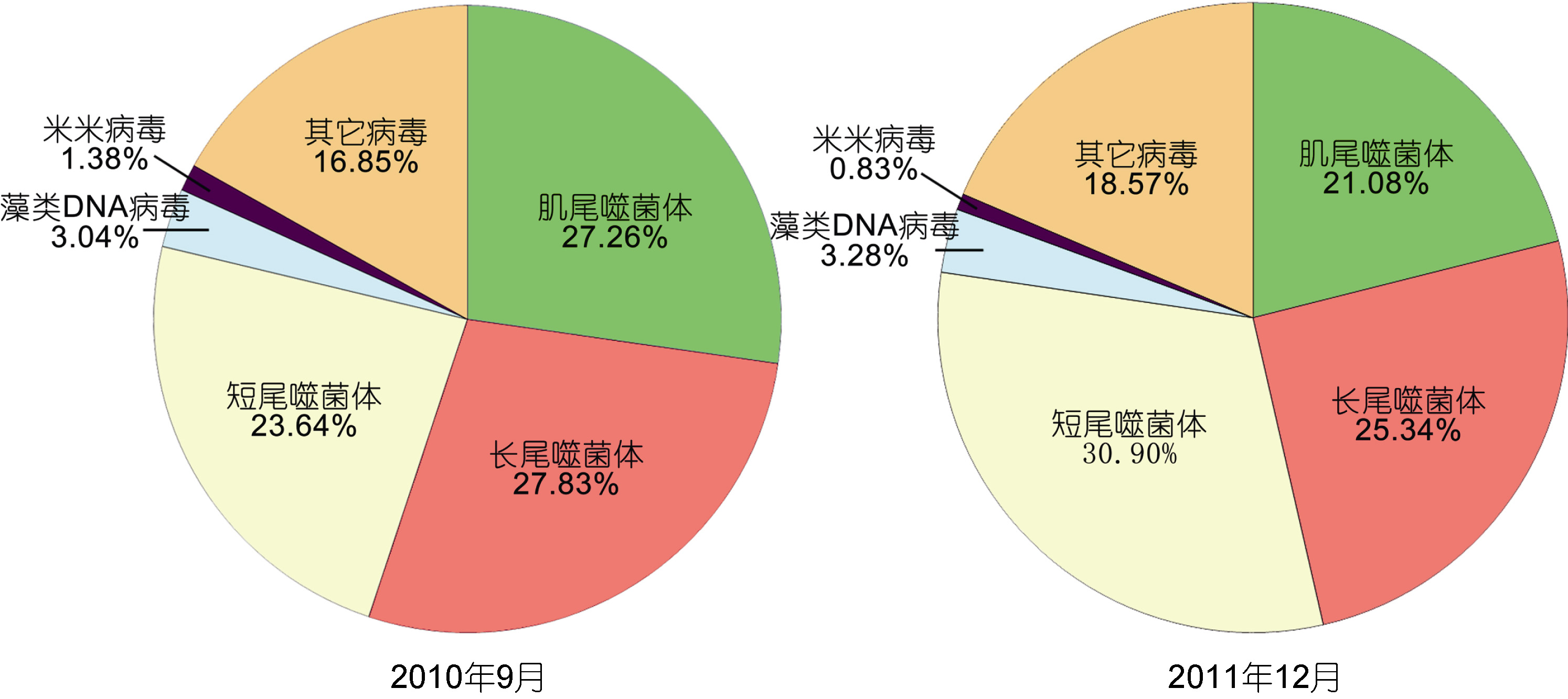 |
| 图 2 病毒物种科级分类 Fig. 2 Viral taxonomy in family |
两个样品中总序列数排在前十的病毒种(图 3)为Puniceispirillum phage HMO-2011(5.26%和6.97%)、Pelagibacter phageHTVC008M(3.58%和2.77%)、Pelagibacter phage HTVC010P(2.33%和3.01%)、cyanophage KBS-S-2A(1.82%和1.15%)、Cellulophaga phage phi38:1(0.54%和1.97%)、Prochlorococcus phage P-SSM2(1.17%和1.52%)、Synechococcus phage S-SM2(1.86%和0.51%)、Pelagibacter phage HTVC011P(0.60%和1.38%)、Pelagibacter phage HTVC019P(0.70%和1.17%)、Idiomarinaceae phage 1N2-2(0.98%和0.93%)。这十个种的病毒序列占到总序列数的20.32%。
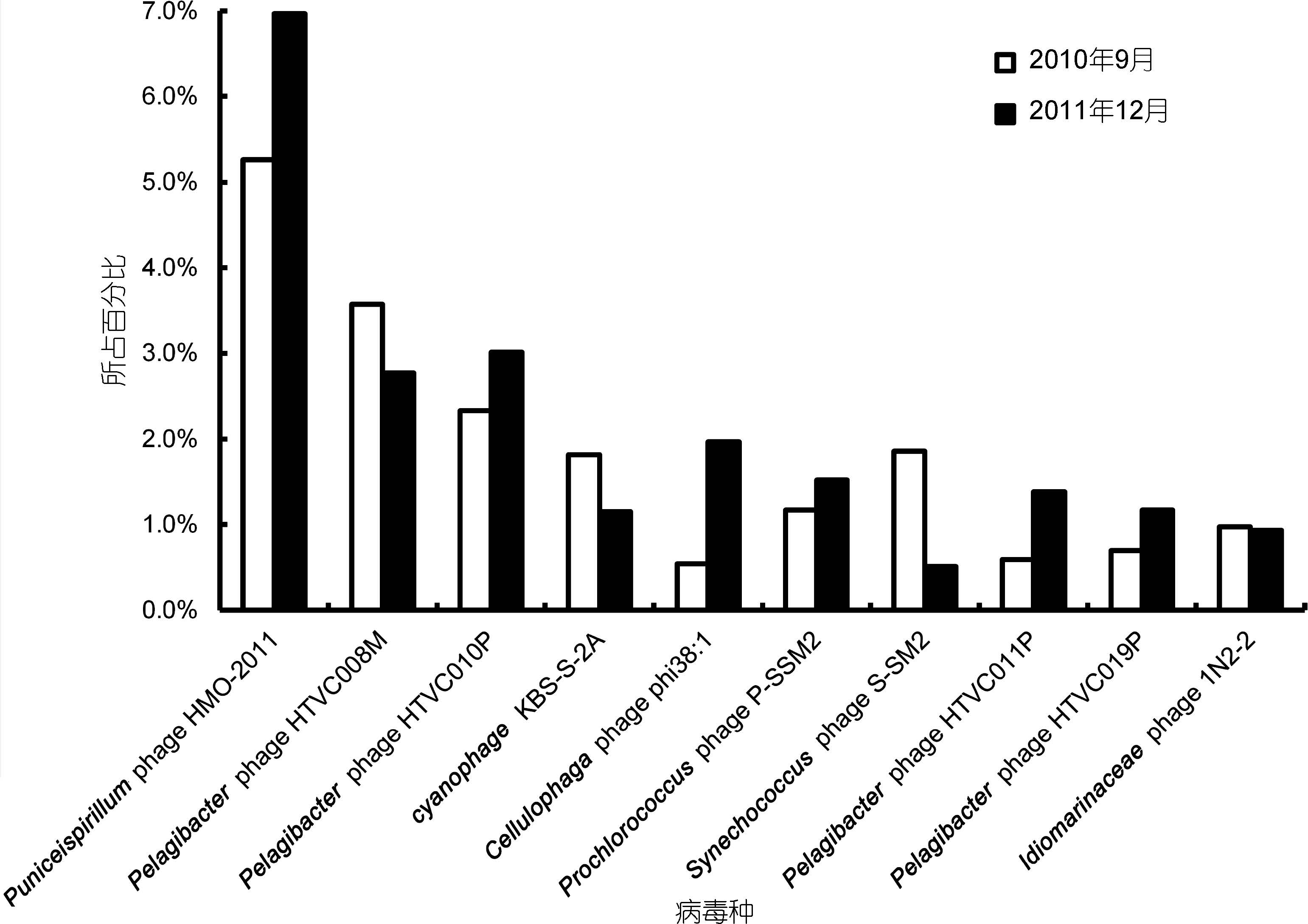 |
| 图 3 序列数量丰富度前十的病毒种 Fig. 3 The top 10 virus species in sequence abundance |
序列按照主要宿主来划分病毒大类(图 4),Synechococcus phage(共33个种,10.28%)、Pelagibacter phage(共4个种,7.86%)和Puniceispirillum phage(共1个种,6.26%)为数量最多的三大类,以这12类微生物为宿主的病毒序列占到两个样品总序列数的52.01%。而其中在两个样品中差距最大的为Synechococcus phage,2010年9月样品中所占比例为12.75%,而2011年12月的为8.52%。原绿球藻噬藻体(Prochlorococcus phage)所占比例为3.09%至3.73%。
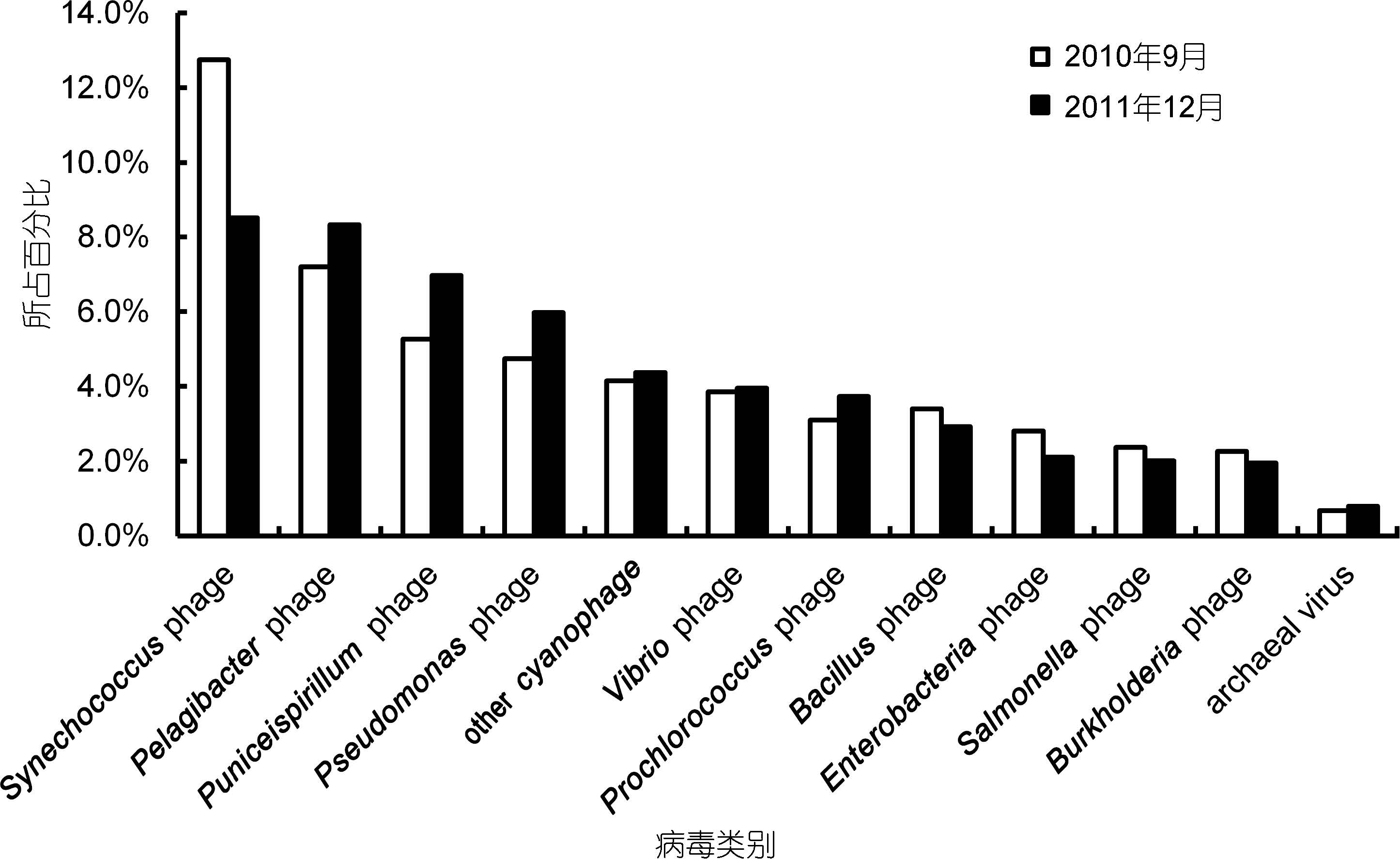 |
| 图 4 以主要宿主分类的前12大类病毒 Fig. 4 The top 12 major viral groups classified in by hosts |
另外,对于存在于渤海病毒组中不占优势的病毒大类,共有27条序列比对上单链DNA病毒的8个种,5条比对上单链RNA病毒5个种。
物种多样性指数采用Shannon-Wiener指数H′,以序列的个数作为病毒个体数进行计算,2010年9月病毒组为5.87nats,2011年12月的为5.83nats。Pielou’s均匀度J′冬季为0.612,秋季为0.639。
2.2 病毒功能基因多样性将所有预测到的ORF在COG(Clusters of Orthologous Groups of proteins,直系同源蛋白簇)数据库中进行比对(blastp,e-value<10-3)。共有20.85%(2010年9月)和9.39%(2011年12月)的ORF在COG数据库中比对上。在具有特定功能的基因中,以复制、结合和修复蛋白最为丰富,其次为细胞壁/细胞膜/包膜合成蛋白,以及转录蛋白等(表 2)。渤海冬季病毒群落复制、结合和修复蛋白所占比例远高于秋季,在北黄海的9个病毒组中呈现相同的现象(汪俭,2015)。
| 类别 | 描述 | 2010年9月 | 2011年12月 |
| L | 复制、结合和修复蛋白 | 16.39% | 33.77% |
| M | 细胞壁/细胞膜/包膜合成蛋白 | 6.19% | 7.46% |
| K | 转录蛋白 | 5.23% | 4.01% |
| O | 翻译后修饰、蛋白转换和分子伴侣蛋白 | 4.02% | 4.92% |
| F | 核酸转运和代谢蛋白 | 3.26% | 3.65% |
| E | 氨基酸转运和代谢蛋白 | 4.64% | 0.84% |
| C | 能量产生和转化蛋白 | 4.09% | 0.50% |
| G | 碳水化合物转运和代谢蛋白 | 3.24% | 0.47% |
| J | 翻译、核糖体结构和合成蛋白 | 2.85% | 1.07% |
| P | 无机盐转运和代谢蛋白 | 2.88% | 0.30% |
| 注: 只列出有特定功能的、比对上的ORF总数排在前十位的基因 | |||
将全部ORF与各标记基因比对(blastp,e-value< 10-3),有多个ORF被比对上,结果见表 3,其中以DNA末端酶大亚基TerL最多,其次为T7噬菌体尾丝蛋白gp17和DNA聚合酶Polβ。选取比对结果最好的两个标记基因Polβ和TerL建立N-J系统进化树(图 5,6)。结果显示,渤海病毒组的ORF在已分离的病毒基因中,亲缘关系与聚球藻噬藻体(Synechococcus phage)、原绿球藻噬藻体(Prochlorococcus phage)、根瘤菌噬菌体(Sinorhizobium phage)、远洋杆菌噬菌体(Pelagibacter phage)较近。
| 样品 | 保守结构域 | ||||||||
| AVS | G20 | GP23 | MCP | PhoH | Polβ | Polβ2 | T7gp17 | TerL | |
| 2010年9月 | 23 | 41 | 41 | 0 | 27 | 49 | 3 | 68 | 529 |
| 2011年12月 | 14 | 13 | 14 | 0 | 26 | 45 | 23 | 94 | 747 |
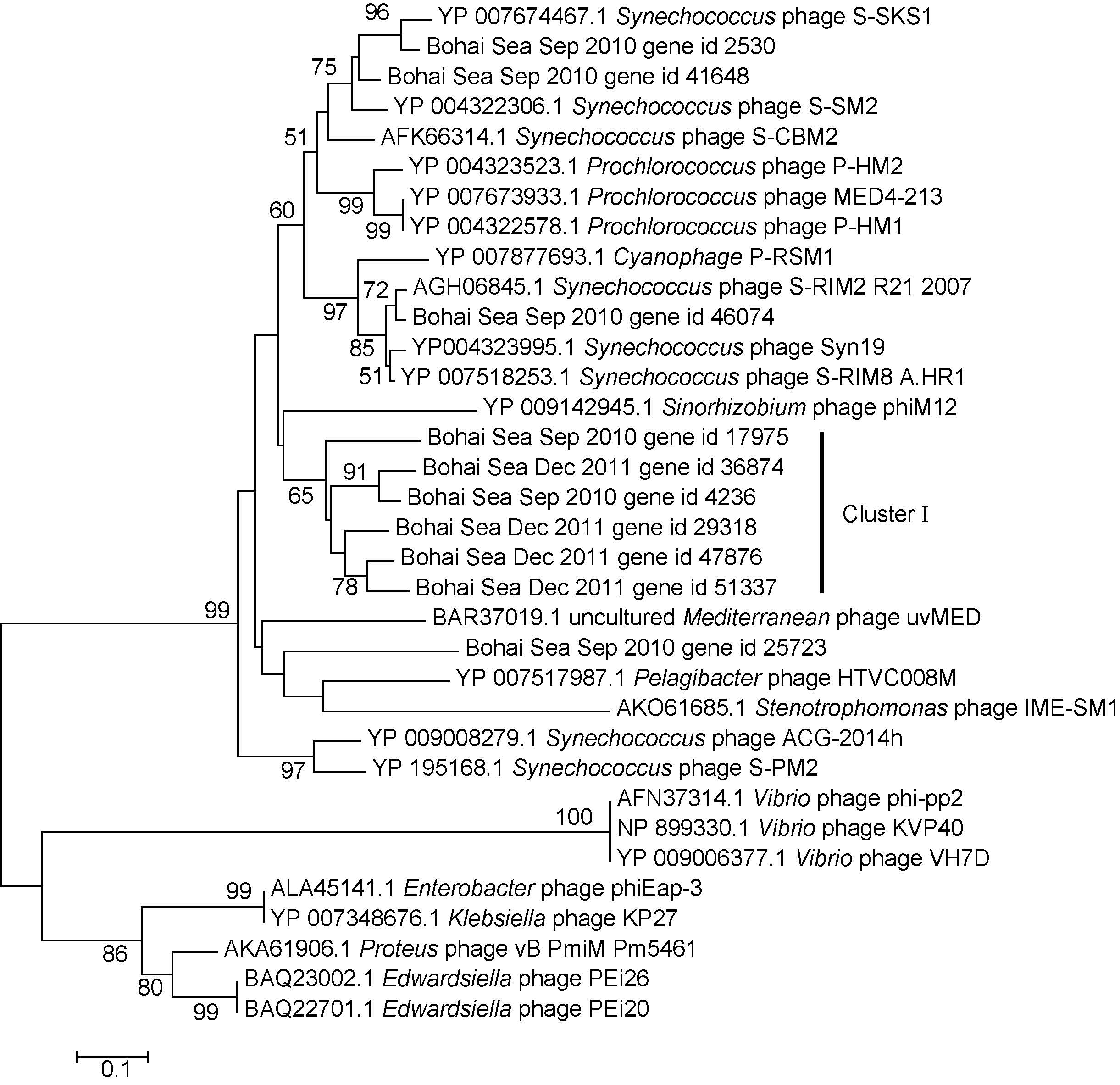 |
| 图 5 渤海病毒与已知病毒Polβ(DNA聚合酶β)保守结构域基因的N-J系统进化树 Fig. 5 Neighbor-joining tree based on conserved domain Polβ(DNA polymerase β)from known virus |
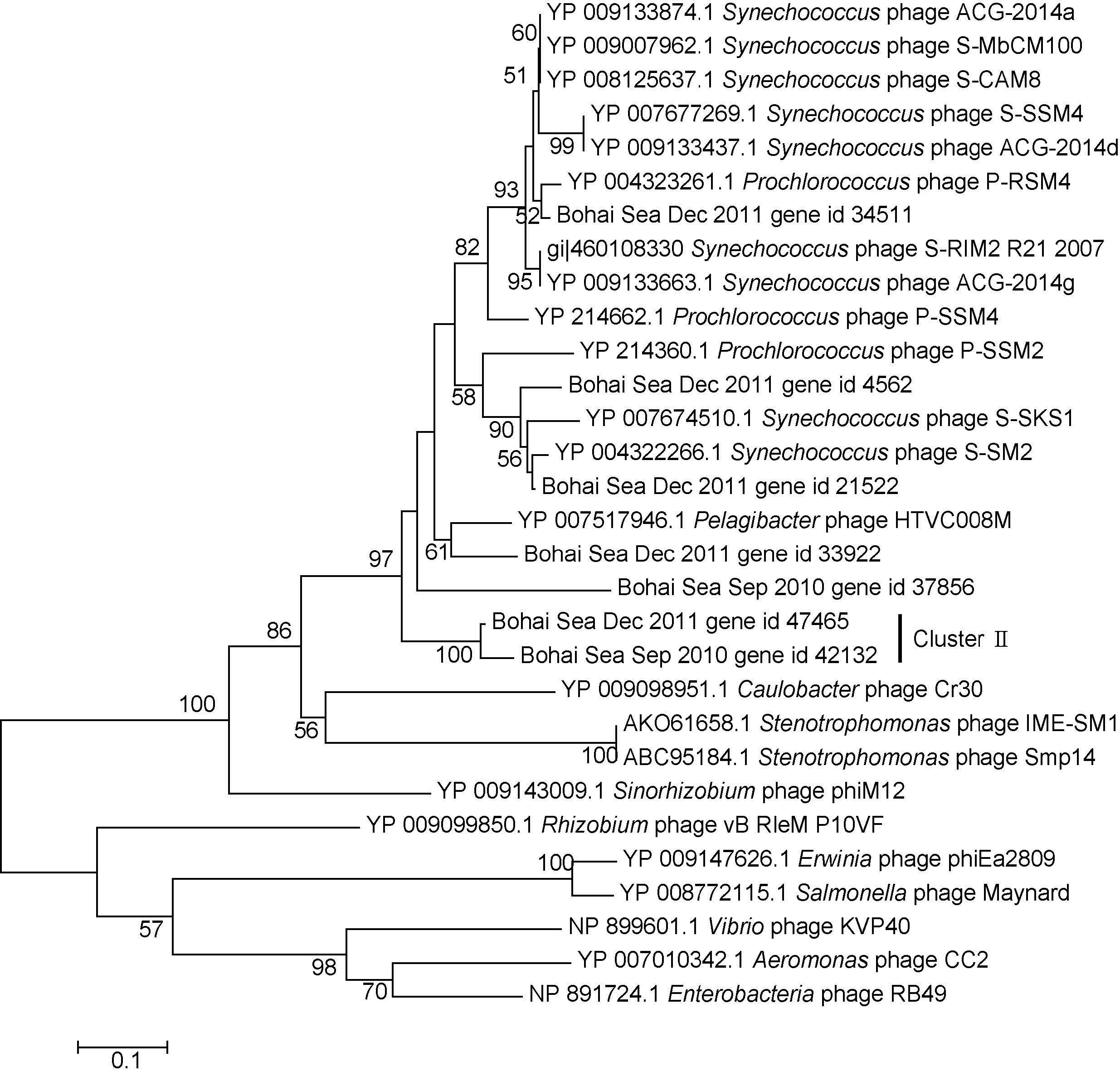 |
| 图 6 渤海病毒与已知病毒TerL(DNA末端酶大亚基)保守结构域基因的N-J系统进化树 Fig. 6 Neighbor-joining tree based on conserved domain TerL(DNA terminase large subunit)from known virus |
在以Polβ建的系统进化树中,一支Cluster I与已知的蓝藻噬藻体亲缘关系较远,与之最近的一个种为Sinorhizobium phage phiM12。以TerL建的系统进化树中,渤海病毒组的序列较为分散,其中Cluster II 一支的bootstrap值为100%,聚类极为可信,其进化地位同样在T4-like virus的HTVC008M与phiM12之间,但不与phiM12在同一分支上。
3 讨论在渤海表层海水2010年9月和2011年12月的两个病毒宏基因组中,大部分的病毒序列属于双链DNA病毒,其中肌尾病毒科、长尾病毒科、短尾病毒科、藻DNA病毒科占据主导地位。类似结果也出现在近期的印度洋和太平洋东北部真光层海水病毒组中,渤海各级分类与太平洋物种分类结果接近一致,原因可能是均没有对样品DNA进行扩增,最大程度地还原了病毒群落结构的真实情况(Steward et al,2011)。而印度洋虽然同样以双链DNA病毒—有尾噬菌体目占绝对主导地位,但其扩增后的病毒组中肌尾病毒科占据54.3%与渤海病毒组相差较大(Williamson et al,2012)。
噬藻体(cyanophage)能够侵染蓝细菌中聚球藻属和原绿球藻属两个重要的初级生产者,在自然水体中数量极为丰富(Bergh et al,1989),其对宿主的致死率在1%至8%( Garza et al,1998),是微食物环(microbial loop)中的重要成员。噬藻体序列在病毒组序列中占有相当大的比例,通过系统进化分析指明,渤海噬藻体中存在未知的物种分支Cluster I(图 5)和Cluster II(图 6),且很有可能属于肌尾噬菌体— T4-like病毒。对未知物种分支的探索,可为将来的病毒分离提供指导,并完善病毒宏基因组中的“Unknown”物种部分。
聚球藻(Synechococcus)对于海洋初级生产力的贡献为25%(Li,1994; Smith et al,2001),是海洋中重要的微生物类群。在黄海海域,由于水温降低,聚球藻冬季的数量小于秋季数量(Zhao et al,2011)。聚球藻噬藻体呈现出与宿主相同的变化趋势,2011年12月所占比例小于2010年9月。另一个重要的大类,原绿球藻噬藻体(Prochlorococcus phage)所占比例为3.09%至3.73%。但相关研究指出,原绿球藻仅分布于夏季水温较高的亚热带和热带海域(焦念志,2006),对中国海域原绿球藻生长的实验研究也表明,其最适生长水温范围在25—30°C(冯宪栋等,2007)。渤海B47站2011年夏季表层海水水温为18.0°C,且本实验室并没有以流式细胞仪测出原绿球藻的存在。可能的原因是原绿球藻噬藻体宿主范围较广,能够侵染其他属蓝细菌,或是对马暖流分支将部分聚球藻从亚热带海域携带至渤海。
在冬季和秋季渤海病毒组中,4个病毒种Puniceispirillum phage HMO-2011、Pelagibacter phage HTVC010P、Pelagibacter phage HTVC008M和Synechococcus phage S-SM2占据优势地位。海洋真光层细菌其中一个主要分支为SAR116(Candidatus Puniceispirillums类群),它对于甲基营养和光合异养代谢有着潜在影响(Grote et al,2011; Giovannoni et al,2012)。Puniceispirillum phage HMO-2011序列在印度洋的3个病毒组以及太平洋的4个病毒组中占到病毒序列总数的10.3%—25.3%,为丰富度第一或第二的病毒种(Kang et al,2013),与之前报道的太平洋病毒组(POV)中开阔海域和近岸的SAR11噬菌体Pelagibacter phage HTVC010P总数结合来看(Zhao et al,2013),大洋真光层水体中最占优势的病毒种为HMO-2011和HTVC010P。其次为Pelagibacter phage HTVC008M和Synechococcus phage S-SM2(Kang et al,2014)。因此处于太平洋西岸的渤海,样品中这四种已分离纯化病毒的基因序列比例占据相当大的优势应为正常现象。但渤海病毒组与其它海域不同的是,cyanophage KBS-S-2A(1.43%)、Cellulophaga phage phi38:1(1.38%)和Idiomarinaceae phage 1N2-2(0.95%)为序列数排在前十位的优势种。
仅有极少数的单链DNA病毒存在于渤海病毒组中,这与MetaVir病毒宏基因组数据库中大部分海域的病毒组相似,但也与部分海域差异很大,如2005年马尾藻海的ssDNA序列占到了总病毒序列的22.43%。27条单链DNA序列中最丰富的病毒科为丝杆噬菌体(Inoviridae)。有研究表明,海洋中存在大量单链DNA病毒(Labonté et al,2013),但由于其高突变率难以被分离纯化,数据库中仅有少数病毒种的全基因组序列。在病毒组研究中,将以发掘海水中未知单链DNA病毒种为未来的一个方向。
在冬季病毒种数(1093)高于秋季(1010)、病毒序列数(13599)也高于秋季(9716)的情况下,冬季病毒物种多样性低于秋季,原因是冬季优势种的优势度更大。从Pielou’s均匀度J′(冬季为0.612,秋季为0.639)可以看出,秋季各物种序列数更为平均。冬季功能基因中的复制、结合和修复基因所占比例却是秋季的两倍多,说明渤海病毒群落(特别是劣势种)在冬季水温较低的情况下代谢活性减弱,但以能够适应低温条件的微生物为宿主的病毒则更具优势。例如Puniceispirillum phage和Pelagibacter phage两个主要的大类,在冬季的比例明显高于秋季。聚球藻噬藻体在冬季数量降低,是由于宿主聚球藻的数量与水温呈显著相关,在中国海域数量有冬季<秋季的规律(赵苑,2010)。
进化关系中,与Cluster I最近的一个种为Sinorhizobium phage phiM12,宿主Sinorhizobium meliloti 1021能在土壤中与根系共生,达到固氮的作用(Stroupe ME et al,2014),属于有尾噬菌体目-肌尾噬菌体科-T4-like病毒属。T4-like噬菌体有着可收缩尾部因而宿主范围广,从肠杆菌至蓝细菌噬藻体,广泛分布于海水和淡水中(Mann et al,2005; Sullivan et al,2005; Weigele et al,2007; Dreher et al,2011)。Cluster I可能代表海洋真光层中新的一类病毒,从Polβ进化地位在同属于T4-like 病毒的Pelagibacter phage HTVC008M 与Sinorhizobium phage phiM12之间可以推断,Cluster I所代表的一类病毒很可能属于T4-like 病毒。Cluster II极为可信,进化地位在HTVC008M与phiM12之间,可能属于Cluster I以外的另一支T4-like病毒类群。
在将来的海洋病毒宏基因组研究中,采样和实验方法需要联系最新的研究进展不断改善,如最近的Fe离子沉降病毒法(John et al,2011)可使病毒宏基因组采样步骤简化。数据分析方面,除了对群落的基础分析,更应在结合环境因子和宿主序列,拓宽研究病毒大类领域,明确病毒群落的海洋生态学地位方面有所突破。
4 结论综上所述,渤海B47站的表层海水病毒宏基因组中双链DNA病毒占据主要地位,前五位的病毒科依次为肌尾病毒科、长尾病毒科、短尾病毒科、藻类DNA病毒科、米米病毒科,病毒种类与太平洋、大西洋、印度洋等远洋相比,有一定的独特性。渤海秋季病毒物种多样性和均匀性都比冬季更高,且功能基因分析结果表明秋季病毒代谢活性更强。原绿球藻一般情况下只存在于水温较高的亚热带海域,但在秋季和冬季水温较低的渤海海域,病毒组中却存在原绿球藻噬藻体,值得进一步研究探讨原因。在系统进化分析结果中发现噬藻体中可能属于未知T4-like噬菌体的病毒分支,可为噬菌体分离提供指导。
 2016, Vol. 47
2016, Vol. 47


