
中国海洋湖沼学会主办。
文章信息
- 陈琼, 李贵阳, 罗坤, 孔杰, 莫照兰, 栾生, 李杰, 曹宝祥, 张玉玲. 2017.
- CHEN Qiong, LI Gui-Yang, LUO Kun, KONG Jie, MO Zhao-Lan, LUAN Sheng, LI Jie, CAO Bao-Xiang, ZHANG Yu-Ling. 2017.
- 凡纳滨对虾(Litopenaeus vannamei)亲虾繁殖期水体微生物多样性
- MICROBIAL DIVERSITY IN BROODSTOCK WATERS OF THE TWO GENDERS OF LITOPENAEUS VANNAMEI
- 海洋与湖沼, 48(1): 130-138
- Oceanologia et Limnologia Sinica, 48(1): 130-138.
- http://dx.doi.org/10.11693/hyhz20160700176
-
文章历史
- 收稿日期:2016-08-21
- 收修改稿日期:2016-09-22
2. 中国水产科学研究院黄海水产研究所 农业部海洋渔业资源可持续利用重点开放实验室 青岛 266071;
3. 青岛海洋科学与技术国家实验室 海洋渔业科学与食物产出过程功能实验室 青岛 266071
2. The Key Laboratory for Sustainable Utilization of Marine Fisheries Resources, Ministry of Agriculture, Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China;
3. Laboratory for Marine Fisheries Science and Food Production Processes, Qingdao National Laboratory for Marine Science and Technology, Qingdao 266071, China
凡纳滨对虾(Litopenaeus vannamei),俗称南美白对虾,具有生长迅速、个体大、营养要求较低、抗病性较强、对水环境变化适应强等特点。1988 年我国引进凡纳滨对虾,目前在全国各地广泛养殖(王兴强等,2004)。随着对虾养殖业的深入发展,养殖集约化日益加深,大量的残饵和粪便导致养殖水体恶化,各种病害频发,严重制约了对虾养殖产业的可持续发展。养殖水体是水产动物赖以生存的环境,水体中的微生物在养殖生态系统的物质循环和能量流动中发挥着巨大作用(郑天凌等,1994)。细菌是水体中的主要微生物类群,对于对虾而言,养殖水体中气单胞菌(Aeromonas)和假单胞菌(Pseudomonadaceae)保持一定数量时,有利于对虾生长; 而一些弧菌(Vibrio)、单胞菌、假单胞菌等大量存在时,可导致对虾患病。有研究表明,养殖水体中弧菌数量达到105indi./mL 以上时,可能会引发弧菌病(朱根福等,1999)。一些有益微生物,如乳酸菌(lactic acid bacteria,LAB)和酵母菌(Yeast)等数量增多,也可以改善养殖水环境质量,为高效、高产的对虾养殖奠定基础。因此,了解对虾养殖水体的微生物组成,对养殖水环境进行监控,通过人为调控养殖水体微生物菌群结构来保持水体的微生态平衡,可以为人们提供绿色健康的养殖模式。1986 年,Kozasa 首次将分离自土壤的1 株芽孢杆菌(Bacillus)用于水产养殖(Kozasa,1986)。目前,在水产养殖中已经认识的有益微生物有光合细菌(Photosynthetic Bacteria Abbr,PSB)、芽孢杆菌、酵母菌等,这些有益微生物在调节养殖水体质量、促进养殖生物生长、提高养殖生物抗病性等方面发挥着重要作用。
由于大多数环境微生物在常规培养基上无法生长,故传统的培养法只能分离和鉴定总微生物量的0.1%-10%(Torsvik et al,1998)。基于进化中相对保守的核酸分子序列的分析技术逐渐用于研究环境微生物多样性(Pace et al,1997)。例如,通过变性梯度凝胶电泳(Denaturing Gradient Gel Electrophoresis,DGGE)、温度梯度凝胶电泳(Temperature Gradient Gel Electrophoresis,TGGE)等分析16S rRNA 基因序列的点突变来反应环境样品的微生物多样性。如,王亭芳等(2012)采用DGGE技术对凡纳滨对虾养殖水体中的微生物多样性进行研究; 高平平等(2003)采用TGGE技术研究废水处理系统活性污泥细菌种群动态变化及多样性。但这些传统的分子生物学技术存在重复性较差、无法定量分析、信息不全等缺陷,故对环境中绝大部分微生物的发掘仍不全面(李祎,2013)。高通量测序技术(High-throughput sequencing,HTS),又称为“下一代”测序(next generation sequencing,NGS)技术,可以一次分别对几百万至上千万条DNA 进行测序(Shendure et al,2008),它的迅速发展弥补了传统分子生物学的不足,能更加全面的反映样品的微生物多样性。高通量测序法具有测序成本低,测序深度高,更有利于低丰度群落物种鉴定的优点,从而提高了微生物群落研究的完整性。
在凡纳滨对虾育苗时期,对亲虾的管理至关重要,亲虾养殖水体中致病微生物达到一定数量则会使虾患病,从而降低生产。养殖水体微生物多样性的研究可以对水体质量进行评价,为及时采取措施调控水体微生态平衡提供条件。为了深入了解凡纳滨对虾繁殖时期亲虾养殖水体的微生物群落结构,本实验采用Illumina HiSeq2500 测序平台,对凡纳滨对虾亲虾养殖水体微生物16S rRNA 基因的两个高变区(V3-V4 区)进行测序分析,从而更全面地分析亲虾养殖水体中的微生物群落组成,不仅可以为亲虾繁殖期的健康养殖提供理论支持,还为我们寻找具有特殊功能的微生物菌种提供丰富的生物资源。
1 材料与方法 1.1 水样采集和预处理水样采自于中国水产科学研究院黄海水产研究所水产遗传育种中心(山东省青岛市即墨鳌山卫)的凡纳滨对虾亲虾养殖池。养殖水泥池的面积为9m2,池深1.2m,水深约为0.6m。虾池每天上午换水一次,换水量为总水量的2/3,对虾按时投喂经高锰酸钾消毒处理的鲜活沙蚕(Sipunculus nudus),日投喂量为体重的30%。取样时水样温度为26℃,盐度为35(W/V)。选取雄虾池(A7 池,42 尾)、雌虾池(A8 池,37 尾)进行水样采集。每池各选取水深为0.2m 处的3 个不同的采样点,于2015 年10 月23 日上午8 时,用采样器取每个取样点水2L 于灭菌的三角烧瓶中,编号分别为A7(X1.1、X2.2、X3.2)、A8(C1.2、C2.2、C3.2)。所有样品迅速运回实验室并保存在4℃ 条件下,及时用孔径为0.22μm 的醋酸纤维素滤膜过滤收集水体中的微生物。将过滤好的滤膜保存在灭菌的50mL 离心管中,并保存在-20℃ 条件下,用于微生物宏基因组DNA 的提取。
1.2 水样微生物宏基因组DNA 的提取水样微生物宏基因组DNA 的提取采用酚氯仿法和细菌基因组提取试剂盒(艾德莱,北京)。具体为: 剪碎滤膜,放入1.5mL 无菌离心管中,加600μL 无菌STE 缓冲液(10mmol/L Tris-HCl,100mmol/L NaCl,1mmol/L EDTA,pH8.0)混匀,涡旋1min。加60μL 溶菌酶,涡旋10s,置于37℃ 水浴锅水浴30min,每10min 摇晃一次。加入60μL 10%的SDS,混匀,加6μL 蛋白酶K,涡旋10s,65℃ 水浴20min,每10min 摇晃一次。1400g 条件下离心15min,吸取上清液于新的离心管中。加 600μL 酚︰氯仿︰异戊醇(25︰24︰1),混匀后在15294g 转速下离心10min。吸取上清液转入新的离心管,加 600μL 酚︰氯仿︰异戊醇(25︰24︰1),混匀后在15294g 转速下离心10min,重复此步骤两次。加入2 倍体积无水乙醇,-20℃ 过夜。之后采用细菌基因组提取试剂盒的方法过柱处理。提取好的DNA 采用Nanodrop 2000(Thermo Fisher,美国)测定其浓度,将合格样品(OD260/280=1.8-2.0,浓度≥50ng/μL,总量≥2μg)送至诺禾致源公司(北京)进行高通量测序。
1.3 PCR 扩增及扩增产物的纯化取适量DNA 于离心管中,使用无菌水稀释样品至1ng/μL,以稀释后的基因组DNA 为模板,选择加入Barcode 标签的341F(CCTACGGGNGGCWGCAG)和805R(GACTACHVGGGTATCTAATCC)作为引物,利用高效高保真酶(New England Biolabs,NEB)扩增16S rRNA 基因V3-V4 区。PCR 产物使用2%浓度的琼脂糖凝胶进行电泳检测; 根据PCR 产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测混合的PCR 产物,利用胶回收试剂盒(QIAGEN)对目的条带进行回收。
1.4 文库构建和上机测序使用TruSeq® DNA PCR-Free Sample Preparation Kit 建库试剂盒(Illumina)进行文库构建,构建好的文库经过Qubit和Q-PCR 定量,使用HiSeq2500(诺禾致源公司,北京)进行测序。
1.5 测序数据的处理与分析 1.5.1 高通量序列的处理采用Illumina HiSeq 测序平台得到原始数据(raw data),然后进行拼接和质控,得到有效数据(clean data),再进行嵌合体过滤,得到可用于后续分析的有效数据(effective tags)。然后,基于有效数据进行OTUs(operational taxonomic units)聚类。
1.5.2 统计分析利用Uparse 软件对所有样品的全部effective tags 进行聚类,以97%的一致性将序列聚类成OTUs。根据OTUs 聚类结果,对每个OTU 的代表序列做物种注释,得到对应的物种信息,根据物种注释结果,选取每个样品在门、纲、目、科、属5个分类级别上最大丰度排名前10 的物种,生成物种相对丰度柱形累加图。分别统计了雌雄虾养殖水体的所有样品中均存在的OTU,找出其核心微生物菌群(core microbiome,指两个或多个与某一特定环境相关的微生物集群中所共有的成员)(Turnbaugh et al,2007)。从物种分类信息和样品间差异两个层面进行聚类并绘制成热图(heatmap)。采用热图对核心微生物群在各分类水平上进行可视化展示。进一步对OTUs 进行丰度分析、α-多样性(alpha diversity)和β-多样性计算等,以得到样品内物种丰富度和均匀度信息等。α- 多样性用于分析样品内的微生物群落多样性(Holmström et al,1999),通过单样本的多样性分析可以反映样品内微生物群落的丰富度和多样性。使用Qiime 软件(Version 1.7.0)计算α-多样性指数,包括Observed-species,Shannon,Simpson,Chao1,ACE,Goods-coverage 指数。对不同样品在97%一致性阈值下的α-多样性分析指数进行统计。β-多样性研究中,基于weighted unifrac 距离和unweighted unifrac 距离进行了主坐标分析(Principal Coordinates Analysis,PCoA)分析,并选取贡献率最大的主坐标组合进行作图展示。LEfSe(LDA Effect Size)分析能够在组与组之间寻找具有统计学差异的Biomarker(Segata et al,2011),即组间差异显著的物种。利用LEfSe 软件进行数据分析,并采用LDA 值分布柱状图和进化分支图(系统发育分布)来展示LEfSe 的统计结果。为进一步挖掘分组样品间的群落结构差异,选用T-test 、Metastats、Anosim和MRPP等统计分析方法对分组样品的物种组成和群落结果进行差异显著性检验。同时,结合环境因素进行CCA/RDA 分析和多样性指数与环境因子的相关性分析,得到显著影响组间群落变化的环境影响因子。
2 结果与分析 2.1 高通量测序数据从雌雄虾池获取的6 个水样(X1.1、X2.2、X3.2、C1.2、C2.2、C3.2))经过Illumina HiSeq2500 平台测序后分别得到各自的下机数据Raw PE,经过拼接和质控后得到Clean Tags,经过嵌合体过滤后得到Effective Tags。6 个水样经高通量测序后共得到230398 条可用于分析的序列,包含4603 条频数为1 且无法被聚类到OTUs 的序列,3 条没有获得注释信息的序列和225792条可用于构建OTUs 并且获得注释信息的序列。以97%的一致性,将每个样品所得的序列聚类成OTU,其中雄虾池3 个样品的平均OTU 数目为396; 雌虾池3 个样品的平均OTU 数目为475。根据物种注释情况,统计每个样品注释到各分类水平上的序列数目,在门、纲、目、科、属、种6 个水平上,雄虾养殖水体序列数分别平均为41583、41463、411494、40709、2496和148 条; 雌虾养殖水体序列数分别平均为33578、33512、32981、32295、7823和113 条。
2.2 雌雄虾养殖水体细菌群落特征 2.2.1 物种相对丰度展示对于雄虾养殖水体,优势门中变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria)的相对丰度分别为76.15%、21.28%和1.65%; 优势纲中α-变形菌纲(Alphaproteobacteria)、Saprospirae、γ-变形菌纲(Gammaproteobacteria)、Flavobacteriia和放线菌纲(Actinobacteria)的相对丰度分别为73.11%、19.89%、2.30% 、1.29%和1.42%; 优势目中红杆菌目(Rhodobacterales)、腐螺旋菌目(Saprospirales)、放线菌目(Actinomycetales)和黄杆菌目(Flavobacteriales)的相对丰度分别为71.24%、19.89%、1.42%和1.29%;优势科中红杆菌科(Rhodobacteraceae)和腐螺旋菌科(Saprospiraceae)的相对丰度分别为71.20%和19.89%;优势属Pseudoruegeria和Marivita 的相对丰度分别为2.02%和1.43%。对于雌虾养殖水体,优势门变形菌门、拟杆菌门和放线菌门的相对丰度分别为66.01%、29.02%和3.82%; 优势纲中α-变形菌纲、Saprospirae、Flavobacteriia、γ-变形菌纲和放线菌纲的相对丰度分别为58.93%、19.14%、9.14%、5.51%和3.48%; 优势目中红杆菌目、腐螺旋菌目、黄杆菌目和放线菌目的相对丰度分别为53.87%、19.14%、9.14%和3.48%;优势科中红杆菌科、腐螺旋菌科、Cryomorphaceae 的相对丰度分别为53.76%、19.11%和4.87%; 优势属Pseudoruegeria、Marivita、HTCC2207和Sediminicola 的相对丰度分别为8.41%、5.33%、2.03%和1.80%。图 1 是选取门(图 1a)和属(图 1b)水平最大丰度排名前10 的物种生成的物种相对丰度柱形图,从图中可知,雌雄虾养殖水体中丰度最高的2 个门均是变形菌门和拟杆菌门,丰度最高的2 个属均是Pseudoruegeria和Marivita。
| Sample name | Total tags | Taxon tags | Unclassified tags | Unique tags | OUT number |
| X1.1 | 45190 | 44539 | 0 | 651 | 418 |
| X2.2 | 45247 | 44636 | 0 | 611 | 402 |
| X3.2 | 36245 | 35795 | 1 | 449 | 367 |
| C1.2 | 37496 | 36370 | 0 | 1126 | 465 |
| C2.2 | 34588 | 33683 | 2 | 903 | 496 |
| C3.2 | 31632 | 30769 | 0 | 863 | 463 |
| 注: Total Tags. 指每个样品的总的Tags 数目(等同于Effective Tags, 每个样品用于OTU 聚类等后续分析的有效数据); Taxon Tags. 指用于构建OTUs并且获得注释信息的Tags 数目; Unclassified Tags. 指没有获得注释信息的Tags 数目; Unique Tags. 指频数为1, 并且无法被聚类到OTUs 的Tags 数目(无法聚类到OTUs 的序列将不用于后续分析); OTU number. 指每个样品得到的OTUs 数目。 | |||||
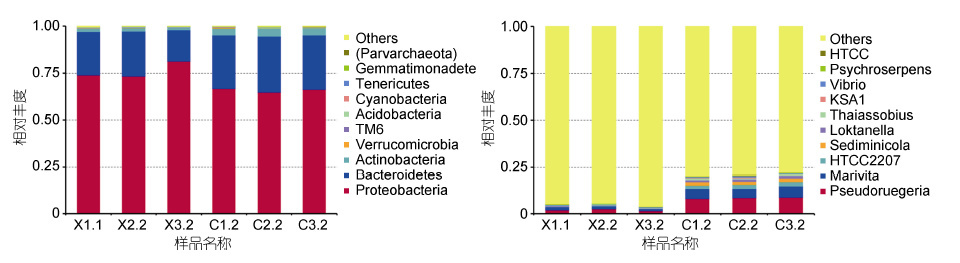 |
| 图 1 样品在门水平(a)和属水平(b)物种相对丰度 Fig. 1 Relative abundance of species in phylum(a)and genus(b)level 注: Others 表示图中这10 个门(属)之外的其他所有门(属)的相对丰度之和。 |
核心微生物分析表明,雌雄虾养殖水体的核心OTU 数目分别为475和396。图 2 是核心微生物群在属水平上的热图。从图中可以看出,雌雄虾养殖水体的核心微生物群(属水平)明显不同。雄虾养殖水体的核心属主要包括Glaciecola、Octadecabacter、Methylobacterium、Psychroserpens、Olleya; 雌虾养殖水体的核心属主要包括Thalassomonas、Thalassobius、Marinicella、Fluviicola、Sediminicola、Marivita、Pseudoruegeria、HTCC2207、Pseudomonas、Vibrio、Synechococcus、Owenweeksia等,其中Vibrio、Pseudomonas和Owenweeksia等属中包含了部分对虾病原菌或条件性致病菌种类。
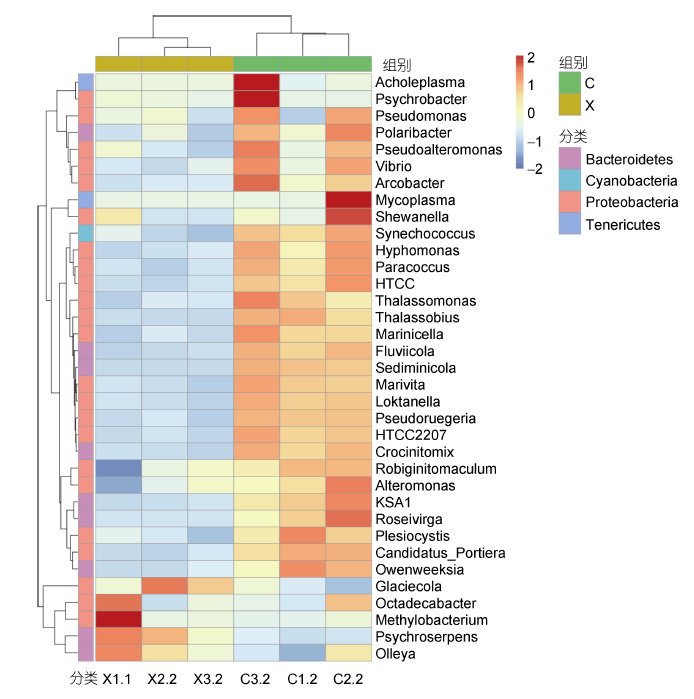 |
| 图 2 核心微生物群在属水平上的热图 Fig. 2 The heatmap of core microbiome in genus level 注: 图中横向为物种注释信息,纵向为样品信息,左侧的聚类树为物种聚类树,上方的聚类树为样品组间的聚类树,中间热图对应的值为每一行物种相对丰度经过标准化处理后得到的Z 值(即一个样品在某个分类上的Z 值为样品在该分类上的相对丰度和所有样品在该分类的平均相对丰度的差除以所有样品在该分类上的标准差所得到的值)。 |
如表 2 所示,雌虾池3 个样品(C1.2、C2.2、C3.2)群落丰度指数ACE 指数(471.484、495.638和444.435)和Chao1 指数(458.628、488和428.484)略高于雄虾池3个样品(X1.1、X2.2、X3.2)的ACE 指数(486.375 、422.907和376.52)和Chao1 指数(447.939 、387.511和353.13)。雌虾池3 个样品(C1.2、C2.2、C3.2)的Shannon 多样性指数和Simpson 多样性指数也略高于雄虾池3 个样品(X1.1、X2.2、X3.2)。比较雌雄虾养殖水体细菌群落的α-多样性指数得知,雌虾养殖水体细菌群落多样性和丰度均高于雄虾水体。对雌雄虾养殖水体细菌群落α-多样性指数进行T-test 检验得知,Chao1、ACE和Goods-coverage 指数无显著差异(P>0.05); Observed-species、Shannon和Simpson 指数差异显著(P<0.05)。
| Sample name | Observed-species | Shannon | Simpson | Chao1 | ACE | Goods-coverage |
| X1.1 | 368 | 3.06 | 0.716 | 447.939 | 486.375 | 0.996 |
| X2.2 | 343 | 2.94 | 0.701 | 387.511 | 422.907 | 0.997 |
| X3.2 | 319 | 2.617 | 0.602 | 353.13 | 376.52 | 0.998 |
| C1.2 | 415 | 4.735 | 0.919 | 458.628 | 471.484 | 0.997 |
| C2.2 | 443 | 4.966 | 0.93 | 488 | 495.638 | 0.997 |
| C3.2 | 419 | 4.842 | 0.927 | 428.484 | 444.435 | 0.998 |
基于weighted unifrac和unweighted unifrac 主坐标分析时,前两个主坐标的累计方差贡献率分别为98.42%和64.58%。其中,在weighted unifrac 主坐标分析中,第一主坐标PC1和第二主坐标PC2 分别占总变异来源的91.39%和7.03%。对雌雄虾养殖水体细菌群落β-多样性指数进行T-test 检验得知,基于weighted unifrac 分析,β-多样性无显著差异(P>0.05); 基于unweighted unifrac 分析,β-多样性差异显著(P<0.05)。从LDA 值分布柱状图(图 4a)可以看出,Alphapro teobacter i a 、Proteobacteria、Rhodopbacterales、Rhodopbacteraceae、Actinomycetales、Actinobacteria、Rickettsiales、Pelagibacteraceae等是雌雄虾养殖水体中差异显著的物种。在雄虾养殖水体中,Alphaproteobacteria、Proteobacteria、Rhodopbacterales、Rhodopbacteraceae 的LDA 值均接近于4.8; 而对于雌虾养殖水体而言,Flavobacteriales 、Bacteroidetes 、Flavobacteriia 、Pseudoruegeria等LDA 值均接近于4.8。由进化分支图(图 4b)知,Cryomorphaceae、Flavobacteriaceae、Flavobacteriales、Flavobacteriia 在进化上属于同一个门Bacteroidetes; Pelagibacteraceae 、Rickettsiales 、Rhodopbacterales 、Rhodopbacteraceae 、Alphaproteobacteria 、Gammaproteobacteria 、Alteromonadales 在进化上属于同一个门Proteobacteria; 而Actinomycetales 在进化上属于单独的一个门Actinobacteria 。Actinomycetales 、Cryomorphaceae、Flavobacteriaceae、Flavobacteriales、Flavobacteriia 、Rickettsiales 、Alteromonadales 、Pelagibacteraceae、Gammaproteobacteria 是雌虾养殖水体中的重要微生物类群; 而Rhodopbacterales、Rhodopbacteraceae 、Alphaproteobacteria 、Proteobacteria 是雄虾养殖水体的重要微生物类群。
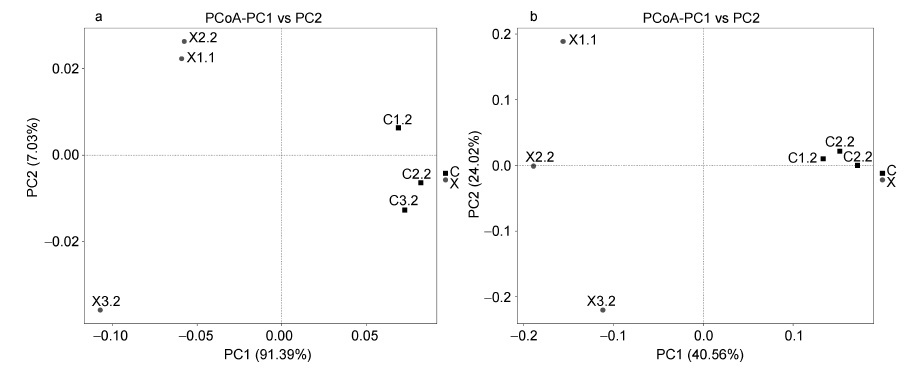 |
| 图 3 基于Weighted Unifrac 距离(a)和Unweighted Unifrac 距离(b)的主坐标分析 Fig. 3 Principal Coordinates Analysis in weighted unifrac distance(a)and unweighted unifrac distance(b) 注: 横坐标表示第一主成分,百分比则表示第一主成分对样品差异的贡献值; 纵坐标表示第二主成分,百分比表示第二主成分对样品差异的贡献值; 图中的每个点表示一个样品,其中,黑色方块代表雌虾水体样品,红色圆圈代表雄虾水体样品,同一个组的样品使用同一种颜色表示 |
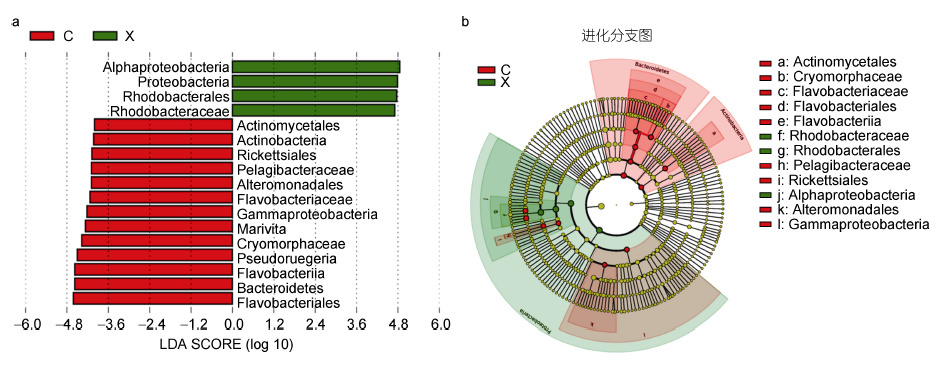 |
| 图 4 LDA 值分布柱状图(a)和进化分支图(b) Fig. 4 The distribution histogram in LDA value(a)and the phylogenetic tree(b) 注: LDA 值分布柱状图中展示了LDA Score 大于设定值(默认设置为4)的物种,即组间具有统计学差异的Biomarker。展示了不同组中丰度差异显著的物种,柱状图的长度代表差异物种的影响大小(即为 LDA Score)。在进化分支图中,由内至外辐射的圆圈代表了由门至属(或种)的分类级别。在不同分类级别上的每一个小圆圈代表该水平下的一个分类,小圆圈直径大小与相对丰度大小呈正比。着色原则: 无显著差异的物种统一着色为黄色,差异物种Biomarker 跟随组进行着色,红色节点表示在红色组别中起到重要作用的微生物类群,绿色节点表示在绿色组别中起到重要作用的微生物类群。红色和绿色分别表示雌雄虾养殖水体,图中英文字母表示的物种名称在右侧图例中进行展示 |
高通量测序技术在宏基因组学(Metagenomics)的研究中具有重大意义,宏基因组学又称为微生物环境基因组学、元基因组学,是一门直接研究自然环境下微生物群落(包含了可培养的和不可培养的细菌、真菌和病毒等基因组的总和的学科)(Shade et al,2012)。宏基因组学研究的对象不是某特定的微生物或其细胞中的总DNA,而是特定环境中的总DNA,不需要对微生物进行分离培养和纯化,可以全面地对微生物进行分析,对于我们认识和利用未培养微生物提供了一条新的途径。本研究提取了凡纳滨对虾养殖水体微生物宏基因组DNA,采用Illumina HiSeq2500 测序平台分析了凡纳滨对虾养殖水体菌群多样性。得到有效序列31632-45190 条,可归纳为367-496 个分类操作单元。基于测序数据,评估了凡纳滨对虾雌雄养殖水体中细菌群落的特征,比较了两个养殖池水体微生物多样性和群落结构的差异。雌雄虾养殖池水体细菌群落的α-多样性指数中Chao1、ACE和Goods-coverage 指数无显著差异; Observedspecies 、Shannon和Simpson 指数差异显著。β-多样性指数中,基于weighted unifrac 的β-多样性无显著差异; 基于unweighted unifrac 的β-多样性差异显著。
雌雄虾养殖水体细菌群落组成有一定差异,这可能与雌雄虾养殖池的虾数目和虾的大小有一定关系,雄虾数目多于雌虾,但雌虾体重均高于雄虾。雌雄虾养殖池水体的微生物物种相对丰度排列前10 的分别均是变形菌门、拟杆菌门、放线菌门、疣微菌门(Verrucomicrobia)、 候选菌门(TM6)、酸杆菌门(Acidobacteria)、蓝细菌门(Cyanobacteria)、软壁菌门(Tenericutes)、芽单胞菌门(Gemmatimonadetes)和(Parvarchaeota)。变形菌门和拟杆菌门为各个样品的优势菌,在雄虾养殖池水体中的平均相对丰度分别为76.15%和21.28%; 在雌虾养殖池水体中平均相对丰度分别为66.01%和29.02%。变形菌门是海洋环境和海洋沉积物中的主要类群(Whittaker,1972)。雄虾养殖池水体中α-变形菌纲占73.11%,β-变形菌纲占0.23%; 雌虾养殖池水体中α-变形菌纲占58.93%,β-变形菌纲占0.46%。α-变形菌纲和β-变形菌纲对于多环芳烃(Polycyclic Aromatic Hydrocarbons,PAHs)具有降解作用,可以利用PAHs 进行生长(袁军,2008)。拟杆菌门是海洋环境中的重要微生物类群,是溶解性有机物的主要消费者(Cottrell et al,2000)。在养殖环境中,残留的饵料以及养殖动物的粪便在水体中未被及时分解,便在养殖水体中沉积下来,造成养殖池底部拟杆菌门细菌含量增加(Rosselló-Mora et al,1999)。拟杆菌门中的 Flavobacteriia 纲是海水中高分子有机物的主要降解者,雌虾养殖水体Flavobacteriia 纲所占的比例(9.14%)大于雄虾养殖水体(1.29%),这可能是因为雌虾养殖池中残留饵料较多而导致的。有机物降解菌群的大量存在可加快养殖水体中有机物的的降解,从而减轻水产动物所面临的环境压力。疣微菌门在雌雄虾养殖水体中所占的比例分别为0.34%和0.10%。海洋环境中广泛分布着疣微菌门细菌,其数量约占水体细菌总量的2%,该门类细菌大多属于不可培养的微生物,其功能目前尚不明确(Freitas et al,2012)。
本研究表明,雌雄虾养殖水体的核心微生物(属水平)明显不同(图 2),特别是雌虾养殖水体中包含了一些致病菌或条件性致病菌,如弧菌、Pseudomonas和Owenweeksia,但这些属不是雄虾养殖水体的核心属。其中,弧菌是海洋环境中常见细菌,也是海水养殖对虾肠道的优势菌群(Gatesoupe,1999; Oxley et al,2002)。研究表明,弧菌对于养殖水体中有机物的降解具有重要意义,同时也具有硝酸盐异化作用(赵晓伟等,2015)。但有些弧菌对水产动物甚至人类具有潜在致病性,例如,副溶血弧菌(Vibrio Parahaemolyticus)、哈维氏弧菌(Vibrio harveyi)、创伤弧菌(Vibrio vulnificus)、溶藻弧菌(Vibrio alginolyticus)、鳗弧菌(Vibrio anguillarum)等。有研究表明,导致凡纳滨对虾养殖过程中“早期死亡综合征”(Early mortality syndrome,EMS)的病原可能是副溶血弧菌的特异变种(杨先乐等,2005; Tran et al,2013)。因此,采取有效措施控制养殖环境中致病弧菌的数量对于水产动物的健康至关重要。
4 结论本研究利用HiSeq 测序技术对凡纳滨对虾亲虾繁殖期养殖水体的微生物进行分析,揭示了其养殖环境中微生物的多样性和丰度,为凡纳滨对虾繁殖期亲虾养殖过程中的病害防治及新的微生物资源的开发提供基础数据。
| 王兴强, 马甡, 董双林, 2004. 凡纳滨对虾生物学及养殖生态学研究进展. 海洋湖沼通报, (4) : 94–100 |
| 王亭芳, 金凤杰, 马士禹, 等, 2012. DGGE技术对南美白对虾养殖水体中微生物多样性的研究. 生物技术通报, (10) : 131–136 |
| 朱根福, 宣晓冬, 江富二, 1999. 杭州西湖水体中微生物生理群生态分布的初步研究. 生态学报, 19 (3) : 435–440 |
| 李祎, 郑伟, 郑天凌, 2013. 海洋微生物多样性及其分子生态学研究进展. 微生物学通报, 40 (4) : 655–668 |
| 杨先乐, 郑宗林, 2005. 南美白对虾病害流行的新动向及其思考. 淡水渔业, 35 (3) : 3–6 |
| 郑天凌, 王海黎, 洪华生, 1994. 微生物在碳的海洋生物地球化学循环中的作用. 生态学杂志, 13 (4) : 47–50 |
| 赵晓伟, 丁君, 窦妍, 等, 2015. 基于MiSeq测序技术分析红鳍东方鲀养殖环境菌群多样性. 生态学杂志, 34 (10) : 2965–2970 |
| 袁军, 2008. 印度洋深海多环芳烃降解菌的多样性分析及降解菌新种的分类鉴定与降解机理初步研究. 厦门:厦门大学博士学位论文, 1-2 |
| 高平平, 晁群芳, 张学礼, 等, 2003. TGGE分析焦化废水处理系统活性污泥细菌种群动态变化及多样性. 生态学报, 23 (10) : 1963–1969 |
| Cottrell M T, Kirchman D L, 2000. Natural assemblages of marine proteobacteria and members of the Cytophaga-Flavobacter cluster consuming low- and high-molecularweight dissolved organic matter. Applied and Environmental Microbiology, 66 (4) : 1692–1697 DOI:10.1128/AEM.66.4.1692-1697.2000 |
| Freitas S, Hatosy S, Fuhrman J A, et al, 2012. Global distribution and diversity of marine Verrucomicrobia. The ISME Journal, 6 (8) : 1499–1505 DOI:10.1038/ismej.2012.3 |
| Holmström C, Kjelleberg S, 1999. Marine Pseudoalteromonas species are associated with higher organisms and produce biologically active extracellular agents. FEMS Microbiology Ecology, 30 (4) : 285–293 DOI:10.1111/fem.1999.30.issue-4 |
| Kozasa M, 1986. Toyocerin (Bacillus toyoi) as growth promoter for animal feeding. Microbiologie Aliments Nutrition, 4 (2) : 121–135 |
| Oxley A P A, Shipton W, Owens L, et al, 2002. Bacterial flora from the gut of the wild and cultured banana Prawn, Penaeus merguiensis. Journal of Applied Microbiology, 93 (2) : 214–223 DOI:10.1046/j.1365-2672.2002.01673.x |
| Pace N R, 1997. A molecular view of microbial diversity and the biosphere. Science, 276 (5313) : 734–740 DOI:10.1126/science.276.5313.734 |
| Rosselló-Mora R, Thamdrup B, Schäfer H, et al, 1999. The response of the microbial community of marine sediments to organic carbon input under anaerobic conditions. Systematic and Applied Microbiology, 22 (2) : 237–248 DOI:10.1016/S0723-2020(99)80071-X |
| Segata N, Izard J, Waldron L, et al, 2011. Metagenomic biomarker discovery and explanation. Genome Biology, 12 (6) : R60 DOI:10.1186/gb-2011-12-6-r60 |
| Shade A, Handelsman J, 2012. Beyond the Venn diagram:the hunt for a core microbiome. Environmental Microbiology, 14 (1) : 4–12 DOI:10.1111/j.1462-2920.2011.02585.x |
| Shendure J, Ji H, 2008. Next-generation DNA sequencing. Nature Biotechnology, 26 (10) : 1135–1145 DOI:10.1038/nbt1486 |
| Tran L, Nunan L, Redman R M, et al, 2013. EMS/AHPNS:infectious disease caused by bacteria. Global Aquaculture Advocate, : 18–20 |
| Torsvik V, Daae F L, Sandaa R A, et al, 1998. Novel techniques for analysing microbial diversity in natural and perturbed environments. Journal of Biotechnology, 64 (1) : 53–62 DOI:10.1016/S0168-1656(98)00103-5 |
| Turnbaugh P J, Ley R E, Hamady M, et al, 2007. The human microbiome project. Nature, 449 (7164) : 804–810 DOI:10.1038/nature06244 |
| Whittaker R H, 1972. Evolution and measurement of species diversity. Taxon, 21 (2-3) : 213–251 |
 2017, Vol. 48
2017, Vol. 48


