中国海洋湖沼学会主办。
文章信息
- 常城, 韩慧宗, 王腾腾, 马海涛, 刘阳, 王斐, 张明亮, 王力勇, 姜海滨. 2017.
- CHANG Cheng, HAN Hui-Zong, WANG Teng-Teng, MA Hai-Tao, LIU Yang, WANG Fei, ZHANG Ming-Liang, WANG Li-Yong, JIANG Hai-Bin. 2017.
- 单环刺螠(Urechis unicinctus)微卫星标记开发及5个地理种群遗传结构分析
- MICROSATELLITE MARKER ISOLATION AND GENETIC STRUCTURE ANALYSIS FOR FIVE POPULATIONS OF URECHIS UNICINCTUS
- 海洋与湖沼, 48(3): 498-507
- Oceanologia et Limnologia Sinica, 48(3): 498-507.
- http://dx.doi.org/10.11693/hyhz20161100238
-
文章历史
- 收稿日期:2016-11-02
- 收修改稿日期:2017-01-12
2. 山东省海洋资源与环境研究院山东省海洋生态修复重点实验室 烟台 264006;
3. 中国科学院南海海洋研究所 广州 510301;
4. 烟台市水产研究所 烟台 264003
2. Shandong Provincial Key Laboratory of Restoration for Marine Ecology, Shandong Marine Resource and Environment Research Institute, Yantai 264006, China;
3. South China Sea Institute of Oceanology, Chinese Academy of Sciences, Guangzhou 510301, China;
4. Yantai Fisheries Research Institute, Yantai 264003, China
单环刺螠, 俗称“海肠子”, 隶属于螠虫动物门(Echiurida)、螠纲(Echiurida)、无管螠目(Xenopneusta)、刺螠科(Urichidae), 主要分布于中国、俄罗斯、日本及朝鲜半岛的沿海潮间带下区及潮下带浅水区域, 是无管螠目在我国沿海分布的唯一种类。单环刺螠味道鲜美、营养丰富、食药用价值高(Jo et al, 2008; Wang et al, 2010), 而且具改良底质能力(Kang et al, 2010), 是一种有较大养殖开发前景的海洋生物。近年来单环刺螠野生资源已无法满足市场需求的增加, 商品价格不断攀升, 其种质也呈现出一定程度的退化。因此, 单环刺螠人工繁育技术研究引起国内外学者的普遍关注, 亲本来源主要是在当地沿海滩涂捕获, 这种方式极易导致群体遗传多样性降低(Miller et al, 2015), 因此积极开展单环刺螠遗传多样性相关研究和调查, 避免种质衰退, 成为当前面临的迫切任务。
微卫星标记, 又称简单重复序列(simple sequence repeats, SSR), 因其具有多态性高、共显性遗传、易于检测且等位条带易于识别等特点, 被广泛应用于水产经济动物的群体遗传多样性分析、系谱认证、遗传连锁图谱构建及数量性状连锁定位等工作(Feng et al, 2014; Mohanty et al, 2014)。迄今为止, 对于单环刺螠的研究大多集中于人工养殖与繁育、营养组分分析等方面(王雷等, 2011), 有关微卫星标记开发及群体多样性的研究仍未见报道。本研究利用高通量测序的方法从单环刺螠基因组中开发了22对多态性高的微卫星标记, 并对采自我国不同海域的5个地理群体进行遗传多样性分析, 旨在摸索筛选单环刺螠微卫星的方法, 开发更多的单环刺螠微卫星标记, 为单环刺螠种质资源保护、良种培育及增殖放流提供科学依据。
1 材料与方法 1.1 实验材料单环刺螠群体分别采自烟台、秦皇岛、潍坊、青岛和大连等附近海域(见表 1), 每个地点各取30头。活体解剖取其体壁, 立即提取基因组DNA。
采用酚/氯仿抽提方法提取基因组DNA(孙孝德, 2011), 1%琼脂糖电泳检测其完整性, NanoDrop2000紫外分光光度计测DNA纯度和浓度, 选取一个DNA浓度适合、完整度好和纯度高的秦皇岛样本送至北京诺禾致源生物公司进行测序并构建RAD文库; 剩余的DNA模板稀释至30ng/μL, 4℃保存备用。
1.3 微卫星引物设计和筛选在RAD文库中, 选取微卫星核心序列大小在24bp以上的合适序列用Primer 5.0软件设计引物。引物长度控制在20-25bp之间, 产物长度控制在90-300bp之间, Tm值控制在40-60℃, 正反引物相差不超过10℃, GC含量控制在40%-60%, 正反引物相差不超过10%。引物送由上海生工生物工程有限公司合成。
首先选用6个个体DNA模板对合成引物进行初步筛选, 选出能稳定扩增出目的条带的微卫星标记, 并通过温度梯度PCR优化引物退火温度。其次用30个秦皇岛群体模板对初选引物进行PCR扩增, 筛选出具多态性的微卫星引物。然后用筛选出的引物在剩余4个群体中PCR扩增, 并对单环刺螠5个地理群体的遗传多样性进行评价。PCR反应体系如下: 2μLDNA模板(30ng/μL), 2.5μL10×buffer(Mg2+Plus), 引物各1μL(50μmol/L), 0.5μLdNTP(10mmol/L), 0.3μLTaq DNA聚合酶(5U/μL), 17.7μL灭菌水, 共计25μL的PCR反应体系。反应程序为: 94℃预变性5min, 94℃变性45s, 退火反应45s, 72℃延伸1min, 35个循环, 最后72℃延伸10min。
1.4 数据统计与处理Gel-Por Analyzer软件统计各微卫星DNA条带分子量大小; PopGene32软件计算每个位点的等位基因数(Na)、有效等位基因数(Ne)、观测杂合度(Ho)、期望杂合度(He); Cervus软件计算多态信息含量(PIC); GenePop软件检验Hardy-Weinberg平衡, 并经Bonferroni校正; FSTAT 2.9.3软件以Nei(1972)参数计算两两群体间遗传分化指数(FST); 运用TFPGA软件根据Nei(1972)参数进行群间遗传距离(DS)的计算, 并进行UPGMA聚类分析; 利用IBDWS软件中的Mantel检验评估单环刺螠群体间遗传距离和地理距离的相关性。
2 结果 2.1 DNA提取结果提取的单环刺螠组织DNA的整体浓度在400-700ng/μL, OD260/OD280值在1.9-2.1之间, 1%琼脂糖凝胶电泳检测, 个别样本有拖带及胶孔蛋白残留现象, 但不影响PCR扩增。
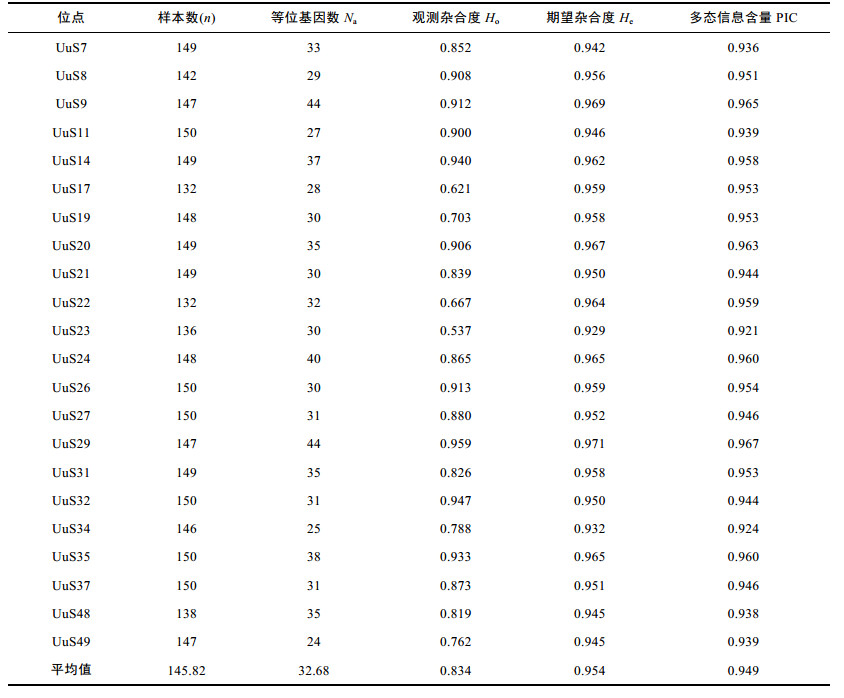
2.2 微卫星标记筛选与评价Primer 5.0软件设计引物200对, 挑选50对引物进行合成, 利用30个秦皇岛野生群体DNA样本对其进行筛选和多态性分析, 共筛选出了22对能稳定扩增且具多态性的微卫星标记, 其余28对引物扩增产物呈单态或无产物, 结果见表 2。根据Weber(1990)提出的微卫星分类标准, 22个微卫星序列全部为完美型。其中二核苷酸微卫星5个(22.73%), 三核苷酸微卫星14个(63.64%), 四核苷酸微卫星2个(9.09%), 五核苷酸微卫星1个(4.55%)。每个微卫星位点的平均等位基因数(Na)为10.6364, 平均期望杂合度(He)为0.8826, 平均多态信息含量(PIC)为0.8535。其中一个微卫星引物UuS27在群体中扩增的电泳图如图 1。

|
 |
| 图 1 微卫星引物UuS27在单环刺螠中扩增的电泳图谱 Fig. 1 Electrophoretic patterns of PCR products amplified by primer UuS27 in U. unicinctus |
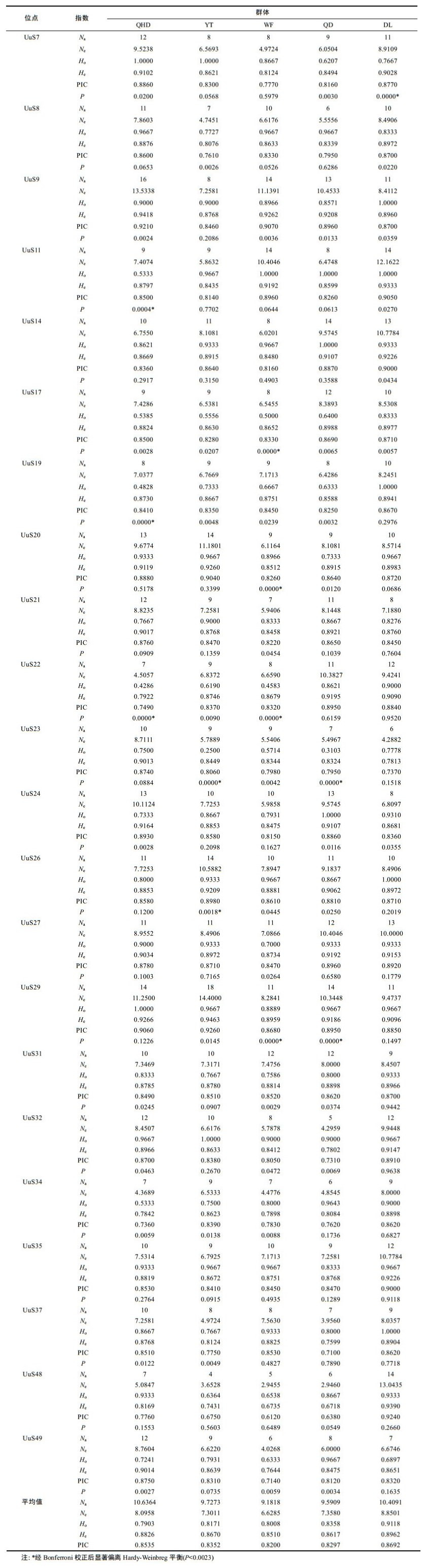
从5个群体整体来看, 22对微卫星位点的遗传统计指标见表 3, 共检测到719个, 等位基因数(Na)介于24-44, 有效等位基因数(Ne)介于2.95-14.40, 观测杂合度(Ho)和期望杂合度(He)范围分别是0.537-0.959和0.929-0.971, 每个位点的多态信息含量(PIC)介于0.921-0.967之间。
|
22个微卫星位点在5个单环刺螠地理群体中的遗传多样性信息见表 4, 从表 4可以看出, 平均等位基因数差别很小, 由高到低依次为QHD群体(10.64)、DL群体(10.41)、YT群体(9.73)、QD群体(9.59)、WF群体(9.18);平均多态信息含量的顺序与平均等位基因数排序一致。DL群体的平均观测杂合度(Ho=0.9118)、期望杂合度(He=0.8962) 及多态信息含量(PIC=0.8692) 均最大, WF群体的平均期望杂合度(He=0.8510)、多态信息含量(PIC=0.8200) 及等位基因数均最小。以上结果表明, 5个单环刺螠群体的遗传多样性较为丰富, 其中DL群体的遗传多样性在5个群体中最高, 而WF群体最低。
|
基于Hardy-Weinberg平衡定律对5个地理群体中的每个位点基因平衡状态进行检测(表 4), 经Bonferroni校正(P < 0.0023) 后, 在QHD群体中, 有3个位点显著Hardy-Weinberg平衡, 分别为UuS11、UuS19、UuS22;在YT群体中, 有2个位点显著偏离, 分别为UuS23、UuS26;在WF群体中, 有4个位点显著偏离平衡, 分别为UuS17、UuS20、UuS22、UuS29;在QD群体中, 有2个位点显著偏离, 分别为UuS23、UuS29;在DL群体中, 除UuS7位点外, 其余位点等位基因频率均符合。连锁不平衡检测表明, 各位点间无连锁不平衡现象。
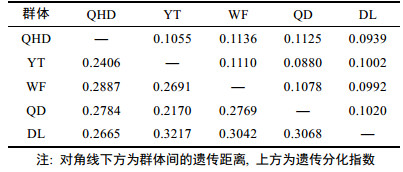
2.4 群体遗传结构分析5个单环刺螠群体间的平均遗传分化指数(FST)和遗传距离(DS)如表 5所示。不同群体间的遗传分化指数FST值范围为0.0880-0.1136, 各群体间FST值差异不显著(P>0.05);从Nei’s标准遗传距离来看, YT和DL群体遗传距离最远(DS=0.3217), 说明两群体间亲缘关系最远; YT和QD群体遗传距离最近(DS= 0.2170), 说明两群体间亲缘关系较近; DL群体与其他群体均有较远的遗传距离。基于Nei遗传距离采用UPGMA方法构建系统发育树(图 2), YT与QD群体首先聚为一起, 再与QHD群体聚为一支, 然后跟WF群体聚为一支, DL群体为外围群(outgroup), 再聚为一支。IBD分析结果显示单环刺螠群体之间遗传距离和地理距离不存在显著相关(见图 3, R2=0.1600, P=0.876>0.05), 表明基因流动不符合地理距离隔离模式。
|
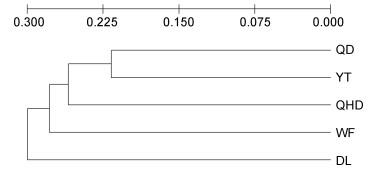 |
| 图 2 单环刺螠5个群体的UPGMA聚类图 Fig. 2 UPGMA dendrogram of 5 U. unicinctus populations |
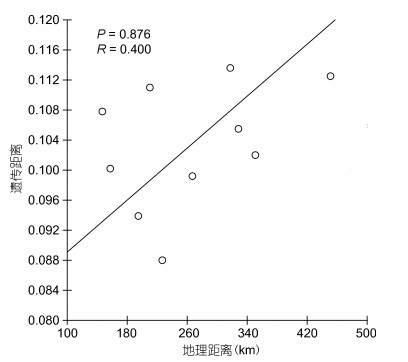 |
| 图 3 单环刺螠群体间遗传距离和地理距离的相关性(y=0.0805+8.647×10–4x, R2= 0.160, P = 0.876) Fig. 3 Relationship between geographical and genetic distances in U. unicinctus populations by IBD analysis |
微卫星标记在水产动物遗传分析和育种实践中被广泛使用, 但有关单环刺螠微卫星标记的开发及应用尚未见报道。本研究采用RAD测序法获得微卫星标记, 这种方法与传统的磁珠富集法相比, 不仅节约实验成本, 而且可以快速精确的定位出大量微卫星序列。本研究中得到1287个微卫星序列, 并从中挑选了重复单元、重复次数都较多的微卫星序列进行引物设计。选用此类微卫星核心序列, 能避免筛选出过多低多态性微卫星位点(韩承慧等, 2016)。本研究中获得的微卫星位点均为完美型(Perfect), 而未得到非完美型(Imperfect)和复合型(Compound)微卫星片段, 原因可能是RAD测序法进行酶切时破坏了后两种微卫星片段, 此现象在菲律宾蛤仔(Ruditapes philippinarum)的微卫星开发中也有报道(闫路路等, 2015)。而在长臀鮠(Cranoglanis bouderius)(孔杰等, 2016)、虾夷扇贝(Patinopecten yessoensis)(赵莹莹等, 2006)的磁珠富集法微卫星开发中则得到了更丰富的微卫星类型。主要原因可能是获取微卫星位点的方法存在明显差异。
多态信息含量(PIC)是衡量基因座位在群体多态性的指标, 当PIC>0.50时, 为高度多态(Ortí et al, 1997), 本研究中22个多态位点(平均PIC为0.921-0.967) 全部表现为高多态性。群体的等位基因数(N)及遗传杂合度(H)是评估和度量群体遗传多样性程度的重要参数。依据微卫星选择标准(Barker, 1994), 当等位基因数(Na)>4个时能较好的反映群体遗传变异水平, 本研究中5个群体中检测的平均Na为9.18-10.64, 平均Ne为6.63-8.85, 分析其等位基因保存较好的原因, 主要是单环刺螠生存环境适宜且受到的人为破坏较少。Leberg(2002)研究发现期望杂合度(He)比观测杂合度(Ho)更能准确衡量群体多样性大小, 本研究中5个地理群体的平均Ho为0.7903-0.9118, 平均He为0.8510-0.8962,与 王蕾等(2010)调查的渤海牙鲆(Paralichthys olivaceus)群体多样性(Ho=0.532-0.895; He=0.635-0.902) 相比, He值相对更大, 表明黄渤海单环刺螠在自然海区仍保持较高的遗传多样性。在所筛选出的22个微卫星标记中, 有12个位点在不同群体中出现了偏离Hardy-Weinberg平衡的现象, 可能原因是单环刺螠活动能力不强, 营定居式生活, 可能存在近亲交配, 导致群体内杂合子过剩或缺失; 也可能是实验选用样本数较少, 对位点平衡有一定的影响, 对长牡蛎(Crassostrea gigas) (Yan et al, 2015)、美洲帘蛤(Mercenaria mercenaria) (Yu et al, 2010)等固着型和埋栖型贝类利用微卫星标记进行遗传分析时也存在微偏离Hardy-Weinberg平衡的现象。
本研究利用基因遗传分化指数FST来评价群体遗传分化程度。按照Balloux等(2002)的评判标准, FST在0.00-0.05时代表群体间分化程度弱; 0.05-0.15代表分化程度中等; 0.15-0.25代表分化程度较大; 0.25-1.00代表分化程度极大。本研究5个地理群体间的遗传分化指数水平为0.0880-0.1136, 说明群体分化水平并不高, 遗传变异主要来自群体内的个体之间, 与杨智鹏等(2015)报道的我国沿海脉红螺(Rapana venosa) 9个群体间遗传分化特性(FST为0.0122-0.0936) 得到类似的结果。本研究结果表明, YT和DL群体遗传距离最大(0.3217), YT和QD遗传距离最小(0.2170), IBD检验结果显示单环刺螠群体不符合距离隔离模式, 即群体间的遗传分化未随地理距离的增加而增加, 说明群体间可能存在较强的基因流, 原因可能是黄、渤海海区海流增加了地理群体间基因流的机会性, 此结果与倪乐海(2011)对我国北方中国蛤蜊9个地理群体的遗传多样性与遗传结构分析一致。对其他海洋生物如多鳞鱚(Sillago sihama)(郭昱嵩等, 2013)、近江牡蛎(Crassostrea ariakensis)(Xiao et al, 2010)等也有过IBD模式的报道。同时Melchinger等(1990)认为在遗传距离小于0.54时, 亲本间的遗传距离越大, 杂交子代的基因杂合度越高, 杂种优势越强。本研究遗传距离范围在0.2170-0.3217, 均满足以上条件。在未来进行单环刺螠人工选育时选取遗传距离较远且性状优良的群体间进行杂交, 可能选育出优良单环刺螠新品种。
4 结论本研究开发的22对高多态性微卫星标记可以用于单环刺螠微卫星多态性、群体多样性、遗传学分析和种群进化等研究, 从分子标记水平表明秦皇岛、烟台、潍坊、青岛和大连5个野生地理群体的杂合度较高, 遗传多样性丰富, 种群遗传结构相对稳定。单环刺螠种群在黄、渤海海区多样性水平虽然较高, 但应及时采取措施加强单环刺螠种质资源保护与管理, 以缓解或避免由于过度滥捕造成自然单环刺螠种群遗传多样性的丢失。同时本研究也为下一步良种人工选育工作提供指导。
| 王雷, 刘海梅, 彭鸽, 等, 2011. 单环刺螠营养成分及体内活性物质的研究进展. 鲁东大学学报(自然科学版), 27(4): 342–345 |
| 王蕾, 张立冬, 万玉美, 等, 2010. 牙鲆微卫星标记的筛选及群体遗传结构分析. 遗传, 32(10): 1057–1064 |
| 孔杰, 蒋晓红, 周洲, 等, 2016. 磁珠富集法开发长臀鮠微卫星分子标记. 贵州农业科学, 44(7): 14–17 |
| 闫路路, 秦艳杰, 闫喜武, 等, 2015. 基于转录组平台的蛤仔微卫星标记筛选. 生态学报, 35(5): 1573–1580 |
| 孙孝德, 2011. 刺参(Apostichopus japonicus)微卫星标记的筛选及群体分析. 上海: 上海海洋大学硕士学位论文, 21-28 |
| 杨智鹏, 于红, 于瑞海, 等, 2015. 中国沿海脉红螺群体遗传多样性及其遗传结构. 水产学报, 39(10): 1443–1449 |
| 赵莹莹, 朱晓琛, 孙效文, 等, 2006. 磁珠富集法筛选虾夷扇贝微卫星序列. 中国水产科学, 13(5): 749–755 |
| 倪乐海, 2011. 中国蛤蜊群体遗传结构与蛤蜊科贝类系统发育研究. 青岛: 中国海洋大学硕士学位论文, 42-52 |
| 郭昱嵩, 颜程钟, 张宇岚, 等, 2013. 多鳞鱚(Sillago sihama)4个野生地理群体的微卫星标记分析. 海洋与湖沼, 44(2): 267–276 DOI:10.11693/hyhz201302001001 |
| 韩承慧, 马海涛, 姜海滨, 等, 2016. 许氏平鲉(Sebastes schlegeli)微卫星标记开发及野生、养殖群体遗传多样性分析. 海洋与湖沼, 47(1): 213–220 |
| Balloux F, Lugon-Moulin N, 2002. The estimation of population differentiation with microsatellite markers. Molecular Ecology, 11(2): 155–165 DOI:10.1046/j.0962-1083.2001.01436.x |
| Barker J S F, 1994. Sequential gel electrophoretic analysis of esterase-2 in two populations of Drosophila buzzatii. Genetica, 92(3): 165–175 DOI:10.1007/BF00132535 |
| Feng L Y, Hu L P, Fu X T, et al, 2014. An integrated genetic and cytogenetic map for Zhikong scallop, Chlamys farreri, based on microsatellite markers. PLoS One, 9(4): e92567 DOI:10.1371/journal.pone.0092567 |
| Jo H Y, Jung W K, Kim S K, 2008. Purification and characterization of a novel anticoagulant peptide from marine echiuroid worm, Urechis unicinctus. Process Biochemistry, 43(2): 179–184 DOI:10.1016/j.procbio.2007.11.011 |
| Kang K H, Zhang Z F, Min K J, et al, 2010. Effects of Urechis unicinctus juveniles on chemical characteristics of organically contaminated coastal sediment. Journal of Ocean University of China, 9(1): 48–52 DOI:10.1007/s11802-010-0048-2 |
| Leberg P L, 2002. Estimating allelic richness:effects of sample size and bottlenecks. Molecular Ecology, 11(11): 2445–2449 |
| Melchinger A E, Lee M, Lamkey K R, et al, 1990. Genetic diversity for restriction fragment length polymorphisms:relation to estimated genetic effects in maize inbreds. Crop Science, 30(5): 1033–1040 DOI:10.2135/cropsci1990.0011183X003000050016x |
| Miller P A, Elliott N G, Koutoulis A, et al, 2015. Genetic diversity of cultured, naturalized, and native pacific oysters, Crassostrea gigas, determined from multiplexed microsatellite markers. Journal of Shellfish Research, 31(3): 611–617 |
| Mohanty P, Sahoo L, Pillai B R, et al, 2014. Genetic divergence in Indian populations of M. rosenbergii using microsatellite markers. Aquaculture Research, 47(2): 472–481 |
| Ortí G, Pearse D E, Avise J C, 1997. Phylogenetic assessment of length variation at a microsatellite locus. Proceedings of the National Academy of Sciences of the United States of America, 94(20): 10745–10749 DOI:10.1073/pnas.94.20.10745 |
| Wang D L, Jiang H Z, Wang R L, et al, 2010. Purification and characterization of a novel fibrinolytic enzyme from a marine animal, Urechis unicinctus. China Biotechnology, 30(8): 42–51 |
| Weber J L, 1990. Informativeness of human (dC-dA)n·(dG-dT)n polymorphisms. Genomics, 7(4): 524–530 DOI:10.1016/0888-7543(90)90195-Z |
| Xiao J, Cordes J F, Wang H Y, et al, 2010. Population genetics of Crassostrea ariakensis in Asia inferred from microsatellite markers. Marine Biology, 157(8): 1767–1781 DOI:10.1007/s00227-010-1449-x |
| Yan W, Wang A M, Guo X M, 2015. Development and characterization of polymorphic microsatellite markers for the northern quahog Mercenaria mercenaria (Linnaeus.. 1758). Journal of Shellfish Research, 29(1): 77–82 |
| Yu Z N, Wang Y H, Fu D K, 2010. Development of fifty-one novel EST-SSR loci in the Pacific oyster, Crassostrea gigas by data mining from the public EST database. Conservation Genetics Resources, 2(S1): 13–18 DOI:10.1007/s12686-009-9127-3 |
 2017, Vol. 48
2017, Vol. 48