中国海洋湖沼学会主办。
文章信息
- 巩超彦, 南芳茹, 冯佳, 吕俊平, 刘琪, 谢树莲. 2017.
- GONG Chao-Yan, NAN Fang-Ru, FENG Jia, LÜ Jun-Ping, LIU Qi, XIE Shu-Lian. 2017.
- 串珠藻目植物rbcL基因的适应性进化分析
- ADAPTIVE EVOLUTIONARY ANALYSIS ON rbcL GENE OF BATRACHOSPERMALES
- 海洋与湖沼, 48(3): 527-535
- Oceanologia et Limnologia Sinica, 48(3): 527-535.
- http://dx.doi.org/10.11693/hyhz20161200277
-
文章历史
- 收稿日期:2016-12-14
- 收修改稿日期:2017-02-08
绝大多数红藻为海产, 只有少数生于淡水。这些淡水红藻是海陆演变过程中残留在淡水中的孑遗生物, 多生长在清洁、温度偏低、环境比较稳定的水体中, 其分布区狭窄, 且具有一定的封闭性, 因此, 在对环境的长期适应过程中形成了一些珍稀特有种类(梅洪等, 2004)。淡水红藻是植物进化中的一个重要类群, 串珠藻目(Batrachospermales)是其中最主要的代表类群(施之新, 2006)。
核酮糖1, 5二磷酸羧化酶/加氧酶(Rubisco)是植物中含量最丰富的蛋白质, 约占植物可溶性蛋白含量的50%以上(Kapralov et al, 2007), 这也是对其相当低的催化效率的补偿(Spreitzer et al, 2002)。该酶在植物光合作用中具有双重功能, 一是催化核酮糖-1, 5二磷酸(RUBP)的羧化反应, CO2与RUBP共价结合生成不稳定的六碳化合物, 分裂出2分子3-磷酸甘油酸, 二是在光呼吸中催化RUBP的加氧反应, O2与RUBP反应生成1分子3-磷酸甘油酸和1分子2-磷酸乙醇酸(Portis, 1992; Gutteridge et al, 1995)。全酶由8个大亚基和8个小亚基组成, 其活性中心位于Rubisco大亚基, 特别是大亚基的C末端区域在功能上作用重要, 由叶绿体rbcL基因编码(Hartman et al, 1994)。以往的一些研究表明, 不同物种Rubisco的催化效率有差异(Jordan et al, 1981), 这种差异的原因主要是自然环境的选择压力, 而不是系统发生(Galmés et al, 2005)。
分析具有重要功能的蛋白质的适应性进化有助于深刻了解基因的变异和蛋白结构与功能的改变及物种的进化史(Nei et al, 2000)。在适应性进化的分析研究中, 可以通过度量核苷酸序列的非同义替换率(dN)和同义替换率(dS)的比值(ω)来判断自然选择对氨基酸位点的选择压力(Yang, 2006)。同义替换指不导致氨基酸变异的核苷酸突变, 一般发生在密码子的第三位, 非同义替换指导致氨基酸变异的核苷酸突变, 一般发生在密码子的第一、二位。当dN=dS, 即ω=1, 表示非同义突变和同义突变的固定速率相同, 则认为选择对适合度没有影响; 当dN < dS, 即ω < 1, 认为非同义突变有害, 那么进化选择就会降低这些突变的固定速率; 当dN > dS, 即ω > 1, 认为发生的非同义突变有利于选择, 那么它们会以比同义突变更快的速率被固定。所以, ω > 1的显著性可被当做蛋白质发生适应性进化的证据。Nielsen等(1998)、Yang等(2000b)和Wong等(2004)针对编码蛋白质的DNA序列, 先后建立、改进了允许不同氨基酸位点取不同ω值的一系列似然进化模型。其中, Yang(2007)开发了PAML(phylogenetic analysis by maximum likelihood)软件, 该软件是一个用最大似然法来对DNA和蛋白质序列进行系统发育分析的综合程序包, 其中的Codeml程序可以估算编码蛋白质的DNA序列的同义和非同义替代率并发现DNA序列中的正选择位点, 即对DNA序列进行适应性进化分析。经由这些模型验证存在正向选择的位点后, 可进一步通过贝叶斯途径将发生正向选择的位点加以鉴定。
鉴于Rubisco大亚基的重要功能, 本文对串珠藻目的rbcL基因进行了适应性进化分析, 目的在于探讨自然选择对串珠藻目植物Rubisco大亚基的选择作用, 即在其进化过程中Rubisco大亚基是否经历过正选择及正选择发生的氨基酸。
1 材料与方法 1.1 序列数据采集及系统发育树构建本研究所用数据包括作者采集样本提取和从GenBank收集的rbcL基因共39条(表 1)。用Clustal X软件(Thompson et al, 1997)进行序列对位排列, 经人工检查校对, 得到每条序列均由381个密码子组成的序列数据供系统发育分析。
|
利用MEGA5.0(Tamura, 2004)分析序列特征, 运行Modeltest软件(Posada et al, 2004), 筛选核苷酸最优进化模型。采用最大似然法(ML)(Rannala et al, 1996)运行PhyML3.0(Guindon et al, 2010)构建系统发育树。
1.2 统计分析利用最大似然法得到ML树, 运行PAML4.8软件包(Yang, 2007)中的Codeml程序中的分支模型、位点模型、分支-位点模型进行分析。
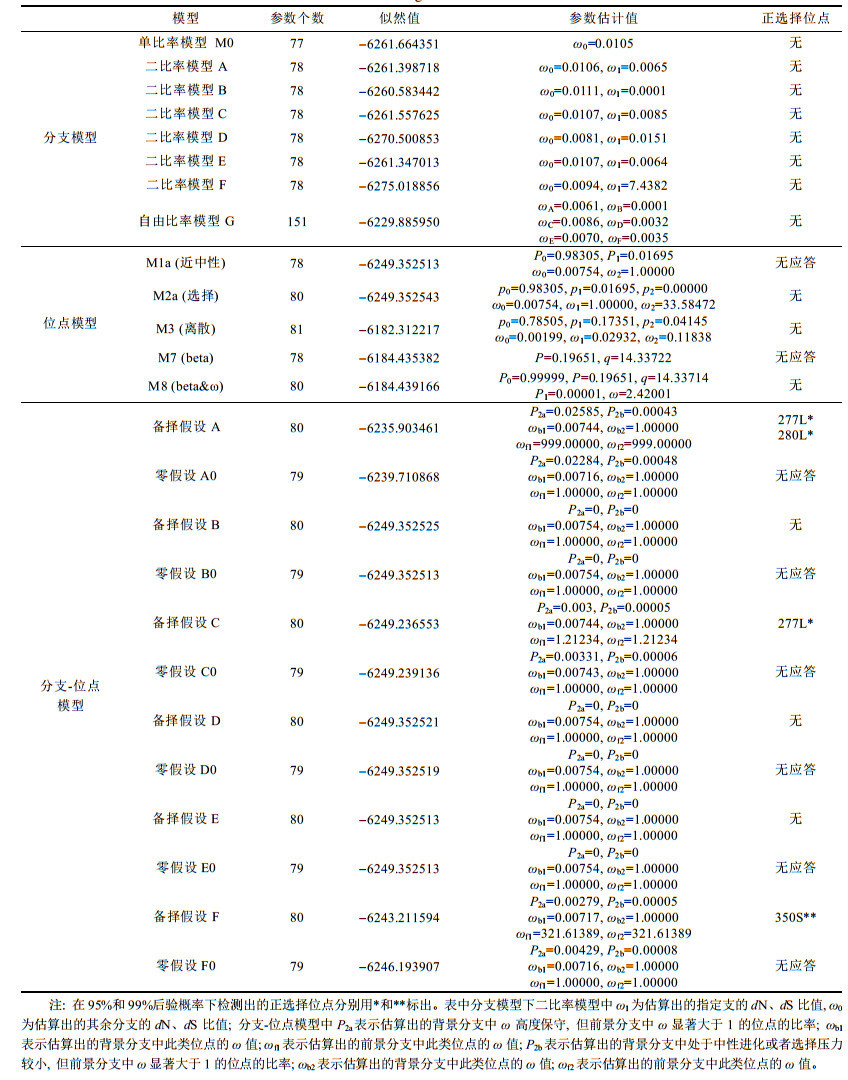
在分支模型(Yang, 1998)中, 假定不同分支有不同的ω值, 以检测各分支的选择压力。单比率(one ratio)模型是最简单的模型, 该模型假定进化树上所有分支的比率相同, 为ω0。自由比率(free ratio)模型是最全面的模型, 该模型假定不同的分支有不同的比率。在这两种模型之间本研究还运用了二比率模型。
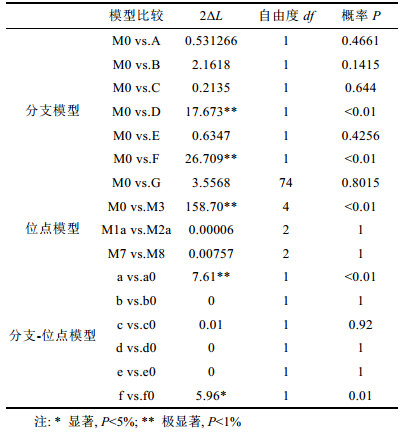
位点模型(Yang et al, 2000a)是用于检验rbcL基因是否存在经受正选择(ω > 1) 和负选择(ω < 1) 的位点。在位点模型中, 允许不同位点有不同的选择压力, 而在系统树的不同分支之间则无差异。本研究采用了3对模型, 即M1a(近中性)对M2a(选择), M0(单一比值)对M3(离散), M7(beta)对M8(beta&ω)(Nielsen et al, 1998; Yang et al, 2000b), 后者均为备择假设, 前者是后者的零假设。对3对模型进行LRT检验(likelihood ratio testes), 是通过在相对的自由度(两模型参数之差)下, 二倍对数似然值之差的绝对值的χ2检验来确定备择模型是否可靠(Yang et al, 2000a)。
分支-位点模型(Hong et al, 1997)用于检验指定分支中是否存在正选择位点。将系统发育树分为目标前景支和背景支(除了目标前景支之外的分支)。分支-位点模型的LRT检验, 根据文献(Yang, 2007)选择了test2模型, 将MA和无效模型(ω设置为1) 进行比较。
1.3 三维结构建模选取代表性物种胶串珠藻Batrachospermum gelatinosum (串珠藻目, 串珠藻科, 串珠藻属的模式种, 登录号为AF029141) 的rbcL序列为参考序列, 翻译为氨基酸序列, 提交瑞士生物信息研究所(European Bioinformatics Institute: http://www.isb-sib.cn/) Swiss-model, 基于同源建模原理预测Rubisco大亚基的三维结构(周媛, 2011)。
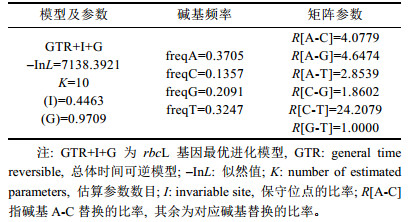
2 结果与分析 2.1 系统发育分析基于选取的rbcL基因序列, 通过Modeltest软件筛选得到核苷酸最优进化模型为GRT+I+G(假设核苷酸位点替换具有可逆性, G代表gamma分布的形状参数, I代表不变位点的比例), 模型参数见(表 2)基于此模型构建了系统发育树, 使用的构树方法为最大似然法(ML)(图 1), 以Galdieria maxima和Cyanidium caldarium为外类群。从系统树可以看出, 除了外类群, 所有内类群聚集为6个分支。其中, 分支A为暗紫红毛菜(Bangia atropurpurea)的4个品系, 其他5个分支聚为1个大的分支, 包括所有串珠藻目的种类。分支B为6株胶串珠藻(Batrachospermum gelatinosum), 后验概率为100%, 显示它们之间具有密切的亲缘关系。分支C为7株扁圆串珠藻(Batrachospermum helminthosum), 后验概率同样为100%。分支D为熊野藻属(Kumanoa)的6个种, 后验概率为99.2%, 与此前的有关报道一致(南芳茹等, 2015)。分支E为4种共6株连珠藻属(Sirodotia)植物, 后验概率为97.5%。分支F为6株弧形西斯藻(Sheathia arcuata), 后验概率为100%。据此, 选定这6个分支供后面的适应性进化分析。
|
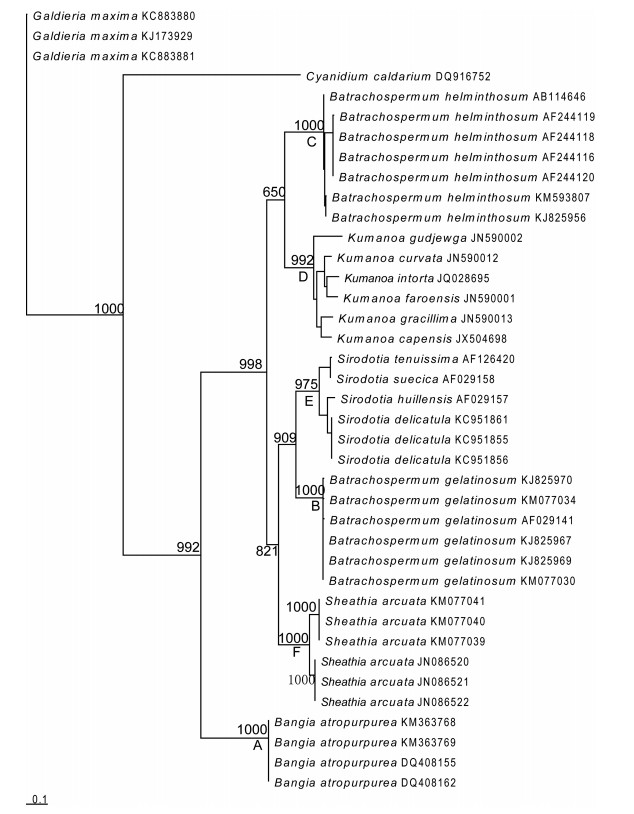 |
| 图 1 基于rbcL基因序列构建的系统发育树 Fig. 1 Phylogenetic tree established based on the rbcL gene sequence 注:节点处的数字代表最大似然法歩靴值, A、B、C、D、E、F代表选定的分支 |
分支模型中自由比率模型显示, 分支A、B、C、D、E、F的ω值均小于1, 意味着各分支均处于负选择下。但是, 经过与单比率模型进行LRT检验, 表明此模型并不可靠。二比率模型中, 指定分支A、B、C、D、E、F为前景枝, 其余为背景枝, 估算各前景枝的ω值。结果显示, 只有F分支的ω > 1, ωF=7.4382, 显示这一分支中可能有正选择位点的存在, LRT检验也支持这点。
在位点模型中, 模型M2a(选择)、模型M3(离散)、模型M8(beta&ω)均允许ω值大于1。经LRT检验, M2a和M8模型均没有对应的可靠零假设, M3模型则显著优于其对应的零假设。但是, 比较模型并非用来检测正选择位点, 而是检验各位点是否取不同的ω值。本研究显示各位点的ω值是不同的, 在位点模型下均没有检测出正选择位点, 表现出rbcL基因受到强烈的负选择。
分支-位点模型是研究指定分支相较于其它分支是否存在适应性进化。本文6个分支中, 结果显示只有分支A(Bangia atropurpurea)的277L和280L, 分支F(Sheathia arcuata)的350S被鉴定为正选择位点。LRT检验也肯定了其存在。另外, 分支C(Batrachospermum helminthosum)检测出了277L为正选择位点, 但经LRT检验, 认为备择假设不可靠, 拒绝其存在。分支B、D、F均不比单比率模型更合适。
2.3 正选择位点定位以胶串珠藻Batrachospermum gelatinosum (串珠藻目, 串珠藻科, 串珠藻属的模式种, 登录号为AF029141) 的rbcL序列为参考序列, 基于同源建模原理预测出Rubisco大亚基的参考三维结构(图 2)。经过对PDB数据库进行Blast搜索模板, 得到一种喜温红藻Geldieria partita的Rubisco三维结构(PDB ID: 1BWV)(Sugawara et al, 1999), 相似度83.64%, 符合同源建模的可靠性要求。
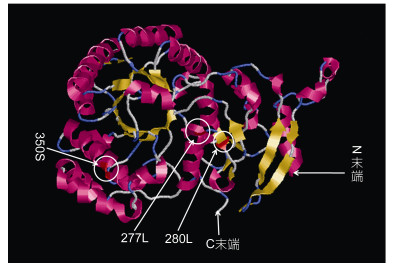 |
| 图 2 胶串珠藻(AF029141) Rubisco大亚基参考三维结构 Fig. 2 Reference three -dimensional structure of Rubisco large subunits of Batrachospermum gelatinosum (AF029141) 注:正选择位点在图中用白色圆圈标出 |
将用于建模的序列AF029141、程序分析序列PAML(379个氨基酸位点)和模板Rubisco大亚基对应氨基酸序列用Bioedit软件进行比对, 确定了被检测出的正选择位点的相对位置(图 2)。运用Raswin软件(Sayle et al, 1995)将正选择位点标定在构建出的Rubisco大亚基的参考三维模型中。如图 3所示, 被选出的正选择位点277L、280L位于Rubisco大亚基羧基末端保守的8个α螺旋和8个β片层构成的α/β桶状结构域中第7个α螺旋和第7个β片层之间的loop结构上, 350S位于羧基末端邻近α/β桶状结构域的一个α螺旋上。
 |
| 图 3 Rubisco大亚基氨基酸序列对位排列 Fig. 3 Alignment of the amino acid sequence of Rubisco 注:对应氨基酸下方箭头表示对应的Rubisco的二级结构。图中字母第一行代表PAML序列, 第二行代表模板序列, 第三行代表本文建模序列 |
被选出的正选择位点277L、280L位于Rubisco大亚基羧基末端活性中心区域, 350S位于羧基末端邻近α/β桶状结构域的一个α螺旋上。已有研究表明Rubisco的一个大亚基和一个小亚基组成的二聚体是该酶最小的功能单位, 小亚基氨基端的功能结构域由5个β折叠和相邻的2个α螺旋组成(Farber et al, 1990), 它们和大亚基羧基端的由8个α螺旋和8个β折叠组成功能结构域组成该酶的功能活性中心(Soper et al, 1988; Knight et al, 1990)。这都提示这些位点可能对Rubisco功能有影响, 也为今后研究Rubisco的功能提供了优先选择位点。
本研究中分支-位点模型下, 分支C检测出了277L为正选择位点, 但经LRT检验, 备择假设不可靠, 拒绝了正选择位点的存在; 分支模型下检测出分支D可能有正选择位点, 但在分支-位点模型中没有检测出该支有正选择位点。Suzuki等(2001, 2002)曾指出似然法分析可能得出假阳性结果, Wong等(2004)基于模拟研究对似然法的可信度和统计效力也提出了质疑, Zhang(2004)曾怀疑PAML中分支-位点模型易产生假阳性(Yang et al, 2002)。之后, Zhang等(2005)对该模型进行了改进, 很好地解决了假阳性问题。基于已有文献, 作者推测造成该结果的原因不是由于假阳性的问题, 而可能是由于选择压力放松造成的。同时, 这些结果也充分显示了基于ω比值检验DNA编码序列分子适应的可靠性和有效性。
位点模型下未能检测出正选择位点, 可能的原因是rbcL基因具有十分重要的功能, 需要保持相当的稳定性, 因此比较保守, 也有可能该基因适应性进化发生在较早期, 以至后来适应性进化信号被极其普遍的中性选择或者净化选择所淹没。相关研究也指出, 在藻类植物的Rubisco中几乎没有发现正选择位点的存在, 而在陆生植物中却广泛存在, 该酶结构上的差异不足以解释这个现象(Kapralov et al, 2007), 而可能的一个重要原因是它们生境的不同。藻类植物生长于稳定的水生环境, 具有特有的CO2浓缩机制, 即碳酸氢盐泵, 该机制利用溶解在水中的碳酸氢盐并且抑制Rubisco的补氧活性, 使得Rubisco执行功能的气态环境相当稳定(Lambers et al, 2008)。
分支模型下检测出了分支F可能有正选择位点, 分支-位点模型也同时检测出了这一支确实有正选择位点。分支A在分支模型下显示没有正选择位点, 在分支-位点模型中却显示有两个正选择位点(277L和280L), 但它们的后验概率不高, 分别为73.4%和64%, 可能是分支模型未能检测出正选择的原因。另外, 分支A是暗紫红毛菜, 分支F是弧形西斯藻, 两种都是淡水红藻中分布范围较广泛的种, 说明其对环境有较好的适应, 这可能是在它们中检测出正选择位点的一个原因。
以往在研究酶的重要功能位点时往往采用生物化学方法对目标位点进行突变筛选以及突变体回复试验(Hong et al, 1997; Du et al, 2003)。例如, 有研究鉴定出凤尾蕨科旱生植物rbcL基因的365F为正选择位点(周媛等, 2011), 该位点位于βH折叠区, 苯丙氨酸可能与βH折叠中的其他氨基酸形成氢键, 该区域的空间变化可能与植物对生境的适应有关(刘念等, 2010)。而365F位点与本研究鉴定出的位点相近。此外有实验表明, Rubisco α/β桶状结构域中的loop6对该酶的催化作用、决定羧化作用和氧化作用的比值至关重要(Chen et al, 1989, 1991)。本研究鉴定出的277L和280L, 虽然不位于loop6但位于loop7, 由此可见, 本研究鉴定出的位点可能对酶的功能的研究产生积极意义。这些正选择位点仍有待更深入的研究, 今后的定向诱变研究和遗传相关试验也可以优先关注这些位点。
适应性突变的发生虽然是偶然的, 但突变位点的正选择作用则是自然环境的选择, 环境条件必然对酶的功能及活性产生影响。淡水红藻是海陆演变过程中残留在淡水中的孑遗生物, 在世界各地的淡水中分布稀少且分布区狭窄。海洋生境同淡水生境差异巨大, 淡水红藻对生境的要求较高, 一般为弱光照下洁净、清冷、高溶氧的流动水体(施之新, 2006)。因此, 淡水红藻rbcL基因的进化压力来源可能就是这些环境因素, rbcL的适应性进化使其更加适应淡水环境。
4 结论本文利用PAML4.8软件, 运行位点模型、分支模型及分支-位点模型, 对选取淡水红藻类群的rbcL基因进行适应性进化分析, 得到如下结论:
(1) 通过最大似然法构建的系统发育树显示, 所有内类群聚集为6个分支, 其中, 分支A为暗紫红毛菜, 分支B为胶串珠藻, 分支C为扁圆串珠藻, 后验概率同样为100%, 分支D为熊野藻属, 分支E为连珠藻属, 分支F为弧形西斯藻;
(2) 分支-位点模型中, 在3个分支中分别鉴定出350S、277L和280L为正选择位点;
(3) 在构建出的Rubisco大亚基的参考三维模型中, 277L和280L位于Rubisco大亚基羧基末端保守的8个α螺旋和8个β片层构成的α/β桶状结构域中第7个α螺旋和第7个β片层之间的loop结构上, 350S位于羧基末端邻近α/β桶状结构域的一个α螺旋上。
(4) 研究结果显示了基于ω比值检验基因适应性进化的准确性和有效性, 同时也揭示了串珠藻目植物rbcL基因确实发生了适应性进化, 对串珠藻目适应特殊生存环境产生了有益的作用。
| 刘念, 王庆彪, 陈婕, 等, 2010. 麻黄属rbcL基因的适应性进化检测与结构模建. 科学通报, 55(14): 1341–1346 |
| 周媛, 王博, 高磊, 等, 2011. 凤尾蕨科旱生蕨类rbcL基因的适应性进化和共进化分析. 植物科学学报, 29(4): 409–416 |
| 南芳茹, 冯佳, 谢树莲, 2015. 基于叶绿体psaA和psbA基因的中国熊野藻属植物系统发育分析. 水生生物学报, 39(1): 155–163 DOI:10.7541/2015.20 |
| 施之新, 2006. 中国淡水藻志(第十三卷)——红藻门和褐藻门. 北京: 科学出版社, |
| 梅洪, 赵先富, 郭斌, 等, 2004. 中国淡水藻类生物多样性研究进展. 生态科学, 22(4): 356–359 |
| Chen Z X, Spreitzer R J, 1989. Chloroplast intragenic suppression enhances the low CO2/O2 specificity of mutant ribulose-bisphosphate carboxylase/oxygenase. The Journal of Biological Chemistry, 264(6): 3051–3053 |
| Chen Z X, Yu W Z, Lee J H, et al, 1991. Complementing amino acid substitutions within loop 6 of the α/β-barrel active site influence the carbon CO2/O2 specificity of chloroplast ribulose-1, 5-bisphosphate carboxylase/oxygenase. Biochemistry, 30(36): 8846–8850 DOI:10.1021/bi00100a017 |
| Du Y, Peddi S R, Spreitzer R J, 2003. Assessment of structural and functional divergence far from the large subunit active site of ribulose-1, 5-bisphosphate carboxylase/oxygenase. Journal of Biological Chemistry, 278(49): 49401–49405 DOI:10.1074/jbc.M309993200 |
| Farber G K, Petsko G A, 1990. The evolution of α/β barrel enzymes. Trends in Biochemical Sciences, 15(6): 228–234 DOI:10.1016/0968-0004(90)90035-A |
| Galmés J, Flexas J, Keys A J, et al, 2005. Rubisco specificity factor tends to be larger in plant species from drier habitats and in species with persistent leaves. Plant, Cell & Environment, 28(5): 571–579 |
| Guindon S, Dufayard J F, Lefort V, et al, 2010. New algorithms and methods to estimate maximum-likelihood phylogenies:assessing the performance of PhyML 3.0. Systematic Biology, 59(3): 307–321 DOI:10.1093/sysbio/syq010 |
| Gutteridge S, Gatenby A A, 1995. Rubisco synthesis, assembly, mechanism, and regulation. The Plant Cell, 7(7): 809–819 DOI:10.1105/tpc.7.7.809 |
| Hartman F C, Harpel M R, 1994. Structure, function, regulation, and assembly of D-ribulose-1, 5-bisphosphate carboxylase/oxygenase. Annual Review of Biochemistry, 63(1): 197–232 DOI:10.1146/annurev.bi.63.070194.001213 |
| Hong S, Spreitzer R J, 1997. Complementing substitutions at the bottom of the barrel influence catalysis and stability of ribulose-bisphosphate carboxylase/oxygenase. Journal of Biological Chemistry, 272(17): 11114–11117 DOI:10.1074/jbc.272.17.11114 |
| Jordan D B, Ogren W L, 1981. Species variation in the specificity of ribulose biphosphate carboxylase/oxygenase. Nature, 291(5815): 513–515 DOI:10.1038/291513a0 |
| Kapralov M V, Filatov D A, 2007. Widespread positive selection in the photosynthetic Rubisco enzyme. BMC Evolutionary Biology, 7: 73 DOI:10.1186/1471-2148-7-73 |
| Knight S, Andersson I, Brändén C I, 1990. Crystallographic analysis of ribulose 1, 5-bisphosphate carboxylase from spinach at 2·4Å resolution:subunit interactions and active site. Journal of Molecular Biology, 215(1): 113–160 DOI:10.1016/S0022-2836(05)80100-7 |
| Lambers H, Chapin Ⅲ F S, Pons T L, 2008. Plant Physiological Ecology. 2nd ed. New York:Springer |
| Nei M, Kumar S, 2000. Molecular Evolution and Phylogenetics. Oxford: Oxford University Press, |
| Nielsen R, Yang Z, 1998. Likelihood models for detecting positively selected amino acid sites and applications to the HIV-1 envelope gene. Genetics, 148(3): 929–936 |
| Portis A R, 1992. Regulation of ribulose 1, 5-bisphosphate carboxylase/oxygenase activity. Annual Review of Plant Physiology and Plant Molecular Biology, 43: 415–437 DOI:10.1146/annurev.pp.43.060192.002215 |
| Posada D, Buckley T R, 2004. Model selection and model averaging in phylogenetics:advantages of Akaike information criterion and Bayesian approaches over likelihood ratio tests. Systematic Biology, 53(5): 793–808 DOI:10.1080/10635150490522304 |
| Rannala B, Yang Z H, 1996. Probability distribution of molecular evolutionary trees:a new method of phylogenetic inference. Journal of Molecular Evolution, 43(3): 304–311 DOI:10.1007/BF02338839 |
| Sayle R A, Milner-White E J, 1995. RASMOL:biomolecular graphics for all. Trends in Biochemical Sciences, 20(9): 374–376 DOI:10.1016/S0968-0004(00)89080-5 |
| Soper T S, Mural R J, Larimer F W, et al, 1988. Essentiality of Lys-329 of ribulose-1, 5-bisphosphate carboxylase/oxygenase from Rhodospirillum rubrum as demonstrated by site-directed mutagenesis. Protein Engineering, Design and Selection, 2(1): 39–44 DOI:10.1093/protein/2.1.39 |
| Spreitzer R J, Salvucci M E, 2002. Rubisco:structure, regulatory interactions, and possibilities for a better enzyme. Annual Review of Plant Biology, 53(1): 449–475 DOI:10.1146/annurev.arplant.53.100301.135233 |
| Sugawara H, Yamamoto H, Shibata N, et al, 1999. Crystal structure of carboxylase reaction-oriented ribulose 1, 5-bisphosphate carboxylase/oxygenase from a thermophilic red alga, Galdieria partita. Journal of Biological Chemistry, 274(22): 15655–15661 DOI:10.1074/jbc.274.22.15655 |
| Suzuki Y, Nei M, 2001. Reliabilities of parsimony-based and likelihood-based methods for detecting positive selection at single amino acid sites. Molecular Biology and Evolution, 18(12): 2179–2185 DOI:10.1093/oxfordjournals.molbev.a003764 |
| Suzuki Y, Nei M, 2002. Simulation study of the reliability and robustness of the statistical methods for detecting positive selection at single amino acid sites. Molecular Biology and Evolution, 19(11): 1865–1869 DOI:10.1093/oxfordjournals.molbev.a004010 |
| Tamura K, Peterson D, Peterson N, et al, 2011. MEGA5:molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular Biology and Evolution, 28(10): 2731–2739 DOI:10.1093/molbev/msr121 |
| Thompson J D, Gibson T J, Plewniak F, et al, 1997. The CLUSTAL_X windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools. Nucleic Acids Research, 25(24): 4876–4882 DOI:10.1093/nar/25.24.4876 |
| Wong W S W, Yang Z, Goldman N, et al, 2004. Accuracy and power of statistical methods for detecting adaptive evolution in protein coding sequences and for identifying positively selected sites. Genetics, 168(2): 1041–1051 DOI:10.1534/genetics.104.031153 |
| Yang Z, 1998. Likelihood ratio tests for detecting positive selection and application to primate lysozyme evolution. Molecular Biology and Evolution, 15(5): 568–573 DOI:10.1093/oxfordjournals.molbev.a025957 |
| Yang Z H, Swanson W J, Vacquier V D, 2000a. Maximum-likelihood analysis of molecular adaptation in abalone sperm lysin reveals variable selective pressures among lineages and sites. Molecular Biology and Evolution, 17(10): 1446–1455 DOI:10.1093/oxfordjournals.molbev.a026245 |
| Yang Z H, Bielawski J P, 2000b. Statistical methods for detecting molecular adaptation. Trends in Ecology & Evolution, 15(12): 496–503 |
| Yang Z H, Nielsen R, 2002. Codon-substitution models for detecting molecular adaptation at individual sites along specific lineages. Molecular Biology and Evolution, 19(6): 908–917 DOI:10.1093/oxfordjournals.molbev.a004148 |
| Yang Z H, 2006. Computational Molecular Evolution. Oxford: Oxford University Press, |
| Yang Z H, 2007. PAML 4:phylogenetic analysis by maximum likelihood. Molecular Biology and Evolution, 24(8): 1586–1591 DOI:10.1093/molbev/msm088 |
| Zhang J Z, 2004. Frequent false detection of positive selection by the likelihood method with branch-site models. Molecular Biology and Evolution, 21(7): 1332–1339 DOI:10.1093/molbev/msh117 |
| Zhang J Z, Nielsen R, Yang Z H, 2005. Evaluation of an improved branch-site likelihood method for detecting positive selection at the molecular level. Molecular Biology and Evolution, 22(12): 2472–2479 DOI:10.1093/molbev/msi237 |
 2017, Vol. 48
2017, Vol. 48