中国海洋湖沼学会主办。
文章信息
- 黄郁葱, 丁燏, 梁秀全, 蔡双虎, 吴灶和, 简纪常. 2017.
- HUANG Yu-Cong, DING Yu, LIANG Xiu-Quan, CAI Shuang-Hu, WU Zao-He, JIAN Ji-Chang. 2017.
- 红笛鲷(Lutjanus sanguineus)CD4和CD4-2基因克隆与诱导表达
- MOLECULAR CLONING AND INDUCED EXPRESSION OF CD4 AND CD4-2 GENES FROM HUMPHEAD SNAPPER LUTJANUS SANGUINEUS
- 海洋与湖沼, 48(3): 589-599
- Oceanologia et Limnologia Sinica, 48(3): 589-599.
- http://dx.doi.org/10.11693/hyhz20161200276
-
文章历史
- 收稿日期:2016-12-14
- 收修改稿日期:2017-01-23
2. 广东省水产经济动物病原生物学及流行病学重点实验室 湛江 524088;
3. 仲恺农业工程学院 广州 510225;
4. 广东省湛江市霞山区农业技术推广中心 湛江 524000
2. Guangdong Provincial Key Laboratory of Pathogenic Biology and Epidemiology for Aquatic Economic Animals, Zhanjiang 524088, China;
3. Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;
4. Agricultural Technology Promotion Center of Xiashan District, Zhanjiang 524000, China
CD4分子是T淋巴细胞重要的表面标志分子之一, 作为T细胞辅助受体参与抗原识别和信号转导, 同时还具有调节免疫应答的功能, 在介导机体的适应性免疫应答中起重要作用。哺乳类CD4分子为单链跨膜糖蛋白, 属于免疫球蛋白超家族成员, 主要表达于Th细胞、调节性T细胞、胸腺细胞及某些B细胞、EB病毒转化的B细胞、单核/巨噬细胞表面、树突状细胞和脑细胞膜(Parnes, 1989)。哺乳类CD4由信号肽、胞外区、跨膜区和胞浆区组成, 胞外区包含4个IgSF结构域(D1—D4), 其中D1和D3结构域为V样结构域, D2和D4为C样结构域。在抗原识别过程中, CD4分子的D1和D2结构域可与MHC Ⅱ类分子非多肽区结合(Parnes, 1989), 有助于增强TCR与抗原肽-MHCⅡ复合物之间的相互作用并辅助TCR识别抗原。胞内区的CXCP基序是Src家族成员酪氨酸蛋白激酶p56lck的结合部位, 参与T细胞活化、增殖和信号转导(Smith-Garvin et al, 2009)。CD4分子也是人类免疫缺陷病毒(Human Immunodeficiency Virus, HIV)受体, 其D1结构域可与HIV gp120结合。此外, CD4还是T细胞IL-16的受体, 可调节CD4+T细胞的增殖和趋化(Lynch et al, 2003)。
2004年, Suetake等(2004)首先报道了红鳍东方鲀(Takifugu rubripes)的CD4分子, 继而多种鱼类的CD4分子先后得到克隆鉴定(Laing et al, 2006; Buonocore et al, 2008; Patel et al, 2009; Kato et al, 2013)。研究发现此类鱼类CD4分子胞外区大多含有典型的4个Ig样结构域。此外, 在还发现另一种鱼类特有的CD4分子, 该CD4分子胞外区仅含两个或三个Ig样结构域, 命名为CD4REL或CD4-2 (Dijkstra et al, 2006; Laing et al, 2006; Edholm et al, 2007; vergrd et al, 2010)。CD4-2分子与CD4分子在染色体上呈线性排列, 分布在GAPDH、LPREL2和TAPBP-R之间。CD4分子和CD4-2分子在结构上有很多的相似之处, 如CD4和CD4-2分子胞内区虽然都含有与p56lck结合基序, 但鱼类CD4和CD4-2分子在一些重要结构和组织表达模式存在差异, 因此两者的功能可能并不相同。此外, 有关CD4和CD4-2在抗菌免疫中的作用及其免疫调节功能仍有待进一步研究加以阐明。
红笛鲷(Lutianus sanguineus)是我国南方沿海网箱和池塘养殖重要海水养殖鱼类之一, 然而近年来细菌病和寄生虫病频繁暴发, 严重制约了其养殖业的健康可持续发展(Zhang et al, 2011)。提高机体免疫力是鱼病防治的基础, 探求其免疫防御机制对于鱼病防治具有重要的指导意义。然而, 到目前为止尚未有红笛鲷T细胞表面标志基因的报道。本研究克隆了红笛鲷CD4和CD4-2的cDNA全长序列, 应用荧光定量PCR (Real Time Quantitative PCR, qPCR)检测了CD4和CD4-2在健康鱼组织中的分布和哈维氏弧菌疫苗诱导后红笛鲷免疫相关组织中的表达变化, 为进一步研究红笛鲷T细胞免疫功能奠定基础。
1 材料与方法 1.1 实验鱼健康红笛鲷购自湛江南三岛某海水养基地, 体质量约100g/尾, 活力好, 摄食正常, 暂养于水温25—28 ℃的海水养殖试验水泥池中, 24h连续充氧, 每天早晚投喂商品配合饲料各一次, 日换水量三分之一。暂养1周后用于实验。
1.2 哈维氏弧菌疫苗的免疫及样品采集将哈维氏弧菌强毒株ZJ0603接种于TSB (2% NaCl)培养基中, 28 ℃振荡培养12h, 用平板计数法计数菌液浓度, 剩余菌液缓慢加入0.3%福尔马林, 28 ℃灭活24h, 灭活完全后将哈维氏弧菌疫苗菌液用PBS (0.01mol/L, pH 7.4) 调整浓度为1×109 CFU/mL, 置2—8 ℃保存备用。将实验鱼分成2组, 免疫组每尾鱼腹腔注射哈维氏弧菌疫苗菌液0.2mL, 对照组注射等量无菌PBS, 于免疫后的4、8、12、24、48和72h分别采集胸腺、头肾、脾脏、肝脏、鳃、皮肤、后肾和肠8个组织, 每个时间点采集5尾红笛鲷, 置于RNAlater –80 ℃保存备用, 收集的组织主要用于荧光定量分析。同时取5尾健康鱼的胸腺、鳃、头肾、肝、脾、中肾、心脏、肠、脑、胃、皮肤和肌肉组织立即置液氮中, 用于cDNA全长克隆和组织表达分析。
1.3 头肾淋巴细胞的制备及体外刺激试验分别取5尾健康红笛鲷用MS-222麻醉后, 75%酒精短暂全身浸泡消毒, 无菌条件下取出头肾组织, 迅速置于RPMI1640(含100U青霉素, 100μg/mL链霉素和0.5%胎牛血清), 用无菌的剪刀将其剪成小块后置于100目的无菌细胞筛网上, 用一次性无菌的注射器头轻轻研磨头肾组织, 用RPMI1640冲洗筛网, 去掉细胞碎片, 获取头肾细胞悬液备用。用无菌枪头吸取2 mL头肾细胞悬液沿管壁小心缓慢地加到等34%和51%不连续密度梯度Percoll细胞分离液界面上。于4 ℃、500g离心30min, 小心吸取两层Percoll细胞分离液中间层白细胞, 用RPMI1640洗涤2次, 再用血球计数板进行计数, 调整所收集到的细胞悬液浓度为5.0×105, 以每孔接种1mL接种到24孔板中, 25 ℃培养4h, 收集细胞悬液后, 重新接种到新的24孔板中。分别在每孔中加入终浓度分别为10µg/mL的脂多糖(Lipopolysaccharide, LPS)和刀豆蛋白A (Concanavalin A, ConA), 空白组加入PBS, 每种处理组设3个平行, 分别于4、8、12和24h取样。将样品于4 ℃、500g条件下离心10min去除培养基, 加入500μL TRIzol –80 ℃保存备用。
1.4 总RNA提取和cDNA一链合成参考TRIzol(全式金)说明书分别提取红笛鲷各组织总RNA, 紫外分光光度计测定其浓度, 使其OD260/OD280在1.8—2.0。按照M-MLV(TaKaRa)说明书合成cDNA一链。另取脾脏的总RNA, 按RACE试剂盒(Clontech)说明书分别合成用于RACE-PCR的cDNA一链。
1.5 红笛鲷CD4和CD4-2 cDNA克隆 1.5.1 基因部分序列克隆依据GenBank已提交的鱼类CD4基因cDNA序列的保守区域设计简并引物CD4F与CD4R(表 1), 运用降落PCR从红笛鲷头肾cDNA第一链中扩增红笛鲷CD4基因的部分序列, 反应条件为: 95 ℃预变性3min, 94 ℃ 30s, 62 ℃ 30 s, 72 ℃ 1min 5个循环; 94 ℃ 30s, 59 ℃ 30s, 72 ℃ 1min 5个循环; 94 ℃ 30s, 56 ℃ 30s, 72 ℃ 1min 25个循环, 72 ℃延伸6min。PCR产物经1.2%琼脂糖电泳, 目的条带切胶后用GeneJET Kit (Thermo Scientific)进行回收, 与pMD-19T载体连接后转化DH5α感受态, 阳性菌落送生工生物工程(上海)股份有限公司测序。
根据简并引物扩增获得的CD4基因部分序列和红笛鲷头肾转录组高通量测序获得CD4-2基因部分序列设计特异性巢式3'RACE和5'RACE引物(表 1)。将3'CD4F1、3'CD4-2F1和5'CD4R1、5'CD4-2R1分别与UPM Mix引物进行3'RACE与5'RACE第一轮降落PCR扩增。反应条件为: 95 ℃预变性3min, 94 ℃ 30s, 68 ℃ 2min 5个循环; 94 ℃ 30s, 65 ℃ 30s, 72 ℃ 2min 5个循环; 94 ℃ 30s, 62 ℃ 30s, 72 ℃ 2min 25个循环, 72 ℃延伸6min。将第一轮PCR产物稀释10倍为模板, 将3'CD4F2、3'CD4-2F2和5'CD4R2、5'CD4-2R2分别与NUP进行第二轮PCR扩增, 反应条件为: 95 ℃预变性3min, 94 ℃ 30s, 65 ℃ 30s, 72 ℃ 2min 30个循环, 72 ℃延伸6min。PCR产物回收和测序同1.5.1。
1.6 生物学信息分析Blast程序进行序列同源性分析; protparam软件预测分子量(Mw)和理论等电点(pI)等; SignalP 4.0 Server软件预测蛋白质分子信号肽; TMHMM Server 2.0软件预测蛋白质分子跨膜区; NetGlyc1.0软件预测蛋白质分子N-糖基化位点; SMART软件分析蛋白质功能结构域; Clustalw 2及MEGA 5.0软件以邻位相连法(Neighbor-Joining)构建系统发育树。物种CD4分子序列号见表 2。
将合成的cDNA第一链以1︰5—10稀释后作为qPCR的模板, 根据红笛鲷CD4和CD4-2基因ORF设计qPCR引物reCD4F、reCD4R和reCD4-2F、reCD4-2R(表 1), β-actin作为内参基因, 反应和分析在Bio-Rad iQ5实时荧光定量PCR仪进行。每个样本设3个平行。qPCR反应条件为: 95 ℃ 20s, 62 ℃ 20s, 72 ℃ 20s, 40个循环。通过2-△△CT法计算红笛鲷CD4和CD4-2基因的相对表达量, 应用SPSS 11.0 for windows软件对获得的实验数据进行进行ANOVA单因素方差分析和Tukey HSD多重比较。*表示与同期对照组相比差异显著(P < 0.05)。
2 结果 2.1 CD4和CD4-2基因的cDNA克隆与序列分析红笛鲷CD4基因cDNA全长2216bp (GenBank登录号: KF285565), 包含180bp 5′ UTR、1431bp ORF和605bp 3′ UTR, 推导编码476个氨基酸, 预测分子量和等电点分别为53.02kDa和9.44。红笛鲷CD4分子含有4个N-糖基化位点。红笛鲷CD4-2基因cDNA全长为1520bp (GenBank登录号: KY020127), 包含62bp 5′ UTR、933bp ORF和525bp 3′ UTR, 推导编码310个氨基酸, 预测其蛋白分子量和等电点分别为33.80kDa和9.69。红笛鲷CD4-2分子不含有N-糖基化位点。
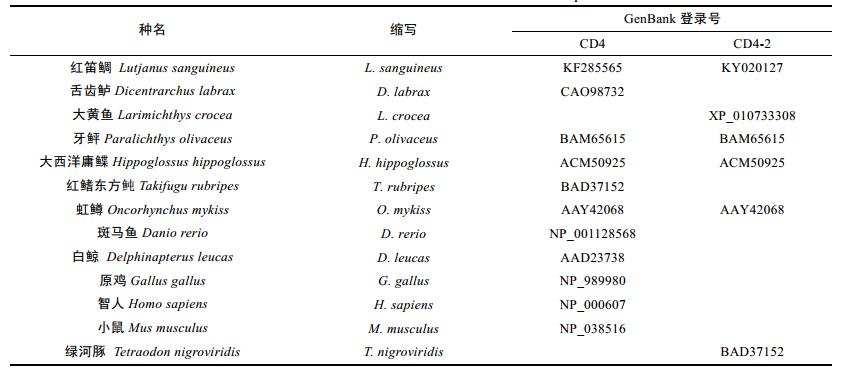
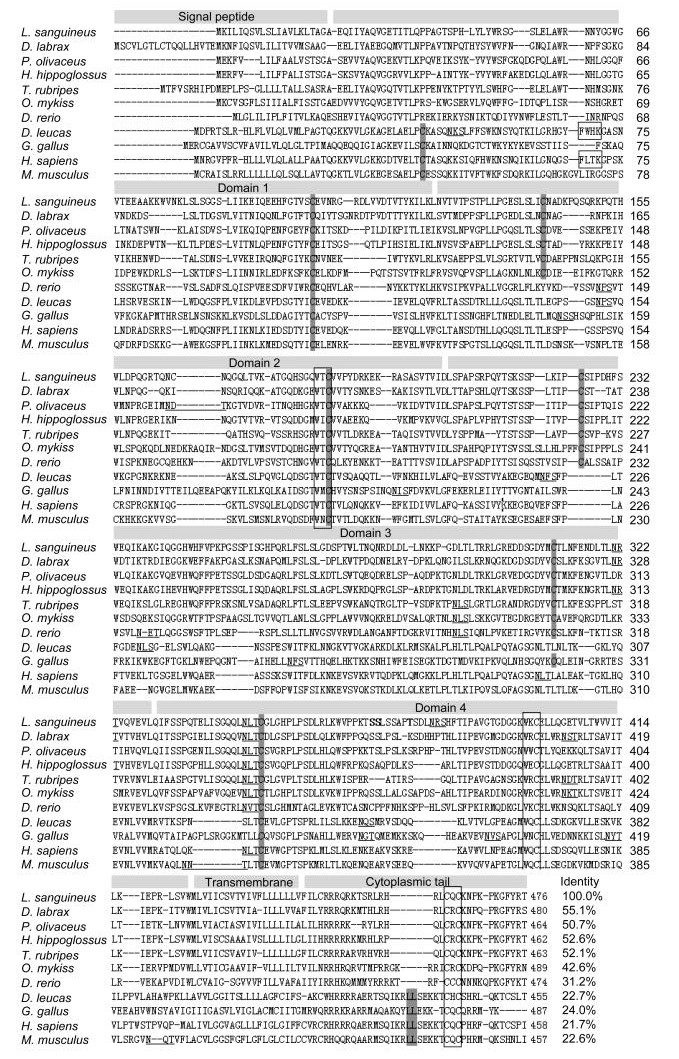
红笛鲷CD4和CD4-2均属于Ⅰ型跨膜蛋白分子, 包含信号肽、胞外区、跨膜区和胞浆区四个结构域。CD4分子胞外区为4个Ig样区(D1-C1-D2-C2)。红笛鲷的胞外区含有7个在鱼类或与哺乳动物之间保守的Cys残基(Cys101, Cys141, Cys185, Cys225, Cys310, Cys349和Cys399)。CD-2分子蛋白胞外区包含2个Ig样区(D1-D2) 和连接肽区。红笛鲷的胞外区含有6个在鱼类保守的Cys残基(Cys34, Cys94, Cys127, Cys168, Cys213和Cys216)。鱼类中保守的色氨酸残基和CXXC基序分别存在于CD4-2分子的Ig样区(D1—D2) 和连接肽区。在红笛鲷CD4和CD4-2胞浆区均存在高度保守的p56lck的结合位点CXC基序(图 1, 图 2)。
 |
| 图 1 红笛鲷CD4与其他物种CD4氨基酸序列比较 Fig. 1 Multiple alignments of snapper CD4 and other species CD4 sequences 注:保守氨基酸以灰色背景突出显示, 功能基序加方框显示, 预测的N-糖基化位点以下划线进行标示 |
 |
| 图 2 红笛鲷CD4-2与其他物种CD4-2氨基酸序列比较 Fig. 2 Multiple alignments of snapper CD4-2 and other species CD4-2 sequences 注:保守氨基酸以灰色背景突出显示, 功能基序加方框标示, 预测的N-糖基化位点以下划线标示 |
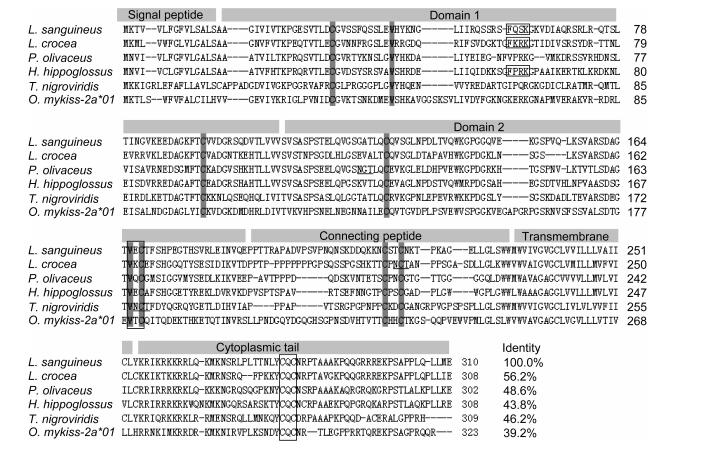
利用BLAST程序将红笛鲷CD4和CD4-2的氨基酸序列与NCBI数据库已登录的其他物种的CD4和CD4-2分别进行同源性比较, 结果显示红笛鲷与其他物种的CD4具有不同程度的同源性, 与斜带石斑鱼的CD4同源率最高达58.5%, 与智人(Homo sapiens)的同源性最低为21.7%(图 1)。其中红笛鲷CD4-2和大黄鱼(Larimichthys crocea)的CD4-2同源率最高达56.2%, 与其他鱼类的CD4-2同源率达39.2%—48.6%(图 2)。利用MEGA 6.0的Neighbor-joining法构建系统进化树, 结果如图 3显示, 鸟类和哺乳动物聚成簇, 与鱼类形成独立的进化分支。红笛鲷CD4和CD4-2与硬骨鱼鲈形目的斜带石斑鱼(Epinephelus coioides)及大黄鱼聚为一支, 表明它们亲缘关系最近, 与鸟类和哺乳类遗传距离较远, 七鳃鳗(Petromyzon marinus)CD4与其他鱼类的CD4-2聚在一起, 表明该分子可能是已知CD4分子的原基。
 |
| 图 3 邻接法构建的红笛鲷与其他物种CD4和CD4-2分子系统进化树 Fig. 3 Phylogenetic tree of CD4 and CD4-2 snapper and other vertebrate constructed in neighbor-joining algorithm |
应用qPCR技术检测CD4和CD4-2在健康红笛鲷不同组织中的表达水平, 结果显示, CD4和CD4-2在鳃、头肾、肝、脾、肾、心脏、肠、脑、胃、皮肤和肌肉等组织中均有表达, 在胸腺的表达量最高, 其次为中肾、鳃、皮肤、脾、头肾和肠, 其他组织表达量较低(图 4)。
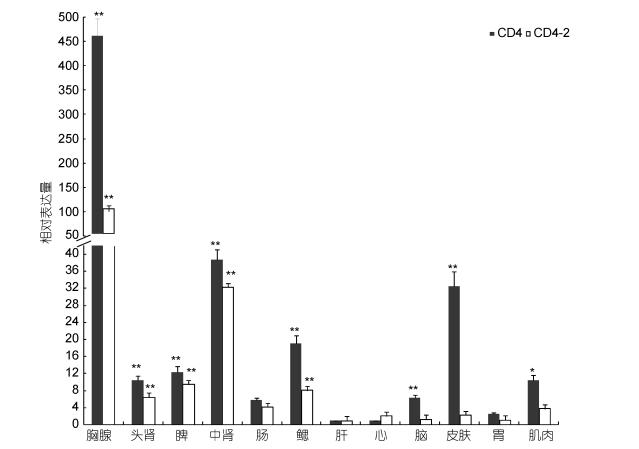 |
| 图 4 红笛鲷CD4和CD4-2的组织分布 Fig. 4 Tissue distribution of snapper CD4 and CD4-2 |
红笛鲷头肾淋巴细胞在体外经LPS和ConA刺激后, CD4和CD4-2表达量在试验期间内均有明显的升高(图 5, 图 6)。在刺激后4h, CD4的表达量开始升高,LPS处理组在12h达到最高, 与对照组有显著差异(P < 0.05), ConA处理组的CD4的表达量在8h和12h时较高, 与对照组有显著差异(P < 0.05), 然后逐渐下降至正常表达水平。与CD4的表达相类似, LPS处理组和ConA处理组的CD4-2在刺激后开始轻微上调表达, 并在12h达到最高表达水平, 与对照组有显著差异(P < 0.05)。
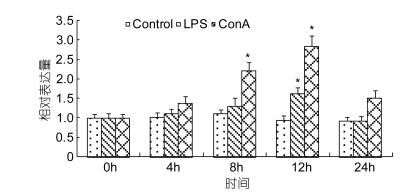 |
| 图 5 红笛鲷头肾淋巴细胞在两种免疫刺激剂体外刺激后CD4基因的表达变化 Fig. 5 Snapper CD4 expression analysis of cultured head kidney leukocytes after in vitro stimulation with LPS and ConA |
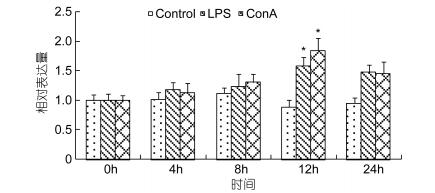 |
| 图 6 红笛鲷头肾淋巴细胞在两种免疫刺激剂体外刺激后CD4-2基因的表达变化 Fig. 6 Snapper CD4-2 expression analysis of cultured head kidney leukocytes after in vitro stimulation with LPS and ConA |
应用荧光定量PCR检测了哈维氏弧菌疫苗免疫后4、8、12、24、48和72h后CD4和CD4-2在红笛鲷鳃、头肾、脾脏和肠的表达变化。结果显示, 哈维氏弧菌疫苗免疫后的8h, CD4基因在鳃和肠中显著上调表达(P < 0.05), 并在12h达到最高, 在头肾和脾脏中的表达量均于24h达到最高(P < 0.05) (图 7, 图 8)。CD4-2基因表达量在鳃和肠于免疫后的12h达到峰值(P < 0.05), 在头肾和脾脏则于24h达到峰值(P < 0.05)。
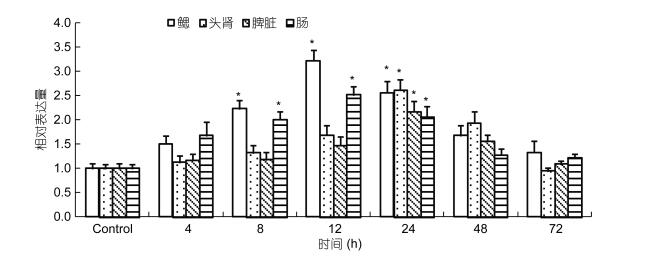 |
| 图 7 哈维氏弧菌感染后鳃、头肾、脾和肠中CD4的表达 Fig. 7 The relative expression level of CD4 in gills, head kidney, spleen and intestine after V. harveyi immunization |
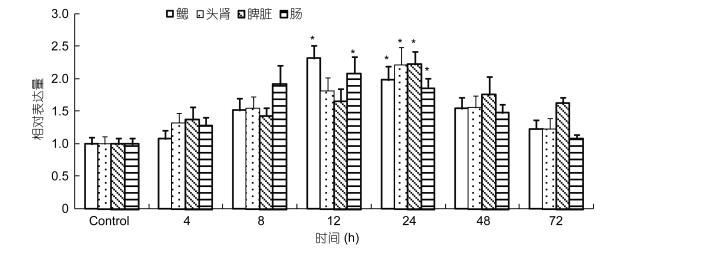 |
| 图 8 哈维氏弧菌感染后鳃、头肾、脾和肠中CD4-2的表达 Fig. 8 The relative expression level of CD4-2 in gills, head kidney, spleen and intestine after V. harveyi immunization |
本研究通过RACE-PCR技术获得了红笛鲷CD4和CD4-2的cDNA全长序列, 分别编码462和223个氨基酸, 与已发现的其他鱼类和哺乳动物的CD4结构相类似, 红笛鲷CD4和CD4-2分子均含有信号肽、胞外区、跨膜区和胞浆区, 具有一些高度保守的氨基酸残基、N-糖基化位点和功能基序, 对于T细胞分化、成熟和信号转导起关键作用(Parnes, 1989)。
红笛鲷CD4胞外区主要是由4个Ig样结构域(D1-D4) 构成。除D1区外, 其他3个区都含有两个保守的半胱氨酸残基, 用以形成具有稳定Ig折叠作用的结构域内二硫键。与哺乳动物和鸟类不同的是, 鱼类的D1区只含有一个半胱氨酸残基, 该区域不能形成二硫键连接。在D2和D4区, 鱼类和哺乳动物的半胱氨酸残基相对比较保守, 但斑马鱼、白鲸和鸟类D2区却缺少一个半胱氨酸残基, 导致鱼类CD4分子在空间构象上可能不同于高等脊椎动物CD4分子(Romano et al, 1999)。由于D1与D2是CD4分子在抗原识别过程中与MHC Ⅱ分子主要结合部位(Clayton et al, 1989; Huang et al, 1997), 因此不同物种CD4分子构象不同可能影响Th细胞的功能(Sun et al, 2007)。此外, 红笛鲷D3区也存在硬骨鱼类所特有的一对半胱氨酸残基, 形成了一个额外的二硫键, 而鸟类和哺乳类在该区域均缺失了两个半胱氨酸残基, 因此对该二硫键的功能有待进一步研究。
人CD4分子D1区的FXXK基序是MHC Ⅱ分子的结合位点, 其中结合能主要由F提供, 但鸟类、小鼠和一些鱼类缺乏该此位点, 相反红笛鲷、大黄鱼和大西洋庸鲽的CD4-2分子的D1区均含有该基序, 因此鱼类CD4与MHC Ⅱ分子可能存在一种不同于人的的结合方式。此外所有物种的CD4和CD4-2分子D2区均存在一个特有的WXC基序, 并且该基序在脊椎动物的淋巴细胞活化基因-3(lymphocyte activation gene 3, LAG-3) 的D4区也同样保守, 据此推测CD4和LAG-3可能来源于同一个祖先, 并通过含有两个Ig样结构域的CD4-2分子通过基因内复制产生(Triebel et al, 1990; Laing et al, 2006)。红笛鲷CD4-2连接肽区含有一个CXXC基序, 该基序在目前已知硬骨鱼类如大黄鱼、大西洋庸鲽、红鳍东方鲀和虹鳟中高度保守, 用以形成连接二聚体的二硫键, 目前已证明CD4二聚化在CD4介导的T细胞激活中必不可少, 而且可以促进与MHC Ⅱ链接(Moldovan et al, 2006)。同时鱼类CD4-2连接肽区富含脯氨酸, 使肽链伸更易于伸展弯曲接触MHC Ⅱ(Buonocore et al, 2008)。与所有的鸟类、哺乳动物和鱼类CD4相类似, 红笛鲷CD4和CD4-2胞内区均含有一个高度保守的p56lck结合基序CXC, 该基序通过锌指拉链结构与p56lck的CXXC基序连接相连接启动T细胞激活的第一信号(Kim et al, 2003), 进而导致CD3分子胞内域的ITAM部分的磷酸化和fyn以及活化ZAP-70等下游信号分子, 最终T细胞的信号转导和激活得以完成(Leo et al, 2001)。高等脊椎动物的CD4分子胞内区的CXC基序上游含有两个保守的丝氨酸残基和LL基序, 这4个保守氨基酸参与抗原诱导CD4分子的内化(Shin et al, 1990), 但是在鱼类CD4和CD4-2分子中均不存在, 因此有关鱼类控制T淋巴细胞的过度活化以保持免疫系统平衡的机制尚需要进一步研究。
蛋白糖基化影响蛋白质的折叠、成熟、包装、投送、定位和抗原性以及细胞与细胞之间的连接(章晓联, 2004)。糖基化对T细胞的结构和功能同样也起到非常重要的作用, CD4分子的糖基化位点糖基化完全后才能正确折叠和表达于细胞膜上(Rudd et al, 2001)。鱼类CD4分子含多个N-糖基化位点, 位于D3和D4结构域的糖基化位点的在所有物种中均相对保守, 预示鱼类CD4分子与高等脊椎动物CD4分子具有相似的部分功能。
荧光定量PCR检测结果显示红笛鲷CD4和CD4-2在胸腺中表达量最高, 在中肾、鳃、皮肤、肠和头肾等组织中也有中等程度的表达, 这种组织表达模式与舌齿鲈、大西洋庸鲽和虹鳟相似(Laing et al, 2006; Buonocore et al, 2008; Patel et al, 2009; ), 但是与红鳍东方鲀和斑点叉尾鮰不同(Suetake et al, 2004; Edholm et al, 2007), 可能与种属、生活环境及生理状态有关。胸腺是硬骨鱼的中枢免疫器官(谢海侠等, 2003)。CD4的高表达证实其是鱼类T细胞的发生、增殖、分化和成熟的主要场所。鱼类皮肤、鳃和肠是鱼类主要的黏膜淋巴组织, 也是最早接触水体病原的部位, 目前研究发现其中含有大量T淋巴细胞、抗原呈递细胞(Antigen-Presenting Cells, APC)和B淋巴细胞组织(Fournier-Betz et al, 2000; Rombout et al, 2014), 具有引发免疫应答的细胞基础, CD4分子这些组织中的表达表明红笛鲷黏膜组织中同样含有CD4+T细胞, 病原入侵时其可引起局部黏膜免疫应答, 在抵御病原入侵和免疫反应中起重要作用。头肾淋巴细胞在体外经LPS和ConA刺激后, CD4 mRNA显著上调表达, 与小鼠和舌齿鲈上的研究结果相一致(McAleer et al, 2007), 表明鱼类的CD4+T细胞与哺乳类CD4+T具有相似的特性(Toda et al, 2011), 膜上含有丝分裂原受体, 与LPS和ConA结合后可刺激其增殖。
CD4+T细胞在外周器官识别抗原肽与MHC Ⅱ类分子形成的复合物后被激活, 成为辅助T细胞(Th), 参与细胞免疫和体液免疫, 在抗菌感染中发挥重要作用。耶尔森氏菌(Yersinia ruckeri)灭活疫苗浸泡免疫8h后虹鳟CD4显著上调表达(Raida et al, 2008), Kato等(2013)等用胞内病原海豚链球菌(Streptococcus iniae)、迟缓爱德华氏菌(Edwardsiella tarda)和病毒性出血性败血症病毒(Viral Hemorrhagic Septicemia Virus, VHSV)感染牙鲆, 结果发现经上述病原感染后5d中肾CD4 mRNA呈现中等程度的持续上调表达, 而CD4-2 mRNA表达水平显著上升, 其相对表达量高于CD4, 表明主要触发Th1介导的免疫应答, CD4-2+T细胞在感染早期大量增殖, 并认为CD4和CD4-2存在于不同亚型的T细胞, 分别表达于初始Th细胞和记忆Th细胞。本研究中红笛鲷经哈维氏弧菌疫苗免疫24h后鳃、头肾、脾脏和肠CD4和CD4-2表达量均显著上调, 表明哈维氏弧菌疫苗免疫后引起Th大量增殖, 与耶尔森氏菌灭活疫苗浸泡免疫虹鳟的结果相似, 与Kato等(2013)的研究结果不太相同, 可能与病原的种类、感染方式及宿主等有关, 有关CD4-1和CD4-2分子分别表达或共表达以及免疫反应过程中的作用和调节机制仍有待进一步研究。综上所述, CD4和CD4-2在鱼类的免疫应答中发挥重要作用。
4 结论本研究克隆获得了红笛鲷CD4和CD4-2基因, 均由信号肽、免疫球蛋白样结构域构成的胞外区、跨膜区和胞浆区组成, 健康红笛鲷CD4和CD4-2基因在健康鱼的胸腺中表达量最高, 其次是中肾、鳃、皮肤、脾、头肾和肠。红笛鲷头肾淋巴细胞体外经LPS和ConA刺激12h后, CD4和CD4-2表达量显著上调(P < 0.05)。哈维氏弧菌疫苗免疫24h后鳃、头肾、脾脏和肠的表达量显著上升(P < 0.05)。研究结果为进一步制备单克隆抗体、分选不同的T细胞亚群以及不同亚群的功能鉴定提供了基础。
| 章晓联, 2004. 蛋白糖基化与免疫. 中国免疫学杂志, 20(4): 290–293 |
| 谢海侠, 聂品, 2003. 鱼类胸腺研究进展. 水产学报, 27(1): 90–96 |
| Buonocore F, Randelli E, Casani D, et al, 2008. A CD4 homologue in sea bass (Dicentrarchus labrax):molecular characterization and structural analysis. Mol Immunol, 45(11): 3168–3177 DOI:10.1016/j.molimm.2008.02.024 |
| Clayton L K, Sieh M, Pious D A, et al, 1989. Identification of human CD4 residues affecting class Ⅱ MHC versus HIV-1 gp120 binding. Nature, 339(6225): 548–551 DOI:10.1038/339548a0 |
| Dijkstra J M, Somamoto T, Moore L, et al, 2006. Identification and characterization of a second CD4-like gene in teleost fish. Mol Immunol, 43(5): 410–419 DOI:10.1016/j.molimm.2005.03.005 |
| Edholm E S, Stafford J L, Quiniou S M, et al, 2007. Channel catfish, Ictalurus punctatus, CD4-like molecules. Dev Comp Immunol, 31(2): 172–187 DOI:10.1016/j.dci.2006.05.012 |
| Fournier-Betz V, Quentel C, Lamour F, et al, 2000. Immunocytochemical detection of Ig-positive cells in blood, lymphoid organs and the gut associated lymphoid tissue of the turbot (Scophthalmus maximus). Fish Shellfish Immunol, 10(2): 187–202 DOI:10.1006/fsim.1999.0235 |
| Huang B, Yachou A, Fleury S, et al, 1997. Analysis of the contact sites on the CD4 molecule with class Ⅱ MHC molecule:co-ligand versus co-receptor function. J Immunol, 158(1): 216–225 |
| Kato G, Goto K, Akune I, et al, 2013. CD4 and CD8 homologues in Japanese flounder, Paralichthys olivaceus:differences in the expressions and localizations of CD4-1, CD4-2, CD8α and CD8β. Dev Comp Immunol, 39(3): 293–301 DOI:10.1016/j.dci.2012.09.004 |
| Kim P W, Sun Z Y, Blacklow S C, et al, 2003. A zinc clasp structure tethers Lck to T cell coreceptors CD4 and CD8. Science, 301(5640): 1725–1728 DOI:10.1126/science.1085643 |
| Laing K J, Zou J J, Purcell M K, et al, 2006. Evolution of the CD4 family:teleost fish possess two divergent forms of CD4 in addition to lymphocyte activation gene-3. J Immunol, 177(6): 3939–3951 DOI:10.4049/jimmunol.177.6.3939 |
| Leo A, Schraven B, 2001. Adapters in lymphocyte signalling. Curr Opin Immunol, 13(3): 307–316 DOI:10.1016/S0952-7915(00)00220-X |
| Lynch E A, Heijens C A W, Horst N F, et al, 2003. Cutting edge:IL-16/CD4 preferentially induces Th1 cell migration:requirement of CCR5. J Immunol, 171(10): 4965–4968 DOI:10.4049/jimmunol.171.10.4965 |
| McAleer J P, Zammit D J, Lefrançois L, et al, 2007. The lipopolysaccharide adjuvant effect on T cells relies on nonoverlapping contributions from the MyD88 pathway and CD11c+ cells. J Immunol, 179(10): 6524–6535 DOI:10.4049/jimmunol.179.10.6524 |
| Moldovan M C, Sabbagh L, Breton G, et al, 2006. Triggering of T cell activation via CD4 dimers. J Immunol, 176(9): 5438–5445 DOI:10.4049/jimmunol.176.9.5438 |
| Øvergård A C, Nerland A H, Patel S, 2010. Cloning, characterization, and expression pattern of Atlantic halibut (Hippoglossus hippoglossus L) CD4-2, Lck, and ZAP-70. Fish Shellfish Immunol, 29(6): 987–997 DOI:10.1016/j.fsi.2010.08.011 |
| Parnes J R, 1989. Molecular biology and function of CD4 and CD8. Adv Immunol, 44: 265–311 DOI:10.1016/S0065-2776(08)60644-6 |
| Patel S, Øvergård A C, Nerland A H, 2009. A CD4 homologue in Atlantic halibut (Hippoglossus hippoglossus):molecular cloning and characterisation. Fish Shellfish Immunol, 26(3): 377–384 DOI:10.1016/j.fsi.2008.11.013 |
| Raida M K, Buchmann K, 2008. Bath vaccination of rainbow trout (Oncorhynchus mykiss Walbaum) against Yersinia ruckeri:effects of temperature on protection and gene expression. Vaccine, 26(8): 1050–1062 DOI:10.1016/j.vaccine.2007.12.029 |
| Romano T A, Ridgway S H, Felten D L, et al, 1999. Molecular cloning and characterization of CD4 in an aquatic mammal, the white whale Delphinapterus leucas. Immunogenetics, 49(5): 376–383 DOI:10.1007/s002510050510 |
| Rombout J H W M, Yang G W, Kiron V, 2014. Adaptive immune responses at mucosal surfaces of teleost fish. Fish Shellfish Immunol, 40(2): 634–643 DOI:10.1016/j.fsi.2014.08.020 |
| Rudd P M, Elliott T, Cresswell P, et al, 2001. Glycosylation and the immune system. Science, 291(5512): 2370–2376 DOI:10.1126/science.291.5512.2370 |
| Shin J, Doyle C, Yang Z, et al, 1990. Structural features of the cytoplasmic region of CD4 required for internalization. EMBO J, 9(2): 425–434 |
| Smith-Garvin J E, Koretzky G A, Jordan M S, 2009. T cell activation. Annu Rev Immunol, 27(1): 591–619 DOI:10.1146/annurev.immunol.021908.132706 |
| Suetake H, Araki K, Suzuki Y, 2004. Cloning, expression, and characterization of fugu CD4, the first ectothermic animal CD4. Immunogenetics, 56(5): 368–374 |
| Sun X F, Shang N, Hu W, et al, 2007. Molecular cloning and characterization of carp (Cyprinus carpio L.) CD8β and CD4-like genes. Fish Shellfish Immunol, 23(6): 1242–1255 DOI:10.1016/j.fsi.2007.06.004 |
| Toda H, Saito Y, Koike T, et al, 2011. Conservation of characteristics and functions of CD4 positive lymphocytes in a teleost fish. Dev Comp Immunol, 35(6): 650–660 DOI:10.1016/j.dci.2011.01.013 |
| Triebel F, Jitsukawa S, Baixeras E, et al, 1990. LAG-3, a novel lymphocyte activation gene closely related to CD4. J Exp Med, 171(5): 1393–1405 DOI:10.1084/jem.171.5.1393 |
| Zhang X Z, Dai L P, Wu Z H, et al, 2011. Molecular cloning, mRNA expression, and characterization of heat shock protein 10 gene from humphead snapper Lutjanus sanguineus. Mar Genomics, 4(3): 143–150 DOI:10.1016/j.margen.2010.10.001 |
 2017, Vol. 48
2017, Vol. 48