中国海洋湖沼学会主办。
文章信息
- 熊尚凌, 邢峻梁, 范东洋, 袁洋洋, 朱四东, 林旦璆, 杨季芳, 陈吉刚. 2017.
- XIONG Shang-Ling, XING Jun-Liang, FAN Dong-Yang, YUAN Yang-Yang, ZHU Si-Dong, LIN Dan-Qiu, YANG Ji-Fang, CHEN Ji-Gang. 2017.
- 锯缘青蟹(Scylla serrata)呼肠孤病毒5个结构蛋白互作分析
- MAPPING INTERACTIONS OF SCYLLA SERRATA REOVIRUS IN FIVE STRUCTURAL PROTEINS
- 海洋与湖沼, 48(5): 1108-1113
- Oceanologia et Limnologia Sinica, 48(5): 1108-1113.
- http://dx.doi.org/10.11693/hyhz20170300070
-
文章历史
- 收稿日期:2017-03-24
- 收修改稿日期:2017-06-03
青蟹呼肠孤病毒(mud crab reovirus, MCRV)首次报道于广东青蟹养殖区(Weng et al, 2007), 其后在浙江青蟹养殖区发现类似病毒, 并将其命名为锯缘青蟹呼肠孤病毒(Scylla serrata reovirus, SsRV)(陈吉刚等, 2008; Chen et al, 2011)。根据病毒粒子形态和基因组序列同源性, 判定MCRV和SsRV为不同地区分离株(Chen et al, 2012; Deng et al, 2012)。SsRV基因组为12节段双链(ds)RNA(S1—S12), 可能编码13种病毒蛋白(p160, p100, p96, p50, p25, p61, p67, p46, p32, p40, p35, p23和p29) (Chen et al, 2012)。SDS-PAGE结合Modi TOF/TOF质谱分析表明, p160, p96, p67, p40, p29和p23六个蛋白为SsRV病毒粒子的结构蛋白(Yuan et al, 2016)。在上述13种病毒蛋白中, 只有p160和p40的功能较为清楚。p160蛋白为病毒的RNA依赖的RNA聚合酶(RdRp), p35蛋白具有病毒孔蛋白样功能, 在病毒复制的后期发挥重要作用(Chen et al, 2012; Zhang et al, 2015; Yuan et al, 2017)。
MCRV的三维结构已被初步解析(Huang et al, 2012)。该病毒衣壳由T=13外层衣壳和T=2内层衣壳两层衣壳组成, 其结构特征与RDV、SA1114F和BTV相似。在MCRV完整病毒颗粒的三维结构中, 外衣壳蛋白包含表面小突起的同源三聚体蛋白260个、衣壳表面六邻体中央蛋白120个; 内层衣壳具有一种蛋白的两种构象体A和B各60个, 以及5次轴顶点下方的可能由RNA聚合酶和其它蛋白形成的转录复合物。然而目前仅根据上述研究结果还无法确定青蟹呼肠孤病毒衣壳蛋白的具体组成, 更不清楚这些病毒蛋白间的相互作用关系及其对病毒结构和生物学发生功能的影响。呼肠孤病毒衣壳蛋白之间通常存在某种相互作用, 以维系病毒蛋白的层次结构并维持病毒形态的稳定性(Miyazaki et al, 2005)。基于此, 本研究通过酵母双杂交技术对SsRV 5个结构蛋白间的相互作用进行分析, 研究结果为进一步揭示SsRV病毒结构和形态发生提供理论依据。
1 材料与方法 1.1 菌株、载体及主要试剂大肠杆菌菌株E. coli DH5α购自北京全式金生物有限公司; 酵母双杂交试剂盒购自Clontech公司; 携带SsRV S3, S6, S9, S11或S12的重组质粒PMD-S3、PMD-S6、PMD-S9、PMD-S11和PMD-S12由本实验室构建并保存; 鲑鱼精DNA购自索莱宝生物科技有限公司; Minimal SD Base、SD/-Trp、SD/-Leu、SD/-Leu-Trp、SD/-Leu-Trp-Ade-His、Aureobasidin A、X-α-gal、PCR相关试剂、EcoR I、Xhol I、Sal I、BamH I均购自TaKaRa公司; DNA纯化试剂盒和质粒提取试剂盒SanPrep购自上海生物工程有限公司。其它试剂均为国产分析纯。
1.2 方法 1.2.1 猎物载体及诱饵载体的构建及鉴定由于SsRV和MCRV为同一病毒不同地区分离株, 为了达到SsRV和MCRV病毒蛋白描述上的统一, 本文采用MCRV病毒蛋白命名方式(VPx, x对应基因组中节段序号)对SsRV蛋白进行命名。根据SsRV的VP3(p96), VP6(p67), VP9(p40), VP11(p23), VP12(p29) 蛋白对应的基因序列并结合猎物载体pGADT7和诱饵载体pGBKT7多克隆位点设计6对特异引物(表 1), 分别以重组载体PMD-S3、PMD-S6、PMD-S9、PMD-S11和PMD-S12为模板进行PCR扩增。PCR产物电泳回收纯化后, 经双酶切后, 分别克隆于猎物载体pGADT7和诱饵载体pGBDT7, 转化感受态细胞DH5α, 并利用氨苄青霉素或卡那霉素抗性筛选阳性克隆, 挑选阳性克隆小量抽提质粒, 利用PCR和双酶切进行鉴定后, 送上海桑尼生物科技有限公司进行测序。
| 引物名称 | 载体名称 | 引物序列(5′—3′)* | 酶切位点 |
| VP3-AD-F | AD-VP3 | CGGAATTCATGGCTAGTACAACTAGACTTG | EcoR I |
| VP3-AD-R | CCGCTCGAGTTTATCTCACATATTCGACCACAC | Xhol I | |
| VP3-BD-F | BD-VP3 | CGGAATTCATGGCTAGTACAACTAGACTTG | EcoR I |
| VP3-BD-R | ACGCGTCGACGTTATCTCACATATTCGACCACAC | Sal I | |
| VP6-F | BD-VP6/AD-VP6 | CGGAATTCATGGCTTTCGAATTTCGTCTATC | EcoR I |
| VP6-R | CGGGATCCTATTCTTGAAAGTTTTCCAAATATGTACC | BamH I | |
| VP9-F | BD-VP6/AD-VP6 | CGGAATTCATGGCAGACAGTATCACTG | EcoR I |
| VP9-R | CGGGATCCCTCAGTAATAAACATCAGCCTTAGT | BamH I | |
| VP11-F | BD-VP11/AD-VP11 | CGGAATTCATGAATTGGTCAAAAGCAATAAAC | EcoR I |
| VP11-R | CGGGATCCCTTAACTTTGGTTTTTCCAAGAGAG | BamH I | |
| VP12-F | BD-VP12/AD-VP12 | CGGAATTCATGAACCTGGAAATTAACAACTTTG | EcoR I |
| VP12-R | CGGGATCCCTCAGTAATCGAGAACCCAC | BamH I | |
| 注: *加粗字体代表内切酶识别位点 | |||
将含有目的基因的重组载体pGBKT7-VPx, 简称BD-VPx (x代表3, 6, 9, 11, 12)分别与pGADT7共转感受态细胞Y2H Gold, 以及将pGADT7-VPx, 简称AD-VPx (3, 6, 9, 11, 12) 分别与pGBKT7共转Y2H Gold感受态细胞, 进行目的蛋白的自激活检测。实验设pGBKT7-53 & pGADT7-T共转感受态细胞为阳性对照组, 设pGBKT7-lam & pGADT7-T共转转感受态细胞, 以及pGBKT7 & pGADT7共转感受态细胞为阴性对照组。吸取适量的共转化酵母涂布于SD/-Trp-Leu (DDO)平板, 30℃恒温倒置培养3—5d, 待菌落长出后, 挑取单菌落同时接种到二缺板(SD/-Trp-Leu + X-α-Gal + Aureobasidin A, DDO/X/A)和四缺板(SD/-Trp-Leu-Ade-His + X-α-Gal + Aureobasidin A, QDO/X/A), 30℃培养3—5d, 观菌落生长及菌落颜色变化情况。若转化酵母菌在DDO/X/A和QDO/X/A平板上均能生长, 且菌落颜色为蓝色, 则说明目的蛋白具有自激活活性; 若转化酵母菌在DDO/X/A板上生长且菌落不变蓝, 而在QDO/X/A板上不能生长, 则说明该蛋白不具有自激活能力, 可用于后续蛋白互作实验。
1.2.3 SsRV 5个结构蛋白之间的互作对5个结构蛋白25种互作对(双向蛋白间互作对20个, 自身互作对5个)两两共转酵母感受态细胞, 通过观察转化酵母菌在DDO、DDO/X/A和QDO/X/A平板上的生长及菌落颜色变化情况, 分析5个结构蛋白之间以及蛋白自身的互作情况。每组互作对均设pGBKT7-53 & pGADT7-T共转感受态细胞为阳性对照组, 设pGBKT7-lam & pGADT7-T共转转感受态细胞, 以及pGBKT7 & pGADT7共转感受态细胞为阴性对照组。
2 实验结果 2.1 阳性克隆菌株PCR检测结果重组载体目的基因VPx的PCR及双酶切鉴定如图 1所示。从图中可以看出, 扩增产物及酶切产物中均出现与5个目的片段预期分子量相符的条带, 说明目的基因被成功引入猎物载体pGADT7和诱饵载体pGBDT7。
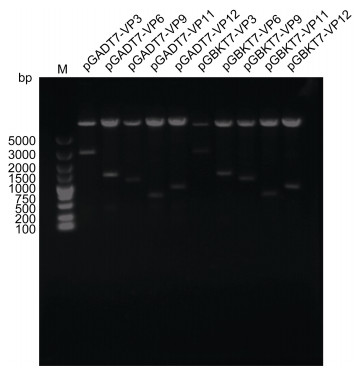 |
| 图 1 重组猎物载体及诱饵载体双酶切及PCR鉴定 Fig. 1 Digestion and PCR identification of recombined prey vectors and bait vectors 注: M为DNA分子量标准 |
含重组载体AD-VP3、AD-VP6、AD-VP9、AD-VP11或AD-VP12的阳性克隆, 以及含BD-VP3、BD-VP6、BD-VP9、BD-VP11或BD-VP12的阳性克隆在培养基中, 经30℃振荡培养24 h后, 测得菌液OD600均大于0.8, 表明猎物蛋白及诱饵蛋白对酵母细胞均无毒性作用, 可用于后续实验。
2.3 结构蛋白自激活检测将重组载体BD-VPx和载体pGADT7, 以及将载体pGBKT7和重组载体AD-VPx两两共转酵母菌, 通过判断His3、Ade2、MEL1报告基因是否表达来判断目的蛋白是否具有自激活活性。如果目的蛋白具有自激活活性, 将会导致转化酵母菌中报告基因His3和Ade2的表达, 酵母菌因此能够在缺失His和Ade的QDO/X/A培养基上生长, 同时由于蛋白自激活导致MEL1基因的表达会促使菌落表面的X-α-Gal变为蓝色。反之, 如果目的蛋白不具备自激活活性, DDO/X/A平板上的菌落不会变蓝, 亦不能在QDO/X/A培养基上生长。实验结果显示(图 3, 图 3), 所有的实验组酵母菌均能在DDO和DDO/X/A平板上生长, 且在DDO/X/A平板上菌落颜色不变蓝, 说明重组捕获载体和重组诱饵载体均成功转入酵母菌, 同时也初步表明5个结构蛋白无论在诱饵载体, 还是在猎物载体中均无自激活活性。更加严格的QDO/X/A平板筛选结果表明, 酵母菌均不能在QDO/X/A板上生长, 进一步说明目的蛋白没有自激活活性。阳性对照组和阴性对照组与预期结果相符, 即阳性组均能够在DDO、DDO/X/A和QDO/X/A平板上生长, 且菌落在DDO/X/A和QDO/X/A平板上均呈现蓝色, 而阴性对照组能够在DDO和DDO/X/A平板上生长, 但不能在QDO/X/A平上生长, 且菌落在DDO/X/A平板上不变蓝。
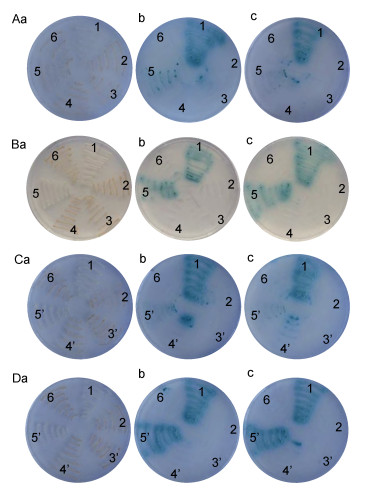 |
| 图 3 酵母双杂交分析结构蛋白两两互作 Fig. 3 Y2H screening of binary interaction in SsRV structural proteins 注: A. AD-VP11 & BD-VP6; B. AD-VP11 & BD-VP12; C. BD-VP11 & AD-VP6; D. BD-VP11 & AD-VP12。a. DDO培养基; b. DDO/X/A培养基; c. QDO/X/A培养基。1.阳性对照pGBKT7-53 & pGADT7-T; 2.阴性对照pGBKT7-Lam & pGADT7-T; 3.自激活验证pGADT7-VP11 & pGBKT7; 4.自激活验证pGBDT7-VPx & pGAKT7; 5. pGAKT7-VP11 & pGBDT7-VPx; 6.阴性对照pGBKT7 & pGADT7; 3’.自激活验证pGBDT7-VP11 & pGAKT7; 4’.自激活验证pGADT7-VPx & pGBKT7; 5’. pGBKT7-VP11 & pGADT7-VPx。x代表6或12 |
将5种蛋白25对互作对分别两两转化感受态酵母Y2H Gold, 并分别涂布于DDO、DDO/X/A和QDO/X/A平板, 在30℃恒温培养箱中培养3—5d, 观察记录转化酵母菌生长及颜色变化。如果转化酵母菌能在DDO和DDO/X/A平板上生长, 而不能在QDO/X/A板上生长, 且菌落在DDO/X/A平板上不变蓝, 则说明目的蛋白之间或自身不存在相互作用。反之, 如果转化菌能在DDO、DDO/X/A和QDO/X/A平板上生长, 且菌落在DDO/X/A和QDO/X/A平板上均呈现蓝色, 则说明蛋白存在相互作用。如图 2所示, AD-VP3 & BD-VP3、AD-VP11 & BD-VP11或AD-VP12 & BD-VP12两两共转的酵母菌均能在DDO、DDO/X/A和QDO/X/A平板上生长, 且菌落在DDO/X/A和QDO/X/A平板上均呈现蓝色, 说明VP3、VP11和VP12存在自身互作。AD-VP11 & BD-VP6、AD-VP11 & BD-VP12、BD-VP11 & AD-VP6或BD-VP11 & AD-VP12两两共转的酵母菌均能在DDO、DDO/X/A和QDO/X/A平板上生长, 且菌落在DDO/X/A和QDO/X/A平板上均呈现蓝色, 说明VP11 & VP6、VP11 & VP12存在双向互作(图 3)。此外, 阳性对照组和阴性对照组与预期结果相符(图 2, 图 3), 说明本实验结果可靠。
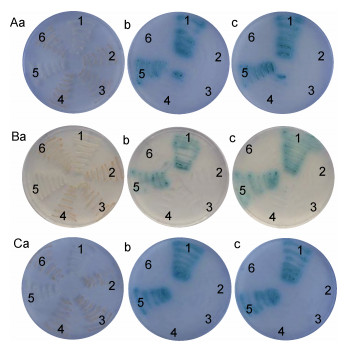 |
| 图 2 酵母双杂交分析结构蛋白自身互作 Fig. 2 Y2H screening of self-association interaction in SsRV structural proteins 注: A. VP3 & VP3; B. VP11 & VP11; C. VP12 & VP12。a. DDO培养基; b. DDO/X/A培养基; c. QDO/X/A培养基。1.阳性对照pGBKT7-53 & pGADT7-T; 2.阴性对照pGBKT7-Lam & pGADT7-T; 3.自激活检测pGBKT7-VPx & pGADT7; 4.自激活检测pGBKT7 & pGADT7-VPx; 5. pGBKT7-VPx & pGADT7-VPx; 6.阴性对照pGBKT7 & pGADT7。x代表3, 11或12 |
SsRV全病毒SDS-PAGE图谱显示, VP3、VP6、VP12蛋白条带最浓, 其次为VP9和VP11蛋白, 而VP1条带最弱(Chen et al, 2012; Yuan et al, 2016)。同源分析表明, 优势蛋白VP6与LNV病毒外衣壳蛋白VP4蛋白的同源性为25%, 与MRV-2病毒吸附蛋白sigma1的同源性为25%。糖基化位点预测显示, VP6蛋白含有呼肠孤病毒外衣壳蛋白普遍所具有的糖基化位点。有文献报道, 外衣壳蛋白上的糖基化位点有助于病毒与细胞表面的吸附与侵入(Shaw et al, 1996)。基于上述分析, 推断该蛋白为病毒的外衣壳蛋白(Chen et al, 2012)。酵母双杂交结果显示VP6蛋白能够与VP11蛋白互作。VP11蛋白除了能与VP6蛋白互作外, 还能与VP12蛋白互作。免疫电镜结果表明, VP12蛋白定位于病毒粒子表面, 是病毒的外衣壳蛋白(Xu et al, 2015)。对于SsRV结构蛋白VP9, SVPProt蛋白功能预测将该蛋白归类于分子内转移酶家族, 该家族成员具有激酶活性, 也具有二磷酸和核苷酸的转移活性。此外, VP9蛋白部分序列RNA结合蛋白及甲基化转移酶存在同源性, 以此推断该蛋白可能为病毒的甲基转移酶, 参与了SsRV mRNA的5'端加帽(Chen et al, 2012; 张昭等, 2014)。然而, MCRV全病毒的SDS-PAGE电泳图谱中未发现VP9蛋白(Deng et al, 2012), 究其原因可能是VP9作为病毒转录复合体的组成成分, 由于其在MCRV病毒粒子中的含量较低而被遗漏。酵母双杂交未发现VP9存在自身互作, 也未发现该蛋白与其它结构蛋白存在相互作用。对于SsRV VP3蛋白, SVMProt蛋白功能预测将该蛋白归类于催化形成碳-碳键的酶家族。当对其它呼肠孤病毒蛋白进行SVMProt预测时, 发现唯有BTV内衣壳蛋白VP3属于催化形成碳-碳键的酶家族, 因此我们推测VP3蛋白可能是SsRV内衣壳蛋白组成成分(Chen et al, 2012)。酵母双杂交结果显示, VP3蛋白存在自身互作, 这为内衣壳蛋白的多聚体的形成提供了可能。综合上述分析, 笔者推测: VP6、VP11和VP12均为SsRV的外衣壳组成成分, 而VP3和VP9分别为SsRV内衣壳和病毒转录复合体的组成成分。
呼肠孤病毒衣壳蛋白通过自身互作形成多聚体(Liu et al, 2007)。然而, 酵母双杂交结果表明, 作为潜在外衣壳蛋白的VP6并不存在自身互作。病毒外衣壳蛋白与内衣壳蛋白间往往通过相互作用来维持结构的稳定性, 然而酵母双杂交并未检测到潜在内衣壳蛋白VP3与外衣壳蛋白之间的互作。笔者认为导致上述结果的可能原因是由于酵母双杂交系统的局限性。在酵母细胞中, 通过验证报告基因的表达情况能够精确验证蛋白之间微弱的相互作用关系, 且其互作是在活细胞中进行能够在一定程度上代表细胞内的真实情况, 此外目的基因在酵母细胞中的产物积累效应, 能够显著提高检测的灵敏性, 对于微弱的或暂时性的相互作用也能够呈现出表观的效果(Rajagopala, 2015)。虽然该技术在蛋白互作研究方面具有上述诸多优点, 但其仍具有一定的局限性, 首先是诱饵蛋白与猎物蛋白的相互作用发生在细胞核内, 对于一些不能入核的蛋白无法检测, 其次是酵母中表达的蛋白质只能进行有限的翻译后修饰, 对于一些需要多种翻译后修饰的蛋白质作用有限(Rajagopala, 2015)。因此, 后续实验还需要通过GST-pull down, Far-western和双分子荧光互补等其他蛋白互作技术, 对SsRV结构蛋白互作进一步分析。
4 结论本研究采用Clontech公司的Matchmaker系统对SsRV 5个结构蛋白的互作进行了分析。利用Matchmaker系统携带的三个启动子, 四个报告基因(His3, Ade2, MEL1和AUR1-C2), 以及显色反应的筛选, 有效地减少了假阳性的发生。另外, 通过两个方向的酵母双杂交实验, 验证了蛋白之间的相互作用, 结果真实可靠。VP3, VP6, VP9, VP11和VP12五个结构蛋白自身及之间的相互作用, 与维系SsRV病毒形态发生、结构稳定性有密切关系。研究结果为进一步探讨该病毒对宿主的感染和致病机制奠定了坚实的基础。
| 陈吉刚, 杨季芳, 王海丽, 等, 2008. 养殖锯缘青蟹呼肠孤样病毒粒子的电镜观察. 海洋学研究, 26(4): 93–96 |
| 张昭, 朱四东, 井晓欢, 等, 2014. 锯缘青蟹呼肠孤病毒p40蛋白的体外表达与单克隆抗体制备. 上海海洋大学学报, 23(4): 487–491 |
| Chen J G, Xiong J, Cui B J, et al, 2012. Molecular characterization of eight segments of Scylla serrata reovirus (SsRV) provides the complete genome sequence. Archives of Virology, 157(8): 1551–1557 DOI:10.1007/s00705-012-1298-7 |
| Chen J G, Xiong J, Yang J F, et al, 2011. Nucleotide sequences of four RNA segments of a reovirus isolated from the mud crab Scylla serrata provide evidence that this virus belongs to a new genus in the family Reoviridae. Archives of Virology, 156(3): 523–528 DOI:10.1007/s00705-010-0852-4 |
| Deng X X, Lü L, Ou Y J, et al, 2012. Sequence analysis of 12 genome segments of mud crab reovirus (MCRV). Virology, 422(2): 185–194 DOI:10.1016/j.virol.2011.09.029 |
| Huang Z W, Deng X X, Li Y Y, et al, 2012. Structural insights into the classification of Mud Crab Reovirus. Virus Research, 166(1-2): 116–120 DOI:10.1016/j.virusres.2012.02.025 |
| Liu H J, Wei C H, Zhong Y W, et al, 2007. Rice black-streaked dwarf virus outer capsid protein P10 has self-interactions and forms oligomeric complexes in solution. Virus Research, 127(1): 34–42 DOI:10.1016/j.virusres.2007.03.017 |
| Miyazaki N, Hagiwara K, Naitow H, et al, 2005. Transcapsidation and the conserved interactions of two major structural proteins of a pair of phytoreoviruses confirm the mechanism of assembly of the outer capsid layer. Journal of Molecular Biology, 345(2): 229–237 DOI:10.1016/j.jmb.2004.10.044 |
| Rajagopala S V, 2015. Mapping the protein-protein interactome networks using yeast two-hybrid screens. Advances in Experimental Medicine and Biology, 883: 187–214 DOI:10.1007/978-3-319-23603-2 |
| Shaw A L, Samal S K, Subramanian K, et al, 1996. The structure of aquareovirus shows how the different geometries of the two layers of the capsid are reconciled to provide symmetrical interactions and stabilization. Structure, 4(8): 957–967 DOI:10.1016/S0969-2126(96)00102-5 |
| Weng S P, Guo Z X, Sun J J, et al, 2007. A reovirus disease in cultured mud crab, Scylla serrata, in southern China. Journal of Fish Diseases, 30(3): 133–139 DOI:10.1111/jfd.2007.30.issue-3 |
| Xu H D, Su H J, Zou W B, et al, 2015. Identification of mud crab reovirus VP12 and its interaction with the voltage-dependent anion-selective channel protein of mud crab Scylla paramamosain. Fish & Shellfish Immunology, 44(1): 224–231 |
| Yuan Y Y, Fan D Y, Zhang Z, et al, 2016. Identification and RNA segment assignment of six structural proteins of Scylla serrata reovirus. Virus Genes, 52(4): 556–560 DOI:10.1007/s11262-016-1319-9 |
| Yuan Y Y, Fan D Y, Zhu S D, et al, 2017. Identification and characterization of host cell proteins interacting with Scylla serrata reovirus non-structural protein p35. Virus Genes, 53(2): 317–322 DOI:10.1007/s11262-016-1418-7 |
| Zhang Z, Yuan Y Y, Fan D Y, et al, 2015. Scylla serrata reovirus p35 protein expressed in Escherichia coli cells alters membrane permeability. Virus Genes, 51(1): 69–76 DOI:10.1007/s11262-015-1218-5 |
 2017, Vol. 48
2017, Vol. 48


