
中国海洋湖沼学会主办。
文章信息
- 王先锋, 林承刚, 许强, 宋肖跃, 张海建, 汝少国, 杨红生. 2017.
- WANG Xian-Feng, LIN Cheng-Gang, XU Qiang, SONG Xiao-Yue, ZHANG Hai-Jian, RU Shao-Guo, YANG Hong-Sheng. 2017.
- 利用18S rDNA分子方法分析浒苔绿潮过境期间长牡蛎摄食情况的变化
- IMPACT OF ENTEROMORPHA PROLIFERA GREEN TIDE ON OYSTER FEEDING USING 18S rDNA MOLECULAR METHOD
- 海洋与湖沼, 48(6): 1362-1370
- Oceanologia et Limnologia Sinica, 48(6): 1362-1370.
- http://dx.doi.org/10.11693/hyhz20170700189
-
文章历史
- 收稿日期:2017-07-20
- 收修改稿日期:2017-09-28
2. 中国科学院海洋生态与环境科学重点实验室(中国科学院海洋研究所) 青岛 266071;
3. 海南大学 海口 570228;
4. 日照市渔业通讯管理站 日照 276800
2. CAS Key Laboratory of Marine Ecology and Environmental Sciences, Institute of Oceanology, Chinese Academy of Sciences, Qingdao 266071, China;
3. Hainan University, Haikou 570228, China;
4. Rizhao Fishery Communication Management Station, Rizhao 276800, China
滤食性海洋双壳贝类在近岸大型底栖生物群体中处于优势地位, 它们通常充当“生态系统工程师”(Jones et al, 1996), 并可以在浮游生产者和底栖生物之间建立重要联系。双壳贝类的食物营养级很广, 但对它的了解却较少。生态学家们曾尝试研究出它们全部的食物摄取情况, 但他们面临的最直接挑战就是它们所摄取的食物过于微小, 还有其随时空的变化((Lotsy, 1895; Galtsoff, 1964)。通常判断滤食性贝类的食物成分非常困难, 这是因为传统的显微镜检测所需的样本量较大, 同时还存在着较多胃含物碎片难以鉴别的问题(Fukumori et al, 2008)。早期有关海洋双壳贝类的食物成分调查表明其食物源中有重要的初级生产者, 例如硅藻和鞭毛藻(Galtsoff, 1964)。大量近期调查研究发现了其食物源也含有次级消费者, 例如桡足类, 无脊椎动物幼虫(Davenport et al, 2000; Lehane et al, 2006)。了解双壳贝类的食物组成对于研究海岸带食物网和生态基础管理等方面至关重要。尤其对判断海洋牧场生态系统将会发生怎样的改变尤其重要(Fukumori et al, 2008)。
传统分类学将浒苔划分为绿藻门、绿藻纲、石莼目、石莼科、浒苔属(Enteromorpha), 但近年来取消了浒苔属, 而将原浒苔属物种合并为石莼属(Ulva)(Hiraoka et al, 2004), 并认为导致绿潮暴发的优势藻类以石莼属为主(Callow et al, 1997; Hiraoka et al, 2004), 因此本研究也将石莼属和浒苔属归为一类。浒苔属和石莼属等大型海洋绿藻大量增殖而导致的海洋生态异常现象, 被称为“绿潮”。绿潮暴发会引发一系列次生灾害, 如藻体腐败散发出难闻气味污染环境, 藻类堆积沉降会引起缺氧和底质腐败, 导致生物死亡。位于日照的平岛海域是重要的海洋牧场, 每年大规模浒苔都在此有规律的暴发, 浒苔的出现对当地渔民的劳动生产造成很大干扰。平岛周围自然生长着大量的牡蛎和贻贝这两种重要的经济贝类, 其中属长牡蛎(Ostrea gigas Thunberg)分布最广、数量最多。长牡蛎属于滤食性贝类, 目前并没有研究表明浒苔碎屑是否可以作为长牡蛎食物源或对长牡蛎食物源产生影响。浒苔的成熟藻体较大, 高可达1m, 因此无法作为长牡蛎的食物源, 但在海风海浪和浒苔自身衰败的作用下会产生浒苔丝状体碎屑, 这些悬浮在水体中的有机物质大小可以为长牡蛎所摄食, 并且浒苔的成熟藻体能产生大量的微观繁殖体, 浒苔微观繁殖体作为浒苔生长发育的早期形成产物, 主要包括孢子、配子、合子这些无细胞壁保护的裸露单细胞, 在适宜的温盐等条件下, 微观繁殖体遇到合适附着基就会固着, 进而萌发生长成藻丝体, 固着和萌发是浒苔生长发育的必要阶段(Chapman, 1986; Fletcher et al, 1992; Santelices et al, 2002)。而微观繁殖体的直径与小型浮游植物相近。考虑到滤食性贝类以水体中浮游生物和有机碎屑等为主要食物(张继红, 2008), 水体中浒苔丝状体碎屑和微观繁殖体增加, 贝类的食物来源可能发生变化。本研究通过高通量DNA测序技术研究了长牡蛎的真核食物成分, 从长牡蛎的摄食状况角度为预测相关生态风险提供理论依据与数据支持。
1 材料与方法 1.1 样品收集采样区域站位见图 1。2016年在中国海州湾平岛太平湾(34°38′N, 119°54′E)同一地点共采集4次不同月份的长牡蛎(Ostrea gigas Thunberg)样品, 共41个, 分别是5月10个, 6月11个, 7月6个, 10月14个。2016年平岛浒苔暴发期为6月25日—7月15日, 这四次样品的采集时间涵盖了浒苔暴发前期, 浒苔暴发期和浒苔暴发后期三个阶段。平岛水温5月15.88℃, 6月20.32℃, 7月27.1℃, 10月22.24℃。盐度5月30.42, 6月30.66, 8月30.55, 10月30.48。叶绿素5月2.5μg/L, 6月5.4μg/L, 7月3.8μg/L, 10月3.2μg/L。平岛面积0.148km2, 水深15m, 最近处距离海岸42km。所有调查研究中采集的长牡蛎均为自然状态下繁殖生长的长牡蛎, 并且岛附近没有其他河流影响。
 |
| 图 1 采样站位区域分布图 Fig. 1 Map of sampling site |
采集长牡蛎后用尺子量取其背腹缘最大距离并记录。
1.3 浮游生物采集在长牡蛎样品收集区域附近设置3个采集点, 使用浮游植物网和浮游动物网垂直采集浮游生物, 其中浮游植物网网径为76μm, 口径为37cm, 浮游动物网网径为500μm, 口径为50cm。采集的浮游生物样品加入甲醛固定带回实验室等待检测。
1.4 胃含物样品所有采集的长牡蛎都现场解剖, 提取消化道部分, 转移至1.5mL冻存管中并做标号, 将冻存管投于液氮罐中带回实验室等待进一步处理。
1.5 基因组DNA提取采用CTAB方法对样本的基因组DNA进行提取(Tel-Zur et al, 1999), 之后利用琼脂糖凝胶电泳检测DNA的纯度和浓度, 取适量的样品于离心管中, 使用无菌水稀释样品至1ng/μL。
1.6 PCR扩增以稀释后的基因组DNA为模板, 使用特异引物扩增18S V4区DNA。通常引物序列为528F (5′-GCGG TAATTCCAGCTCCAA-3′)和706R (5′-AATCCRAGA ATTTCACCTCT-3′) ( Cheung et al, 2010)。使用New England Biolabs公司的Phusion® High-Fidelity PCRMaster Mix with GC Buffer酶和缓冲液。使用高效和高保真的酶进行PCR, 能确保扩增效率和准确性(Magoč et al, 2011)。最后设置PCR反应体系和程序(30μL): Phusion Master Mix(2×) 15μL, Primer (2μmol/L) 3μL (6μmol/L), gDNA (1ng/μL) 10μL (5—10ng), H 2O2μL。反应程序: 98℃预变性1min; 30个循环包括(98℃, 10s; 50℃, 30s; 72℃, 30s); 72℃, 5min。PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测; 根据PCR产物浓度进行等质量混样, 充分混匀后使用1×TAE浓度2%的琼脂糖胶电泳纯化PCR产物, 选择主带大小在400—450bp之间的序列, 割胶回收目标条带。产物纯化试剂盒使用的是Thermo Scientific公司GeneJET胶回收试剂盒。
1.7 文库构建和上机测序使用Illumina公司TruSeq DNA PCR-Free Library Preparation Kit建库试剂盒进行文库的构建, 构建好的文库经过Qubit定量和文库检测, 合格后, 使用HiSeq进行上机测序。
1.8 数据分析测序得到的原始数据(Raw Data), 存在一定比例的干扰数据(Dirty Data), 为了使信息分析的结果更加准确、可靠, 首先对原始数据进行拼接、过滤, 得到有效数据(Clean Data)。
基于有效数据进行OTUs (Operational taxonomic units)聚类和物种分类分析, 根据OTUs聚类结果, 一方面对每个OTU的代表序列做物种注释, 得到对应的物种信息和基于物种的丰度分布情况。同时, 对OTUs进行丰度、Alpha多样性计算等分析, 以得到样品内物种丰富度和均匀度信息、不同样品或分组间的共有和特有OTUs信息等(Bokulich et al, 2013; Mueller et al, 2014)。另一方面, 通过PCoA和PCA等降维图和样品聚类树进行展示(Backeljau et al, 1996; Zhang et al, 2016)。
2 结果 2.1 壳高每个月份长牡蛎的壳高是不同的但是都在成年长牡蛎范围之内, 其壳高平均为49.16mm, 其中最小的45.0mm, 最大的53.1mm。这四个月的长牡蛎可能的年龄结构变化或者长牡蛎的增长率未知。
2.2 浮游生物丰度浮游植物(表 1)和浮游动物(表 2)的结果显示本地浮游动物生物丰度远低于浮游植物。其中硅藻最多的月份是5月, 甲藻最多的月份为7月。浮游植物各个月的丰度分别是5月55561.7cells/m3, 6月13035cells/ m3, 7月81927.7cells/m3, 10月62508cells/m3。浮游动物各个月份的丰度分别为5月304.7cells/m3, 6月64.77cells/m3, 7月1638.7cells/m3, 10月140.7cells/m3。
| 浮游植物 | 站位 | ||||||||||||||
| 5月 | 6月 | 7月 | 10月 | ||||||||||||
| A1 | A2 | A3 | A1 | A2 | A3 | A1 | A2 | A3 | A1 | A2 | A3 | ||||
| 硅藻 | 55080 | 37291 | 52189 | 9840 | 4017 | 21920 | 43002 | 39187 | 60198 | 10287 | 29102 | 18917 | |||
| 甲藻 | 6480 | 10927 | 4718 | 395 | 701 | 2018 | 59810 | 32761 | 10825 | 297 | 1078 | 2827 | |||
| 金藻 | — | — | — | 84 | 84 | 46 | — | — | — | — | — | — | |||
| 浮游动物 | 站位 | ||||||||||||||
| 5月 | 6月 | 7月 | 10月 | ||||||||||||
| A1 | A2 | A3 | A1 | A2 | A3 | A1 | A2 | A3 | A1 | A2 | A3 | ||||
| 刺胞动物 | 4.2 | 2.1 | 2.4 | 18 | 7.5 | 14 | 22 | 14 | 5 | 9 | 3 | 1 | |||
| 桡足类 | 288.4 | 104.1 | 219 | 27.8 | 12.2 | 3 | 1300 | 309 | 741 | 138 | 42 | 69 | |||
| 翼足类 | 32 | 12 | 18 | — | — | — | — | — | — | — | — | — | |||
| 毛颚类 | 93.8 | 49.2 | 30.2 | 11.4 | 4 | 1 | 640 | 381 | 419 | 29 | 21 | 17 | |||
| 被囊类 | 3 | 2 | 4 | — | — | — | — | — | — | — | — | — | |||
| 浮游幼体 | 7.6 | 20 | 22 | 38.4 | 44 | 12 | 466 | 217 | 402 | 18 | 21 | 13 | |||
| 十足类 | — | — | — | 0.2 | 0 | 1.8 | — | — | — | — | — | — | |||
| 海樽类 | — | — | — | — | — | — | — | — | — | 27 | 10 | 4 | |||
剔除无效样品和消化道内容物中长牡蛎自身的OTU后总共获得53347个有效序列片段, 每一个样品中的有效序列片段数量从2984到10742不等, 平均为4445.58, OTU的数量展示了胃含物中的生物物种丰度情况。
所有样品中总共获得105个OTU, 2016年5月(WH1)有57—80个OTU, 6月(WH2)有38—65个OTU, 7月(WH3)有44—56个OTU, 10月(WH4)有45—84个OTU。根据OTUs的数目做稀释曲线和Chao1曲线(图 2a, b)显示所有样品都趋向饱和, 如图显示, 所有样品之间的Shannon多样性指数差异不显著(P > 0.05, 图 2c)。这些结果显示浒苔发生前和浒苔发生后长牡蛎胃含物的真核生物丰度差异并不显著, 但是胃含物成分不同。
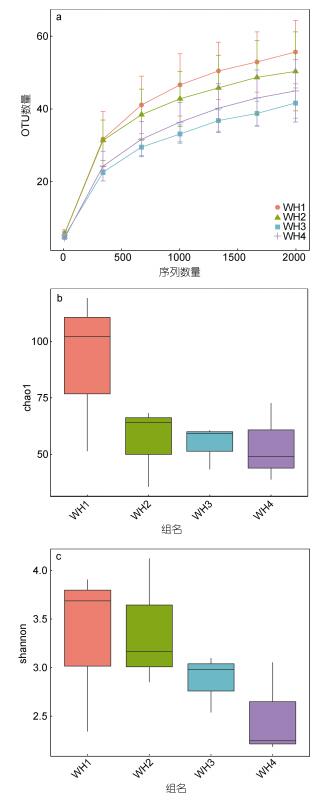 |
| 图 2 样品稀释曲线(a)、Chao1指数(b)、4组样品的Shanon多样性指数(c) Fig. 2 Richness and diversity: a. Sample rarefaction curves. b. Chaos1 curves. c. Shannon-Wiener indices of the four groups 注: WH1: 5月样品; WH2: 6月样品, WH3: 7月样品, WH4: 10月样品 |
所有样品中共鉴定出15个门类, WH1样品11个门, WH2样品12个门, WH3样品11个门, WH4样品14个门。每组样品中未鉴别的真核生物比例为5.9%—63.7%。前10个丰度最高的门类各占总体的99.082%—99.16%, 如图 3。各个胃含物样品之间门水平上的丰度差异显著, 其中链形植物门(Streptophyta)是长牡蛎胃含物中整体丰度最高的门类, 最低23.86%, 最高49.66%, 平均35.10%, 链型植物是植物中的一大类群, 包括轮藻门(广义上的轮藻)和有胚植物(现存的陆生植物:苔藓维管植物)两大类。5月(WH1)胃含物占20%以上的门类有链形植物门和子囊菌门(Ascoaamycota)分别占32.65%和26.32%, 占10%以上的门类有绿藻门(Chlorophyta)和囊泡虫门(Protalveolata)分别占14.31%和17.7%。在6月(WH2)胃含物占20%以上的门类有链形植物门(Streptophyta)为49.66%, 占10%以上的门类是子囊菌门(Ascoaamycota)占18.14%, 7月(WH3)胃含物占20%以上的门类有链形植物门和囊泡虫门(Protalveolata)分别占34.07%和44.72%。10月(WH4)胃含物样品中占20%以上的门类为链形植物门23.89%。所有组样品中除了WH3中囊泡虫门含量最多, 其他3组样品中都是链形植物门含量最多。
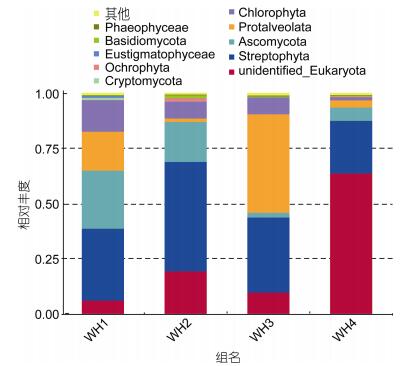 |
| 图 3 胃含物中10个丰度最高门类相对丰度图 Fig. 3 Relative abundance of 10 most abundant phyla in stomach content 注: “其他”代表除了最高的10个门外所有物种丰度 |
结果表明在OTU水平上样品之间差异明显, 在表 3中显示了OTU丰度大于10%的样品组, 如图可以得知丰度大于10%的物种只有5种。4组胃含物样品中的链形植物门(Streptophyta)都大于20%, 表明链形植物门是长牡蛎胃含物中的主要食物成分。
| 门类 | 最高丰度(%) | 丰度最高的组 | 丰度大于10% 的组数量 |
| 链形植物门 | 49.66 | WH2 | 4 |
| 子囊菌门 | 26.32 | WH1 | 2 |
| 囊泡虫门 | 44.71 | WH3 | 2 |
| 绿藻门 | 14.34 | WH1 | 1 |
根据前10个丰度最高的属聚类分析发现5月、6月、7月和10月真核生物成分差别明显, 表明胃含物中真核生物成分显著的季节性不同, 有一些成分只能鉴别到纲。如图 4, 并没有石莼属成分。WH1中梨属(Pyrus), 四胞藻纲(Trebouxiophyceae)达到最多; WH2样品中座囊菌属(Dothidea)含量都达到所有样品的最高值。WH3样品中共甲藻属(Syndiniales)、菥蓂属(Thlaspi)成分达到最多。WH4样品中沟鞭藻属(Biecheleria)的含量达到最高值。
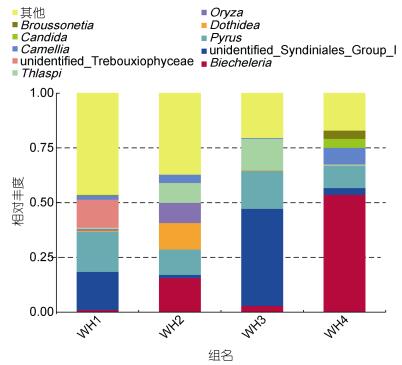 |
| 图 4 属水平20个丰度最高门类相对丰度图 Fig. 4 The relative abundance of 20 most abundant genus in stomach content 注: “其他”代表除了最高的20个属外所有物种丰度 |
通过PCoA(主成分分析)也可以确定样品之间的关系(图 5)。PCoA结果显示4组样品之间是不同的, 但也有相似之处。根据第一主成分可以看出5月和6月份样品较为接近, 从第二主成分来看7月与其他月份差别最大。
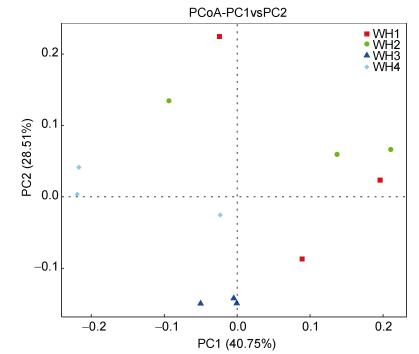 |
| 图 5 组间PCoA主坐标分析 Fig. 5 PCoA plots among the groups |
长牡蛎属于滤食性贝类, 且食物源较复杂。使用传统的显微镜技术鉴定这个物种的胃含物真核生物成分非常困难。我们使用了高通量DNA序列技术作为研究方法来鉴定长牡蛎胃含物中真核食物成分。目前已有研究通过18S rDNA方法鉴别海参胃含物的真核生物组成(Zhang et al, 2016)。我们共鉴定出15个门类的105个OTU, 每组样品的OTU数量范围从38—84不等, 代表了11—14个门, 而在属水平共鉴定出105个属, 表明长牡蛎胃含物真核生物成分较复杂, 食物源丰富。我们对石莼发生前后长牡蛎胃含物的研究结果显示并没有石莼属成分。因此表明石莼属不太可能作为长牡蛎的直接食物源。
现已有的关于胃含物的鉴定方法有COI(Leray et al, 2013; Bade et al, 2014; Brown et al, 2014), ITS (Bachy et al, 2013)和18S rDNA序列分析(Martin et al, 2006; Suzuki et al, 2008; Maloy et al, 2009; Riemann et al, 2010)三种。但是COI和ITS扩增片段与18S rDNA扩增相比更长, 且变异度较高, 只适用于摄食水平单一的物种胃含物分析, 由于长牡蛎的摄食成分较复杂因此选用18S rDNA序列分析。使用这种方法可以鉴别到属水平而运用传统的显微镜检查大多只能观察到门水平, 例如硅藻门(Lotsy, 1895); Galtsoff, 1964), 因此这种方法更加精确全面, 可以为海洋动物摄食研究提供宝贵经验(Leray et al, 2013)。但这种方法也有其缺点, 一般食物在体内经过几个小时或者几天就会被完全消化, 因此这种方法只能提供短期的胃含物成分研究, 具有一定偶然性, 而稳定同位素分析和脂肪酸分析等方法可以对长牡蛎几个月内甚至几年内主要的食物来源进行对比分析, 想要研究长期的食物变化还需要结合稳定同位素分析和脂肪酸分析等方法共同分析。
研究结果显示浒苔过境前后4组不同时间相同地点的胃含物成分有着显著变化。在四组样品中通过18S rDNA分子方法分析长牡蛎的胃含物结果是不同的, 考虑到采样时间跨越春季夏季和秋季三个季节, 可能导致胃含物成分的显著不同但不同组之间的生物丰度变化不显著(P > 0.05), 胃含物中基本都是初级生产者而未发现次级消费者, 根据我们的检测结果显示长牡蛎主要摄食被子植物和小型浮游植物(如硅藻)这些初级生产者, 这与Fukumori等(2008)的研究结果有不一致之处。在之前的研究中发现长牡蛎具有广泛的营养可塑性, 因其食物源不止含有初级生产者还包括藤壶类, 甲壳类等次级消费者(Davenport et al, 2000; Lehane et al, 2006; Fukumori et al, 2008), 其不适合充当是一个初级生产力的表征。本次研究发现了胃含物中具有很高比例的高等植物, 例如菥蓂属、梨属、稻属, 这种情况在已有的相关研究中并没有提到过, 但并没有检测出浮游动物成分, 这可能与浮游动物丰度较低有关, 根据浮游动物检测结果(表 2)显示浮游动物与浮游植物相比含量显著偏低, 这在某种程度上也说明了长牡蛎的营养可塑性, 表明长牡蛎的食物源不仅仅来源于海洋而且还摄食陆源有机质。由于本次研究地点位于离岸小岛, 最近处距离海岸42km, 而且附近没有其他河流注入因此这些高等植物不太可能来自大陆, 推测高含量的被子植物可能来自小岛自身的植被, 平岛面积0.148km2, 目前在岛上没有发现种植大量稻属植物和梨属植物, 但岛周围地势险恶无法近距离观察, 不排除有野生稻属植物和梨属植物在此生长。平岛周围海域属于当地公司承保养殖区, 常年有4—6条渔船停靠或活动在岛附近, 船员会将吃剩的饭菜或变质的食物直接倒入海水中, 经过海流的传播, 这些剩菜剩饭的降解颗粒可能被附近的长牡蛎滤食, 可能会造成一定的污染, 推测这也可能是稻属植物可能的来源之一。在岛上有大量军事碉堡等建筑, 并且在岛的最高处流动住有2—5名士兵, 其生活垃圾都集中处理不会倒入海中, 因此不太可能来自岛上垃圾。菥蓂属通常是一种野外生长的植物其花粉会随着海风传播到海水中, 其叶子等植物组织也会掉落腐败后随着雨水流入海水中被长牡蛎滤食, 这可能是长牡蛎滤食大量菥蓂属的原因。因此未来使用双壳贝类作为初级生产力的表征时, 应该考虑到长牡蛎的生活史和摄食模式的不同。
有研究表明浒苔的繁殖多以配子形式出现, 每平方厘米单层藻体叶片可以产生2.84×106—6.62×106个孢子或配子(陈群芳等, 2011)。也有研究显示浒苔的生殖囊释放出的游孢子大小约为(9—12.5)μm× (5.3—6)μm; 配子相对较小, 约(5.8—8.5)μm×(2—2.8)μm (张华伟等, 2011), 孢子或两个配子结合后形成的合子, 合子以成熟藻体为附着基继续发育成一株新的藻体。一般浮游生物可分为六类:巨型浮游生物, 大于1cm, 如海蜇; 大型浮游生物, 5—10mm, 如大型桡足类、磷虾类; 中型浮游生物, 1—5mm, 如小型水母、桡足类; 小型浮游生物, 50μm—1mm, 如硅藻、蓝藻; 微型浮游生物, 5—50μm, 如甲藻、金藻; 超微型浮游生物, 小于5μm, 如细菌。因此我们推测浒苔繁殖体的粒径与小型浮游植物和微型浮游植物相当。从浒苔微观繁殖体的体积来看其可以作为长牡蛎的食物源, 由于本研究所使用的浮游植物网的网径为76μm, 大于一般的浒苔微观繁殖体大小, 因此这可能是采集浮游植物时并没有检测到微观繁殖体的原因, 但是大量浒苔出现在平岛海域后, 长牡蛎的胃含物中也没有检测到石莼属或浒苔属成分, 推测可能有两种可能: (1)平岛属于浒苔暴发后自南向北漂移的通道区, 并没有浒苔腐败沉降因此没有碎屑产生或产生量极少, 因此长牡蛎无法摄食浒苔。(2)此时的浒苔未到成熟期并没有微观繁殖体产生, 因此长牡蛎并没有摄食。有研究表明黄海绿潮暴发过程中, 石莼属绿藻的微观繁殖体在海水和沉积物中是广泛分布的, 并且生物量不容忽视。Liu等(2010, 2012)将采集到的微观繁殖体带回实验室内重培养, 对长出的幼苗进行计数从而解决了微观繁殖体的定量问题。张华伟等(2011)曾经指出在绿潮繁盛期, 一株飘浮的浒苔藻体可以产生2.31×107个配子。Wang等(2013)利用荧光定量PCR的方法, 对绿潮暴发期间黄海石莼属绿藻微观繁殖体生物量的时空分布进行了定量研究, 结果发现微观繁殖体的生物量在5月迅速上升, 平均数量为300—105cells/L, 5月中旬达到顶峰, 约为107cells/L。Zhang等(2015)曾指出自由漂浮的浒苔藻体在5月份生物量急剧攀升, 因为浒苔微观繁殖体的分布与水体中的浒苔藻体生物量呈现明显正相关。
根据本次研究记录平岛浒苔出现至海面上可见浒苔消散的时间为6月11日—7月10日, 此时出现的浒苔比其微观繁殖体数量的顶峰时期晚了将近一个月的时间, 虽然有大量浒苔藻体存在, 可能其微观繁殖体数量已经开始减少。也有研究表明浒苔的配子具有正趋光性, 这趋光性使其更容易在光线较强的海面聚集并在漂浮藻体上附着生长, 浒苔成熟体有大量囊状结构, 这些结构有利于藻体更好地漂浮在水面接受光照(张华伟等, 2011)。而长牡蛎生长在潮间带, 当海水涨潮时长牡蛎生活在水下3—6m, 本次研究地点长牡蛎生长在中潮间带区, 只有落潮时长牡蛎才会浮出水面并且只有此时才能采集到长牡蛎样品, 浒苔的这种生长特性与长牡蛎生长区在空间上产生一定分隔。因此也说明了水体中有大量浒苔而长牡蛎胃含物中没有浒苔成分的原因。
4 结论综上所述, 浒苔暴发并不会直接影响长牡蛎的摄食情况, 因此推测浒苔通道区平岛海洋牧场受浒苔危害影响较小, 应该更多关注浒苔漂移后的沉降区域并做好灾害预防工作。使用高通量测序技术可以准确地鉴定胃含物成分但这种方法更多关注的是其短期的摄食情况, 未来研究双壳贝类食物网时应该结合同位素分析和脂肪酸分析等多种分析方法, 这些方法可以关注到贝类的长期摄食情况, 以助于更好地了解双壳贝类食物网结构。由于海州湾大规模浒苔暴发已经呈现常态化, 从长期角度来看可能会影响整个海洋渔业资源环境进而直接或间接对双壳贝类营养成分造成影响, 因此应该全面加强监测浒苔对海洋环境的影响并做好生态风险评估。
| 张华伟, 马家海, 胡翔, 等, 2011. 绿潮漂浮浒苔繁殖特性的研究. 上海海洋大学学报, 20(4): 600–606 |
| 张继红, 2008. 滤食性贝类养殖活动对海域生态系统的影响及生态容量评估. 青岛: 中国科学院海洋研究所博士学位论文, 20-25 http://cdmd.cnki.com.cn/article/cdmd-80068-2008117213.htm |
| 陈群芳, 何培民, 冯子慧, 等, 2011. 漂浮绿潮藻浒苔孢子/配子的繁殖过程. 中国水产科学, 18(5): 1069–1076 |
| Bachy C, Dolan J R, López-García P, et al, 2012. Accuracy of protist diversity assessments:morphology compared with cloning and direct pyrosequencing of 18S rRNA genes and ITS regions using the conspicuous tintinnid ciliates as a case study. ISME J, 7(2): 244–255 |
| Backeljau T, De Bruyn L, De Wolf H, et al, 1996. Multiple UPGMA and neighbor-joining trees and the performance of some computer packages. Mol Biol Evol, 13(2): 309–313 DOI:10.1093/oxfordjournals.molbev.a025590 |
| Bade L M, Balakrishnan C N, Pilgrim E M, et al, 2014. A genetic technique to identify the diet of cownose rays, Rhinoptera bonasus:analysis of shellfish prey items from North Carolina and Virginia. Environ Biol Fish, 97(9): 999–1012 DOI:10.1007/s10641-014-0290-3 |
| Bokulich N A, Mills D A, 2013. Improved selection of internal transcribed spacer-specific primers enables quantitative, ultra-high-throughput profiling of fungal communities. Appl Environ Microbiol, 79(8): 2519–2526 DOI:10.1128/AEM.03870-12 |
| Brown D S, Burger R, Cole N, et al, 2014. Dietary competition between the alien Asian Musk Shrew (Suncus murinus) and a re-introduced population of Telfair's Skink (Leiolopisma telfairii). Mol Ecol, 23(15): 3695–3705 DOI:10.1111/mec.12445 |
| Callow M E, Callow J A, Pickett-Heaps J D, et al, 1997. Primary adhesion of Enteromorpha (chlorophyta, ulvales) propagules:quantitative settlement studies and video microscopy. J Phycol, 33(6): 938–947 DOI:10.1111/j.0022-3646.1997.00938.x |
| Chapman A R O, 1986. Population and Community Ecology of Seaweeds. In:Blaxter J H S, Southward A J eds. Advances in Marine Biology. London:Academic Press, 1-161 |
| Cheung M K, Au C H, Chu K H, et al, 2010. Composition and genetic diversity of picoeukaryotes in subtropical coastal waters as revealed by 454 pyrosequencing. ISME J, 4(8): 1053–1059 DOI:10.1038/ismej.2010.26 |
| Davenport J, smith R J J W, Packer M, 2000. Mussels Mytilus edulis:significant consumers and destroyers of mesozooplankton. Mar Ecol Prog Ser, 198: 131–137 DOI:10.3354/meps198131 |
| Fletcher R L, Callow M E, 1992. The settlement, attachment and establishment of marine algal spores. British Phycol J, 27(3): 303–329 DOI:10.1080/00071619200650281 |
| Fukumori K, Oi M, Doi H, et al, 2008. Food sources of the pearl oyster in coastal ecosystems of japan:evidence from diet and stable isotope analysis. Estuar, Coastal Shelf Sci, 76(3): 704–709 |
| Galtsoff P S, 1964. The American oyster Crassostrea virginica Gmelin. Fish Bull, 64: 1–480 |
| Hiraoka M, Ohno M, Kawaguchi S, et al, 2004. Crossing test among floating ulva, thalli forming green tide in japan. Hydrobiologia, 512(1-3): 239–245 DOI:10.1023/B:HYDR.0000020332.12641.a2 |
| Jones C G, Lawton J H, Shachak M, 1996. Organisms as Ecosystem Engineers. In:Samson F B, Knopf F eds. Ecosystem Management. New York, USA:Springer Press, 130-147 |
| Lehane C, Davenport J, 2006. A 15-month study of zooplankton ingestion by farmed mussels (Mytilus edulis) in Bantry Bay, Southwest Ireland. Estuar Coastal Shelf Sci, 67(4): 645–652 DOI:10.1016/j.ecss.2005.12.015 |
| Leray M, Yang J Y, Meyer C P, et al, 2013. A new versatile primer set targeting a short fragment of the mitochondrial COI region for metabarcoding metazoan diversity:application for characterizing coral reef fish gut contents. Front Zool, 10(1): 34 DOI:10.1186/1742-9994-10-34 |
| Liu F, Pang S J, Zhao X B, et al, 2012. Quantitative, molecular and growth analyses of Ulva microscopic propagules in the coastal sediment of Jiangsu province where green tides initially occurred. Mar Environ Res, 74: 56–63 DOI:10.1016/j.marenvres.2011.12.004 |
| Liu D Y, Keesing J K, Dong Z J, et al, 2010. Recurrence of the world's largest green-tide in 2009 in Yellow Sea, China:Porphyra yezoensis aquaculture rafts confirmed as nursery for macroalgal blooms. Mar Pollut Bull, 60(9): 1423–1432 DOI:10.1016/j.marpolbul.2010.05.015 |
| Lotsy J P, 1895. The food of the oyster, clam, and ribbed mussel. Rept US Comm Fish, 1893(19): 375 |
| Magoč T, Salzberg S L, 2011. FLASH:fast length adjustment of short reads to improve genome assemblies. Bioinformatics, 27(21): 2957–2963 DOI:10.1093/bioinformatics/btr507 |
| Maloy A P, Culloty S C, Slater J W, 2009. Use of PCR-DGGE to investigate the trophic ecology of marine suspension feeding bivalves. Mar Ecol Prog Ser, 381: 109–118 DOI:10.3354/meps07959 |
| Martin D L, Ross R M, Quetin L B, et al, 2006. Molecular approach (PCR-DGGE) to diet analysis in young Antarctic krill Euphausia superba. Mar Ecol Prog Ser, 319: 155–165 DOI:10.3354/meps319155 |
| Mueller R C, Paula F S, Mirza B S, et al, 2014. Links between plant and fungal communities across a deforestation chronosequence in the Amazon rainforest. ISME J, 8(7): 1548–1550 DOI:10.1038/ismej.2013.253 |
| Riemann L, Alfredsson H, Hansen M M, et al, 2010. Qualitative assessment of the diet of European eel larvae in the Sargasso Sea resolved by DNA barcoding. Biol Lett, 6(6): 819–822 DOI:10.1098/rsbl.2010.0411 |
| Santelices B, Aedo D, Hoffmann A, 2002. Banks of microscopic forms and survival to darkness of propagules and microscopic stages of macroalgae. Revista Chilena de Historia Natural, 75: 547–555 |
| Suzuki N, Hoshino K, Murakami K, et al, 2008. Molecular diet analysis of phyllosoma larvae of the Japanese spiny lobster Panulirus japonicus (Decapoda:Crustacea). Mar Biotechnol, 10(1): 49–55 DOI:10.1007/s10126-007-9038-9 |
| Tel-Zur N, Abbo S, Myslabodski D, et al, 1999. Modified CTAB procedure for DNA isolation from epiphytic cacti of the genera Hylocereus and Selenicereus (Cactaceae). Plant Mol Biol Rep, 17(3): 249–254 DOI:10.1023/A:1007656315275 |
| Wang R J, Wang Y, Zhou J, et al, 2013. Algicidal activity of Ulva pertusa and Ulva prolifera on Prorocentrum donghaiense under laboratory conditions. African J Microbiol Res, 7(34): 4389–4396 |
| Zhang Q C, Liu Q, Yu R C, et al, 2015. Application of a fluorescence in situ hybridization (FISH) method to study green tides in the Yellow Sea. Estuar Coast Shelf Sci, 163: 112–119 DOI:10.1016/j.ecss.2014.09.005 |
| Zhang H Y, Xu Q, Zhao Y, et al, 2016. Sea cucumber (Apostichopus japonicus) eukaryotic food source composition determined by 18S rDNA barcoding. Mar Biol, 163: 153 DOI:10.1007/s00227-016-2931-x |
 2017, Vol. 48
2017, Vol. 48


